近年来随着生活水平的提高,人们对营养与健康的关注度正不断提升,益生菌因具有多种健康功效备受市场关注。益生菌是指一类对人体有益的微生物,这类微生物可以有效调节肠道稳态[1]、维持免疫平衡[2]、促进神经发育[3]、改善代谢过程[4]等,对人体健康产生显著影响。同时,随着益生菌相关研究的深入,食品中益生菌添加的种类也愈加丰富,出现了多菌复配菌粉、多菌协同发酵食品等复杂食品体系,产品形式从传统的发酵食品扩展到婴配食品、保健食品、特医食品等各个领域。
由于益生菌的功能与安全性均具有菌株特异性,因此对含益生菌食品的市场监管,也需要精确到菌株水平;然而,现有的技术标准体系(GB 4789.34—2016《食品安全国家标准 食品微生物学检验 双歧杆菌检验》、GB 4789.35—2023《食品安全国家标准 食品微生物学检验乳酸菌检验》等)尚无法同时实现上述目标,其技术缺陷主要体现在:a)采用平板计数法、流式细胞计数法仅能对食品体系中总活菌量进行测定,无法区分特定菌株的含量;b)通过分离培养纯菌株进行鉴定,检测周期较长;若涉及多菌体系(尤其是同种菌株),单菌株分离纯化难度较大。近年来,有研究挖掘菌株的蛋白分子标签,并利用荧光分子标记与流式细胞计数于复杂生物体系中进行菌株鉴别与定量检测,然而该类方法目前仅能进行菌种水平的检测[5],并且局限于流式细胞计数的可测定样本类型。此外,也有尝试利用测序技术来进行复杂菌相的分析比如利用16S rRNA[6]或GroEL管家基因[7]等靶标的方法,然而此类分子标签种内相似度>97%,且部分菌株>99.9%[8],仅能进行菌种水平的鉴定与相对含量的评估;在此基础上衍生出来的宏基因组拼接法直接对样本中的微生物进行测序并组装全基因组[9],尽管能在一定程度上实现菌株的鉴别和相对定量,且在加入外源内参菌株后可实现绝对定量[10],但其准确度尚未得到广泛验证;同时,该方法检测周期长、技术壁垒高,不适于产业和政府监管层面的推广应用。
当前,亟待建立一套可用于复杂生物体系中特定菌株的鉴定与定量方法体系,以确保产品中的益生菌菌株信息准确可靠。本研究拟以鼠李糖乳酪杆菌MP108为研究对象。该菌株为我国自主研发的、可应用于婴幼儿食品的益生菌之一。本研究拟通过生物信息学手段对大量食品用菌种(益生菌)核酸信息数据开展遗传进化关系分析获取基因分子标签,并以之为靶标,协同多种技术手段建立完善的菌株水平鉴定系统,实现复杂生物样品中鼠李糖乳酪杆菌MP108的鉴别与绝对定量,以期为其他菌株在复杂生物体系中的鉴别和定量提供理论和技术参考。
1 材料与方法
1.1 材料与试剂
鼠李糖乳酪杆菌MP108、77株鼠李糖乳酪杆菌及16种验证菌株保藏于江南大学食品学院生物技术中心菌种保藏库。
胰蛋白胨、酵母提取物,英国OXOID公司;无水葡萄糖、无水乙酸钠、七水合硫酸镁、一水合硫酸锰、柠檬酸氢二铵、三水合磷酸氢二钾、吐温80、甘油、琼脂糖、琼脂、氯化钠、75%(体积分数)乙醇、95%乙醇、1%(体积分数)次氯酸钠溶液,国药集团化学试剂有限公司;2×Taq Master Mix,北京康为世纪生物科技有限公司;qPCR引物,上海生工生物工程有限公司;细菌基因组DNA提取试剂盒,北京天根生化科技有限公司;2×ChamQ SYBR qPCR Master Mix,南京诺唯赞生物科技有限公司;粪便基因组提取试剂盒(Fast DNA SPIN Kit for Feces),美国MP公司。
1.2 仪器与设备
B300-N型电子天平,上海Mettler Toledo公司;MS3型涡旋振荡仪,德国IKA公司;TOMY SX-700型高压蒸汽灭菌锅,日本TOMY公司;DGG-9123A型电热恒温鼓风干燥箱,上海森信实验仪器有限公司;ZHJH-C1115B型超净工作台,上海智诚分析仪器制造有限公司;ULT 1386-3-V31型超低温冰箱、NanoDrop 2000微量分光光度计,美国Thermo Fisher公司;Centrifuge 5424 R型小型台式高速离心机,德国Eppendorf公司;T100型PCR扩增仪、水平琼脂糖凝胶电泳系统、Universal hood I型凝胶成像仪和CFX实时荧光定量仪,美国Bio-Rad公司;高通量组织研磨器,宁波新芝生物科技股份有限公司;Apogee Micro Plus超灵敏纳米流式分析仪,英国Apogee Flow公司。
1.3 实验方法
1.3.1 鼠李糖乳酪杆菌MP108基因组测序及分析
将实验菌株活化后的菌悬液使用TIANamp Bacteria DNA Kit进行总DNA提取,DNA浓度、准确性分别使用NanoDrop和16S rRNA进行检测。检测无误的DNA样品存于-20 ℃备用。将样品送至上海美吉生物有限公司进行denovo测序并质控后进行拼接组装,对拼接完成的基因组序列进行Prokka[11]注释与GC含量分析,以供下一步分析。
1.3.2 鼠李糖乳酪杆菌泛基因组及同源基因分析
基于77株鼠李糖乳酪杆菌及MP108的基因组核酸信息,首先使用FastANI分析其种内平均核苷酸相似度(average nucleotide similarity,ANI),排除ANI<95%的菌株;再使用PGAP v1.2.1[12]进行泛基因组和核心基因组计算,根据Heap’s law构建泛基因组模型,利用基因家族和泛基因组方法将每个基因组的开放阅读框组织成功能基因簇,构建核心基因组模型;并利用核心基因序列构建系统发育树,使用MATTF对核心基因进行比对,再使用FastTree[13]采用最大似然法构建系统发育树以分析鼠李糖乳酪杆菌中不同菌株的系统发育关系。
使用OrthoMCL v2.0.9[14]将MP108与同种乳酪杆菌进行同源基因比对(保持50%同一性;截点参数E-value为1e-4),并利用马尔可夫聚类算法(Markov clustering algorithm, MCL)(膨胀指数为2.50)对具有相同功能的蛋白质家族进行聚类,最终得到MP108的种内同源基因与菌株特有基因。
1.3.3 菌株特异引物设计
基于泛基因组与同源基因分析结果,将MP108备选的特有基因上传至NCBI的RefSeq数据库进行BLAST比对,无同源性或低同源性结果的则为其特有序列。使用Primer Premier 6为筛选得到的特有序列进行引物设计,并上传至NCBI数据库进行Primer-BLAST比对验证,选取无同源性产物结果的引物备用。
1.3.4 引物特异性及qPCR定量能力验证
选取与MP108同种的鼠李糖乳酪杆菌和食品体系中可能存在的其他菌株,使用设计的引物对上述菌株进行PCR扩增验证,并使用琼脂糖凝胶电泳检测DNA是否能够被引物特异性扩增。PCR采用25 μL扩增体系,其中包括2×Taq Master Mix 12.5 μL,上下游引物各1 μL(10 μmol/L),模板DNA 1 μL,补足体系至25 μL的ddH2O。PCR条件为预变性:95 ℃预热3 min;循环程序为95 ℃变性30 s、52 ℃退火15 s、72 ℃延伸30 s,共循环34次;72 ℃延伸10 min;12 ℃保温2 min。
将已知细胞浓度的待测菌株对数期菌悬液按照梯度稀释,使用细菌DNA提取试剂盒提取上述梯度稀释菌液的全基因组,并使用特异引物基于qPCR程序对上述梯度稀释菌液的基因组样本进行检测,验证含梯度浓度待测菌株的qPCR-Ct值与其浓度之间的线性关系以建立菌液浓度与qPCR-Ct值的标准曲线。qPCR采用20 μL扩增体系,包括2×ChamQ SYBR qPCR Master Mix 10 μL、上下游引物各1 μL(10 μmol/L)、模板DNA1 μL,补足体系至20 μL的ddH2O。qPCR程序为:95 ℃变性3 min;循环程序为95 ℃变性10 s,在引物特异性温度下退火15 s,72 ℃延伸15 s,共循环40次;在65 ℃和95 ℃之间进行熔融曲线分析。
2 结果与分析
2.1 鼠李糖乳酪杆菌MP108全基因组分析
基于Prokka注释结果,使用Proksee[15]软件分析MP108全基因组信息(图1-A)。结果表明,MP108基因组全长为2 896 747 bp,总GC含量为46.76%,共编码2 708个开放阅读框,与NCBI数据库中已鉴定的鼠李糖乳酪杆菌基本一致(基因全长为2.9 Mbp,总GC含量为46%左右,存在约2 650~2 750个编码基因),符合鼠李糖乳酪杆菌的全基因组特征[16];同时,对MP108与经16S rRNA鉴定的77株鼠李糖乳酪杆菌进行ANI分析,结果如图1-B所示,鼠李糖乳酪杆菌种内出现4个聚类群,聚类群内的菌株核苷酸水平上具有高度相似性,无法通过ANI实现菌株区分。然而,MP108与种内77株鼠李糖乳酪杆菌的ANI仅为97.15%~97.98%,并未与其余鼠李糖乳酪杆菌出现聚类,说明MP108为鼠李糖乳酪杆菌[8]但在核苷酸水平上其与同种菌株并未高度相似,基于此可以推测MP108具有潜在的种内特异性核酸序列。

A-鼠李糖乳酪杆菌MP108全基因组编码基因及GC含量分析;B-鼠李糖乳酪杆菌种内ANI分析
图1 鼠李糖乳酪杆菌MP108全基因组分析
Fig.1 Whole-genome analysis of L.rhamnosus MP108
2.2 鼠李糖乳酪杆菌泛基因组分析
基于78株鼠李糖乳酪杆菌菌株全基因组预测的蛋白序列进行泛基因组分析并构建基于菌种核心基因的发育进化树。结果如图2所示,鼠李糖乳酪杆菌泛基因组总基因数为9 031个,其中核心基因数为1 751个,且随着菌株数目的增加逐渐趋于稳定;然而,随着菌株数目的增加不断有新的基因被纳入鼠李糖乳酪杆菌泛基因组中,呈现出典型的开放型泛基因组特征,展现了种内的基因多样性和潜在的代谢多样性[17]。
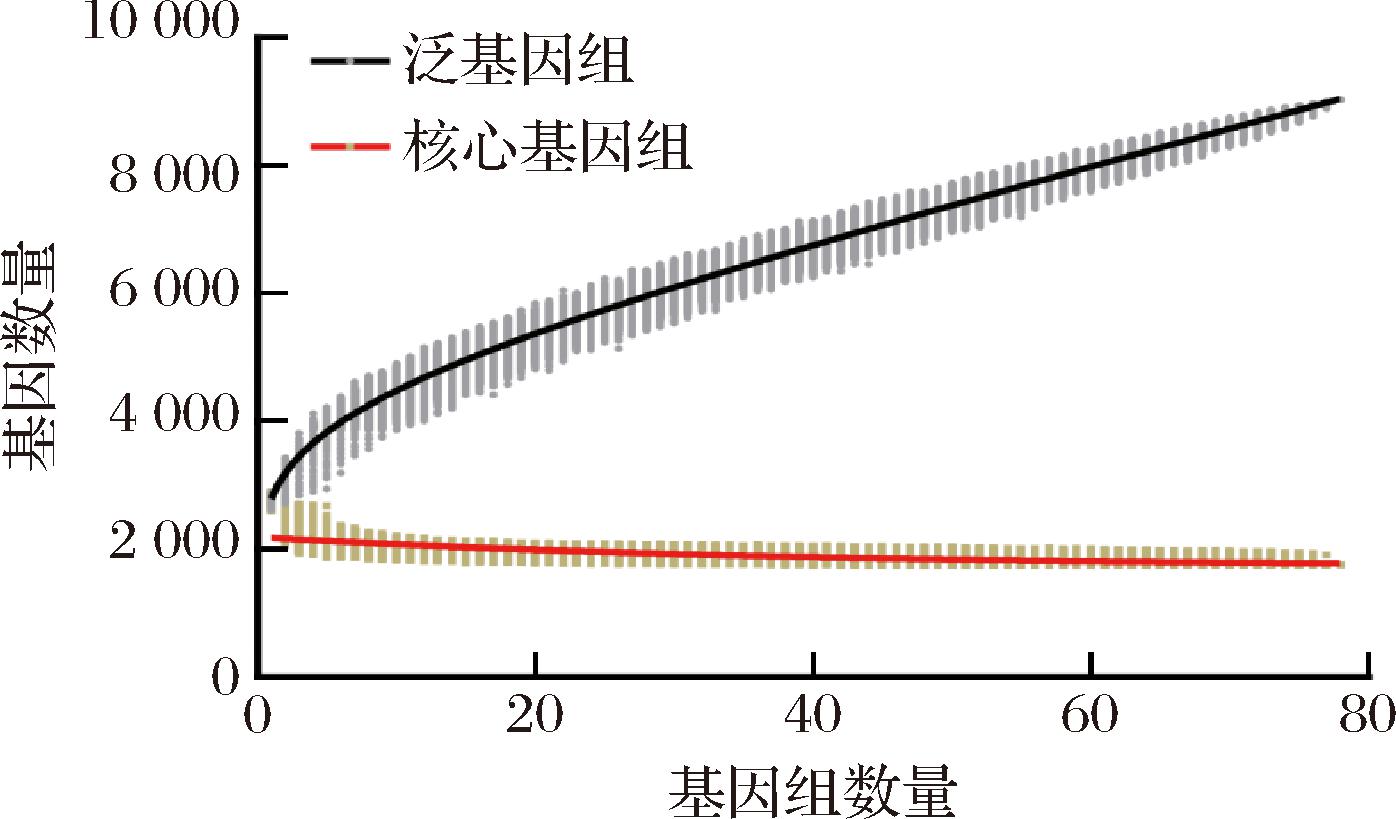
图2 鼠李糖乳酪杆菌泛基因组分析
Fig.2 Pangenomic analysis of L.rhamnosus
其次,基于78株鼠李糖乳酪杆菌泛基因组分析得到的1 751个核心基因,采用极大似然值法进行系统发育分析,从进化同源性角度分析鼠李糖乳酪杆菌MP108与其余鼠李糖乳酪杆菌的亲缘关系。如图3所示,MP108并未与其他鼠李糖乳酪杆菌聚类分簇,具有进化出菌株特异基因的潜力。综上所述,鼠李糖乳酪杆菌具有种内基因多样性,且MP108与本研究中分析的鼠李糖乳酪杆菌进化距离较远,说明MP108具有潜在的菌株特异基因。
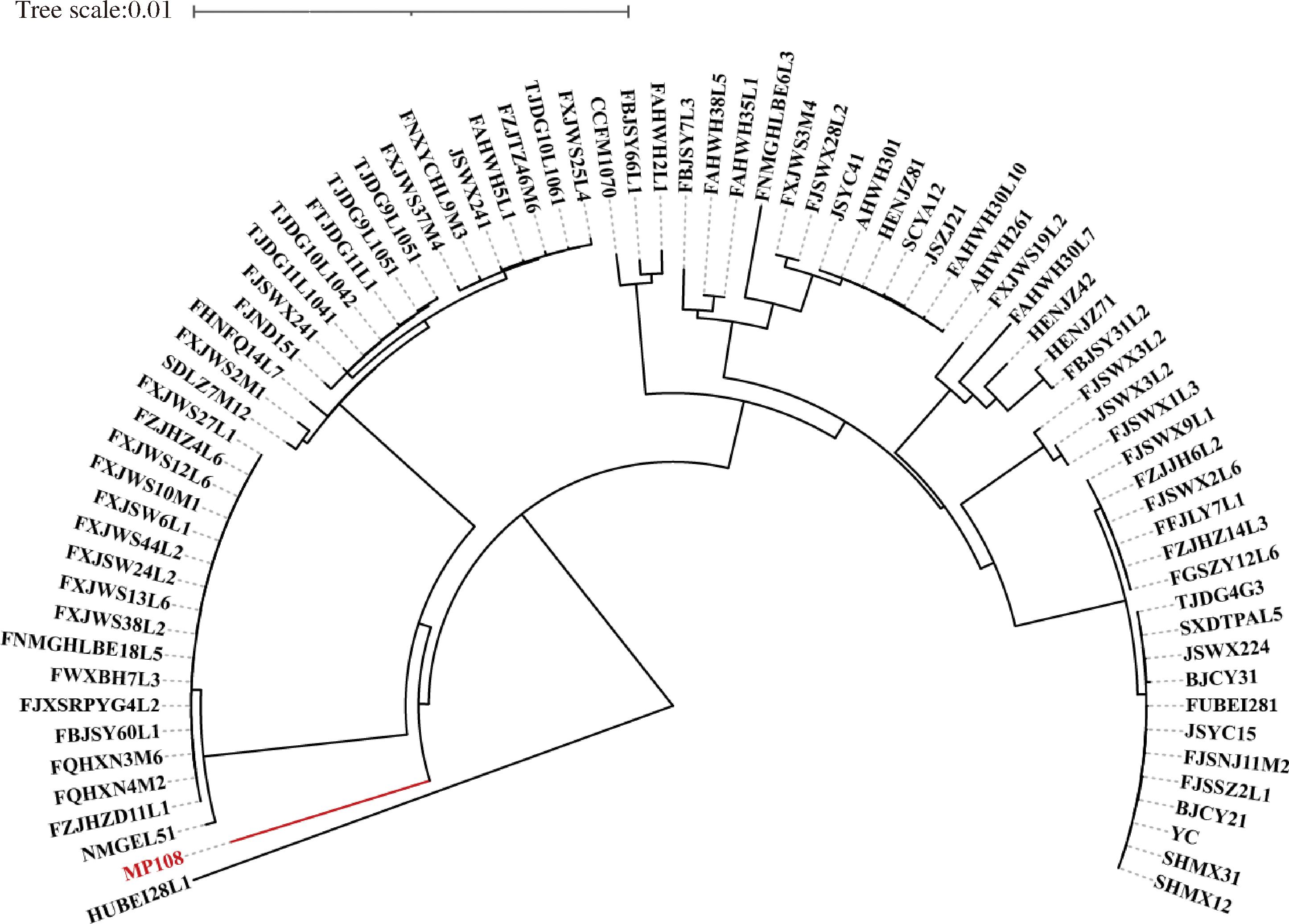
图3 78株鼠李糖乳酪杆菌全基因组系统发育树
Fig.3 Whole-genome phylogenetic tree of 78 L.rhamnosus strains
2.3 MP108特异序列发掘及引物验证
通过2.1节与2.2节推断MP108具有潜在的特异基因,因此基于MP108的全基因组信息挖掘菌株的特异核酸分子标签,并以之为靶标设计特异性引物。同源聚类结果如图4-B所示,这78株鼠李糖乳酪杆菌拥有1 821段同源序列,每个独立菌株均具有3~60不等的菌株特有序列,与2.2节中泛基因组分析结果相符。MP108相较于其余77株鼠李糖乳酪杆菌拥有48个特异基因(核酸序列),均为潜在的特异性引物设计靶点;已有研究采用菌株特异性核酸分子标签为靶标鉴定与定量益生菌菌株,例如BARRATT等[18]分析目标长双歧杆菌婴儿亚种EVC001与同时分离出的菌株的全基因组差异,以实现EVC001于婴儿肠道内的精确定量;然而,此类研究仅囊括了少量菌株的全基因组,并未考虑到纳入分析的菌株增加后靶标分子标签是否仍具有菌株特异性。因此,将具有潜在特异性的48个基因与NCBI数据库中同种菌株的公开基因组进行核酸序列比对,根据序列相似度判断核酸序列的特异性。最终,确定了1段序列为MP108核酸分子标签并以之为模板设计特异性引物(表1)。
表1 鼠李糖乳酪杆菌MP108菌株特异性引物信息
Table 1 Information for strain-specific primers of
L.rhamnosus MP108
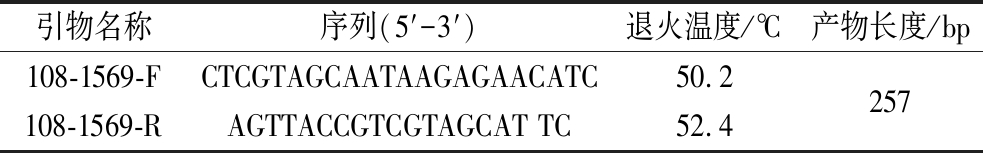
引物名称序列(5′-3′)退火温度/℃产物长度/bp108-1569-F108-1569-RCTCGTAGCAATAAGAGAACATCAGTTACCGTCGTAGCAT TC50.252.4257
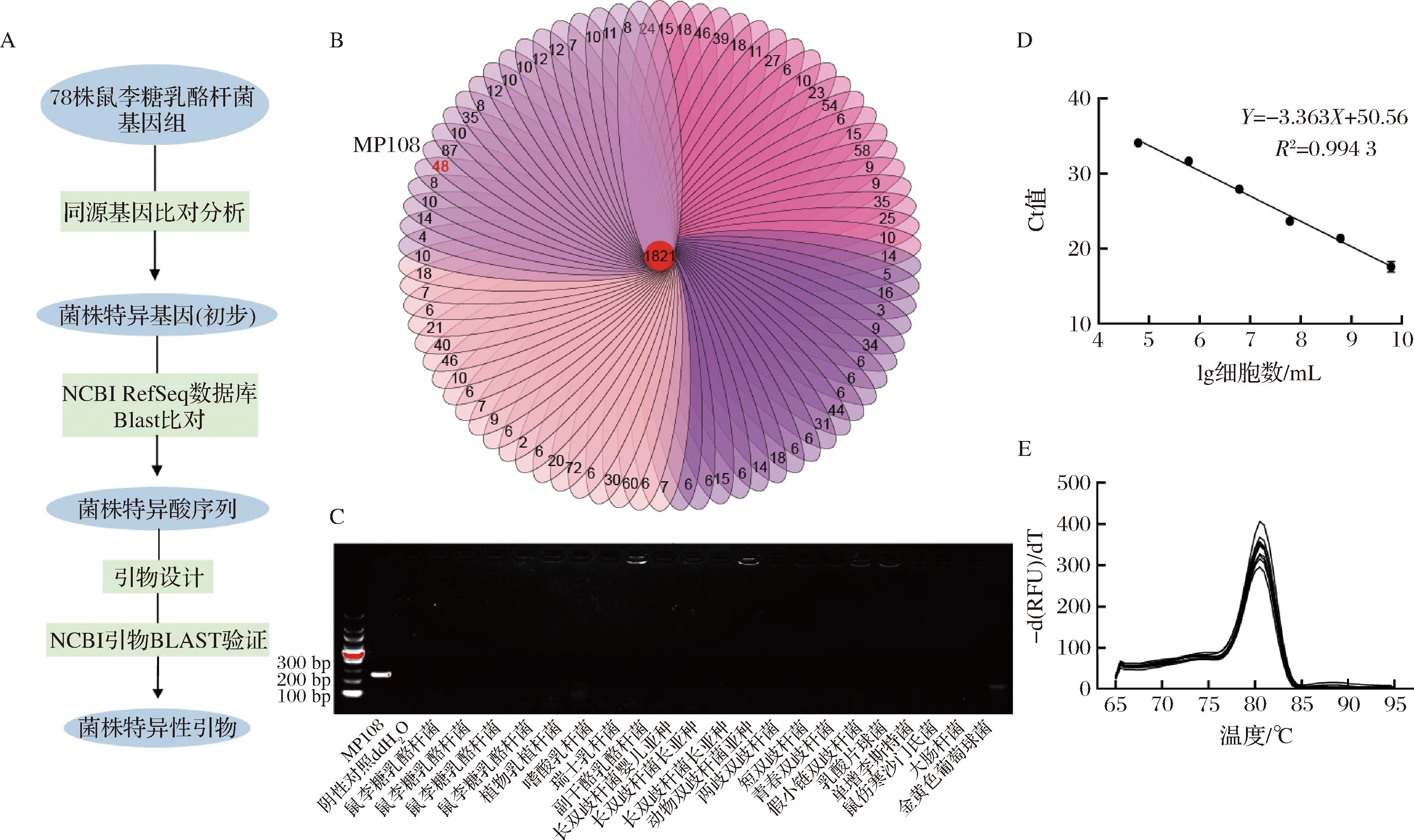
A-菌株特异性引物设计流程图;B-78株鼠李糖乳酪杆菌同源基因聚类分析;C-菌株特异性引物PCR-琼脂糖凝胶验证;D-MP108纯培养物DNA定量标准曲线;E-MP108纯培养物DNA qPCR产物熔解曲线分析
图4 菌株特异性引物设计及验证
Fig.4 Design and verification of strain-specific primers
其次,对该菌株特异性引物进行特异性验证,以MP108菌株为阳性对照,ddH2O为阴性对照,利用多株鼠李糖乳酪杆菌、多种乳杆菌与双歧杆菌以及常见致病菌对MP108菌株水平特异性引物进行PCR与琼脂糖凝胶电泳验证(详细菌种见图4-C)。验证结果如图4-C所示,仅MP108所在泳道出现了亮色条带且该条带位于200~300 bp,这与特异性引物预期设计的产物长度相符合,且阴性对照与其余泳道的菌株未出现亮色条带,这证明该引物于退火温度为52 ℃时具有针对鼠李糖乳酪杆菌MP108的特异性。
最后,为防止菌泥中出现过多死菌干扰后续qPCR定量结果,于指数期末期(8~9 h)收集MP108菌泥,重悬并梯度稀释后进行平板计数。对上述已知活菌数的梯度稀释液进行qPCR定量,以模板细胞数量为横坐标,Ct值为纵坐标建立Ct值-细胞浓度标准曲线。结果如图4-D所示,qPCR定量标准曲线的拟合方程为Y=-3.363X+50.56,于105~1010细胞数/mL区间内表现出很好的线性关系(R2>0.99),且qPCR产物的扩增熔解曲线(图4-E)在80.5 ℃时达到最高峰,且峰型单一,无杂条带干扰,证明该引物具有于菌株水平定量鼠李糖乳酪杆菌MP108的潜力。
2.4 复杂生物体系中菌株特异性引物定量能力验证
本方法以实现食品体系中菌株水平检测与定量为目标,考虑到含益生菌食品类型与菌种的多样化,本研究采用组分更加复杂、微生物更加多样的人类粪便为检测背景,为排除个体粪便样品的差异对检测结果的影响,本研究将20~30岁男女各2人的粪便样品混样后用作检测背景;此外,相较于不同食品类型的DNA提取方法,粪便DNA提取有广泛认可且便捷的方法,能够尽量避免DNA提取过程中的损耗使结果更为准确;同时,考虑到益生菌具有剂量依赖性,即一定数量的益生菌才能够发挥功效,且市场上的益生菌产品的益生菌含量均不低于107 CFU/mL,本研究对于该方法检出限的分析限制于104 CFU/mL。
为了进一步验证该引物对MP108鉴定与定量的潜力,对已知细胞浓度为109 CFU/mL的菌液进行梯度稀释,并向不含MP108的空白人类粪便样品中分别加入该系列梯度稀释液。提取上述样品的DNA后,使用菌株特异引物对上述所得100 μL DNA溶液中的1 μL进行qPCR定量,根据结果的线性判断其检出限(图5)。
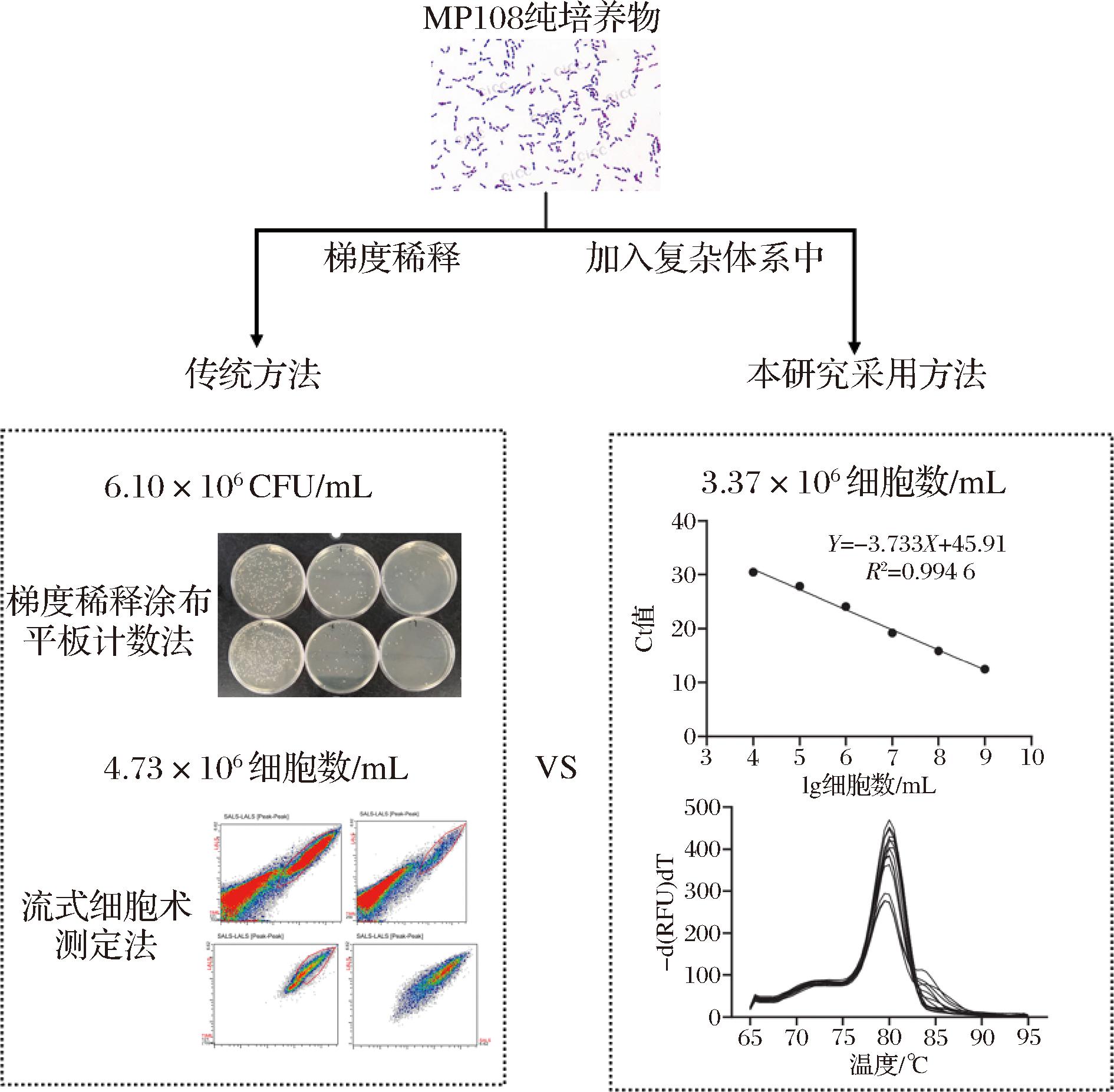
图5 MP108绝对定量检测及检出限分析
Fig.5 Absolute quantitative detection and detection limit analysis of MP108
如图5所示,以模板细胞数量为横坐标,Ct值为纵坐标建立Ct值-细胞浓度标准曲线,qPCR定量标准曲线拟合方程为Y=-3.733X+5.91,该特异性引物于104~1010细胞数/mL区间内具有良好的线性关系(R2>0.99),且qPCR产物的扩增熔解曲线(图5-B)在80 ℃时达到最高峰,且峰型单一,无杂条带干扰。因此,该标准曲线的检出限低于104细胞数/mL,其灵敏度为10细胞数/μL DNA溶液;该结果与XIAO等[19]采用的长双歧杆菌菌株定量方法的灵敏度相似,但此研究在进行标准曲线构建时并未排除死菌对qPCR结果的干扰。上述100 μL DNA溶液由0.1 g粪便的基因组及已知量的MP108构成,因此,可以证明该特异性引物于复杂微生物体系中检测MP108的检出限为104细胞数/g。
为验证其定量检测准确度,本研究选取2组样品(105与106两个数量级)分别采用梯度稀释平板计数法与流式细胞计数法进行纯培养物定量检测,并加至复杂生物体系(粪便样品)中进行qPCR定量检测。结果如图6所示,经平板计数后得出的细胞数对数值分别为6.78、5.78,纳米流式细胞计数的细胞数对数值为6.66、5.68,特异性引物qPCR定量的细胞数对数值分别为6.64、5.57,显著性差异分析结果均未表现出显著差异(P>0.05),能够实现复杂食品体系中益生菌数量级的绝对定量。
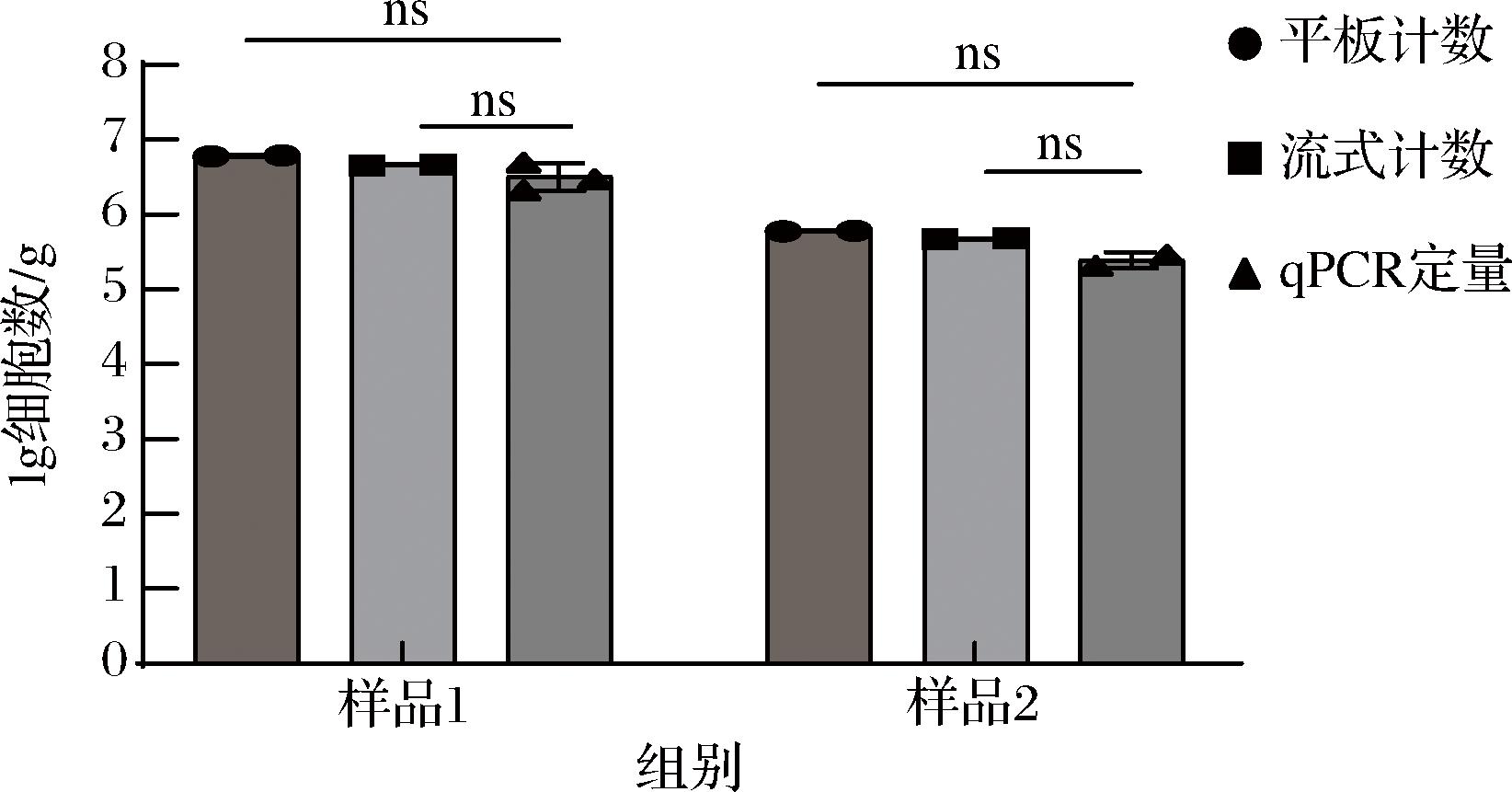
图6 菌株特异性引物定量准确度分析
Fig.6 Quantitative accuracy analysis of strain-specific primers
注:ns表示无显著性差异(P>0.05)。
3 结论
本研究构建了一种复杂生物体系中菌株水平鉴别与定量检测的方法,能够实现复杂食品体系中目标菌株的鉴别与绝对定量。本方法在复杂体系中的定量准确度与基于纯培养菌株的平板计数法、流式细胞计数法对照值无显著性差异,能够实现数量级水平的精确检测;并且其检出限为104细胞数/g,灵敏度为10 copies/μL DNA溶液;然而,本研究所采用的qPCR技术的检出限偏高,无法满足食品体系中某些致病菌的低检出限需求,为满足此需求须将株特异性分子标签与更为精准的鉴定方法与仪器结合。考虑到益生菌食品中的益生菌含量均大于107 CFU/mL(g),且qPCR定量技术操作简便、成本较低,因此本方法足以适用于大部分食品体系中益生菌的定量检测。综上,本研究基于鼠李糖乳酪杆菌MP108建立了一套菌株鉴别与定量检测体系,有望为益生菌食品检测细致化、精确化的技术升级提供理论参考。
[1] MUKHERJI A, KOBIITA A, YE T, et al.Homeostasis in intestinal epithelium is orchestrated by the circadian clock and microbiota cues transduced by TLRs [J].Cell, 2013, 153(4):812-827.
[2] HONDA K, LITTMAN D R.The microbiota in adaptive immune homeostasis and disease[J].Nature, 2016, 535(7610):75-84.
[3] AHRENS A P, HYÖTYL INEN T, PETRONE J R, et al.Infant microbes and metabolites point to childhood neurodevelopmental disorders[J].Cell, 2024, 187(8):1853-1873:e15.
INEN T, PETRONE J R, et al.Infant microbes and metabolites point to childhood neurodevelopmental disorders[J].Cell, 2024, 187(8):1853-1873:e15.
[4] TAN S, SANTOLAYA J L, WRIGHT T F, et al.Interaction between the gut microbiota and colonic enteroendocrine cells regulates host metabolism[J].Nature Metabolism, 2024, 6(6):1076-1091.
[5] JERYE K, LÜKEN H, STEFFEN A, et al.Activity-based protein profiling identifies protein disulfide-isomerases as target proteins of the volatile salinilactones[J].Advanced Science, 2024, 11(18):e2309515.
[6] JOHNSON J S, SPAKOWICZ D J, HONG B Y, et al.Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J].Nature Communications, 2019, 10(1):5029.
[7] 胡陆军. 复杂样品中双歧杆菌的高通量检测和菌种高效分离方法的研究[D]. 无锡: 江南大学, 2017.
HU L J. Study on Qualcomm detection of Bifidobacterium in complex samples and efficient separation of strains[D]. Wuxi: Jiangnan University, 2017.
[8] JAIN C, RODRIGUEZ-R L M, PHILLIPPY A M, et al.High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries[J].Nature Communications, 2018, 9(1):5114.
[9] KIRSCH J M, HRYCKOWIAN A J, DUERKOP B A.A metagenomics pipeline reveals insertion sequence-driven evolution of the microbiota[J].Cell Host &Microbe, 2024, 32(5):739-754.e4.
[10] ST MMLER F, GL
MMLER F, GL SNER J, HIERGEIST A, et al.Adjusting microbiome profiles for differences in microbial load by spike-in bacteria[J].Microbiome, 2016, 4(1):28.
SNER J, HIERGEIST A, et al.Adjusting microbiome profiles for differences in microbial load by spike-in bacteria[J].Microbiome, 2016, 4(1):28.
[11] SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics, 2014, 30(14):2068-2069.
[12] ZHAO Y B, WU J Y, YANG J H, et al.PGAP:Pan-genomes analysis pipeline[J].Bioinformatics, 2012, 28(3):416-418.
[13] PRICE M N, DEHAL P S, ARKIN A P.FastTree 2:Approximately maximum-likelihood trees for large alignments[J].PLoS One, 2010, 5(3):e9490.
[14] LI L, STOECKERT C JJr, ROOS D S.OrthoMCL:Identification of ortholog groups for eukaryotic genomes[J].Genome Research, 2003, 13(9):2178-2189.
[15] GRANT J R, ENNS E, MARINIER E, et al.Proksee:In-depth characterization and visualization of bacterial genomes[J].Nucleic Acids Research, 2023, 51(W1):W484-W492.
[16] ZHENG J S, WITTOUCK S, SALVETTI E, et al.A taxonomic note on the genus Lactobacillus:Description of 23 novel Genera, emended description of the genus Lactobacillus beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4):2782-2858.
[17] HORESH G, TAYLOR-BROWN A, MCGIMPSEY S, et al.Different evolutionary trends form the twilight zone of the bacterial pan-genome[J].Microbial Genomics, 2021, 7(9):000670.
[18] BARRATT M J, NUZHAT S, AHSANK, et al. Bifidobacterium infantis treatment promotes weight gain in Bangladeshi infants with severe acute malnutrition[J].Science Translational Medicine, 2022, 14(640):eabk1107.
[19] XIAO Y, WANG C, ZHAO J X, et al.Quantitative detection of Bifidobacterium longum strains in feces using strain-specific primers[J].Microorganisms, 2021, 9(6):1159.