食源性致病菌是指可以通过食物或饮水感染人体的细菌,单核细胞增生李斯特氏菌(单增李斯特菌,Listeria monocytogenes)、大肠杆菌(Escherichia coli)、金黄色葡萄球菌、沙门氏菌(Salmonella)、蜡样芽孢杆菌、空肠弯曲杆菌等,进入人体后能导致胃肠炎、败血症、脑膜炎等食源性疾病。食源性致病菌与宿主肠道之间的相互作用是引发食源性疾病的关键步骤。宿主中存在不同的自然机制,如胃酸度、肠蠕动、肠道酶对细菌的破坏、胆汁和免疫球蛋白A的释放等可以防止入侵细菌在肠道上定植。肠道微生物群、黏液层、肠上皮细胞单层共同形成的物理屏障、以及肠道免疫系统提供的内层化学屏障,能防止细菌扩散到宿主组织内部。肠道与食源性致病菌的互作研究一直是研究热点和重点,例如食源性致病菌对肠道不同部位的侵袭和趋向规律,食源性致病菌的免疫逃逸机制,不同食源性致病菌菌株与肠道微生物之间的互作规律,肠道内部环境对食源性致病菌的响应和影响规律等,这些相互作用机制仍需进一步阐明。
动物模型是研究肠道与致病菌的主要方法,但由于种源差异,动物和人类的肠道生理结构和生化环境不同,在重现宿主与病原体之间的相互作用方面存在许多缺陷,此外还具有伦理质疑。体外人体细胞模型,是更为简便的研究宿主肠道与病原体相互作用的工具。其中,体外2D细胞培养模型被广泛应用,具有低成本、易用性和高通量的优点,但不能准确地再现人类生理或病理特征,无法复制原生组织中存在的细胞-细胞和细胞-环境相互作用。相比之下,三维(3D)细胞培养模型显示出更多生理相关的基因和蛋白质表达谱,能提供更真实的人体组织或器官的形态、功能和微环境特征。3D培养模型有很多形式,包括简单的多细胞球体或更复杂的类器官、器官芯片和3D生物打印技术,用于不同的应用领域。目前关于肠道3D体外模型的研究多综述了体外肠上皮细胞的新范式、肠道细菌感染、炎症性肠病中微生物组-宿主相互作用以及肠道疾病的药物筛选与治疗等,缺乏对肠道宿主-食源性致病菌相互作用、模型在食源性致病菌应用的综述。而体外3D培养模型为深入解析宿主-致病菌相互作用提供了一个强大的体外平台。
因此,本文以食源性致病菌的研究为切入点,首先介绍肠道的基本构造以及肠道不同层次结构中的细胞亚型特点;其次,分析并讨论了代表性食源性致病菌黏附入侵肠道时与宿主之间的相互作用。最后,重点综述并讨论了4种主要3D模型的构建原理,以及这些模型在研究肠道和与食源性致病菌相互作用中的应用、优点和局限性。本文将为食源性致病菌肠道感染机制等方面的研究中最优模型的选择提供科学参考。
1 肠道的基本生理构造和细胞亚型特征
肠道是人体重要的消化器官,同时是抵御外界环境的主要屏障。肠道、肠道微生物、免疫系统、肠道神经系统和其他器官之间形成了一个复杂相互关系网,通过多种途径的相互影响,维持肠道健康和免疫平衡,抵御外界食源性致病菌等病原体的侵入[1]。肠道主要由负责消化吸收的小肠(十二指肠、空肠和回肠)和负责排泄、部分吸收功能的大肠(盲肠、结肠、直肠和肛门)2部分组成。肠道具有4层组织层(图1-a),从内到外分别是黏膜层(肠上皮细胞层、固有层和黏膜肌层)、黏膜下层、肌肉层(内环肌层、肠肌从和外纵肌层)和浆膜[3]。小肠黏膜层的上皮层由一层复杂类型的肠细胞群组成,形成细胞间连接的天然屏障,将内部和外部环境分开;该屏障选择性地吸收营养物质和电解质,同时分泌黏液和激素,防止潜在有害物质(抗原、毒素和微生物副产物)进入体内[4]。具有代表性的是小肠上皮细胞(图1-b),与其他上皮细胞相比,结构简单而规则,细胞紧密排列形成手指状的突起,称为小肠绒毛。绒毛结构增加了肠道表面积,从而提高了对营养物质的吸收和消化能力,并起到防御病原菌的作用[5]。
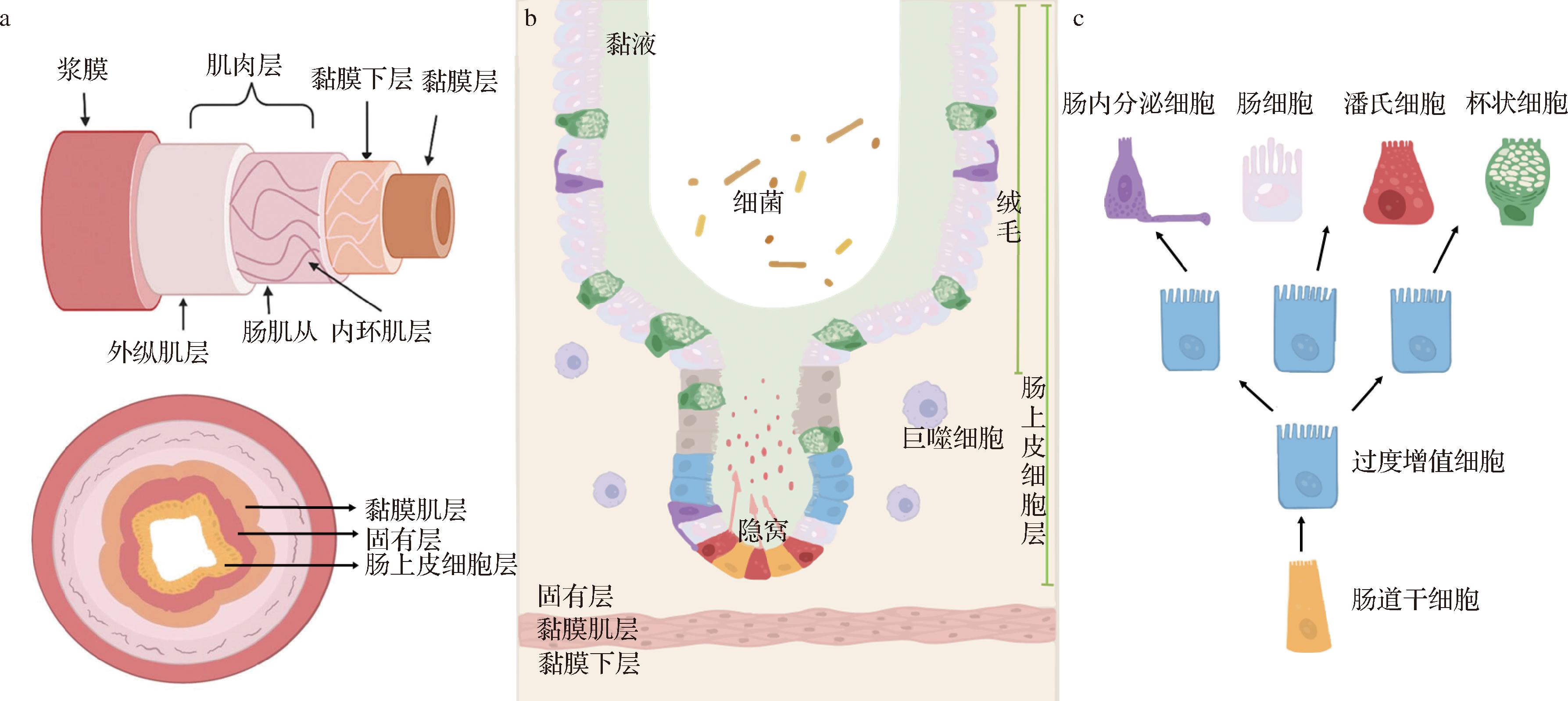
a-肠道组织层;b-肠上皮细胞层;c-肠道干细胞分化
图1 肠道结构示意图[2]
Fig.1 Schematic diagram of the intestinal structure[2]
注:根据文献[2]改绘。
小肠黏膜层下凹部分称为隐窝,位于绒毛之间,富含肠干细胞。肠干细胞具有自我更新和分化的能力,可以分化为不同类型的成熟上皮细胞(图1-c),包括吸收性肠细胞、分泌酶的肠内分泌细胞、分泌黏液的杯状细胞和产生溶菌酶的潘氏细胞[6]。上皮细胞是人体内具有最强再生能力的组织,上皮细胞借助干细胞每3~5 d完成一次更新,以此维持上皮的结构完整性和肠道稳态。杯状细胞,占小肠上皮的10%~15%和结肠上皮的50%,主要分泌产物是高糖基化的黏蛋白,将其分泌到肠腔中,并与水结合产生黏液以保护黏膜,黏液也可以阻断上皮细胞与细菌等微生物之间的接触[7]。潘氏细胞占肠上皮细胞群的一小部分,可以主动感知细菌的存在,并通过释放溶菌酶和抗菌肽调节细菌共生[8]。肠内分泌细胞占肠道上皮细胞群总数的1%,散布在肠道的整个长度,包括隐窝和绒毛,会通过检测肠腔内代谢物、消化产物等变化,从而分泌各种激素,如胃动素、胆囊收缩素,以调节肠道运动或调节营养物质的代谢和吸收[9]。成熟细胞在末梢分化后约3 d到达绒毛尖端,发生凋亡,最终脱落进入肠腔。肠道黏膜层的固有层含有丰富的间充质细胞、巨噬细胞、免疫细胞和胶原蛋白,在修复肠道损伤方面也起着重要作用。肠道黏膜层的黏膜肌层的作用是产生躁动和蠕动,为黏膜提供支撑和活动性[10]。黏膜下层由一层结缔组织组成,将肌肉层与黏膜层连接起来,黏膜下层有着丰富的血管和神经。肌肉层由2层组成,即内环肌层和外纵肌层,中间是肠肌丛,控制蠕动。2个肌肉层相互协调向前移动食物,为食物的消化、团块向下运动、胃部搅动以及摄入的食物通过肠道的运动以及通过直肠排出废物提供机械力[3]。
由此可知,人体肠道结构和生理功能非常复杂、肠道细胞类型种类多样。为了充分研究食源性致病菌与肠道的互作机制,在体外构建的细胞模型中,需要尽可能包含多种肠道细胞亚型,以获得人体肠道生理结构层和微环境,这是体外模型构建成功与否的关键。传统的2D细胞模型中一般只含有1~2种细胞亚型,而最新的肠道类器官中可含有多种的肠上皮细胞亚型。3D打印技术则可以人工加入不同细胞亚型,理论上可以构建任意肠道结构体系。这些3D模型在模拟人体肠道生理结构和微环境方面取得了前所未有的进展。
2 食源性致病菌与肠道互作机制
肠道微生物群是防止致病菌入侵的第一道屏障,占据所有的营养生态位,形成优先效应,比致病菌更有效地获取营养,从而阻碍致病菌建立自己的生态位,在排除入侵细菌、防止致病菌定植、过度生长方面起关键作用[11]。此外,肠道微生物群也可以通过产生特定的代谢物,改变宿主的环境条件,从而直接损害病原体的生长或毒力。比如梭状芽胞杆菌可以将糖从复合碳水化合物转化为有机酸和短链脂肪酸,而短链脂肪酸通过细菌膜扩散和降低细胞内pH值而具有直接的抗菌活性[12]。肠道微生物群还能够分泌具有广泛生物活性的小分子,如细菌素的抗菌肽,选择性地杀死、抑制或调节竞争细菌的行为。如苏云金拟杆菌能够分泌一种菌素(苏云金杆菌CD),该菌素可以直接靶向形成孢子的芽孢杆菌和梭状芽胞杆菌[13]。
肠上皮的官腔表面的黏液层将上皮细胞与微生物群分开,使其免受密集的细菌群的影响,在宿主-致病菌相互作用和肠道稳态维持中起重要作用。GARC A-D
A-D AZ等[14]构建了一个体外模型,将分泌的黏液层和上皮屏障包含在3D绒毛状水凝胶支架中,以此增强黏液的分泌,研究发现黏液层可以防止与克罗恩病相关的致病性大肠杆菌LF82的黏附入侵、减轻感染时宿主细胞的反应。小肠上皮细胞群(即肠上皮细胞、杯状细胞和潘氏细胞等)的细胞之间的紧密连接构成的物理屏障将顶端与基底外侧隔室完全分开,以减少组织损伤和炎症的出现[15],因此,不同细胞亚型与食源性致病菌之间相互作用也是重要的研究方向。
AZ等[14]构建了一个体外模型,将分泌的黏液层和上皮屏障包含在3D绒毛状水凝胶支架中,以此增强黏液的分泌,研究发现黏液层可以防止与克罗恩病相关的致病性大肠杆菌LF82的黏附入侵、减轻感染时宿主细胞的反应。小肠上皮细胞群(即肠上皮细胞、杯状细胞和潘氏细胞等)的细胞之间的紧密连接构成的物理屏障将顶端与基底外侧隔室完全分开,以减少组织损伤和炎症的出现[15],因此,不同细胞亚型与食源性致病菌之间相互作用也是重要的研究方向。
肠道相关淋巴组织是肠道中重要的免疫系统,包括Peyer斑块、存在于固有层中的浆细胞和淋巴细胞等。当屏障完整性被破坏,肠道稳态失调时,该组织可通过内分泌物、肠道微生物组、免疫系统以及肠道干细胞之间的微妙相互作用实现维持肠道稳态。入侵的病原体一旦通过肠上皮层屏障,黏膜先天免疫细胞(如巨噬细胞、树突状细胞)会对其识别、吞噬和消除,并在健康状态下被清除[16]。若无法根除入侵的病原体,导致细菌的全身易位时,肠上皮细胞会对入侵的病原体产生先天免疫反应,这种免疫应答会产生和分泌促炎细胞因子、Ⅰ型和Ⅲ型干扰素(interferon,IFN),在其刺激下会产生由T细胞、自然杀伤细胞或抗原呈递细胞(antigen presenting cell,APC)分泌的细胞因子,从而减轻全身感染[17]。
3 常见食源性致病菌与肠道互作机制
3.1 单增李斯特菌
L.monocytogenes是唯一能引起人类李斯特菌病的致病菌,定植于肠道中,穿过肠道屏障后,再通过血液传播到靶器官,可导致易感人群出现败血症、脑膜炎、孕妇流产死产、新生儿死亡等严重后果。L.monocytogenes面对肠道中胃酸、高渗透压、胆盐等多种环境压力时,会通过应激反应因子(alternative sigma factor,SigB)独立调节机制诱导数百个与一般应激反应(general stress response,GSR)相关的基因对遇到的肠道环境和宿主做出应激反应[18]。如面对酸性环境,L.monocytogenes会通过增加排斥系统基因的转录来提高细胞内的pH值,如谷氨酸脱羧酶和α-酮戊二酸脱氢酶等排阻系统的转录[19]。在肠道中与宿主肠道微生物群竞争生存时,肠道菌群通过产生抗菌肽的细菌素来抑制细菌定植;而L.monocytogenes会通过诱导乙醇胺代谢等次级代谢途径或产生李斯特菌溶血素S(listeriolysin S,LLS)调节肠道微生物群,帮助李斯特菌跨越肠道屏障[20]。L.monocytogenes侵入肠道上皮屏障主要有3种途径:通过M细胞介导的吞噬作用进入Peyer斑块后,感染巨噬细胞和树突状细胞;转胞吞作用:通过侵袭杯状细胞,在较小程度上侵袭位于绒毛顶端的肠细胞;通过LAP-Hsp60通路以及InlA-E-cadherin相互作用,使L.monocytogenes从肠腔转移到固有层[21]。
3.2 肠出血性大肠杆菌
肠出血性大肠杆菌(Enterohemorrhagic E.coli,EHEC)是一种危害严重的食源性致病菌,会引起人类腹泻、出血性结肠炎和溶血性尿毒症综合征等。肠出血性大肠杆菌感染机制的2个主要特征是志贺毒素的产生和在肠上皮细胞上形成的附着和消失(attaching and effacing,AE)病变,志贺毒素是导致溶血性尿毒症综合症相关临床症状的原因[22]。AE病变特征是微绒毛损伤、附着细菌下方的肌动蛋白“基座”的形成以及细菌与宿主细胞紧密附着[22]。EHEC通过Ⅲ型分泌系统(type III secretion system,T3SS)将转移的整合素受体(translocated intimin receptor,Tir)输送到宿主上皮细胞膜内,并作为EHEC膜上的吸附蛋白的受体,这种Tir/内膜连接使得EHEC能够附着在宿主细胞上[23]。Tir还与宿主细胞骨架结合,并诱导肌动蛋白聚合,最终形成细菌下方肌动蛋白的基座,进一步导致上皮微绒毛被消除,肠道远端组织的细胞损伤和炎症反应[24]。
3.3 沙门氏菌
沙门氏菌是最常见的食源性致病菌之一,主要污染鸡蛋、猪肉、水产品等,可引起胃肠炎和伤寒等。沙门氏菌对肠道宿主的防御机制(如胃酸)作出反应并存活,入侵肠上皮屏障引发炎症或穿透肠道屏障并扩散到整个网状内皮系统引发伤寒[25]。沙门氏菌入侵肠上皮屏障主要是通过沙门菌毒力岛1(Salmonella pathogenicity islands 1,SPI1)和沙门菌毒力岛2(Salmonella pathogenicity islands 2,SPI2),分别编码不同类型的T3SS[26]。SPI1编码的T3SS将早期的效应蛋白转运到上皮细胞内,激活宿主细胞GTP酶,从而刺激宿主细胞的肌动蛋白细胞骨架重排,导致沙门氏菌进入上皮细胞。宿主细胞GTP酶的激活促进宿主产生炎症反应,沙门氏菌进入细胞后,宿主细胞的肌动蛋白细胞骨架会恢复正常形态,炎症反应也会被减轻。细胞内的沙门氏菌能够抵抗宿主中固有免疫反应的杀伤作用,并存在于一种特殊的囊泡中,被称为“含沙门菌的囊泡”(Salmonella-containing vacuole,SCV)[27]。SPI2编码的T3SS将效应蛋白转运到真核细胞的细胞质中,通过维持SCV膜和SCV在细胞核附近的定位等帮助沙门氏菌在细胞内存活和增殖[27]。沙门氏菌穿过肠道上皮细胞后,巨噬细胞将其摄取并通过细胞因子激活免疫系统,表现出显著增强的杀菌、降解和分泌功能。
4 基于肠道3D细胞培养模型的宿主与食源
性致病菌互作研究
4.1 多细胞球体
多细胞球体是多个细胞简单的聚集体形式,是细胞通过整合素与细胞外基质相互作用而自组织形成的,该过程与细胞黏附和细胞分化有关。多细胞球体是异质性细胞群落,包含增殖、静止、缺氧、凋亡和坏死等各阶段的细胞[28](图2)。多细胞球体的最内层包含坏死细胞和其旁边的凋亡细胞,由于营养物质的缺乏和有毒废物的积累而失去活性;中间层细胞处于缺氧或静止状态,由于它们从培养基中获得的氧气、营养物质和其他基本化合物较少,所以代谢活性较小,但暴露于营养物质后会变得活跃;而最外层细胞高度暴露于培养基中,是增殖的细胞和静止的可存活细胞[30](图2-a)。该模型能够模仿真实组织的结构和功能,研究细胞-细胞和细胞-细胞外基质相互作用,模拟胚胎发生、形态发生和器官发生等体内过程,能形成废弃物、氧气、营养物质和信号因子的梯度[31](图2-a)。与2D单细胞层模型相比,该模型更适合研究肠道与食源性致病菌之间的相互作用。
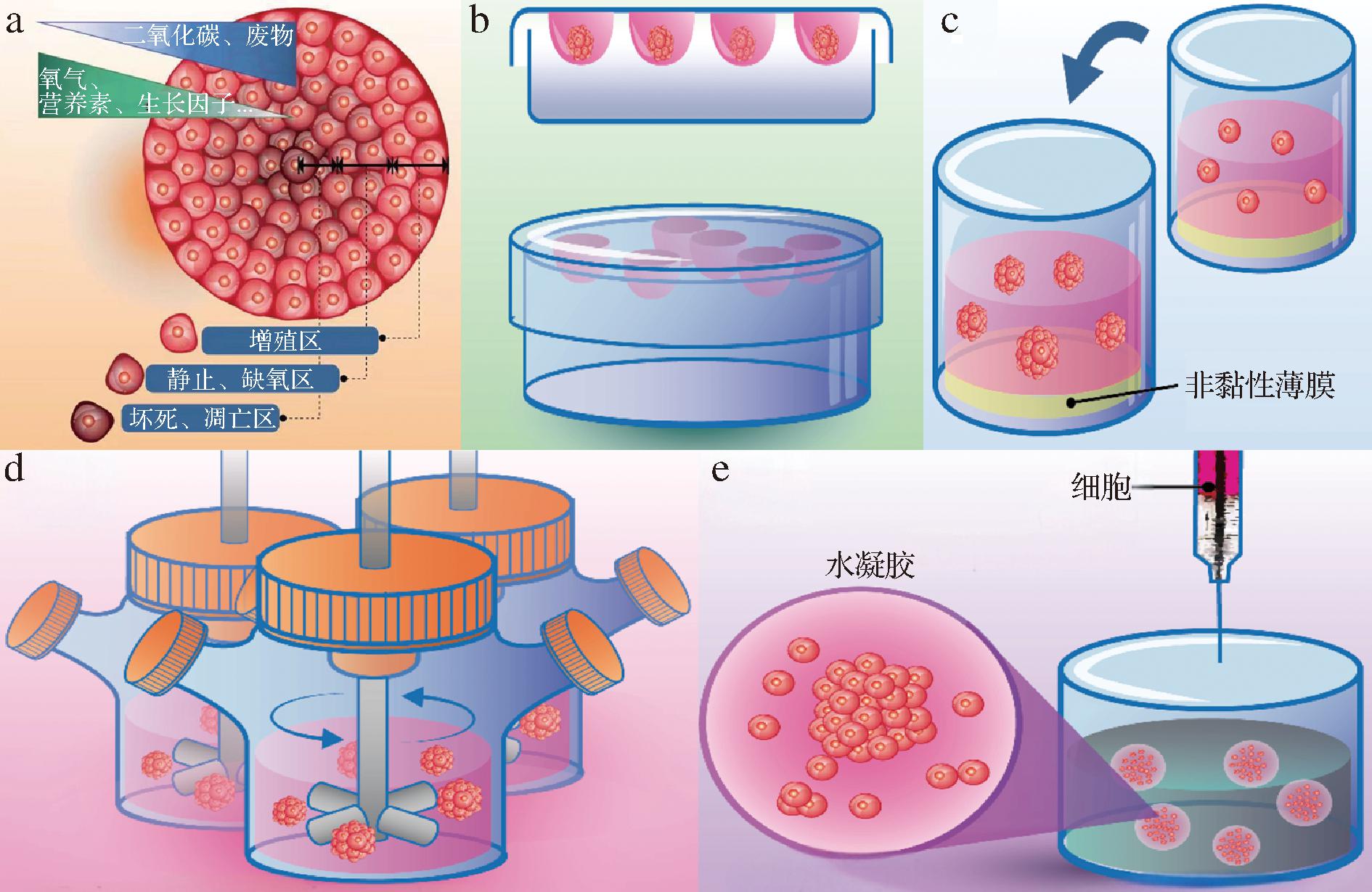
a-球状体的细胞区域示意图;b-悬滴法;c-非黏性表面法;d-旋转瓶生物反应器法;e-水凝胶包埋法
图2 3D球状体细胞区域示意图和不同的培养技术示意图[29]
Fig.2 Schematic diagram of the 3D spheroid cellular region and different culturing techniques[29]
多细胞球体的培养方法有悬滴法、非黏性表面培养法、旋转瓶或旋转生物反应器培养法、以及水凝胶包埋法等,其性能、优缺点如表1所示。悬挂液滴法(图2-b)是一种基于重力和表面张力原理的简单培养方法,一般从单层细胞培养开始,然后将细胞制备为悬浮液,然后使用移液器将细胞悬液滴到培养板上,将培养板倒置,在重力作用下聚集在液滴底部形成一个球体[32]。该方法可形成大小均匀的球体,但需要大量的劳动力且无法进行大规模生产。非黏性表面培养法(图2-c)是阻断细胞附着并使悬浮细胞聚集成可见的球状体,该方法简单、可大规模生产,但缺乏细胞外基质支持[33]。旋转瓶或生物反应器培养法(图2-d)通过连续旋转防止细胞黏附,从而大量生产3D球体,可以调节烧瓶的转速以获得所需尺寸的球体,可以输送营养和氧气,去除废物,然而,连续的旋转可能会对细胞造成机械损伤[31]。上述3种方法操作简单、快速、经济,但缺乏细胞和细胞外基质的相互作用。
表1 多细胞球体培养方法的优缺点
Table 1 Advantages and disadvantages of multicellular spheroid culturing methods

技术方法性能优点缺点参考文献旋转生物反应器培养法模拟微重力将细胞悬浮在动态液体中,持续恒定转速搅拌形成球体长期培养、大规模生产、球体尺寸可控、多细胞共培养高速搅拌可能造成机械损伤、缺乏细胞-基质相互作用[31]悬挂液滴法利用表面张力和重力可控制球状体体积、操作简单、重现性高耗时、劳动力大、无法大规模生产、缺乏细胞-基质相互作用[32]非黏性表面培养法使细胞聚集成球体,悬浮不贴壁生长简单、长期培养、可与异质细胞共培养缺乏细胞-细胞-基质相互作用[33]水凝胶包埋法利用物理特性提供细胞外基质可进行多细胞共培养、价格低廉、重现性高凝胶结构会随时间变化,不稳定[34]
水凝胶具有丰富的细胞外基质蛋白和细胞支持基质网络,可为多细胞球体提供细胞外基质,以调整多细胞球体的微环境。可以通过二维水凝胶表面和三维水凝胶包埋法培养多细胞球体。二维水凝胶表面是将水凝胶聚合物(无明显的细胞结合位点)涂覆在基材上,从而导致细胞无法附着在底物上,并促进细胞与细胞的结合形成聚集体[35]。水凝胶包埋法(图2-e)是将细胞嵌入多孔水凝胶中,水凝胶充当细胞外基质,细胞会沉入底部中并能够聚集形成球状体。此外,也可应用微流体技术形成3D多细胞球体,该方法能帮助形成均匀大小的球体,减小机械力对细胞的损伤,增强了细胞增殖、存活、代谢能力等[33]。
多细胞球体模型已被应用于研究食源性致病菌感染肠道,宿主-致病菌的相互作用并揭示感染机制。NICKERSON等[36]使用旋转壁容器生物反应器构建了肠道3D多细胞聚集体的培养模型,证明了3D肠道细胞对沙门氏菌感染反应与同一细胞系的传统2D单层模型研究结果有很大不同,包括细菌黏附和侵袭、细胞凋亡等。BARRILA等[37]报告了一种结肠上皮细胞和巨噬细胞的3D多细胞共培养感染模型,对具有不同宿主适应性和疾病表型的沙门氏菌定植机制进行了研究。多细胞球体模型还用于研究宿主-食源性致病菌-共生微生物之间的相互作用。共生微生物可以通过复杂机制自然增强肠黏膜屏障功能,防止致病菌定植。DE等[38]使用人类结肠上皮的3D多细胞培养模型,研究罗伊氏乳杆菌在保护肠道免受沙门氏菌感染的潜在作用,发现其可通过免疫调节、竞争结合位点,以及将甘油转化为抗菌物质来保护肠道上皮细胞免受沙门氏菌感染。
总之,3D多细胞球体模型能够比2D单层模型更准确地模拟体内肠道的生理结构和功能,更真实地复制食源性致病菌感染肠道时的复杂环境,推进宿主-致病菌的相互作用研究。但悬浮液滴和非黏性表面等多种传统方法由于分子扩散,形成的球体直径有限,直径过大时,球体中间由于缺乏营养和氧气以及代谢废物的积累会出现坏死区域。新型生成球状体技术包括水凝胶和微流控技术是模拟人体微环境更有效的方法,但成本较高,操作难度大,这些局限性也阻碍了多细胞球体生成技术的广泛应用。
4.2 肠道类器官
肠道类器官是目前研究的最为成熟的类器官之一,是自我更新和分化的干细胞衍生的3D细胞结构,具有自我更新、自组织、在体外准确复制肠上皮的结构和细胞组成、可稳定传代培养等特点[39]。肠道类器官主要有2种构建方法,一种由人或小鼠小肠或结肠组织中收获含有隐窝的成体干细胞、或分离的单个表达lgr5的肠道干细胞衍生的肠道类器官(图3),这种类器官具有较为成熟的特征。如SATO等[41]首次使用小鼠的单个Lgr5+干细胞开发的类器官,重现了小鼠肠道的生理和组织结构,当移植到小鼠体内时,也成功重建了隐窝绒毛单元。另一种是由诱导多能干细胞、胚胎干细胞或含有上皮细胞和间充质细胞的碎组织分化为肠道类器官(图3),这种类器官更近似与胎儿的特征。比如SPENCE等[42]首次将人类胚胎干细胞和诱导多能干细胞分化为肠道类器官用以模拟胚胎肠道发育,重现了干细胞到极化肠上皮的分化过程,分化的极化肠上皮形成绒毛状结构和隐窝状增殖区,并包含所有主要上皮细胞类型(杯状细胞、潘氏细胞和内分泌细胞等)。这2种构建方法都可以形成明确的3D结构,包括小肠的绒毛和隐窝微结构,并能够长时间进行自我更新和自我组织。肠道类器官能够模拟人体肠道组织结构、细胞组成,保留了区域的特异性,同时也能稳定遗传,因此在研究食源性致病菌与肠道互作方面潜力巨大。
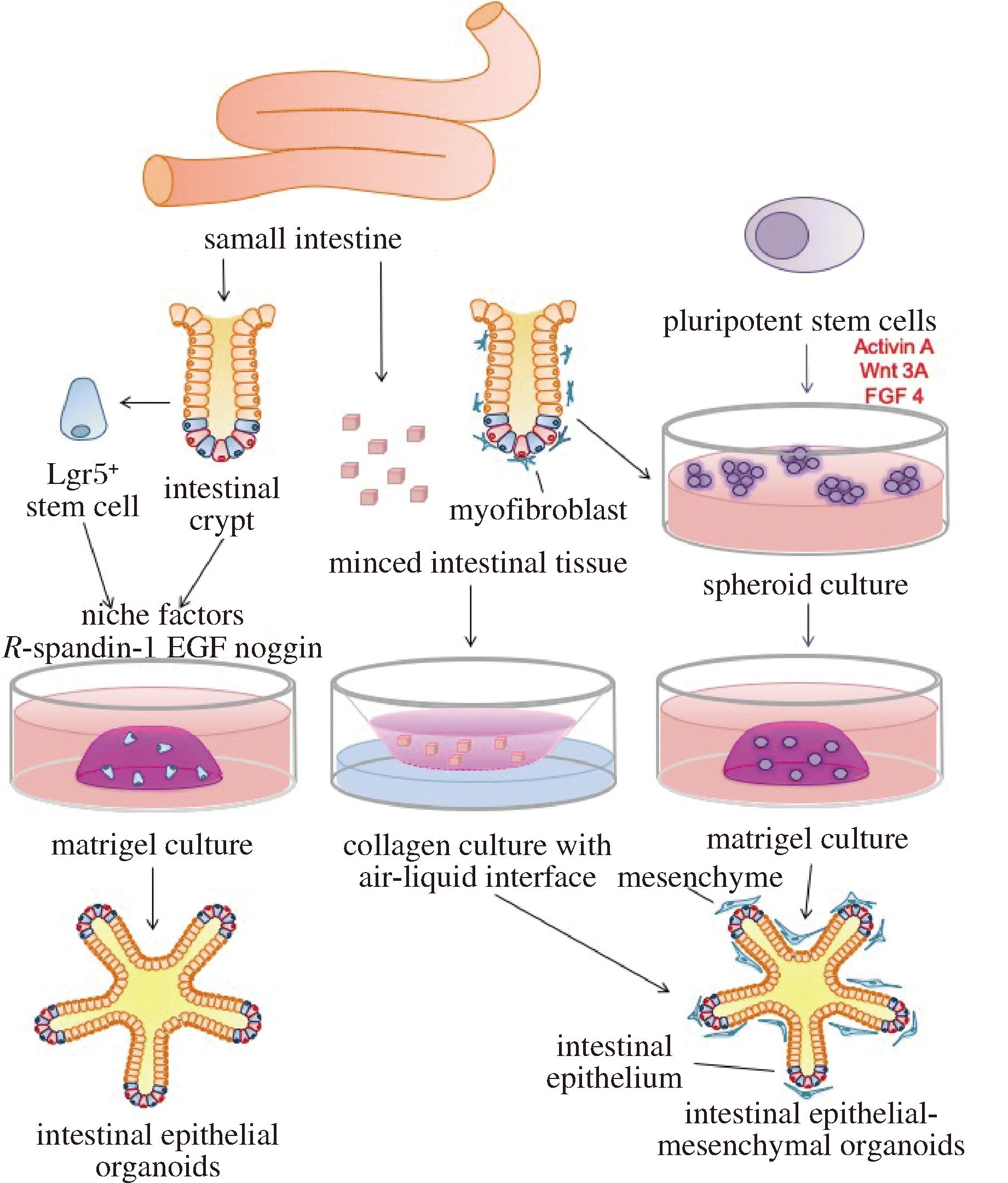
图3 肠道类器官示意图[40]
Fig.3 Schematic diagram of intestinal organoids[40]
注:small intestine-小肠;Lgr5+ stem cell-Lgr5+干细胞;intestinal crypt-肠道隐窝;niche factors-生态位因子;R-spandin-1-Wnt信号放大器和Lgr5配体;EGF(epidermal growth factor)-表皮生长因子;noggin-一种分泌型糖蛋白,可诱导沿隐窝-绒毛轴形成异位隐窝;matrigel culture-基质胶中培养;intestinal epithelial organoids-肠上皮类器官; myofibroblast-成肌纤维细胞;minced intestinal tissue-切碎的肠道碎组织;collagen culture with air-liquid interface-在气-液界面处的胶原蛋白中培养;pluripotent stem cells-多能干细胞;activin A-激活素A,用来促进确定内胚层的分化;FGF 4(fibroblast growth factor 4)-成纤维细胞生长因子 4,用来诱导间充质
细胞从中胚层分化;spheroid culture-培养成球体;mesenchyme-间充质细胞;intestinal epithelium-肠上皮细胞;intestinal epithelial-mesenchymal organoids-肠上皮-间充质类器官[40]。
在肠上皮与致病菌相互作用的研究中获得了一些前所未有的实验结论。ZHANG等[43]利用隐窝来源的小鼠肠道类器官分析鼠伤寒沙门氏菌与上皮细胞的相互作用,发现病原体感染的肠道类器官中上皮紧密连接被破坏,炎症反应被激活以及干细胞标志物Lgr5减少,重现了许多体内模型的结果。HUANG等[44]利用小鼠小肠类器官建L.monocytogenes的入侵模型,结果表明L.monocytogenes减少类器官出芽率、增加了类器官的死亡率、影响上皮细胞的增殖和分泌细胞的数量、降低了Lgr5+干细胞的数量。ROUCH等[45]利用人肠隐窝类器官培养分化出富含有功能性M细胞的上皮层,发现S.typhimurium会优先感染肠上皮层中的M细胞、并诱导M细胞分化。
肠道类器官也成为研究食源性致病菌-肠道微生物群相互作用机制的新工具。PRANHAN等[46]构建了人胚胎干细胞H1来源的人肠类器官,评估了益生菌大肠杆菌Nissle会激活人体防御机制,使宿主免受致病性E.coli介导的上皮屏障破坏、氧化应激增加和细胞凋亡。LU等[47]使用小鼠肠道类器官证明嗜酸乳杆菌可以减轻S.typhimurium引起的肠上皮损伤,并通过抑制S.typhimurium介导的杯状细胞和潘氏细胞的过度扩张,促进了肠上皮细胞的分化,也调节了S.typhimurium介导的过度激活的Wnt/β-catenin信号通路,产生更多的黏液和抗菌肽来加强黏膜屏障。
类器官比上文多细胞球体模型产生了更多的组织水平表型,具有更高的组织复杂性,为研究宿主与食源性致病菌相互作用机制提供了更为强大的工具。然而,肠道类器官仍然存在一定的局限性,如缺乏复杂的间充质异质性和结构、血管系统、神经组织、免疫细胞和机械力(流体流动和蠕动),这些都是正常肠道生理学和疾病发展的关键因素,因此无法完全复制宿主与致病菌相互作用的整体复杂性。另外,自然形成的肠类器官本质上是一个封闭管腔,限制致病菌与上皮细胞顶端的接触,难以完成取样、转运研究、药物暴露。尽管微注射技术已被开发用于克服这个问题,然而这是一个困难耗时的过程。为此,CO等[48]开发了一种反转肠上皮极性的技术,利用螯合剂乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA)破坏细胞外基质(extracellular matrix,ECM),顶端表面朝外避开介质,从而使致病菌更容易与类器官的接触。总体而言,肠道类器官的发展推进了食源性致病菌致病机制和宿主响应方面的研究。
4.3 微流控器官芯片
微流控器官芯片是将微流控技术和生物材料相结合构建的仿生模型,能在体外模拟组织和器官的基本特征,提供相关的生理结构、机械力和微环境[49],为宿主-食源性致病菌相互作用的研究开辟了新的途径。
4.3.1 单一肠芯片
肠芯片的结构和流路设计直接影响其是否可以有效模拟人体肠道的生理功能和3D结构。目前,肠芯片结构主要有3种类型(图4):2D三明治结构、平面平行结构和3D支架结构。2D三明治结构(图4-a)是由多孔半透膜分开2个通道(上下层),通道可以设计成直的、曲折的或其他复杂的结构。上通道培养肠上皮细胞形成屏障,下通道培养基质细胞(如内皮细胞、成纤维细胞或免疫细胞)来模拟细胞-细胞相互作用,半透膜可以促进可溶性分子和营养物质在肠腔和血管之间的运输[53]。2D三明治结构满足了大多数肠道研究的需求,但由于膜透明度的限制,很难通过这种肠芯片获得高分辨率的图像,同时它不能很好地模拟绒毛隐窝结构,芯片与真实肠道环境之间存在一定差距。基于平面平行结构(图4-b)的肠道芯片通常由3个平行的微通道组成,包括上皮通道、凝胶通道和内皮细胞通道[54]。外基质凝胶填充在中央微通道中,使肠道细胞与细胞外基质凝胶充分结合,形成完整的肠道屏障。使用细胞外基质凝胶降低了芯片制造难度,使其适合高通量分析[55]。这种结构也适合添加血管内皮细胞、免疫细胞和肠道微生物,以增加芯片的复杂度,拓宽其应用领域。黏附在基质凝胶上的细胞层平面平行于显微镜的视角,普通显微镜不能直接观察细胞层的状态,需要使用共聚焦激光扫描显微镜等,才能获得整个芯片的清晰3D图像[56]。3D支架结构的体外模型(图4-c)通过使用合成/天然水凝胶材料模拟肠绒毛的支架模型,为共培养系统创造了一个3D微环境,可以控制绒毛的大小和分部,更接近人体肠道的绒毛隐窝结构,可诱导肠上皮细胞高度分化[57]。该芯片的主要优点是持续灌注流动介质,有助于营养物质、气体和废物的流动,引导3D模型中一系列细胞行为(如细胞黏附、增殖、分化)来生成组织,模拟宿主-微生物群落相互作用[58]。具有3D支架的肠道芯片可以培养与肠上皮细胞密切联系的细胞,如内皮细胞、免疫细胞和肌成纤维细胞。因此,在条件允许的情况下,采用3D支架结构设计的芯片肠道是研究宿主与食源性致病菌相互作用的更好选择。
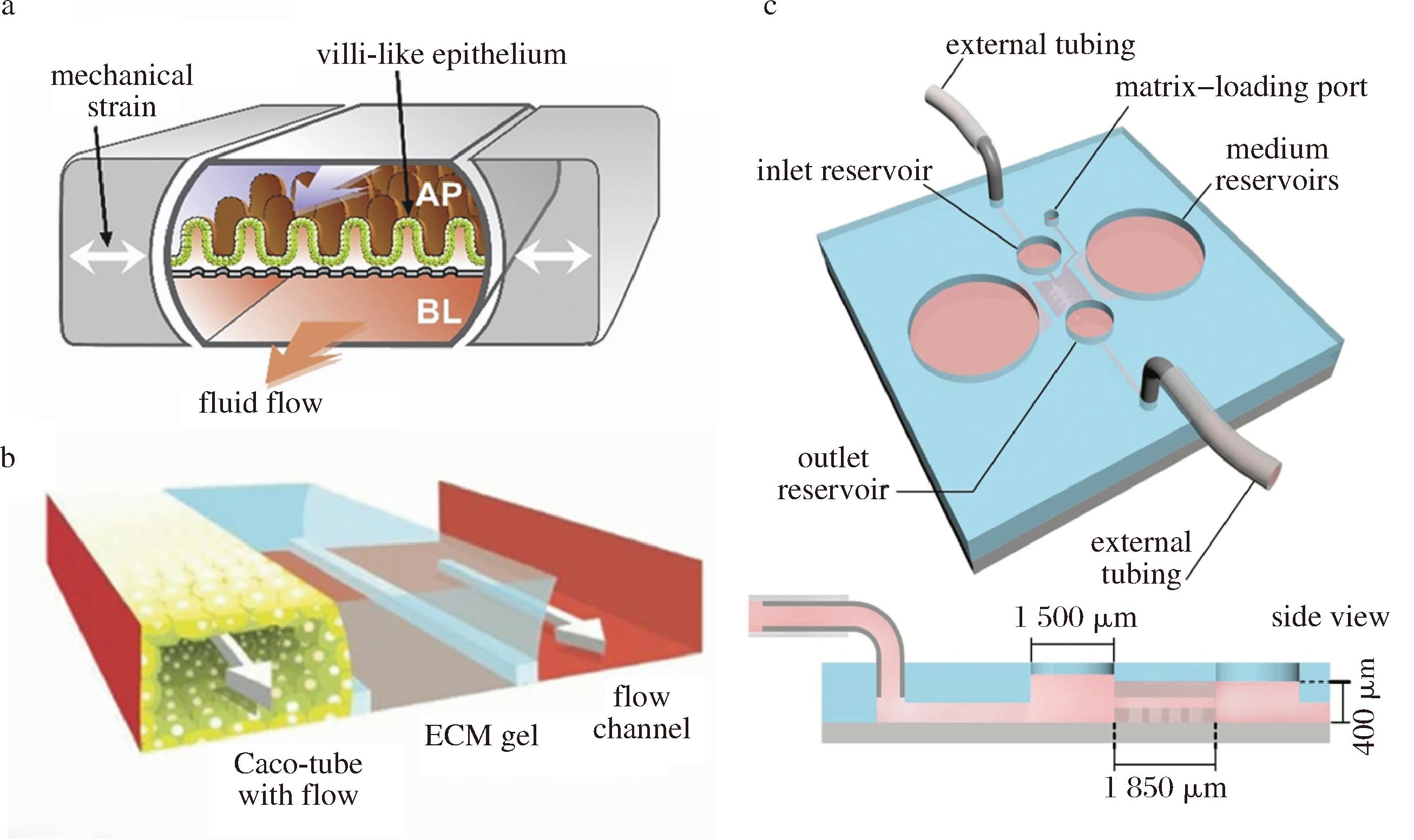
a-2D三明治结构;b-平面平行结构;c-3D支架结构
图4 肠芯片不同结构示意图[50-52]
Fig.4 Schematic diagram of the different structures of intestine-on-a-chip[50-52]
注:mechanical strain-机械应力;villi-like epithelium-上皮绒毛;fluid flow-液体流动;Caco-tube with flow-流动的Caco通道;ecm gel-细胞外基质凝胶;flow channel-流动的通道;external tubing-外部管道;matrix-loading port-加入胶原蛋白的孔;inlet reservoir-进口储液槽;outlet reservoir-出口储液槽;medium resevoirs-培养基储液槽[50-52]。
单一肠道芯片已经作为新工具用于研究宿主-肠道菌-食源性致病菌互作关系(表2),并得到了其他体内外模型不能获得的一些重要结论。肠道器官芯片已被应用于益生菌抑制致病菌定植和感染的机制方面的研究。JEON等[61]设计了三通道微流体的肠道芯片模型,证明了益生菌植物乳杆菌对肠道屏障完整性的保护作用。KIM等[65]使在商业化的肠道芯片,发现益生菌治疗制剂(含有8种益生菌菌株的混合物)可以增强肠道屏障功能,减少肠侵袭性E.coli对肠腔的损伤。JING等[66]使用三通道层压微流控芯片,发现干酪乳杆菌可以防止E.coil(ATCC11775)的过度生长,避免其对肠道屏障的破坏,显著缓解了E.coil引起的糖萼和微绒毛的损伤,增加了闭合蛋白水平,并降低了肠腔和血管腔中促炎细胞因子的分泌水平。此外,肠道芯片也被用来研究致病菌与肠道的互作关系。GRASSART等[59]使用2D三明治肠道芯片发现流动和蠕动会显著增加志贺氏菌(Shigella)在结肠上皮内的感染与侵袭,且志贺氏菌会从根尖侧感染肠细胞且迅速定植于隐窝。SUNUWAR等[63]在器官芯片中培养人空肠肠道类固醇单层,并暴露于肠毒素大肠杆菌热稳定肠毒素A,证明了机械刺激可以控制E.coil的热稳定肠毒素循环GMP信号。GAZZANIGA等[64]构建了小鼠结肠芯片,证明肠芯片可以较好地模拟鼠伤寒沙门氏菌和肠道之间的互作关系。单个肠道芯片虽具有诸多优势,但是其无法反映食源性致病菌对人体其他器官的影响,将肠道和其他器官串联建模,也为宿主-肠道菌-食源性致病菌相互作用的研究开辟了新途径。
表2 不同肠道芯片在宿主与食源性致病菌相互作用研究中的应用
Table 2 Applications of different intestine-on-a-chip in the study of host-borne pathogenic bacteria interactions
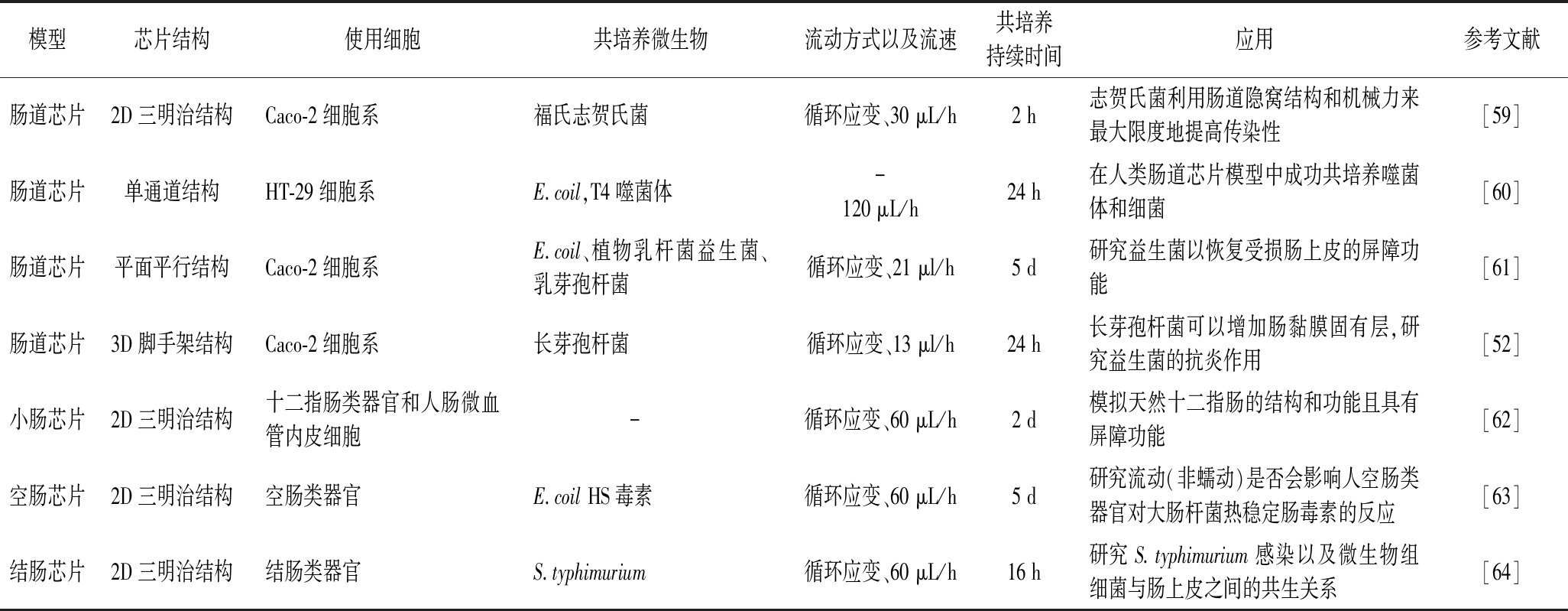
模型芯片结构使用细胞共培养微生物流动方式以及流速共培养持续时间应用参考文献肠道芯片2D三明治结构Caco-2细胞系福氏志贺氏菌循环应变、30 μL/h2 h志贺氏菌利用肠道隐窝结构和机械力来最大限度地提高传染性 [59]肠道芯片单通道结构HT-29细胞系E.coil,T4噬菌体-120 μL/h24 h在人类肠道芯片模型中成功共培养噬菌体和细菌 [60]肠道芯片平面平行结构Caco-2细胞系E.coil、植物乳杆菌益生菌、乳芽孢杆菌循环应变、21 μl/h5 d研究益生菌以恢复受损肠上皮的屏障功能 [61]肠道芯片3D脚手架结构Caco-2细胞系长芽孢杆菌循环应变、13 μl/h24 h长芽孢杆菌可以增加肠黏膜固有层,研究益生菌的抗炎作用 [52]小肠芯片2D三明治结构十二指肠类器官和人肠微血管内皮细胞 -循环应变、60 μL/h2 d模拟天然十二指肠的结构和功能且具有屏障功能 [62]空肠芯片2D三明治结构空肠类器官E.coil HS毒素循环应变、60 μL/h5 d研究流动(非蠕动)是否会影响人空肠类器官对大肠杆菌热稳定肠毒素的反应 [63]结肠芯片2D三明治结构结肠类器官S.typhimurium循环应变、60 μL/h16 h研究S.typhimurium感染以及微生物组细菌与肠上皮之间的共生关系 [64]
注:-表示无数据(下同)。
4.3.2 多器官芯片
宿主与致病菌之间的复杂作用不可避免地会影响其他器官的功能,然而传统的体外模型,如球状体、类器官和单器官芯片无法模拟器官和组织之间的相互作用。多器官芯片模型是基于微流体技术将多个器官连接在一起,一般构建方法是使用器官特异性培养基单独培养单器官,达到所需的成熟或分化水平且满足各个器官整合的条件时,再用微流控技术使其相互连接,形成多个模块化配置[67]。BRICKS等[68]基于Caco-2和肝(HepG2)细胞分别模拟肠道和肝脏区室,开发了一种肠-肝芯片,研究了肠-肝脏器官之间的药物(对乙酰氨基酚)代谢关联。器官越多,系统的复杂性也随之增加。MASCHMEYER等[69]构建的器官芯片扩展到了4个器官,包括肝脏、皮肤、肾脏和肠道,研究药物在小肠中的系统吸收和代谢、肝脏的代谢、肾脏的排泄以及体外皮肤活检,从而得出药物的毒性。
多器官芯片适合用于研究药物治疗食源性致病菌感染,对肠道、肝脏、肾脏等器官引起的继发毒性和对身体的损伤。为研究抗生素对其治疗效果,LEE等[70]开发了肠-肾芯片,共培养Caco-2和肾脏(HKC-8)细胞,用产志贺毒素E.coli进行感染,结果表明环丙沙星治疗比庆大霉素治疗会对人体细胞造成更多的损害。JIE等[71]使用Caco-2细胞和HepG2细胞构建了肠-肝芯片,研究组合药物(染料木黄酮和达卡巴嗪)的肠道吸收和肝脏代谢,结果表明高浓度的药物组合抑制了HepG2细胞的生长,而中等浓度的药物组合维持了肝细胞的正常代谢功能。目前的多器官芯片模型多用于研究药物吸收、药物转运和代谢研究,而致病菌上的研究较少。
尽管微流控肠芯片模型有效地模拟了人类肠道的许多不同表型和反应,但肠芯片的肠壁成分有限,不能充分反映肠道微生态学,可能会影响宿主-肠道菌-食源性致病菌互作关系的研究。因此,未来的研究中应考虑添加更多的细胞,如结缔组织成分、肌肉细胞和肠神经系统细胞。当前器官芯片重要的挑战是微流控器件的开发时间长,芯片设计与制造的专业性和繁琐性限制大规模使用。最近器官芯片技术已开始商业化,这为扩大制造规模、提高稳健性、降低成本等相关的问题提供了可能性。生物材料的选择也是挑战之一,开发出生物相容性、惰性、非吸附性和不浸出的材料将是一个主要的研究方向。多器官芯片有可能成为致病菌感染机制研究、治疗药物筛选、以及疾病建模等研究领域的范式转变对象。
4.4 3D生物打印
多细胞球体、类器官、器官芯片等模型的精度和可控性有限,不能精确重现天然组织的微观结构和尺寸。3D生物打印使用计算机技术高度可重复地选择性分布细胞、生物材料、生长因子等,精确地控制结构组成和空间分布,以创建复杂的组织和器官[72]。将要打印的细胞嵌入到生物相容的基质中,该基质由聚合物、营养素、生长因子和功能性多肽组成,统称为生物墨水,使用不同的方法和模式将该生物墨水打印成所需的形状和尺寸,以产生仿生3D组织结构[73](表3)。
表3 四种3D细胞培养模型优缺点的比较
Table 3 Comparison of the advantages and disadvantages of four 3D cell culture models

模型特点优点缺点实例参考文献多细胞球体依赖于细胞之间的黏附、聚集、自组装低成本;高通量筛选;重复性高缺乏干细胞,无法进行自我更新与分化、球状体内核缺氧沙门氏菌对肠道黏附、侵袭[36]沙门氏菌与结肠[37]肠道类器官干细胞自组织更新形成、具有高度相似的组织学特征,如隐窝-绒毛结构可长期稳定传代增殖培养、保持原代基因组学稳定性、分子的高通量筛选官腔结构封闭,内环境不稳定、缺乏特异性细胞类型、组织结构的生理确定性差沙门氏菌与肠道上皮层[43]L.monocytogenes与肠道上皮层[44]E.coli与肠道上皮屏障[46]肠芯片动态连续灌注培养的微环境、自发分化成具有3D绒毛结构的单层3D仿生微环境、提供共培养、流体剪切力等、成本较低、成熟稳定的细胞系、相对成本较高、芯片测试通量低、难以收集细胞用于分析E.coli与肠道上皮屏障[65]志贺氏菌与结肠[59]E.coli与空肠[63]沙门氏菌与肠道[64]生物细胞打印打印出具有一定形貌的三维仿生结构精确且快速、高通量、高细胞活力成本高、无法打印复杂结构和多种细胞类型的组织、精度难以保持-[73]
生物打印技术主要有3种:基于挤出、喷墨和激光辅助的生物打印(图5)。挤出生物打印(图5-a)是最为广泛使用的打印技术,通过气动、活塞驱动或螺杆驱动将载有细胞的生物墨水以连续流的形式沉积在基底上[75]。主要优点是可以打印复杂的结构,该方法已成功用于打印皮肤、神经、肝脏、骨骼肌和内皮结构。但挤出打印速度慢,分辨率相对较低,产生的剪切应力大,导致细胞存活率较低(40%~80%)。喷墨生物打印(图5-b)是一种非接触式打印技术,利用热、电磁或压电将生物墨水滴沉积到基底上[76]。喷墨生物打印的主要优点在于系统的简单性和相对较低的成本,与挤出式打印相比,喷墨式打印的液滴细小、分辨率高以及细胞活力较高(80%~90%),然而,细胞密度相对较低,只能打印低黏度的生物墨水。激光辅助生物打印(图5-c)通过激光脉冲作用于能量吸收层上,产生的高压气泡推动生物墨水到基体上[77]。主要优点是高分辨率和细胞高存活率(>95%),但系统复杂,成本较高。3D生物打印技术可以打印出肠道组织或器官的等效物。如MADDEN等[78]使用生物打印技术生成了可以模拟肠道的人类原代肠上皮细胞和肌成纤维细胞组成的三维组织,KIM等[79]使用挤出生物打印技术和双细胞打印系统制造了带有毛细血管的3D肠绒毛模型,这为研究食源性致病菌生物膜与宿主之间的相互作用提供了另一种途径。
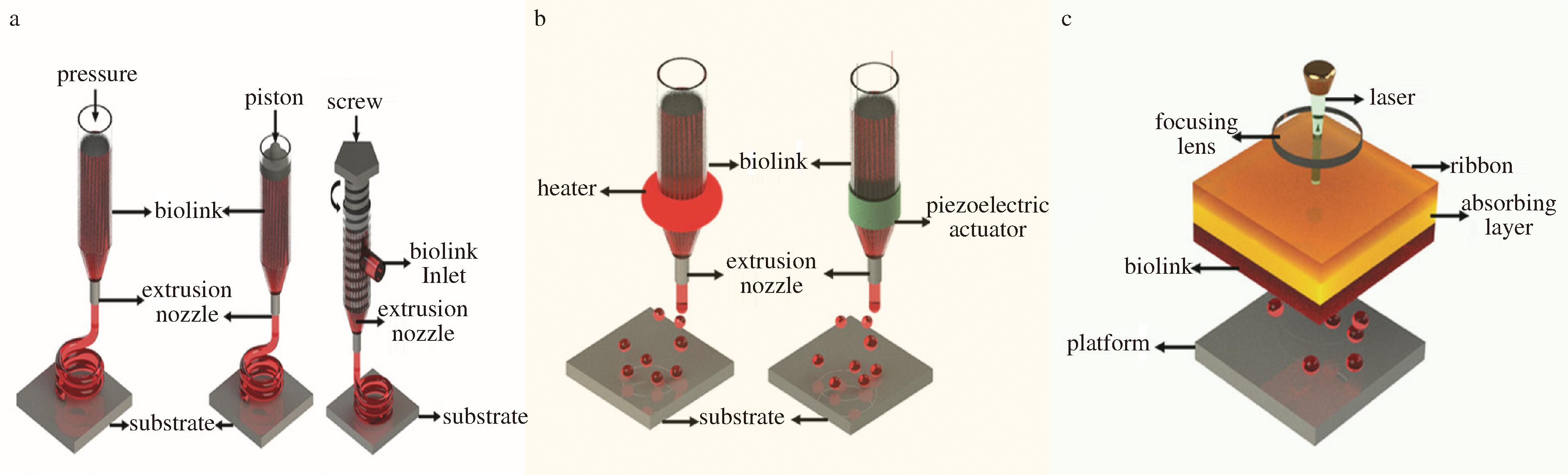
a-挤出生物打印;b-喷墨生物打印;c-激光辅助生物打印
图5 不同3D生物打印技术示意图[74]
Fig.5 Schematic diagram of different 3D bioprinting techniques[74]
注:pressure-按压;bioink-生物墨水;extrusion nozzle-挤压喷嘴;substrate-基底;piston-活塞;screw-螺杆;bioink inlet-生物墨水入口;heater-加热器;piezolectric actuator -压电制动器;laser-激光;ribbon-功能区; focusing lens-聚焦透视镜;absorbing layer-吸收层;platform -基底[74]。
生物打印技术虽进展迅速,但该系统分辨率低于天然组织,仍无法完全模仿天然器官的复杂性和功能性。3D生物打印在创建更复杂的肠道体外3D组织模型方面潜力巨大,然而,目前还未有基于生物打印技术创建的简单肠道器官模型进行宿主-食源性致病菌互作方面的研究,未来可进一步开拓该领域的研究。
5 结论与展望
3D细胞培养模型从简单的球状体到复杂的类器官和肠道芯片,在研究肠道-致病菌、肠道-肠道菌-致病菌互作方面不仅提供了与体内模型相似的研究结果,同时也得到了目前体内模型以往没有发现的新结论,对解析食源性致病菌的感染机制和开发预防策略和新的治疗方案至关重要。
尽管体外3D肠道细胞培养模型取得了较多进展,并且在生理病理学研究和药物开发方面具有重要意义,但仍然存在一些挑战。a)优化共培养参数:共培养增加了3D细胞培养模型的复杂性,为了保持所有细胞类型的最佳生长,与生物学和疾病相关性保持一致,需优化加入3D模型中的细胞共培养物的各种参数,比如细胞比例、细胞培养基组成、培养细胞需求的培养基补充剂等。b)整合免疫成分:食源性致病菌和宿主免疫系统之间的相互作用对于复制感染结果至关重要,应评估免疫细胞浸润(如淋巴细胞、巨噬细胞)、活化和细胞因子分泌对抗致病菌的能力。c)开发更加有效标准的3D细胞培养模型:理想的模型应该能够反映食源性致病菌感染的关键因素,例如准确复制肠道组织机械性能、再现氧梯度、致病菌黏附侵袭位点、并且必须允许增殖、分化和迁移。以此推进3D细胞模型的开发,可更加准确地重现体内状况。d)开发先进监测技术:对肠道模型的实时监测可以准确捕获系统的动态反应,包括屏障完整性、细胞健康和蛋白质表达等,将大大改善当前的3D细胞培养模型。目前,与3D模型相关的广泛研究仅限于学术界,未来,还需要开发更兼容、成本效益高和可重复的系统,使更多的研究成果转化为实际的成果还需要克服巨大的障碍。
[1] KIM R, SUNG J H.Recent advances in gut- and gut-organ-axis-on-a-chip models [J].Advanced Healthcare Materials, 2024,13(21):e23032777.
[2] MCCOY R, OLDROYD S, YANG W, et al.In vitro models for investigating intestinal host-pathogen interactions[J].Advanced Science, 2024, 11(8):2306727.
[3] STEINWAY S N, SALEH J, KOO B K, et al.Human microphysiological models of intestinal tissue and gut microbiome [J].Frontiers in Bioengineering and Biotechnology, 2020, 8:725.
[4] MALIJAUSKAITE S, CONNOLLY S, NEWPORT D, et al.Gradients in the in vivo intestinal stem cell compartment and their in vitro recapitulation in mimetic platforms [J].Cytokine &Growth Factor Reviews, 2021, 60:76-88.
[5] TORRAS N, ZABALO J, ABRIL E, et al.A bioprinted 3D gut model with crypt-villus structures to mimic the intestinal epithelial-stromal microenvironment [J].Biomaterials Advances, 2023, 153:213534.
[6] ZACHOS N C, KOVBASNJUK O, FOULKE-ABEL J, et al.Human enteroids/colonoids and intestinal organoids functionally recapitulate normal intestinal physiology and pathophysiology [J].Journal of Biological Chemistry, 2016, 291(8):3759-3766.
[7] GAO J W, CAO B, ZHAO R Y, et al.Critical signaling transduction pathways and intestinal barrier:Implications for pathophysiology and therapeutics [J].Pharmaceuticals, 2023, 16(9):1216.
[8] GROOTJANS J, THUIJLS G, VERDAM F, et al.Non-invasive assessment of barrier integrity and function of the human gut [J].World Journal of Gastrointestinal Surgery, 2010, 2(3):61-69.
[9] MÉNARD S, LACROIX-LAMANDÉ S, EHRHARDT K, et al.Cross-talk between the intestinal epithelium and Salmonella typhimurium [J].Frontiers in Microbiology, 2022, 13:906238.
[10] ALLAM-NDOUL B, CASTONGUAY-PARADIS S, VEILLEUX A.Gut microbiota and intestinal trans-epithelial permeability [J].International Journal of Molecular Sciences, 2020, 21(17):6402.
[11] VONAESCH P, ANDERSON M, SANSONETTI P J.Pathogens, microbiome and the host:Emergence of the ecological Koch’s postulates [J].FEMS Microbiology Reviews, 2018, 42(3):273-292.
[12] MIRZAEI R, DEHKHODAIE E, BOUZARI B, et al.Dual role of microbiota-derived short-chain fatty acids on host and pathogen [J].Biomedicine &Pharmacotherapy, 2022, 145:112352.
[13] ABT M C, PAMER E G.Commensal bacteria mediated defenses against pathogens [J].Current Opinion in Immunology, 2014, 29:16-22.
[14] GARC A-D
A-D AZ M, CENDRA M D M, ALONSO-ROMAN R, et al.Mimicking the intestinal host-pathogen interactions in a 3D in vitro model:The role of the mucus layer [J].Pharmaceutics, 2022, 14(8):1552.
AZ M, CENDRA M D M, ALONSO-ROMAN R, et al.Mimicking the intestinal host-pathogen interactions in a 3D in vitro model:The role of the mucus layer [J].Pharmaceutics, 2022, 14(8):1552.
[15] SANTOS R L.Pathobiology of Salmonella, intestinal microbiota, and the host innate immune response [J].Frontiers in Immunology, 2014, 5:252.
[16] MOSSER D M, EDWARDS J P.Exploring the full spectrum of macrophage activation [J].Nature Reviews Immunology, 2008, 8(12):958-969.
[17] SAEZ A, HERRERO-FERNANDEZ B, GOMEZ-BRIS R, et al.Pathophysiology of inflammatory bowel disease:Innate immune system [J].International Journal of Molecular Sciences, 2023, 24(2):1526.
[18] GUERREIRO D N, ARCARI T, O’BYRNE C P.The σB-mediated general stress response of Listeria monocytogenes:Life and death decision making in a pathogen [J].Frontiers in Microbiology, 2020, 11:1505.
[19] GAHAN C G M, HILL C.Listeria monocytogenes:Survival and adaptation in the gastrointestinal tract [J].Frontiers in Cellular and Infection Microbiology, 2014, 4:9.
[20] QUEREDA J J, DUSSURGET O, NAHORI M A, et al.Bacteriocin from epidemic Listeria strains alters the host intestinal microbiota to favor infection [J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(20):5706-5711.
[21] QUEREDA J J, MOR N-GARC
N-GARC A A, PALACIOS-GORBA C, et al.Pathogenicity and virulence of Listeria monocytogenes:A trip from environmental to medical microbiology [J].Virulence, 2021, 12(1):2509-2545.
A A, PALACIOS-GORBA C, et al.Pathogenicity and virulence of Listeria monocytogenes:A trip from environmental to medical microbiology [J].Virulence, 2021, 12(1):2509-2545.
[22] LIU B, JIANG L Y, LIU Y T, et al.Enterohaemorrhagic E.coli utilizes host- and microbiota-derived L-malate as a signaling molecule for intestinal colonization [J].Nature Communications, 2023, 14(1):7227.
[23] GARMENDIA J, FRANKEL G, CREPIN V F.Enteropathogenic and enterohemorrhagic Escherichia coli infections:Translocation, translocation, translocation [J].Infection and Immunity, 2005, 73(5):2573-2585.
[24] LANGE M E, UWIERA R R E, DOUGLAS INGLIS G.Enteric Escherichia coli O157:H7 in cattle, and the use of mice as a model to elucidate key aspects of the host-pathogen-microbiota interaction:A review [J].Frontiers in Veterinary Science, 2022, 9:937866.
[25] JAJERE S M.A review of Salmonella enterica with particular focus on the pathogenicity and virulence factors, host specificity and antimicrobial resistance including multidrug resistance [J].Veterinary World, 2019, 12(4):504-521.
[26] YIN Y B, ZHOU D G.Organoid and enteroid modeling of Salmonella infection [J].Frontiers in Cellular and Infection Microbiology, 2018, 8:102.
[27] BAKOWSKI M A, BRAUN V, BRUMELL J H.Salmonella-containing vacuoles:Directing traffic and nesting to grow[J].Traffic, 2008, 9(12):2022-2031.
[28] KIM J B.Three-dimensional tissue culture models in cancer biology [J].Seminars in Cancer Biology, 2005, 15(5):365-377.
[29] CHAICHAROENAUDOMRUNG N, KUNHORM P, NOISA P.Three-dimensional cell culture systems as an in vitro platform for cancer and stem cell modeling [J].World Journal of Stem Cells, 2019, 11(12):1065-1083.
[30] CHIEW G G Y, WEI N, SULTANIA S, et al.Bioengineered three-dimensional co-culture of cancer cells and endothelial cells:A model system for dual analysis of tumor growth and angiogenesis [J].Biotechnology and Bioengineering, 2017, 114(8):1865-1877.
[31] LIN R Z, CHANG H Y.Recent advances in three-dimensional multicellular spheroid culture for biomedical research [J].Biotechnology Journal, 2008, 3(9-10):1172-1184.
[32] NETO A I, CORREIA C R, OLIVEIRA M B, et al.A novel hanging spherical drop system for the generation of cellular spheroids and high throughput combinatorial drug screening [J].Biomaterials Science, 2015, 3(4):581-585.
[33] CUI X, HARTANTO Y, ZHANG H.Advances in multicellular spheroids formation [J].Journal of the Royal Society Interface, 2017, 14(127):20160877.
[34] CUI X, DINI S, DAI S, et al.A mechanistic study on tumour spheroid formation in thermosensitive hydrogels:Experiments and mathematical modelling [J].RSC Advances, 2016, 6(77):73282-73291.
[35] BIA KOWSKA K, KOMOROWSKI P, BRYSZEWSKA M, et al.Spheroids as a type of three-dimensional cell cultures-examples of methods of preparation and the most important application [J].International Journal of Molecular Sciences, 2020, 21(17):6225.
KOWSKA K, KOMOROWSKI P, BRYSZEWSKA M, et al.Spheroids as a type of three-dimensional cell cultures-examples of methods of preparation and the most important application [J].International Journal of Molecular Sciences, 2020, 21(17):6225.
[36] NICKERSON C A, GOODWIN T J, TERLONGE J, et al.Three-dimensional tissue assemblies:Novel models for the study of Salmonella enterica serovar typhimurium pathogenesis [J].Infection and Immunity, 2001, 69(11):7106-7120.
[37] BARRILA J, YANG J, CRABBÉ A, et al.Three-dimensional organotypic co-culture model of intestinal epithelial cells and macrophages to study Salmonella enterica colonization patterns [J].NPJ Microgravity, 2017, 3:10.
[38] DE WEIRDT R, CRABBÉ A, ROOS S, et al.Glycerol supplementation enhances L.reuteri′s protective effect against S.typhimurium colonization in a 3-D model of colonic epithelium [J].PLoS One, 2012, 7(5):e37116.
[39] 宋仪洋,吴梦洁,董庆利,等.食源性致病菌感染体内外肠道模型研究进展[J].食品与发酵工业,2024,50(5):340-349.
SONG Y Y, WU M J, DONG Q L, etc.Research progress on in vivo and external intestinal models of foodborne pathogenic bacteria infection[J].Food and Fermentation Industries,2024,50(5):340-349.
[40] CHUSILP S, LI B, LEE D, et al.Intestinal organoids in infants and children [J].Pediatric Surgery International, 2020, 36(1):1-10.
[41] SATO T, VRIES R G, SNIPPERT H J, et al.Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche [J].Nature, 2009, 459(7244):262-265.
[42] SPENCE J R, MAYHEW C N, RANKIN S A, et al.Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro [J].Nature, 2011, 470(7332):105-109.
[43] ZHANG Y G, WU S P, XIA Y L, et al.Salmonella-infected crypt-derived intestinal organoid culture system for host-bacterial interactions [J].Physiological Reports, 2014, 2(9):e12147.
[44] HUANG J, ZHOU C, ZHOU G H, et al.Effect of Listeria monocytogenes on intestinal stem cells in the co-culture model of small intestinal organoids [J].Microbial Pathogenesis, 2021, 153:104776.
[45] ROUCH J D, SCOTT A, LEI N Y, et al.Development of functional microfold (M) cells from intestinal stem cells in primary human enteroids [J].PLoS One, 2016, 11(1):e0148216.
[46] PRADHAN S, WEISS A A.Probiotic properties of Escherichia coli nissle in human intestinal organoids [J].mBio, 2020, 11(4):e01470-20.
[47] LU X X, XIE S, YE L L, et al.Lactobacillus protects against S.typhimurium-induced intestinal inflammation by determining the fate of epithelial proliferation and differentiation [J].Molecular Nutrition &Food Research, 2020, 64(5):e1900655.
[48] CO J Y, MARGALEF-CATAL M, LI X N, et al.Controlling epithelial polarity:A human enteroid model for host-pathogen interactions [J].Cell Reports, 2019, 26(9):2509-2520.e4
M, LI X N, et al.Controlling epithelial polarity:A human enteroid model for host-pathogen interactions [J].Cell Reports, 2019, 26(9):2509-2520.e4
[49] 李向阳, 史鹏程, 张乐, 等.肠芯片在宿主-微生物互作中的研究进展 [J].生物工程学报, 2024,40(9):2916-2933.
LI X Y, SHI P C, ZHANG L, et al.Research progress of intestinal chip in host-microbe interaction [J].Chinese Journal of Biotechnology, 2024,40(9):2916-2933.
[50] SHIN W, HINOJOSA C D, INGBER D E, et al.Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow-dependent physical cues in a microengineered gut-on-a-chip [J].IScience, 2019, 15:391-406.
[51] TRIETSCH S J, NAUMOVSKA E, KUREK D, et al.Membrane-free culture and real-time barrier integrity assessment of perfused intestinal epithelium tubes [J].Nature Communications, 2017, 8(1):262.
[52] NIKOLAEV M, MITROFANOVA O, BROGUIERE N, et al.Homeostatic mini-intestines through scaffold-guided organoid morphogenesis [J].Nature, 2020, 585(7826):574-578.
[53] KASENDRA M, LUC R, YIN J Y, et al.Duodenum Intestine-Chip for preclinical drug assessment in a human relevant model [J].eLife, 2020, 9:e50135.
[54] ZHANG D X, QIAO L.Intestine-on-a-chip for intestinal disease study and pharmacological research [J].View, 2023, 4(1):20220037.
[55] BEAURIVAGE C, NAUMOVSKA E, CHANG Y X, et al.Development of a gut-on-a-chip model for high throughput disease modeling and drug discovery [J].International Journal of Molecular Sciences, 2019, 20(22):5661.
[56] LIANG D, SU W T, TAN M Q.Advances of microfluidic intestine-on-a-chip for analyzing anti-inflammation of food [J].Critical Reviews in Food Science and Nutrition, 2022, 62(16):4418-4434.
[57] COSTELLO C M, SORNA R M, GOH Y L, et al.3-D intestinal scaffolds for evaluating the therapeutic potential of probiotics [J].Molecular Pharmaceutics, 2014, 11(7):2030-2039.
[58] LIU H T, WANG Y Q, CUI K L, et al.Advances in hydrogels in organoids and organs-on-a-chip [J].Advanced Materials, 2019, 31(50):e1902042.
[59] GRASSART A, MALARDÉ V, GOBAA S, et al.Bioengineered human organ-on-chip reveals intestinal microenvironment and mechanical forces impacting Shigella infection[J].Cell Host &Microbe, 2019, 26(3):435-444.e4
[60] CHIN W H, KETT C, COOPER O, et al.Bacteriophages evolve enhanced persistence to a mucosal surface [J].Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(27):e2116197119.
[61] JEON M S, CHOI Y Y, MO S J, et al.Contributions of the microbiome to intestinal inflammation in a gut-on-a-chip [J].Nano Convergence, 2022, 9(1):8.
[62] KASENDRA M, TOVAGLIERI A, SONTHEIMER-PHELPS A, et al.Development of a primary human Small Intestine-on-a-Chip using biopsy-derived organoids [J].Scientific Reports, 2018, 8(1):2871.
[63] SUNUWAR L, YIN J Y, KASENDRA M, et al.Mechanical stimuli affect Escherichia coli heat-stable enterotoxin-cyclic GMP signaling in a human enteroid intestine-chip model [J].Infection and Immunity, 2020, 88(3):e00866-19.
[64] GAZZANIGA F S, CAMACHO D M, WU M, et al.Harnessing colon chip technology to identify commensal bacteria that promote host tolerance to infection [J].Frontiers in Cellular and Infection Microbiology, 2021, 11:638014.
[65] KIM H J, LI H, COLLINS J J, et al.Contributions of microbiome and mechanical deformation to intestinal bacterial overgrowth and inflammation in a human gut-on-a-chip [J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(1):E7-15.
[66] JING B L, WANG Z A, ZHANG C, et al.Establishment and application of peristaltic human gut-vessel microsystem for studying host-microbial interaction [J].Frontiers in Bioengineering and Biotechnology, 2020, 8:272.
[67] ROGAL J, PROBST C, LOSKILL P.Integration concepts for multi-organ chips:How to maintain flexibility?! [J].Future Science OA, 2017, 3(2):FSO180.
[68] BRICKS T, PAULLIER P, LEGENDRE A, et al.Development of a new microfluidic platform integrating co-cultures of intestinal and liver cell lines [J].Toxicology in Vitro, 2014, 28(5):885-895.
[69] MASCHMEYER I, LORENZ A K, SCHIMEK K, et al.A four-organ-chip for interconnected long-term co-culture of human intestine, liver, skin and kidney equivalents [J].Lab on a Chip, 2015, 15(12):2688-2699.
[70] LEE Y, KIM M H, ALVES D R, et al.Gut-kidney axis on chip for studying effects of antibiotics on risk of hemolytic uremic syndrome by shiga toxin-producing Escherichia coli [J].Toxins, 2021, 13(11):775.
[71] JIE M S, LIN H F, HE Z Y, et al.An on-chip intestine-liver model for multiple drugs absorption and metabolism behavior simulation [J].Science China-Chemistry, 2018, 61(2):236-242.
[72] MOTA C, CAMARERO-ESPINOSA S, BAKER M B, et al.Bioprinting:From tissue and organ development to in vitro models [J].Chemical Reviews, 2020, 120(19):10 547-10 607.
[73] OZBOLAT I T.Bioprinting scale-up tissue and organ constructs for transplantation [J].Trends in Biotechnology, 2015, 33(7):395-400.
[74] HEINRICH M A, LIU W J, JIMENEZ A, et al.3D bioprinting:From benches to translational applications [J].Small, 2019, 15(23):e1805510.
[75] MANDRYCKY C, WANG Z J, KIM K, et al.3D bioprinting for engineering complex tissues [J].Biotechnology Advances, 2016, 34(4):422-434.
[76] CRIS WILSON W Jr, BOLAND T.Cell and organ printing 1:Protein and cell printers [J].The Anatomical. Record Part A,Discoveries in Molecular, Cellular, and Evolutionary Biology, 2003, 272(2):491-496.
[77] DUOCASTELLA M, COLINA M, FERN NDEZ-PRADAS J M, et al.Study of the laser-induced forward transfer of liquids for laser bioprinting [J].Applied Surface Science, 2007, 253(19):7855-7859.
NDEZ-PRADAS J M, et al.Study of the laser-induced forward transfer of liquids for laser bioprinting [J].Applied Surface Science, 2007, 253(19):7855-7859.
[78] MADDEN L R, NGUYEN T V, GARCIA-MOJICA S, et al.Bioprinted 3D primary human intestinal tissues model aspects of native physiology and ADME/tox functions [J].iScience, 2018, 2:156-167.
[79] KIM W, KIM G.Intestinal villi model with blood capillaries fabricated using collagen-based bioink and dual-cell-printing process[J].ACS Applied Materials &Interfaces, 2018, 10(48):41185-41196.