β-烟酰胺单核苷酸(β-nicotinamide mononucleotide,β-NMN)是一种生物活性核苷酸,作为NAD+和NADP+的前体广泛存在于动物体内[1]。NAD+是一种重要的辅因子,在生物系统中发挥着关键作用。NAD+的缺乏会导致阿尔兹海默症、心脑疾病、Ⅱ型糖尿病以及肥胖症等疾病[2-3]。β-NMN作为NAD+的前体物质,与这些疾病有着广泛的联系。2010年以前,NMN主要以单磷酸腺苷、四乙酰核糖和烟酰胺为原料,通过化学合成大规模生产[4-5]。但是由于化学合成法使用大量有机溶剂所带来的环境污染,以及手性异构体分离困难等问题,逐渐被生物合成法所取代[6]。烟酰胺核糖激酶(nicotinamide riboside kinase,Nrk)可以催化烟酰胺核糖(nicotinamide riboside, NR)磷酸化,该方法可在温和的条件下仅需一步酶催化反应生成β-NMN[7]。HE等[8]研究了来源于Saccharomyces cerevisiae的Nrk,并在大肠杆菌中构建其异源表达体系,酶活力可达14.75 U/mL。钟锦潮等[9]在发酵罐中放大培养Nrk生产菌株,并分阶段将OUR、发酵液中ORP和乙酸浓度中的至少一者控制在预定范围内,最终使得其酶活力最高可达30.3 U/mL。因此,通过表达与发酵优化提高Nrk重组菌酶活力十分重要。
核糖体结合位点(ribosome binding site,RBS)是一种重要的表达元件。它与核糖体结合,将其定位到正确的位置,与起始翻译速度和效率息息相关[10]。在原核生物中,RBS序列位于mRNA起始密码子AUG上游约3至10个氨基酸处,常常含有一段与5′-GGAGG-3′类似,富含嘌呤的序列,被称为Shine Dalgarno序列[10]。不同的RBS序列可以调控异源蛋白的表达水平。MAO等[11]通过RBS序列优化,将枯草芽孢杆菌中细胞外几丁二糖脱乙酰酶的活性从3 682.2 U/mL提高至4 807.6 U/mL,是优化前的1.3倍。启动子作为另一重要的表达元件,调控位于基因上游的转录从而达到调控蛋白表达的目的[12]。近年来,许多研究通过启动子工程来提高重组蛋白的表达量[13]。ZHANG等[14]在枯草芽孢杆菌中通过构建PHpaI-PamyQ双启动子表达系统,使得β-环糊精葡萄糖基转移的酶活力从24.2 U/mL提升至30.5 U/mL,是单启动子表达水平的1.26倍。
本研究通过对表达质粒进行筛选,构建了Nrk在大肠杆菌中的异源可溶性表达系统,并针对其表达元件进行适配性优化,进一步提升重组菌的酶活力。同时本文对Nrk的酶学性质进行研究,并在优化诱导发酵体系的基础上对其进行培养条件优化和发酵罐放大产酶研究,以挖掘其工业应用潜力。
1 材料与方法
1.1 材料与试剂
1.1.1 质粒、菌株和引物
菌株Escherichia coli BL21(DE3)、质粒pET-3b、pET-28a(+)、pET-32a(+)、pRSFDuet-1及来源于Kluyveromyces lactis的编码基因Nrk均为本实验室保藏。研究中涉及到的主要引物如表1所示。
表1 本研究所用引物
Table 1 Primers used in this study

引物名称引物序列(5′-3′)pET-28a(+)-Nrk-FGTGGACAGCAAATGGGTCGCGGATCCATGACGACAACTAAAGTTAAATTpET-28a(+)-Nrk-RCAAGCTTGTCGACGGAGCTCGAATTCTTAGTTTGCATCCAAATGGGACpET-3b-Nrk-FAACTTTAAGAAGGAGATATACATATGGAaATGACGACAACTAAAGTTAAApET-3b-Nrk-RTTTCGGGCTTTGTTAGCAGCCGGATCCTTAGTTTGCATCCAAATpET-32a(+)-Nrk-FggccatggctgatatcggatccATGACGACAACTAAAGTTAApET-32a(+)-Nrk-RttgtcgacggagctcgaattcTTAGTTTGCATCCAAATGGGpRSFDuet-1-Nrk-FtcatcaccacagccaggatccgATGACGACAACTAAAGTTAAApRSFDuet-1-Nrk-RtgcggccgcaagcttgtcgacTTAGTTTGCATCCAAATGGGA5000-FCCTTTATTCTTACCAATTCGGAAACTTTTTTTatgggcagcagccatcaccat5000-RAAAAAAAGTTTCCGAATTGGTAAGAATAAAGGattaaagttaaacaaaattat7500-FACTCTAAATTATCCAGGGGAGACGTTCAATATatgggcagcagccatcaccat7500-RATATTGAACGTCTCCCCTGGATAATTTAGAGTattaaagttaaacaaaattattt10000-FGTACAGAAGTAAAACACATCCTTAGGAGAAACTAACGAatgggcagcagccatca10000-RTCGTTAGTTTCTCCTAAGGATGTGTTTTACTTCTGTACattaaagttaaacaaaatta12500-FtAAGTTATTCTAAATAAGGAGGCGCTTAAatgggcagcagccatcacc12500-RTTAAGCGCCTCCTTATTTAGAATAACTTattaaagttaaacaaaatta15000-FAAAGACAATCCAATTCAAACAATATTAAGGAAGCCTTTCatgggcagcagccatca15000-RGAAAGGCTTCCTTAATATTGTTTGAATTGGATTGTCTTTattaaagttaaacaaaatta17500-FAGCCGGACGAATCTCAGGAGGTCTTTTTACatgggcagcagccatcacca17500-RGTAAAAAGACCTCCTGAGATTCGTCCGGCTattaaagttaaacaaaatta20000-FCTCAAAGTAGGAATCGAGACTTAAGGAGCTAACTatgggcagcagccatcacca20000-RAGTTAGCTCCTTAAGTCTCGATTCCTACTTTGAGattaaagttaaacaaaatta25000-FACTACGATTAATCGACAGGAGCCGTATCAAatgggcagcagccatcacca25000-RTTGATACGGCTCCTGTCGATTAATCGTAGTattaaagttaaacaaaatta30000-FACAACTCACTACCATCCCTATCAGGAGCAATTAAatgggcagcagccatcacca30000-RTTAATTGCTCCTGATAGGGATGGTAGTGAGTTGTattaaagttaaacaaaatta35000-FACTTCACGCAATTCAGATAAAGGAGTCTTATatgggcagcagccatcacc35000-RATAAGACTCCTTTATCTGAATTGCGTGAAGTattaaagttaaacaaaattat40000-FCATCGGCGTCACCGACATCCTCAAGACATCATAAGGAGGGTTAAatgggcagc40000-RTTAACCCTCCTTATGATGTCTTGAGGATGTCGGTGACGCCGATGattaaagttaaa80000-FCGACAGCACGACATCAACCCACTAAGGAGCGTTTTatgggcagcagccatcacca80000-RAAAACGCTCCTTAGTGGGTTGATGTCGTGCTGTCGattaaagttaaacaaaatta100000-FCTAAATTACACCGTCCCTCGAATAAGTAAGGAGGTCATAatgggcagcagccatcac100000-RTATGACCTCCTTACTTATTCGAGGGACGGTGTAATTTAGattaaagttaaacaaaattaPaceE-T7-RtatagtgagtcgtattagggttattccttatctatPaceE-T7-FatgcgactcctgcattaggaaatttatccagaagatgttgtaaatPalsR-T7-FtgcattaggaaatTCATCTTCCCCGTCCGTGCAAAACPalsR-T7-RtatagtgagtcgtattaCACGTTCTCTTGTCCTTGAAPdnaKJ-T7-RcgactcctgcattaggaaataaggattctcttagtgggaagPdnaKJ-T7-FtatagtgagtcgtattactaaacgtctccactatatattcggtcatcatPgapA-T7-FctcctgcattaggaaatttgctcacatctcactttaatcgtgPgapA-T7-RtatagtgagtcgtattaatattccaccagctatttgttPskp-T7-RgactcctgcattaggaaattggtagtgtagcgatgactPskp-T7-FtatagtgagtcgtattaaataaactccttaccatcccatttgcPssrA-T7-FgactcctgcattaggaaatccagttcctcaccgcgcctcPssrA-T7-Rtatagtgagtcgtattaaatgtgtaaaggtaagt
1.1.2 主要试剂
胰蛋白胨、酵母粉,Thermo Fisher 公司;NaCl、琼脂粉、琼脂糖、Na2HPO4、NaH2PO4、K2HPO4、KH2PO4,国药集团化学试剂有限公司;氨苄青霉素,上海生工生物工程有限公司。
1.1.3 培养基与缓冲液
LB培养基(g/L):蛋白胨10、NaCl 10、酵母粉5,用于重组E.coli BL21(DE3)在摇瓶中进行发酵培养;
发酵培养基(g/L):蛋白胨20、NaCl 0.5、酵母粉5、KCl 0.19、MgCl2 0.95、葡萄糖10、微量元素2 mL/L,调节pH值至7.0,用于重组E.coli BL21 (DE3)在发酵罐中进行放大产酶研究;
微量元素(g/L):七水合硫酸锌0.58、二水合氯化铜0.2、乙二胺四乙酸0.5、六水合氯化铁6;
补料培养基(g/L):葡萄糖700、硫酸镁14、微量元素14 mL/L,用于补料发酵中重组E.coli BL21(DE3)的培养。
PBS缓冲液(g/L):NaCl 8、KCl 0.2、Na2HPO4 1.42、KH2PO4 0.27,调节pH值为7.0。
1.2 仪器与设备
回旋式摇床,太仓市强乐实验设备有限公司;凝胶成像仪,美国UVP公司;高效液相色谱仪,安捷伦公司;超声破碎仪,宁波新芝生物科技股份有限公司;低温高速离心机,Eppendorf公司;生化培养箱,上海博迅实业有限公司;核酸电泳,COSMO BIO公司;金属浴,杭州奥盛仪器有限公司;pH计,METTLER TOLEDO公司;酶标仪,Bio-Rad公司。
1.3 实验方法
1.3.1 烟酰胺核糖激酶异源表达载体构建
以本实验室保存的基因片段作为模板,对Nrk基因进行PCR扩增,反应条件为:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸5 min,34个循环;72 ℃终延伸60 min。利用在snapgene上选择的酶切位点,对空载质粒进行双酶切,酶切温度37 ℃,时间1 h。将酶切产物和PCR产物分别利用电泳仪进行核酸电泳,并利用胶回收试剂盒对其进行回收。将回收产物构建同源重组体系(表2),利用水浴锅进行反应,温度37 ℃,时间1 h,得到同源重组后的新质粒。
表2 同源重组体系
Table 2 Homologous recombination system

同源重组体系体积/μL质粒X目的基因Y5×CEⅡ Buffer4Exnase Ⅱ2ddH2O20
注:X=(0.02×克隆载体碱基对数) ng;Y=(0.04×插入片段碱基对数) ng。
利用热激法[15]将质粒转化至菌株E.coli BL21 (DE3)中,涂布后,在培养箱中37 ℃倒置培养12 h。
1.3.2 发酵培养及酶活力测定
将得到的阳性转化子接种于10 mL含50 μg/mL卡那霉素的LB培养基培养10 h。之后,以1%(体积分数)接种于30 mL LB培养基中,37 ℃,220 r/min培养至OD600值为0.6~0.8后加入一定量的异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)。发酵12 h后,将所得菌液4 ℃离心。向沉淀中加入10 mL PBS溶液,充分振荡后重悬,重复2次。用超声破碎仪破碎菌液50 min,结束后离心10 min,保留上清液。
配制1.2 mol/L NR,1.2 mol/L ATP,0.6 mol/L MgCl2,各取50 μL,加入750 μL ddH2O和50 μL粗酶液,37 ℃,1 500 r/min振荡,反应10 min。反应结束后加入100 μL 25% HCl溶液终止反应。通过高效液相色谱检测NMN含量[16],采用Dikma Technology(中国北京)C18色谱柱,以含0.06%四丁基氢氧化铵的水溶液及甲醇为流动相进行梯度洗脱,在254 nm波长处检测色谱峰,并与NR和NMN标准品的色谱峰检测保留时间进行对比,计算样品中NMN的含量。一个单位的NRK活性定义为每分钟催化NR生成1 μmol β-NMN的酶量,用U/mL表示。
1.3.3 RBS序列的设计与替换
利用RBS Calculator(https://www.denovodna.com/software/predict_rbs_calculator)对pRSFDuet-Nrk自带的RBS序列进行翻译速率的计算,得到翻译起始速率分别为5 000、7 500、10 000、12 500、15 000、17 500、20 000、25 000、30 000、35 000、40 000、80 000、100 000 a.u.的13条不同的RBS序列。以pRSFDuet-Nrk为模板,按照表1中的引物利用反向PCR替换质粒中原始的序列,构建含有不同RBS序列的工程菌。
1.3.4 双启动子工程菌的构建
利用表1中的引物从大肠杆菌基因组中通过PCR分别获得PdnaKJ、PalsR、PssrA、Pskp、PgapA、PaceE启动子,经同源重组将其连接到1.3.3节中得到的质粒上,并转化至大肠杆菌中,测序后选择正确的重组菌株进行发酵培养和酶活力测定。
1.3.5 酶学性质表征
1.3.5.1 酶蛋白的纯化
离心发酵结束后的菌体,并用PBS缓冲液重悬,重复2次。将所得菌液经超声破碎后离心,收集上清液并通过Ni2+亲和层析进行分离纯化,通过SDS-PAGE验证酶液纯度,通过Bradford法测定蛋白浓度。
1.3.5.2 温度、pH和Mg2+浓度对酶活力的影响
分别在30、37、45、50、55、60 ℃下进行酶活力测定,将50 ℃下纯酶的酶活力设定为100%,计算不同温度下的相对酶活力;配制不同pH的PBS缓冲液(pH 6.0~9.0)作为介质考察pH对催化反应的影响,将pH 7.0时下纯酶的酶活力设定为100%,分别计算不同pH下的相对酶活力;Nrk催化的磷酸化反应需要Mg2+参与才能进行。控制反应体系中的Mg2+浓度为1、5、10、15、30 mmol/L,计算不同条件下纯酶的酶活力,将10 mmol/L时下纯酶的酶活力设定为100%,分别计算其相对酶活力。
1.3.6 诱导表达条件的优化
为了研究重组菌最佳的产酶条件,本研究对诱导温度和诱导剂浓度进行了优化。在如1.3.2节所述的发酵过程中,当菌体浓度达到OD600值为0.6~0.8时,向其中添加0.5 mmol/L诱导剂,分别将其在16、20、25、30、37 ℃培养12 h后测定其酶活力,并将25 ℃时下纯酶的酶活力设定为100%,计算其相对酶活力。类似的,在如1.3.2节所述的发酵过程中,当菌体浓度达到OD600为0.6~0.8时,分别加入终浓度为0.1、0.3、0.5、0.8、1.0 mmol/L的IPTG,在25 ℃培养12 h后测定其酶活力,并以添加0.5 mmol/L IPTG时的酶活力为对照,设置为100%,分别计算其相对酶活力。
1.3.7 发酵罐放大培养体系
基于上述优化的最佳培养条件,在5 L发酵罐中,对重组菌进行分批发酵和补料发酵的放大工艺研究。培养基选择1.1.3节中所述的发酵培养基和补料培养基,在发酵过程中,诱导后每隔2 h取样,通过测定OD600和酶活力以检测其生长和产酶情况。
1.4 数据分析
上述所有的催化反应设置3个平行样进行HPLC检测,以计算得到3个样品中的Nrk酶活力的平均值作为最终的数据,其标准偏差通过误差棒进行展示。
2 结果与分析
2.1 Nrk的高可溶性异源表达体系构建
为了实现Nrk基因在大肠杆菌中的异源表达,首先利用质粒pET-3b对其进行重组表达,得到重组质粒pET-3b-Nrk。测序后将其转化至BL21 (DE3)宿主中,成功构建了重组菌E.coli BL21 (DE3)/pET-3b-Nrk。接种并发酵菌株,并对其胞内蛋白与破碎沉淀进行SDS-PAGE分析。结果如图1所示,超声破碎后,其上清液在27.3 kDa左右处有特异条带,但条带较细,破碎沉淀在27.3 kDa有明显的特异条带,表明其可溶性表达水平较低(图1-d)。因此继续更换质粒以实现Nrk的高效可溶性表达。

a-质粒构建示意图;b-Nrk在不同质粒中表达的可溶性蛋白的SDS-PAGE分析图谱(M:蛋白Marker,1:pET-32a(+),2:pET-28a(+),3:pRSFDuet-1,4:pET-3b);c-Nrk在不同质粒中表达的非可溶性蛋白的SDS-PAGE(M:蛋白Marker,1:pET-3b,2:pET-32a(+),3:pET-28a(+),4:pRSFDuet-1);d-Nrk通过不同载体表达后的粗酶液催化活性
图1 Nrk在不同质粒中的异源表达
Fig.1 Heterologous expression of Nrk in different vectors
不同拷贝数的质粒会导致重组蛋白的表达量差异,在异源蛋白的表达体系中,需要找到菌体生长和产物形成的良好平衡,以实现高表达效率[17]。在该研究中分别选择了不同拷贝数的质粒,构建了Nrk的异源表达体系。将该基因片段分别连接至含有诱导型T7启动子的载体pET-28a、pET-32a、pRSFDuet-1上,得到重组质粒pET-28a-Nrk、pET-32a-Nrk、pRSFDuet-Nrk。将验证正确的质粒热激转化到大肠杆菌E.coli BL21 (DE3)宿主中,成功构建了重组菌E.coli BL21 (DE3)/pET-28a-Nrk、E.coli BL21 (DE3)/pET-32a-Nrk、 E.coli BL21 (DE3)/pRSFDuet-Nrk。接种并发酵重组菌株,超声破碎后通过SDS-PAGE检测重组蛋白在上清液中的表达水平,并观察不同菌株的破碎沉淀。图1-b表明,不同重组菌的上清液均在27.3 kDa左右处有特异条带,而重组菌株E.coli BL21 (DE3)/pRSFDuet-Nrk的上清液中Nrk条带较粗,且沉淀中的Nrk条带较细,表明E.coli BL21 (DE3)/pRSFDuet-Nrk的Nrk可溶性表达水平较高,且经过酶活力测定,其粗酶液具有最高的催化活性,酶活力为2.14 U/mL(图1-d),因此选择该质粒进行后续研究。
2.2 Nrk的表达元件优化
2.2.1 不同翻译速率的RBS序列对Nrk酶活力的影响
起始阶段是翻译过程中的关键限速部分,不同的RBS由于强度差异可以在很广泛的范围内控制翻译效率,因此RBS是确定蛋白表达准确性和整体效率的重要控制元件[18-19]。本实验利用网站RBS Calculator对E.coli BL21 (DE3)/pRSFDuet-Nrk自身携带的RBS序列进行翻译起始速率的预测,并以此为模板,设计了13个不同速率的RBS序列,调控Nrk的表达。以初始菌株E.coli BL21 (DE3)/pRSFDuet-Nrk为模板,构建了翻译起始速率为5 000~100 000 a.u.的RBS序列的重组菌,并分别命名为R1~R13。将其发酵培养12 h后,分别测定胞内Nrk的酶活力,结果如图2所示。结果表明,翻译起始速率为35 000 a.u.的RBS序列最适合Nrk的表达,酶活力较对照菌E.coli BL21 (DE3)/pRSFDuet-Nrk提升最多,达到4.28 U/mL,是对照菌株的1.5倍,将其命名为35000-Nrk。其次是含有翻译起始速率为40 000 a.u.的RBS序列的重组菌株,是对照菌株的1.3倍。随着翻译起始速率的逐渐增加,重组菌株的酶活力未持续增长,这说明了Nrk酶活力与RBS强度不呈线性关系。含有不同翻译起始速率RBS序列的重组菌株具有相似的生长情况,这说明Nrk在胞内的积累不影响E.coli BL21 (DE3)的生长。
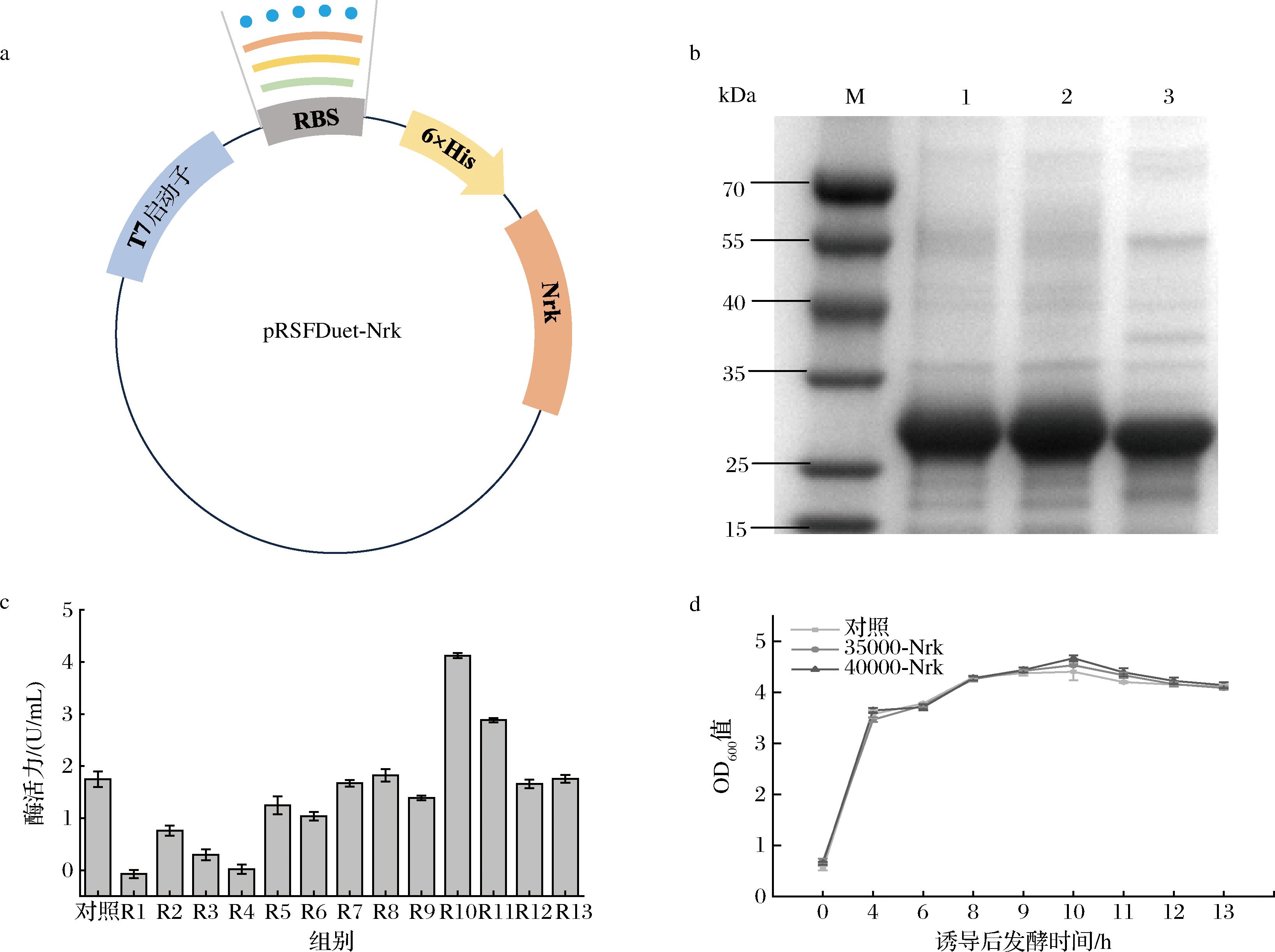
a-RBS适配性优化示意图;b-不同RBS重组菌中Nrk的蛋白表达(M:蛋白Marker,1:40000-Nrk,2:35000-Nrk,3:初始菌株);c-不同RBS重组菌Nrk发酵液的酶活力;d-35000-Nrk、40000-Nrk与初始菌株的生长曲线
图2 不同RBS序列对重组菌表达Nrk的影响
Fig.2 The influence of different RBS sequences on the expression of Nrk in recombinant strains
2.2.2 启动子工程对酶活力的影响
本研究从大肠杆菌基因组中选择了6种不同的启动子PdnaKJ、PalsR、PssrA、Pskp、PgapA、PaceE,分别与T7启动子进行串联,以经过RBS适配性优化后得到的35 000-Nrk菌株为模板,构建重组菌株,并将其命名为P1~P6。发酵培养12 h后,检测其酶活力,结果如图3所示。结果表明,PdnaKJ、PssrA、PgapA和Pskp启动子与T7串联后,重组菌酶活力明显降低,其中PgapA和Pskp与T7启动子串联导致Nrk基本失去活性。而含双启动子PaceE-T7、PalsR-T7的重组菌株表现出较高的催化活性(图3-b),其中PalsR与T7启动子串联后得到的菌株,酶活力提升最为明显,约为5.56 U/mL,较35000-Nrk提升30%,将其命名为35000-PalsR-Nrk。
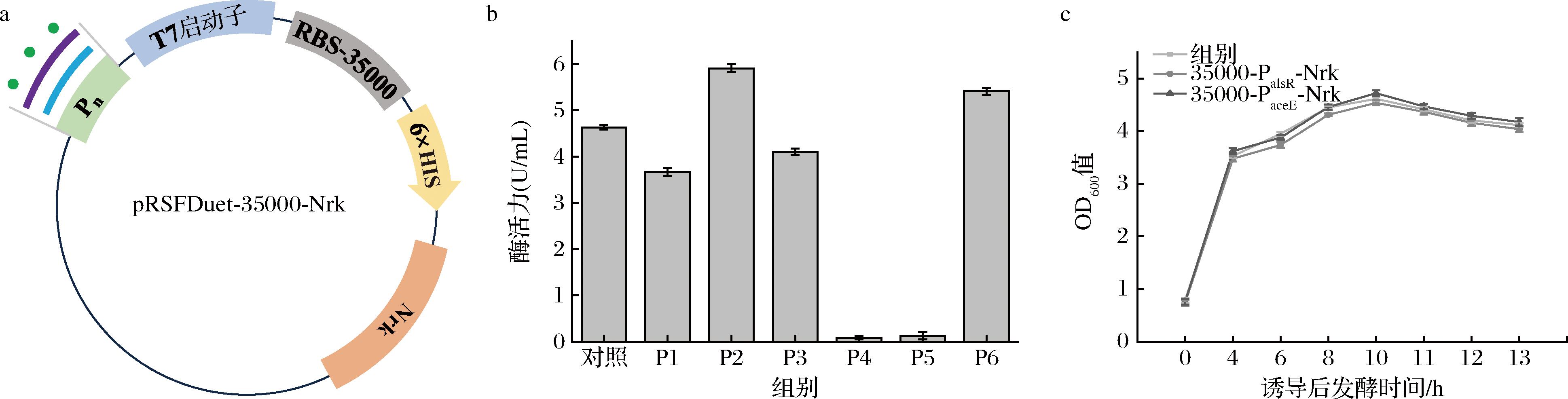
a-启动子优化示意图;b-含不同启动子重组菌中Nrk发酵液的酶活力;c-PaceE-T7、PalsR-T7和初始菌株的生长曲线
图3 串联启动子对Nrk表达的影响
Fig.3 The impact of tandem promoters on Nrk expression
比较对照菌株的生长曲线(图3-c),仅在生长后期略有差异。因此,PaceE、PalsR的引入并不会对菌株生长带来明显的影响。
2.3 生物转化体系优化和酶学性质研究
通过Ni2+亲和层析获得纯化后的Nrk,并对其最适反应温度和pH进行探究。如图4所示,反应温度从37 ℃升高至50 ℃,Nrk的酶活力随着温度升高而增加,并在50 ℃达到最高,随后下降。该酶在50 ℃下的酶活力约为37 ℃的1.6倍。这可能是由于温度越高,分子布朗运动越剧烈,从而有利于反应的进行。温度继续升高,其酶活力骤降,可能是由于酶在高温条件下结构受到了热变性破坏,因此选择50 ℃作为Nrk的后续反应温度。
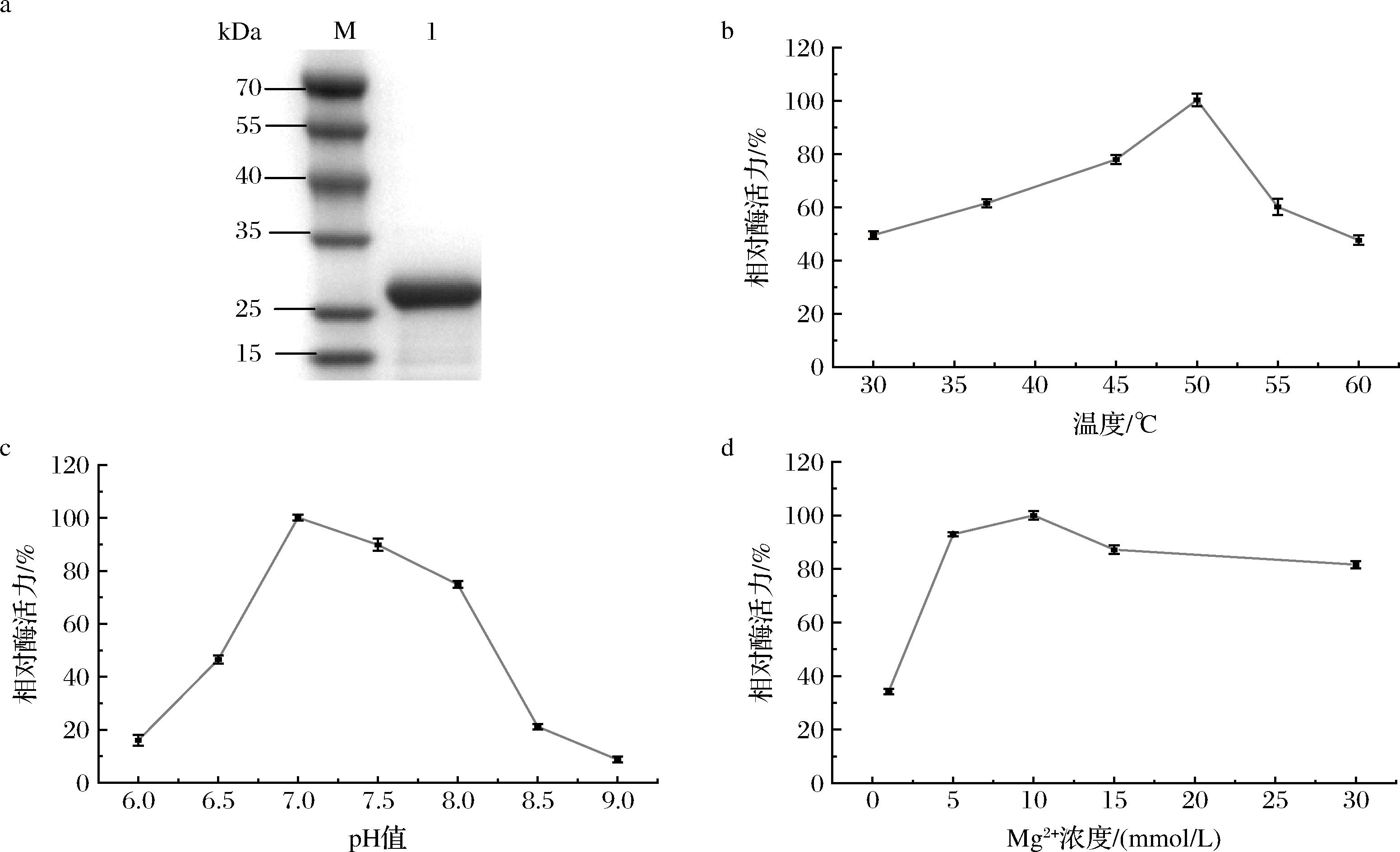
a-纯化Nrk的SDS-PAGE;b-Nrk在不同温度下的相对酶活力;c-Nrk在不同pH下的相对酶活力;d-Nrk在不同Mg2+浓度下的相对酶活力
图4 Nrk的酶学性质研究
Fig.4 Investigation of the enzymatic properties of Nrk
图4-c展示了Nrk在不同pH的反应体系下酶活力的变化。随着pH由6.0逐渐升高至7.0,其酶活力也逐渐升高,并在pH 7.0时达到最高,表明Nrk的最适反应pH约为7.0。在pH为6.0的环境中,该酶只能保留约10%的活性。当pH超过7.0并持续升高时,酶活力迅速降低,当pH升高至9.0时,该酶仅保留约8.7%的活性。
金属离子在催化反应中常与酶分子结合,通过不同的方式参与反应[20]。Nrk催化的磷酸化反应需要Mg2+参与,作为该酶的激活剂,Mg2+与反应的另一底物ATP形成螯合物作为该酶的实际底物。因此,本研究通过调整Mg2+添加浓度来优化催化体系。如图4-d所示,Mg2+浓度过高或过低都会对反应带来不利影响,当其浓度为10 mmol/L时,可以使Nrk达到最高酶活力。当Mg2+浓度从10 mmol/L升高至30 mmol/L时,其酶活力呈现下降趋势。
2.4 发酵条件优化及罐上放大培养
2.4.1 诱导剂浓度对重组菌产酶的影响
实验中构建的重组菌E.coli BL21 (DE3)/35000-PalsR-Nrk需要IPTG进行诱导表达,因此本文探究了不同浓度的IPTG对该菌株产酶的影响。
图5-a展示了经不同浓度的IPTG诱导后重组菌的酶活。随着IPTG浓度从0 mmol/L逐渐增加至0.5 mmol/L,重组菌的活性也不断提高,当诱导剂的浓度继续增加,酶活力呈现下降的趋势,这说明过高浓度的诱导剂不利于该菌株的产酶。这是因为过高浓度的IPTG对菌体具有一定毒性,而过低的IPTG浓度可能会导致蛋白的表达水平过低[21-22]。

a-诱导剂浓度对E.coli BL21 (DE3) 35000-PalsR-Nrk发酵液酶活力的影响;b-诱导时间对35000- PalsR -Nrk发酵液酶活力的影响;c-35000- PalsR -Nrk分批发酵菌液浓度及酶活力检测;d-35000- PalsR -Nrk补料发酵菌液浓度及酶活力检测
图5 E.coli BL21 (DE3)/35000-PalsR-Nrk发酵条件优化及罐上放大培养
Fig.5 Optimization of E.coli BL21 (DE3)/35000-PalsR-Nrk fermentation conditions and scale-up fermentation
2.4.2 诱导温度对重组菌产酶的影响
诱导温度影响蛋白的表达,较高的温度会使菌株生长速度过快,并加快蛋白质的合成速度,但可能会导致包涵体的积累[23]。为了实现该酶高效的可溶性表达,本研究在不同温度下对其进行培养,并检测其酶活力水平。
本研究分别在16、20、25、30、37 ℃下诱导E.coli BL21 (DE3)/35000-PalsR-Nrk培养12 h,在发酵结束后测定其酶活力。结果如图5-b所示,随着温度不断升高,重组菌的酶活力呈现增长的趋势,当诱导温度为25 ℃时,酶活力最高,这可能是由于随着温度的升高Nrk的表达量增加。当温度继续上升,酶活力下降,这可能是温度过高使蛋白质发生错误折叠或部分变性导致的。因此选用25 ℃用作后续诱导温度。
2.4.3 重组菌罐上产酶放大工艺研究
本研究应用5 L发酵罐以上述的最优培养条件对重组菌E.coli BL21 (DE3)/35000-PalsR-Nrk进行分批发酵,以进一步提高Nrk的酶活力。以7.5%(体积分数)的接种量将其接入发酵罐培养基中,37 ℃下培养至OD600值达到4时,降温至25 ℃,并加入0.5 mmol/L的IPTG进行诱导表达。在分批发酵的过程中,控制培养pH在7.0左右,并控制发酵罐转速为400 r/min。约3 h后OD600值达到4,进行诱导。诱导后12 h,菌体浓度持续增长,葡萄糖被快速消耗,12 h后OD600值最后平稳于25左右,此时,葡萄糖已全部消耗完毕。发酵过程中定期取样检测酶活力,重组菌E.coli BL21 (DE3)/35000-PalsR-Nrk经过分批发酵培养,酶活力最高可达到23.6 U/mL,与摇瓶水平相比提升3.2倍。
分批发酵中提供的营养物质有限,因此采取补料发酵以进一步探究重组菌在工业生产中的潜力。以相同的接种量和培养温度培养该重组菌,当OD600值达到约10时,加入0.5 mmol/L的IPTG进行诱导表达。随着菌体不断生长,葡萄糖被逐步消耗,当其浓度低于2 g/L时,进行流加补料。经过补料发酵,重组菌35000-PalsR-Nrk的OD600值在18 h最高可达到62.2,此时的酶活力也达到最高72.33 U/mL,达到摇瓶水平的13.1倍,超过目前专利报道的罐上最高水平30.3 U/mL[9]。发酵后期,随着时间延长,菌体浓度和酶活力都呈下降趋势。高密度发酵使得Nrk重组菌的酶活力呈现明显提升,充分挖掘了其在催化反应中的潜力,为β-NMN的合成提供了高酶活性的Nrk重组菌株。
3 结论
补充β-NMN被视为提高NAD+水平的有效方法[24],本研究通过筛选不同载体,实现了烟酰胺核糖激酶在大肠杆菌中的可溶异源表达,初始酶活力为2.14 U/mL。通过RBS适配性优化和启动子串联,得到了工程菌E.coli BL21 (DE3)/35000-PalsR-Nrk,使得Nrk的表达水平明显提升,酶活力提升至初始水平的2.6倍。随后对其进行发酵条件优化及高密度发酵,使最终酶活力达到72.33 U/mL,是摇瓶水平的13.1倍。Nrk的酶学性质进行表明其最适反应条件为pH 7.0、50 ℃和10 mmol/L Mg2+。以上研究结果探究了该重组菌表达Nrk的潜力,为生物酶法合成β-NMN奠定了坚实基础。
[1] SHADE C.The science behind NMN-A stable, reliable NAD+activator and anti-aging molecule[J].Integrative Medicine (Encinitas), 2020, 19(1):12-14.
[2] YOSHINO J, BAUR J A, IMAI S I.NAD+ intermediates:The biology and therapeutic potential of NMN and NR[J].Cell Metabolism, 2018, 27(3):513-528.
[3] WANG X S, HE H J, XIONG X, et al.NAD+ in Alzheimer′s disease:Molecular mechanisms and systematic therapeutic evidence obtained in vivo[J].Frontiers in Cell and Developmental Biology, 2021, 9:668491.
[4] MARINESCU G C, POPESCU R G, STOIAN G, et al.β-nicotinamide mononucleotide (NMN) production in Escherichia coli[J].Scientific Reports, 2018, 8(1):12278.
[5] SUGIYAMA K, IIJIMA K, YOSHINO M, et al.Nicotinamide mononucleotide production by fructophilic lactic acid bacteria[J].Scientific Reports, 2021, 11(1):7662.
[6] LEE J, CHURCHIL H, CHOI W B, et al.A chemical synthesis of nicotinamide adenine dinucleotide (NAD+)[J].Chemical Communications, 1999(8):729-730.
[7] HUANG Z S, LI N, YU S Q, et al.Systematic engineering of Escherichia coli for efficient production of nicotinamide mononucleotide from nicotinamide[J].ACS Synthetic Biology, 2022, 11(9):2979-2988.
[8] HE J J, LIU X X, LI Y, et al.High level expression of nicotinamide nucleoside kinase from Saccharomyces cerevisiae and its purification and immobilization by one-step method[J].Frontiers in Bioengineering and Biotechnology, 2023, 11:1134152.
[9] 钟锦潮, 陈必钦, 严必能, 等.一种烟酰胺核糖激酶及烟酰胺单核苷酸的合成方法:中国, CN112522232A[P].2020-12-07.
ZHONG J C, CHEN B Q, YAN B N, et al.A type of nicotinamide riboside kinase and a method for synthesizing nicotinamide mononucleotide:China, CN112522232A[P].2020-12-07.
[10] CHEN Y L, WEN J D.Translation initiation site of mRNA is selected through dynamic interaction with the ribosome[J].Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(22):e2118099119.
[11] MAO X Z, HUANG Z Y, SUN G Y, et al.High level production of diacetylchitobiose deacetylase by refactoring genetic elements and cellular metabolism[J].Bioresource Technology 2021, 341:125836.
[12] XIAO S, SHILOACH J, BETENBAUGH M J.Engineering cells to improve protein expression[J].Current Opinion in Structural Biology, 2014, 26:32-38.
[13] FIRAT DUZENLI O, OKAY S.Promoter engineering for the recombinant protein production in prokaryotic systems[J].AIMS Bioengineering, 2020, 7(2):62-81.
[14] ZHANG K, SU L Q, DUAN X G, et al.High-level extracellular protein production in Bacillus subtilis using an optimized dual-promoter expression system[J].Microbial Cell Factories, 2017, 16(1):32.
[15] FROGER A, HALL J E.Transformation of plasmid DNA into E.coli using the heat shock method[J].Journal of Visualized Experiments, 2007(6):253.
[16] HE Z H, YANG X S, TIAN X, et al.Yeast cell surface engineering of a nicotinamide riboside kinase for the production of β-nicotinamide mononucleotide via whole-cell catalysis[J].ACS Synthetic Biology, 2022, 11(10):3451-3459.
[17] ROSANO G L, CECCARELLI E A.Recombinant protein expression in Escherichia coli:Advances and challenges[J].Frontiers In Microbiology, 2014, 5:172.
[18] THIEL K, MULAKU E, DANDAPANI H, et al.Translation efficiency of heterologous proteins is significantly affected by the genetic context of RBS sequences in engineered Cyanobacterium Synechocystis sp.PCC 6803[J].Microbial Cell Factories, 2018, 17(1):34.
[19] RODNINA M V.The ribosome in action:Tuning of translational efficiency and protein folding[J].Protein Science, 2016, 25(8):1390-1406.
[20] RIORDAN J F.The role of metals in enzyme activity[J].Annals of Clinical and Laboratory Science, 1977, 7(2):119-129.
[21] DVORAK P, CHRAST L, NIKEL P I, et al.Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway[J].Microbial Cell Factories, 2015, 14:201.
[22] MüHLMANN M, FORSTEN E, NOACK S, et al.Optimizing recombinant protein expression via automated induction profiling in microtiter plates at different temperatures[J].Microbial Cell Factories, 2017, 16(1):220.
[23] SAN-MIGUEL T, PéREZ-BERMúDEZ P, GAVIDIA I.Production of soluble eukaryotic recombinant proteins in E.coli is favoured in early log-phase cultures induced at low temperature[J].Springerplus, 2013, 2(1):89.
[24] LIU Y, GONG J S, MARSHALL G, et al.Protein engineering of NADH pyrophosphatase for efficient biocatalytic production of reduced nicotinamide mononucleotide[J].Frontiers in Bioengineering and Biotechnology, 2023, 11:1159965.