谷胱甘肽是生物体内一种重要的调节代谢物质,含有活性基团巯基,可参与体内多种生化反应[1]。谷胱甘肽有还原型(glutathione,GSH)和氧化型(oxidized glutathione,GSSG)2种形式,在生理条件下,还原型占绝大多数[2]。谷胱甘肽还原酶可将GSH转化为GSSG,该酶还可以为磷酸戊糖旁路代谢提供NADPH[3-4]。谷胱甘肽是一种重要的抗氧化剂,可保证体内蛋白质和酶等分子发挥正常的生理功能,起到增强免疫力、延缓衰老、抗肿瘤等作用[5-7]。谷胱甘肽可与某些药物、毒物等结合,具有整合解毒作用[8-9]。谷胱甘肽在药物、食品和化妆品添加剂等领域中具有广泛的应用[10-11]。
目前GSH的生物合成主要通过两步依赖于ATP的酶催化反应进行[12],首先是L-谷氨酸和L-半胱氨酸在γ-谷氨酰半胱氨酸合成酶(γ-glutamylcysteine synthetase,GSHA)的催化下生成γ-谷氨酰半胱氨酸,然后与甘氨酸经谷胱甘肽合成酶(glutathione synthetase,GSHB)催化反应生成GSH[13-14]。研究表明,产物GSH的积累对GSHA的酶活性具有反馈抑制作用,即GSHA为其生物合成过程中的限速酶[15-16]。目前天然存在的GSHA催化活性较低,不满足实际生产的需求,因此对天然酶进行分子改造,以实现高效催化GSH的生物合成。ZHANG等[13]选取来自血链球菌的双功能谷胱甘肽合成酶,进行双酶级联生产谷胱甘肽,在一锅中合成了8.8 g/L的GSH,转化率达到了81.4%。
本研究通过定点突变策略对GSHA进行分子改造。首先通过Pymol对GSHA与过渡态类似物N-磷酰亚砜亚胺(N-phosphorylsulfoxide,P2S)进行可视化,对关键的氨基酸结合位点进行分析[17]。通过丙氨酸扫描对蛋白质结构中存在氢键作用的氨基酸进行替换,以缓解GSH的产物抑制。其次使用Pymol软件分析GSHA的表面电荷,选取位于蛋白表面带正电或不带电的非保守氨基酸残基,突变为带负电的氨基酸残基,以提高GSHA的活性及稳定性。综合以上方法进行关键突变位点筛选,对能够提升活性的关键位点进行定点突变,以及组合突变,最终获得了酶活力提高的突变菌株,相比于野生型酶活提高了6倍,并对突变体的酶学性质进行了分析。将筛选出的最佳突变体应用于谷胱甘肽的合成中,最终的GSH产量为76.8 mmol/L,转化率达到了96%,实现了高效生物合成GSH的目的。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本研究所用大肠杆菌来源的GSHA基因的氨基酸序列来自国家生物技术信息中心(National Center for Biotechnology Information,NCBI),表达宿主菌均为大肠杆菌Escherichia coli BL21(DE3)、克隆宿主菌为E.coli BL21(DE3),质粒为pET28a,重组质粒pET28a-GSHA-WT为本实验室构建和保存。
1.1.2 培养基
LB培养基(g/L):蛋白胨10、酵母提取物5、氯化钠10。
TB培养基(g/L):蛋白胨12、酵母提取物24、甘油5、磷酸二氢钾17 mmol/L、磷酸氢二钾72 mmol/L。
固体培养基:于液体LB培养基中添加20 g/L的琼脂粉。
1.1.3 试剂
蛋白胨、酵母提取物,英国OXOID公司;PrimeSTAR DNA Polymerase、DNA聚合酶rTaq、Dpn I限制性内切酶、Agarose Gel DNA Purification Kit,TaKaRa(大连)有限公司;质粒抽提试剂盒,Axygen(杭州)有限公司;卡那霉素、异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG),生工(上海)股份有限公司;其他试剂,国药集团化学试剂有限公司。
1.2 实验方法
1.2.1 突变位点的选择
为分析GSHA的蛋白质结构,以研究其三维结构及关键活性位点,在PDB蛋白数据库中检索GSHA的三维结构模型。使用Pymol软件对GSHA与过渡态类似物P2S进行可视化,分析在活性中心区域参与底物识别与催化的氨基酸残基[18]。选择存在氢键作用的氨基酸残基,通过丙氨酸扫描对关键氨基酸进行丙氨酸替换,以提高酶的催化活性[19-20]。另外,在蛋白质稳定性改造中,常通过在蛋白表面的非保守区域内,将一些带正电荷或不带电荷的氨基酸突变为带负电荷的氨基酸,从而提高酶的稳定性,使用ProteinTools在线蛋白结构分析网站对表面电荷进行分析,引入负电荷氨基酸来提高酶的稳定性[21-22]。
1.2.2 原核表达载体的构建
设计含有突变位点氨基酸的两条互补引物,构建单点突变体,并在单点突变的基础上进行组合突变,以实验室前期构建的pET28a-GSHA-WT质粒为模板,通过全质粒PCR引入突变位点,PCR产物通过琼脂糖核酸凝胶电泳进行验证。将扩增后的质粒用Dpn I酶进行消化,消化产物通过热激法转化到E.coli BL21(DE3)感受态中,涂布于含卡那霉素抗性的平板上,挑取单菌落过夜培养,提取质粒进行测序验证,并在大肠杆菌中表达培养。本研究所用引物及测序均由通用生物(安徽)股份有限公司完成(表1)。
表1 本研究使用的引物
Table 1 Primers used in this study

序号引物名称引物序列(5′-3′)1E27A-FCAGCGTGGGCTGGCACGAAATTTGCE27A-RGCAAAGTTTCGCGTGCCAGCCCACGCTG2E29A-FGTGGGCTGGAGCGCGCAACTTTGCGTGTTAATGE29A-RCATTAACACGCAAAGTTGCGCGCTCCAGCCCAC3D60A-FGGATTACTACCGCATTTGCGGAAGCATTGD60A-RCAATGCTTCCGCAAATGCGGTAGTAATCC4E67A-FGGAAGCATTGCTGGCATTCATTACACCAGE67A-RCTGGTGTAATGAATGCCAGCAATGCTTCC5Y131A-FGCTTTAAAACGCTGGCACGTGAAGGGCTGY131A-RCAGCCCTTCACGTGCCAGCGTTTTAAAGC6I146A-FGCTGATGCAAACCGCATCCGGCGTGCACTACI146A-RGTAGTGCACGCCGGATGCGGTTTGCATCAGC7H150A-FCATTTCCGGCGTGGCATACAATTTCTCH150A-RGAGAAATTGTATGCCACGCCGGAAATG8R235A-FGCGACCTCTCTTGCATTGAGCGATCR235A-RGATCGCTCAATGCAAGAGAGGTCGC9Y241A-FGAGCGATCTCGGCGCAACCAATAAATCGCY241A-RGCGATTTATTGGTTGCGCCGAGATCGCTC10Y300A-FGATTGAAAACGAACTGGCAGCGCCGATTCGTCY300A-RGACGAATCGGCGCTGCCAGTTCGTTTTCAATC11R330A-FGAATATATTGAAGTGGCATCGCTGGACATCR330A-RGATGTCCAGCGATGCCACTTCAATATATTC12A109E-FCATGCTACATCGAGGAAGGTCAGGACA109E-RGTCCTGACCTTCCTCGATGTAGCATG13G111E-FCATCGCAGAAGAGCAGGACATCGAACG111E-RGTTCGATGTCCTGCTCTTCTGCGATG14A170E-FGATATCTCGGGCGAGGATGCCAAAGAGA170E-RCTCTTTGGCATCCTCGCCCGAGATATC15A172D-FGGCGCTGATGACAAAGAGAAAATTTCA172D-RGAAATTTTCTCTTTGTCATCAGCGCC16S368D-FGAAATGAGCAGTAGCGCACTTGCCTGTACACS368D-RGTGTACAGGCAAGTGCGCTACTGCTCATTTC17A511D-FGATACCGAACCGTTTGATGTGTGGCTGGA511D-RCCAGCCACACATCAAACGGTTCGGTATC
1.2.3 突变体的表达及培养
将测序正确的GSHA突变菌株,接种于含50 μg/mL卡那霉素的LB培养基中,在37 ℃、200 r/min的振荡培养箱中培养12 h后,将种子液以1%的接种量转接入TB培养基中,于37 ℃、200 r/min振荡培养至OD600为0.6~0.8后,加入终浓度为0.2 mmol/L的IPTG进行诱导,在28 ℃、150 r/min条件下培养12 h后,离心收集菌体,放入-80 ℃冰箱保存。
1.2.4 酶活力的测定
采用四氧嘧啶法测定酶活力[23],测定体系为:100 mmol/L Tris-HCl buffer(pH 7.0)、90 mmol/L L-Glu、90 mmol/L Gly、80 mmol/L L-Cys、180 mmol/L ATP、40 mmol/L MgCl2,在37 ℃预热3 min,在10 mL反应体系中加入300 μL GSHA粗酶液(100 g/L)和过量的GSHB粗酶液,最后加入1 g/L的四氧嘧啶。在37 ℃反应30 min后,放入沸水中3 min终止反应,离心取上清液,在305 nm处测定吸光度。一个酶活力单位(U)定义为每分钟催化生成1 μmol产物所需的酶量。
1.2.5 酶学性质研究
为了确定不同条件对酶活的影响,对该酶的最适MgCl2浓度、ATP浓度、pH值以及温度进行测定。将反应体系分别放入25~60 ℃温度下反应30 min,测定酶活力;在pH值为3.0~9.0的缓冲液中,于最适温度下进行反应,30 min后测定酶活力;在反应体系中分别加入不同浓度的ATP(20~200 mmol/L),在最适温度下反应30 min,测定酶活;加入不同浓度的MgCl2(30~210 mmol/L),在最适温度下反应30 min,测定酶活力,最高酶活力定义为100%,用相对酶活力表示,每个反应做3次平行实验。
1.2.6 谷胱甘肽的合成
将菌体重悬于Tris-HCl(pH 7.0)溶液中,菌液质量浓度为100 g/L,在冰水浴中超声破碎,8 000 r/min离心10 min,收集上清液,即为粗酶液。催化合成谷胱甘肽反应体系为:100 mmol/L Tris-HCl buffer(pH 7.0)、90 mmol/L L-Glu、90 mmol/L Gly、80 mmol/L L-Cys、最佳浓度的ATP和MgCl2,以及15 g/L GSHA粗酶液、5 g/L GSHB粗酶液。在37 ℃条件下反应,加入等体积15% HCl溶液终止反应,离心取上清液,经0.22 μm滤膜过滤后使用高效液相色谱检测谷胱甘肽生成的量。HPLC检测方法:Agilent C18色谱柱(250 mm×4.6 mm,5 μm),流动相A为pH 3.0的50 mmol/L KH2PO4与10 mmol/L庚烷磺酸钠混合液,流动相B为色谱级甲醇,以V(A)∶V(B)=97∶3,流速0.5 mL/min,柱温25 ℃,紫外波长210 nm进行检测[24]。
1.3 数据处理
HPLC的检测结果通过与1 g/L的谷胱甘肽标准品的峰面积相比较,对产物进行定量。酶活力测定根据酶活力公式进行计算,每个实验均设置3个平行,使用Origin 8.5软件进行单因素方差分析并作图。
2 结果与分析
2.1 突变体的筛选
2.1.1 单点突变体的筛选
HIBI等[25]已研究报导过E.coli GSHA与过渡态类似物P2S的晶体结构,分析P2S与周围氨基酸残基的氢键作用力,如图1所示,发现P2S与E27、E29、D60、E67、Y131、I146、H150、R235、Y241、Y300和R330残基之间都存在氢键作用,将这11个氨基酸位点分别突变为丙氨酸破坏相互作用力缓解产物抑制[26]。另外,蛋白表面负电荷的增加可以有效提高蛋白稳定性,基于此原理在来自E.coli的GSHA蛋白表面选择一些非保守的氨基酸位点将其突变为带负电的氨基酸[27]。通过Pymol对GSHA的表面电势进行可视化分析,选择位于非保守区的带正电或不带电荷的氨基酸残基,突变为带负电荷的氨基酸,选定的具体的突变位点为A109E、G111E、A170E、A172D、S368D和A511D,根据选定的突变位点构建相应的突变体。
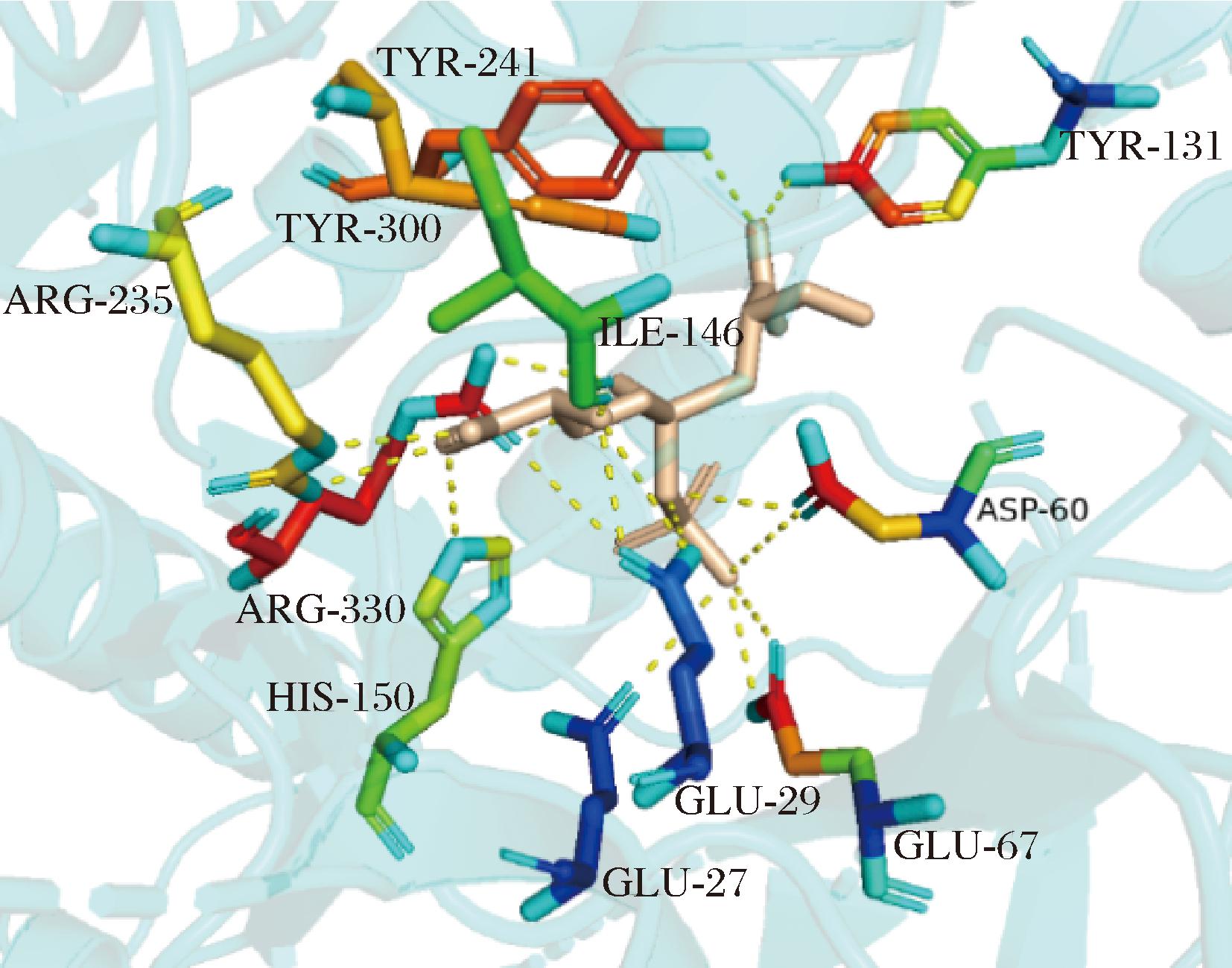
图1 GSHA与P2S的氢键作用力分布
Fig.1 Distribution of hydrogen bonding force between GSHA and P2S
2.1.2 单点突变体的构建与表达
利用设计的引物进行全质粒PCR,产物通过琼脂糖凝胶核酸电泳分析,结果如图2所示,所有突变体都得到了预期大小的目的条带。将PCR产物消化后转化入大肠杆菌感受态中,通过进一步测序验证,确定得到构建成功的单点突变体。将构建的突变体通过IPTG诱导进行表达,收集菌体进行超声破碎后得到粗酶液,用于后续实验。
图2 突变体的琼脂糖凝胶电泳
Fig.2 Agarose gel electrophoresis of mutants
在构建的17个突变体中,选择正确的突变菌株进行酶活测定。结果如图3所示,突变位点为E27A、E29A、D60A、E67A、I146A、H150A、R235A、Y241A、Y300A和R330A相对应的突变体的酶活力均小于野生型菌株,突变位点为Y131A的突变菌株酶活力较野生型菌株有较大提高,对底物的亲和能力及催化效率也相应提高。在选择的6个含有负电荷氨基酸的突变体中,A170E、A511D和S368D突变体的酶活力相较于野生型都有明显提高,说明负电荷氨基酸的引入对酶的活性有较好的提升效果。
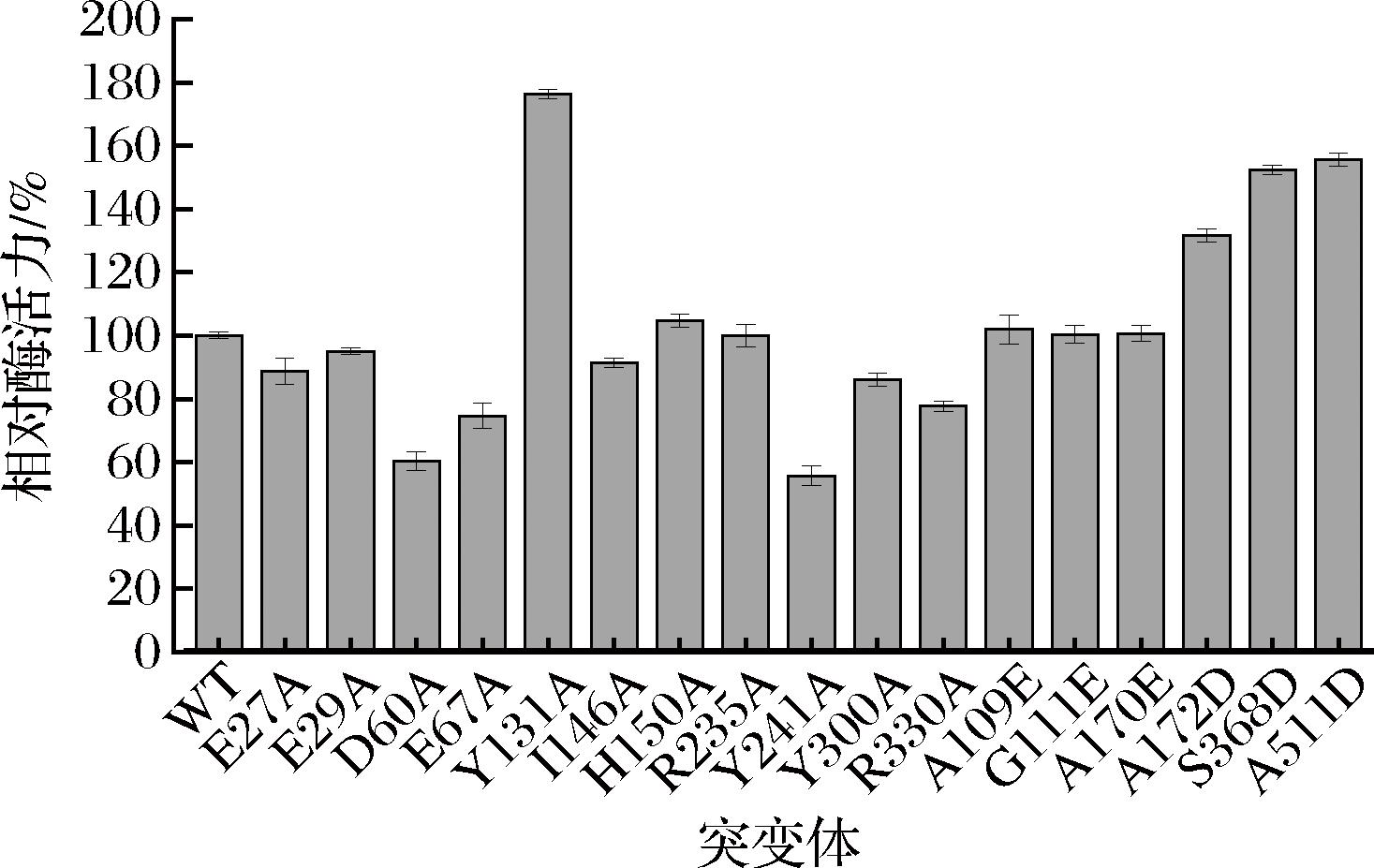
图3 单点突变体的相对酶活力
Fig.3 Relative enzyme activity of a single point mutant
2.1.3 组合突变体的筛选
为进一步提高酶的催化效果,将酶活力提高的Y131A突变体与3个提高稳定性的位点两两组合进行组合突变,共得到3个组合突变体:Y131A-A511D、Y131A-A170E、Y131A-S368D。对组合突变体的酶活力进行测定,结果如表2显示,组合突变体Y131A-A511D的酶活力最高,相对于野生型酶活力提高了6倍,展现出较好的底物催化效率。因此,选择最佳突变体Y131A-A511D进行后续实验。
表2 组合突变体的酶活
Table 2 Enzyme activity of combined mutants
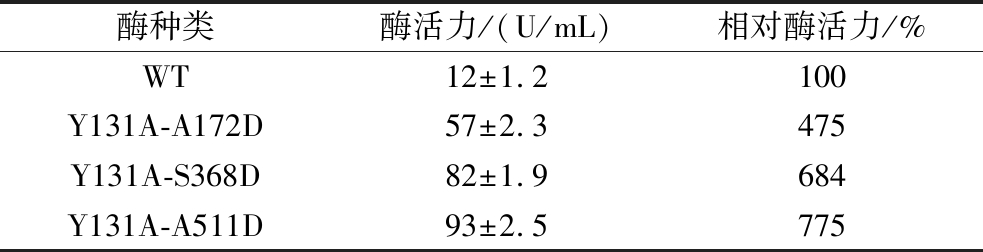
酶种类酶活力/(U/mL)相对酶活力/%WT12±1.2100Y131A-A172D57±2.3475Y131A-S368D82±1.9684Y131A-A511D93±2.5775
2.2 酶学性质分析
2.2.1 温度对酶活力的影响
在25~60 ℃下分别测定酶的活力,如图4-a所示,该酶在不同温度下均表现出较好的活力,在40 ℃以下,相对酶活力随着温度的升高而增大,最高酶活力在90%以上,当温度升高到40 ℃以上时,相对酶活力则逐渐下降,在60 ℃时,突变体的相对酶活力仅剩43.5%。温度过高会导致酶的蛋白质结构发生变化,对酶的活力具有较大影响。
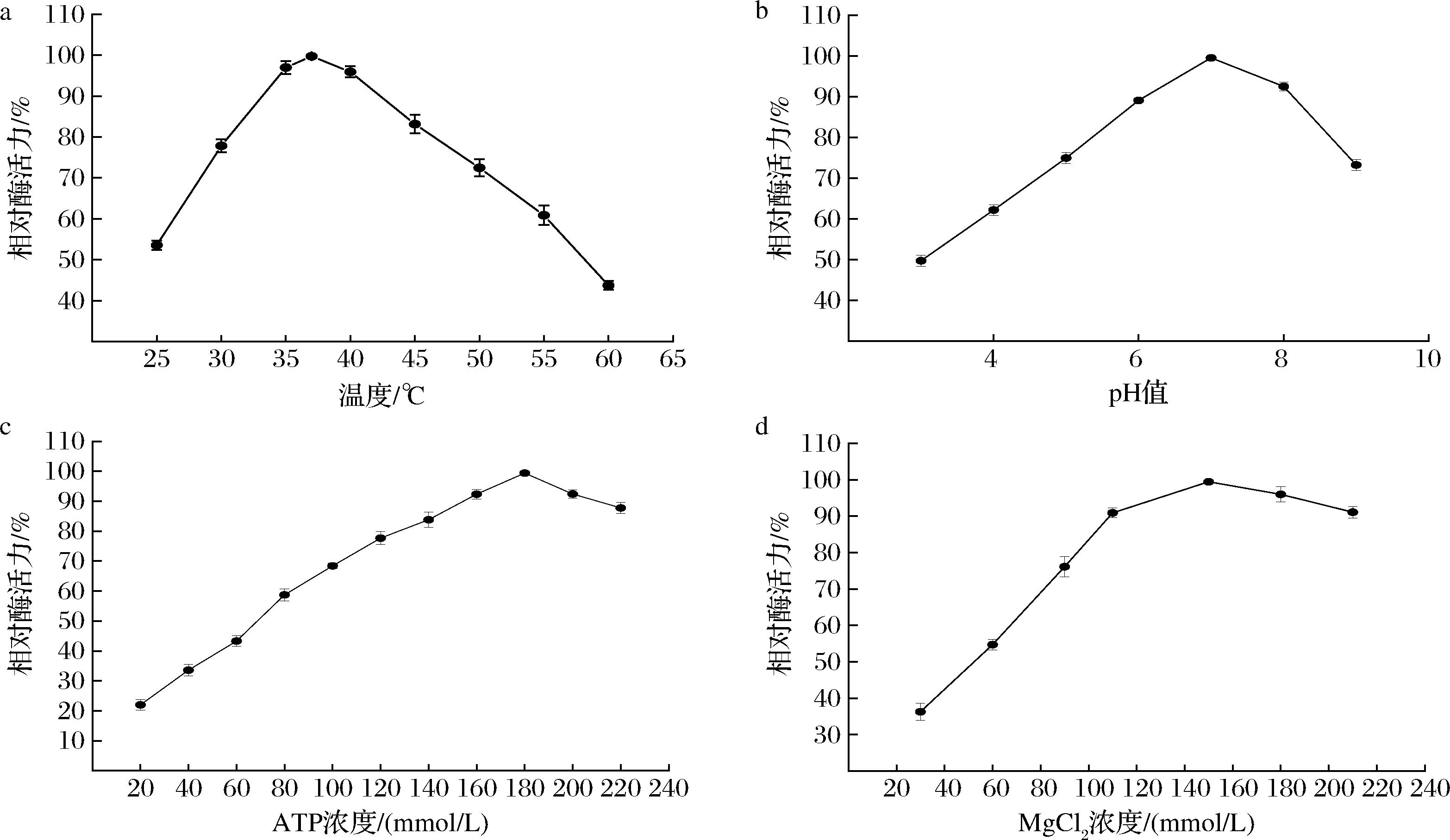
a-不同温度下的相对酶活力;b-不同pH值下的相对酶活力;c-不同浓度ATP下的相对酶活力;d-不同浓度MgCl2下的相对酶活力
图4 突变体的酶学性质
Fig.4 Enzymatic properties of the mutant
2.2.2 pH值对酶活力的影响
测定不同pH(3.0~9.0)下突变体的相对酶活力,在37 ℃反应30 min,结果如图4-b所示,当pH值在6.0~8.0时,突变体的相对酶活力较高,均保持在90%以上,说明该突变体在中性条件下酶活力较高。在pH值过高或过低的条件下,相对酶活力均有所下降,可能是由于酶催化过程中的关键基团的解离受到影响。
2.2.3 ATP浓度对酶活力的影响
ATP是双酶催化反应体系中的关键辅酶因子,为生物催化反应提供能量。ATP的浓度对反应体系的催化效率会产生影响,并且过多的ATP会增加生产的成本,理论上每摩尔底物转化需要消耗2 mol的ATP。测定ATP浓度对结果如图4-c所示,ATP浓度从140 mmol/L增加到180 mmol/L,相对酶活力逐渐升高,继续增加ATP浓度,相对酶活力逐渐下降,说明在此范围内,当ATP浓度为180 mmol/L时,酶活力最高,同时反应的催化效率最高。ATP浓度增加,GSH的生产效率反而降低,说明过高的ATP浓度对两酶偶联催化起到一定的抑制作用。
2.2.4 MgCl2浓度对酶活力的影响
在使用ATP依赖性酶进行催化反应时,加入Mg2+能够中和多磷酸链上的一些负电荷,减少了酶和核苷酸的多磷酸基团之间的非特异性离子的相互作用,ATP与Mg2+之间的结合能够促进磷酸盐的转移。因此,合适的MgCl2浓度对反应效率有着重要的影响。MgCl2浓度对酶活力影响的测定结果如图4-d所示,当MgCl2浓度为150 mmol/L时,相对酶活力较高,继续增加MgCl2浓度,酶活力不再继续提高。
2.3 利用突变酶催化生产谷胱甘肽
将改造后的酶应用于谷胱甘肽的生产中,构建双酶级联反应体系。将筛选的GSHA酶的最佳突变体Y131A-A511D应用于催化反应中,构建5 L谷胱甘肽催化体系,在37 ℃、pH7.0条件下进行反应,每小时取样进行HPLC检测产物浓度。如图5所示,反应6 h后底物转化率为96%,最终得到76.8 mmol/L(23.6 g/L)谷胱甘肽,继续反应产量无明显增高,可能是随着催化时间的增长,酶活力逐渐下降。与野生型酶的催化效率相比提高了2倍,提高了谷胱甘肽的产量。
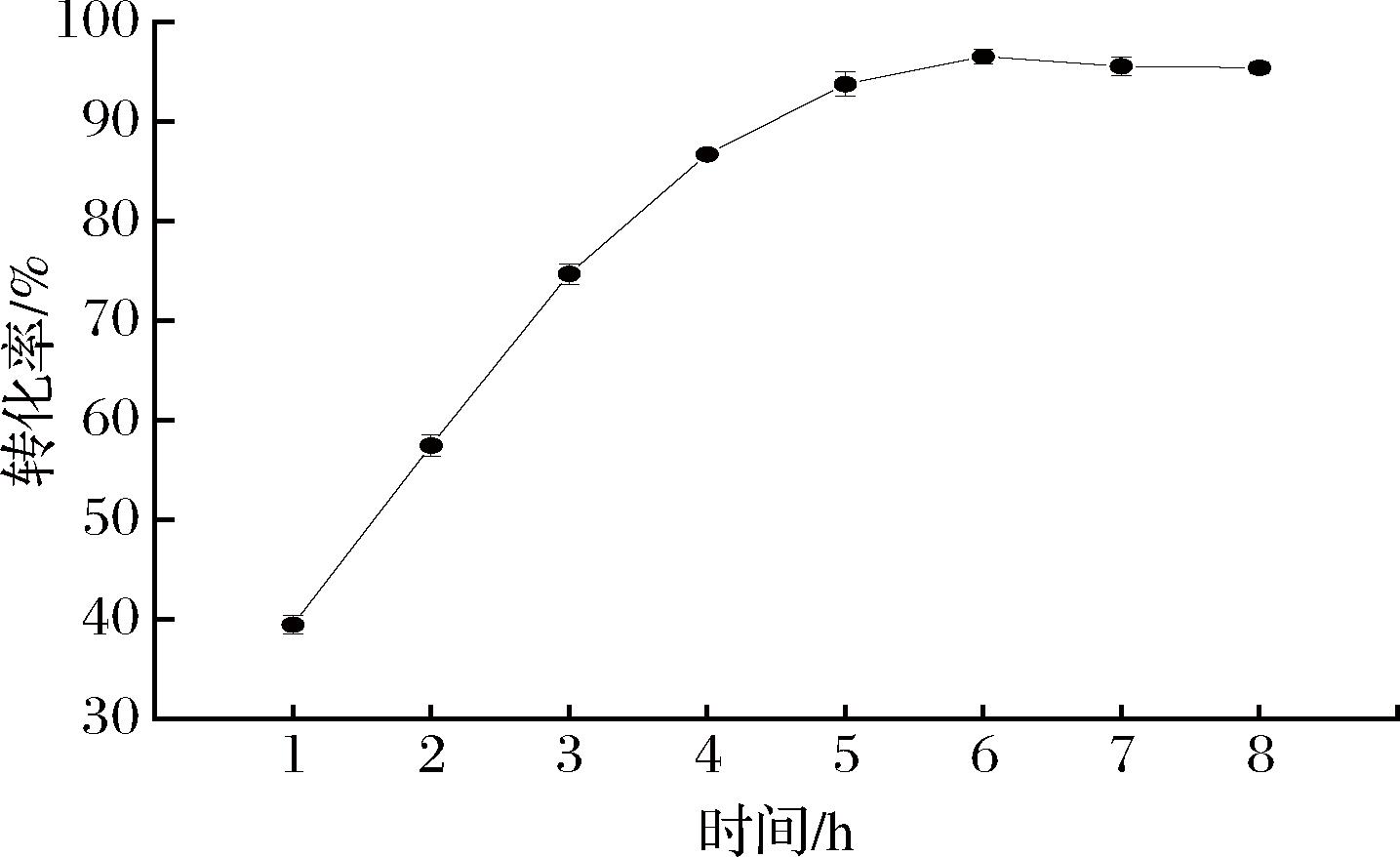
图5 谷胱甘肽的合成
Fig.5 The process of glutathione synthesis
3 结果与讨论
传统的酶法合成谷胱甘肽是通过两种酶进行催化,其中由GSHA催化合成中间体γ-GC的反应是限速步骤,该酶会受到产物的反馈抑制,催化效率不能满足生产需求。为获得高效的谷胱甘肽合成方法,有研究者通过两阶段反应来解除底物对酶的反馈抑制作用,在经条件优化后,30 h内谷胱甘肽的产量为3.44 g/L,但存在产量较低且产物易降解的问题[28]。近年来,在一些革兰氏阳性菌中发现了一种谷胱甘肽双合成酶,可单独催化合成谷胱甘肽[29]。JIANG等[30]将来自嗜热链球菌的GshF在大肠杆菌中进行表达,在3 h内生产的谷胱甘肽达到了18.3 g/L。但利用GshF催化存在效果不稳定、效率较低等问题。
本文以大肠杆菌来源的GSHA为对象,通过定点突变的方法,提高了该酶的催化活性与稳定性,扩大了底物的利用范围,减弱了产物的反馈抑制。成功筛选出酶活力提高的突变体,与野生型相比,突变体Y131A-A511D的酶活提高了6倍。对改造后的酶进行酶学性质分析,确定了该酶的最佳催化条件。最终在谷胱甘肽的合成中,通过5 L的催化反应,得到76.8 mmol/L(23.6 g/L)的谷胱甘肽。本研究构建了高效的谷胱甘肽合成酶,后续将通过与ATP再生系统进行联合,构建多酶级联生产谷胱甘肽的体系,降低生产成本,实现大规模工业生产谷胱甘肽的目的。
[1] KENDALL E C, MCKENZIE B F, MASON H L.A study of glutathione[J].Journal of Biological Chemistry, 1929, 84(2):657-674.
[2] JONES D P.Redox potential of GSH/GSSG couple:Assay and biological significance[J].Methods in Enzymology, 2002, 348:93-112.
[3] PENNINCKX M J.An overview on glutathione in Saccharomyces versus non-conventional yeasts[J].FEMS Yeast Research, 2002, 2(3):295-305.
[4] CARMEL-HAREL O, STORZ G.Roles of the glutathione and thioredoxin-dependent reduction systems in the Escherichia coli and Saccharomyces cerevisiae responses to oxidative stress[J].Annual Review of Microbiology, 2000, 54(1):439-461.
[5] HOMMA T, FUJII J.Application of glutathione as anti-oxidative and anti-aging drugs[J].Current Drug Metabolism, 2015, 16(7):560-571.
[6] FANG Y Z, YANG S, WU G.Free radicals, antioxidants, and nutrition[J].Nutrition, 2002, 18(10):872-879.
[7] LEI X G.In vivo antioxidant role of glutathione peroxidase:Evidence from knockout mice[J].Methods in Enzymology, 2002, 347:213-225.
[8] WU G Y, LUPTON J R, TURNER N D, et al.Glutathione metabolism and its implications for health[J].The Journal of Nutrition, 2004, 134(3):489-492.
[9] NIU B Y, LIAO K X, ZHOU Y X, et al.Application of glutathione depletion in cancer therapy:Enhanced ROS-based therapy, ferroptosis, and chemotherapy[J].Biomaterials, 2021, 277:121110.
[10] TOWNSEND D M, TEW K D, TAPIERO H.The importance of glutathione in human disease[J].Biomedicine &Pharmacotherapy, 2003, 57(3-4):145-155.
[11] GUAN X M. Glutathione and glutathione disulfide - their biomedical and pharmaceutical applications[J]. Medicinal Chemistry Research, 2023, 32(9): 1972-1994.
[12] HARA K Y, KONDO A.ATP regulation in bioproduction[J].Microbial Cell Factories, 2015, 14:198.
[13] ZHANG J, QUAN C, WANG C, et al.Systematic manipulation of glutathione metabolism in Escherichia coli for improved glutathione production[J].Microbial Cell Factories, 2016, 15(1):38.
[14] LU S C.Regulation of glutathione synthesis[J].Current Topics in Cellular Regulation, 2000, 36:95-116.
[15] RICHMAN P G, MEISTER A.Regulation of gamma-glutamyl-cysteine synthetase by nonallosteric feedback inhibition by glutathione[J].Journal of Biological Chemistry, 1975, 250(4):1422-1426.
[16] MURATA K, KIMURA A.Overproduction of glutathione and its derivatives by genetically engineered microbial cells[J].Biotechnology Advances, 1990, 8(1):59-96.
[17] BITEROVA E I, BARYCKI J J.Mechanistic details of glutathione biosynthesis revealed by crystal structures of Saccharomyces cerevisiae glutamate cysteine ligase[J].Journal of Biological Chemistry, 2009, 284(47):32 700-32 708.
[18] CHOI J M, KIM H S.Structure-guided rational design of the substrate specificity and catalytic activity of an enzyme[J].Methods in Enzymology, 2020, 643:181-202.
[19] ANAND P, NAGARAJAN D, MUKHERJEE S, et al.ABS-Scan:In silico alanine scanning mutagenesis for binding site residues in protein-ligand complex[J].F1000Research, 2014, 3:214.
[20] LIU X, PENG L, ZHOU Y F, et al.Computational alanine scanning with interaction entropy for protein-ligand binding free energies[J].Journal of Chemical Theory and Computation, 2018, 14(3):1772-1780.
[21] YANG J X, WANG B, GE L, et al.The enhancement of surface activity and nanoparticle stability through the alteration of charged amino acids of HGFI[J].Colloids and Surfaces B:Biointerfaces, 2019, 175:703-712.
[22] ANUMALLA B, PRABHU N P.Surface hydration and preferential interaction directs the charged amino acids-induced changes in protein stability[J].Journal of Molecular Graphics and Modelling, 2020, 98:107602.
[23] CUI C X, MING H, LI L J, et al.Fabrication of an in-situ co-immobilized enzyme in mesoporous silica for synthesizing GSH with ATP regeneration[J].Molecular Catalysis, 2020, 486:110870.
[24] 张浩月, 穆玉敏, 秦浩杰, 等.多酶体系中谷胱甘肽HPLC分析方法研究[J].煤炭与化工, 2023, 46(10):151-155.
ZHANG H Y, MU Y M, QIN H J, et al.Study on HPLC analysis method of glutathione in multienzyme system[J].Coal and Chemical Industry, 2023, 46(10):151-155.
[25] HIBI T, NII H, NAKATSU T, et al.Crystal structure of γ-glutamylcysteine synthetase:Insights into the mechanism of catalysis by a key enzyme for glutathione homeostasis[J].Proceedings of the National Academy of Sciences, 2004, 101(42):15 052-15 057.
[26] SONG Z D, ZHANG Q F, WU W H, et al.Rational design of enzyme activity and enantioselectivity[J].Frontiers in Bioengineering and Biotechnology, 2023, 11:1129149.
[27] STEINER K, SCHWAB H.Recent advances in rational approaches for enzyme engineering[J].Computational and Structural Biotechnology Journal, 2012, 2(3):e201209010.
[28] LI W, LI Z M, YE Q.Enzymatic synthesis of glutathione using yeast cells in two-stage reaction[J].Bioprocess and Biosystems Engineering, 2010, 33(6):675-682.
[29] LI W, LI Z M, YANG J H, et al.Production of glutathione using a bifunctional enzyme encoded by gshF from Streptococcus thermophilus expressed in Escherichia coli[J].Journal of Biotechnology, 2011, 154(4):261-268.
[30] JIANG Y, TAO R S, SHEN Z Q, et al.Enzymatic production of glutathione by bifunctional γ-glutamylcysteine synthetase/glutathione synthetase coupled with in vitro acetate kinase-based ATP generation[J].Applied Biochemistry and Biotechnology, 2016, 180(7):1446-1455.