自然发酵乳是以原料乳固有微生物在一定环境下自然发酵而得,其微生物多样性丰富,主要分布于新疆、内蒙、西藏等乳源较广区域,以牛乳和非牛乳两大来源为主[1-2]。其中,自然发酵驼乳是高原牧区沿袭而来的特色传统食品,历史悠久,通常被用于治疗水肿、黄疸、结核病、哮喘和利什曼病或黑热病等一系列疾病[3]。近年来,以非牛乳为来源的自然发酵乳制品中,自然发酵驼乳因其优异的功能特性,其中包括抗菌、抗氧化(ABTS、DPPH)、抗肥胖、抗高血压(ACE酶抑制特性)、抗糖尿病(α-淀粉酶、α-葡萄糖苷酶抑制特性)、抗炎等[4-6],成为科研领域挖掘优良乳酸菌和研究微生物多样性的一大热点。卓娜等[7]研究发现,不同区域自然发酵酸驼乳微生物群落丰度均较高,自然发酵酸驼乳中分离的乳酸菌主要为肠球菌(Enterococcus)、乳球菌(Lactococcus)、乳杆菌(Lactobacillus)等[8]。骆驼乳及其发酵制品微生物多样性丰富,是分离筛选优良乳酸菌发酵剂的较佳选择[9-10]。
青稞酒糟及黄水是高原青稞酒酿造过程中产生的主要副产物,酒糟产量高,且多用作饲料;黄水是在酒醅发酵过程中,淀粉逐渐被消耗,其中所含水分及发酵产生的水分沉积到窖底形成的一种黄褐色黏稠液体[11],多为废液排出,利用价值低,探讨酒糟及黄水微生物组成并挖掘优良菌种发酵剂,不仅可实现酿酒副产物的高值化利用,同时可提高经济效益[12-13]。研究表明,黄水微生物资源及风味物质丰富,且优势菌属与黄水风味来源密切相关,其中乳杆菌属是核心菌属[14]。酒糟中的微生物来源丰富,包括大曲、窖泥等,罗爱国等[12]发现清香型堡子酒酒糟中细菌多样性及丰度均高于真菌,细菌由10个门、86个属和124个种组成。青稞大曲中的优势细菌属主要为泛菌属(Pantoea)、链球菌属(Staphylococcus),且乳杆菌属(Lactobacillus)为核心微生物,在后期酒醅副产物中占主导地位[15];不同区域清香型大曲中优势细菌属主要包括Bacillus Burkholderia、魏斯氏菌属(Weissella)、Lactobacillus等[16];不同原料大曲微生物组成明显不同,区域的差异性也对微生物组成有所影响[17]。青稞酒因其独特酿造环境(原料与季节特征),其细菌丰度高,天佑德青稞酒厂分析表明,青稞酒酿造微生物主要包括酵母菌、霉菌和细菌三大类。但目前对青稞酒糟及黄水副产物的微生物菌群结构及多样性分析的相关研究鲜见报道。
目前以自然发酵驼乳及青稞酒糟和黄水为原材料,对其微生物多样性及菌群结构差异性和关联性分析的相关研究还未发现。本研究以青稞酒酿造副产物(酒糟及黄水)和取自不同季度的自然发酵驼乳为原料,从中分离鉴定乳酸菌;并基于Illumina NovaSeq二代测序技术,进行物种结构和多样性分析,同时测定乳酸菌理化特性,探究青稞酒糟与自然发酵驼乳微生物组成差异以及共有的核心发酵菌株,为下一步从青稞酒糟中分离纯化用于骆驼乳发酵的乳酸菌做基础准备。本试验不仅对青稞酒糟和自然发酵驼乳的微生物相关分析提供新的借鉴,同时丰富了自然菌种发酵库。
1 材料与方法
1.1 材料与试剂
采集于2个不同季度的自然发酵驼乳,其中R、X分别为夏季六月、九月采集的样品,W为冬季采集样品,所有样品均采自德令哈。青稞酒糟样品(J)及黄水(H)均采自天佑德青稞酒厂。所有样品均采用无菌工具分别采集,即时置于低温环境运往实验室进行后续试验。采集过程中,自然发酵驼乳样品置于无菌冻存管,青稞酒糟及黄水样品分别置于灭菌袋及无菌瓶,所有样品在无菌条件下采集后用于乳酸菌分离培养试验。
MRS固体培养基、MRS液体培养基,北京奥博星生物技术有限责任公司;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;PCR扩增试剂盒,艾科瑞生物工程有限公司;引物27F/1492R,生工生物工程股份有限公司;凝胶电泳所用试剂和酶类及Marker,塞维尔生物科技有限公司。
1.2 仪器与设备
YM高压蒸汽灭菌锅,江苏迅迪仪器科技有限公司;SW-CJ-1D超净工作台,苏州净化设备有限公司;小型高速台式离心机,德国Sigma公司;SPX-250F恒温培养箱,宁波普朗特仪器有限公司;ABI GeneAmp®9700型梯度PCR扩增仪,美国ABI公司;Multiskan SkyHigh酶标仪,赛默飞世尔科技公司;JY600电泳仪,北京君意东方电泳仪设备公司;Gel Doc XR+凝胶成像分析系统,美国ABI公司。
1.3 实验方法
1.3.1 乳酸菌分离、纯化
采用涂布平板法分离纯化乳酸菌。10倍梯度稀释法处理自然发酵驼乳及青稞酒糟和黄水样品,选择稀释度为10-3、10-4、10-5的样品稀释液振荡混匀后,涂布于MRS固体培养基,37 ℃恒温倒置培养48 h。挑取不同形态单菌落划线纯化,将革兰氏阳性,外表呈椭圆、乳白色的单菌落初步确定为乳酸菌。所得细菌纯培养物转移至MRS液体培养基进行传代培养,活化至2代以增强细菌活性。活化所得乳酸菌置于20%甘油和80% MRS液体培养基混合液中培养2 h,并置于-80 ℃超低温冰箱保藏。
1.3.2 样品DNA的提取及PCR扩增
1.3.2.1 细菌基因组DNA的提取
保藏菌株活化两代后经4 ℃,3 800 r/min低速离心5 min,弃掉废液,加入PBS缓冲液清洗2遍,弃掉滤液;最后1次加入PBS缓冲液,振荡混匀吸取1 mL菌悬液到离心管内,12 000 r/min离心2 min,移出废液,参照说明书步骤依次操作,利用细菌基因组DNA提取试剂盒提取DNA,并置于-20 ℃贮存备用。完成基因组DNA抽提后,通过微量紫外分光光度计和1%琼脂糖凝胶电泳测定所提DNA的浓度以及纯度。
1.3.2.2 细菌16S rRNA基因PCR扩增
根据PCR扩增试剂盒各步骤制备细菌PCR扩增反应体系:25 μL 2×Pro TaqMaster Mix(dye 1×plus);2 μL DNA模板(100 ng/μL);1 μL正向引物27F(5′-AGAGTT TGATCTGGCTCAG-3′);1 μL反向引物1492R(5′-ACCTTGTTACGACTT-3′);21 μL ddH2O。
反应条件:90 ℃预变性30 s;98 ℃变性10 s,55 ℃退火30 s,72 ℃延伸1 min,共25~30个循环;72 ℃末端延伸2 min。
1.3.3 细菌16S rRNA V3~V4区测序及微生物多样性分析
PCR产物用1%琼脂糖凝胶电泳检测,细菌16S rRNA产物条带在1 500~2 000 bp之间,所得条带清晰且明亮,无拖尾现象,PCR扩增成功。经检测的PCR产物送往塞维尔生物科技有限公司测序。双端序列使用Seqman软件拼接后在NCBI(www.ncbi.nlm.nih.gov)中进行BLAST(basic local alignment search tool)比对,从中选择相似度和覆盖率均较高的序列,确定其菌种。
基于Illumina NovaSeq测序平台,通过对下机原始Reads拼接、过滤、聚类或去噪,并进行物种注释及丰度分析,从而揭示样品的物种构成;进一步进行α多样性分析、β多样性分析、显著物种差异分析等,挖掘样品之间的差异。
1.3.4 数据统计与分析
采用Excel 2016进行数据计算和分析,使用QIIME2软件进行样品α多样性分析,基于R语言平台绘制样本堆积柱状图以分析样本细菌门、属组成,选取数目最多的前80个物种,基于Rheatmap绘制聚类热图,乳酸菌测序鉴定结果在NCBI数据库进行BLAST比对,使用MEGA 11构建系统发育树。
2 结果与分析
2.1 系统发育树的构建及乳酸菌分离鉴定结果
将获得的青稞酒糟、黄水及自然发酵驼乳16S rRNA基因序列在NCBI数据库中进行BLAST比对之后,选择序列长度相近,同源性>99%的相似菌株确定为该种,并使用MEGA 11构建系统发育树。由图1可知X(夏季自然发酵驼乳)、W(冬季自然发酵驼乳)、H(黄水)、J(青稞酒糟)分别鉴定为粪肠球菌(Enterococcus faecium)、乳酸球菌(Lactococcus lactis)、戊糖片球菌(Pediococcus pentosaceus)、乳酸片球菌(Pediococcus acidilactici)。

图1 五株乳酸菌分离株16S rRNA基因序列系统发育树
Fig.1 Phylogenetic tree of 16S rRNA gene sequence of five lactic acid bacteria isolates
2.2 青稞酒糟、黄水和自然发酵驼乳的测序分析结果
2.2.1 样品OTU结果分析
对采集的3个不同自然发酵驼乳样品和青稞酒糟及黄水样品进行基因测序和质量控制分析后,获得362 055个细菌16S rRNA基因序列,平均每个样品产生72 411个序列,序列的平均长度460 bp。根据97%的相似性对所有高质量的细菌序列进行OTU分类,共鉴定出3 577个细菌OTUs,其中H样品获得的代表性OTUs最少,为327条;J样品获得的代表性OTUs最多,为1 265条,OTUs数量与测序深度呈正相关;平均每个样品获得715条代表性序列,5份不同样品中的高相似度OTUs共7条,分别占R、W、X、H、J组样品的0.81%、1.62%、0.86%、2.14%、0.55%,这些微生物为5份不同样品中的核心微生物(图2)。进一步利用稀疏曲线(图3-a)和Shannon指数曲线(图3-b)对样品中的细菌测序量进行分析,由图3可知,当样品测序深度逐渐趋于平缓时,随着测序深度的增加,样本中的细菌测序量基本不再变化,表明样品的测序深度足以揭示样品中的大部分细菌序列,能够满足后续生物信息学分析的要求。
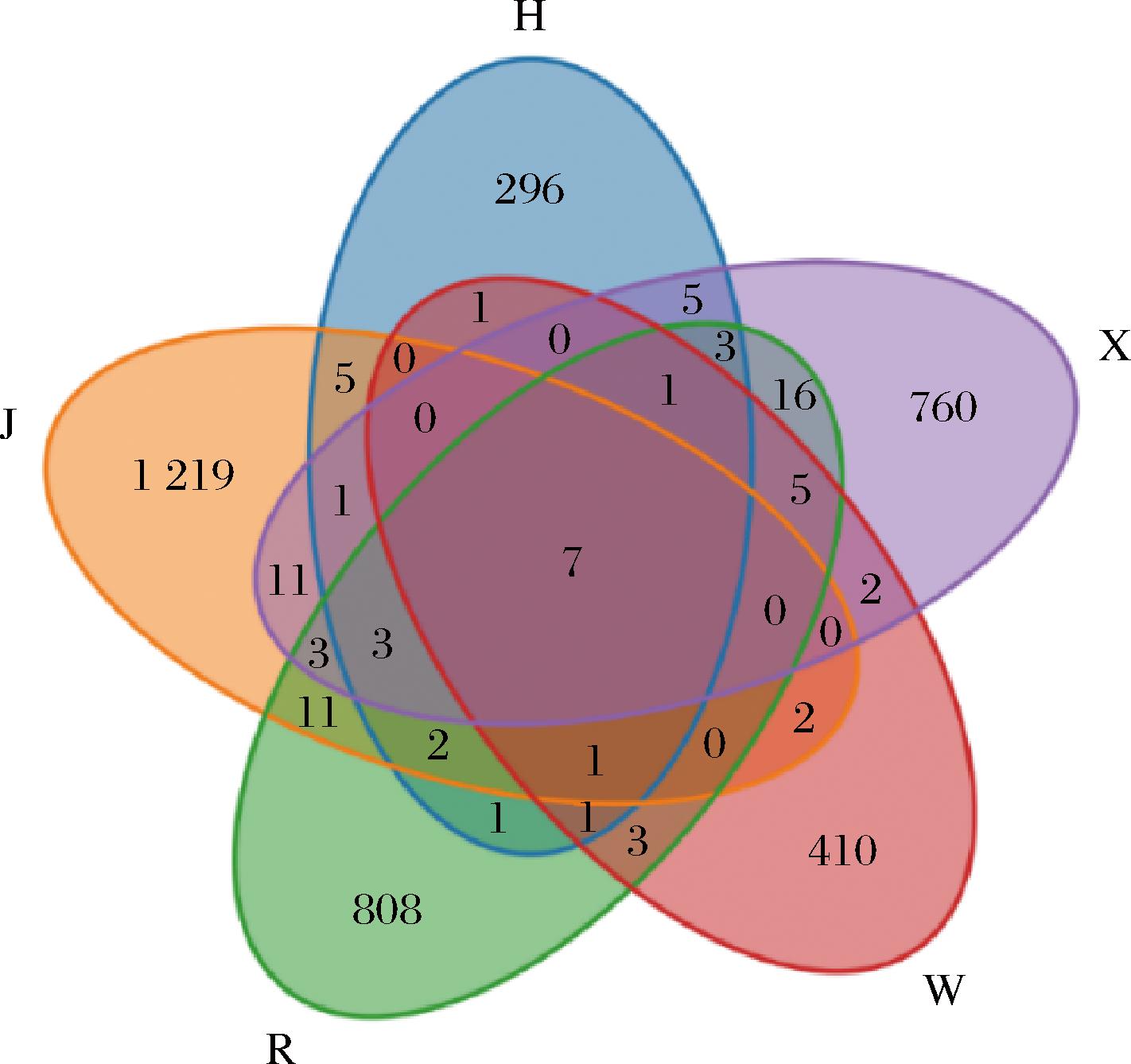
图2 自然发酵驼乳及青稞酒糟和黄水样品细菌OTUs韦恩图
Fig.2 Venn diagram of bacteria in naturally fermented camel milk, barley lees, and yellow water samples
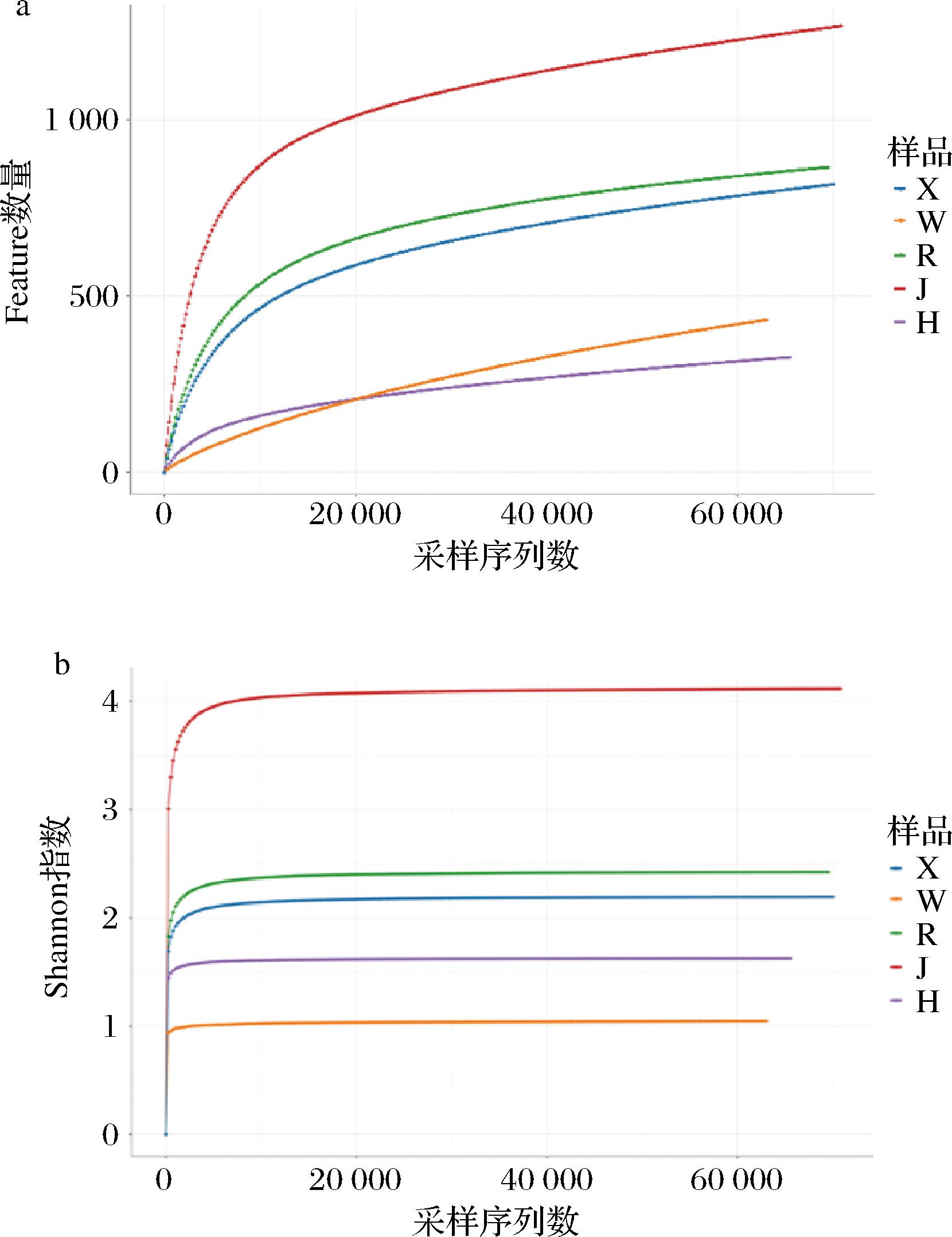
a-稀释曲线;b-香农曲线
图3 自然发酵驼乳样品及青稞酒糟和黄水样品稀释曲线及香农曲线
Fig.3 Dilution curve and Shannon curve of naturally fermented camel milk, barley lees, and yellow water samples
2.2.2 青稞酒糟、黄水和自然发酵驼乳的乳酸菌多样性分析结果
对5份样品进行α多样性分析,由表1可知,3份自然发酵驼乳的ACE指数和Chao1指数相近,表明3份自然发酵驼乳微生物群落丰富度相似;而青稞酒糟ACE指数和Chao1指数最高,微生物丰度最高,黄水具有最低的微生物丰度。香农指数和辛普森指数是微生物多样性的主要指示指标,青稞酒糟辛普森指数和香农指数最高,表明酒糟微生物多样性最高;W样品的香农指数较小,微生物多样性较低。
表1 自然发酵驼乳样品及青稞酒糟和黄水样品细菌序列信息和微生物多样性
Table 1 Bacterial sequence information and microbial diversity of naturally fermented camel milk samples, barley lees, and yellow water samples
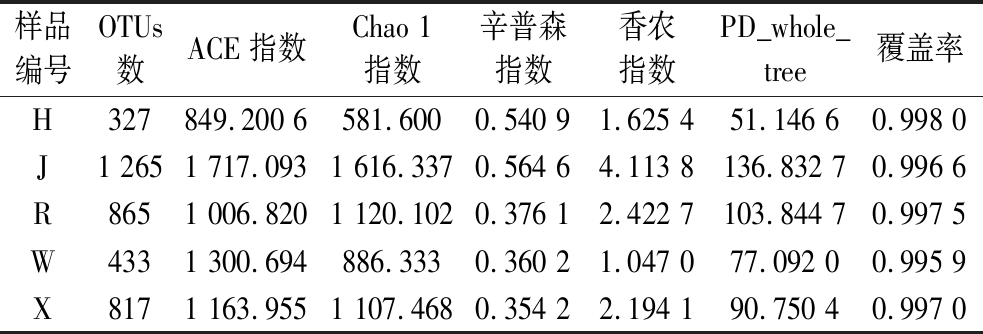
样品编号OTUs数ACE指数Chao 1指数辛普森指数香农指数PD_whole_tree覆盖率H327849.200 6581.6000.540 91.625 451.146 60.998 0J1 2651 717.0931 616.3370.564 64.113 8136.832 70.996 6R8651 006.8201 120.1020.376 12.422 7103.844 70.997 5W4331 300.694886.3330.360 21.047 077.092 00.995 9X8171 163.9551 107.4680.354 22.194 190.750 40.997 0
2.2.3 青稞酒糟、黄水和自然发酵驼乳细菌群落结构分析结果
为了确认样品中微生物组成的相似性与差异性,对微生物组成进行分析。由图4-a可知,在门水平上,5份样品经测序鉴定后归属于38个细菌门,其中主要的优势菌门依次为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria),与WANG等[18]探究不同轮次酱香型白酒堆积发酵微生物多样性结果相似。占比分别为85.82%、3.87%、2.5%、1.18%,其中厚壁菌门是主要的优势菌门,其余次要菌门(others)的相对含量都相对较少。
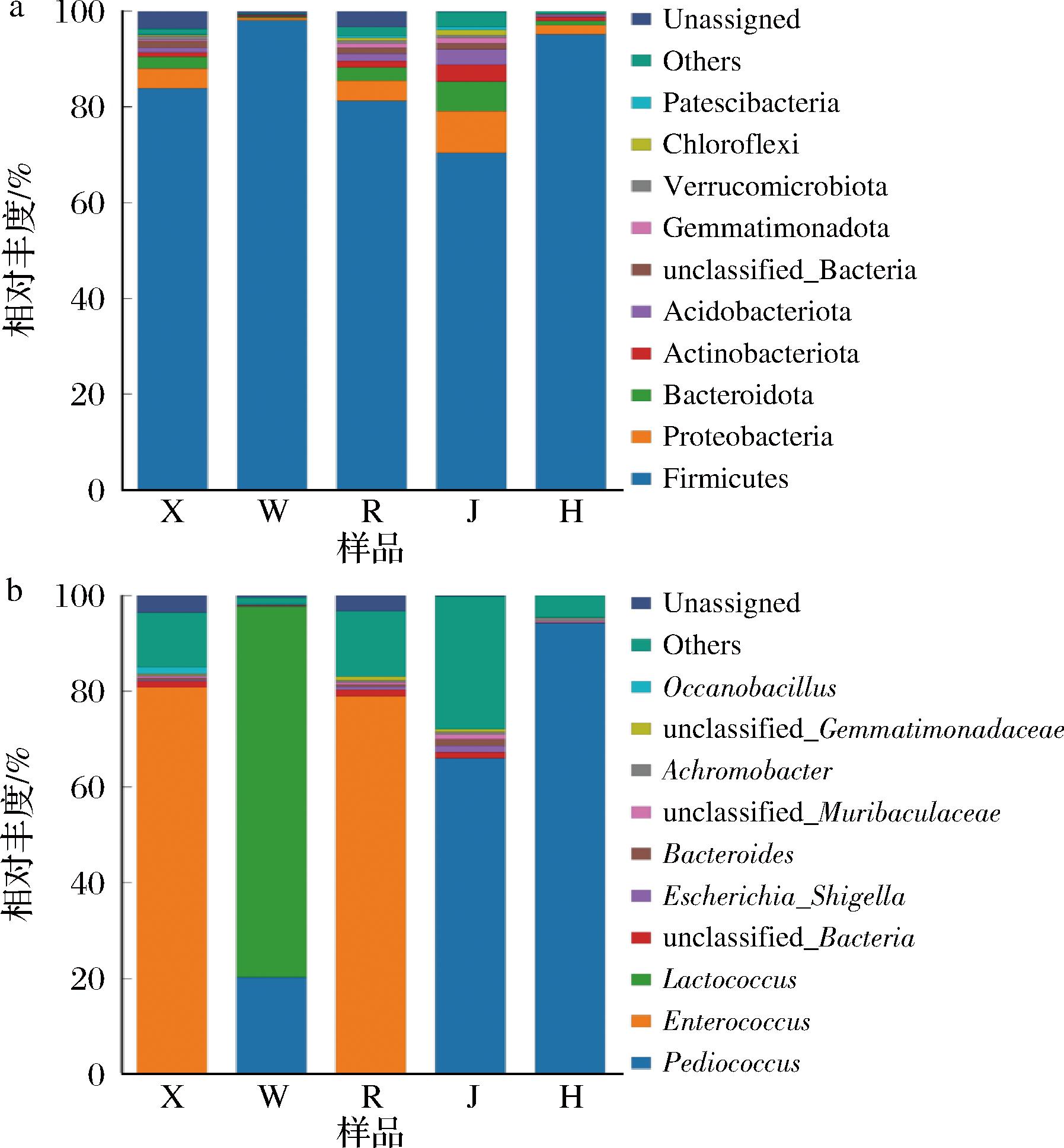
a-门水平;b-属水平
图4 门水平和属水平的细菌群落组成
Fig.4 Bacterial community composition at phylum level and genus level
在属水平上,5份样品共检测到779个属,其中相对含量较高的优势菌属主要包括片球菌属(Pediococcus,36.12%)、肠球菌属(Enterococcus,31.98%)、乳球菌属(Lactococcus,15.49%)、others。Enterococcus是X、R两个样品的主要优势菌属,其相对含量分别为80.87%、79.01%;而W中的优势菌属包括Pediococcus和Lactococcus,分别占20.28%、77.43%;J、H两个样品中Pediococcus相对含量最高,分别为66.03%、94.25%,为其中的关键优势菌属。可见,3份自然发酵驼乳由于采集地域不同,发酵条件不同,其微生物组成也存在较大差异;此外,相比X、R两份自然发酵驼乳,W样品与酒糟、黄水具有更相似的微生物结构。
2.2.4 青稞酒糟、黄水和自然发酵驼乳细菌聚类结果分析
为了更好的探究5份样品中的优势细菌,基于属水平,进一步作物种丰度聚类分析,结果如图5所示。由图5可知,青稞酒糟中优势细菌丰度较大,其微生物组成丰度较高,其优势菌主要包括另枝菌属(Alistipes)、Staphylococcus、棒状杆菌属(Corynebacterium)、厌氧支原体属(Anaeroplasma)、别样海源菌属(Aliidiomarina)、粪杆菌属(Facealibacterium)等;而W样品细菌物种丰度最低,主要优势菌为Alistipes;H样品丰度较高的菌有栖水菌属(Enhydrobacter)、无色杆菌属(Achromobacter)、Pediococcus等;X、R样品中的细菌丰度及结构较为相似,其中,肠杆菌科(unclassified-Enterobacteriaceae)、海洋芽胞杆菌属(Oceanobacillus)在X中的丰度较高,黄杆菌属(Flavobacterium)、Faecalibaculum、气单胞菌属(Aeromonas)为R中的优势菌。由此可推测酒糟及黄水中的细菌在发酵驼乳环境中具有生存的可能。
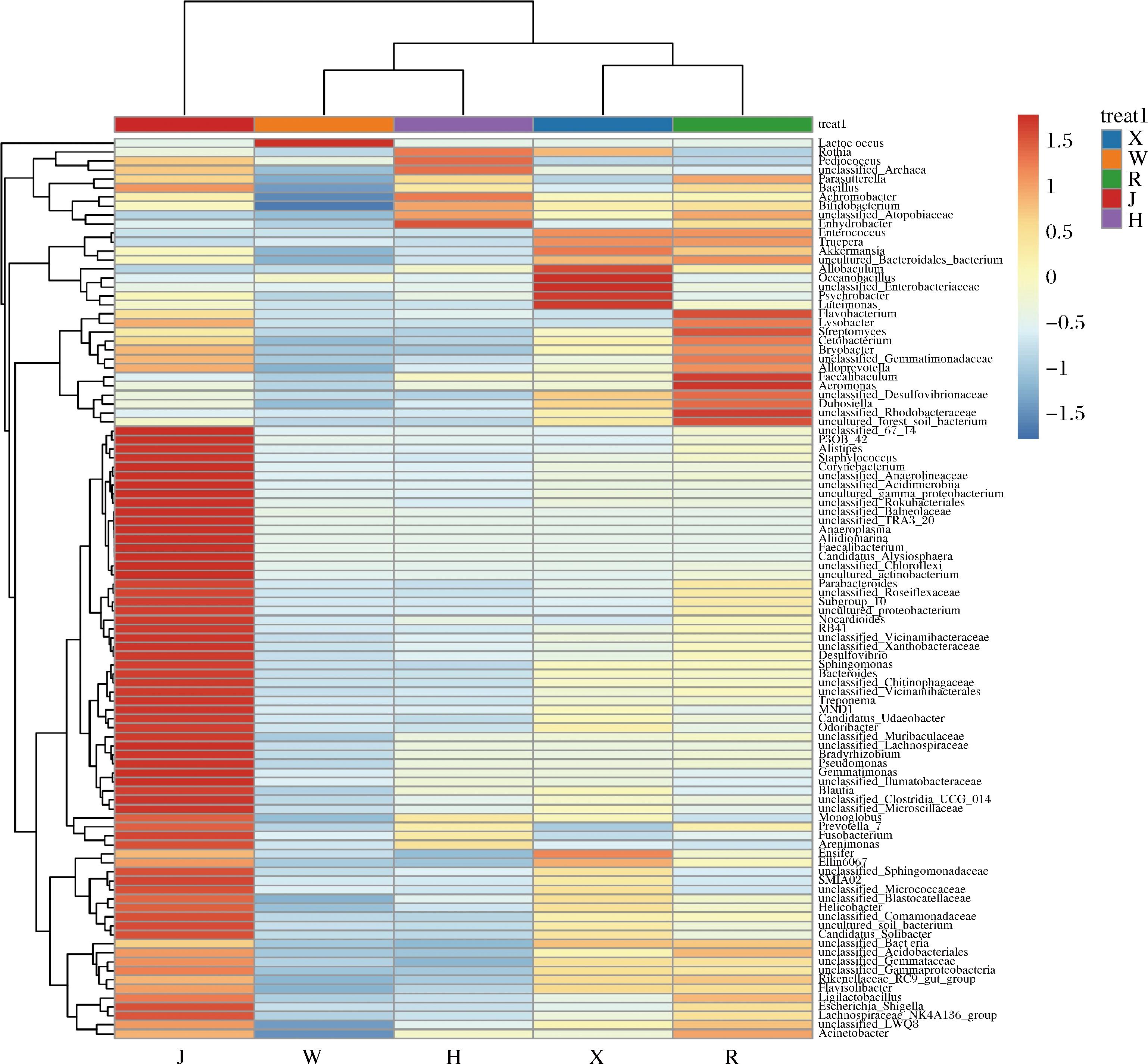
图5 自然发酵驼乳样品及青稞酒糟和黄水样品物种丰度聚类热图
Fig.5 Clustering heatmap of species abundance of three naturally fermented camel milk samples, barley lees and yellow water
2.3 乳菌耐酸、耐盐特性及生长曲线绘制
青稞酒糟、黄水和自然发酵驼乳中分离的5株乳酸菌经鉴定后,从中选择3株菌,测定其耐酸及耐盐性,3株菌在酸性及胆盐环境下生长结果如图6。由各菌株生长曲线可知,在pH 2或pH 3条件下培养,黄水中乳酸菌表现出最佳耐酸性,在后续生长过程中,其光密度值仍有所上升,而R和J中所得的2株菌在酸性条件下,其活性完全受到抑制。与此相似,3株菌在0.1%及0.3%的牛胆盐环境下培养,R中所分离的乳酸菌耐盐性能较差,而H与J中的乳酸菌在牛胆盐环境下仍能保持一定活性。
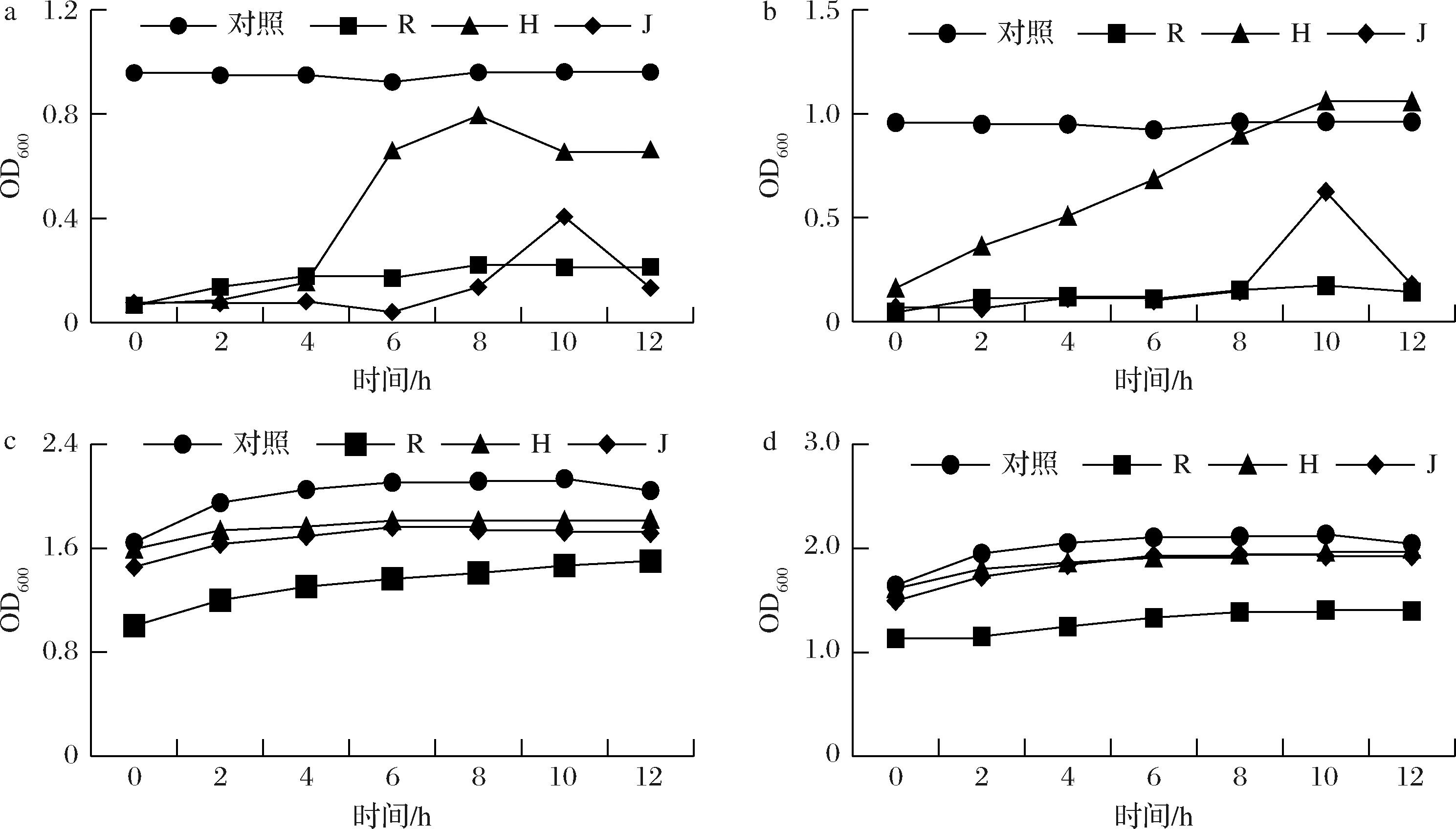
a-pH 2;b-pH 3;c-0.1%牛胆盐;d-0.3%牛胆盐
图6 三株乳酸菌对不同酸及胆盐的耐受力
Fig.6 Tolerance of three lactic acid bacteria to different acids and bile salts
3 讨论
中国发酵制品极其丰富,而在众多发酵品中,发酵乳制品和酿酒产业在居民生活中消费较广。酸驼乳及青稞酒是高原特色产品,因其独特的加工条件、气候、工艺、环境等,使得酸驼乳及青稞酒酿造副产物中微生物极为丰富。发酵驼乳中乳酸菌占据主要优势,乳杆菌属是其中的核心优势菌属[19-20]。与本研究结果有所不同,可能是乳酸菌纯培养环境(温度、操作方式)所致。酒糟中的微生物主要来源于酒曲及发酵环境等。酒曲中以Oceanobacillus、枝芽孢杆菌属(Virgibacillus)、芽孢杆菌属(Bacillus)等为优势菌[21],窖泥中的优势菌主要为Pediococcus、Bacillus、Corynebacterium、Enterococcus、链霉菌属(Streptomyces)等[22]。本研究结果表明,青稞酒糟中微生物多样性及细菌丰度均为最高,主要优势菌门为Firmicutes,优势菌属为Alistipes、Pediiococcus、Staohylococcus、others等。W、X、R样品次之,H样品微生物多样性及丰度最低,可能是在酒糟发酵后期,发酵环境酸性增强,微生物群落减少。测序结果表明,自然发酵驼乳及青稞酒酿造副产物中存在共有核心菌,且在微生物组成上存在相似菌,表明某些菌株在酒糟及驼乳发酵环境下有均可成活的可能。
目前对自然发酵驼乳中乳酸菌进行分离筛选,从而制备优良乳酸发酵菌株是研究的一大热点,其中乳酸球菌是一类产酸性能较好,具有安全性的发酵菌株[23]。乳品在发酵过程中,发酵菌株分解糖类产生乳酸,酸度增大;同时,对酸及胆盐的耐受性是保证益生菌进入人体发挥益生功能的前提条件[24-26]。因此,分析乳酸菌的耐酸及耐胆盐性是筛选优良发酵菌株的基础。本研究分析了所选3株菌的耐酸、耐盐性,其中,青稞酒糟及黄水中的乳酸菌在酸性及胆盐环境下存活率更高。此外,区域和季度的差异性是微生物群落组成差异的两大影响因素。内蒙古4个地区传统发酵乳中分离的乳酸菌均属于Lactobacillus、Entercoccus等,但乳酸菌数量及种类存在差异[27];内蒙古阿尤恩地区自然发酵乳样品中则以Lactococcus为优势菌属,Lactococcus lactis为优势菌种[28],不同区域采集自然发酵驼乳样品乳酸菌结构及多样性存在差异。与本研究结果相似,不同季度采集自然发酵驼乳也会对细菌群落造成较大影响,采自夏季的样品中细菌丰度及多样性均高于冬季。
4 结论
本文基于Illumina NovaSeq二代测序技术,主要探究青稞酒酿造副产物及自然发酵驼乳的乳酸菌组成及群落多样性。结果表明,3类样品微生物群落均有所差异,其中,采自夏季的自然发酵驼乳样品较为相似,青稞酒糟样品细菌丰度及多样性均为最高,而在冬季采集酸驼乳中均较低。3类样品细菌结构由38门、779属、914种组成,其中,Firmicutes、Proteobacteria、Bacteroidetes、Actinobacteria为样品中的优势菌门;Pediococcus、Enterococcus 、Lactococcu、others为优势菌属。3株乳酸菌中源于青稞酒的Pediococcus acidilactici对酸及胆盐耐受性能较好。目前,对青稞酒酿造副产物微生物多样性的研究尚少,后续可针对青稞酒酿造不同楂次采集青稞酒糟样品,并分析其微生物群落演变规律,从中分离筛选乳酸菌,实现酿酒副产物高值化利用。本研究解析了青稞酒糟、黄水和自然发酵驼乳的细菌结构差异及多样性,为后续从中挖掘优良乳酸菌发酵菌株奠定基础,同时丰富了自然菌种库。
[1] XIA A N, JIANG Y, LI B K, et al.Indigenous Chinese fermented dairy products:Microbial diversity, flavour, and health benefits[J].International Dairy Journal, 2022, 135:105479.
[2] ZHONG Z, HOU Q, KWOK L, et al.Bacterial microbiota compositions of naturally fermented milk are shaped by both geographic origin and sample type[J].Journal of Dairy Science, 2016, 99(10):7832-7841.
[3] IZADI A, KHEDMAT L, MOJTAHEDI S Y.Nutritional and therapeutic perspectives of camel milk and its protein hydrolysates:A review on versatile biofunctional properties[J].Journal of Functional Foods, 2019, 60:103441.
[4] AYYASH M, AL-DHAHERI A S, AL MAHADIN S, et al.In vitro investigation of anticancer, antihypertensive, antidiabetic, and antioxidant activities of camel milk fermented with camel milk probiotic:A comparative study with fermented bovine milk[J].Journal of Dairy Science, 2018, 101(2):900-911.
[5] AYYASH M, AL-NUAIMI A K, AL-MAHADIN S, et al.In vitro investigation of anticancer and ACE-inhibiting activity, α-amylase and α-glucosidase inhibition, and antioxidant activity of camel milk fermented with camel milk probiotic:A comparative study with fermented bovine milk[J].Food Chemistry, 2018, 239:588-597.
[6] KHAKHARIYA R, SAKURE A A, MAURYA R, et al.A comparative study of fermented buffalo and camel milk with anti-inflammatory, ACE-inhibitory and anti-diabetic properties and release of bioactive peptides with molecular interactions:In vitro, in silico and molecular study[J].Food Bioscience, 2023, 52:102373.
[7] 卓娜, 伊丽, 浩斯娜, 等.基于16S rRNA基因序列分析法比较苏尼特双峰驼和阿拉善双峰驼自然发酵酸驼乳的微生物多样性[J].微生物学报, 2019, 59(10):1948-1959.
ZHUO N, YI L, HAO S N, et al.Application of 16S rRNA high-throughput sequencing for comparative study of the microbial diversity of traditional fermented Bactrian camel milk from Alxa Bactrian camel and Sonid Bactrian camel[J].Acta Microbiologica Sinica, 2019, 59(10):1948-1959.
[8] 柳青, 史迪, 刘文俊, 等.摩洛哥自然发酵驼乳中乳酸菌分离鉴定及特性研究[J].食品科学技术学报, 2022, 40(4):85-95;137.
LIU Q, SHI D, LIU W J, et al.Isolation, identification and characterization of lactic acid bacteria in naturally fermented camel milk from Morocco[J].Journal of Food Science and Technology, 2022, 40(4):85-95;137.
[9] SHORI A B.Camel milk and its fermented products as a source of potential probiotic strains and novel food cultures:A mini review[J].PharmaNutrition, 2017, 5(3):84-88.
[10] ANUSHA SIDDIQUI S, MAHMOOD SALMAN S H, ALI REDHA A, et al.Physicochemical and nutritional properties of different non-bovine milk and dairy products:A review[J].International Dairy Journal, 2024, 148:105790.
[11] GAO Z Z, WU Z Y, ZHANG W X.Effect of pit mud on bacterial community and aroma components in yellow water and their changes during the fermentation of Chinese strong-flavor liquor[J].Foods, 2020, 9(3):372.
[12] 罗爱国, 郗鑫瑞, 郑同庆, 等.基于Illumina MiSeq高通量测序分析清香型白酒酒糟微生物群落多样性[J].中国酿造, 2022, 41(1):98-102.
LUO A G, XI X R, ZHENG T Q, et al.Analysis on diversity of the microbial community of light-flavor Baijiu distiller’s grains based on Illumina Mi Seq high-throughput sequencing[J].China Brewing, 2022, 41(1):98-102.
[13] 赵亮亮, 盛伟喜, 时晓, 等.浓香型白酒酿造生境中微生物群落多样性及群落间相互作用[J].中国酿造, 2023, 42(11):15-21.
ZHAO L L, SHENG W X, SHI X, et al.Research progress on microbial community diversity and interaction in strong-flavor Baijiu brewing habitat[J].China Brewing, 2023, 42(11):15-21.
[14] GAO L, XIE F, REN X, et al.Correlation between microbial diversity and flavor metabolism in Huangshui:A by-product of solid-state fermentation Baijiu[J].LWT, 2023, 181:114767.
[15] 黄和强, 车富红, 陈占秀, 等.青稞大曲微生物菌群的多样性及其核心菌群的判定[J].食品与发酵工业, 2021, 47(23):305-310.
HUANG H Q, CHE F H, CHEN Z X, et al.Determination of microbial diversity and core microbiota in highland barley Daqu[J].Food and Fermentation Industries, 2021, 47(23):305-310.
[16] XIANG F S, CAI W C, HOU Q C, et al.Comparative analysis of the microbial community structure in light-flavor Daqu in Taiyuan and Suizhou regions, China[J].LWT, 2023, 177:114599.
[17] PENG C T, SUN Z H, SUN Y R, et al.Characterization and association of bacterial communities and nonvolatile components in spontaneously fermented cow milk at different geographical distances[J].Journal of Dairy Science, 2021, 104(3):2594-2605.
[18] WANG H, HUANG Y G, HUANG Y L.Microbiome diversity and evolution in stacking fermentation during different rounds of Jiang-flavoured Baijiu brewing[J].LWT, 2021, 143:111119.
[19] 张哲, 刘小鸣, 陈卫.内蒙古传统发酵驼乳中乳酸菌和酵母菌的分离鉴定及其生物多样性分析[J].中国食品学报, 2018, 18(7):230-238.
ZHANG Z, LIU X M, CHEN W.Isolation and identification of lactic acid bacteria and yeast and biodiversity analysis of traditional fermented camel milk in Inner Mongolia[J].Journal of Chinese Institute of Food Science and Technology, 2018, 18(7):230-238.
[20] 熊磊, 赵建新, 范大明, 等.酸驼乳发酵乳酸菌菌株的筛选及其发酵特性研究[J].现代食品科技, 2016, 32(9):84-89;274.
XIONG L, ZHAO J X, FAN D M, et al.Screening and fermentation characteristics of a lactic acid bacterial strain in acid camel milk[J].Modern Food Science and Technology, 2016, 32(9):84-89;274.
[21] 孙羊羊, 尹亚格, 吴雨甍, 等.基于高通量测序技术分析天津高温大曲微生物菌群多样性[J].中国酿造, 2023, 42(5):28-34.
SUN Y Y, YIN Y G, WU Y M, et al.Analysis of microbial community diversity of Tianjin high-temperature Daqu based on high-throughput sequencing technology[J].China Brewing, 2023, 42(5):28-34.
[22] 杨少勇, 贺子豪, 李娜, 等.人工培养窖泥中微生物菌群多样性解析[J].中国酿造, 2023, 42(12):99-103.
YANG S Y, HE Z H, LI N, et al.Microbial flora diversity in artificially cultivated pit mud[J].China Brewing, 2023, 42(12):99-103.
[23] FUGL A, BERHE T, KIRAN A, et al.Characterisation of lactic acid bacteria in spontaneously fermented camel milk and selection of strains for fermentation of camel milk[J].International Dairy Journal, 2017, 73:19-24.
[24] FAHAD Shah, 徐鸿洋, 智超, 等.阿拉善双峰驼酸驼乳中乳酸菌的分离鉴定及耐酸和耐盐研究[J].食品研究与开发, 2020, 41(18):189-194.
SHAH F, XU H Y, ZHI C, et al.Separation, identification, acid-resistent and halotolerant characterization of lactic acid bacteria from shubat of Alashan bactrian camel[J].Food Research and Development, 2020, 41(18):189-194.
[25] 杨博, 柏吉, 靳亚梅, 等.新疆地区驴乳源优良乳酸菌发酵剂的筛选及菌株益生特性[J].食品科学, 2022, 43(2):224-232.
YANG B, BAI J, JIN Y M, et al.Screening of lactic acid bacteria from donkey milk in Xinjiang for use as starter culture and their probiotic characteristics[J].Food Science, 2022, 43(2):224-232.
[26] BAIG M A, TURNER M S, LIU S Q, et al.Heat, cold, acid, and bile salt induced differential proteomic responses of a novel potential probiotic Lactococcus garvieae C47 isolated from camel milk[J].Food Chemistry, 2022, 397:133774.
[27] 代牡兰, 锡林高娃, 吴金花, 等.内蒙古地区传统发酵乳中乳酸菌的分离与鉴定[J].中国乳品工业, 2023, 51(5):31-35.
DAI M L, XI L, WU J H, et al.Isolation and identification of lactic acid bacteria from traditional fermented milk in Inner Mongolia[J].China Dairy Industry, 2023, 51(5):31-35.
[28] 史迪, 党娜, 武岳, 等.阿尤恩地区自然发酵牛乳中乳酸菌分离鉴定及多样性研究[J].中国乳品工业, 2021, 49(3):4-9;41.
SHI D, DANG N, WU Y, et al.Isolation, identification, and biodiversity analysis of lactic acid bacteria in naturally fermented cow milk from Laayoune, Morocco[J].China Dairy Industry, 2021, 49(3):4-9;41.