虾酱作为东亚及东南亚地区著名的传统发酵食品之一,富含蛋白质、氨基酸、虾青素和多种微量元素,以其独特的风味和发酵香气深受消费者喜爱[1-2]。发酵虾酱是用新鲜小虾和食盐作为原辅料,通过附着在原料上的天然微生物自发酿造而成[3]。在虾酱的酿造过程中,微生物主导发酵进程,参与碳水化合物、蛋白质和脂肪酸的代谢,产生小分子鲜味肽、游离氨基酸和脂肪酸等多种代谢产物,直接影响虾酱的品质[4]。解析虾酱酿造过程中微生物群落的变化及其作用,有助于虾酱生产从自发发酵向受控发酵转变,实现品质的稳定。
基于高通量测序的免培养技术被广泛用于探究发酵食品的微生物群落多样性及演替,其只需提取发酵食品的DNA,便能够知晓全面的微生物群落信息,具有高效率、高准确性和高灵敏度的优点[5]。微生物群落演替是指早期定植的微生物群落被后续群落取代,已在多种发酵食品中被观察到[6-8]。微生物群落演替主要受外源环境(如温度和盐度)和内源环境(如酸度和氨基酸态氮含量)的共同影响,其中内源环境与原料的固有成分有关[3,9-10]。段泽瑞等[9]发现不同食盐添加量是引起虾酱微生物群落结构和理化特性差异的主要因素。GAO等[4]发现四联球菌属(Tetragenococcus)和葡萄球菌属(Staphylococcus)是虾酱的优势微生物,影响其理化特性。LYU等[11]发现假交替单胞菌属(Pseudoalteromonas)和发光杆菌属(Photobacterium)是虾酱发酵前期的主要优势微生物,而四联球菌属、葡萄球菌属和海洋芽胞杆菌属(Oceanobacillus)在发酵后期逐渐占据主导地位。目前,关于虾酱酿造过程中微生物群落演替的驱动因素仍不明确。因此,亟需从环境因素的角度深入探讨其驱动机制,为精准调控提供策略。
本研究监测了虾酱酿造过程中内源环境指标的变化,利用高通量测序技术解析微生物群落的演替特征,并通过多元统计分析探究内源环境因子与微生物群落演替之间的互作关系。本研究为控制发酵虾酱微生物群落演替提供理论基础。
1 材料与方法
1.1 材料与试剂
河虾,本地农贸市场,品种为青虾,产地为小阮水库,体表完整、无外伤;精制食用盐,中国盐业集团有限公司;NaOH、酚酞、甲醛、邻苯二甲酸氢钾、MgO、硼酸、三氯乙酸、乙二胺四乙酸二钠、硫代巴比妥酸、三氯甲烷(分析纯),国药集团化学试剂有限公司;乙腈、丙酮、乙酸、乙酸胺(色谱纯),天津科密欧化学试剂有限公司;色胺盐酸盐、酪胺盐酸盐、组胺盐酸盐、苯乙胺盐酸盐等标准品(纯度>98%),上海阿拉丁生化科技股份有限公司;PowerSoil® DNA提取试剂盒,德国QIAGEN公司;16S rRNA V3~V4引物,北京百迈客生物科技有限公司。
1.2 仪器与设备
FE28 pH计、MA204/A电子分析天平,瑞士梅特勒托利多科技公司;HR-06万能高速粉碎机,浙江哈瑞工贸有限公司;1260高效液相色谱仪,美国安捷伦公司;JC-1181多功能酶标仪,青岛聚创环保集团有限公司;DYCP-31CN水平电泳仪、WD-9413B凝胶成像分析系统,北京六一仪器厂。
1.3 方法
1.3.1 虾酱制备
选取新鲜河虾(青绿色,外壳清澈透明、有光泽),去头,洗净沥干,加入质量分数为20%的精制食用盐,置于万能高速粉碎机打浆,加入质量分数为1%的高度白酒,混匀后装入1 L玻璃发酵罐,双层纱布封口,置于春季室外自然发酵35 d,每隔7 d收集样品,将样品分为2份,1份用于DNA提取,1份用于内源环境指标检测。
1.3.2 内源环境测定
pH:称取2 g虾酱,加入10 mL去离子水,涡旋混匀,静置10 min,直接用pH计进行测定;氨基酸态氮:参考GB 5009.235—2016《食品安全国家标准 食品中氨基酸态氮的测定》进行测定;挥发性盐基氮:参考GB 5009.228—2016《食品安全国家标准 食品中挥发性盐基氮的测定》进行测定;丙二醛:参照GB 5009.181—2016《食品安全国家标准 食品中丙二醛的测定》进行测定;色泽:称取2 g虾酱,加入5 mL去离子水,涡旋混匀,静置10 min,用色差仪进行测定。
生物胺:称取5 g虾酱,加入15 mL三氯乙酸溶液(100 g/L),涡旋混匀,4 ℃反应2 h,4 500 r/min离心15 min,收集上清液,虾酱残渣重复提取2次,合并上清液,过0.22 μm微孔滤膜;采用丹磺酰氯进行衍生,N2吹干,加入1 mL乙腈,涡旋混匀,使残留物完全溶解,过0.22 μm微孔滤膜,待测;参考GB 5009.208—2016《食品安全国家标准 食品中生物胺的测定》进行测定。
1.3.3 DNA提取及高通量测序
根据PowerSoil® DNA Isolation Kit说明书对不同发酵时间的虾酱样品中微生物总DNA进行提取,分别采用超微量分光光度计和10 g/L琼脂糖凝胶电泳测定DNA的浓度和完整性,利用正向引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)和反向引物806R(5′-GGACTACHVGGGTWTCTAAT-3′)对微生物16S rRNA V3-V4基因进行聚合酶链式反应(polymerase chain reaction,PCR)扩增,PCR扩增体系:5×FastPfu缓冲液4 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)(2.5 mmol/L)2 μL,上下游引物(5 μmol/L)各1 μL,DNA聚合酶1 μL,DNA模板10 ng,双蒸水(ddH2O)10 μL,补充至20 μL[12];PCR扩增程序为:95 ℃预变性3 min,95 ℃变性30 s,55 ℃退火20 s, 72 ℃延伸30 s,30次循环[12]。用20 g/L琼脂糖凝胶电泳检测扩增结果,并通过切胶回收PCR产物,寄至北京百迈客生物科技有限公司,并委托进行16S rDNA序列高通量测序。
1.3.4 序列分析
应用qiime2[13]生信分析软件处理高通量测序数据,具体如下:使用Trimmomatic插件对原始序列进行质控,与条形码完全匹配的原始序列被分配给各自样品,被识别为优质序列,利用DADA2插件切除测序引物片段,对双端序列进行拼接,并除去嵌合体,获得扩增子序列变体(amplicon sequence variants,ASVs),与Silva 138标准数据库进行比对,对代表性序列进行分类学注释,得到ASV表格[14]。
1.4 统计分析
所有实验重复3次,数据用“平均值±标准差”形式表示;采用GraphPad Prism 10软件进行可视化分析,使用SPSS 21.0软件对实验数据进行单因素方差分析(analysis of variance,ANOVA);利用R 4.0.1软件对不同发酵时间样品的微生物群落α-多样性和β-多样性进行分析,利用主坐标分析(principal co-ordinates analysis,PCoA)、样本聚类分析和相似性分析(analysis of similarities,ANOSIM)对不同发酵时间样品微生物群落差异进行分析,采用线性判别分析(linear discriminant analysis effect size,LEfSe)对不同发酵时间微生物标志物进行鉴定,使用相关性分析和冗余分析(redundancyanalysis,RDA)评估优势微生物属和各内源性环境之间的互作关系。
2 结果与分析
2.1 虾酱发酵过程中内源性环境的变化
新鲜河虾含有活性较高的内源性酶,在发酵过程中能迅速将虾肉中的蛋白质降解为游离氨基酸和小分子肽,有利于虾酱鲜味和香气的形成。pH和氨基酸态氮是评估虾酱发酵过程中微生物代谢活性的重要指标,与其营养价值和感官特性密切相关[9]。由图1可知,虾酱的初始pH值和氨基酸态氮含量分别为7.24和0.06 g/100 g。发酵期间,pH在发酵0~28 d内持续升高,随后在28~35 d内趋于稳定,与GAO等[15]研究结果相一致。发酵35 d后,虾酱的pH值稳定在7.84。水产品中原生微生物,尤其是蛋白质水解菌株,在发酵初期迅速繁殖,产生大量多肽和氨基酸,导致pH值上升[3]。同时,氨基酸等含氮化合物的分解产生挥发性氮类化合物也会促进pH值升高[15]。氨基酸态氮含量与pH变化趋势一致,随着发酵时间的延长,其含量逐渐增加,发酵35 d时可达1.21 g/100 g。随着发酵的不断进行,微生物代谢活性逐渐增强,分泌的胞外蛋白酶增多,使大分子含氮化合物被水解为小分子氨基酸,从而提高虾酱的氨基酸态氮含量[4]。而虾酱发酵后期氨基酸态氮含量变化减缓,可能与碳水化合物的美拉德反应和糖基化反应有关[16]。
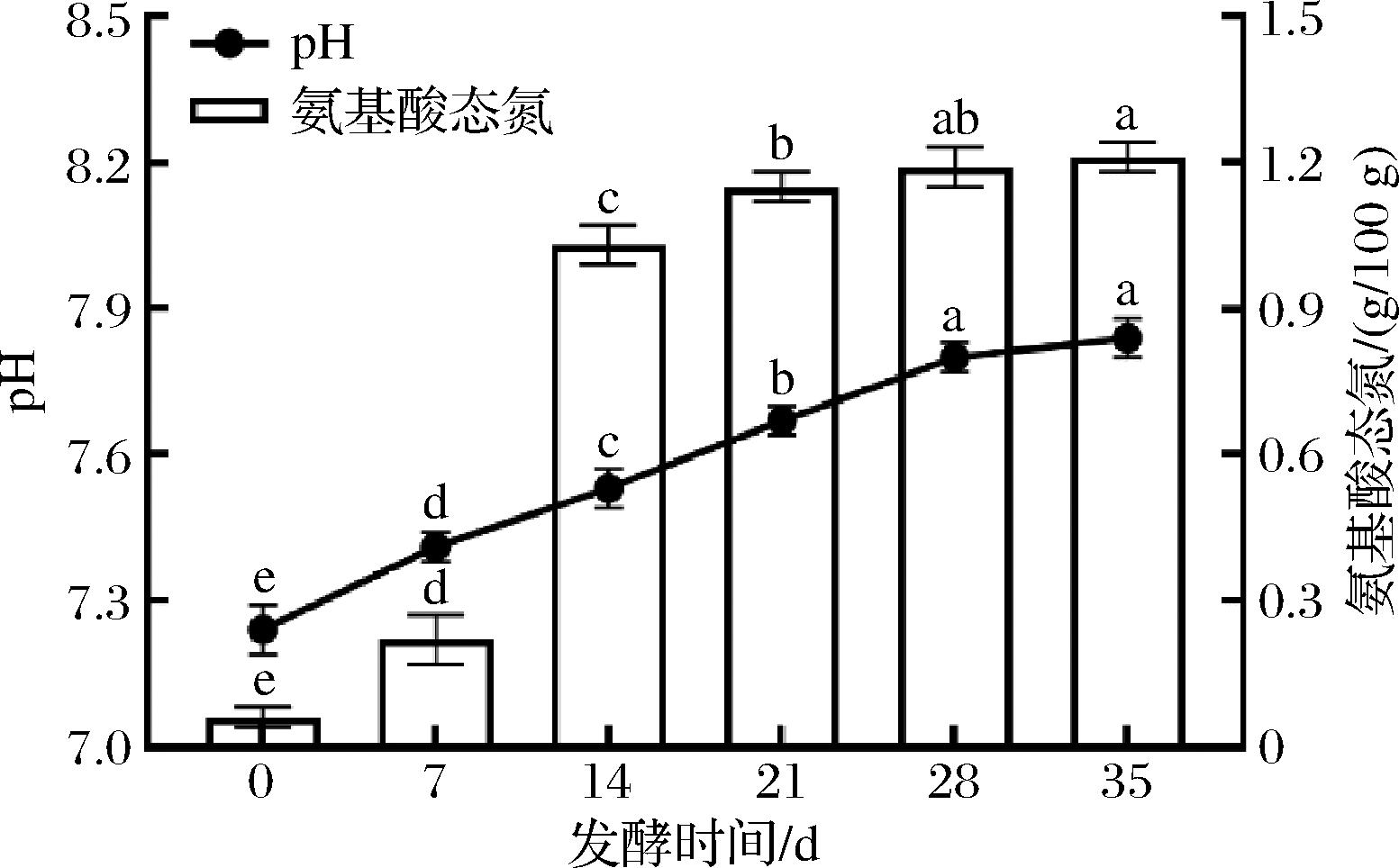
图1 虾酱发酵过程中pH和氨基酸态氮含量的变化
Fig.1 Changes in pH and amino acid nitrogen content during shrimp paste fermentation
注:不同小写字母表示差异显著(P<0.05)(下同)。
挥发性盐基氮和丙二醛是衡量水产品质量和新鲜度的重要指标,与腐败微生物的代谢和内源性酶的活性密切有关[2]。如图2所示,虾酱的初始挥发性盐基氮含量仅为26.52 mg/100 g,随着发酵的进行,挥发性盐基氮含量在发酵0~21 d内迅速增加至81.95 mg/100 g,随后在21~35 d内缓慢增加至90.33 mg/100 g,低于安全限量标准的450 mg/100 g[17]。与腐败相关的蛋白质水解微生物的繁殖,尤其是肠杆菌、光杆菌和乳杆菌,其代谢产物会引起水产品不良挥发性风味物质的增加,带来不良感官特征和潜在的食品安全风险[4]。此外,虾酱的丙二醛含量在发酵0~14 d内快速升高至0.53 mg/kg,随后在14~21 d内迅速增加至1.25 mg/kg,之后随着发酵的不断进行而缓慢增加,并在发酵28~35 d趋于稳定,维持在1.62 mg/kg。丙二醛的增加主要源于脂质氧化,易受到外界发酵环境的影响,导致由自由基介导的脂质氧化,引起食品安全隐患[18]。
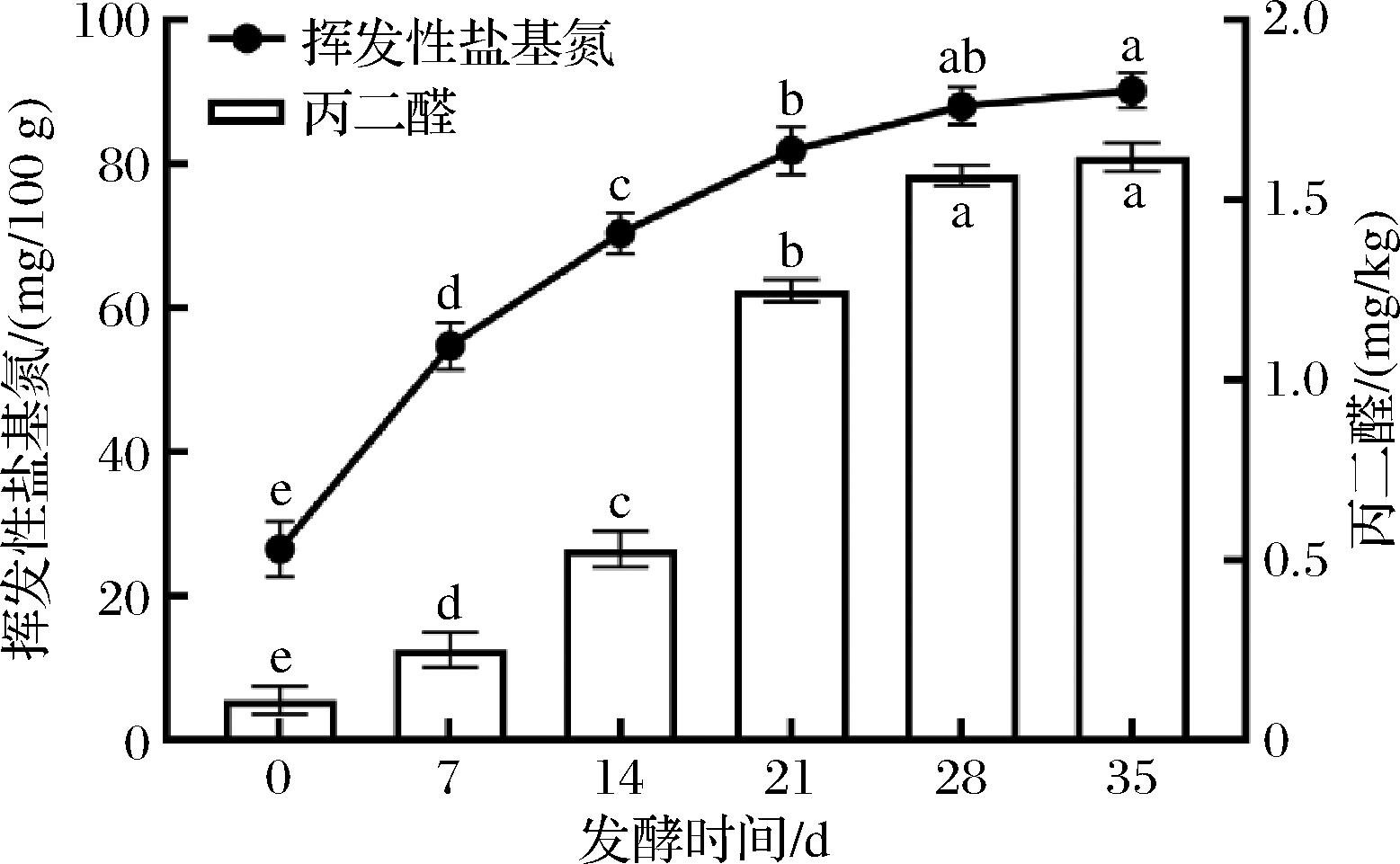
图2 虾酱发酵过程中挥发性盐基氮和丙二醛含量的变化
Fig.2 Changes in volatile basic nitrogen and malondialdehyde contents during shrimp paste fermentation
色泽是评估水产品感官品质的重要指标之一,也是消费者对水产品的第一印象[18]。L*值、a*值和b*值分别表示虾酱的明暗、红绿和黄蓝的程度[18]。根据表1可知,虾酱的初始L*、a*和b*的值分别为58.66、10.35和11.81,与檀茜倩等[18]研究结果相一致。随着发酵时间的延长,虾酱的L*值和b*值逐渐降低,而a*值逐渐升高,表明发酵使虾酱的色泽变暗。发酵35 d后,虾酱的L*、a*和b*的值分别为49.47、11.92和11.03,与发酵初始差异显著(P<0.05)。虾酱色泽变化与美拉德反应产物的形成有关,美拉德反应产物使虾酱呈现棕红色[18]。此外,虾青素含量的变化也会影响虾酱的色泽,发酵过程中微生物分泌蛋白酶破坏虾青素与蛋白质的结合,导致虾青素含量增加,从而改变色泽[9]。
表1 虾酱发酵过程中色泽的变化
Table 1 Changes in color during shrimp paste fermentation
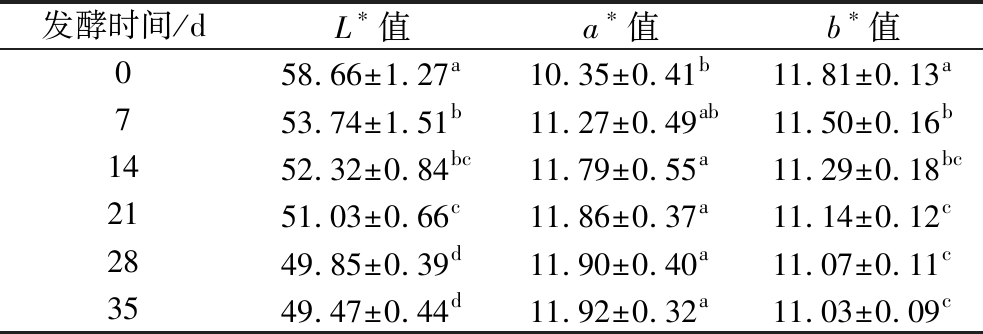
发酵时间/dL∗值a∗值b∗值058.66±1.27a10.35±0.41b11.81±0.13a753.74±1.51b11.27±0.49ab11.50±0.16b1452.32±0.84bc11.79±0.55a11.29±0.18bc2151.03±0.66c11.86±0.37a11.14±0.12c2849.85±0.39d11.90±0.40a11.07±0.11c3549.47±0.44d11.92±0.32a11.03±0.09c
注:同列不同小写字母表示差异显著(P<0.05)(下同)。
生物胺是一类含氮有机化合物,广泛存在于水产品中,过量摄入会带来安全隐患,危害人体健康[19]。生物胺种类繁多,代谢途径和毒性各不相同[19]。通常,新鲜河虾的生物胺含量较低,符合食品安全限量要求。如图3所示,色胺、酪胺、组胺和β-苯乙胺是虾酱发酵过程中主要的生物胺,其初始含量分别为6.37、5.16、2.89和0.74 mg/kg。随着发酵的进行,4种生物胺含量均逐渐增加,但增幅有所不同,其中,色胺和β-苯乙胺变化较大,而酪胺和组胺变化较小。发酵35 d后,色胺和β-苯乙胺含量分别是发酵初始的5.3倍和8.5倍,而酪胺和组胺分别增加了63.18%和96.19%。尽管各国对生物胺的安全限量有所不同,综合来看,β-苯乙胺、组胺和酪胺的安全限量分别为30、100和100~800 mg/kg,且总生物胺含量不应超过900 mg/kg[20]。相比之下,虾酱中生物胺及其总含量均在安全范围内。综上,虾酱的自然发酵显著影响其内源性指标,发酵前后差异显著(P<0.05)。
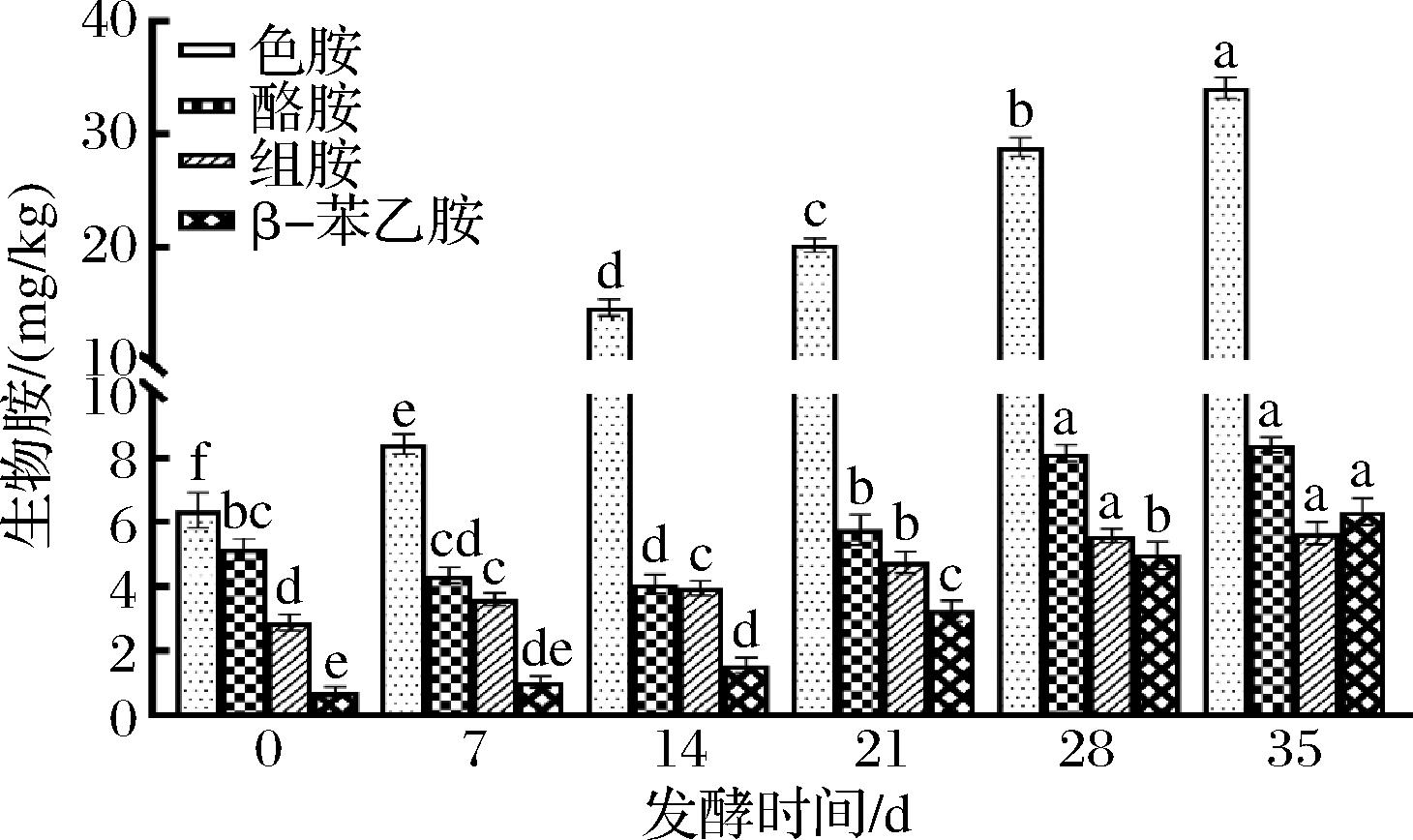
图3 虾酱发酵过程中生物胺含量的变化
Fig.3 Changes in biogenic amine content during shrimp paste fermentation
2.2 虾酱发酵过程中微生物多样性的变化
采用Miseq测序分析不同发酵时间虾酱样品的微生物多样性,由表2可知,共检索到的1 254 948条优质序列,各发酵时间样品的优质序列数目在67 512~74 068条,样品间无显著差异(P>0.05)。微生物群落覆盖率在99.89%~99.97%,表明测序深度足以覆盖虾酱中的大多数微生物,满足后续分析需求[12]。通过Chao1指数和Shannon指数进行Alpha多样性分析,其中,Chao1指数反映物种丰富度,Shannon指数用于反映物种多样性[1]。初始虾酱的Chao1指数和Shannon指数分别为407.19和4.12。随着发酵的进行,两者呈下降趋势。发酵35 d后,虾酱的Chao1指数和Shannon指数分别下降至171.55和2.73,显著低于其他发酵时间的样品(P<0.05)。通常,虾酱的发酵离不开食盐,其高盐度不仅有利于产品的贮藏,也是抑制腐败微生物和限制微生物群落多样性的重要因素[21]。发酵时间延长导致水分流失、盐度增加,抑制不耐盐微生物生长,显著降低微生物数量[17]。总体而言,虾酱发酵过程中原核微生物的丰富度和多样性随时间显著下降(P<0.05)。
表2 虾酱发酵过程中高通量测序质量及Alpha多样性的分析
Table 2 Analysis of high-throughput sequencing quality and Alpha diversity during shrimp paste fermentation
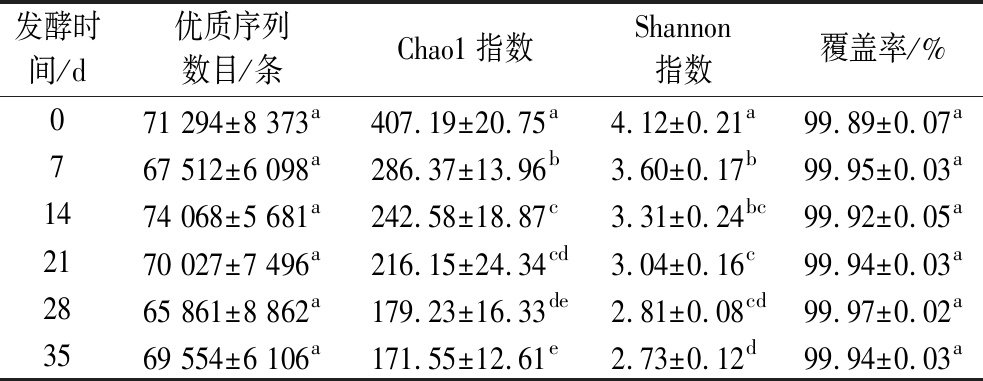
发酵时间/d优质序列数目/条Chao1指数Shannon指数覆盖率/%071 294±8 373a407.19±20.75a4.12±0.21a99.89±0.07a767 512±6 098a286.37±13.96b3.60±0.17b99.95±0.03a1474 068±5 681a242.58±18.87c3.31±0.24bc99.92±0.05a2170 027±7 496a216.15±24.34cd3.04±0.16c99.94±0.03a2865 861±8 862a179.23±16.33de2.81±0.08cd99.97±0.02a3569 554±6 106a171.55±12.61e2.73±0.12d99.94±0.03a
通过基于布雷柯蒂斯距离的PCoA分析虾酱发酵过程中微生物群落组成差异,结果见图4-a。发酵0 d、7 d和14 d的虾酱样品分别位于坐标轴的第三、第二和第一象限,而其他发酵时间的样品位于坐标轴的第四象限,表明虾酱的微生物群落组成具有明显的时间演替规律。这与LI等[3]研究结果相一致。PCoA第一轴可解释物种方差变异为73.87%,而PCoA第二轴则为18.35%,两者之和高达92.22%。如图4-b所示,根据微生物群落组成的相似性,可将虾酱的发酵过程分为3个阶段,包括发酵初期(0 d)、发酵中期(7~14 d)和发酵后期(21~35 d)。发酵时间对组间差异的影响大于组内差异,表明发酵时间是微生物群落演替的主要驱动因素。不同发酵阶段微生物的代谢活动差异导致发酵环境变化,驱动群落演替[11]。综上,虾酱发酵过程中原核微生物群落组成发生了明显变化。
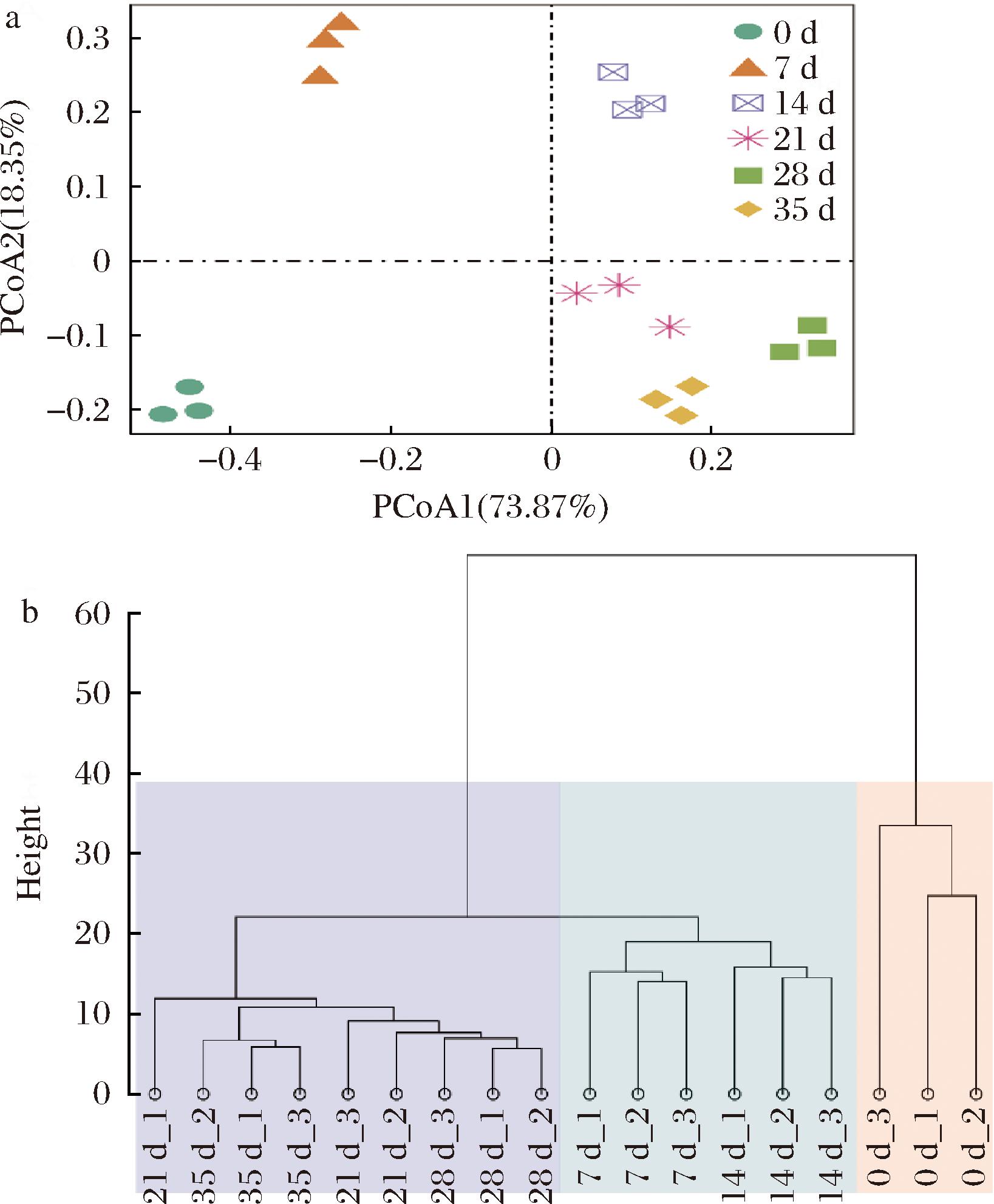
a-主坐标分析;b-样本聚类分析
图4 虾酱发酵过程中原核微生物群落组成
Fig.4 Prokaryotic microbial community during shrimp paste fermentation
2.3 虾酱发酵过程中微生物群落结构的演替
新鲜河虾的微生物群落相对简单且稳定,适合酿造微生物的定向增殖,从而促进优良发酵菌群的生态位优势建立。根据图5可知,所有虾酱共鉴定5个门,分别为厚壁菌门(Firmicutes)、盐厌氧菌门(Halanaerobiaeota)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其中厚壁菌门、盐厌氧菌门和变形菌门的相对丰度之和超过98%,占据主导地位。这一结果与LYU等[11]研究结果相一致。随着发酵的进行,厚壁菌门的相对丰度从发酵初期的29.26%增加至发酵21 d时的60.29%,在发酵35 d时降低至52.04%,而盐厌氧菌门的相对丰度从发酵初期的1.66%持续增加至发酵35 d时的47.92%。变形菌门的相对丰度则持续下降,从发酵初期的67.63%下降至发酵21 d时的1.68%,发酵35 d时几乎检测不到。由此可知,厚壁菌门、盐厌氧菌门和变形菌门是虾酱发酵的主要原核微生物,其丰度变化推动了虾酱的成熟。

图5 虾酱发酵过程中原核微生物群落在门水平的演替
Fig.5 Succession of prokaryotic microbial community at phylum level during shrimp paste fermentation
为进一步解析虾酱发酵过程中原核微生物的群落结构,对属水平进行了分析,如图6所示,虾酱发酵过程中相对丰度较高的是盐厌氧菌属(Halanaerobium)、芽孢杆菌属(Bacillus)、发光杆菌属(Photobacterium)、四联球菌属(Tetragenococcus)、乳杆菌属(Lactobacillus)、肠球菌属(Enterococcus)、假交替单胞菌属(Pseudoalteromonas)、弧菌属(Vibrio)、魏斯氏菌属(Weissella)、漫游球菌属(Vagococcus)、变形杆菌属(Proteus)、嗜盐嗜碱菌属(Alkalibacterium)和盐弧菌属(Salinivibrio)。发光杆菌属、弧菌属和漫游球菌属主要来源于虾的肠道[22],发酵0 d时,虾酱中发光杆菌属、弧菌属和漫游球菌属的相对丰度分别为50.77%、7.73%和9.06%,其中弧菌属是食品中的致病微生物,易引发腹泻、呕吐、头疼及急性胃肠炎等健康问题[23]。随着发酵的进行,这些菌属的相对丰度逐渐下降,发酵35 d后已检测不到,表明虾酱的发酵环境不利于这些微生物的生长繁殖,消除了安全隐患。肠球菌属和魏斯氏菌属具有较强的耐盐特性,对虾酱风味、质地和安全性产生显著影响[4]。魏斯氏菌属和肠球菌属的相对丰度在虾酱发酵过程中呈先增加后下降的变化趋势,分别在发酵7 d和14 d时达到最大,其相对丰度分别为5.37%和18.56%。它们能够利用虾中碳水化合物及蛋白质等营养物质进行生长繁殖,在发酵中期迅速占领生态位,并通过代谢活动产生蛋白酶、酯酶和葡萄糖苷酶,分解原料中大分子物质,为其他微生物的生长提供营养[24]。芽孢杆菌属的相对丰度也呈先增加后下降的变化趋势,从发酵初期的0.36%增加至发酵21 d时的28.31%,随后在发酵35 d时降低至11.55%。芽孢杆菌属利用肠球菌属和魏斯氏菌属代谢产生的小分子营养物质,为原核微生物群落演替提供了新的生态位[25]。此外,芽孢杆菌属适应性强,能够在较广的温度和pH范围内生长,并分泌多种代谢产物,有助于虾酱的成熟[18]。在发酵后期,盐厌氧菌属、四联球菌属和乳杆菌属成为的绝对优势菌群,其相对丰度之和从发酵初期的1.82%增加至发酵14 d时的43.84%,随后持续增加至发酵35 d时的86.78%,这些菌属表现出良好的耐盐性,能够在高盐发酵环境中共存,对虾酱的成熟起到协同作用。乳杆菌属通过产生乳酸和乙酸等有机酸,维持虾酱的pH稳定,抑制腐败菌生长,提升虾酱的风味和保质期。盐厌氧菌属和四联球菌属广泛存在于多种发酵海产品中,能够分解有机物、调节pH、产生风味物质,共同促进发酵产品的成熟[26]。综上,虾酱发酵过程中原核微生物群落结构呈现动态变化。

图6 虾酱发酵过程中原核微生物群落在属水平的演替
Fig.6 Succession of prokaryotic microbial community at genus level during shrimp paste fermentation
2.4 虾酱发酵过程中差异原核微生物的分析
为进一步揭示虾酱发酵过程中不同发酵时间的原核微生物差异,应用LEfSe分析鉴定差异类群,结果见图7。总共存在48个显著差异类群(P<0.05)。其中,变形菌门、γ-变形菌纲(Gammaproteobacteria)、梭菌目(Clostridiales)、拟杆菌科(Bacteroidaceae)、链球菌科(Streptococcaceae)、盐单胞菌属(Halomonas)、Marinilactibacillus、变色菌属(Roseovarius)和弧菌属等在虾酱发酵前期的相对丰度显著高于其他发酵时期,这些微生物主要来源于虾的肠道[22]。在虾酱发酵中期,肠杆菌目(Enterobacteriales)、明串球菌科(Leuconostocaceae)、肠球菌属、魏斯氏菌属和假交替单胞菌属等的相对丰度显著高于其他发酵时期。虾酱发酵后期表现出较高的拟杆菌门、杆菌纲(Bacilli)、盐厌氧菌目(Halanaerobiales)、乳杆菌科(Lactobacillaceae)的相对丰度,这与盐厌氧菌属、四联球菌属、芽孢杆菌属和乳杆菌属在虾酱发酵后期富集有关。因此,虾酱发酵过程中不同发酵微环境对原核微生物类群的选择不同,导致不同发酵时期的类群差异显著。
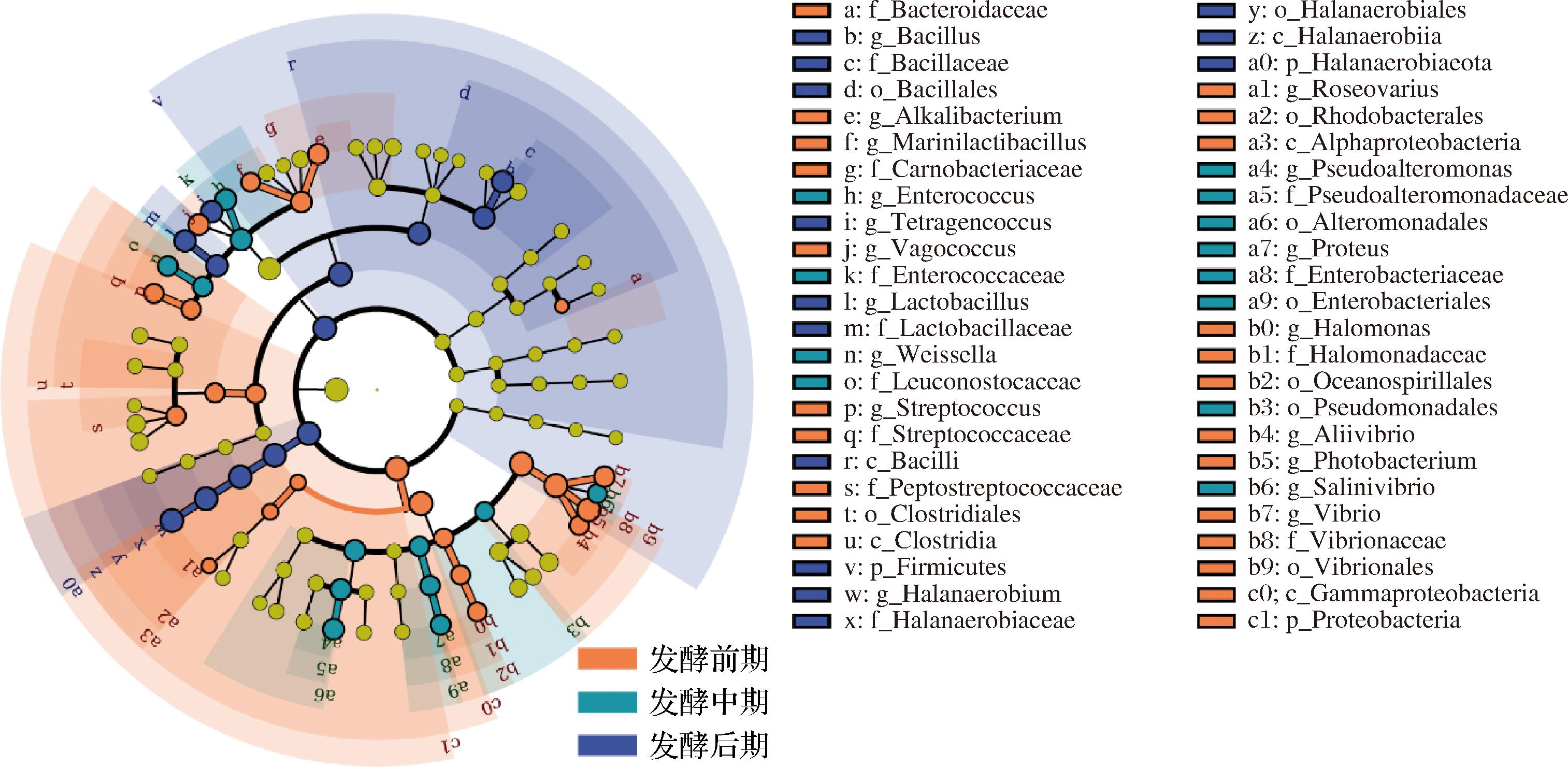
图7 虾酱发酵过程中不同发酵时期差异类群的分析
Fig.7 Analysis of differential taxa in different fermentation periods during shrimp paste fermentation
2.5 虾酱发酵过程中内源环境与原核微生物群落的相关性分析
基于RDA探究虾酱发酵过程中原核微生物群落与内源环境的关系,结果见图8。RDA1和RDA2两排序轴分别解释了75.34%和18.81%的原核微生物群落与内源环境的变异,总共累计解释了94.15%的变异,表明RDA的前2轴能够较为全面地揭示内源环境与虾酱原核微生物群落之间的内在联系。发酵后期的样品聚集在第一象限中,与其他发酵时期样品的距离较远,表明虾酱发酵后期的原核微生物群落组成变化较小,与图4结果相一致。L*和b*值与发酵前期的样品原核微生物群落呈正相关,群落主要由发光杆菌属、漫游球菌属和弧菌属组成;氨基酸态氮含量与发酵中期的群落呈正相关,群落主要由假交替单胞菌属、变形杆菌属和肠杆菌属组成;而多种生物胺、挥发性盐基氮、pH和丙二醛含量与发酵后期的群落呈正相关,群落主要由盐厌氧菌属、四联球菌属、芽孢杆菌属和乳杆菌属组成。此外,L*和b*值与挥发性盐基氮和丙二醛含量呈负相关,这与常规认识相符。通常,新鲜虾色泽鲜亮、晶莹剔透,挥发性盐基氮和丙二醛含量较低,随着发酵的进行,受酶促反应和微生物共同作用下,虾体中蛋白质和脂肪逐渐被分解,生成氨、胺类等碱性含氮物质及醛类化合物,致虾酱色泽显著变化[27]。综上,多个内源环境共同驱动了虾酱发酵过程中原核微生物的群落演替。
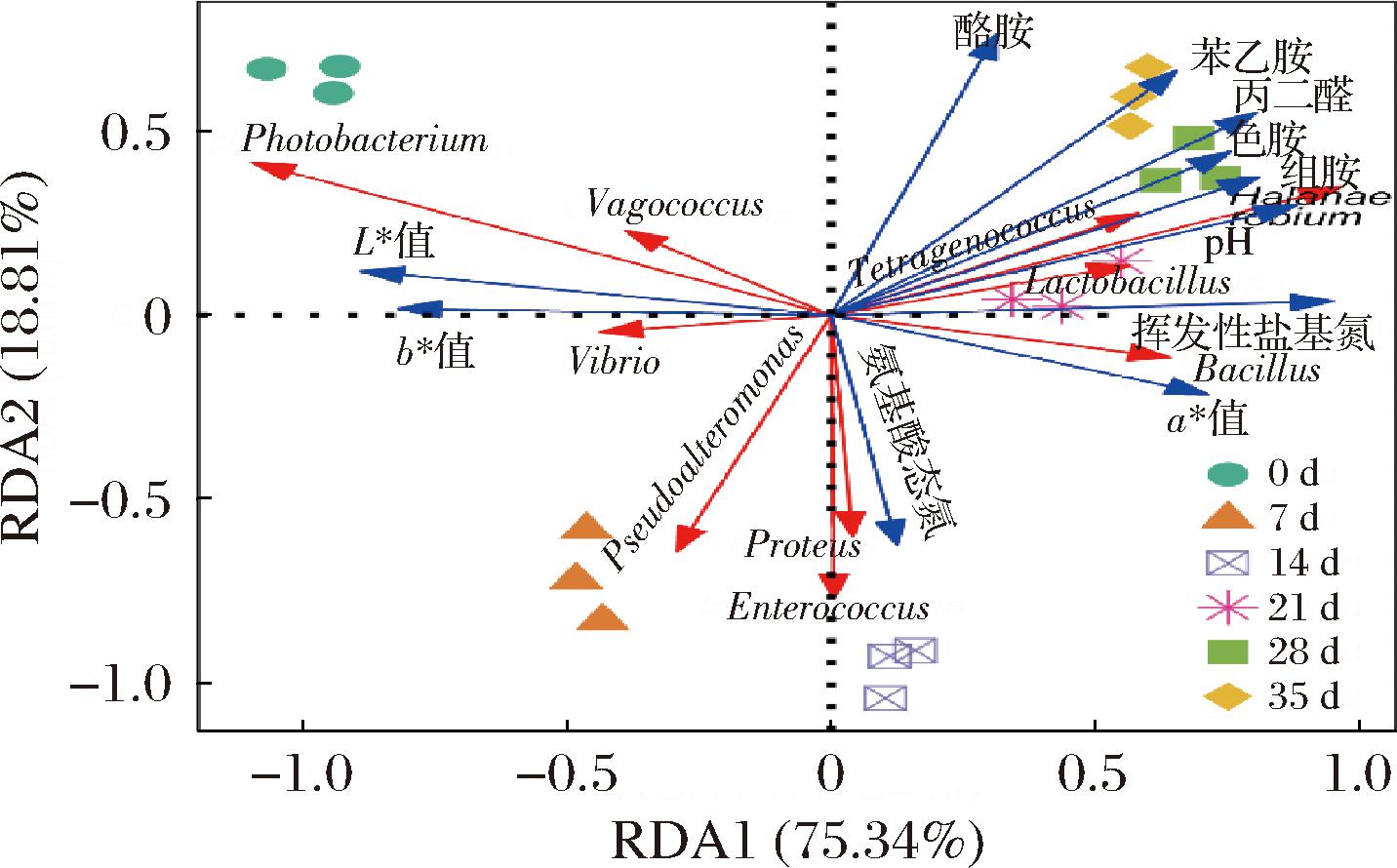
图8 虾酱发酵过程中原核微生物群落与内源环境的冗余分析
Fig.8 Redundancy analysis of prokaryotic microbial community and endogenous environment during shrimp paste fermentation
通过相关性分析进一步阐明内源环境与虾酱发酵过程中优势原核微生物属的关系,结果见图9。虾酱发酵过程中发光杆菌属、嗜碱菌属(Alkalibacterium)、Aliivibrio、漫游球菌属、Marinilactibacillus和弧菌属与L*和b*值呈极显著正相关(P<0.001),与挥发性盐基氮、a*值、pH和多种生物胺呈显著负相关(P<0.05、P<0.01或P<0.001)。这一结果与GAO等[4]的研究结果相一致,表明这些来源于虾肠道的原核微生物无法适应虾酱的发酵环境。此外,魏斯氏菌属与L*和b*值呈显著正相关(P<0.05)。肠球菌属和变形杆菌属与氨基酸态氮呈显著正相关(P<0.05),与酪胺呈显著负相关(P<0.05或P<0.01)。同时,盐厌氧菌属、葡萄球菌属、乳杆菌属、Jeotgalicoccus、四联球菌属和芽孢杆菌属与挥发性盐基氮、a*值、pH和多种生物胺呈显著正相关(P<0.05、P<0.01或P<0.001)。因此,虾酱发酵过程中不同内源环境因子与各原核微生物属之间存在显著差异的互作关系。在虾酱发酵过程中,盐厌氧菌属、四联球菌属、芽孢杆菌属和乳杆菌属等微生物通过分解蛋白质、脂肪和碳水化合物等大分子物质,产生氨基酸、生物胺、丙二醛和有机酸等小分子代谢产物,引起发酵环境的多个内源环境因素发生变化,促进有益发酵菌群优势演替,使发酵过程更加稳定。最终,虾酱呈现出成熟的黏稠状态、紫红色泽以及鲜香浓郁的特征。
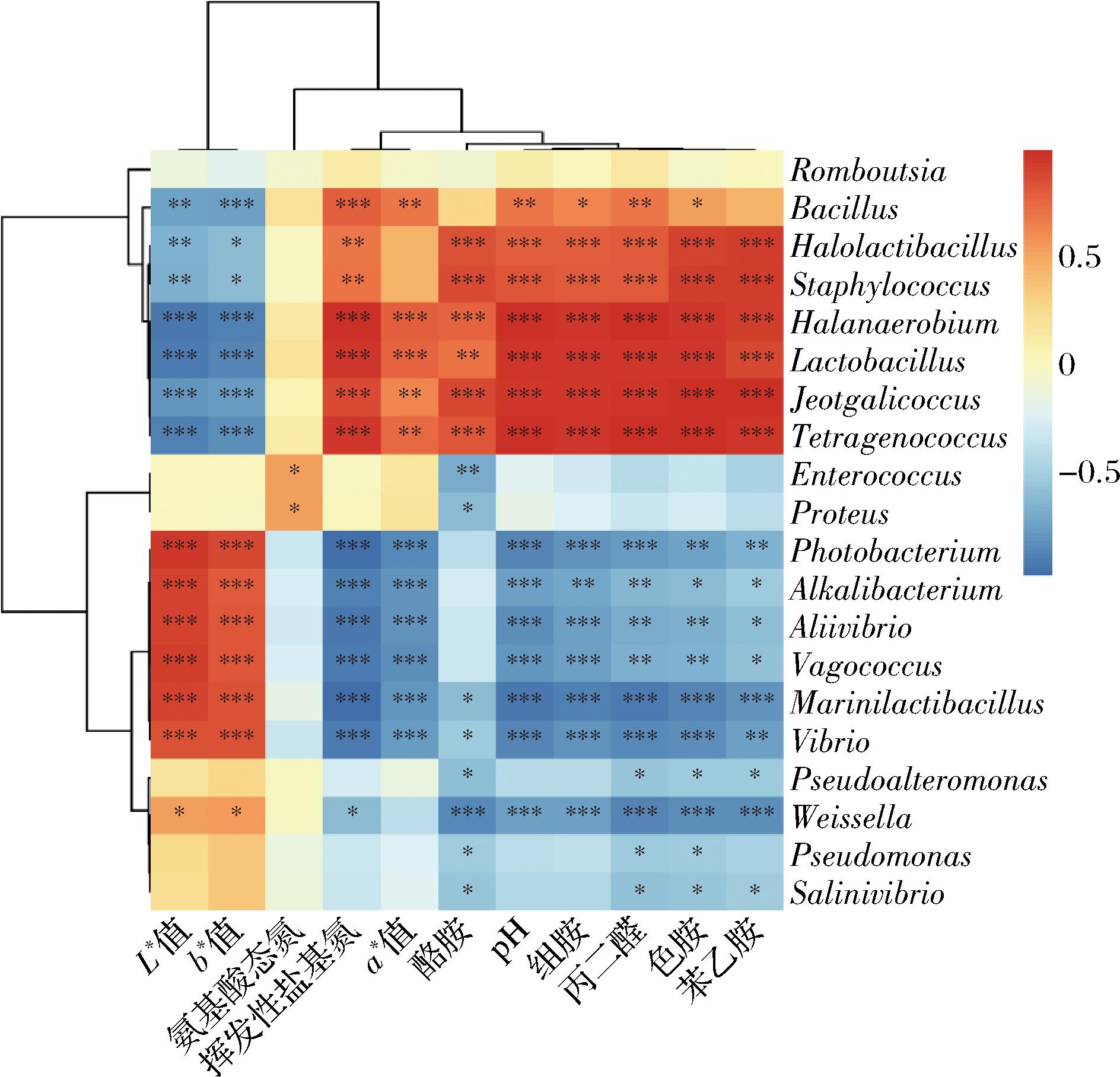
图9 虾酱发酵过程中主要属水平微生物与内源环境之间的相关性分析
Fig.9 Correlation analysis between main genus level microorganisms and endogenous environment during shrimp paste fermentation
3 结论
本研究探究了虾酱发酵过程中原核微生物群落及内源环境的演替规律,确定了两者的互作关系。随着发酵的进行,pH、氨基酸态氮、挥发性盐基氮和丙二醛含量逐渐增加。色胺是虾酱中主要的生物胺,约占总生物胺含量的60%。虾酱原核微生物群落的多样性和丰富度均随发酵时间的延长而逐渐下降。基于原核微生物群落信息,可将整个虾酱发酵过程分为3个发酵时期,不同发酵时期的群落结构和微生物类群存在显著差异。盐厌氧菌属、四联球菌属和乳杆菌属在发酵后期占据绝对优势,发酵35 d后,其相对丰度之和高达86.78%。冗余分析结果表明,多种内源性环境共同驱动了虾酱发酵过程中原核微生物的群落演替,其中盐厌氧菌属、葡萄球菌属、乳杆菌属、四联球菌属和芽孢杆菌属与挥发性盐基氮、a*值、pH和多种生物胺呈显著正相关,与L*和b*值呈显著负相关。综上,本研究深入解析了虾酱发酵过程中原核微生物群落结构的演替及其驱动因素,为改进虾酱发酵工艺提供了理论依据。
[1] 班雨函, 杨兵兵, 马爱进, 等.不同香辛料对低盐虾酱中微生物群落和理化性质的影响[J].食品科学, 2023, 44(8):143-151.
BAN Y H, YANG B B, MA A J, et al.Effects of different spices on microbial community and physicochemical properties in low-salt shrimp paste[J].Food Science, 2023, 44(8):143-151.
[2] LI W Y, LU H Q, HE Z H, et al.Quality characteristics and bacterial community of a Chinese salt-fermented shrimp paste[J].LWT, 2021, 136:110358.
[3] LI Y, LENG W J, XUE J N, et al.A multi-omics-based investigation into the flavor formation mechanisms during the fermentation of traditional Chinese shrimp paste[J].Food Research International, 2023, 166:112585.
[4] GAO R C, LIU H J, LI Y, et al.Correlation between dominant bacterial community and non-volatile organic compounds during the fermentation of shrimp sauces[J].Food Science and Human Wellness, 2023, 12(1):233-241.
[5] 胡梓晴, 刘晓艳, 白卫东, 等.高通量测序技术在白酒微生物多样性中的研究进展[J].中国酿造, 2023, 42(5):15-21.
HU Z Q, LIU X Y, BAI W D, et al.Research progress of high-throughput sequencing technology in microbial diversity of Baijiu production[J].China Brewing, 2023, 42(5):15-21.
[6] 郑雪珂, 陈晖, 朱静, 等.商城筒鲜鱼发酵过程中微生物多样性及品质特性分析[J].食品与发酵工业, 2024, 50(13):91-97.
ZHENG X K, CHEN H, ZHU J, et al.Microbial diversity and quality characteristics analysis of Shangcheng Tongxian fish during fermentation[J].Food and Fermentation Industries, 2024, 50(13):91-97.
[7] 刘敏, 林佳卫, 焦昱玮, 等.基于高通量测序技术分析蒙古族传统奶酪微生物菌群多样性[J].中国酿造, 2024, 43(3):116-120.
LIU M, LIN J W, JIAO Y W, et al.Microbial diversity of Mongolian traditional cheese based on high-throughput sequencing technology[J].China Brewing, 2024, 43(3):116-120.
[8] 王镓璇, 郝淑月, 任清.中国传统发酵食品发酵系统中的未培养微生物研究进展[J].食品与发酵工业, 2023, 49(11):306-314.
WANG J X, HAO S Y, REN Q.Advances in the uncultured microorganisms in the fermentation system of traditional Chinese fermented food[J].Food and Fermentation Industries, 2023, 49(11):306-314.
[9] 段泽瑞, 田梅兰, 刘杨柳, 等.食盐添加量对虾酱理化性质和细菌多样性的影响[J].食品科学, 2024, 45(15):85-93.
DUAN Z R, TIAN M L, LIU Y L, et al.Effect of salt concentration on physicochemical properties and bacterial diversity of shrimp paste[J].Food Science, 2024, 45(15):85-93.
[10] 杨兵兵, 王利文, 陈智慧, 等.发酵温度对低盐虾酱中主要风味物质的影响[J].食品科学, 2023, 44(20):300-308.
YANG B B, WANG L W, CHEN Z H, et al.Effect of fermentation temperature on the main flavor substances in low-salt shrimp paste[J].Food Science, 2023, 44(20):300-308.
[11] LYU X R, LI Y, CUI T Q, et al.Bacterial community succession and volatile compound changes during fermentation of shrimp paste from Chinese Jinzhou region[J].LWT, 2020, 122:108998.
[12] 唐艳平, 高秀兵, 倪婕, 等.白酒发酵副产物黄水原核微生物组成及理化特性研究[J].中国酿造, 2024, 43(3):72-78.
TANG Y P, GAO X B, NI J, et al.Prokaryotic microbial compositions and physicochemical characteristics of Huangshui, a by-product of Baijiu fermentation[J].China Brewing, 2024, 43(3):72-78.
[13] BOLYEN E, RIDEOUT J R, DILLON M R, et al.Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2[J].Nature Biotechnology, 2019, 37(8):852-857.
[14] 杨希. 盐浓度对蚕豆酱发酵过程中原核微生物多样性及理化因子的影响[J].食品与发酵工业, 2022, 48(4):200-206.
YANG X.Effects of salt concentration on prokaryotic microbial diversity and physicochemical properties during broad bean paste fermentation[J].Food and Fermentation Industries, 2022, 48(4):200-206.
[15] GAO R C, XUE J N, SHI T, et al.Effects of ‘bask in sunlight and dewed at night’ on the formation of fermented flavor in shrimp paste after maturation[J].Food Chemistry, 2024, 452:139546.
[16] FAN Y, YIN L A, XUE Y, et al.Analyzing the flavor compounds in Chinese traditional fermented shrimp pastes by HS-SPME-GC/MS and electronic nose[J].Journal of Ocean University of China, 2017, 16(2):311-318.
[17] SANG X, LI K X, ZHU Y L, et al.The impact of microbial diversity on biogenic amines formation in grasshopper sub shrimp paste during the fermentation[J].Frontiers in Microbiology, 2020, 11:782.
[18] 檀茜倩, 王丹, 麻冰玉, 等.芽孢杆菌发酵对虾酱品质和菌群的影响[J].食品工业科技, 2024, 45(21):103-110.
TAN X Q, WANG D, MA B Y, et al.Effect of Bacillus fermentation on the quality and bacteria diversity of shrimp paste[J].Science and Technology of Food Industry, 2024, 45(21):103-110.
[19] W JCIK W,
JCIK W,  UKASIEWICZ M, PUPPEL K.Biogenic amines:Formation, action and toxicity-a review[J].Journal of the Science of Food and Agriculture, 2021, 101(7):2634-2640.
UKASIEWICZ M, PUPPEL K.Biogenic amines:Formation, action and toxicity-a review[J].Journal of the Science of Food and Agriculture, 2021, 101(7):2634-2640.
[20] YANG J, DING X W, QIN Y R, et al.Safety assessment of the biogenic amines in fermented soya beans and fermented bean curd[J].Journal of Agricultural and Food Chemistry, 2014, 62(31):7947-7954.
[21] MOKASHE N, CHAUDHARI B, PATIL U.Operative utility of salt-stable proteases of halophilic and halotolerant bacteria in the biotechnology sector[J].International Journal of Biological Macromolecules, 2018, 117:493-522.
[22] RAJEEV R, ADITHYA K K, KIRAN G S, et al.Healthy microbiome:A key to successful and sustainable shrimp aquaculture[J].Reviews in Aquaculture, 2021, 13(1):238-258.
[23] WU W, JING Z H, YU X Q, et al.Recent advances in screening aquatic products for Vibrio spp[J].TrAC Trends in Analytical Chemistry, 2019, 111:239-251.
[24] WU Q, ZHU Y, FANG C, et al.Can we control microbiota in spontaneous food fermentation?-Chinese liquor as a case example[J].Trends in Food Science &Technology, 2021, 110:321-331.
[25] BLASCHE S, KIM Y, MARS R A T, et al.Metabolic cooperation and spatiotemporal niche partitioning in a kefir microbial community[J].Nature Microbiology, 2021, 6(2):196-208.
[26] BELLEGGIA L, OSIMANI A.Fermented fish and fermented fish-based products, an ever-growing source of microbial diversity:A literature review[J].Food Research International, 2023, 172:113112.
[27] JIANG P F, LIU Y, YANG J Y, et al.Feasibility of circular fermentation as a new strategy to accelerate fermentation and enhance flavor of Antarctic krill paste[J].Current Research in Food Science, 2024, 9:100838.