玛格孜木和波杂分别是新疆伊犁地区哈萨克族和柯尔克孜族的传统谷物发酵饮料,同时也是伊犁特克斯县非物质文化遗产。玛格孜木通常以牧区种植的塔尔米(糜子)或玉米、青稞为原料,经粉碎、煮制后自然发酵调制的“土饮料”,营养丰富,具有醒脑、消暑、抗氧化、治疗失眠和贫血的作用,深受当地群众欢迎[1-2]。波杂是由小米、玉米、大米、小麦等自然发酵而成的一种低酒精度、高酸度、质地浓稠的液体饮料,其具有解腻、促消化等功效[3]。
目前,我国的发酵饮料主要是以乳为基质,谷物为辅的混合型乳酸菌谷物发酵饮料,而单纯以谷物为基质的发酵饮料相对较少,其研究主要在于原料类型的不同而分为传统型(主要以玉米、小麦、大麦等为原料)和新型(主要以藜麦、燕麦、青稞等为原料),并且绝大多数生产工艺仍然采用自然发酵模式[4]。国外对于谷物发酵饮料研究较多,例如西非的发酵食品 Ogi 是以小米、高粱等利用乳酸菌发酵制得的一种口感顺滑,香味独特的食品[5];韩国的 Jeungpyun[6]是以玉米为主要原料经过乳酸发酵制得酸甜可口、口感滑润的米浆制品;其他还包括土耳其的Shalgam[7]、非洲的 Mahewu[8]、保加利亚的波杂[9-10]等。
目前国内外谷物乳酸发酵饮料的发酵工艺仍然以自然发酵为主,生产仍然以家庭或者小作坊制作为主,没有实现工业化生产[11]。自然发酵由于其菌群结构不明确及参与发酵的菌种不纯,发酵过程不可控制等因素,导致产品品质不可控,因而影响其工业化生产[12]。谷物发酵饮料要实现工业化生产,重点方向应在于对参与发酵的有益菌之间相互作用、菌株分离和发酵特性等研究,以及基于菌株间互作关系制成发酵剂从而实现可控的工业化及标准化发酵[13]。因此,利用现代分子生物学技术对谷物发酵饮料进行系统的深入研究,探明其发酵过程中发酵菌株间互作关系,将会进一步促进发酵剂的研制[11]。
因此,本研究使用高通量测序技术对新疆伊犁地区两种传统型自然发酵的谷物饮料中微生物进行研究,以期对这两种谷物发酵饮料中起关键作用的微生物及其功能进行解析,为玛格孜木与波杂饮料发酵剂的研制及该民族饮料工业化、标准化生产提供参考依据。
1 材料与方法
1.1 试剂和仪器
MRS琼脂、马铃薯葡萄糖琼脂(含氯霉素)/PDAC、PCA琼脂培养基,青岛高科技工业园海博生物技术有限公司;Phusion® High-Fidelity PCR MasterMix,安诺伦(北京)生物科技有限公司;通用型DNA纯化回收试剂盒,天根生化科技(北京)有限公司;NEB Next® UltraTM Ⅱ FS DNA PCR-free Library Prep Kit建库试剂盒,纽英伦生物技术(北京)有限公司。
SW-CJ-2D型双人净化工作台,浙江孚夏医疗科技有限公司;HPX-9272MBE电热恒温培养箱,上海博讯实业有限公司;Biometra TAdvanced 96SG梯度高速PCR仪,德国耶拿分析仪器股份公司;DYCP-31DN水平电泳仪,北京六一生物科技有限公司;pHS-25 PH计,上海雷磁仪器厂;2-16R台式高速冷冻离心机,湖南恒诺仪器设备有限公司。
1.2 实验方法
1.2.1 样品采集
玛格孜木样品来自伊犁特克斯县喀拉达拉镇的一户生产玛格孜木产品的家庭作坊(43°13′N,82°5′E),该从业者为哈萨克族非物质文化遗产传承人。波杂样品来自伊犁特克斯县阔克铁热克柯尔克孜族乡的一户生产波杂产品的柯尔克孜族家庭作坊(42°57′N,81°38′E),该从业者也是该产品的非物质文化遗产传承人。采样时间为2023年2月10日(晴),玛格孜木原料为糜米,波杂原料为糜米、高粱、玉米,两者皆室温发酵1 d后,使用无菌离心管从生产车间分别采集发酵完成的玛格孜木(A)与波杂(B)样品,所有样品4 ℃、24 h内运回实验室进一步分析处理,-20 ℃保存。
1.2.2 菌落总数及pH值测定
参考林瑛兰等[14]的方法,样品混匀取10-3、10-4、10-5菌悬液各100 μL分别接种至MRS、PDAC、PCA固体培养基上,每个梯度做3个平行,37 ℃倒置培养48 h后计数。pH值用pH计测定。
1.2.3 基因组DNA提取与PCR扩增
采用CTAB方法对基因组DNA进行提取,每个样品5个重复。利用1%琼脂糖凝胶电泳检测DNA的纯度和浓度,取适量的样本DNA于离心管中,使用无菌水稀释样本至1 ng/μL。细菌16S rRNA基因V4区扩增使用引物对515F (5′-GTGCCAGCMGCC-GCGGTAA-3′)/806R(5′-GGACTACHVGGGTWTCTAAT-3′)[15];真菌ITS1-1F区扩增使用ITS1-1F-F (5′-CTTGGTCATTTAGAGGAAGTAA-3′)/ITS1-1F-R(5′-GCTGCGTTCTTCATCGATGC-3′)。PCR混合液加入15 μL Phusion®High-Fidelity PCR MasterMix、0.2 μmol/L引物和10 ng基因组DNA模板,扩增程序为预变性98 ℃(1 min),然后在98 ℃(10 s)、50 ℃(30 s)和72 ℃(30 s)下进行30次循环,最后在72 ℃下保持5 min。PCR产物使用20 g/L的琼脂糖凝胶进行电泳检测;对检测合格的PCR产物进行磁珠纯化,采用酶标定量,根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物,对目的条带使用通用型DNA纯化回收试剂盒回收产物。
1.2.4 文库构建与上机测序
使用NEB Next® UltraTM Ⅱ FS DNA PCR-free Library Prep Kit建库试剂盒进行文库构建, 构建好的文库经过Qubit和Q-PCR定量,合格后,使用NovaSeq 6000进行PE 250上机测序。
1.2.5 数据质量控制
根据barcode进行拆分获得每个样品的原始数据,并去除barcode和引物,随后通过FLASH(版本1.2.11,http://ccb.jhu.edu/software/FLASH/)[16],对每个样本的reads进行拼接,得到的拼接序列为原始Tags数据(Raw Tags)。使用fastp软件(版本0.23.1)对拼接得到的Raw Tags进行质控得到高质量的Tags数据(Clean Tags)[17]。Tags序列通过与物种注释数据库(Silva database,https://www.arb- silva.de/for 16S/18S; Unite database,https://unite.ut.ee/for ITS)进行比对检测嵌合体序列,去除其中的嵌合体序列,得到最终的有效数据(Effective Tags)[18]。
1.2.6 ASVs降噪和物种注释
对以上得到的Effective Tags,使用QIIME2(版本QIIME2-202006)软件中的DADA2模块进行降噪获得特征序列[19]。采用QIIME2的classify-sklearn算法[17]对每个特征序列使用预先训练好的Naive Bayes分类器进行物种注释(16S rRNA数据库为 Silva 138.1,ITS数据库为Unite v8.2)。使用QIIME2软件进行快速多序列比对,最后对各样本的数据进行均一化处理,以样本中数据量最少的为标准进行均一化处理。
1.2.7 数据分析
基于均一化处理后的数据使用QIIME2 软件分析样品复杂度,计算Observed OTUs、Shannon-Wiener、Simpson、Chao1指数,评估群落组成的复杂性并比较样本之间的差异;使用R软件(版本4.0.3)中的 ade4软件包和ggplot2软件包进行PCoA分析,并进行t检验来实现各个分类水平上的物种显著差异性分析;利用R软件计算得到物种相关性;选用PICRUST2、FunGuild软件对样本中的微生物群落进行功能预测分析;使用Origin 2021软件作图。
2 结果与分析
2.1 菌落总数及pH值
如表1所示,玛格孜木与波杂在PDAC培养基中的活菌数均为106,表明其真菌活菌数没有明显差异,在MRS和PCA培养基上的菌落总数均明显高于PDAC培养基,其中玛格孜木高2个数量级,波杂高1个数量级,说明两种饮料中细菌活菌数更高,在发酵过程中占主导地位。玛格孜木pH值略低于波杂,可能与乳酸菌数量高有关。
表1 样品菌落总数及pH值
Table 1 Total colonies and pH value
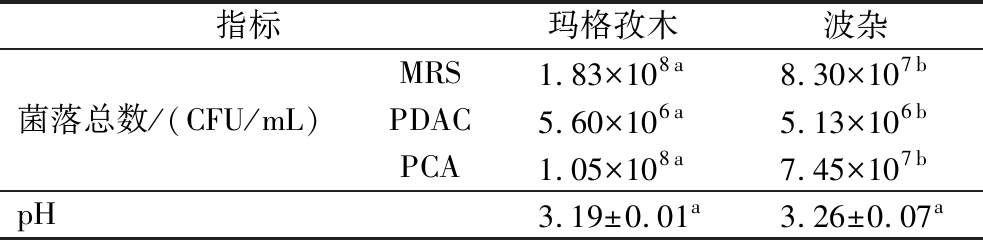
指标玛格孜木波杂菌落总数/(CFU/mL)MRS1.83×108a8.30×107bPDAC5.60×106a5.13×106bPCA1.05×108a7.45×107bpH3.19±0.01a3.26±0.07a
注:不同小写字母表示差异显著(P<0.05)。
2.2 细菌和真菌群落组成
图1显示了门和属水平的注释信息以及相对丰度前10位的类群,在门水平及属水平玛格孜木和波杂中细菌群落结构组成基本相同,但相对丰度差异较大。由图1-a可知,在门水平两组样品均检测出3个优势细菌门(相对丰度>1%),为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和蓝藻门(Cyanobacteria),在玛格孜木中相对丰度分别占48.88%、31.40%和18.15%,而在波杂中分别占90.79%,5.17%和2.35%,波杂中厚壁菌门的丰度远高于玛格孜木。由图1-b可知,在门水平上,玛格孜木检测出2个优势真菌门,为子囊菌门(Ascomycota)和担子菌门(Basidiomycota),分别占78.11%和14.35%;从波杂中检测出3个优势真菌门,为子囊菌门、毛霉门(Mucoromycota)和担子菌门,分别占79.26%、17.16%和2.31%。子囊菌门为两组样品中的绝对优势真菌门。
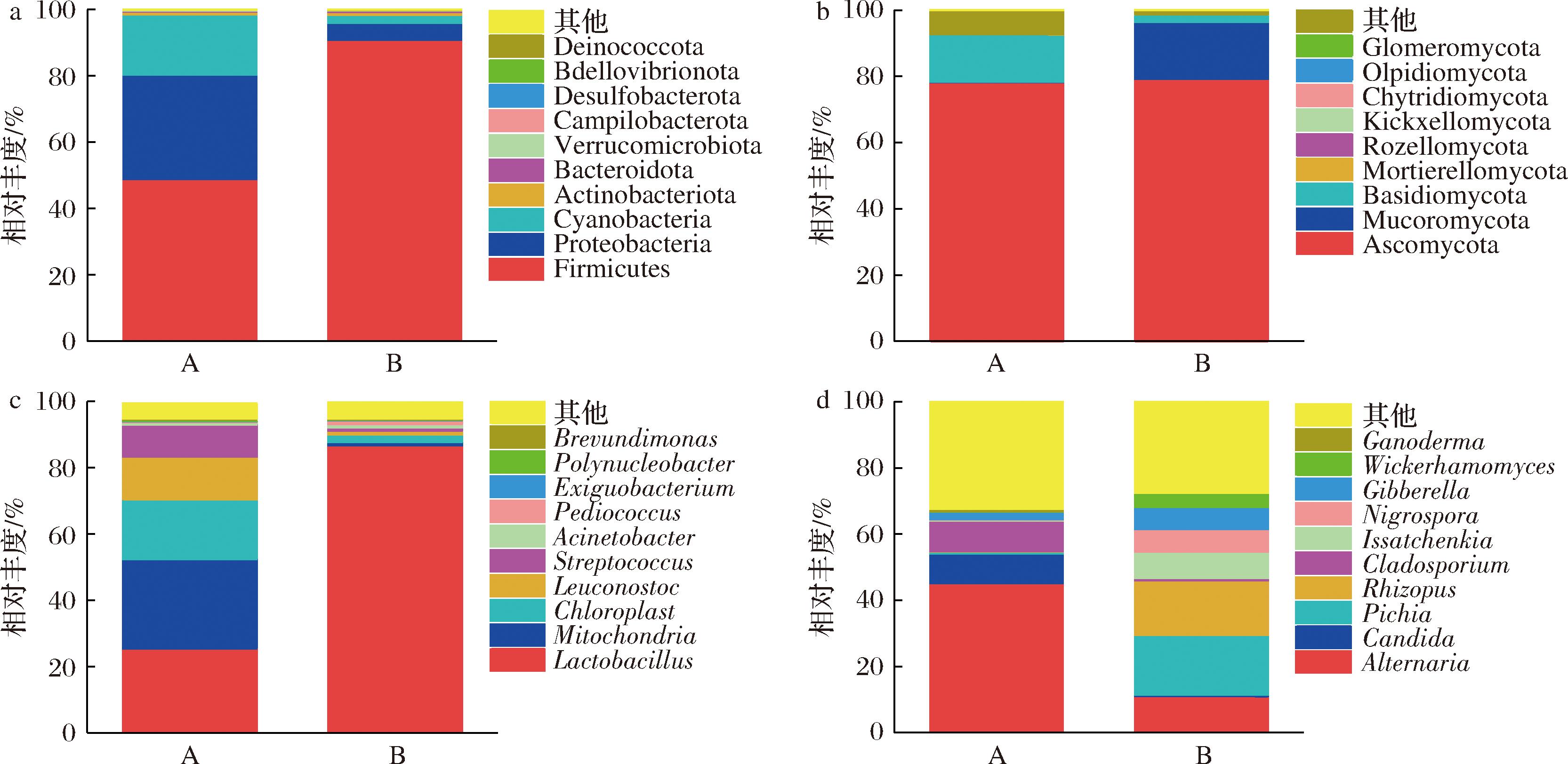
a-细菌门水平;b-真菌门水平;c-细菌属水平;d-真菌属水平
图1 玛格孜木和波杂微生物门水平和属水平的相对丰度
Fig.1 Relative abundance of microbial phyla and genera levels of Magzim and Boza
注:A-玛格孜木;B-波杂(下同)。
由图1-c可知,在属水平,除线粒体(Mitochondria)和叶绿体(Chloroplast)外,玛格孜木中有3个优势细菌属,为乳酸杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)和链球菌属(Streptococcus),相对丰度分别占25.46%、12.88%和9.60%,其余均小于1%;波杂中有5个优势细菌属,除乳酸杆菌属、明串珠菌属、链球菌属外,还有不动杆菌属(Acinetobacter)和片球菌属(Pediococcus),其中乳酸杆菌属丰度达到86.67%,其次片球菌属(1.10%)、不动杆菌属(1.07%)、明串珠菌属(1.06%)和链球菌(1.01%),其他菌属相对丰度较低。由图1-d可知,在玛格孜木中优势真菌属有链格孢属(Alternaria)、枝孢属(Cladosporium)、假丝酵母属(Candida)和赤霉属(Gibberella)分别占45.15%、9.21%、8.99%和2.31%;波杂中的优势真菌属有7个,包括相对丰度18.11%的毕氏酵母属(Pichia),16.41%的根霉属(Rhizopus),10.97%的链格孢属,7.95%的伊萨酵母属(Issatchenkia),6.80%的黑孢霉属(Nigrospora),6.69%的赤霉属(Gibberella),4.37%的威克汉姆酵母属(Wickerhamomyces)。
2.3 细菌和真菌Alpha多样性
由图2可以看出玛格孜木和波杂样本中细菌和真菌的丰度不同。细菌Alpha多样性结果显示波杂的Chao1和Observed OTUs指数均显著高于玛格孜木,而Shannon指数和Simpson指数正好相反,表明波杂的细菌物种丰度显著高于玛格孜木,而玛格孜木的细菌群落多样性更高,物种分布更均匀。真菌Alpha多样性与细菌相反,玛格孜木的Chao1指数和Observed OTUs指数均显著高于波杂,而Shannon-Wiener指数和Simpson指数显著低于波杂,表明玛格孜木的真菌物种丰度高于波杂,而波杂的真菌群落多样性更高,物种分布更均匀。对所有样本的有效数据进行聚类后,图2-e和图2-f结果显示细菌和真菌分别有2 043个和1 723个OTUs;其中,17.62%(360/2 043)和12.83%(221/1 723)是两组样本共有的,说明两组样本之间细菌和真菌群落组成差异显著。

a, b, e-细菌;c, d, f-真菌
图2 玛格孜木和波杂Alpha多样性指数统计与操作分类单元的韦恩图
Fig.2 Alpha diversity index statistics and Venn plot of operational classification units for Magzim and Boza
注:**表示P<0.01,***表示P<0.001。

a-细菌门水平;b-真菌门水平;c-细菌属水平;d-真菌属水平
图3 玛格孜木和波杂的优势菌属相关性
Fig.3 Correlation of dominant genera in Magzim and Boza
2.4 属水平间的相关性分析
选取Top10的微生物属进行Spearman相关系数的计算后,在P<0.05的显著性水平下,细菌及真菌属之间的相关性如图3所示。由图3-a可知,玛格孜木中乳酸杆菌属、链球杆菌属和明串珠菌属3个优势细菌属与其他细菌属共有14对呈正相关,10对呈负相关。其中乳酸杆菌属与马赛菌属(Massilia)呈现显著正相关;链球杆菌属与不动杆菌属(Acinetobacter)呈现显著正相关;明串珠菌属与其他菌均呈现负相关,并且与马赛菌属呈现显著负相关。由图3-b可知,波杂中5个优势细菌属与其他细菌属共有22对呈正相关,12对呈负相关。与玛格孜木不同的是,除不动杆菌属外,乳酸杆菌属与其他属均呈负相关,特别是与明串珠菌属为显著负相关;链球菌属与假单胞菌属显著正相关。
由图3-c可以看出,玛格孜木中链格孢属、枝孢属、假丝酵母属和赤霉属4个优势真菌属与其他真菌属共有6对呈正相关,12对呈负相关,有4对相关系数为0,其中链格孢属与假丝酵母属显著负相关;枝孢属与根霉属显著负相关。另外毕赤酵母属与根霉属、黑孢霉属均极显著正相关。由图3-d可知,波杂中的7个优势真菌属与其他真菌属共有13对呈正相关,18对呈负相关,有1对相关系数为0。其中链格孢属与赤霉属、威克汉姆酵母属均为显著正相关,与伊萨酵母属显著负相关;毕赤酵母属与伊萨酵母属、黑孢霉属显著负相关;伊萨酵母属与威克汉姆酵母属显著负相关,黑孢霉属与赤霉属显著正相关;威克汉姆酵母属与赤霉属也显著正相关。
2.5 细菌和真菌群落的功能预测
根据样品在数据库中的功能注释及丰度信息,选取丰度排名前25的功能信息生成功能相对丰度柱状堆积图。如图4-a所示,代谢相关基因的丰度在两组样本中最高,达到46%,说明代谢在微生物群落中起着重要作用,微生物的快速生长与代谢基因的丰度密切相关[20]。2级KEGG通路分析如图4-b所示,所有细菌种群都存在复制和修复、膜运输、碳水化合物和氨基酸代谢相关的基因,且相对丰富≥8%,每一组样本中的核苷酸代谢、能量代谢、辅助因子和维生素代谢以及脂质代谢途径也较丰富,凸显了微生物的代谢和快速生长等功能的生物学意义。经FUNGuild平台解析,除未定类群(unassigned)外,所有样本共涉及病原型、腐生型、病原-腐生型、病原-共生型、腐生-共生型、病原-腐生-共生型6种营养方式的41个生态功能群如图4-c所示。玛格孜木以动物病原体-内生菌-植物病原体-木材腐生菌(animal pathogen-endophyte-plant pathogen-wood saprotroph)(50.00%) 为主,其次为内生菌-植物病原体(endophyte-plant pathogen)(10.40%)。波杂主要包括动物内共生未定义腐生体(animal endosymbiont-undefined saprotroph)(17.71%)及动物病原体-内生菌-植物病原体-木材腐生菌(animal pathogen-endophyte-plant pathogen-wood saprotroph)(11.37%)。

a-细菌功能注释柱形图(第1级);b-细菌功能注释柱形图(第2级);c-真菌功能注释柱形图(Guild)
图4 功能注释相对丰度柱形图
Fig.4 Bar graph of the relative abundance of the functional annotation

a-细菌;b-真菌
图5 Spearman相关性分析热图
Fig.5 Heat map of the Spearman Correlation analysis
2.6 物种多样性与pH值、经纬度的关系
为进一步明确物种与环境因子的相互变化关系,对属分类单元丰度前25的细菌和真菌分别与环境因子进行 Spearman 秩相关分析,结果如图5所示,乳酸杆菌属、片球菌属、魏斯氏菌属、根霉属、毕赤酵母属、伊萨酵母属、黑孢霉属、赤霉属、威克汉姆酵母属、棒孢酵母属与pH值呈显著正相关(P<0.05),与经纬度呈显著负相关(P<0.05)。而明串珠菌属、链球菌属、乳球菌属、链格孢属、枝孢菌属、线黑粉酵母属、球腔菌属、平脐蠕孢属、维希尼克氏酵母属与pH值呈显著负相关(P<0.05),与经纬度呈显著正相关(P<0.05)。
3 讨论
3.1 微生物群落组成及多样性
通过对细菌16S rRNA基因,真菌ITS基因的测序,可以有效地揭示样品中微生物的群落结构。然而,到目前为止,新疆伊犁地区的玛格孜木的微生物多样性尚未见报道,对波杂样品的微生物分析多采用传统可培养法结合分子生物学分析,关于其微生物多样性分析尚不充分。因此,本研究通过对16S rRNA和ITS基因的高通量测序,分析了来自伊犁特克斯县的10份样本的微生物群落组成、高通量测序结果清楚地表明,玛格孜木中的厚壁菌门、变形菌门为优势细菌门,优势细菌属有3个,分别为乳酸杆菌属、明串珠菌属、链球菌属;优势真菌属有4个,包括链格孢属、枝孢属、假丝酵母属和赤霉属。这一结果与同样以糜米为主要原料的酸粥相似,王玉荣等[20-21]研究结果表明厚壁菌门和变形菌门均为内蒙古鄂尔多斯地区的酸粥的优势细菌门,但相对丰度不同,其厚壁菌门丰度约为玛格孜木的两倍;优势菌属差别较大,酸粥中的乳酸杆菌属是玛格孜木的3倍多;王成等[22]、史学敏等[23]利用高通量测序技术对不同地区酸粥样品进行多样性分析,同样得到乳酸杆菌属在各样品中占比最高,为42.63%~97.49%,都高于玛格孜木,但玛格孜木物种更丰富。而酸粥优势真菌属主要为84.3%的假丝酵母属[20-21],其相对丰度是玛格孜木的3倍多。
波杂有5个优势细菌属(乳酸杆菌属、明串珠菌属、链球菌属、不动杆菌属、片球菌属),7个优势真菌属(毕氏酵母属、根霉属、链格孢属、伊萨酵母属、黑孢霉属、赤霉属、威克汉姆酵母属)。波杂中原料种类较多,这或许是影响其物种组成的因素之一。国内外已有较多关于波杂的研究,但与本实验结果仍有不同。UCAK等[9]对土耳其的波杂样品进行宏基因组分析,发现乳球菌属、乳酸杆菌属、明串珠菌属和链球菌属为优势属,富集后乳球菌属、肠球菌属、大肠埃氏菌属-志贺氏菌属、芽孢杆菌和乳杆菌属占优势。OSIMANI等[24]采用可培养法从索非亚波杂中分出副干酪乳杆菌亚属、乳酸杆菌属、毕赤酵母属;从科伊纳雷的波杂中分离出乳杆菌属、片球菌属、副干酪乳杆菌亚属、季也蒙迈耶氏酵母菌属、毕赤酵母属及有孢圆酵母属;从保加利亚波杂中分离出副干酪乳杆菌亚属、乳酸杆菌属、毕赤酵母属、有孢圆酵母属;阿特尔古丽·沙布尔江[25]采用传统可培养法对新疆5个县自然发酵的波杂进行分离,鉴定出魏斯氏菌属(6.25%)、片球菌属(31.25%)、乳球菌属(12.5%)、乳酸杆菌属(50.00%);真菌有3个属,包括酿酒酵母菌属(83.33%)、毕赤酵母属(13.33%)、隐球酵母属(3.33%)。总之,乳酸杆菌属在多数谷物自然发酵饮料中属于绝对优势菌群,当然不同地域自然发酵菌种存在一定的差异性,可能与不同地区的环境、温度、发酵原料,水源,样品分析处理方法等因素有关[9-10, 22]。
3.2 微生物群落组成相关性分析
细菌及真菌属水平间的相关性结果表明,玛格孜木中的乳酸杆菌属、链球菌属与片球菌属之间具有相互促进作用,与明串珠菌属具有抑制作用;假丝酵母属对病原菌链格孢属和镰刀菌属极显著负相关,酿酒酵母属对曲霉属、线黑粉酵母属与地霉属具有抑制作用,而毕赤酵母属与根霉属相互促进。与玛格孜木不同的是波杂中的乳酸杆菌属与其他属均呈负相关,对条件致病菌黄杆菌属极显著负相关,邹恬杏等[26]同样发现酒醅中的乳杆菌属与其他细菌属均呈现负相关,可能是因为乳酸菌在谷物发酵过程中产生乳酸、乙酸及细菌素等代谢产物,抑制其他微生物的生长[27]。
环境因素对微生物群落组成也有影响,本实验结果中乳酸杆菌属、片球菌属、魏斯氏菌属、根霉属、毕赤酵母属、伊萨酵母属、黑孢霉属、赤霉属与pH值呈显著正相关,胡此海等[28]分析泡菜母水中优势微生物和理化特性的相关性结果相似,乳酸杆菌属、毕赤酵母属与pH值正相关,并且乳酸杆菌属与乳酸和乙酸具有极显著正相关性。
3.3 优势微生物群落功能预测
PICRUSt2可以根据16S rRNA测序数据进行基于KEGG数据库的功能预测,包括细菌在代谢、遗传信息处理、环境信息处理、细胞过程、人类疾病和生物体系统6个领域的生物学功能。FUNGuild是基于ITS的功能预测软件,根据真菌的营养方式将真菌分为三大类:病理营养型(pathotroph)、共生营养型(symbiotroph)、腐生营养型(saprotroph)。基于三大营养方式,又进一步细分为若干个guilds,其中包括动物病原菌、植物病原菌、木质腐生菌等12类。功能预测分析显示,所有细菌种群都存在复制和修复、膜运输、碳水化合物和氨基酸代谢相关的基因,且相对丰富≥8%,每一组样本中的核苷酸代谢、能量代谢、辅助因子和维生素代谢以及脂质代谢途径也较丰富,该结果与夏亚男等[29]对酸马奶中微生物功能预测结果相似。玛格孜木和波杂均注释出一些与人类疾病相关的基因,动、植物病原菌及木质腐生菌差异显著,可能与链格孢属、黑孢属、赤霉属、曲霉属、镰刀菌属、球腔菌属等病原菌的分布有关。通过对细菌和真菌群落的功能预测,可以进一步评估样品中细菌和真菌携带致病基因的潜在风险,以确保不同发酵食品的可食用安全性。
4 结论
本文对新疆伊犁地区民族传统谷物发酵饮料的微生物多样性和功能预测方面进行了深入研究。结果表明不同原料类型的自然发酵谷物饮料中微生物群落组成,多样性和功能差异显著,原料单一的玛格孜木存在3个优势细菌属和4个优势真菌属;3种原料发酵的波杂有5个优势细菌属和7个优势真菌属,玛格孜木与波杂微生物种群分布具有显著差异,波杂的细菌物种丰度显著高于玛格孜木,玛格孜木的真菌物种丰度显著高于波杂。细菌对于发酵的影响起主导作用,主要参与碳水化合物和氨基酸代谢,但也存在危害人体健康的基因,此外,微生物间相互作用显著影响谷物发酵品质和安全性。本研究具有一定的实践和理论价值,为新疆少数民族谷物发酵饮料发酵剂的研制及工业化生产和饮料安全性评价提供参考,对于推动相关领域的发展和促进当地传统文化的保护与传承具有重要意义。
[1] 刘玉梅, 曹军, 王萱, 新疆哈萨克传统食品塔尔米营养成分研究[J].食品工业科技,2009,30(3):310-312.
LIU Y M, CAO J, WANG X.Study on nutritional components of Ta-er-mi from a traditional food of Kazakh in Xinjiang[J].Science and Technology of Food Industry, 2009, 30(3):310-312.
[2] 赵正梅, 拉升·再尼西, 敬思群,等.塔尔米的抗氧化性及其在苏打饼干中的应用[J].食品与发酵工业,2017,43(9):166-170.
ZHAO Z M, ZAINIXI L, JING S Q, et al.Antioxidant effect of Ta-er-mi and its application in soda biscuit[J].Food and Fermentation Industries, 2017, 43(9):166-170.
[3] 努尔古丽·热合曼, 华长春, 朱晓莹,等.新疆柯尔克孜族传统发酵饮料博扎中微生物群落结构的PCR-DGGE分析[J].食品科学,2012,33(1):111-114.
RHMAN N, HUA C C, ZHU X Y, et al.PCR-DGGE analysis of m icrobial community structure of Boza, a traditional Kyrgyz fermented beverage[J].Food Science, 2012, 33(1):111-114.
[4] 张瑞, 刘敬科, 常世敏, 等.谷物饮料的研究进展[J].食品科技, 2023, 48(8):152-158.
ZHANG R, LIU J K, CHANG S M, et al.Research progress on grain beverages[J].Food Science and Technology, 2023, 48(8):152-158.
[5] CHIBUZOR-ONYEMA I E, EZEOKOLI O T, SULYOK M, et al.Metataxonomic analysis of bacterial communities and mycotoxin reduction during processing of three millet varieties into Ogi, a fermented cereal beverage[J].Food Research International, 2021, 143:110241.
[6] JANG S, SHIN W K, KIM Y.Texture of steamed rice cake prepared via soy residue and hydroxypropyl methylcellulose supplementation[J].Cereal Chemistry, 2019, 96(1):57-65.
[7] COSKUN F.A traditional Turkish fermented non-alcoholic beverage, “Shalgam”[J].Beverages, 2017, 3(4):49.
[8] PSWARAYI F, G NZLE M G.Composition and origin of the fermentation microbiota of Mahewu, a Zimbabwean fermented cereal beverage[J].Applied and Environmental Microbiology, 2019, 85(11):e03130-e03118.
NZLE M G.Composition and origin of the fermentation microbiota of Mahewu, a Zimbabwean fermented cereal beverage[J].Applied and Environmental Microbiology, 2019, 85(11):e03130-e03118.
[9] UCAK S, YURT M N Z, TASBASI B B, et al.Identification of bacterial communities of fermented cereal beverage Boza by metagenomic analysis[J].LWT, 2022, 153:112465.
[10] ALTAY F, KARBANC OGLU-GÜLER F, DASKAYA-DIKMEN C, et al.A review on traditional Turkish fermented non-alcoholic beverages:Microbiota, fermentation process and quality characteristics[J].International Journal of Food Microbiology, 2013, 167(1):44-56.
OGLU-GÜLER F, DASKAYA-DIKMEN C, et al.A review on traditional Turkish fermented non-alcoholic beverages:Microbiota, fermentation process and quality characteristics[J].International Journal of Food Microbiology, 2013, 167(1):44-56.
[11] 陈雨春, 王妍, 王畅, 等.谷物乳酸发酵饮料工艺研究进展[J].食品安全导刊, 2020(33):155-156.
CHEN Y C, WANG Y, WANG C, et al.Research progress of cereal lactic acid fermented beverage technology[J].China Food Safety Magazine, 2020(33):155-156.
[12] BYAKIKA S, MUKISA I M, BYARUHANGA Y B, et al.Influence of food safety knowledge, attitudes and practices of processors on microbiological quality of commercially produced traditional fermented cereal beverages, a case of Obushera in Kampala[J].Food Control, 2019, 100:212-219.
[13] TEKLE B, ANURADHA JABASINGH S, FANTAW D, et al.An insight into the Ethiopian traditional alcoholic beverage:Tella processing, fermentation kinetics, microbial profiling and nutrient analysis[J].LWT, 2019, 107:9-15.
[14] 林瑛兰, 张悦, 李阔, 等.传统酸粥中乳酸菌的分离筛选及其体外抗氧化能力分析[J].食品工业科技, 2023, 44(20):126-134.
LIN Y L, ZHANG Y, LI K, et al.Screening lactic acid bacteria in traditional sour porridge and analysis of their antioxidant activity[J].Science and Technology of Food Industry, 2023, 44(20):126-134.
[15] LEE H B, JEONG D H, CHO B C, et al.Comparative analyses of eight primer sets commonly used to target the bacterial 16S rRNA gene for marine metabarcoding-based studies[J].Frontiers in Marine Science, 2023, 10:1199116.
[16] MAGO T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2957-2963.
T, SALZBERG S L.FLASH:Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2957-2963.
[17] BOKULICH N A, SUBRAMANIAN S, FAITH J J, et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nature Methods, 2013, 10(1):57-59.
[18] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2194-2200.
[19] WANG Y Y, GUO H, GAO X G, et al.The intratumor microbiota signatures associate with subtype, tumor stage, and survival status of esophageal carcinoma[J].Frontiers in Oncology, 2021, 11:754788.
[20] 王玉荣, 折米娜, 刘康玲,等.内蒙古鄂尔多斯地区酸粥真菌多样性研究[J].中国酿造,2018,37(8):50-55.
WANG Y R, ZHE M N, LIU K L, et al.Fungnl divecsity in acidic-gruel from Ordos area of Inner Mongolia[J].China Brewing, 2018, 37(8):50-55.
[21] 王玉荣, 折米娜, 刘康玲,等.基于Mi Seq高通量测序技术内蒙古地区酸粥细菌多样性研究[J].食品工业科技,2018,39(19):124-129.
WANG Y R, ZHE M N, LIU K L, et al.Diversity of bacteria m icroflora in acidic-gruel of Inner Mongolia area by MiSeq high throughput sequencing[J].Science and Technology of Food Industry, 2018, 39(19):124-129.
[22] 王成, 肖晗, 郭浩楠, 等.自然发酵酸粥中细菌多样性分析及优势乳酸菌的分离与鉴定[J].食品科技, 2020, 45(10):46-52.
WANG C, XIAO H, GUO H N, et al.Bacterial diversity analysis, isolation and identification of dominant lactic acid bacteria in natural fermented sour porridge[J].Food Science and Technology, 2020, 45(10):46-52.
[23] 史学敏, 刘珊娜, 杜茜.鄂尔多斯地区酸粥细菌多样性分析及乳酸菌的分离鉴定[J].食品工业科技, 2023, 44(9):153-159.
SHI X M, LIU S N, DU X.Bacterial diversity analysis of sour porridge from Ordos and isolation and identification of lactic acid bacteria[J].Science and Technology of Food Industry, 2023, 44(9):153-159.
[24] OSIMANI A, GAROFALO C, AQUILANTI L, et al.Unpasteurised commercial boza as a source of microbial diversity[J].International Journal of Food Microbiology, 2015, 194:62-70.
[25] 阿特尔古丽·沙布尔江.新疆波杂中优良发酵菌种选育及其发酵工艺的优化[D].乌鲁木齐:新疆农业大学, 2017.
ATEERGULI S B E J.Selection of excellent fermentation strains and optimization of fermentation process of Boza in Xinjiang[D].Urumqi:Xinjiang Agricultural University, 2017.
[26] 邹恬杏, 苏伟, 母应春, 等.洞酿酱香酒第四轮次酒醅发酵过程中微生物群落与挥发性代谢物分析[J].中国酿造, 2023, 42(6):64-71.
ZOU T X, SU W, MU Y C, et al.Analysis of microbial community and volatile metabolites during the fourth round fermented grains fermentation of Dongniang sauce-flavor Baijiu[J].China Brewing, 2023, 42(6):64-71.
[27] QUEIROZ L L, HOFFMANN C, LACORTE G A, et al.Genomic and functional characterization of bacteriocinogenic lactic acid bacteria isolated from Boza, a traditional cereal-based beverage[J].Scientific Reports, 2022, 12(1):1460.
[28] 胡此海, 杨絮, 郭全友, 等.四川泡菜母水的微生物群落与理化特性分析[J].中国食品学报, 2024, 24(2):281-291.
HU C H, YANG X, GUO Q Y, et al.Analysis of microbial community and physicochemical characteristics in Sichuan pickle brine[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(2):281-291.
[29] 夏亚男, 刘皓, 双全.基于宏基因组分析酸马奶的微生物多样性及功能基因[J].中国食品学报, 2022, 22(2):301-309.
XIA Y N, LIU H, SHUANG Q.Analysis on microbiological diversity and functional genes of koumiss based on metagenomic technology[J].Journal of Chinese Institute of Food Science and Technology, 2022, 22(2):301-309.