酸笋是一种以新鲜竹笋为原料发酵而成的带有显著酸感的笋制品。目前酸笋的生产主要是以自然发酵为主,代表性菌属包括乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)、明串珠菌属(Leuconostoc)、乳球菌属(Lactococcus)等乳酸菌[1]。其中乳杆菌属、乳球菌属与酸笋中核心特征风味物质对甲酚呈高度正相关[2]。然而,自然发酵存在发酵周期长、生产过程难以控制的问题[3]。接种发酵技术可有效解决这些问题,其具有加快发酵进程及定向调控发酵蔬菜风味品质的作用,是应用广泛的现代食品发酵技术之一[4]。研究表明,接种柠檬明串珠菌能产生泡菜的特征风味物质甘露醇[5];接种间质明串珠菌于四川泡菜可缩短发酵周期,改善产品风味[6];接种乳酸菌可以缩短酸笋发酵周期,使其乳酸发酵风味更加突出[4]。
宏基因组测序技术能够高效准确系统地挖掘微生物的菌系构成并有效地注释发酵菌系的代谢功能[7],MI等[8]通过宏基因组测序技术研究泡菜中微生物发现一些细菌属与风味物质显著性相关,而真菌与风味物质的相关性不显著;杨柳等[9]以朝鲜族辣白菜为原料,利用宏基因组测序技术研究发现,自然发酵过程中菌群结构与主要风味物质呈正相关。但是,目前通过宏基因组学解析酸笋中微生物代谢及关键风味基因的研究鲜有报道。因此,本文通过对自然发酵酸笋(natural fermented Suansun, NFS)和植物乳杆菌接种发酵酸笋(Lactobacillus plantarum fermented Suansun, LPS)进行宏基因组学分析,从基因层面上探究不同发酵方式的酸笋中菌种的功能分类和代谢通路,阐明植物乳杆菌发酵改善酸笋风味品质的作用机理,旨在为酸笋发酵风味品质调控提供理论基础。
1 材料与方法
1.1 材料与试剂
新鲜麻竹笋,重庆市北碚区天生桥农贸市场;植物乳杆菌,山东中科嘉亿生物工程有限公司;腌制盐,重庆合川盐化工业有限公司;Mag-Bind®土壤DNA试剂盒,美国Omega Bio-Tek公司;NEXTFLEX® Rapid DNA-Seq Prep Kit DNA快速建库试剂盒,美国德克萨斯州奥斯汀Bio Scientific公司;NovaSeq 6000 S4 Kit v1.5试剂盒,因美纳(中国)科学器材有限公司;凝胶回收试剂盒,美国赛默飞生物科技有限公司。
1.2 仪器与设备
TBS-380微型荧光计,北京原平皓生物技术有限公司;NanoDrop 2000超微量分光光度计、Owl电泳系统,美国赛默飞生物科技有限公司;Covaris低通量聚焦超声器,香港基因有限公司;Illumina NovaSeq 6000基因测序仪,美国加利福尼亚州圣地亚哥Illumina公司。
1.3 实验方法
1.3.1 自然发酵和植物乳杆菌接种发酵酸笋的制备
将新鲜麻竹笋的外壳剥去,清洗后将其切成笋片,然后在沸水浴中漂烫15 min,冷却后放入5 L发酵桶中,按2∶3的固液比加入质量分数为6%的盐水,密封,在室温条件下进行自然发酵。同时,另取一部分漂烫冷却后的麻竹笋,然后按照自然发酵固液比加入盐浓度为6%的无菌水,按1%的接种量投入已活化的植物乳杆菌,密封,室温发酵。腌制发酵45 d后,各取3份50 mL发酵液进行下一步分析。
1.3.2 DNA提取和Illumina测序
将样品送往上海美吉生物医药科技有限公司进行DNA提取和Illumina测序。首先通过Mag-Bind®土壤DNA试剂盒对样品进行DNA提取,利用TBS-380和NanoDrop2000测量DNA的浓度和纯度。然后,通过Covaris M220进行DNA片段化,利用NEXTFLEX® Rapid DNA-Seq Prep Kit DNA快速建库试剂盒构建双端文库并将含有完整互补测序引物杂交位点的衔接子连接到片段的钝端,使用NovaSeq 6000 S4 Kit v1.5试剂盒通过 Illumina NovaSeq进行双端测序,序列数据存放在NCBI短读存档数据库中。在此基础上,应用Megahit拼接软件对序列进行组装。选择长度≥300 bp的重叠群作为最终组装结果,用于进一步的基因预测和注释。
1.3.3 微生物群落分析
首先用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)扩增样品16S rDNA基因的V3~V4区,然后在72 ℃下延伸10 min,最后在10 ℃下储存以备后用。每个样品复制3次后,使用2%琼脂糖凝胶电泳检测PCR产物,根据凝胶回收试剂盒说明纯化PCR产物。接着使用DNA快速文库制备试剂盒创建文库,利用Illumina的Miseq PE300平台进行测序,再用UPARSE软件对OTU序列进行聚类,结果符合97%的相似度。最后,基于RDP分类器和Silva 16S rDNA基因数据库对微生物进行分类注释。
1.3.4 基因预测和功能注释
通过MetaGene预测每个组装重叠群的开放阅读框(open reading frame, ORF),使用NCBI翻译表检索长度≥100 bp的预测ORF并将其翻译成氨基酸序列;然后用CD-HIT构建一个序列同一性为90%、覆盖率为90%的非冗余基因目录;将高质量读段与非冗余基因目录进行比对,以使用SOAPaligner计算具有95%同一性的基因丰度;使用Diamond在直系同源蛋白分组比对数据库(eggNOG)中执行具有代表性序列的直系同源蛋白质组(cluster of orthologous groups of proteins, COG)注释簇,e值截止值为10-5;使用Diamond对《京都基因和基因组百科全书》(Kyoto Encyclopedia of Genes and Genomes, KEGG)进行注释,e值截止值为10-5;利用hmmscan对碳水化合物活性酶数据库(CAZy)进行碳水化合物活性酶注释,e值截止值为10-5;将基因集与eggNOG、KEGG、CAZy等数据库进行比对,获得基因的物种注释信息和功能注释信息,基于基因集丰度得到功能丰度和物种丰度。
1.4 数据处理
使用美吉云平台对数据进行可视化处理,进行微生物多样性分析。使用R软件绘制酸笋微生物群落的Alpha多样性指数图和物种丰度图;采用Perl软件绘制酸笋微生物群落的Circos图,采用Origin2021软件对eggNOG、KEGG、CAZy数据库注释结果作图。
2 结果与分析
2.1 NFS与LPS的微生物多样性分析
两组样品经宏基因组测序后共获得序列数为405 974 170的原始数据,然后进行了清理、汇集和重新组装。平均而言,93.33%的总原始读数通过了质量控制。进一步优化数据的读数,共获得378 885 756个高质量的宏基因组读数。经过MEGAHIT v1.1.2软件拼接组装后,最大重叠群长度151 685 bp,最小重叠群长度为300 bp。上述结果表明不同发酵方式下的酸笋样品的测序数据量合理,基因组的测序深度已经足够,能够保证后续分析结果的准确性。
图1所示为NFS以及LPS在门水平上4个具有代表性的Alpha多样性指数。其中图1-a、图1-b的Sobs和Ace指数反映了微生物群落丰富度,图1-c、图1-d的Shannon和Simpson指数反映了微生物群落多样性。Sobs指数主要反映了生态系统中物种数量变化的趋势,Ace指数则可以用来描述样品的微生物群落丰度。由图可知,NFS的微生物群落丰富度与物种数量变化趋势均略低于LPS。Simpson多样性指数的值越小,代表群落多样性程度越高;Shannon指数则恰恰相反,值越大代表群落多样性越高。从Shannon指数可知,NFS的群落多样性程度仅为LPS的十分之一,Simpson指数也展现出类似的趋势。LPS无论是微生物群落丰富度还是多样性均高于NFS,这说明LP改变了自然发酵酸笋的代谢环境,其核心乳酸菌菌群共生互作较自然发酵酸笋相比更多样,也更复杂。
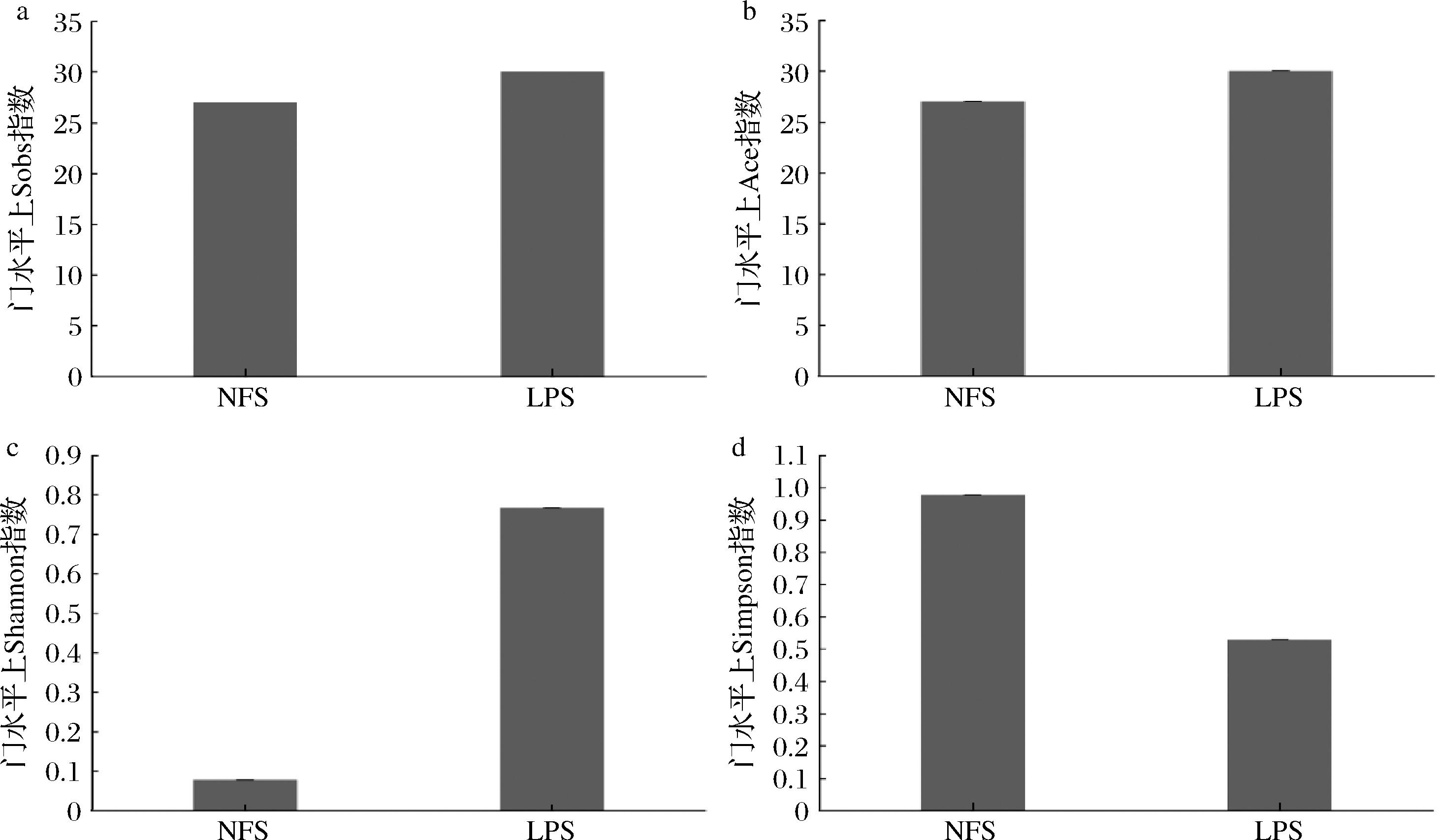
a-Sobs指数;b-Ace指数;c-Shannon指数;d-Simpson指数
图1 NFS和LPS的Alpha多样性指数图
Fig.1 Alpha diversity index of NFS and LPS
2.2 NFS与LPS发酵过程中微生物群落结构分析
微生物群落结构分析可以提供发酵过程微生物组装的轮廓,以了解微生物在发酵过程中的物种组成。根据物种丰度表和物种注释表,计算2种酸笋发酵样品在门、属、种水平上的相对丰度比例,选取丰度较高的物种分类,将每个样本各组分的相对丰度以堆叠柱状图展示。如图2所示为NFS和LPS第45天在门、属、种水平上的物种丰度注释图以及在种水平上两种样品的组成重叠情况。
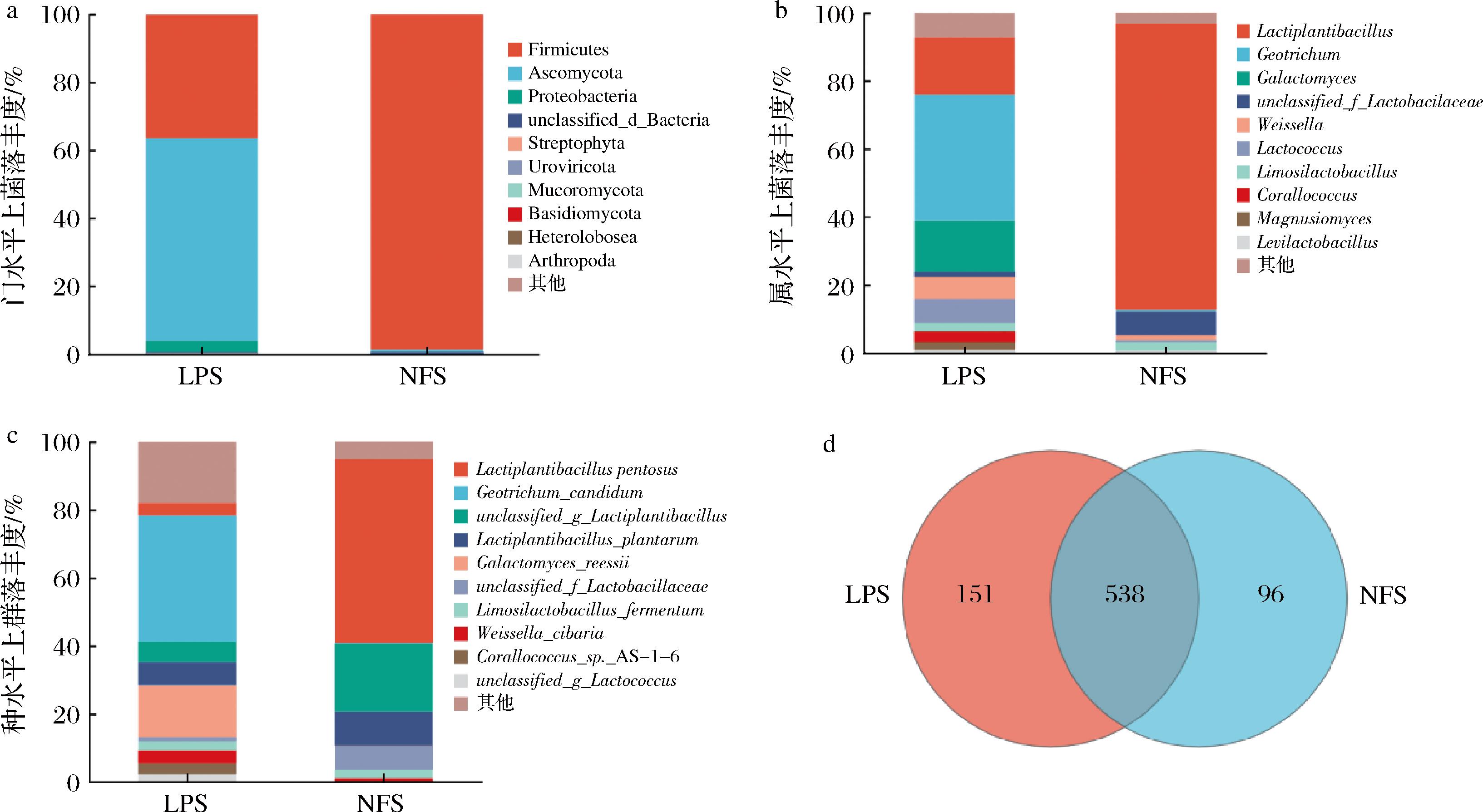
a-门水平上微生物群落柱形图;b-属水平上微生物群落柱形图;c-种水平上微生物群落柱形图;d-种水平上Veen图
图2 NFS与LPS在门、属、种水平样品的群落柱形图及种水平上Venn图
Fig.2 Community bar charts of NFS and LPS at the phylum, genus, species level samples, and Venn plot at the species level
在门水平上,NFS中厚壁菌门(Firmicutes)占绝对优势,其相对丰度在98.66%。这一数据与周金沙等[10]的研究结果(99.94%)类似。LI等[11]在对比鲜笋与酸笋的微生物构成时也发现,在发酵样品中厚壁菌门是作为主要细菌群存在的,由此可推断厚壁菌门是自然酸笋发酵中物质转化的核心因素。除此之外还存在变形菌门(Proteobacteria)、子囊菌门(Ascomycota)等,其分别各占有一定比例。相比之下,LPS则以子囊菌门(Ascomycota)和厚壁菌门(Firmicutes)为主要优势菌种,分别占59.64%和36.34%,这与酸菜发酵过程中的优势微生物类似[12]。
在属水平上,NFS中含有植物乳杆菌属(Lactiplantibacillus)、乳杆菌属(Levilactobacillus)、地霉菌属(Geotrichum)、魏斯氏菌属(Weissella)、罗伊氏杆菌属(Limosilactobacillus)等。其中植物乳杆菌属为NFS中的主要菌种,相对丰度达84.23%,这与陈晓东等[13]的研究结果相似。ROMI等[14]在对印度酸笋的研究中也证实,植物乳杆菌在酸笋发酵后期占主导地位,这是由于乳酸和乙酸的积累导致的微生物菌群的构成发生变化[15]。研究发现,植物乳杆菌不仅能够将糖代谢为乳酸、乙醇和乙酸[16],还可提高发酵蔬菜中酒石酸、柠檬酸以及谷氨酸、甘氨酸和苏氨酸的含量[17],在为酸笋提供更清爽的发酵酸感的同时,其产生的高浓度的酸环境还会抑制相关有害微生物的繁殖,保证酸笋的安全性。相比之下,LPS中微生物多样性更强,包含植物乳杆菌属(Lactiplantibacillus)、地霉菌属(Geotrichum)、耐碱酵母属(Galactomyces)、魏斯氏菌属(Weissella)、乳球菌属(Lactococcus)、罗伊氏杆菌属(Limosilactobacillus)、珊瑚球菌属(Corallococcus)、乳杆菌属(Levilactobacillus)等20余种菌属。
在种水平上,NFS以戊糖乳杆菌(Lactiplantibacillus pentosus)、植物乳杆菌(Lactiplantibacillus plantarum)、发酵乳杆菌(Limosilactobacillus fermentum)、食窦魏斯氏菌(Weissella cibaria)等为主要菌种。LPS包含戊糖乳杆菌(Lactiplantibacillus pentosus)、白地霉(Geotrichum candidum)、植物乳杆菌(Lactiplantibacillus plantarum)、里氏半乳糖菌(Galactomyces reessii)、珊瑚球菌_AS-1-6(Corallococcus sp. AS-1-6)、食窦魏斯氏菌(Weissella cibaria)等菌种,种群多样性高于NFS。从种水平上2种发酵方式酸笋样品的组成重叠情况可得,2种样品在组成上较为相似,重叠度较高,但显然LPS菌种多样性更强。NFS在发酵后期主要以乳杆菌为主,而LPS除此之外还含有白地霉(Geotrichum candidum)、里氏半乳糖菌(Galactomyces reessii)、珊瑚球菌属_AS-1-6(Corallococcus sp. AS-1-6)、食窦魏斯氏菌(Weissella cibaria)等菌种,相对丰度分别为37.10%、15.04%、3.11%、3.99%。有研究发现,食窦魏斯氏菌与植物乳杆菌协同作用可以快速提升发酵蔬菜产品体系中乳酸菌、乳酸和乙酸的含量,从而较好地改善发酵产品的风味与品质[18]。这也造成了LPS中各菌种的协同作用更优,在酸笋代谢反应上更加活跃,最终形成更加丰富的物质种类。
2.3 NFS与LPS发酵过程宏基因组学分析
2.3.1 NFS与LPS的COG分析
研究表明,酸笋中的蛋白质和总糖含量与鲜竹笋相比显著降低,这说明酸竹笋中的微生物群落对其自身营养含量的变化具有显著的驱动作用[19],COG的注释有助于深度了解这一过程。如图3-a、图3-b所示,通过COG的注释,将NFS中22 037个基因和LPS中的26 024个基因按照24个功能大类进行分类,其中NFS基因数大于1 200的有氨基酸转运和代谢、碳水化合物的运输和代谢、翻译,核糖体结构和生物发生、转录、复制、重组和修复、细胞壁/膜/包膜生物发生、常规功能预测;LPS中基因数大于1 200的有氨基酸转运和代谢、碳水化合物的运输和代谢、辅酶转运和代谢、翻译,核糖体结构和生物发生、转录、复制、重组和修复、细胞壁/膜/包膜生物发生、无机离子转运和代谢、常规功能预测。对COG的相对丰度分析表明,2种不同发酵方式的样本有非常相似的功能属性,均以氨基酸代谢和碳水化合物代谢为主要代谢方式。这可能是因为蛋白质和糖类经常被用作微生物发酵的营养物质来源,而微生物类群在消耗这些营养物质的过程中会也引发一系列的产物转运和代谢反应[20],基因与代谢之间相辅相成,在酸笋的各功能表达过程中均有较高响应。LPS代谢的活跃程度略高于NFS,凸显了LP对微生物适应酸笋发酵系统生态位及发挥相应代谢功能的重要性。胞外结构是LPS中所特有的代谢活动,这说明2种样品的差异表达蛋白主要被注释到各种生物分子组成的功能通路中,如蛋白质、多糖类物质和其他细胞外分子等。因此,在COG的注释中,LPS的微生物活动比NFS更加多样化。
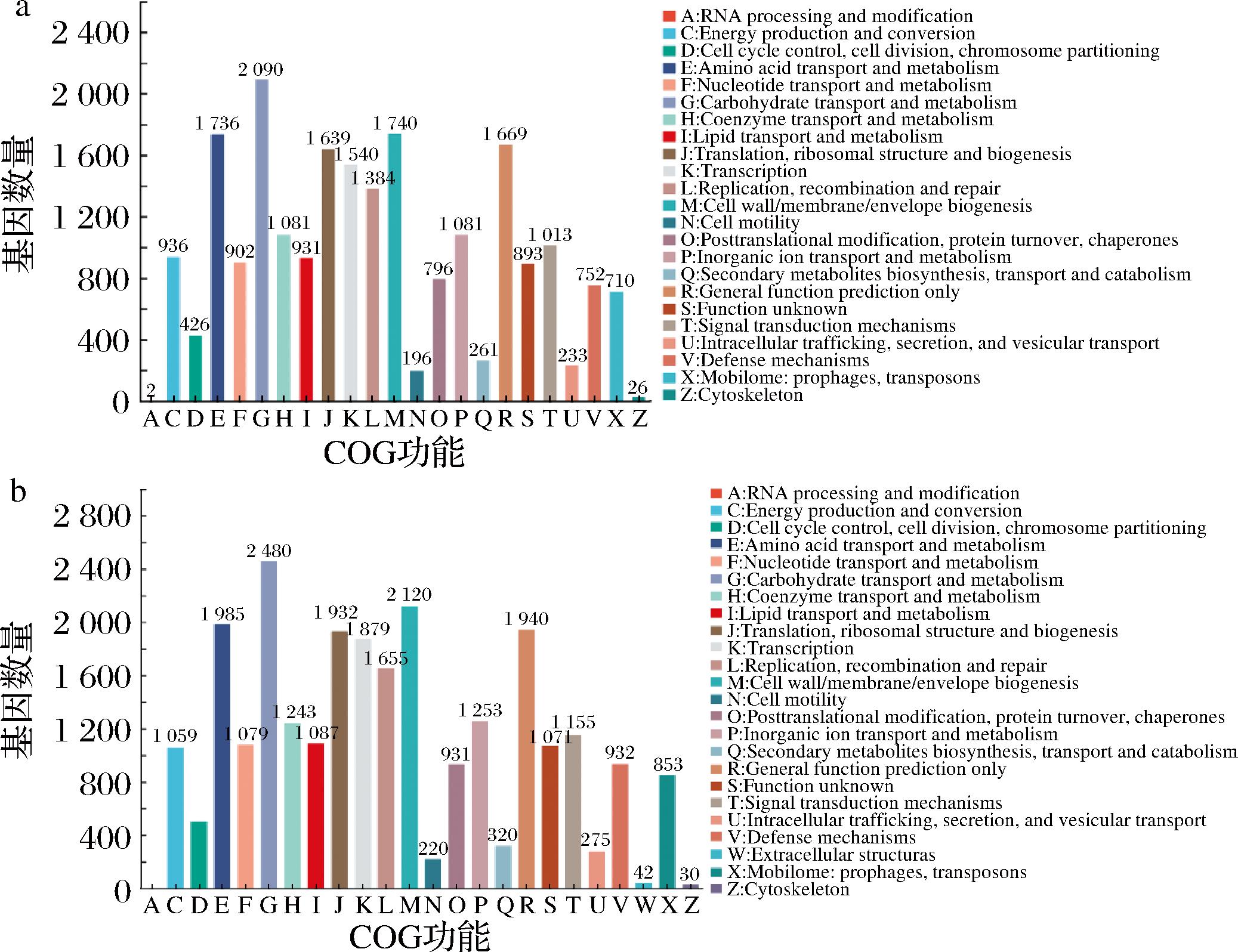
a-NFS的COG注释分类统计;b-LPS的COG注释分类统计
图3 NFS和LPS的COG注释分类统计
Fig.3 COG annotation classification statistics of NFS and LPS
注:A,RNA加工修饰;B,染色质结构和动力学;C,能量生成和转换;D,细胞周期控制、细胞分裂和染色体分裂;E,氨基酸转运代谢;F,核苷酸转运和代谢;G,碳水化合物转运代谢;H,辅酶转运和代谢;I,脂肪转运代谢;J,翻译,核糖体结构和生物合成;K,转录;L,复制、重组和修复;M,细胞壁/膜/被膜的生物合成;N,细胞运动;O,翻译后修饰,蛋白质折叠和伴侣蛋白;P,无机离子转运代谢;Q,次级代谢物生物合成,转运和代谢;R,主要功能预测;S,未知功能;T,信号转导机制;U,胞内转运、分泌和小泡运输;V,抵御机制;W,胞外结构;X,移 动基因组:噬菌体原,转座子;Y,核酸结构;Z,细胞骨架。
2.3.2 NFS与LPS的KEGG分析
为了揭示不同发酵过程中的功能基因分布差异和代谢机制,使用KEGG数据库对NFS和LPS的注释,如图4所示。KEGG富集分析结果表明,在1级KEGG途径中,新陈代谢是NFS和LPS最主要的富集途径。在2级KEGG途径中,NFS基因数在50 000以上的有11个,排名前6的是碳水化合物代谢、全局与概述图谱、膜运输、能量代谢、脂类代谢、氨基酸代谢。而LPS中基因数在50 000以上的只有6个,分别是碳水化合物代谢、全局与概述图谱、能量代谢、氨基酸代谢、脂类代谢、信号转导,说明NFS中微生物的代谢范围比LPS广。二者代谢通路的重合途径主要集中在碳水化合物代谢、能量代谢、氨基酸代谢、脂质代谢这几个方面。在3级KEGG途径中,碳水化合物代谢占主要地位,NFS与LPS所注释到的基因数分别为2 090和2 460,其中丙酮酸代谢在2种发酵环境下均是重要的代谢途径。此外,果糖和甘露糖代谢也是较为重要的代谢通路。研究已证实酸笋中含有甘露醇[21],乳酸菌可通过异型发酵将果糖转化为甘露醇,其独特的清凉口感[22]赋予了酸笋滋味提升的关键作用,并丰富了其风味特性。

a-NFS;b-LPS
图4 NFS和LPS的KEGG分析
Fig.4 KEGG analysis of NFS and LPS
注:Global and overview maps,全局与概述图谱;Carbohydrate metabolism,碳水化合物代谢;Amino acid metabolism,氨基酸代谢;Metabolism of cofactors and vitamins,辅助因子和维生素的代谢;Energy metabolism,能量代谢;Nucleotide metabolism,,核苷酸代谢;Glycan biosynthesis and metabolism,多聚糖的生物合成和代谢;Lipid metabolism,类脂(化合)物代谢作用;Metabolism of other amino acids,其他氨基酸的代谢;Biosynthesis of other secondary metabolites,其他次生代谢物的生物合成;Xenobiotics biodegradation and metabolism,异种生物体的生物降解和代谢;Metabolism of terpenoids and polyketides,萜类和聚酮类物质的代谢;Membrane transport,膜转运;Signal transduction,信号转导;Signaling molecules and interaction,信号分子和相互作用;Replication and repair,复制和修复;Translation,翻译;Folding, sorting and degradation,折叠、整理和降解;Transcription,转录;Information processing in viruses,病毒中的信息处理;Drug resistance:antimicrobial,耐药性:抗菌;Cancer: overview,癌症:概述;Cardiovascular disease,心血管病;Infectious disease: bacterial,传染病:细菌;Endocrine and metabolic disease,内分泌和代谢性疾病;Neurodegenerative disease,神经变性疾病;Infectious disease: parasitic,传染病:寄生虫;Drug resistance: antineoplastic,耐药性:抗肿瘤药物;Infectious disease: viral,传染性疾病:病毒性;Immune disease,免疫性疾病;Cancer:specific types,癌症:特定类型;Substance dependence,药物依赖;Cellular community-prokaryotes,细胞群落原核生物;Cell growth and death,细胞生长和死亡;Transport and catabolism,运输和分解代谢;Cell motility,细胞运动性;Cellular community-eukaryotes,细胞群落-真核生物;Endocrine system,内分泌系统;Aging,衰老;Environmental adaptation,环境适应;Digestive system,消化系统;Nervous system,神经系统;Immune system,免 疫系统;Circulatory system,循环系统;Excretory system,排泄系统;Development and regeneration,发展和再生;Sensory system,感觉系统。

a-NFS的CAZy分析;b-LPS的CAZy分析;c-NFS与LPS的Circos图
图5 NFS和LPS的CAZY分析
Fig.5 CAZy analysis of NFS and LPS
2.3.3 NFS与LPS的CAZy分析
对NFS和LPS的碳水化合物活性酶的注释如图5-a、图5-b所示。在微生物降解多糖的整个过程中,多糖降解酶为关键因素,主要包括以下几大类:糖苷水解酶(glycoside hydrolase, GH)、多糖裂解酶(polysaccharide lyase, PL)、糖基转移酶(glycosyltransferase, GT)、碳水化合物酯酶(carbohydrate esterase, CE)和辅助活性酶类。在几种碳水化合物活性酶中,GH和GT在NFS和LPS中均最高。GH是一类打破多聚糖之间化学连接键的酶,可以精准地识别对应的碳水化合物基团,通过插入一个水分子催化形成糖苷键的断裂[23],它在NFS和LPS中的基因数分别为477和583。GT是产生复杂糖缀合物所必需的酶[24],在NFS和LPS中的基因数分别为465、532,说明相比于LPS,NFS中的GH和GT活性更高,即碳水化合物代谢更旺盛。PL在NFS和LPS中普遍较低,而PL的作用主要是通过β消除机制催化多糖分子内糖苷键的断裂从而产生低聚糖[25],由此可推测在酸笋代谢过程中β消除反应的发生较少。由图5-c可以看出2个样本之间的微生物物种对应关系,其中Lactococcus garvieae、Saprochaete ingens、Magnusiomyces magnusii、Corallococcus sp. AS-1-6、Weissella cibaria为NFS与LPS中共性较大的微生物群落。说明NFS和LPS中含量最高的GH和GT的产生与这些微生物相关性显著。因此,NFS和LPS以碳水化合物的水解、转运为主,在碳水化合物结合的非催化蛋白结构域以及多糖裂解等代谢活动的活跃程度相对较低。
2.3.4 NFS与LPS发酵过程中关键代谢途径分析
如图6所示是NFS和LPS的部分碳水化合物代谢途径分析。在NFS的碳水化合物代谢中共注释到15条相关代谢通路,主要进行的代谢有:淀粉和蔗糖代谢(ko00500)、丙酮酸代谢(ko00620)、氨基糖和核苷酸糖代谢(ko00520)、果糖和甘露糖代谢(ko00051)、糖酵解/糖异生(ko00010),这与关倩倩[3]对传统发酵酸笋的代谢研究结果有相似之处。其中淀粉和蔗糖代谢最为活跃,丙酮酸代谢次之。淀粉和蔗糖的代谢主要伴随着糖酵解的过程进行,目的是将竹笋原料中的淀粉和蔗糖转化成葡萄糖,为微生物的代谢提供能量来源。竹笋中的蔗糖在蔗糖PTS渗透酶的催化下生成蔗糖-6-磷酸,注释到的蔗糖PTS渗透酶的相关微生物有Weissella、Lactobacillus、Lactococcus等。蔗糖-6-磷酸在β-呋喃果糖苷酶催化下生成D-葡萄糖-6磷酸和D-果糖,最终生成α-D-葡萄糖-1-磷酸参与到糖酵解途径中。
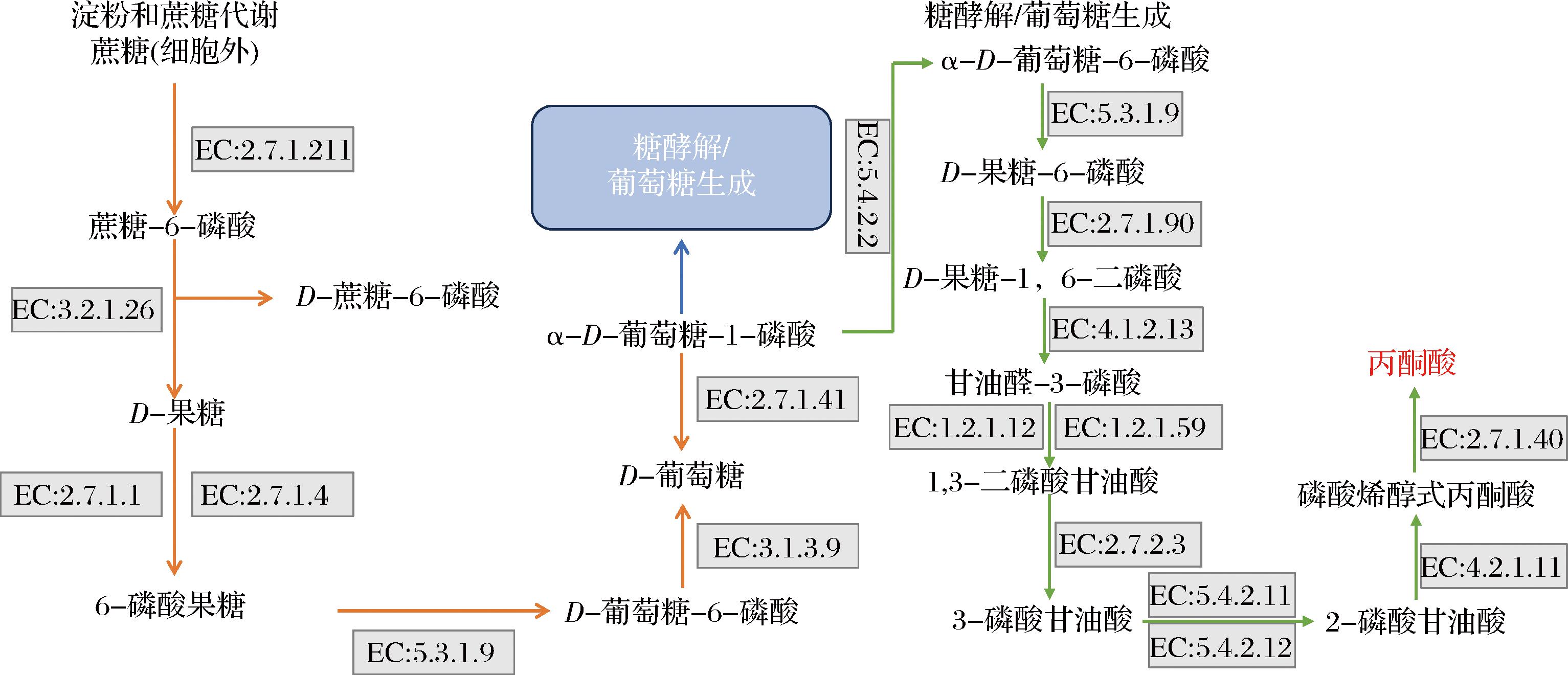
图6 NFS和LPS中酸笋碳水化合物代谢途径分析
Fig.6 Analysis of carbohydrate metabolism pathways in Suansun during NFS and LPS
LPS的碳水化合物代谢中也共注释到15条相关代谢通路,与NFS类型相似,但不同代谢通路在2种样品中的活跃程度不同。LP发酵样品中糖酵解/糖异生最为活跃,淀粉和蔗糖代谢以及丙酮酸代谢其次。糖酵解代谢过程中最主要一环是丙酮酸的生成,其与酸笋主要风味物质乳酸的形成密切相关,由此可推断LP发酵酸笋的酸感更强,更符合大众对酸笋的口味期待。糖酵解合成乳酸的过程为:1-磷酸葡萄糖经葡萄糖磷酸变位酶(EC:5.4.2.2)、葡萄糖-6-磷酸异构酶(EC:5.3.1.9)催化生成6-磷酸果糖,再经过二磷酸依赖性磷酸果糖激酶(EC:2.7.1.90)等9种酶先后催化生成丙酮酸,丙酮酸最后经丙酮酸脱氢酶(EC:1.2.5.1)催化生成乳酸。丙酮酸脱氢酶被注释到与大肠杆菌K-12 MG1655、大肠杆菌K-12 W3110、大肠杆菌K-12 DH10B等基因高度相关,因而这些基因可被认为是与酸笋酸味形成呈正相关的风味基因。研究表明,酸笋注释到KEGG碳水化合物代谢通路上的微生物主要包括植物乳杆菌、戊糖乳杆菌、乳酸乳球菌等乳酸菌,植物乳杆菌注释到的KEGG通路最多,包括柠檬酸循环(ko00020)、磷酸戊糖途径(ko00030)、氨基酸和核苷酸糖代谢(ko00520)等多条代谢通路[26],由此可见,植物乳杆菌的加入可以加速酸笋的发酵进程。
利用KEGG数据库对2种发酵方式下酸笋发酵过程中的氨基酸代谢进行注释,基于注释结果重构NFS和LPS主要氨基酸代谢网络如图7所示。NFS的氨基酸代谢途径共注释到9条,较为活跃的是半胱氨酸和蛋氨酸代谢、酪氨酸代谢、甘氨酸、丝氨酸和苏氨酸代谢、丙氨酸、天冬氨酸和谷氨酸代谢。这些代谢可形成一个完整的代谢网络,其中部分代谢产物起到连接代谢间桥梁的作用。如图7-a所示,在代谢程度最旺盛的半胱氨酸和蛋氨酸代谢中,L-丝氨酸经L-苏氨酸解氨酶(EC:4.3.1.17)催化生成L-半胱氨酸,再经磺基裂解酶(EC:4.4.1.25)催化生成丙酮酸进而进入到丙酮酸代谢中。与之相似,甘氨酸、丝氨酸和苏氨酸代谢过程中的产物丝氨酸也可经L-苏氨酸解氨酶(EC:4.3.1.17)催化形成丙酮酸进入丙酮酸代谢,生成的丙酮酸又可在丙氨酸、天冬氨酸和谷氨酸代谢中经丙氨酸脱氢酶(EC:1.4.1.1)转化为L-丙氨酸,进而进入D-氨基酸代谢中。丙酮酸还可经自身一系列代谢后生成代谢产物丙酮醛,回到甘氨酸、丝氨酸和苏氨酸代谢中参与物质转化。而后代谢产物丝氨酸又经β-合酶(EC:4.2.1.22)、γ-裂解酶(EC:4.2.1.11)生成L-丝氨酸进入到半胱氨酸和蛋氨酸代谢中参与反应。至此,各中间产物均可作为联系其他主要氨基酸代谢的纽带,丙酮酸是该网络的核心枢纽,调控各代谢的发生与转化。酪氨酸代谢是自然发酵酸笋中第二旺盛的代谢,其又可作为合成对甲酚的前体物质[27]。对甲酚作为酸笋中含量最高的挥发性风味物质[28],是赋予酸笋独特臭味的重要特征风味之一。酪氨酸的代谢可通过氨基酸转氨酶转化为4-羟基-苯丙酮酸,在4-羟基苯丙酮酸羧化酶作用下生成对羟基苯乙酸,再转化成对甲酚[27]。此外,半胱氨酸和蛋氨酸代谢产生的含硫化合物也是酸笋中酸臭味的来源之一,这些含硫化合物会进一步通过代谢生成甲硫醇、二甲基二硫醚等物质[29]。
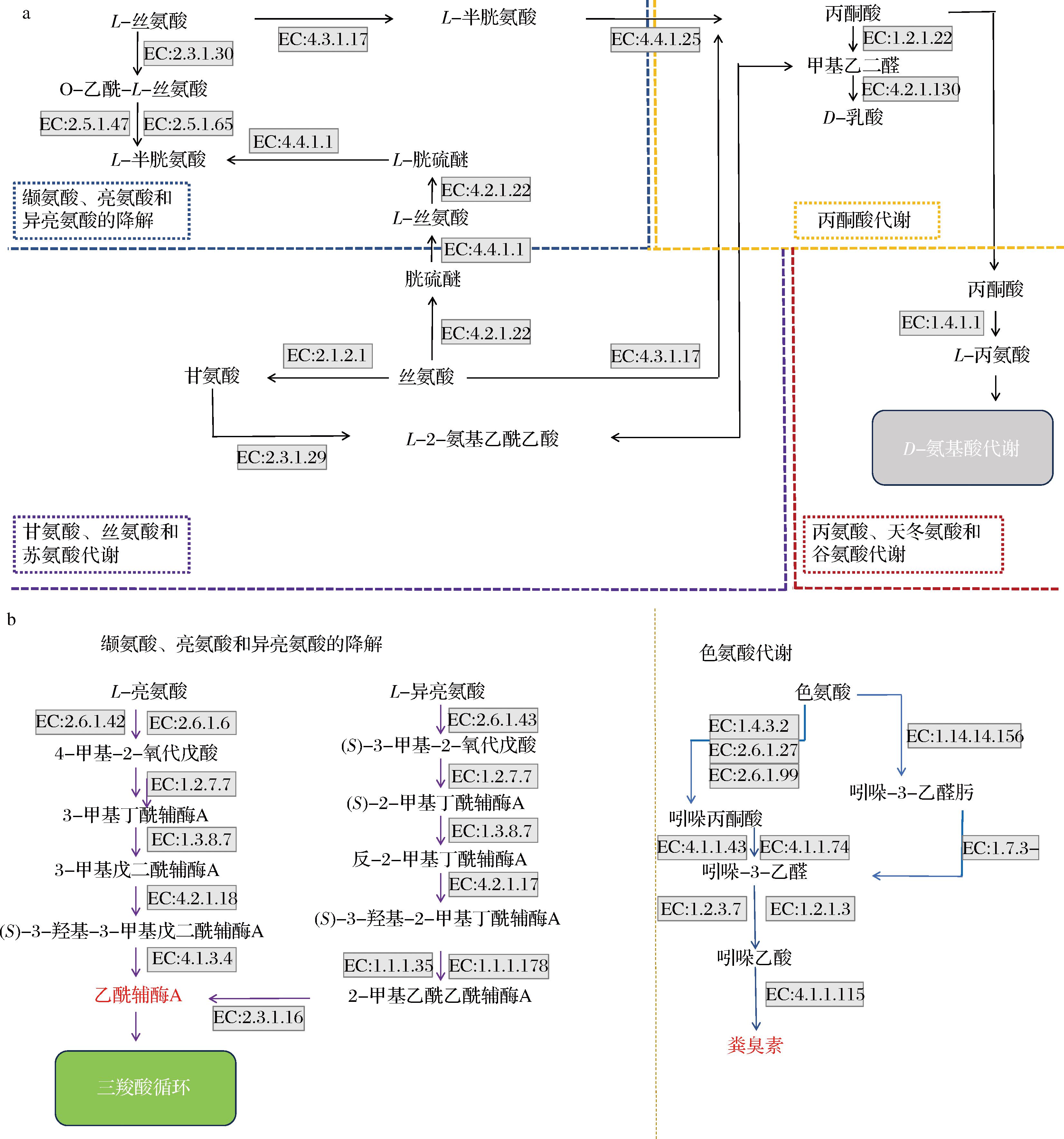
a-NFS;b-LPS
图7 NFS和LPS的关键氨基酸代谢途径分析
Fig.7 Analysis of key amino acid metabolism pathways of NFS and LPS
LPS中的氨基酸代谢中共注释到14条代谢通路,排在前五的有缬氨酸、亮氨酸和异亮氨酸降解(ko00280)、色氨酸代谢(ko00380)、丙氨酸、天冬氨酸和谷氨酸代谢(ko00250)、赖氨酸降解(ko00310)、甘氨酸、丝氨酸和苏氨酸代谢(ko00260)。与NFS相比,LPS中氨基酸代谢种类更多样,缬氨酸、亮氨酸和异亮氨酸降解(ko00280)与色氨酸代谢(ko00380)较NFS相比代谢水平更旺盛。其中最为活跃的是缬氨酸、亮氨酸和异亮氨酸降解,如图7-b所示。L-亮氨酸与L-异亮氨酸在支链氨基酸转氨酶(EC:2.6.1.42)等13种酶的催化下转化成乙酰辅酶A参与到三羧酸循环,为酸笋机体提供能量。此外,色氨酸代谢作为LPS中的次活跃代谢,核心物质色氨酸在L-氨基酸氧化酶(EC:1.4.3.2)、色氨酸转氨酶(EC:2.6.1.27)、色氨酸N-单加氧酶(EC:1.14.14.156)等酶的催化下经2条途径生成吲哚-3-乙醛,后在催化作用下生成粪臭素,是酸笋不悦气味的来源之一。肉毒杆菌BKT015925、梭菌ATCC 25775、德拉克梭菌SL1、发酵厌氧杆菌DY22613、龈乳杆菌DSM 7084被注释为这一代谢过程的关键基因,因此可认为与酸笋特征性臭味形成呈正相关。但与NFS相比,半胱氨酸和蛋氨酸代谢(ko00270)、酪氨酸代谢(ko00350)在LPS中体现程度不高,可见LPS可降低含硫化合物与对甲酚的表达进而抑制酸笋酸臭味的产生,起到改善酸笋风味品质的作用。
3 结论
门水平上,厚壁菌门(Firmicutes)是NFS与LPS共有的优势菌门;属水平上,植物乳杆菌属(Lactiplantibacillus)为NFS中的优势菌属(84.23%),LPS除此之外还含有魏斯氏菌属(Weissella)、乳球菌属(Lactococcus)等优势菌属;从种水平上2种发酵方式的组成重叠情况可得,2种样品在组成上较为相似,但LPS菌种多样性更强。经KEGG注释并重构关键代谢网络发现,碳水化合物代谢及氨基酸代谢是酸笋的主要代谢。大肠杆菌K-12 MG1655、大肠杆菌K-12 W3110等基因被认为与乳酸形成密切相关;肉毒杆菌BKT015925、梭菌ATCC 25775等基因被注释与色氨酸代谢相关,其与酸笋的特征性酸臭味密切相关。此外,LPS可通过抑制半胱氨酸和蛋氨酸代谢(ko00270)和酪氨酸代谢(ko00350)来抑制酸笋不悦气味的产生,从而改善酸笋的风味品质。因此,LP可作为接种发酵的功能性乳酸菌增强酸笋发酵过程中微生物群落的代谢,并一定程度上能够通过抑制相关氨基酸的代谢改善酸笋的风味品质。本文揭示了酸笋自然发酵和植物乳杆菌接种发酵中的微生物多样性,同时从基因层面对其主要的碳水化合物代谢和氨基酸代谢途径进行分析,可以为不同发酵方式的酸笋风味形成机理解析提供理论参考。
[1] 薛冰洁, 胡荣, 吴良如, 等.酸笋中微生物区系演替和风味物质形成机制的研究进展[J].食品与发酵工业, 2024, 50(4):315-321.
XUE B J, HU R, WU L R, et al.Advances on microbial flora succession and flavor substance formation mechanism in Suansun[J].Food and Fermentation Industries, 2024, 50(4):315-321.
[2] ZHANG X Y, WANG A X, YAO H L, et al.Research advancements on the flavor compounds formation mechanism of pickled bamboo shoots in river snails rice noodles[J].LWT, 2023, 186:115226.
[3] 关倩倩. 我国传统酸笋中微生物菌系结构及其对特征风味物质形成的影响[D].南昌:南昌大学, 2021.
GUAN Q Q.Microbial flora structure of traditional sour bamboo shoots in China and its influence on the formation of characteristic flavor substances[D].Nanchang:Nanchang University, 2021.
[4] 谢元, 季家举, 王新权, 等.纯种乳酸菌发酵竹笋技术[J].农产品加工, 2018, 17(11):28-29;33.
XIE Y, JI J J, WANG X Q, et al.The technology of fermented bamboo shoot with pure lactic acid bacteria[J].Farm Products Processing, 2018, 17(11):28-29;33.
[5] 卢宏皓. 异/同型发酵乳酸菌对酸笋接种发酵的影响[D].柳州:广西科技大学, 2022.
LU H H.Effect of heterotypic/homotypic fermentation lactic acid bacteria on inoculation and fermentation of sour bamboo shoots[D].Liuzhou:Guangxi University of Science and Technology, 2022.
[6] 朴泓洁, 黄存辉, 金清.肠膜明串珠菌发酵对四川泡菜品质的影响[J].食品科技, 2018, 43(8):31-35.
PIAO H J, HUANG C H, JIN Q.Effects of Leuconostoc mesenteroides fermentation on quality of Sichuan pickle[J].Food Science and Technology, 2018, 43(8):31-35.
[7] 麻颖垚, 胡萍, 孙利林, 等.宏基因组学分析酱香型白酒窖内发酵优势菌与代谢功能的相关性[J].现代食品科技, 2020, 36(6):128-136.
MA Y Y, HU P, SUN L L, et al.Metagenomics analyze the relationship between the dominant bacteria and fungi and metabolism function in the pit fermentation of Maotai-flavor liquor[J].Modern Food Science and Technology, 2020, 36(6):128-136.
[8] MI T, WANG D K, YAO S J, et al.Effects of salt concentration on the quality and microbial diversity of spontaneously fermented radish Paocai[J].Food Research International, 2022, 160:111622.
[9] 杨柳, 高良锋, 沈明浩, 等.朝鲜族辣白菜在自然发酵过程中菌群结构与主要呈味物质的相关性[J].食品与发酵工业, 2021, 47(17):61-68.
YANG L, GAO L F, SHEN M H, et al.Correlation between microbial community and main flavor components of Korean kimchi during natural fermentation[J].Food and Fermentation Industries, 2021, 47(17):61-68.
[10] 周金沙, 陈晓艺, 谭金萍, 等.基于Illumina MiSeq高通量测序技术分析广西无盐发酵酸笋中细菌多样性[J].中国酿造, 2019, 38(7):85-90.
ZHOU J S, CHEN X Y, TAN J P, et al.Analysis of bacterial diversity in Guangxi non-salt fermentation bamboo shoots based on Illumina Mi Seq sequencing technology[J].China Brewing, 2019, 38(7):85-90.
[11] LI J J, LIU Y, XIAO H, et al.Bacterial communities and volatile organic compounds in traditional fermented salt-free bamboo shoots[J].Food Bioscience, 2022, 50:102006.
[12] 刘鑫, 王家旺, 隋雨萌, 等.中国传统发酵蔬菜中微生物多样性及其对品质形成影响研究进展[J].食品科学, 2024,45(10):290-297.
LIU X, WANG J W, SUI Y M, et al.Microbial diversity and its influence on quality development in Chinese traditional fermented vegetables:A review[J].Food Science, 2024,45(10):290-297.
[13] 陈晓东, 朱志勇, 张韫, 等.基于宏基因组技术分析传统酸笋中微生物多样性[J].食品与生物技术学报, 2020, 39(8):89-98.
CHEN X D, ZHU Z Y, ZHANG Y, et al.Microbial community diversity analysis of traditional fermented bamboo shoots using metagenomic technology[J].Journal of Food Science and Biotechnology, 2020, 39(8):89-98.
[14] ROMI W, AHMED G, JEYARAM K.Three-phase succession of autochthonous lactic acid bacteria to reach a stable ecosystem within 7 days of natural bamboo shoot fermentation as revealed by different molecular approaches[J].Molecular Ecology, 2015, 24(13):3372-3389.
[15] GUAN Q Q, HUANG T, PENG F, et al.The microbial succession and their correlation with the dynamics of flavor compounds involved in the natural fermentation of Suansun, a traditional Chinese fermented bamboo shoots[J].Food Research International, 2022, 157:111216.
[16] LIANG H P, HE Z, WANG X Y, et al.Effects of salt concentration on microbial diversity and volatile compounds during Suancai fermentation[J].Food Microbiology, 2020, 91:103537.
[17] SONG G, HE Z, WANG X Y, et al.Improving the quality of Suancai by inoculating with Lactobacillus plantarum and Pediococcus pentosaceus[J].Food Research International, 2021, 148:110581.
[18] 张楠笛, 祝林, 许琴, 等.食窦魏斯氏菌协同植物乳杆菌改善四川泡菜风味[J].食品科学, 2020, 41(14):102-108.
ZHANG N D, ZHU L, XU Q, et al.Improvement of the flavor of Sichuan pickle by co-fermentation with Weissella cibaria and Lactobacillus plantarum[J].Food Science, 2020, 41(14):102-108.
[19] 朱照华. 酸笋的营养成分检测及其主要风味物质的研究[D].南宁:广西大学, 2014.
ZHU Z H.Determination of nutritional components of sour bamboo shoots and study on their main flavor substances[D].Nanning:Guangxi University, 2014.
[20] 黎婷玉, 陈瑶瑶, 张彦, 等.南宁地区酸笋细菌类群分析[J].食品与发酵工业, 2022, 48(9):71-76.
LI T Y, CHEN Y Y, ZHANG Y, et al.Bacteria community of fermented bamboo shoots from Nanning[J].Food and Fermentation Industries, 2022, 48(9):71-76.
[21] 冯浩森, 李湘銮, 冯爱军, 等.酸笋微生物与风味形成研究进展[J].食品安全质量检测学报, 2023, 14(22):91-99.
FENG H S, LI X L, FENG A J, et al.Research progress on microorganisms and flavor formation of sour bamboo shoots[J].Journal of Food Safety &Quality, 2023, 14(22):91-99.
[22] WISSELINK H W, WEUSTHUIS R A, EGGINK G, et al.Mannitol production by lactic acid bacteria:A review[J].International Dairy Journal, 2002, 12(2-3):151-161.
[23] 原野, 胡彦波, 周义发.糖苷水解酶:生物转化制备活性糖苷与苷元的有效工具[J].微生物学报, 2017, 57(8):1219-1234.
YUAN Y, HU Y B, ZHOU Y F.Glycosidase:An effective tool for the preparation of active glycosides and aglycone[J].Acta Microbiologica Sinica, 2017, 57(8):1219-1234.
[24] JEON J G, ROSALEN P L, FALSETTA M L, et al.Natural products in caries research:Current (limited) knowledge, challenges and future perspective[J].Caries Research, 2011, 45(3):243-263.
[25] 曾良欢. 一种广谱型多糖降解酶的生化特征、酶学特性与催化机理的研究[D].济南:山东大学, 2021.
ZENG L H.Study on biochemical characteristics, enzymatic characteristics and catalytic mechanism of a broad-spectrum polysaccharide degrading enzyme[D].Jinan:Shandong University, 2021.
[26] 陈晓东. 酸笋的宏基因组及其功能基因[D].北京:中国林业科学研究院, 2019.
CHEN X D.Metagenome and functional genes of sour bamboo shoots[D].Beijing:Chinese Academy of Forestry, 2019.
[27] YU L H, BLASER M, ANDREI P I, et al.4-Hydroxyphenylacetate decarboxylases:Properties of a novel subclass of glycyl radical enzyme systems[J].Biochemistry, 2006, 45(31):9584-9592.
[28] XIAO M Y, PENG Z, HARDIE W J, et al.Exploring the typical flavours formation by combined with metatranscriptomics and metabolomics during Chinese Sichuan Paocai fermentation[J].LWT, 2022, 153:112474.
[29] WIECZOREK M N, MAJCHER M A, JELE H H.Identification of aroma compounds in raw and cooked broccoli[J].Flavour and Fragrance Journal, 2021, 36(5):576-583.
H H.Identification of aroma compounds in raw and cooked broccoli[J].Flavour and Fragrance Journal, 2021, 36(5):576-583.