氢能作为一种清洁、高效、安全和可持续发展的新能源,具有来源广泛、清洁环保、可储存和可再生等特点,能够有效转化为电能,广泛应用于汽车、船舶与电力供应等领域[1],从而处理大部分的能源紧缺及环境净化问题。目前,氢气的生产主要依赖于化石燃料,生产过程伴随着大量温室气体的产生[1]。因此,研究生物制氢技术成为新能源研究的理想目标。生物制氢的主体主要是产氢微生物,有着产氢成本低、氢化酶性质稳定、对环境污染较小等优点[2]。目前已发现能够进行发酵产氢的微生物主要包括梭菌属(Clostridium)、肠杆菌属(Enterobacter)和芽孢杆菌属(Bacillus)等[3]。梭菌属细菌具有较高的产氢效率,是良好的产氢发酵菌种,且Clostridium因其底物广泛且易于培养等优势,是当前生物制氢研究广泛关注的对象[3-4]。
本实验室前期在对浓香型白酒窖泥微生物进行筛选的实验中发现了1株具有产氢气能力的菌株——广西梭菌(Clostridium guangxiense)。C.guangxiense的首次报道是ZHAO等[5]在研究氢气高产细菌富集的同时,从广西省柳州龙潭公园龙潭湖湖泊里筛出来的厌氧、中温、生物产氢细菌新物种,故而命名广西梭菌。2019年杨静等[6]探讨了有机氮源的复合配比对梭菌属新菌种C.guangxiense ZGM211T在发酵产氢过程中的生长和产氢的促进作用。C.guangxiense除可以产H2以外,还有很好的产酯性。黄治国等[7]将C.guangxiense与拜氏梭菌、煎盘梭菌进行复配产酯,最终浓香型白酒中的己酸乙酯产量可达322.5 mg/L,丁酸乙酯产量可达1 176.8 mg/L。有学者还将C.guangxiense与其余窖泥中筛选出来的梭菌进行混合制得酯化菌种子液,从而与酵母菌固态发酵优化产己酸乙酯和丁酸乙酯[8]。
截止目前尚未有关于C.guangxiense基因组分析的研究报道,本研究对浓香型白酒窖泥中筛选出的C.guangxiense进行全基因组测序,从基因组的层面解析分析该菌株产氢的潜在机理及其产氢代谢途径,为今后继续深入该菌的应用机理提供参考。
1 材料与方法
1.1 材料与试剂
菌株:前期实验室从浓香型白酒窖泥中分离筛选得到的广西梭菌(C.guangxiense),并命名为WZ-1。
强化梭菌培养基(reinforced clostridial medium,RCM)(g/L):葡萄糖5、NaCl 5、酵母膏5、蛋白胨10、醋酸钠3、牛肉膏10、淀粉1、L-半胱氨酸盐0.5,pH 6.8。
发酵培养基、种子培养基均为RCM培养基。
葡萄糖、NaCl、琼脂粉、醋酸钠,成都市科隆化学品有限公司;蛋白胨、酵母膏、牛肉膏、淀粉、L-半胱氨酸盐酸盐,北京奥博星生物技术有限责任公司。
1.2 仪器与设备
HYQX-Ⅲ厌氧培养箱,上海跃进医疗器械有限公司;LRH-70F生化培养箱,上海捷呈有限公司;HH-2A恒温水浴锅,冠森生物科技有限公司;450-GC气相色谱仪,美国布鲁克·道尔顿公司;5977B-7890B气相色谱-质谱仪,Agilent科技(中国)有限公司。
1.3 全基因组测序和注释
1.3.1 菌株的全基因组测序及其组装与评估
为深入了解WZ-1的生理和代谢特征,将其进行基因组测序与注释。首先,将WZ-1活化好的种子液接种于RCM培养基中,接种量占总体积的5%。随后,培养至对数生长期,即OD600nm 测定值为0.6~0.8。接着,在10 mL离心管中吸取9 mL菌液,并经离心机以5 000 r/min的速度离心5 min,最后收集菌体送至南京派森诺基因科技有限公司进行测序。
对WZ-1菌株进行双末端测序,测序平台采用Illumina NovaSeq。使用荧光染料检测DNA总量,1%(体积分数)琼脂糖凝胶电泳方法来评估DNA的完整性。对检测合格的DNA样品构建所需的基因组上机文库,随后上机进行基因组DNA序列的测定,碱基读取后获得下机数据。为了基因组测序结果的准确性与可靠性,采用AdapterRemoval[9]软件去除接头、低质量的reads序列,采用SOAPec[10]进行质量校正。随后采用A5-MiSeq和SPAdes[11]进行从头拼装,构建Contig和Scaffold。对得到的拼装结果进行评估和比较,并使用Pilon[12]软件进行碱基校正。
1.3.2 基因组分析及功能注释
采用GeneMarkS[13]软件对细菌基因组的蛋白质编码基因进行预测,采用tRNAscan-SE和Barrnap预测tRNA基因和rRNA基因[14]。将预测的基因分别通过GO、COG(Cluster of Orthologous Groups of Proteins)和KEGG数据库进行基因和蛋白质、代谢功能注释与分析,针对预测的蛋白编码基因进行功能注释。采用RAST和Cell-PLoc[15]工具进行菌株的编码蛋白信息及亚细胞定位。利用WZ-1菌株的KEGG代谢途径分析该菌株的氢气、丁酸产生途径,并绘制出WZ-1的产氢途径图所需要的酶类,以进行产氢功能挖掘分析。
1.4 数据处理
1.4.1 产气检测
采用向下排水法测量是否产密度比空气小的气体[6]。先将量筒装满pH值为2的酸性水,然后倒置在装满pH值为2的酸性水槽中,然后将导管伸入水中,待导管出现均匀气泡时再放入倒置的量筒中,等到血清瓶中的气体完全溢出,观察量筒,是否有比空气密度小的气体溢出。
1.4.2 H2含量的测定
气体中的H2组分是通过配备有TCD检测器的布鲁克450-GC气相色谱检测得到。将WZ-1菌种接种于RCM液体培养基内,在35 ℃条件下培养12 h,经过2次传代培养后获得新的种子液。将容量500 mL的血清瓶中先加入300 mL的RCM液体培养基,再将WZ-1的种子液按5%的接种量接种到RCM培养基中,培养3 d,然后利用医用无菌管道将其管道两端分别接入100 mL集气袋与血清瓶中,待血清瓶中气体完全溢出,封存好集气袋,将其送到科学指南针有限公司进行H2含量的检测。其检测方法用气相色谱进样针取0.2 mL血清瓶顶部气体检测,使用N2为载气、进样口、检测器温度分别控制在150和175 ℃,柱箱温度:初温50 ℃,保留11 min,以8 ℃/min的速率升到190 ℃,共计28.5 min。
1.4.3 WZ-1副产物丁酸的测定
采用GC-MS技术对WZ-1发酵液中的丁酸进行定量分析,发酵液在气体收集后继续在厌氧条件下培养5 d,通过0.22 μm滤膜过滤并5 000 r/min离心处理后,取1 mL用于进样测定。
气相色谱条件和质谱条件根据LUO等[16]方法进行测定。定量方法[16]:采用内标法测定发酵液中丁酸含量。定性则采用数据库中的质谱库进行检索。
2 结果与分析
2.1 C.guangxiense WZ-1的发酵产氢能力
通过产气试验的检测,证明WZ-1菌株的代谢产物有比空气密度小的气体。另外,在菌株经过3 d的发酵培养后,将气体收集测氢气含量,剩余液体培养基取1 mL来测丁酸含量。结果表示:在通过气相色谱测定后,可知WZ-1的产氢能力为在100 mL气体里,H2含量为58.990%(体积分数)。另外,也通过气相检测了WZ-1菌株的副产物,其丁酸为1 960 mg/L。图1是H2检测的气相色谱图。图2是副产物丁酸的气相色谱图。
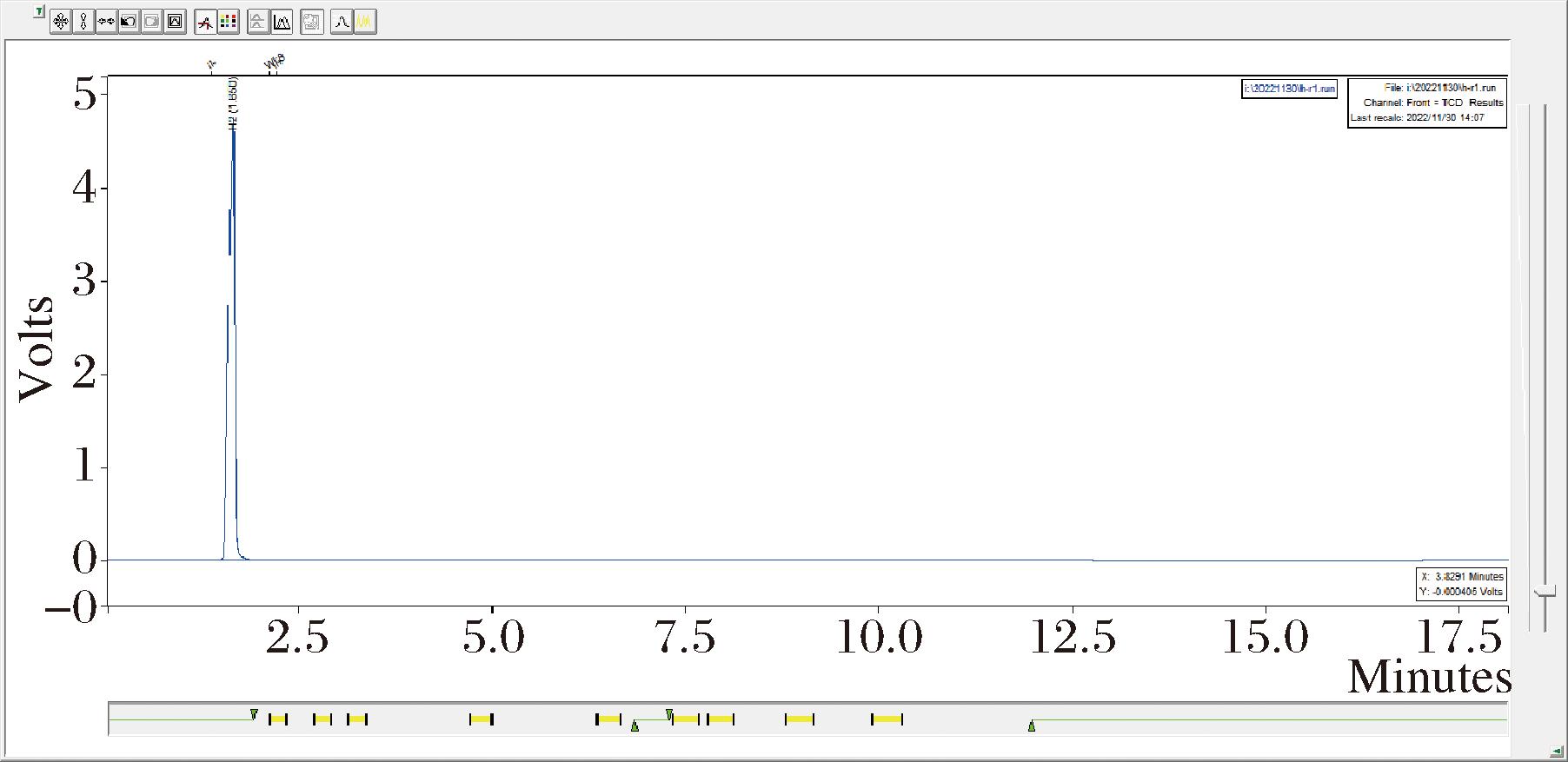
图1 WZ-1氢气检测气相色谱图
Fig.1 WZ-1 hydrogen detection gas chromatogram
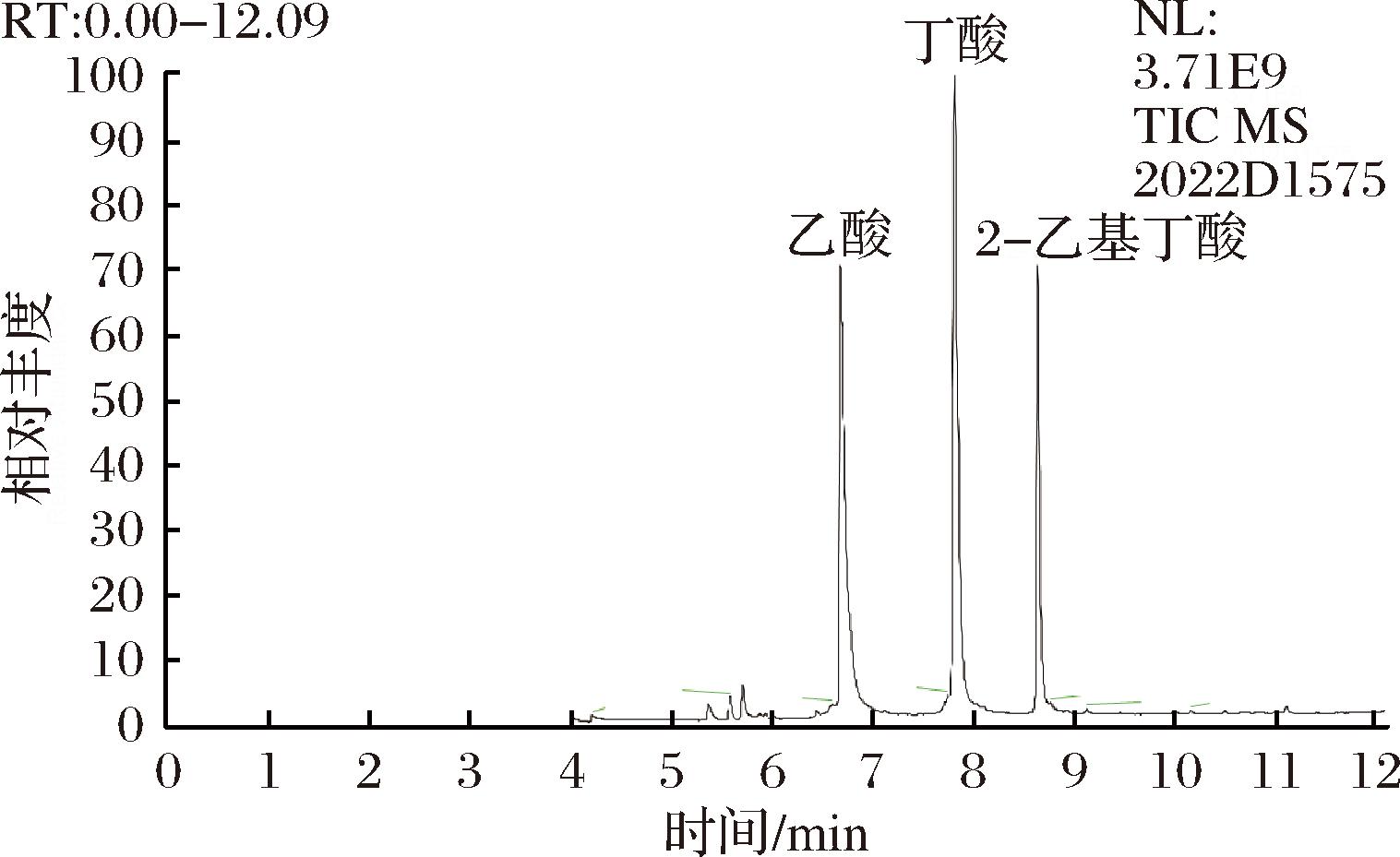
图2 WZ-1副产物气相色谱图
Fig.2 Gas chromatogram of WZ-1 by-products
2.2 C.guangxiense WZ-1的基因组测序与注释
基因组测序结果如表1所示,WZ-1的基因组测序覆盖倍数为237x,基因组总长度为4 406 933 bp,GC含量为31.95%。该基因组包含4 172条编码序列,3个rRNA基因和52个tRNA基因,Contig和Scaffold总序列数量分别为113、99,其N50分别为72 621 bp 和86 360 bp。C.guangxiense WZ-1菌株的核苷酸序列已存入NCBI数据库,登录号为:PRJNA783125,这是当前唯一的C.guangxiense菌株基因组数据。
表1 菌株WZ-1基因组的基本特征
Table 1 Basic features of the genome of strain WZ-1
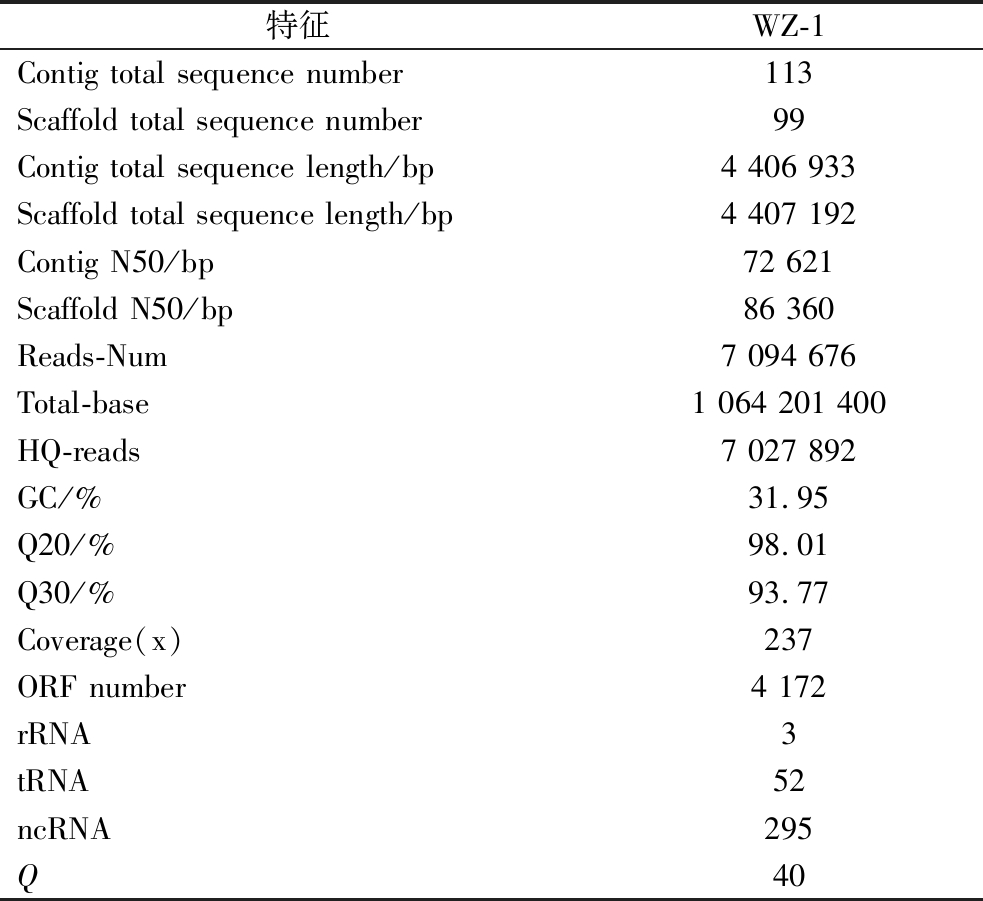
特征WZ-1Contig total sequence number113Scaffold total sequence number99Contig total sequence length/bp4 406 933Scaffold total sequence length/bp4 407 192Contig N50/bp72 621Scaffold N50/bp86 360Reads-Num7 094 676Total-base1 064 201 400HQ-reads7 027 892GC/%31.95Q20/%98.01Q30/%93.77Coverage(x)237ORF number4 172rRNA3tRNA52ncRNA295Q40
注:total sequence number:总序列数量;Coverage(x):测序深度,基因组大小参照拼接得到的基因组序列长度。ORF number:开放阅读框数量。N50:指将序列按长度从长到短排序,逐步累加序列长度,直到累积长度达到所有序列总长度的50%时,所对应的最后一个序列的长度值。Q20:碱基准确率在99%以上所占百分比;Q30:碱基准确率在99.9%以上所占百分比。
eggNOG包括来自原始COG/KOG的功能分类,菌株WZ-1的eggNOG(COG)注释,共有3 572个蛋白编码基因得到注释,占总预测到的编码蛋白基因总数的85.62%(表2)。功能未知(S)是最大的类别,其蛋白编码基因数量为999,占所有蛋白编码基因数量的23.95%,除S外,碳水化合物的转运和代谢(G)和转录(K)是最突出的类群,数量为312和308(图3-a),分别占所有蛋白编码基因数量的7.48%和7.38%。
表2 菌株WZ-1蛋白编码基因功能注释概述
Table 2 Overview of the functional annotation of the protein-coding genes of strain WZ-1

菌株蛋白编码基因在数据库中搜索到的Hits蛋白编码基因数量占比/%WZ-1eggNOG3 57285.618 4GO2 96371.021 1KEGG1 93446.356 7
注:占比为在数据库中能搜索到Hits的蛋白编码基因数量占总预测到的ORF数量的百分比。

a-COG功能基因分类;b-GO功能基因分类;c-KEGG功能基因分类
图3 菌株WZ-1 的COG功能基因分类、GO功能基因分类和KEGG功能基因分类
Fig.3 Classification of COG functional genes, GO functional genes and KEGG functional genes of strain WZ-1
GO的产生是为了解决不同数据库和物种间基因功能定义的混乱,实现基因功能注释的统一和规范化[17]。WZ-1的GO注释中,共有2 963个蛋白编码基因得到注释,占总预测到的编码蛋白基因总数的71.02%(表2);在GO数据库的3大类别——生物学途径、细胞学组件和分子功能中,生物过程、分子功能、细胞氮复合代谢过程、生物合成和离子结合这5个分类中的基因数目最为集中,分别涉及2 798、2 740、1 091、958和945个基因(图3-b)。
KEGG注释旨在揭示物种内部分子之间的相互关系以及各类生化反应构成的网络系统。WZ-1的KEGG注释中,共有1 934个蛋白编码基因在KEGG数据库中得到注释,占总预测到的编码蛋白基因总数的46.36%(表2),统计结果表明代谢途径富集到的数量最多,共1 189个,其中WZ-1菌株最大的3种代谢途径类别分别是397个碳水化合物代谢基因、219个氨基酸代谢基因、122个辅酶因子和维生素代谢基因(图3-c)。
2.3 C.guangxiense WZ-1产氢途径解析
氢化酶是多种细菌中存在的1种关键生物酶,能够通过还原H+来生成H2[18]。根据活性中心金属原子的不同,氢化酶可分为3类:[NiFe]氢化酶、[FeFe]氢化酶和[Fe]氢化酶[18]。其中,[FeFe]氢化酶的产氢活性最高,其效率是其他2种的10~100倍[19]。通过对WZ-1菌株进行RAST注释,所注释文件中发现了[FeFe]氢化酶和[NiFe]氢化酶(EC:1.12.7.2)的氨基酸序列。通过Cell-PLoc[15]对这两类氢化酶定位,发现都位于细胞质中。
通过KEGG注释,WZ-1菌株的产氢代谢途径可以分为4个主要部分,每个部分都涉及不同的酶和步骤来产生H2。
第1部分是丙酮酸脱羧生成乙酰辅酶A过程中,铁氧还蛋白-氢化酶参与产氢(图4-a)。WZ-1菌株首先利用葡萄糖经糖酵解代谢途径(embden meyerhof parnas pathway,EMP)生成丙酮酸。然后在丙酮酸脱羧途径中,有3种酶参与产氢。1)丙酮酸甲酸裂解酶(EC:1.97.1.4)。注释中pf1A基因编码EC:1.97.1.4酶,该酶是1种铁硫蛋白,常见于大肠杆菌(Escherichia coli)和梭菌属(Clostridium)中[20]。它催化丙酮酸生成乙酰辅酶A、甲酸,并释放能量。生成的甲酸在[NiFe]氢化酶或[FeFe]氢化酶催化下转化为H2和CO2[19];2)酮戊二酸铁氧化还原蛋白氧化还原酶(EC:1.2.7.11)。注释中korA基因编码EC:1.2.7.11酶直接催化丙酮酸转化为乙酰辅酶A和CO2,同时生成还原态铁氧还蛋白(Fdred)和H+。Fdred经电子分岔或被[FeFe]氢化酶用来还原质子,生成H2。这一过程在吴志宇[21]的研究中也有所记载;3)丙酮酸铁还蛋白氧化还原酶(EC:1.2.7.1)。注释中porA基因编码EC:1.2.7.1酶,该酶是一种具有氧化还原活性的铁硫蛋白[22]。WZ-1菌株中,丙酮酸被EC:1.2.7.1酶转化为乙酰COA和CO2,并产生Fdred,电子被用来还原NADP+,最终在[FeFe]氢化酶催化下转化为H2和CO2。这与相关报道的梭菌产氢的途径一致[21, 23]。
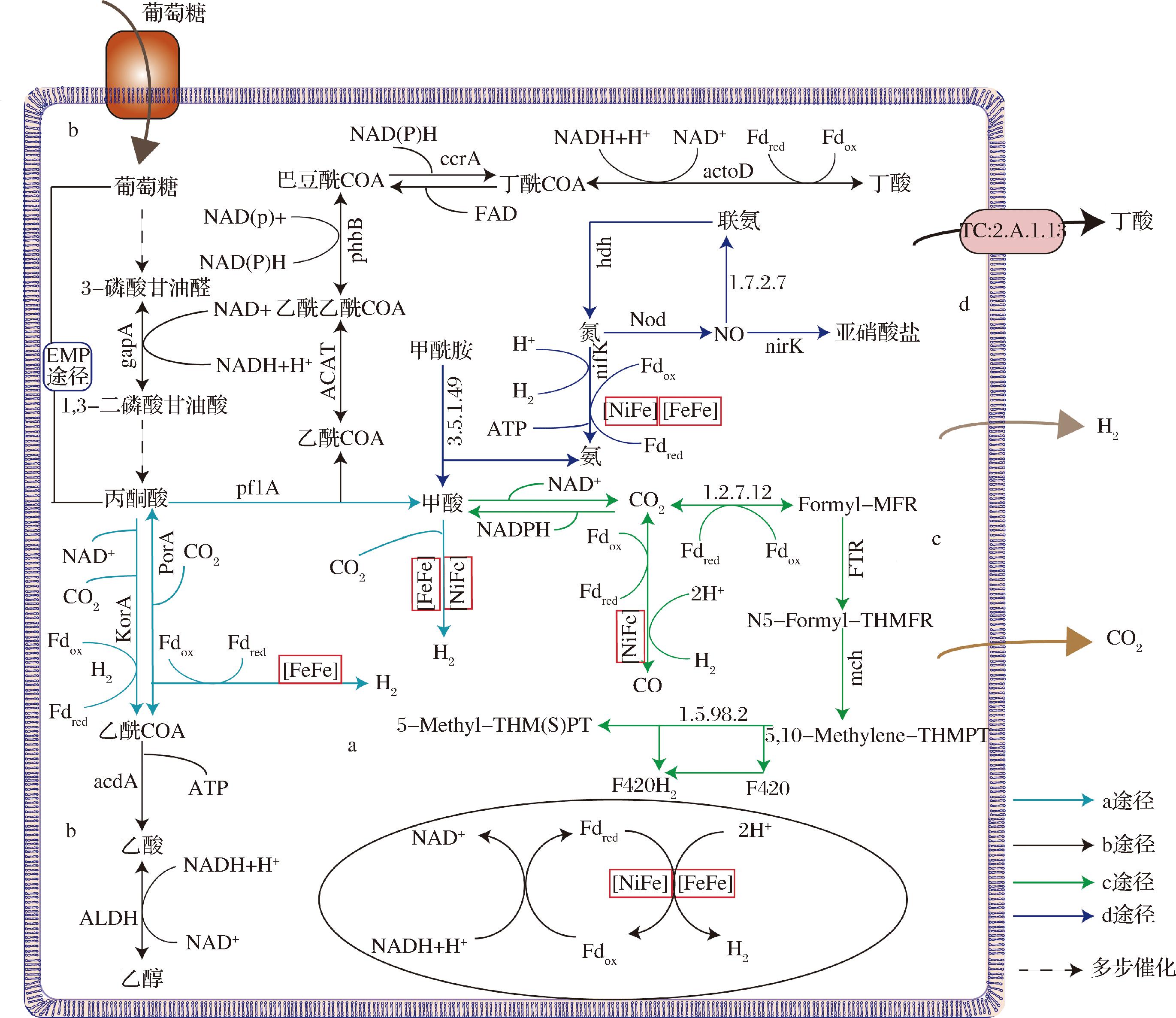
a-丙酮酸脱羧生成乙酰辅酶A中间的铁氧还蛋白-氢化酶途径;b-NADH+H+的氧化还原平衡调节产氢;c-一氧化碳(CO)产氢;d-固氮产氢
图4 WZ-1的产氢途径
Fig.4 Hydrogen production pathway of WZ-1
第2部分是NADH+H+氧化还原平衡调节产氢(图4-b)。在生物体内,NAD+和NADH+H+比例的平衡对细胞的正常生理代谢至关重要[24]。当NADH+H+的生成速率超过其被氧化速度时,可能会导致NADH+H+的积累,从而干扰细胞代谢。为了避免这种干扰,一些发酵细菌会通过释放H2来氧化多余的NADH+H+[24]。如在 WZ-1 菌株中,丙酮酸首先通过一系列酶催化转化为巴豆酰辅酶A(如图4),随后在丁酰辅酶 A 脱氢酶(ccrA)的作用下,巴豆酰辅酶A被还原为丁酰辅酶A。在这个过程中,NADH 作为电子供体参与了反应,并与铁蛋白(Fd)的内源性还原过程相结合。还原的铁蛋白(Fd)再循环产生H2。通过这一对相互关联的氧化还原反应,NADH 的氧化过程得以与 H2 的产生过程相耦合,这不仅有助于维持 NAD+ 和 NADH+H+ 的平衡,还可以额外产生 ATP。总体反应方程:
NADH+2Fdred+3H+→2H2+NAD++2Fdox
在丙酮酸脱羧的过程中,会产生大量的NADH和H+。在多种酶的作用下,丙酮酸进入不同的代谢路径,NADH在这些路径中得以还原为NAD+。同时,NADH释放的电子通过氢化酶的作用转移到H+上,最终形成H2。
第3部分CO产氢(如图4-c)。在研究微生物利用CO产氢的过程中,关键酶是一氧化碳脱氢酶(CODH)[21]。通过对WZ-1菌株进行KAAS注释,发现该菌株具备两种一氧化碳脱氢酶:有氧一氧化碳脱氢酶(EC:1.2.5.3)和厌氧一氧化碳脱氢酶(EC:1.2.7.4)。在WZ-1菌株中,coxS基因负责编码EC:1.2.5.3酶,这种酶在有氧条件下能够催化CO氧化,将其转化为CO2。该酶的结构特征包含两个[2Fe-2S]簇和黄素腺嘌呤二核苷酸,属于黄嘌呤氧化还原酶家族[25]。在转化过程中,产生的CO2被卡尔文循环吸收,而电子则通过黄素腺嘌呤二核苷酸位点转移到醌上,进而通过电子转移链转移到双氧末端受体[25]。这一过程反应式可表示为:
CO+醌+H2O→CO2+醌醇
另一方面,cooS基因编码的EC:1.2.7.4酶在厌氧条件下催化CO的氧化,并将电子转移到铁氧还蛋白上。随后,铁氧还蛋白与[NiFe]氢化酶反应,产生H2。这个过程可细分为两步:
1)CO+H2O+2Fdox→CO2+2Fdred+2H++2e-
2)2H++2Fdred+2e-→H2+2Fdox
在WZ-1菌株中,CO可以通过一氧化碳脱氢酶(EC:1.2.5.3、EC:1.2.7.4)的催化作用,直接转化为CO2和H2。
第4部分固氮产氢(如图4-d)。在WZ-1菌株的基因组注释中,注释到了由nifK基因编码的固氮酶(EC:1.18.6.1),该酶是由两部分(即二氮还原酶和二氮酶)组成的复合体。二氮还原酶是一种含有[4Fe-4S]簇的蛋白质,它在两分子 ATP 的作用下,将电子从铁氧还蛋白转移到二氮酶成分上[26]。二氮酶是一种钼铁蛋白质,它通过重氮和肼将二氮还原成两分子氨,连续进行3次双电子还原,还原开始时会形成一定量的氢[27]。当没有可还原的氮源底物时,固氮酶(EC:1.18.6.1)会将质子还原为H2[21, 28]。在WZ-1菌株的代谢流程中,葡萄糖经EMP途径转化为丙酮酸,同时释放出CO2、H+以及电子。这些电子借助NADH和Fd这两种电子载体,传递给固氮酶。固氮酶则利用这些电子将质子还原,从而产生H2。
综上所述,现将WZ-1菌株中所有参与产氢的酶类总结在表3中。
表3 WZ-1菌株中参与产氢的酶类
Table 3 Enzymes involved in hydrogen production in strain WZ-1
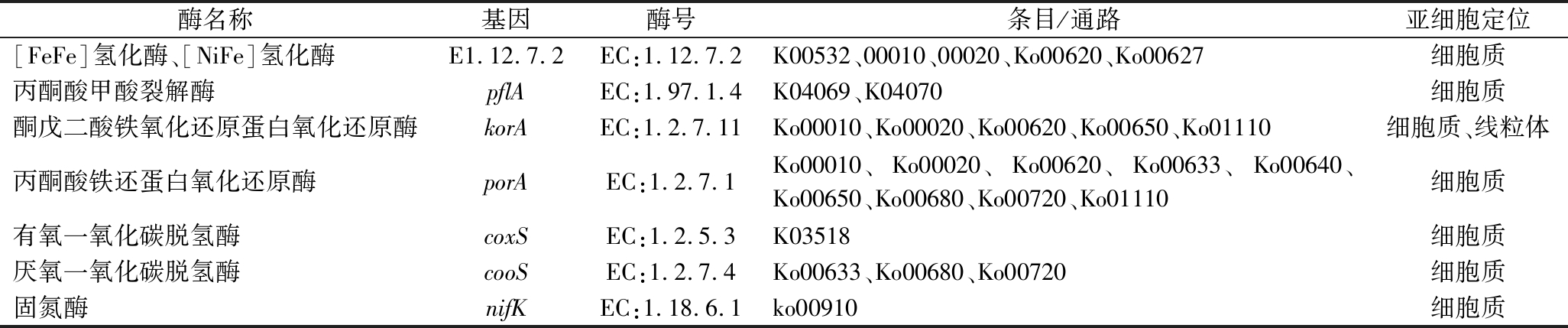
酶名称基因酶号条目/通路亚细胞定位[FeFe]氢化酶、[NiFe]氢化酶E1.12.7.2EC:1.12.7.2K00532、00010、00020、Ko00620、Ko00627细胞质丙酮酸甲酸裂解酶pflAEC:1.97.1.4K04069、K04070细胞质酮戊二酸铁氧化还原蛋白氧化还原酶korAEC:1.2.7.11Ko00010、Ko00020、Ko00620、Ko00650、Ko01110细胞质、线粒体丙酮酸铁还蛋白氧化还原酶porAEC:1.2.7.1Ko00010、Ko00020、Ko00620、Ko00633、Ko00640、Ko00650、Ko00680、Ko00720、Ko01110细胞质有氧一氧化碳脱氢酶coxSEC:1.2.5.3K03518细胞质厌氧一氧化碳脱氢酶cooSEC:1.2.7.4Ko00633、Ko00680、Ko00720细胞质固氮酶nifKEC:1.18.6.1ko00910细胞质
3 结论
本研究旨在深入探究我国传统浓香型白酒生态系统中重要微生物C.guangxiense的产氢代谢机制。通过采用基因组测序技术,并结合KEGG代谢途径分析,全面解析了WZ-1菌株与H2代谢相关的代谢途径和基因。这不仅为今后深入研究白酒梭菌属产氢的生成机理提供了宝贵的基因层面数据支持,同时也为进一步发掘梭菌属产氢的功能应用价值奠定了坚实的基础。
白酒生产过程中,微生物发酵产生的H2不仅可以作为清洁能源加以利用,还能够促进白酒发酵过程的稳定性和风味形成。H2具有还原性,可以创造适宜白酒发酵所需的厌氧环境,同时也能够抑制有害微生物的生长,维持白酒发酵过程中微生物群落的平衡。此外,H2还可以参与白酒风味物质的形成,如通过与醇类、酯类等化合物发生加氢反应,产生一些特殊的风味物质,从而提升白酒的品质和风味。因此,利用微生物发酵制氢技术,不仅能够产生清洁能源H2,还可以实现经济和环境效益的双赢。
尽管目前微生物发酵制氢的产氢量相对较低,但其反应装置和工艺简单、速度快、底物范围广、成本低廉等优势使其在未来生物制氢领域具有广阔的发展前景。通过进一步的研究和技术创新,提高微生物发酵制氢的产氢效率和经济性,必将推动微生物发酵制氢技术的商业化应用,为世界经济的可持续发展做出重大贡献。
总之,本研究通过解析C.guangxiense WZ-1菌株的产氢代谢机制,不仅丰富了人们对白酒生态系统中重要微生物代谢特性的认识,而且为白酒工业的可持续发展提供了新的机遇和挑战。随着对梭菌属产氢机制的不断深入研究和相关技术的进一步创新,微生物发酵制氢在白酒工业及能源领域将发挥着越来越重要的作用,为行业发展和可持续发展贡献力量。
[1] ABE J O, POPOOLA A P I, AJENIFUJA E, et al.Hydrogen energy, economy and storage:Review and recommendation[J].International Journal of Hydrogen Energy, 2019, 44(29):15072-15086.
[2] 叶思岑. 梭状芽孢杆菌属近缘产氢菌种的产氢与代谢差异研究[D].沈阳:东北大学, 2021.
YE S C.Differences in hydrogen production and metabolism in closely related species of Clostridium[D].Shenyang:Northeastern University, 2021.
[3] WANG J L, YIN Y N.Progress in microbiology for fermentative hydrogen production from organic wastes[J].Critical Reviews in Environmental Science and Technology, 2019, 49(10):825-865.
[4] 姜淑敏, 汪吉霞, 王悦佳, 等.光合细菌产氢研究进展[J].现代农业科技, 2023(19):136-142.
JIANG S M, WANG J X, WANG Y J, et al.Research progress on hydrogen production by photosynthetic bacteria[J].Modern Agricultural Science and Technology, 2023(19):136-142.
[5] ZHAO X, LI D Y, XU S H, et al.Clostridium guangxiense sp.nov.and Clostridium neuense sp.nov., two phylogenetically closely related hydrogen-producing species isolated from lake sediment[J].International Journal of Systematic and Evolutionary Microbiology, 2017, 67(3):710-715.
[6] 杨静, 齐男, 赵鑫, 等.复合有机氮对产氢新菌C.guangxiense产氢的促进作用[J].可再生能源, 2019, 37(3):317-321.
YANG J, QI N, ZHAO X, et al.Promoting of compound organic nitrogen on hydrogen production by the novel species of C.guangxiense[J].Renewable Energy Resources, 2019, 37(3):317-321.
[7] 黄治国, 蒲领平, 罗惠波, 等.浓香型白酒酯化菌协同产酯的工艺优化[J].现代食品科技, 2021, 37(2):64-71;146.
HUANG Z G, PU L P, LUO H B, et al.Optimization of the conditions for synergistic production of esters by esterifying bacteria of luzhou-flavor liquor[J].Modern Food Science and Technology, 2021, 37(2):64-71;146.
[8] 郭燕, 邓杰, 任志强, 等.响应面优化酿酒酵母与窖泥酯化细菌协同发酵产丁酸乙酯和己酸乙酯[J].食品科学, 2021, 42(10):209-217.
GUO Y, DENG J, REN Z Q, et al.Optimization of the production of ethyl hexanoate and ethyl butyrate by cofermentation of Saccharomyces cerevisiae and esterifying bacteria from pit mud of Chinese Baijiu using response surface methodology[J].Food Science, 2021, 42(10):209-217.
[9] SCHUBERT M, LINDGREEN S, ORLANDO L.AdapterRemoval v2:Rapid adapter trimming, identification, and read merging[J].BMC Research Notes, 2016, 9:88.
[10] LUO R B, LIU B H, XIE Y L, et al.SOAPdenovo2:An empirically improved memory-efficient short-read de novo assembler[J].GigaScience, 2012, 1(1):18.
[11] BANKEVICH A, NURK S, ANTIPOV D, et al.SPAdes:A new genome assembly algorithm and its applications to single-cell sequencing[J].Journal of Computational Biology, 2012, 19(5):455-477.
[12] WALKER B J, ABEEL T, SHEA T, et al.Pilon:An integrated tool for comprehensive microbial variant detection and genome assembly improvement[J].PLoS One, 2014, 9(11):e112963.
[13] BESEMER J, LOMSADZE A, BORODOVSKY M.GeneMarkS:A self-training method for prediction of gene starts in microbial genomes.Implications for finding sequence motifs in regulatory regions[J].Nucleic Acids Research, 2001, 29(12):2607-2618.
[14] KALVARI I, ARGASINSKA J, QUINONES N, et al.Rfam 13.0:Shifting to a genome-centric resource for non-coding RNA families[J].Nucleic Acids Research, 2017, 46:D335-D342.
[15] CHOU K C, SHEN H B.Cell-PLoc:A package of Web servers for predicting subcellular localization of proteins in various organisms[J].Nature Protocols, 2008, 3(2):153-162.
[16] LUO H, LI T, ZHENG J, et al.Isolation, identification, and fermentation medium optimization of a caproic acid-producing Enterococcus casseliflavus strain from pit mud of Chinese strong flavor Baijiu ecosystem[J].Polish Journal of Microbiology, 2022, 71(4):563-575.
[17] CONESA A, GÖTZ S.Blast2GO:A comprehensive suite for functional analysis in plant genomics[J].International Journal of Plant Genomics, 2008, 2008:619832.
[18] ARRIAZA-GALLARDO F J, ZHENG Y C, GEHL M, et al.Fe]-hydrogenase, cofactor biosynthesis and engineering[J].Chembiochem, 2023, 24(20):e202300330.
[19]  UKAJTIS R, HO
UKAJTIS R, HO OWACZ I, KUCHARSKA K, et al.Hydrogen production from biomass using dark fermentation[J].Renewable and Sustainable Energy Reviews, 2018, 91:665-694.
OWACZ I, KUCHARSKA K, et al.Hydrogen production from biomass using dark fermentation[J].Renewable and Sustainable Energy Reviews, 2018, 91:665-694.
[20] KNAPPE J, BLASCHKOWSKI H P, GRÖBNER P, et al.Pyruvate formate-lyase of Escherichia coli:The acetyl-enzyme intermediate[J].European Journal of Biochemistry, 1974, 50(1):253-263.
[21] 吴志宇. 纳米水铁矿对梭菌菌株BZ-1发酵产氢的促进机制研究[D].烟台:烟台大学, 2022.
WU Z Y.Promoting mechanism of ferrihydrite nanomaterials on hydrogen fermentation by Clostridium sp.BZ-1[D].Yantai:Yantai University, 2022.
[22] MENON A L, HENDRIX H, HUTCHINS A, et al.The delta-subunit of pyruvate ferredoxin oxidoreductase from Pyrococcus furiosus is a redox-active, iron-sulfur protein:Evidence for an ancestral relationship with 8Fe-type ferredoxins[J].Biochemistry, 1998, 37(37):12838-12846.
[23] BRAGA J K, STANCARI R A, MOTTERAN F, et al.Metals addition for enhanced hydrogen, acetic and butyric acids production from cellulosic substrates by Clostridium butyricum[J].Biomass and Bioenergy, 2021, 150:105679.
[24] 尹梦. 丁酸梭菌的产氢影响因素及其代谢特性的研究[D].上海:上海师范大学, 2020.
YIN M.The influencing factors in hydrogen production and metabolic characteristics of Clostridium butyricum[D].Shanghai:Shanghai Normal University, 2020.
[25] WILCOXEN J, ZHANG B, HILLE R.Reaction of the molybdenum- and copper-containing carbon monoxide dehydrogenase from Oligotropha carboxydovorans with quinones[J].Biochemistry, 2011, 50(11):1910-1916.
[26] RAPSON T D, WOOD C C.Analysis of the ammonia production rates by nitrogenase[J].Catalysts, 2022, 12(8):844.
[27] DAVID BRITT R, RAO G D, TAO L Z.Bioassembly of complex iron-sulfur enzymes:Hydrogenases and nitrogenases[J].Nature Reviews.Chemistry, 2020, 4(10):542-549.
[28] WANG Z C, WATT G D.Large anions induce H2-production from the nitrogenase MoFe proteins of Clostridium Pasteurianum and Azotobacter vinelandii[J].Journal of Inorganic Biochemistry, 2020, 208:111075.