香叶基香叶醇(geranylgeraniol,GGOH)是一种直链二萜化合物,具有抗肿瘤、杀菌等活性,主要应用于医药领域[1]。香叶基香叶醇也是多种植物天然产物和多种药物的合成前体和有机合成中间体,如萜类、胆固醇、辅酶Q10、维生素K2等[2]。香叶基香叶醇主要存在于植物油脂中,通过植物提取或化学合成产生香叶基香叶醇不可避免的存在产量低、污染等问题[3]。合成生物学的快速发展使得构建微生物细胞工厂以实现天然萜类化合物的高效合成成为一种有前途的研究方向。与植物提取法和化学合成法相比,微生物合成法具有能耗低、环境友好、产品质量高等优点,是一种可用于大规模工业化生产的方法[4-5]。
酿酒酵母由于其遗传背景清晰、基因操作手段成熟、具有很强的食品安全性,同时具备大规模发酵生产的能力,已被作为底盘细胞生产香叶基香叶醇[6-11],而且酿酒酵母中具有内源的甲羟戊酸途径 (mevalonate pathway, MVA)可以直接为萜类物质合成提供前体,同时,在酿酒酵母中通过同源重组的方式将基因整合到酿酒酵母基因组上,具有较高的遗传稳定性。目前,酿酒酵母生产香叶基香叶醇在摇瓶和5 L发酵罐上的最高产量分别达到了1 271.14 mg/L及6.33 g/L[9-10]。
本研究首先强化了MVA通路的所有基因来增加角鲨烯和香叶基香叶醇的产量。然后,研究了引入外源香叶基香叶基焦磷酸(geranylgeranyl diphosphate,GGPP)合酶crtE、下调角鲨烯合酶基因(squalene synthase, ERG9)的表达减少竞争途径的消耗和敲除不同的调控因子对香叶基香叶醇产量的影响。同时,通过过表达磷酸戊糖途径(pentose phosphate pathway)基因和增加胞质乙酰辅酶A的供应,为香叶基香叶醇的合成提供更多的前体。通过增加融合基因的拷贝数进一步提高了香叶基香叶醇的产量。主要代谢途径如图1所示。最后在5 L发酵罐中通过分批补料发酵高效生产香叶基香叶醇。本研究为香叶基香叶醇的生产提供了一种高效、绿色的工艺。
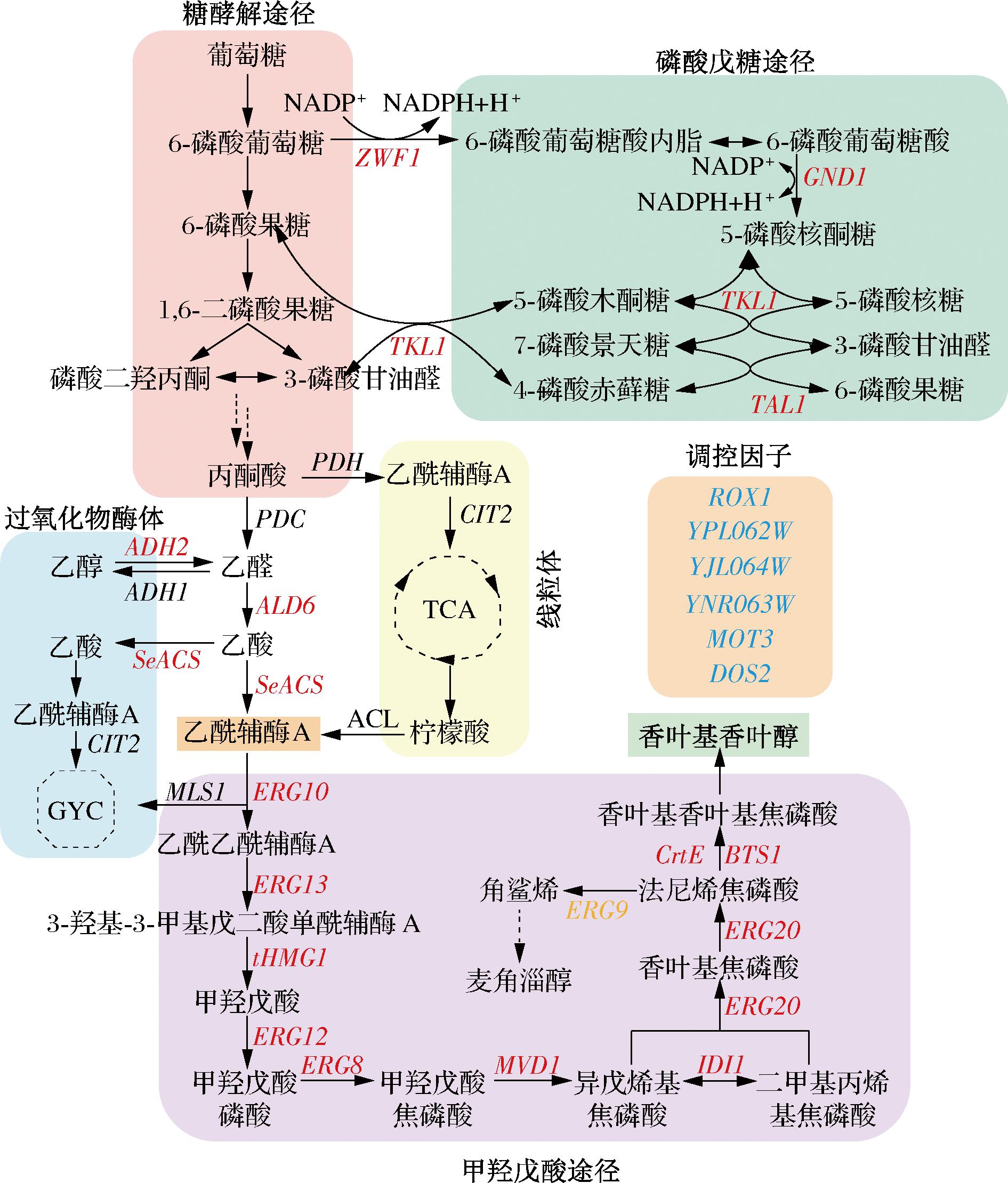
图1 本研究涉及的代谢途径示意图
Fig.1 Schematic diagram of metabolic pathways involved in this study
注:红色字体表示过表达的基因,橙色字体表示下调的基因,蓝色字体表示敲除的基因;GYC:乙醛酸循环途径, glyoxylic acid cycle pathway;TCA:三羧酸循环,tricarboxylic acid cycle;ERG10:乙酰-乙酰辅酶A硫解酶,acetyl-acetyl-CoA thiolase;ERG13:羟甲基戊二酰辅酶A合成酶,hydroxymethyl glutaryl CoA synthetase;tHMG1:截短的羟甲基戊二酰辅酶A还原酶,truncated hydroxymethyl glutaryl CoA reductase;ERG12:甲羟戊酸激酶,mevalonate kinase;ERG8:磷酸甲羟戊酸激酶,phosphomevalonate kinase;MVD1:ERG19,焦磷酸甲羟戊酸脱羧酶,mevalonate pyrophosphate decarboxylase;IDI1:异戊烯焦磷酸异构酶,isopentene pyrophosphate isomerase;ERG20:法尼基焦磷酸合酶,farnesyl pyrophosphate synthase;ERG9:角鲨烯合酶,squalene synthase;BTS1/crtE:香叶基香叶基焦磷酸合酶,geranylgeranyl pyrophosphate synthase;DPP1:磷酸水解酶,phosphohydrolase;SeACS:来自Salmonella enterica的乙酰辅酶A合酶,acetyl-CoA synthase;ALD6:乙醛脱氢酶,aldehyde dehydrogenase;ADH2:乙醇脱氢酶,alcohol dehydrogenase;CIT2:柠檬酸合酶,citrate synthase;MLS1:苹果酸合酶,malate synthase;ZWF1:葡萄糖-6-磷酸脱氢酶,glucose-6-phosphate dehydrogenase;GND1:磷酸葡萄糖酸脱氢酶,phosphogluconate dehydrogenase;TKL1:转酮醇酶,transketolase;TAL1:转醛缩酶,transaldolase。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本研究使用的初始菌株酿酒酵母(Saccharomyces cerevisiae) CEN.PK2-1C为本实验室保藏,大肠杆菌(Escherichia coli) DH5α用于gRNA质粒构建。本研究所使用的所有菌株列于表1。本研究所使用的所有质粒列于表2。
表1 本研究所使用的菌株
Table 1 Strains used in this study

菌株描述来源E.coli DH5α野生型E.coli实验室保藏S.cerevisiaeCEN.PK2-1CMata;ura3-52, trp1-289, leu2-3,112,his3Δ1;MAL2-8c;SUC2实验室保藏YCP1S.cerevisiae CEN.PK2-1C,Δdpp1::PGAP1-BTS1-ERG20-TPGI1-PENO2-tHMG1-TPDC1-PTEF2-IDI1-TENO2本研究YCP2YCP1Δade1::PCCW12-ERG10-TADH1-PTDH3-ERG12-TTDH3-PGPD1-ERG19-TPGK1本研究YCP3YCP2 Δlpp1::TGPM1-ERG20-PPGK1-PTEF1-IDI1-TACT1本研究YCP4YCP3 Δndt80::PRPR1-ERG13-TPGK1-PTPI1-ERG8-TCYC1-PGPD1-BTS1-TPGK1本研究YCP5YCP4 Δgal80本研究YCP6YCP5 Δyprcτ3::PGAL1-tHMG1-TCYC1本研究YG1YCP6 yorwΔ17::PGAL1-crtE-TCYC1本研究YG2YCP6 yorwΔ17::PGAL1-crtE03M-TCYC1本研究YG3YCP6 yorwΔ17::PGAL1-crtE-DPP1-TCYC1本研究YG4YCP6 yorwΔ17::PGAL1-crtE03M-DPP1-TCYC1本研究YCP7YCP6 Δho::PGAL7-tHMG1-TADH1本研究YCP8YG4 ΔPERG9::PERG1-ERG9本研究YCP9YG4 ΔPERG9::PERG7-ERG9本研究YCP10YG4 ΔPERG9::PHXT1-ERG9本研究YCP11YCP8 Δynr063w本研究YCP12YCP8 Δyjl064w本研究YCP13YCP8 Δdos2本研究YCP14YCP8 Δrox1本研究YCP15YCP8 Δmot3本研究YCP16YCP8 Δypl062w本研究YCP17YCP12 Δho::TCYC1-ZWF1-PGAL10-PGAL1-GND1-TADH1本研究YCP18YCP12 ΔXI-3::TCYC1-TKL1-PGAL10-PGAL1-TAL1-TADH1本研究YCP19YCP18 Δho::TCYC1-ZWF1-PGAL10-PGAL1-GND1-TADH1本研究YCP20YCP19 yorwΔ22::PGAL1-SeACSL641P-TPGI1本研究YCP21YCP20 ymrwΔ15::PGAL1-crtE03M-ERG20-TCYC1本研究YCP22YCP21 Δynr063w::PGAL1-crtE03M-ERG20-TCYC1本研究YCP23YCP22 Δx2::PGAL1-crtE03M-DPP1-TCYC1本研究
表2 本研究所使用的质粒
Table 2 Plasmids used in this study
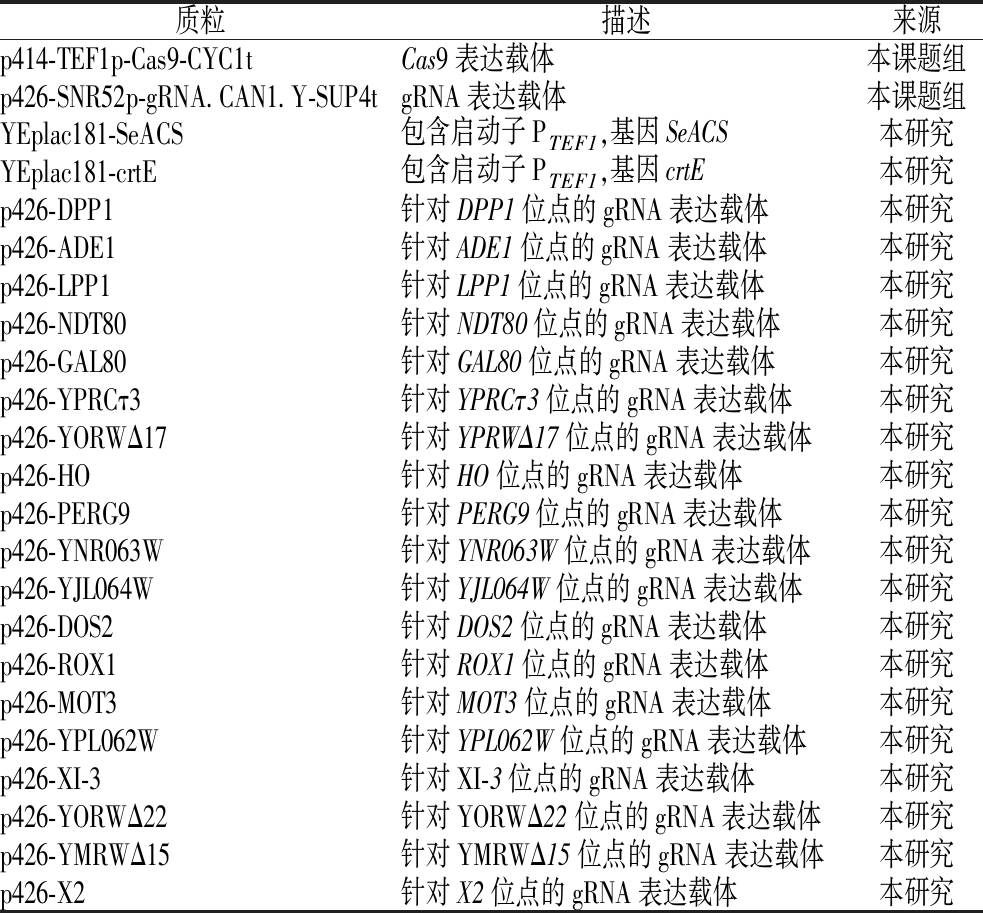
质粒描述来源p414-TEF1p-Cas9-CYC1tCas9表达载体本课题组p426-SNR52p-gRNA.CAN1.Y-SUP4tgRNA表达载体本课题组YEplac181-SeACS包含启动子PTEF1,基因SeACS本研究YEplac181-crtE包含启动子PTEF1,基因crtE本研究p426-DPP1针对DPP1位点的gRNA表达载体本研究p426-ADE1针对ADE1位点的gRNA表达载体本研究p426-LPP1针对LPP1位点的gRNA表达载体本研究p426-NDT80针对NDT80位点的gRNA表达载体本研究p426-GAL80针对GAL80位点的gRNA表达载体本研究p426-YPRCτ3针对YPRCτ3位点的gRNA表达载体本研究p426-YORWΔ17针对YPRWΔ17位点的gRNA表达载体本研究p426-HO针对HO位点的gRNA表达载体本研究p426-PERG9针对PERG9位点的gRNA表达载体本研究p426-YNR063W针对YNR063W位点的gRNA表达载体本研究p426-YJL064W针对YJL064W位点的gRNA表达载体本研究p426-DOS2针对DOS2位点的gRNA表达载体本研究p426-ROX1针对ROX1位点的gRNA表达载体本研究p426-MOT3针对MOT3位点的gRNA表达载体本研究p426-YPL062W针对YPL062W位点的gRNA表达载体本研究p426-XI-3针对XI-3位点的gRNA表达载体本研究p426-YORWΔ22针对YORWΔ22位点的gRNA表达载体本研究p426-YMRWΔ15针对YMRWΔ15位点的gRNA表达载体本研究p426-X2针对X2位点的gRNA表达载体本研究
1.1.2 培养基
LB液体培养基(g/L):蛋白胨10,NaCl 10,酵母提取物5,固体培养基添加20 g/L的琼脂,121 ℃下高压蒸汽灭菌15 min。
YPD培养基(g/L):葡萄糖20,蛋白胨20,酵母提取物10,固体培养基添加20 g/L的琼脂,115 ℃下高压蒸汽灭菌20 min。
SD/-Trp-Ura培养基(g/L):酵母氮源基础(YNB)粉末6.7,葡萄糖20,固体培养基添加20 g/L的琼脂,115 ℃下高压蒸汽灭菌20 min。灭菌后加入过膜除菌的组氨酸80 mg/L,亮氨酸400 mg/L。
SD/-Trp培养基:在SD/-Trp-Ura培养基基础上添加尿嘧啶80 mg/L。
优化培养基(g/L):葡萄糖20,酵母粉30,蛋白胨30,MgSO4·7H2O 2,KH2PO4 2,115 ℃下高压蒸汽灭菌20 min。
1.1.3 主要试剂
2×Taq PCR mix DNA聚合酶和2×phanta flash master mix DNA聚合酶,南京诺唯赞生物科技有限公司;GeneJET柱纯化回收试剂盒,Thermo Fisher Scientific;大肠杆菌超级感受态制备试剂盒、琼脂粉,上海生工生物工程有限公司;质粒小量提取试剂盒,翌圣生物科技;蛋白胨、酵母提取物,英国Oxoid公司;葡萄糖、MgSO4·7H2O、KH2PO4,国药集团化学试剂有限公司;十二烷,上海阿拉丁生化科技股份有限公司;香叶基香叶醇标准品(≥97%),上海甄准生物科技有限公司;角鲨烯标准品(≥98%),上海麦克林生化科技有限公司。
1.2 仪器与设备
Airtech超净工作台,苏州安泰公司;PCR仪、凝胶成像仪,美国Bio-Rad公司;核酸凝胶电泳仪,北京六一电泳仪;紫外可见光分光光度计,上海美普达仪器公司;制冰机,日本Panasonic公司;电子天平,瑞士Mettler-Toledo仪器有限公司;恒温摇床,上海知楚仪器有限公司;恒温培养箱,上海跃进有限公司;高效液相色谱仪,日本Shimadzu公司;5 L发酵罐,上海迪必尔生物工程有限公司。
1.3 实验方法
1.3.1 质粒和菌株的构建
gRNA质粒构建:利用开源工具[CHOPCHOP (uib.no)]获得针对不同位点的特异性gRNA序列。设计引物将特异性gRNA序列插入gRNA表达载体p426-crRNA,采用PCR扩增重组质粒,PCR产物转化大肠杆菌DH5α,挑取正确的转化子于LB培养基中培养用于提取质粒。
菌株构建:利用CRISPR/Cas9系统对酿酒酵母进行基因组编辑。以S.cerevisiae CEN.PK2-1C为模板,扩增目标位点上下游同源臂序列及启动子、终止子序列,将基因序列与上下游同源臂序列、启动子和终止子序列通过融合PCR连接,构建基因表达框。采用醋酸锂法将gRNA质粒和基因表达框转化酿酒酵母,利用含相应缺陷型标记的固体培养基(SD/-Trp/-Ura)对重组菌进行筛选。转化成功后利用含5-氟乳清酸的固体培养基(SD/-Trp+5-FOA)去除gRNA质粒。
1.3.2 菌株培养方法
种子液的培养:从固体琼脂平板中挑选一个单菌落到含有5 mL YPD培养基的无菌离心管中,在30 ℃,220 r/min下培养至对数生长期获得一级种子液,然后按5%的接种量接入装有50 mL YPD培养基的三角瓶中,30 ℃,220 r/min振荡培养至对数生长期,获得二级种子液。
摇瓶发酵培养:取适量种子液以5%的接种量接种于装有50 mL YPD培养基的250 mL摇瓶中,置于30 ℃,220 r/min条件下培养。12 h加入10%(体积比)的十二烷进行双相萃取发酵,发酵周期为144 h。发酵结束后收集有机层进行香叶基香叶醇分析。
5 L发酵罐分批补料发酵:将二级种子液以5%的接种量接种到2 L 优化培养基中。温度保持在30 ℃,通过添加氢氧化铵,pH值保持在5.5。气流量为4 L/min,搅拌速率设置为800 r/min。在葡萄糖质量浓度<20 g/L时,补料高质量浓度(600 g/L)葡萄糖溶液,提供碳源的同时控制葡萄糖质量浓度<20 g/L,避免高浓度葡萄糖对GAL启动子产生抑制[12]。此外,以20 mL/h的恒定速率流加10%酵母提取物溶液,为细胞生长和产物合成提供足够的营养。在发酵20 h后,将10%(体积分数)十二烷的萃取相加入到培养基中,开始两相萃取发酵。
1.3.3 香叶基香叶醇的提取与检测
样品处理:发酵结束后,将发酵液收集在50 mL离心管中,5 000 r/min离心5 min,收集上层有机相,在12 000 r/min下离心2 min,每个样品用正十二烷进行适当稀释,使用0.22 μm的有机滤膜过滤。
样品检测:本实验香叶基香叶醇的测定参考文献[9]采用HPLC进行分析:色谱柱为HypersilTM ODS-2 C18色谱柱(4.6 mm×250 mm,5 μm,Thermo),紫外吸收检测器波长设置为210 nm。流动相为V(甲醇)∶V(乙腈)∶V(水)=90∶5∶5的混合物,流速为0.8 mL/min。柱温设置为40 ℃,持续时间为15 min。使用香叶基香叶醇标准品配制溶液测定标准曲线用于定量。
1.3.4 角鲨烯的提取与检测
样品制备:收集用于提取角鲨烯的酵母菌培养物500 μL转移至2 mL破碎管12 000 r/min 离心2 min后弃去上清液,将保留的细胞沉淀与相同体积的研磨珠(0.5 mm)混合,添加1.5 mL丙酮,使用酿酒酵母FastPrep程序将细胞裂解4~6个周期。角鲨烯在上层有机相中提取用于检测。
角鲨烯的测定:采用GC-MS进行测定,色谱柱为DB-5MS(30 m×0.25 mm,0.25 μm,Agilent)检测条件:初始柱箱温度90 ℃,恒温1 min,以20 ℃/min升温至280 ℃,保持15 min,载气为氦气,流量为1.17 mL/min,扫描范围为35~500 m/z,持续时间为25 min。使用角鲨烯标准品配制溶液测定标准曲线用于定量。
1.4 数据处理
实验结果为3次平行实验的平均值。采用Origin、Adobe Illustrator软件对实验数据进行处理。
2 结果与分析
2.1 强化MVA途径基因表达
本研究使用强组成型启动子将MVA基因进行过表达构建菌株YCP1、YCP2、YCP3、YCP4,在YPD培养基中发酵144 h后,如图2所示,菌株YCP1-4的香叶基香叶醇产量分别为40.9、45.7、53.5、67.1 mg/L,而CEN.PK2-1C中几乎未检测不到香叶基香叶醇及角鲨烯。香叶基香叶醇的HPLC检测图谱如图3所示。接着,本研究探究了在GAL1启动子调控下增加tHMG1的拷贝数对于香叶基香叶醇产量的影响。YCP6产生了168.0 mg/L的香叶基香叶醇,相较于YCP4增加了1.5倍,同时,角鲨烯的积累量(321.0 mg/L)也得到提升,但是,菌株YCP7中的香叶基香叶醇及角鲨烯均出现下降趋势,这可能是因为强化3个拷贝的tHMG1对菌株的生长造成了负担,这与以往所报道的结果相符合[13],因此选择YCP6进行后续实验研究。
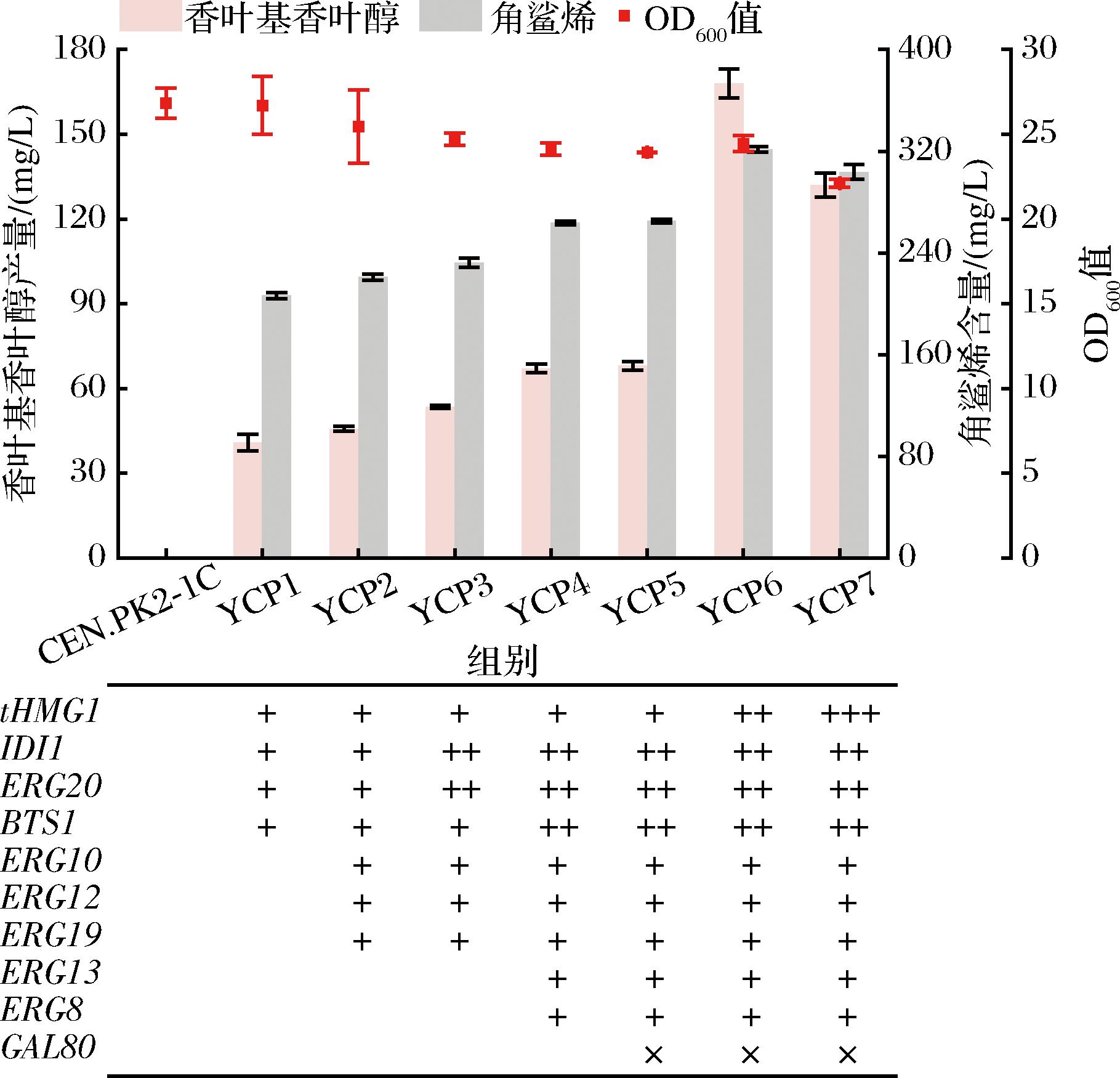
图2 强化MVA途径基因表达对香叶基香叶醇产量的影响
Fig.2 Effect of enhancing MVA pathway gene expression on the production of geranylgeraniol
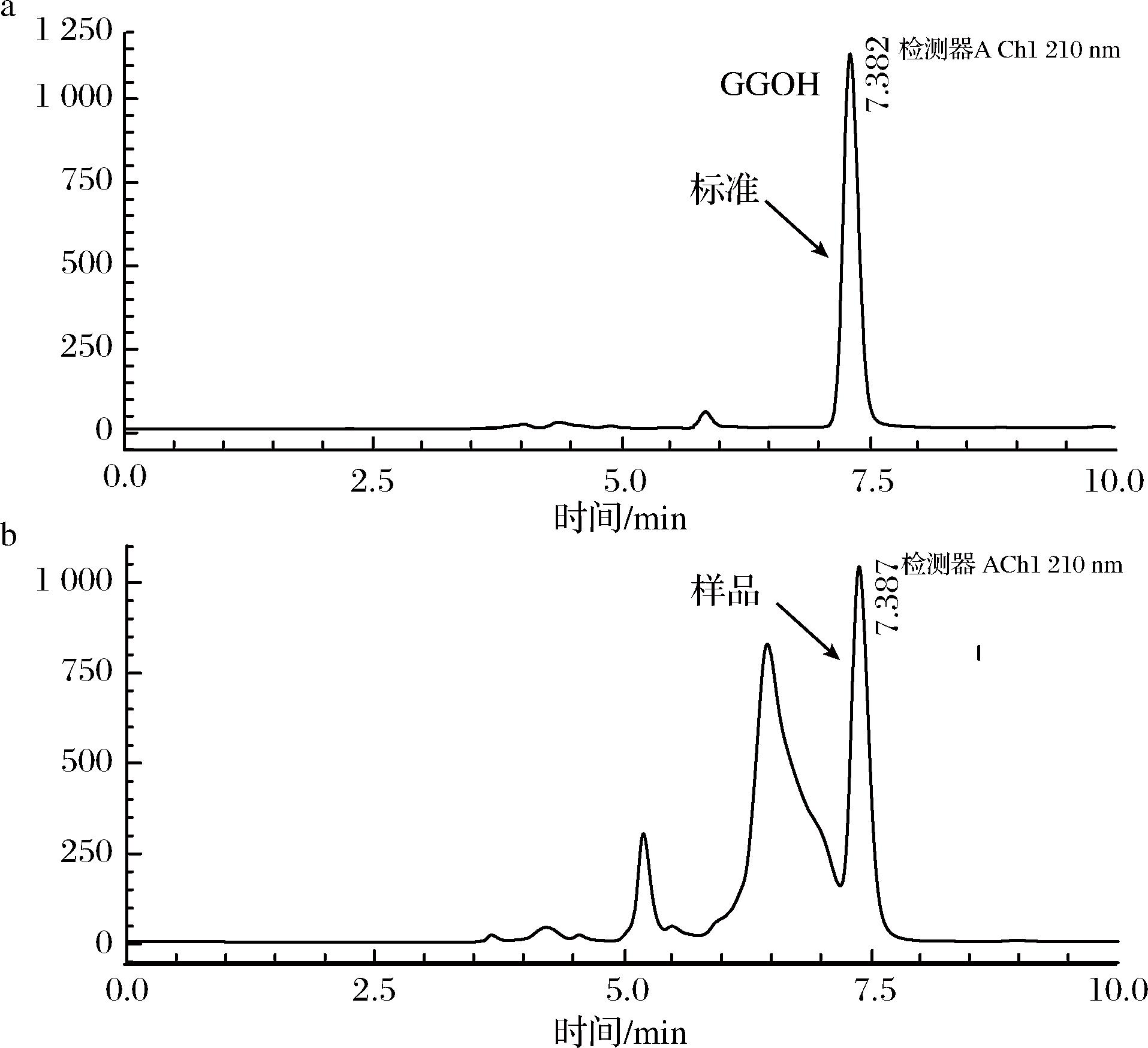
a-香叶基香叶醇;b-样品
图3 香叶基香叶醇样品和标准的高效液相色谱峰图
Fig.3 Chromatographic peaks analyzed by HPLC for the sample and standard of geranylgeraniol
2.2 香叶基香叶基焦磷酸合酶与磷酸水解酶的融合表达
研究证明融合基因BTS1-DPP1的共表达使代谢通量几乎完全从角鲨烯转变为香叶基香叶醇的生产,通过表达BTS1-DPP1使香叶基香叶醇的产量提高了2.9倍[14]。来自红法夫酵母(Phaffia rhodozyma)的香叶基香叶基焦磷酸合酶基因CrtE的突变体CrtE03M由于密码子使用效率的提高而效果更佳[15],本研究将CrtE及突变体CrtE03M与DPP1融合对香叶基香叶醇产量的影响。在GAL1启动子的调控下,将CrtE、CrtE03M、CrtE-DPP1、CrtE03M-DPP1分别整合到菌株YCP6的染色体上构建菌株YG1-4,如图4所示,发酵144 h后YG1-4分别产生186.5、192.5、294.1、312.4 mg/L的香叶基香叶醇,以及213.3、203.0、185.9、175.1 mg/L的角鲨烯,与YCP6相比明显下降,这些结果表明CrtE03M-DPP1融合表达可以促进FPP向GGPP和香叶基香叶醇转化,同时减少FPP向角鲨烯合成路径的消耗。
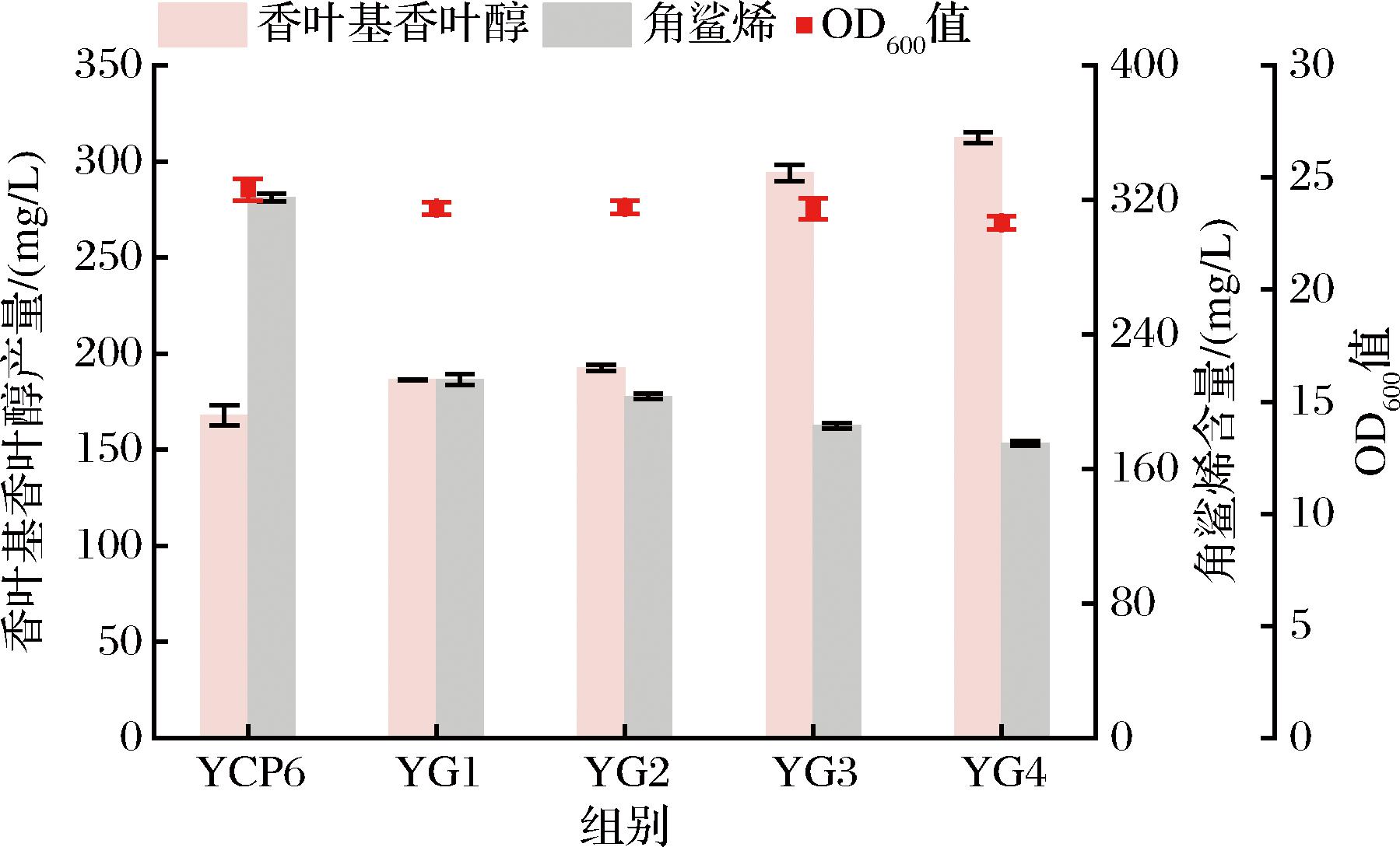
图4 融合基因的表达对香叶基香叶醇产量的影响
Fig.4 Effect of expressing the fusion gene on the production of geranylgeraniol
2.3 下调角鲨烯合成竞争途径
虽然强化MVA通路基因表达和CrtE03M-DPP1融合表达可以增强GGPP和香叶基香叶醇的合成,但是角鲨烯仍有较高的积累量,表明FPP对甾醇通路的消耗仍然限制了香叶基香叶醇的产生。ERG9是酿酒酵母的角鲨烯合酶,它将FPP转化为角鲨烯。它是三萜和麦角甾醇生物合成的分支途径中的第1个酶,其高表达消耗了大量的FPP[16]。但由于麦角甾醇的生物合成是必不可少的。本研究在菌株YG4上使用PERG1、PERG7、PHXT1三种弱启动子代替ERG9启动子,构建菌株YCP8、YCP9、YCP10,如图5所示,在YPD摇瓶培养基中培养144 h后,YCP8、YCP9、YCP10的角鲨烯积累量从175.1 mg/L分别下降至64.6、62.9、49.0 mg/L,香叶基香叶醇的产量分别增加至421.4、330.5、386.9 mg/L。使用弱启动子替换ERG9启动子成功下调了FPP向角鲨烯的转化,使FPP流量更多向GGPP及香叶基香叶醇的合成流入。
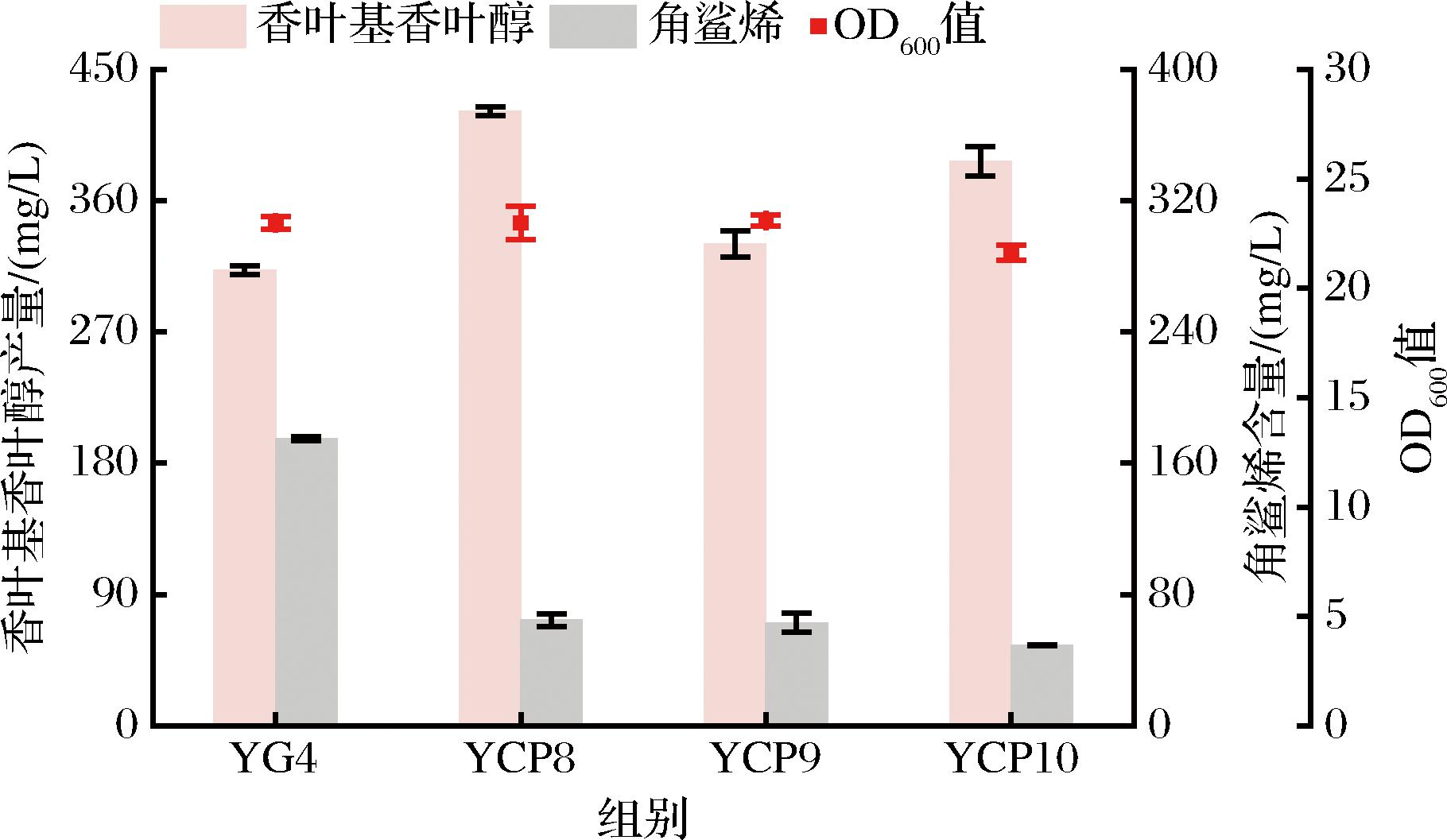
图5 下调角鲨烯合成途径对香叶基香叶醇产量的影响
Fig.5 Effect of downregulation of the squalene synthesis pathway on the production of geranylgeraniol
2.4 敲除全局转录调控因子
本研究选取了文献报道常见的影响MVA通路的基因和转录调控因子靶点YNR063W、YJL064W、DOS2、ROX1、MOT3、YPL062W,在菌株YCP8上分别单独敲除上述靶点构建了菌株YCP11-16,如图6所示,敲除YJL064W后香叶基香叶醇的产量达到576.9 mg/L,相较于YCP8分别提升了36.8%,接下来,在菌株YCP12上敲除了DOS2、YPL062W,构建了菌株YR1、YR2,结果显示,组合敲除YJL064W和YPL062W的菌株YR2的香叶基香叶醇产量达到678.2 mg/L,较YCP12提高了17.5%,较YCP8提高了60.9%,表明组合敲除YJL064W和YPL062W有利于香叶基香叶醇的生产。据报道,远端基因位点YPL062W和YJL064W缺失的酵母菌株质粒维持良好,细胞密度高且具有较高的甲羟戊酸含量[17],YPL062W突变体先前被证明具有较低的糖原水平[18],推测在这种突变体中,以糖原的形式封存的碳较少,更多的乙酰辅酶A可用于其他代谢途径,包括甲羟戊酸途径,从而增加萜类化合物的合成代谢通量。
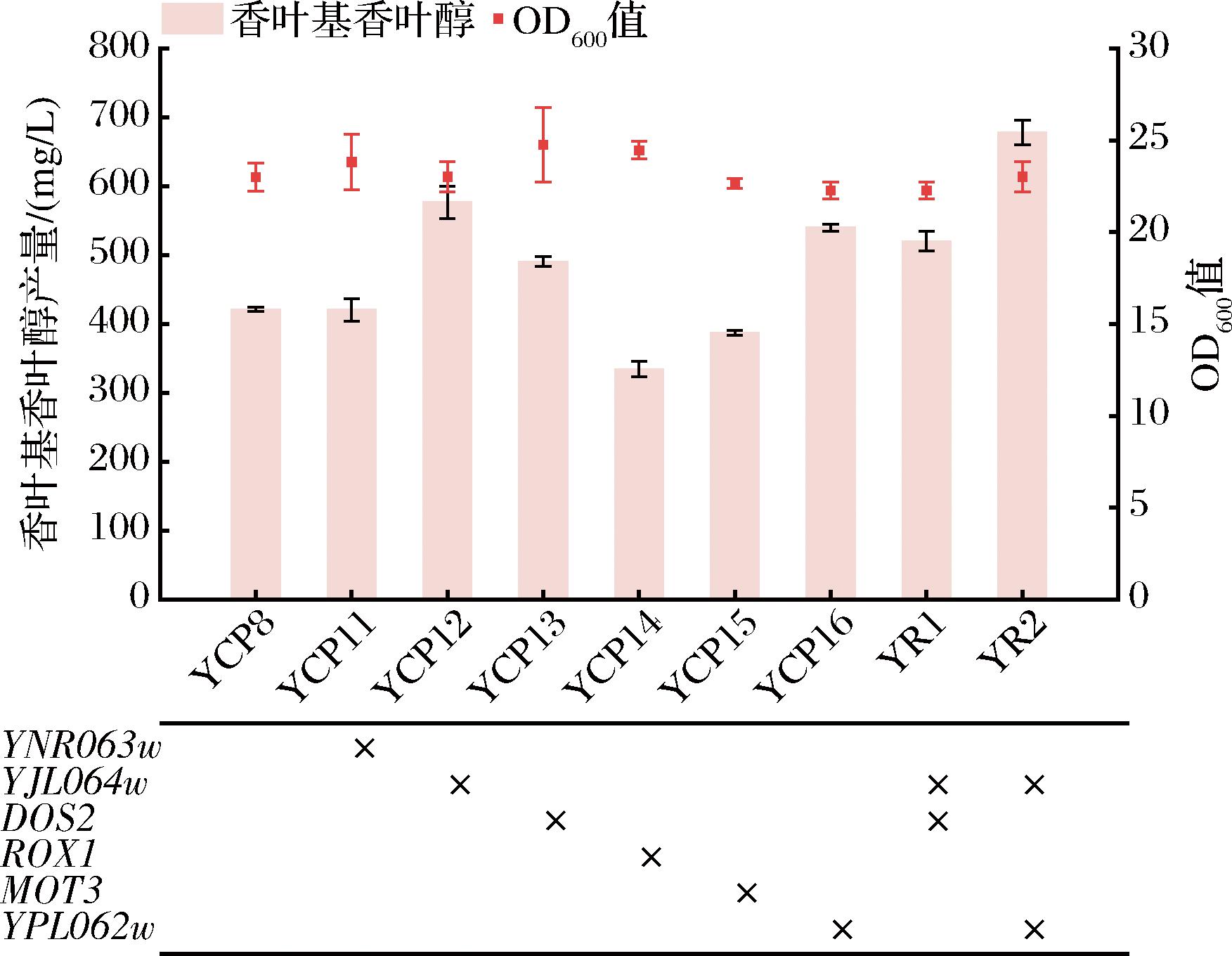
图6 敲除转录调控因子对香叶基香叶醇产量的影响
Fig.6 Effect of knockout of transcription regulatory factors on the production of geranylgeraniol
2.5 增加NADPH及乙酰辅酶A的供应
在香叶基香叶醇的合成过程中伴随着NADPH的消耗,上调磷酸戊糖途径是用来满足NADPH需求的有效策略[19]。因此,本文试图通过过表达参与该途径的几个基因来增强PPP通量,包括:ZWF1、GND1、TKL1和TAL1。构建了菌株YCP17-19,如图7所示,YCP19的香叶基香叶醇的产量达到791.1 mg/L,与YR2相比提高了16.7%,说明过表达PPP途径基因显著提高了NADPH的供应,这对于提高香叶基香叶醇的产量是有效的。本研究过表达了来自肠沙门氏菌(Salmonella enterica)的对乙酰辅酶A反馈不敏感的乙酰辅酶A合成酶基因突变体SeACSL641P来强化胞质中丙酮酸-乙酰辅酶A途径,为MVA途径提供更多的前体,进一步促进香叶基香叶醇的过量生产,构建的菌株YCP20的香叶基香叶醇产量进一步提高至856.9 mg/L。
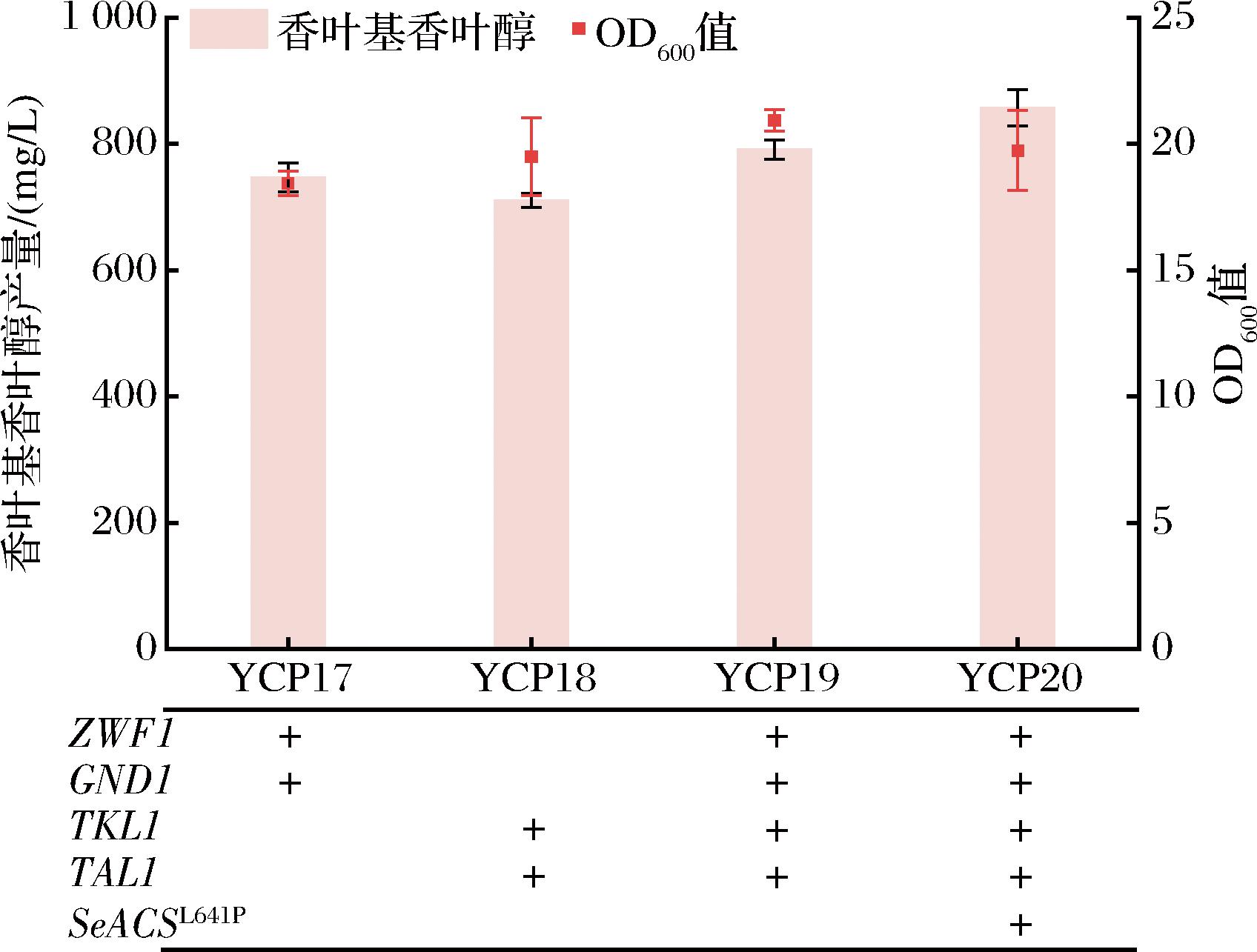
图7 增加NADPH及乙酰辅酶A的供应对香叶基香叶醇产量的影响
Fig.7 Effect of increasing the supply of NADPH and acetyl CoA on the production of geranylgeraniol
2.6 培增加融合基因的拷贝数
研究表明,增加萜类合酶基因额外拷贝的策略可增加前体的利用效率[15]。本研究在菌株YCP20上依次增加了2个拷贝的crtE03M-ERG20和一个拷贝的crtE03M-DPP1构建了菌株YCP21-23在YPD培养基中发酵144 h后,菌株YCP21-YCP23的香叶基香叶醇产量分别达到了1 035.8、1 086.4、1 116.5 mg/L,最终产量较YCP20提高了30.3%。
2.7 5 L发酵罐生产香叶基香叶醇
为了进一步提高香叶基香叶醇的产量,本研究在5 L反应器中进行分批补料发酵。将菌株YCP23接种到2 L优化培养基中,接种量为5%,补料培养基为600 g/L的葡萄糖溶液及100 g/L的酵母粉溶液,为细胞生长及产物积累提供足够的营养。发酵过程中菌株的OD600值变化及葡萄糖、乙醇、香叶基香叶醇的含量如图8所示,生物量在前48 h迅速增加,随后保持稳定,直到120 h达到最大值78.3,香叶基香叶醇的产量随着发酵时间持续累积,直到150 h达到最高11.3 g/L,这是迄今为止酿酒酵母生产香叶基香叶醇的最高产量。在150 h后产量出现下降趋势,可能是由于随着发酵时间的延长,发酵罐中的十二烷在高速搅拌的状态下产生乳化,对香叶基香叶醇的萃取效果下降。
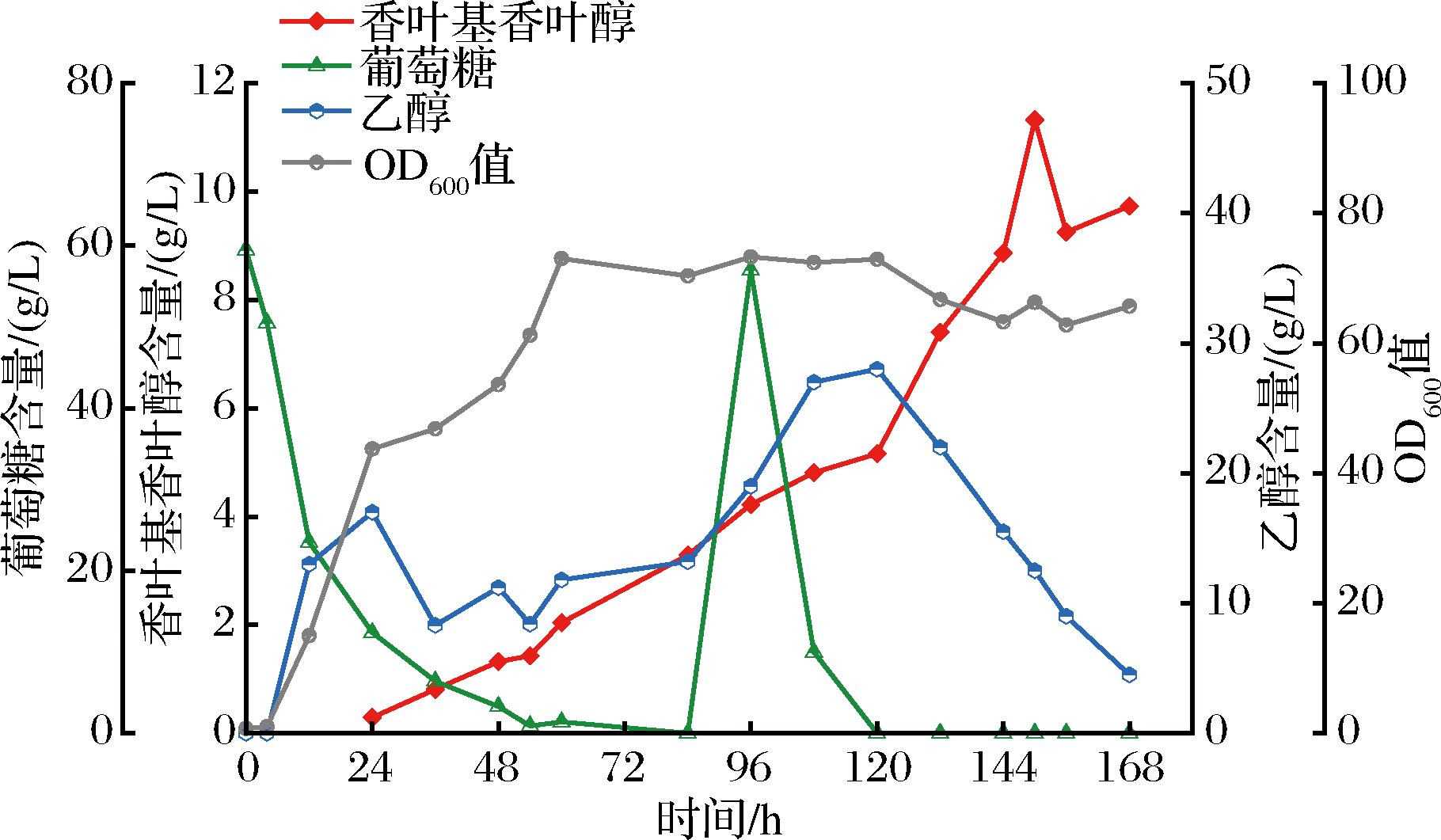
图8 工程菌株5 L发酵罐生产香叶基香叶醇
Fig.8 Production of geranylgeraniol by engineered strains in 5 L fermenters
3 讨论与结论
强化MVA通路基因表达可使香叶基香叶醇的产量逐渐升高,过表达2个tHMG1的拷贝对香叶基香叶醇的合成最有利,而过表达3个拷贝的tHMG1对菌株的生长造成了负担。通过CrtE03M-DPP1融合表达和下调角鲨烯合成途径可以更高效地减少FPP向角鲨烯合成路径的消耗,使FPP流量更多向GGPP及香叶基香叶醇的合成流入。组合敲除YJL064W和YPL062W有利于香叶基香叶醇的合成,香叶基香叶醇产量达到678.2 mg/L。同时,通过过表达磷酸戊糖途径基因和增加胞质乙酰辅酶A的供应,为香叶基香叶醇的合成提供更多的前体,使香叶基香叶醇的产量达到856.9 mg/L。增加融合基因的拷贝数也是提高香叶基香叶醇产量的有效途径,香叶基香叶醇的产量进一步提高至1 116.5 mg/L。最后在5 L发酵罐中进行分批补料发酵,香叶基香叶醇的产量在150 h达到最高11.3 g/L。这是迄今为止酿酒酵母生产香叶基香叶醇的最高产量。
在后续的研究中,可进一步通过响应面实验对各个因素进行系统的优化,探究各因素间的最佳组合;另外,还可以在已构建的营养缺陷型菌株的基础上回补氨基酸编码基因,构建原养型菌株,以实现细胞的高密度发酵,并降低培养的成本。本研究对酿酒酵母生产香叶基香叶醇的工业化应用具有一定的参考意义。
[1] BENFORD H L, FRITH J C, AVRIOLA S, et al.Farnesol and geranylgeraniol prevent activation of caspases by aminobisphosphonates:Biochemical evidence for two distinct pharmacological classes of bisphosphonate drugs[J].Molecular Pharmacology, 1999, 56(1):131-140.
[2] HYATT J A, KOTTAS G S, EFFLER, J.Development of synthetic routes to D,L-alpha-tocopherol (vitamin E) from biologically produced geranylgeraniol[J].Organic Process Research &Development 2002, 6(6):782-787.
[3] MU Y Q, GIBBS R A.Coupling of isoprenoid triflates with organoboron nucleophiles: Synthesis of all trans-geranylgeraniol[J].Tetrahedron Letters, 1995, 36(32):5669-5672.
[4] WAY J C, COLLINS J J, KEASLING J D, et al.Integrating biological redesign:Where synthetic biology came from and where it needs to go[J].Cell, 2014, 157(1):151-161.
[5] JUTURU V, WU J C.Microbial production of lactic acid:The latest development[J].Critical Reviews in Biotechnology, 2016, 36(6):967-977.
[6] MURAMATSU M, OHTO C, OBATA S, et al.Accumulation of prenyl alcohols by terpenoid biosynthesis inhibitors in various microorganisms[J].Applied Microbiology and Biotechnology, 2008, 80(4):589-595.
[7] BRÖKER J N, MÜLLER B, PRÜFER D, et al.Combinatorial metabolic engineering in Saccharomyces cerevisiae for the enhanced production of the FPP-derived sesquiterpene germacrene[J].Bioengineering (Basel), 2020, 7(4):135.
[8] HU T Y, ZHOU J W, TONG Y R, et al.Engineering chimeric diterpene synthases and isoprenoid biosynthetic pathways enables high-level production of miltiradiene in yeast[J].Metabolic Engineering, 2020, 60:87-96.
[9] WANG J H, ZHU L H, LI Y R, et al.Enhancing geranylgeraniol production by metabolic engineering and utilization of isoprenol as a substrate in Saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry, 2021, 69(15):4480-4489.
[10] WANG J H, LI Y R, JIANG W, et al.Engineering Saccharomyces cerevisiae YPH499 for overproduction of geranylgeraniol[J].Journal of Agricultural and Food Chemistry, 2023, 71(25):9804-9814.
[11] SONG T Q, DING M Z, ZHAI F, et al.Engineering Saccharomyces cerevisiae for geranylgeraniol overproduction by combinatorial design[J].Scientific Reports, 2017, 7(1):14991.
[12] PENG B Y, PLAN M R, CARPENTER A, et al.Coupling gene regulatory patterns to bioprocess conditions to optimize synthetic metabolic modules for improved sesquiterpene production in yeast[J].Biotechnology for Biofuels, 2017, 10:43.
[13] PARAMASIVAN K, MUTTURI S.Regeneration of NADPH coupled with HMG-CoA reductase activity increases squalene synthesis in Saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry, 2017, 65(37):8162-8170.
[14] TOKUHIRO K, MURAMATSU M, OHTO C, et al.Overproduction of geranylgeraniol by metabolically engineered Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2009, 75(17):5536-5543.
[15] XIE W P, LV X M, YE L D, et al.Construction of lycopene-overproducing Saccharomyces cerevisiae by combining directed evolution and metabolic engineering[J].Metabolic Engineering, 2015, 30:69-78.
[16] PARAMASIVAN K, MUTTURI S.Progress in terpene synthesis strategies through engineering of Saccharomyces cerevisiae[J].Critical Reviews in Biotechnology, 2017, 37(8):974-989.
[17] BLLGE Ö B, BURD H, LEE T S, et al.Carotenoid-based phenotypic screen of the yeast deletion collection reveals new genes with roles in isoprenoid production[J].Metabolic Engineering, 2013, 15:174-183.
[18] GIAEVER G, CHU A M, NI L, et al.Functional profiling of the Saccharomyces cerevisiae genome[J].Nature, 2002, 418(6896):387-391.
[19] KIM J E, JANG I S, SUNG B H, et al.Rerouting of NADPH synthetic pathways for increased protopanaxadiol production in Saccharomyces cerevisiae[J].Scientific Reports, 2018, 8(1):15820.