酸笋是以新鲜竹笋为原料,经盐水腌制发酵得到的一种发酵蔬菜制品。酸笋中的乳酸菌不仅具有益生菌功能,还能产生丰富的挥发性风味物质、有机酸等,赋予酸笋独特风味[1],深受人们青睐。在发酵蔬菜制作中,盐是不可缺少的材料,其不仅有助于蔬菜脱水和防腐,还能通过调节适当的浓度促进有益菌生长,提高感官品质和保持优良风味[2]。研究表明,在不同盐浓度下泡菜的细菌群落结构表现出显著差异,发酵过程中,变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)减少,厚壁菌门(Firmicutes)增加,成为主导菌门。属水平上,乳酸杆菌和乳球菌的丰度增加,高盐浓度下促进乳酸菌成为泡菜中的主要菌属[3]。MHEEN等[4]指出,泡菜腌制发酵过程中如果盐浓度过低,发酵过程进行过快会导致泡菜酸化和软化;如果盐浓度过高,则乳酸菌代谢受到抑制,泡菜发酵速度变慢,发酵周期延长。因此,探究不同盐浓度对酸笋微生物群落和代谢功能的影响,对于优化酸笋发酵工艺和提高产品品质具有重要意义。
宏基因组学技术可以直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究样品所包含的全部微生物的遗传组成及其群落功能。贾晶晶等[5]使用宏基因组学技术分析不同盐浓度下酸菜的微生物多样性,结果表明在样品中变形菌门和厚壁菌门均为优势菌门,肠杆菌属(Enterobacter)和乳球菌属(Lactococcus)为优势菌属,特别是在1.2 g/L盐质量浓度下,乳球菌属的比例(37%)显著高于0.8 g/L盐质量浓度时的1.4 g/L。还有学者通过宏基因组学技术分析了泡菜中微生物群落与风味物质的相关性[6]。但目前对于盐浓度如何影响酸笋中微生物群落及代谢功能的研究鲜有报道。因此,本研究利用宏基因组学技术对0.2、0.6、1.0 g/L盐浓度条件下酸笋中的微生物多样性、物种丰富度、功能基因和关键代谢途径进行分析,探究盐浓度对酸笋微生物群落结构及代谢功能的影响,旨在为发酵酸笋微生物群落和风味品质的控制提供理论参考。
1 材料与方法
1.1 材料与试剂
新鲜麻竹笋,重庆市北碚区天生桥农贸市场;食盐,中国盐业集团有限公司:0.1 g/L琼脂糖凝胶、磷酸缓冲盐溶液,武汉普诺赛生命科技有限公司;NEXTFLEX® Rapid DNA-Seq Prep Kit for Illumina,美国德克萨斯州奥斯汀Bio Scientific公司;NovaSeq 6000 S4 v1.5试剂盒,因美纳(中国)科学器材有限公司。
1.2 仪器与设备
TG20G台式高速离心机,江苏新春兰科学仪器有限公司;NanoDrop2000超微量分光光度计,美国赛默飞生物科技有限公司;Covaris低通量聚焦超声器,M220香港基因有限公司;Hiseq2500 PE250测序仪,中国诺禾致源生物信息科技有限公司。
1.3 实验方法
1.3.1 酸笋制备
将新鲜麻竹笋的外壳剥去,清洗干净,切成片状,然后在沸水中煮沸15 min。待竹笋片冷却后,置于5 L的发酵罐中,然后分别注入质量浓度为0.2、0.6、1.0 g/L的盐水,原料与盐水质量比为2∶3,加旋盖密封后于25 ℃下进行发酵45 d,分别取出3份50 mL的发酵液进行下一步分析。0.2、0.6、1.0 g/L盐质量浓度发酵酸笋分别命名为S2、S6、S10组。
1.3.2 DNA提取和Illumina测序
将样本送至上海美吉生物医药科技有限公司以进行DNA的提取及Illumina测序,将其置于50 mL大离心管中,进行13 000 r/min高速离心15 min,弃上清液,用磷酸缓冲盐溶液洗涤大离心管中沉淀物,将菌体收集在1.5 mL的离心管中,用与总DNA提取,并对提取的DNA进行浓度及纯度测量。选择待测区域,将DNA打断成300 bp的片段,构建双端文库并将含有完整互补测序引物杂交位点的衔接子连接到片段的钝端,随后进行双端测序,将相关的序列数据上传至NCBI(National Center of Biotechnology Information)的中短读存档数据库中保存,使用Flash软件对测序得到的序列进行拼接,得到高质量的Tags数据,这些数据随后用于进一步的基因预测与注释分析。
1.3.3 微生物群落分析
用引物338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)针对样本的16S rDNA基因V4区进行PCR扩增,然后在72 ℃进行10 min的延伸,PCR扩增产物使用0.1 g/L琼脂糖进行凝胶电泳检查扩增效果,并将产物在10 ℃保存备用,对每个样本进行3次复制实验。然后将样品的PCR产物,在Illumina-Hi Seq平台上进行高通量测序。最后,基于RDP分类器和Silva 16S rDNA数据库对获得的微生物序列进行分类和注释。
1.3.4 基因预测和功能注释
对测序得到的原始数据进行过滤处理,去除低质量数据、污染序列等,得到有效数据,将数据拼接组装并统计组装结果。对重叠群进行基因预测并去冗余,构建基因目录,使用BLASTP与NR数据库进行比对,物种注释方法为Best-hit,获得物种注释信息;此外,通过Hmmscan工具对水化合物活性酶数据库(Carbohydrate-Active Enzymes Database,CAZy)进行碳水化合物活性酶的注释,E值截断为10-5。使用Diamond软件,在直系同源蛋白分组比对数据库(Evolutionary genealogy of genes:Non-supervised Orthologous Groups,eggNOG)中进行查询,以对序列进行直系同源基因蛋白质组(Clusters of Orthologous Groups,COG)注释簇,设置E值截断为10-5。同样使用Diamond对KEGG数据库进行功能注释,E值截断也为10-5。最后,将这些基因与eggNOG、KEGG、CAZy等数据库进行比对,从而获得关于基因的物种和功能注释信息。
1.4 数据处理
采用Perl软件绘制酸笋微生物群落的Circos图,使用Origin 2021软件绘制酸笋微生物群落的Alpha多样性指数图和物种丰度图,对COG、KEGG、CAZy数据库注释结果作图。
2 结果与分析
2.1 不同盐浓度微生物多样性分析
2.1.1 不同盐浓度的宏基因测序结果统计
通过S2、S6、S10酸笋发酵液宏基因组测序获得的数据如表1所示,平均读取长度为568.13 bp,有效读取量达到42.64 M,占原始读取总量的93.73%。此外,有效碱基总量为6.38 G,占原始碱基总量的93.00%。累计而言,宏基因组测序共产生了19.14 G的有效数据量,平均每个样本约为6.38 G,与16 S测序的大约200 bp的读长相比,有效数据量为10~70 M[7],体现出本次测序具有较高的文库覆盖度,有效捕获了样品中的微生物多样性。
表1 不同盐浓度酸笋中微生物宏基因组测序结果
Table 1 Microbial metagenomic sequencing results of Suansun with different salt concentrations
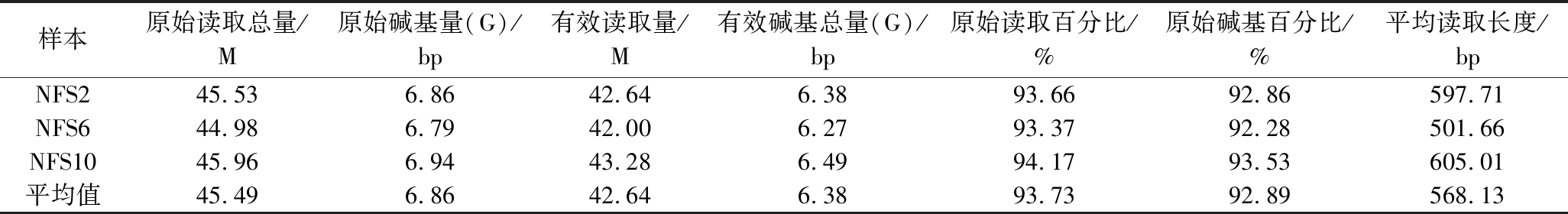
样本原始读取总量/M原始碱基量(G)/bp有效读取量/M有效碱基总量(G)/bp原始读取百分比/%原始碱基百分比/%平均读取长度/bpNFS245.536.8642.646.3893.6692.86597.71NFS644.986.7942.006.2793.3792.28501.66NFS1045.966.9443.286.4994.1793.53605.01平均值45.496.8642.646.3893.7392.89568.13
2.1.2 不同盐浓度微生物群落Alpha多样性分析
Alpha多样性作为评估生态系统内微生物群落丰富度与均匀度的综合指数[8],通过物种观测丰富度、Shannon多样性指数及物种均匀度指数等可以揭示盐浓度对微生物群落结构的影响。如图1所示,所有样品的覆盖度指数达到100%,表明本实验的取样策略能够全面反映各样品微生物群落的组成[9]。在S2、S6样品中,微生物物种观测丰富度分别达到24种和27种,明显高于S10样品观察到的18种。然而,物种均匀度指数在S10样品最高,说明S10样品物种分布更均匀。这表明在1.0 g/L盐质量浓度下促使微生物群落结构更优化,实现了较少物种的均衡共存。Shannon多样性指数反映样品微生物的丰富程度[10],S2和S6的Shannon指数>S10,说明0.2和0.6 g/L盐质量浓度环境下微生物群落的多样性相对较高,而1.0 g/L盐质量浓度条件下生物多样性总体降低。
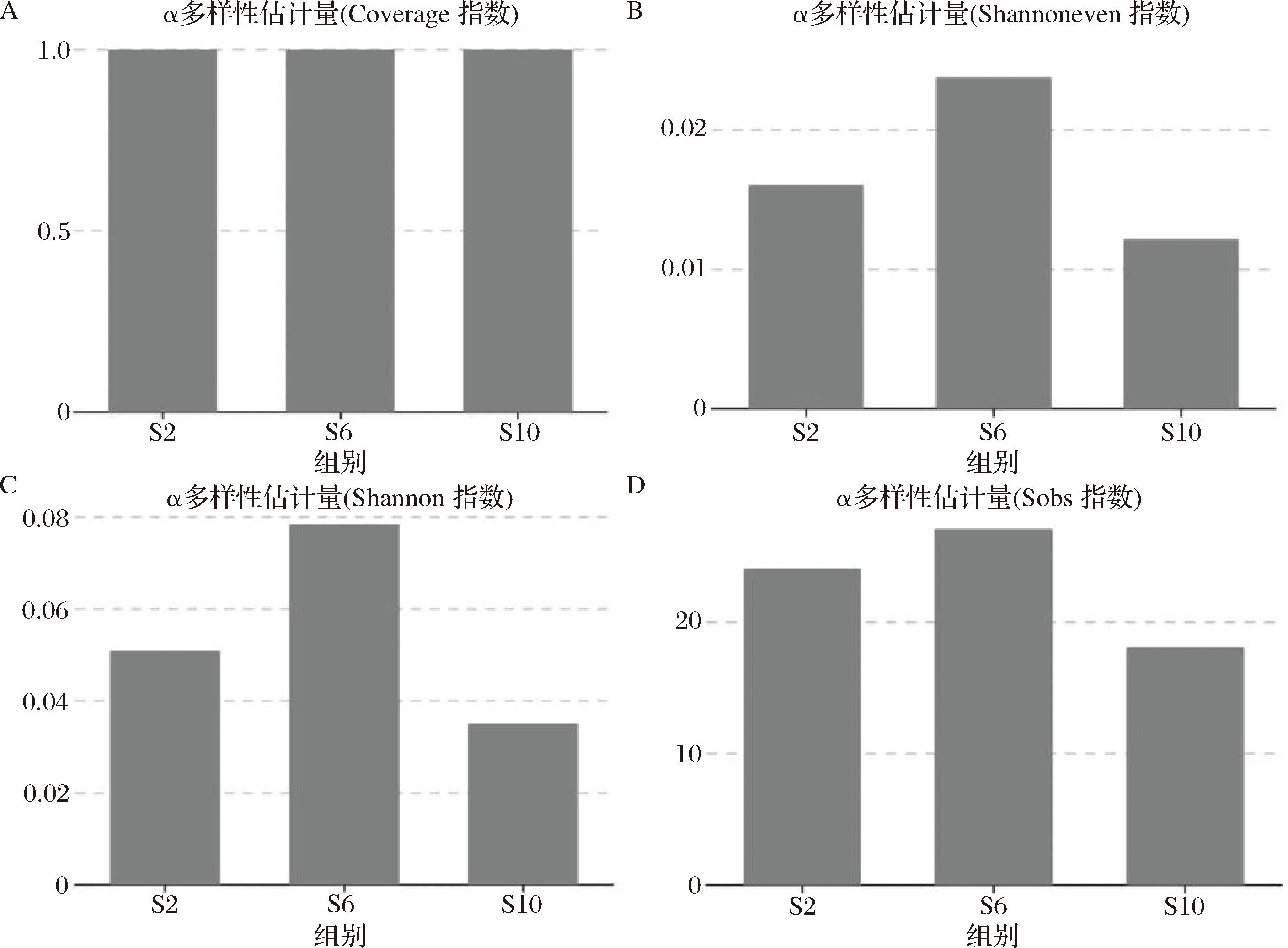
A-Shannon指数统计柱状图;B-Shannoneven指数统计柱状图;C-Coverage统计柱状图;D-Sobs指数统计柱状图
图1 不同盐浓度酸笋的α多样性指数
Fig.1 Alpha diversity index of Suansun with different salt concentrations
2.1.3 不同盐浓度的微生物群落结构分析
本文通过宏基因组学方法,对S2、S6、S10样品的微生物群落结构进行了详细分析;通过分析物种丰度表和注释信息,计算了不同盐浓度酸笋中门、属、种级别排名前10的微生物相对丰度,以比较微生物群落组成的差异。如图2所示,在门水平上,厚壁菌门在S2、S6、S10样品中占绝对优势,相对比例分别为99.15%、99.26%和98.66%,这一结果与钟源[11]的研究结果相似。此外,在不同盐浓度条件下,子囊菌门(Ascomycota)的比例表现出明显差异,S2和S6样品中占比分别为0.27%和0.54%,有研究发现,在含盐环境下子囊菌门的丰富度比非含盐环境高出约1.3倍[12]。然而,S10样品中其比例下降至不到0.1%。这一发现揭示了在0.6 g/L盐质量浓度环境下子囊菌门显示出的独特适应性。
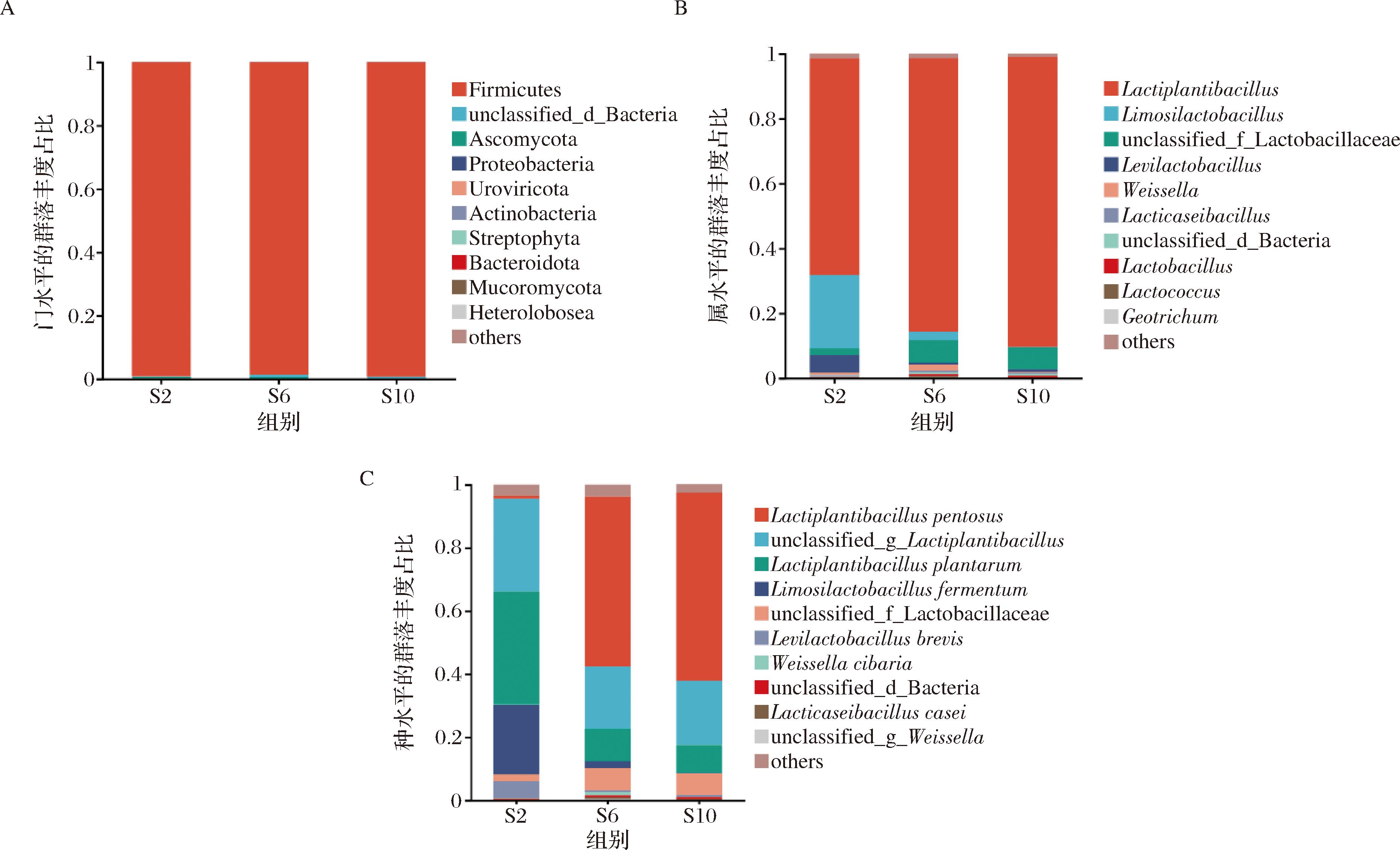
A-门水平上微生物群落丰度图;B-属水平上微生物群落丰度图;C-种水平上微生物群落丰度图
图2 不同盐浓度酸笋的微生物群落结构
Fig.2 Microbial community structure of Suansun with different salt concentrations
在属水平上,不同盐浓度酸笋样本中包含了植物乳杆菌属(Lactiplantibacillus)、乳杆菌属(Levilactobacillus)、魏斯氏菌属(Weissella)、罗伊氏杆菌属(Limosilactobacillus)、乳球菌属等。植物乳杆菌属(Lactiplantibacillus)在S2、S6、S10样品均为优势菌属,其相对丰度分别高达66.71%、84.23%和89.20%,植物乳杆菌(Lactobacillus plantarum)可增加发酵蔬菜中的乳酸、酒石酸、柠檬酸及谷氨酸、甘氨酸和苏氨酸的浓度[13],不仅赋予酸笋更加鲜明的酸感,同时其产生的酸性环境也有效地抑制了潜在有害微生物的生长。此外,S2样品中罗伊氏杆菌属、乳杆菌属、乳球菌属和魏斯氏菌属展现出较S6和S10样品中更高的相对丰度,结果表明乳球菌属和魏斯氏菌属是低盐发酵食品的优势菌属[14],其中乳杆菌属、魏斯氏菌属等是具有益生功能的微生物[15]。酸笋中微生物群落的组成与泡菜、酸菜等传统发酵食品中常见的主要微生物相似[16-17];特别是乳杆菌属,已被证实在发酵乳制品中对酸化过程和风味形成有着显著的贡献,其在酸笋的风味形成中也扮演着重要角色[18]。
在种水平上,S2样品中植物乳杆菌(Lactobacillus plantarum)、发酵乳酸杆菌(Limosilactobacillus fermentum)和短乳杆菌(Lactobacillus brevis)显示出较高的相对丰度,其相对丰度分别为35.86%、22.07%、5.28%,表明这3种菌在酸笋低盐发酵的过程中具有重要作用[19]。随着盐浓度增加,发酵乳酸杆菌和短乳杆菌的相对丰度在酸笋发酵中明显下降,分别从22.07%和5.21%降低到8.86%和0.61%。这一变化可能是由于1.0 g/L盐质量浓度条件下的氧化应激会破坏细胞内的代谢平衡,从而影响乳酸菌的生长和繁殖能力[20];戊糖乳杆菌(Lactiplantibacillus pentosus)在S10样品中成为优势菌种,相对丰度达到59.52%,但其丰度随盐浓度减少而下降,在S2样品中仅为0.94%,这一现象归因于戊糖乳杆菌所展现的亚硝酸盐的降解能力[13],使得戊糖乳杆菌在1.0 g/L盐条件下能够更好地生存。
2.2 不同盐浓度发酵过程宏基因组学分析
2.2.1 不同盐浓度的COG注释分析
S2、S6和S10样本中COG功能单元的变化如图3所示,随着盐浓度增加,微生物在代谢活动上进行了显著的调整[21],特别是碳水化合物转运、细胞壁/膜/包膜的生物合成以及氨基酸的转运与代谢等关键途径;并且在不同盐浓度下酸笋的微生物主要活动与碳水化合物和氨基酸代谢相关,这是因为细胞代谢主要以碳源和氮源为原料,其消耗促进了微生物对营养物质的吸收及相关代谢活动的调节[22]。

A-S2样品;B-S6样品;C-S10样品
图3 不同盐浓度酸笋的COG注释分类统计柱状图
Fig.3 COG annotation classification statistical bar chart of Suansun with different salt concentrations
注:A:RNA加工修饰;B:染色质结构和动力学;C:能量生成和转换;D:细胞周期控制、细胞分裂和染色体分裂;E:氨基酸转运代谢;F:核苷酸转运和代谢;G:碳水化合物转运代谢;H:辅酶转运和代谢;I:脂肪转运代谢;J:翻译,核糖体结构和生物合成;K:转录;L:复制,重组和修复;M:细胞壁/膜/被膜的生物合成;N:细胞运动;O:翻译后修饰,蛋白质折叠和伴侣蛋白;P:无机离子转运代谢;Q:次级代谢物生物合成,转运和代谢;R:主要功能预测;S:未知功能;T:信号转导机制;U:胞内转运、分泌和小泡运输;V:抵御机制;W:胞外结构;X:移动基因组:噬菌体原,转座子;Y:核酸结构Z:细胞骨架。
具体而言,S2样品的微生物群落展现出广泛的代谢活性,尤其在碳水化合物转运、氨基酸转运和代谢、细胞壁/膜/包膜的生物合成等代谢途径中表现突出,并且基因数量均在1 500个以上。在S6样品中,碳水化合物转运代谢的基因数量增至2 090个,细胞壁/膜/包膜生物合成基因数量增加至1 740个,以及其他代谢途径如氨基酸转运和代谢、主要功能预测、翻译及核糖体结构和生物合成的活性提升,表明0.6 g/L盐质量浓度下微生物代谢活性和营养物质转化能力得到进一步优化,显著提升了发酵酸笋风味特性。而S10样品只有碳水化合物转运代谢的基因数量保持在1 500个以上,其基因数量为1 601个,其他代谢途径活性大幅下降。综上,酸笋样品在0.6 g/L盐质量浓度条件下微生物代谢途径的多样性最高,而1.0 g/L盐质量浓度条件下显著抑制了微生物的部分代谢途径。
2.2.2 不同盐浓度的KEGG注释分析
利用KEGG数据库可以对不同盐浓度酸笋中微生物进行功能基因和代谢机制分析。如图4所示,KEGG富集分析揭示了在1级KEGG代谢通路中,新陈代谢是不同盐浓度最主要的富集途径,表明酸笋在发酵过程中微生物代谢活动旺盛。在2级KEGG代谢通路中,对于S2样品,全局与概述图谱、碳水化合物代谢、膜运输、能量代谢、脂质代谢、氨基酸代谢、甘氨酸的生物合成与代谢以及核苷酸代谢途径的基因数量均超过550 000个;而在S6和S10样品中,仅全局与概述图谱、碳水化合物代谢、氨基酸代谢的基因数量超过550 000个,表明S2样品具有更广的代谢活动范围。在3组样品中,碳水化合物和氨基酸代谢是共同的代谢路径。在3级KEGG代谢通路中,碳水化合物代谢通路占主导地位,S2、S6和S10样品所注释到的基因数分别为640、677和472个,S6样品中碳水化合物代谢途径相关基因的数量表现出最高的丰富度。在0.6 g/L盐质量浓度条件下,微生物代谢活性达到了最佳状态。排名第2的氨基酸代谢通路中,在S6样品中相关基因的数量最多为746个,并且在酸笋中主要贡献风味形成的氨基酸包括谷氨酸(Glu)、天冬氨酸(Asp)、丙氨酸(Ala)和缬氨酸(Val)[23],是酸笋风味的重要来源。结果再次揭示0.6 g/L盐浓度条件下微生物代谢活性和风味特性达到最优状态。

A-S2样品;B-S6样品;C-S10样品
图4 不同盐浓度酸笋的KEGG注释分类统计柱状图
Fig.4 KEGG annotation classification statistical bar chart of Suansun with different salt concentrations
注:global and overview maps:全局与概述图谱;carbohydrate metabolism:碳水化合物代谢;amino acid metabolism:氨基酸代谢;metabolism of cofactors and vitamins:辅助因子和维生素的代谢;energy metabolism:能量代谢;nucleotide metabolism:核苷酸代谢;glycan biosynthesis and metabolism:多聚糖的生物合成和代谢;lipid metabolism:类脂(化合)物代谢作用;metabolism of other amino acids:其他氨基酸的代谢;biosynthesis of other secondary metabolites:其他次生代谢物的生物合成;xenobiotics biodegradation and metabolism:异种生物体的生物降解和代谢;metabolism of terpenoids and polyketides:萜类和聚酮类物质的代谢;membrane transport:膜转运;signal transduction:信号转导;signaling molecules and interaction:信号分子和相互作用;replication and repair:复制和修复;translation:翻译;folding, sorting and degradation:折叠、整理和降解;transcription:转录;information processing in viruses:病毒中的信息处理;drug resistance:antimicrobial:耐药性:抗菌;cancer: overview:癌症:概述;cardiovascular disease:心血管病;infectious disease:bacterial:传染病:细菌;endocrine and metabolic disease:内分泌和代谢性疾病;neurodegenerative disease:神经变性疾病;infectious disease:parasitic:传染病:寄生虫;drug resistance:antineoplastic:耐药性:抗肿瘤药物;infectious disease:viral:传染性疾病:病毒性;immune disease:免疫性疾病;cancer:specific types:癌症:特定类型;substance dependence:药物依赖;cellular community-prokaryotes:细胞群落原核生物;cell growth and death:细胞生长和死亡;transport and catabolism:运输和分解代谢;cell motility:细胞运动性;cellular community-eukaryotes:细胞群落-真核生物;endocrine system:内分泌系统;aging:衰老;environmental adaptation:环境适应;digestive system:消化系统;nervous system:神经系统;immune system:免疫系统;circulatory system:循环系统;excretory system:排泄系统;development and regeneration:发展和再生;sensory system:感觉系统。
2.2.3 不同盐浓度的CAZy分析
对不同盐浓度的碳水化合物活性酶的注释如图5-A~图5-C所示,其中主要包括糖基转移酶(glycosyl transferases,GT)、糖苷水解酶(glycoside hydrolases,GH)、多糖裂合酶(polysaccharide lyases,PL)。在不同盐浓度条件下,GT和GH作为碳水化合物代谢主导酶类均展现出高度的相对稳定性,S2样品中分别为411和427个,S6样品中增至465和477个,而S10样品中减至321和372个。其中GT通过转移糖基到受体分子上进行糖基化修饰,对维护代谢过程平衡、稳定和保护细胞具有重要作用[24],GH作用于果聚糖苷键的水解或合成,对果聚糖生物合成和细胞壁代谢等生物过程至关重要[25];结果表明在0.6 g/L盐质量浓度下,GT和GH活力最高,即碳水化合物代谢更为活跃。其中PL在不同盐浓度中相对较低,PL作用于多糖的降解酶,通过β-消去机制作用于1→4糖苷键,进而生成不饱和的寡聚糖[26],由此可推测出,在酸笋代谢中低聚糖不是发酵过程中的微生物营养的主要来源。由图5-D可知,短促生乳杆菌(Levilactobacillus brevis)、发酵黏液乳杆菌(Limosilactobacillus fermentum)为S2、S6、S10中共性较大的微生物群落,说明在S2、S6、S10样品中,含量最高的GH和GT的产生与这些微生物相关性显著。总体而言,酸笋样品在不同盐浓度中主要以碳水化合物转运和水解为主,而在多糖裂解代谢活动中其相对活跃程度较低。
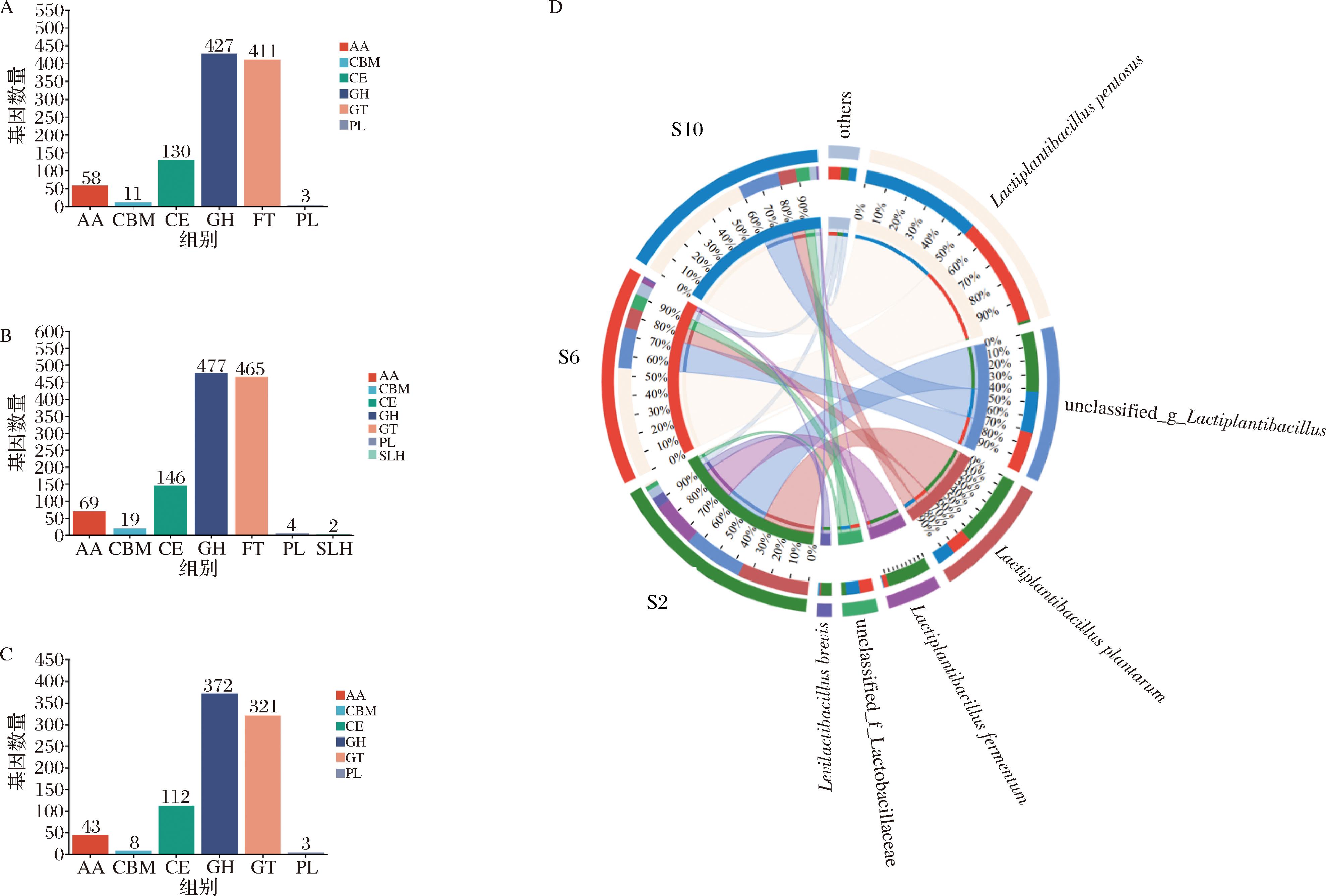
A-S2的Cazy注释分类统计柱状图;B-S6的Cazy注释分类统计柱状图;C-S10的Cazy注释分类统计柱状图;D-S2、S6、S10的Circos图
图5 不同盐浓度酸笋的Cazy分析
Fig.5 Cazy analysis of Suansun with different salt concentrations
注:AA: auxiliary activities,辅助活性;CBM: carbohydrate-binding modules,碳水化合物结合结构域;CE: carbohydrate esterases,碳水化合物酯酶;SLH: cellulosome modules,纤维素小体模块。
2.3 不同盐浓度发酵过程中关键代谢途径分析
2.3.1 碳水化合物代谢相关功能基因 KEGG 注释结果
在不同盐浓度发酵酸笋的碳水化合物代谢中一共注释到15条相关代谢通路,在不同盐浓度下排名前5的主要代谢有:淀粉和蔗糖代谢(ko00500)、氨基糖和核苷酸糖代谢(ko00520)、果糖和甘露糖代谢(ko00051)、糖酵解/糖异生(ko00010)、丙酮酸代谢(ko00620),在不同盐浓度下淀粉和蔗糖代谢最为活跃,糖酵解/糖异生代谢、丙酮酸代谢为次要的代谢活动。在酸笋发酵的碳水化合物代谢过程中(图6),淀粉及蔗糖的转化作用主要通过糖酵解路径进行,目的在于将原材料中的淀粉与蔗糖降解为葡萄糖,从而为微生物代谢活动供能;S2样品有343个淀粉和蔗糖代谢相关基因,S6样品增至405个,而S10样品减至323个,研究结果显示在0.6 g/L盐质量浓度条件下,微生物利用淀粉和蔗糖的效率更高,从而提供了更多转化为风味物质的原料。在糖酵解途径中,乳酸的产生过程起始于1-磷酸葡萄糖,通过葡萄糖磷酸变位酶(EC:5.4.2.2)和葡萄糖-6-磷酸异构酶(EC:5.3.1.9)等一系列酶的作用,转化为6-磷酸果糖,经过多步酶促反应最终形成丙酮酸。丙酮酸代谢是主要风味物质:乳酸、乙醛、乙酸生成的关键步骤,丙酮酸由丙酮酸脱氢酶(EC:1.2.5.1)催化生成乳酸,或者通过丙酮酸脱羧酶(EC:4.1.1.1)、丙酮酸脱氢酶E1组分(EC:1.2.4.1)催化生成乙醛,同时通过醋酸盐-CoA连接酶(EC:6.2.1.13)等4种酶催化生成醋酸。这一复杂的酶促反应凸显了糖酵解途径在能量产生及风味物质生成中的重要作用,同时微生物可以通过乳酸、乙酸的产生调节环境酸度。其中S2样品中参与的丙酮酸代谢基因数量为310个,S6样品中增至344个,而S10样品中减少到239个,说明在0.6 g/L盐质量浓度条件下酸笋酸味特性更加明显,而1.0 g/L盐质量浓度条件下显著抑制其酸味特性。

图6 不同盐浓度酸笋中碳水化合物代谢途径
Fig.6 Metabolic pathways of carbohydrate in acid shoots with different salt concentrations
2.3.2 氨基酸代谢相关功能基因 KEGG 注释结果
使用KEGG数据库注释酸笋发酵中的氨基酸代谢路径,发现共有14条氨基酸代谢途径,在不同盐浓度下关键路径相似,并且随着盐浓度的增加,基因参与代谢途径的数量先增加后减少,其中参与基因数量排名前三的氨基酸代谢路径分别为:半胱氨酸和蛋氨酸代谢(ko00270)、丙氨酸、天冬氨酸和谷氨酸代谢(ko00250)、甘氨酸、丝氨酸和苏氨酸代谢(ko00260),其中丙酮酸代谢起连接代谢之间桥梁的作用(图7-A)。在酸笋发酵过程中,丙氨酸、天冬氨酸和谷氨酸代谢途径为L-天冬氨酸在天冬氨酸4-脱羧酶(EC:4.1.1.12)催化下生成L-丙氨酸,再由丙酮酸转氨酶(EC:2.6.1.44)转化为丙酮酸,之后进入丙酮酸代谢通路通过乳醛脱氢酶(EC:1.2.1.22)等3种酶的催化下生成甲基乙二醛,其中甲基乙二醛作为香气和有色化合物的前体物质在Strecker降解中发挥着重要的作用[27],为酸笋提供特殊的风味。在甘氨酸、丝氨酸和苏氨酸代谢中,甲基乙二醛是由高丝氨酸通过高丝氨酸激酶(EC:2.7.1.39)等经一系列的酶促反应生成,同时高丝氨酸也可在高丝氨酸脱氢酶(EC:1.1.1.3)的催化作用下进入半胱氨酸和蛋氨酸代谢中,由L-天冬氨酸通过天冬氨酸激酶(EC:2.7.2.4)等3种酶经催化反应生成,3种代谢途径形成一个循环的网络体系,丙酮酸代谢在其代谢网络体系中发挥着重要的枢纽作用。不同盐浓度条件下微生物代谢途径的基因表达数量结果显示,0.6 g/L盐质量浓度条件下呈现出最佳状态,S10样品表达的基因数量最少。其中,半胱氨酸与蛋氨酸代谢途径参与的基因数量从S2中的233个增加至S6样品中的281个,而S10样品中降至181个,这一趋势在甘氨酸、丝氨酸与苏氨酸以及丙氨酸、天冬氨酸与谷氨酸的代谢途径中同样显著,表明0.6 g/L盐质量浓度下微生物代谢能力呈现出最优状态,更有利于酸笋风味物质的形成。

A-不同盐浓度酸笋的半胱氨酸和蛋氨酸代谢、丙氨酸、天冬氨酸和谷氨酸代谢、甘氨酸、丝氨酸和苏氨酸代谢、丙酮酸代谢途径;B-不同盐浓度酸笋的色氨酸代谢途径
图7 不同盐浓度酸笋的关键氨基酸代谢途径分析
Fig.7 Analysis of key amino acid metabolism pathways of Suansun with different salt concentrations
在色氨酸代谢通路中(图7-B),色氨酸通过L-色氨酸脱羧酶(EC:4.1.1.105)等7种酶催化生成吲哚-3-乙醇,可能是酸笋酸臭味的主要来源[28]。同时在生成吲哚-3-乙醇时,吲哚-3-乙醛肟作为中间物质也可以通过吲哚乙醛肟脱水酶(EC:4.8.1.3)等3种酶催化生成粪臭素,可能也是酸笋臭味的重要来源。S2样品中参与色氨酸代谢的基因数为54个,S6样品中增至68个,而S10样品中降低至40个,结果表明在0.6 g/L盐质量浓度条件下,微生物群落展现的最佳代谢活性,更有利于提升发酵酸笋的风味品质和酸味特性;而在1.0 g/L盐质量浓度条件下更有利于抑制酸笋产生的酸臭味。
3 结论
本文通过宏基因组学分析了不同盐浓度下酸笋发酵的微生物多样性和关键的碳水化合物与氨基酸代谢路径。门水平上,厚壁菌门是不同盐浓度发酵过程中共有的优势菌门,其占比均超过98%以上;属水平上,植物乳杆菌属为不同盐浓度下的优势菌属,相比于S6和S10样品,S2样品中乳球菌属和魏斯氏菌属的相对丰度更为显著;种水平上,S2样品中L.pentosus的相对丰度远小于S6和S10样品。本文通过对KEGG数据库分析,发现碳水化合物代谢及氨基酸代谢是酸笋的主要代谢;酸笋代谢过程中,随着盐浓度的增加,基因数量先增加后减少;在S6样品中,参与各代谢途径的基因数量最多,表明微生物群落在此条件下展现了最佳的代谢活性,尤其是丙酮酸代谢显著提升了酸笋的酸味,使其在0.6 g/L盐质量浓度条件下酸味特性更为突出。然而在1.0 g/L盐质量浓度条件下通过抑制色胺酸代谢,从而能更好地降低酸笋的酸臭味。以上结果可以为不同盐浓度条件下酸笋微生物群落结构的调控及酸笋风味品质的提升提供理论基础。
[1] 张雅雯. 螺蛳粉酸笋中微生物菌群及风味物质的分析[D].武汉:华中农业大学,2022.ZHANG Y W.Analysis of microbial flora and flavor substances in sour bamboo shoots of Liuzhou river snails rice noodle[D].Wuhan:Huazhong Agricultural University, 2022.
[2] 李凤姿, 张媛, 吴昊, 等.盐对发酵蔬菜微生物群体和发酵质量的影响[J].中国调味品, 2018, 43(3):150-154;200.LI F Z, ZHANG Y, WU H, et al.Effect of salt on microbial population and fermentation quality of fermented vegetables[J].China Condiment, 2018, 43(3):150-154;200.
[3] WANG X Y, SONG G, HE Z, et al.Effects of salt concentration on the quality of Paocai, a fermented vegetable product from China[J].Journal of the Science of Food and Agriculture, 2021, 101(15):6202-6210.
[4] MHEEN T I, KWON T W.Effect of temperature and salt concentration on Kimchi fermentation[J].Korean Journal of Food Science and Technology, 1984, 16(4):443-450.
[5] 贾晶晶, 魏爱丽, 赵虎威, 等.基于高通量测序分析不同食盐浓度的酸菜中微生物多样性[J].中国调味品, 2022, 47(8):167-170;174.JIA J J, WEI A L, ZHAO H W, et al.Analysis of microbial diversity of Chinese sauerkraut with different salt concentrations by high-throughput sequencing[J].China Condiment, 2022, 47(8):167-170;174.
[6] MI T, WANG D K, YAO S J, et al.Effects of salt concentration on the quality and microbial diversity of spontaneously fermented radish Paocai[J].Food Research International, 2022, 160:111622.
[7] 李欣蔚, 丛敏, 武俊瑞, 等.基于16S rRNA基因V3-V4区高通量测序分析东北自然发酵酸菜中细菌群落结构[J].现代食品科技, 2017, 33(2):69-75;82.LI X W, CONG M, WU J R, et al.Analysis of the bacterial community structure in naturally fermented cabbage of Northeast China by high-throughput sequencing[J].Modern Food Science and Technology, 2017, 33(2):69-75;82.
[8] 陈梅春, 朱育菁, 刘波, 等.基于宏基因组茉莉花植株土壤细菌多样性研究[J].农业生物技术学报, 2018, 26(9):1480-1493.CHEN M C, ZHU Y J, LIU B, et al.Diversity research of the soil bacteria of Jasminum sambac Ait based on metagenome[J].Journal of Agricultural Biotechnology, 2018, 26(9):1480-1493.
[9] 陈晓东. 酸笋的宏基因组及其功能基因[D].北京:中国林业科学研究院, 2019.CHEN X D.Metagenomic and its functional genes of fermented bamboo shoot[D].Beijing:Chinese Academy of Forestry, 2019.
[10] XIA F, CHEN X, GUO M Y, et al.High-throughput sequencing-based analysis of endogenetic fungal communities inhabiting the Chinese Cordyceps reveals unexpectedly high fungal diversity[J].Scientific Reports, 2016, 6:33437.
[11] 钟源. 酸笋菌群多样性分析及乳酸菌的筛选、特性研究与应用[D].武汉:华中农业大学, 2022.ZHONG Y.Analysis of the diversity of bacterial flora in sour bamboo shoots and the screening, characterization and application of lactic acid bacteria[D].Wuhan:Huazhong Agricultural University, 2022.
[12] CHEN H H, MA K Y, HUANG Y, et al.Significant response of microbial community to increased salinity across wetland ecosystems[J].Geoderma, 2022, 415:115778.
[13] SONG G, HE Z, WANG X Y, et al.Improving the quality of Suancai by inoculating with Lactobacillus plantarum and Pediococcus pentosaceus[J].Food Research International, 2021, 148:110581.
[14] 周金沙, 陈晓艺, 谭金萍, 等.基于Illumina MiSeq高通量测序技术分析广西无盐发酵酸笋中细菌多样性[J].中国酿造, 2019, 38(7):85-90.ZHOU J S, CHEN X Y, TAN J P, et al.Analysis of bacterial diversity in Guangxi non-salt fermentation bamboo shoots based on Illumina MiSeq sequencing technology[J].China Brewing, 2019, 38(7):85-90.
[15] 陈晓东, 朱志勇, 张韫, 等.基于宏基因组技术分析传统酸笋中微生物多样性[J].食品与生物技术学报, 2020, 39(8):89-98.CHEN X D, ZHU Z Y, ZHANG Y, et al.Microbial community diversity analysis of traditional fermented bamboo shoots using metagenomic technology[J].Journal of Food Science and Biotechnology, 2020, 39(8):89-98.
[16] 赵媛媛. 发酵酸菜的乳杆菌属细菌的筛选及应用[D].大连:大连工业大学, 2015.ZHAO Y Y.Screening and application of Lactobacillus for cabbage fermentation[D].Dalian:Dalian Polytechnic University, 2015.
[17] 朱琳, 高凤, 曾椿淋, 等.萝卜泡菜细菌多样性的高通量测序分析[J].现代食品科技, 2018, 34(2):225-231.ZHU L, GAO F, ZENG C L, et al.The analysis of microbial diversity in the fermentation broth of pickled radish based on the high-throughput DNA sequencing[J].Modern Food Science and Technology, 2018, 34(2):225-231.
[18] LIANG H P, HE Z, WANG X Y, et al.Effects of salt concentration on microbial diversity and volatile compounds during Suancai fermentation[J].Food Microbiology, 2020, 91:103537.
[19] TAMANG J P, SARKAR P K.Microbiology of mesu, a traditional fermented bamboo shoot product[J].International Journal of Food Microbiology, 1996, 29(1):49-58.
[20] 林松洋, 郝利民, 刘鑫, 等.乳酸菌耐盐分子机制研究进展[J].食品科学, 2018, 39(3):295-301.LIN S Y, HAO L M, LIU X, et al.Progress in molecular mechanism of salt tolerance in lactic acid bacteria[J].Food Science, 2018, 39(3):295-301.
[21] FU Z W, FENG Y R, GAO X, et al.Salt stress-induced chloroplastic hydrogen peroxide stimulates pdTPI sulfenylation and methylglyoxal accumulation[J].The Plant Cell, 2023, 35(5):1593-1616.
[22] 卢培利, 杨涵, 丁阿强, 等.碳源与氮源限制下细菌代谢调节研究进展[J].微生物学报, 2023, 63(3):946-962.LU P L, YANG H, DING A Q, et al.Metabolic regulation of bacteria with limited carbon and nitrogen sources[J].Acta Microbiologica Sinica, 2023, 63(3):946-962.
[23] CHEN C X, CHENG G G, LIU Y P, et al.Correlation between microorganisms and flavor of Chinese fermented sour bamboo shoot:Roles of Lactococcus and Lactobacillus in flavor formation[J].Food Bioscience, 2022, 50:101994.
[24] 刘啸尘, 刘护, 张良, 等.细胞代谢过程中的酶促糖基化及其功能[J].中国生物工程杂志, 2018, 38(1):69-77.LIU X C, LIU H, ZHANG L, et al.Enzymatic glycosylation and its function in metabolic process of cells[J].China Biotechnology, 2018, 38(1):69-77.
[25] 陆璐, 陶雅军, 罗学娅, 等.糖苷水解酶32家族结构与功能的研究进展[J].中国酿造, 2019, 38(8):14-19.LU L, TAO Y J, LUO X Y, et al.Research progress on the structure and function of glycoside hydrolase 32 family[J].China Brewing, 2019, 38(8):14-19.
[26] 唐志红, 鞠宝, 林剑, 等.多糖裂合酶及其应用前景[J].生命的化学, 2005, 25(2):163-166.TANG Z H, JU B, LIN J, et al.The development of polysaccharide lyase[J].Chemistry of Life, 2005, 25(2):163-166.
[27] WANG Y, HO C T.Flavour chemistry of methylglyoxal and glyoxal[J].Chemical Society Reviews, 2012, 41(11):4140-4149.
[28] 周杏荣, 陈晓艺, 蒋立文, 等.竹笋自然发酵过程中风味物质变化规律[J].中国酿造, 2019, 38(8):20-24.ZHOU X R, CHEN X Y, JIANG L W, et al.Changes of flavor substances in natural fermentation of bamboo shoots[J].China Brewing, 2019, 38(8):20-24.