蛋白质是人类和其他动物的主要食物成分。在工业生产中,某些蛋白质是食品工业及轻工业的重要原料,蛋白质在农业、畜牧业、水产养殖业领域也极其重要。工业生产中降解蛋白质的方法主要有酶降解、酸碱降解、氧化降解、热降解。其中,酶降解因具有生产周期短、生产效率高、产品纯度高、副产物少等优点在工业生产中具有广阔的应用前景。
蛋白酶是一种重要的水解酶类,其广泛存在于动物内脏、植物茎叶、果实和微生物中。但在工业生产中,蛋白酶的活力和稳定性受到多种环境因素的限制,使其在处理餐厨废水、工业废水与微生物菌肥制作等工艺中降解效果不明显。因此,寻找能够在高盐环境下生长和产生蛋白酶的菌株具有急迫性和重要性。强济宇[1]从土壤中筛选出一株高产碱性蛋白酶的菌株DS5,有较高的耐盐性,能够水解酪蛋白、脱脂牛奶和牛血清白蛋白等多种蛋白;李文静等[2]从鱼露发酵液中分离到一株耐盐且产蛋白酶的菌株,为鱼露的快速发酵提供一种高效的发酵剂和酶制剂;赵谋明等[3]从深海海泥中筛选出一株酶活力高、耐盐且稳定性好的菌株 SWJSS3,将其应用于食品加工领域和工业领域;周佳等[4]分离出一株能高效降解糖精钠废水中有机物的耐盐菌 Halomonas sp.A20,可为高盐、高浓度糖精钠废水的处理提供优良的微生物菌种资源。为了实现高效低耗地处理工业废水,制作菌肥等,耐盐产蛋白酶菌株资源还需大量扩充。
本研究从山东省威海市麻子港海岸带海洋沉积物中取样筛选出一株高产蛋白酶且具有良好耐盐特性的菌株,对其生长条件进行优化并进行酶活力测定,以期利用其产蛋白酶和耐盐的特点,为餐厨废水、工业废水与微生物菌肥制作等领域提供新的菌株和酶试剂。
1 材料与方法
1.1 材料
1.1.1 样品采集
从山东省威海市麻子港海岸带海洋沉积物中采集污泥。
1.1.2 培养基
富集培养基(g/L):胰蛋白胨10,酵母浸粉5,NaCl 10,121 ℃灭菌20 min。
蛋白质初筛培养基(g/L):MgCl2 2.26,MgSO4 3.25,CaCl2 1.15,NaHCO3 0.20,KCl 0.72,NaCl 30,琼脂18,脱脂牛奶20 mL/L,脱脂牛奶巴氏灭菌后加入121 ℃灭菌20 min的初筛培养基。
基础发酵培养基(g/L):豆粕40,玉米粉30,麸皮30,MgCl2 2.26,MgSO4 3.25,CaCl2 1.15,NaHCO3 0.20,KCl 0.72,NaCl 30,121 ℃灭菌20 min。
1.2 仪器与设备
V-1100D紫外可见分光光度计,上海美谱达仪器有限公司;SPX-250B-Z生化培养箱,上海博迅实业有限公司医疗设备厂;HH·S21-4-S电热恒温水浴锅,上海新苗医疗器械制造有限公司;Neo 15R高速冷冻离心机,上海力申科学仪器有限公司;MQD-S3 NR振荡培养箱,上海旻泉仪器有限公司。
1.3 实验方法
1.3.1 耐盐菌株的筛选
取10 g样品装入盛有无菌水的锥形瓶中,40 ℃,150 r/min下振荡2 h,吸取4 mL加入NaCl含量为100 g/L的LB液体培养基中,40 ℃,150 r/min培养48 h。取菌悬液进行10-1~10-7梯度稀释,分别吸取100 μL稀释液涂抹于LB平板,40 ℃下恒温培养24 h,得到的菌株分别接种于NaCl含量为100、110、120、130、140、150、160、170、180、190 g/L的LB固体培养基上,40 ℃培养箱培养24 h,筛选得到耐盐菌株。
1.3.2 产蛋白酶菌株的筛选
将初筛得到的耐盐菌株用三点法点接到蛋白质初筛培养基上,40 ℃培养箱培养24 h,观察有无透明的水解圈及水解圈的大小。
1.3.3 菌株的鉴定
1.3.3.1 菌株的形态学观察及生理生化鉴定
将筛选出来的菌株划线至LB固体培养基平板,40 ℃培养24 h,参考《常见细菌系统鉴定手册》[5]和《伯杰氏细菌鉴定手册》[6],对菌体进行形态学观察及生理生化鉴定实验。
1.3.3.2 菌株分子生物学鉴定
菌株于30 ℃、150 r/min振荡培养24 h,利用细菌基因组DNA提取试剂盒对其全基因组DNA进行提取。并利用提取的DNA作为PCR模板,采用细菌通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-ACGGTTACCTTGTTACGACTT-3′)进行PCR扩增。PCR扩增体系(50 μL):25 μL fast Taq-PCR MasterMix;2 μL 27F(10 μmol/);2 μL 1492R(10 μmol/L);20 μL RNase-Free Water;1 μL全基因组DNA模板。PCR扩增条件:95 ℃预变性5 min;98 ℃变性10 s,55 ℃退火5 s,72 ℃延伸10 s,循环30次;72 ℃温育1 min,4 ℃保存。将获得的16S rDNA片段送至睿博兴科测序,得到结果后用NCBI的BLAST工具分析,利用MEGA11.0软件中邻接(neighbor joining)法构建系统发育树。
1.3.4 菌株的生长曲线测定
菌液以1%的接种量接种到LB培养基中,在最适生长条件下培养,每隔4 h取样,于600 nm处测定吸光值,直至菌株生长至衰亡期,每组3个平行。
1.3.5 菌株的生长条件单因素优化
在初始发酵培养基的基础上考察不同温度、转速、初始pH值、盐度、氮源和碳源种类添加对菌株生长的影响,每组3个平行。试验各因素及水平值设置如表1所示。
表1 单因素实验各因素水平值设置
Table 1 Values of each factor
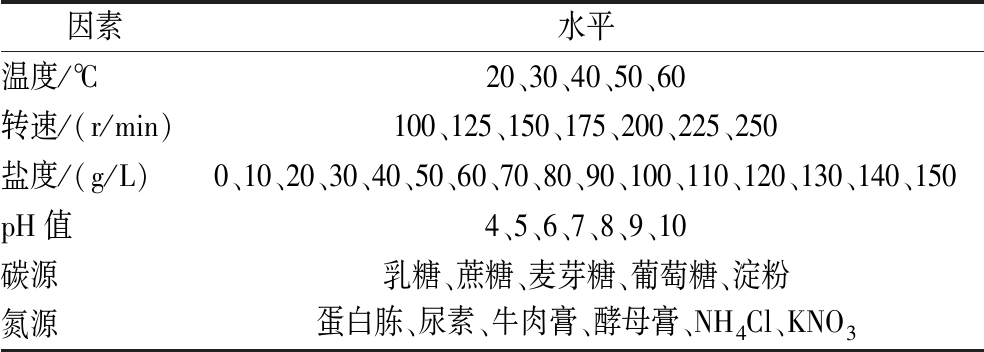
因素 水平温度/℃20、30、40、50、60转速/(r/min)100、125、150、175、200、225、250盐度/(g/L)0、10、20、30、40、50、60、70、80、90、100、110、120、130、140、150pH值4、5、6、7、8、9、10碳源乳糖、蔗糖、麦芽糖、葡萄糖、淀粉氮源蛋白胨、尿素、牛肉膏、酵母膏、NH4Cl、KNO3
1.3.6 菌株的生长条件响应面优化
对单因素实验中的6个因素进行Plackett-Burman实验设计,每个因素取高、低2个水平,比较各因素对菌株生长影响的显著性,以OD600值为响应值,选出3个显著因素运用Design Expert 13.0软件设计Box- Behnken实验,以此确定菌株的最佳生长条件。
1.3.7 菌株蛋白酶活力测定
酪氨酸标准曲线绘制采用GB/T 23527.1—2023 《酶制剂质量要求 第1部分:蛋白酶制剂》。以酪氨酸浓度为横坐标、吸光度为纵坐标,绘制标准曲线。取10 mL培养液离心10 min (条件:10 000 r/min,4 ℃冷冻离心)。试验组:取0.5 mL粗酶液加入10 g/L酪蛋白溶液1 mL,30 ℃水浴反应10 min,加入1.5 mL三氯乙酸溶液,静置10 min,用0.22 μm滤器过滤上清液,吸取1 mL上清液依次加入碳酸钠溶液5.0 mL,福林酚试剂1 mL,30 ℃水浴显色20 min,于680 nm下测定吸光值;对照组:先加三氯乙酸使蛋白酶失活再加酪蛋白溶液,其他操作均与实验组一致。
1.3.8 酶活力的定义及计算
参考冯璨等[7]和赵华等[8]的方法。蛋白酶活力定义:30 ℃条件下,每分钟水解酪蛋白产生1 μg酪氨酸所需要的蛋白酶量为一个酶活力单位(U/mL)。蛋白酶活力计算如公式(1)所示[9]:
(1)
式中:U,样品的蛋白酶活力,U/mL;A,样品的OD680值代入标准曲线回归方程所得的酪氨酸质量浓度,μg/mL;3,反应液的总体积,mL;N,酶液稀释总倍数;t,酶促反应时间,min。
1.3.9 菌株的基因组测序
将对数增长期的菌液低速离心后取菌体沉淀弃上清液,将样品送至北京诺禾致源科技股份有限公司进行基因组测序。对测序得到的基因组结果进行基本特征分析,并将预测得到的基因序列与GO、COG(cluster of orthologous group)、KEGG等数据库进行比对,得到基因功能注释结果[10]。研究分析与A1菌株降解蛋白质和耐盐性有关的基因和代谢通路。
2 结果与分析
2.1 菌株的筛选
2.1.1 耐盐菌株的筛选
共筛选出耐盐菌株25株,其耐盐性及编号见表2。最高耐受盐度在150 g/L以上的有18株,选取这些菌株进行后续的蛋白质水解圈实验。
表2 耐盐菌的初筛
Table 2 Primary screening of salt-tolerant bacteria

注:-:不生长;+:少量生长;++:生长一般;+++:生长良好。
菌株NaCl质量浓度/(g/L)1001101201301401501601701801901++++++++++++++----2+++++++++------3++++++++++++-----4++++++++++++++----5++++++++++++++++++--6+++++++++++-----7++++++++++++++++++++++++8++++++++++++++++++++-9+++++++++++++++++++++++++++10++++++++++++++++++++++++++++1121+++++++++++++++++++++++-12++++++++++++++++++++++++13++++++++++++++++++++++++a-1+++++++++++++++++++++++-a-2+++++++++++++++++++++++B++++++++++++++++++++++++++++C++++++++++++++++++--D++++++++++++++++++++++++A1+++++++++++++---A2++++++++++++++++++++--A3++++++++++++++----A4+++++++++++++++++++++-A5+++++++++++++++++++--A6++++++++++++++----A7++++++++++++++++++++-
2.1.2 耐盐产蛋白酶菌株的筛选
经过筛选共得到5株具有水解圈的菌株,分别为A1、B、C、D、1121(图1)。其中A1的水解圈最大,因此将A1菌株作为后续生长条件和蛋白酶活力测定的实验菌。

a-菌株A1;b-菌株B;c-菌株C;d-菌株D;e-菌株1121
图1 各菌株的蛋白质水解圈
Fig.1 Protein hydrolysis circles of each strain
2.2 菌株的形态学观察
菌落外部呈白色,边缘不规则,略有凸起,表面光滑湿润有光泽,通过革兰氏染色和光学显微镜观察确定A1为革兰氏阳性菌,菌体呈短小杆状,有芽孢;A1菌体的透射电镜观察结果如图2所示,菌体呈杆状,无鞭毛。
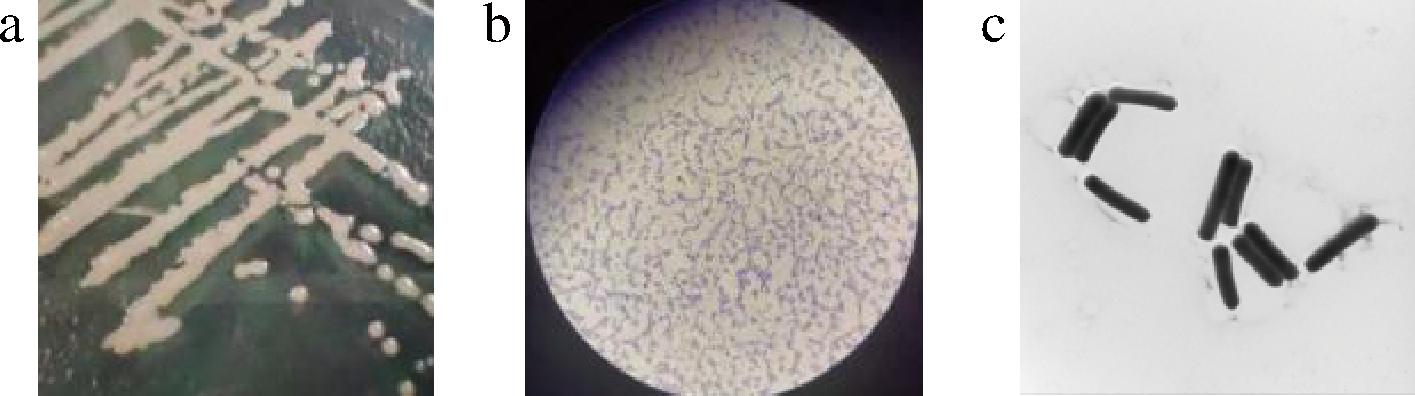
a-菌株A1的外部形态;b-菌株A1的革兰氏染色;c-菌株A1的透射电镜观察
图2 菌株A1的外部形态和革兰氏染色及透射电镜观察
Fig.2 External morphology, gram staining and transmission electron microscopic observation of strain A1
2.3 菌株的分子生物学鉴定结果
利用生理生化试验鉴定管对菌株A1生理生化特征进行探究,结果如表3所示。A1菌株对甘露醇、尿素酶、甘油、邻硝基苯-β-D-吡喃半乳糖苷(o-nitrophenyl β-D-galactopyranosid,ONPG)等呈现阳性反应,对半固体琼脂、黏液酸盐、氰化钾等呈阴性反应,不能水解淀粉,这与孙倩[11]关于高地芽孢杆菌(Bacillus altitudinis)的生理生化鉴定结果一致。
表3 菌株A1的生理生化鉴定结果
Table 3 Physiological and biochemical characteristics of strain A1

注:+:阳性;-:阴性。
鉴定项目结果鉴定项目结果半固体琼脂-尿素酶+甘露醇+黏液酸盐-棉子糖-水杨苷+甘油+七叶苷+ONPG+氰化钾-葡萄糖胺-山梨醇-西蒙氏枸橼酸盐-丙二酸-鸟氨酸脱羧酶-卫矛醇-淀粉水解-
2.4 菌株的生理生化鉴定结果
将获得的16S rDNA序列在NCBI数据库中进行相似度比对。比对结果显示该菌株为B.altitudinis。使用MEGA11.0软件采用邻接(neighbor joining)法构建系统发育树,如图3,显示A1与标准菌种空气芽孢杆菌(Bacillus aerius)形成一个独立小分支,亲缘关系最近;这2株菌形成的小分支与B.altitudinis在同一个大分支上,亲缘关系较近,结合BLAST分析与系统发育树结果表明该菌株为B.altitudinis。

图3 菌株A1的系统发育树
Fig.3 Phylogenetic tree of strain A1
2.5 菌株A1的生长曲线
由图4可知,菌株在0~4 h生长速率较缓慢,为生长迟缓期,在4~20 h生长速率最快,为对数生长期,在20~32 h菌体数量基本不变,菌体死亡速率和新生速率基本持平,这一阶段为平稳期,32 h以后,由于营养物质的逐渐消耗,菌体量少,进入衰亡期。
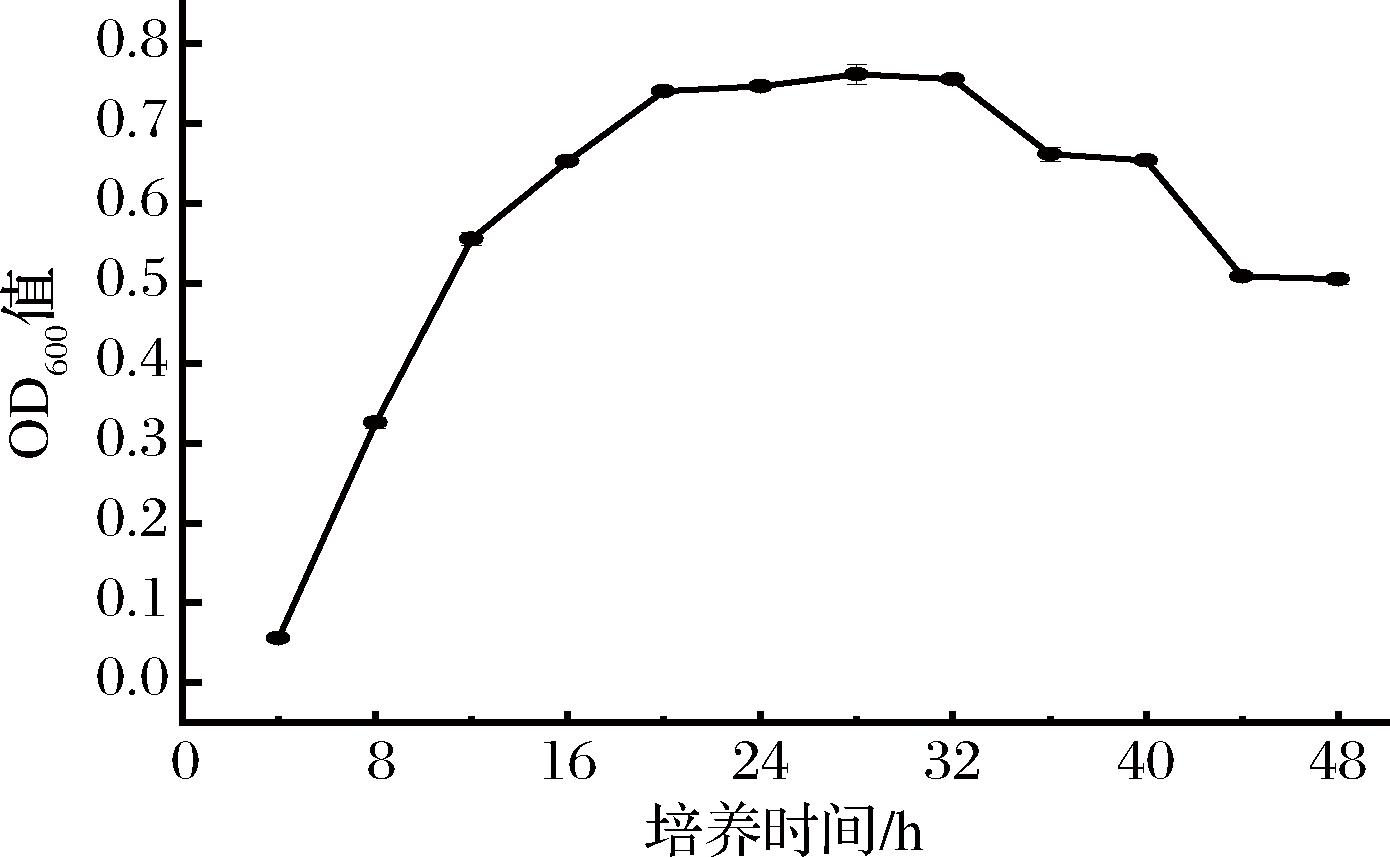
图4 菌株A1的生长曲线
Fig.4 Growth curve of strain A1
2.6 菌株的生长条件优化结果
2.6.1 温度、转速、pH值、盐度对菌株生长的影响
如图5所示,菌株在30 ℃生长最佳,且具有耐热性;菌株的最适转速为225 r/min;菌株的最适pH值为9,有较好的pH稳定性,适宜在中性和碱性的环境下生长。菌株的最适盐度为30 g/L,在10~80 g/L时生长良好。冯曌卓等[12]筛选出的NY1在40 ℃下的吸光度低于20~35 ℃,耐热性比本实验菌株弱。马文花等[13]从石油化工厂排放的污泥和废水中分离得到的HB2最高耐盐度达100 g/L,吴民熙等[14]从含菜籽饼的堆肥样品中筛选得到的菌株,最高耐盐浓度达到100 g/L,均比本实验菌株的最高耐受盐度低。
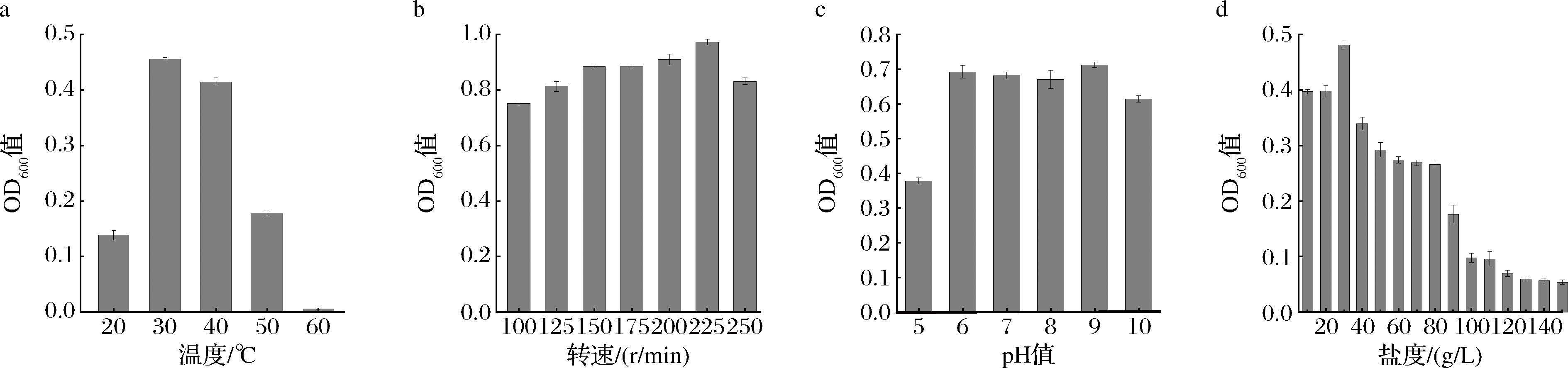
a-温度对菌株生长的影响;b-转速对菌株生长的影响;c-pH值对菌株生长的影响;d-盐度对菌株生长的影响
图5 温度、转速、pH值、盐度对菌株A1生长的影响
Fig.5 Effects of temperature, shaker speed, pH, and salinity on the growth of strain A1
2.6.2 不同氮源、碳源的添加对菌株生长的影响
由图6可知,菌株的最适添加氮源为蛋白胨,硝酸钾次之,而尿素的吸光度接近于0,说明该菌株对无机氮和有机氮均能很好利用且尿素对该菌株的生长可能有抑制作用。当添加的碳源为葡萄糖时,菌株生长最好,其次是蔗糖,最低是淀粉,这与张程等[15]对淀粉芽孢杆菌生长培养基所需的最适碳源的研究结果一致。
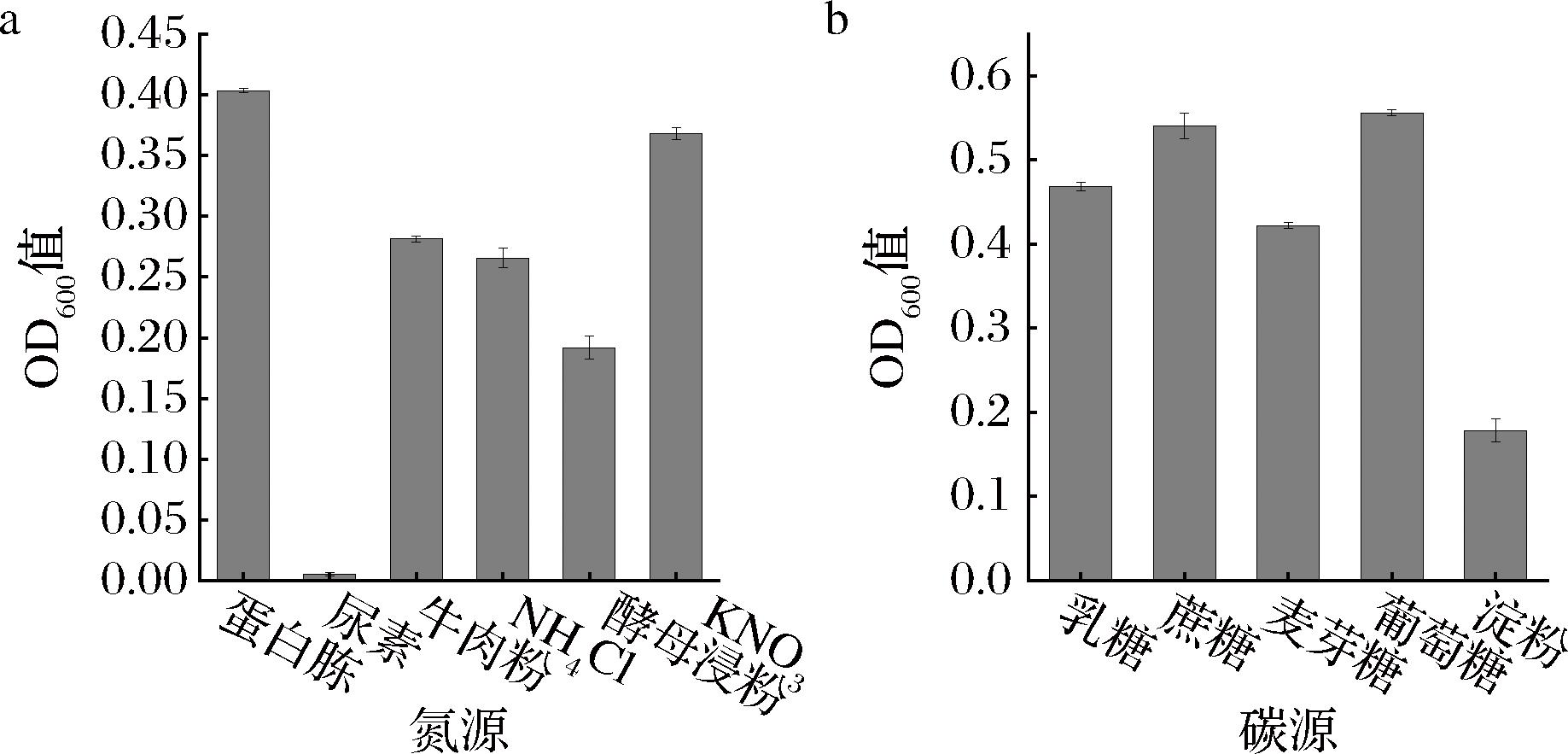
a-氮源对菌株生长的影响;b-碳源对菌株生长的影响
图6 不同氮源、碳源的添加对菌株A1生长的影响
Fig.6 Effects of addition of different nitrogen and carbon sources on the growth of strain A1
2.6.3 响应面实验设计
2.6.3.1 Plackett-Burman试验
Plackett-Burman试验设计及结果见表4。各因素对菌株A1生长的显著性结果见表5。分析可知,该回归模型F=14.96、P<0.01,线性回归模型极显著。由P值可知,在发酵优化分析中各因素对菌株A1生长条件的影响重要性排序为:温度>碳源>转速>pH值>盐度>氮源,其中温度、碳源的P值均小于0.01,对A1的生长有极显著影响,而转速的P值<0.05,对A1的生长有显著影响。因此选择这3个因素作为主要的影响因子进行后续试验。
表4 Plackett-Burman试验组合设计及结果
Table 4 Plackett-Burman experimental combination design and results
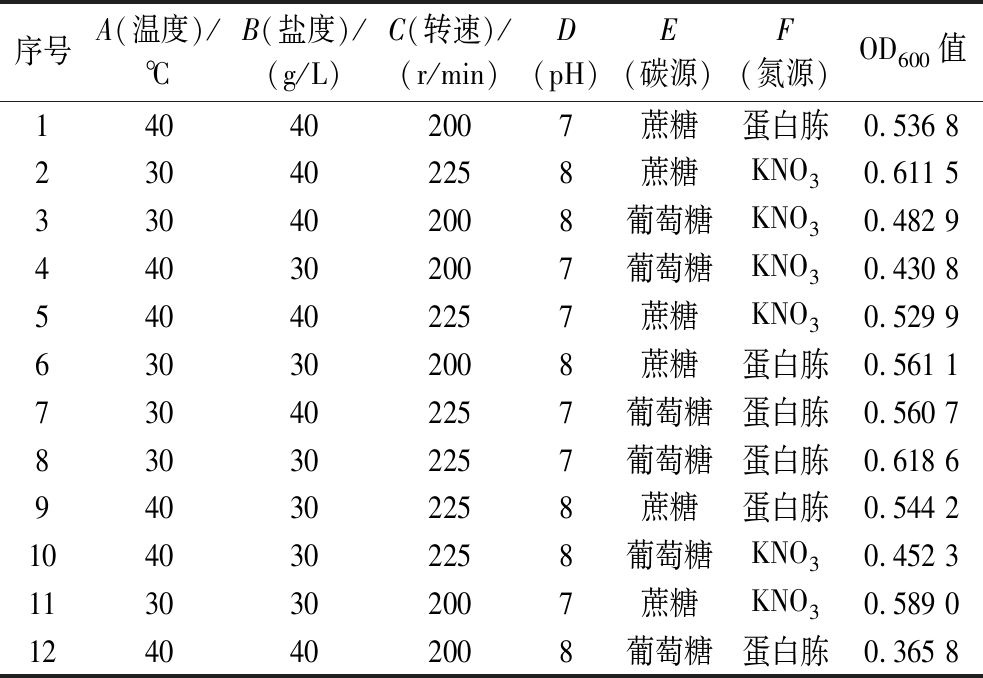
序号A(温度)/℃B(盐度)/(g/L)C(转速)/(r/min)D(pH)E(碳源)F(氮源)OD600值140402007蔗糖蛋白胨0.536 8230402258蔗糖KNO30.611 5330402008葡萄糖KNO30.482 9440302007葡萄糖KNO30.430 8540402257蔗糖KNO30.529 9630302008蔗糖蛋白胨0.561 1730402257葡萄糖蛋白胨0.560 7830302257葡萄糖蛋白胨0.618 6940302258蔗糖蛋白胨0.544 21040302258葡萄糖KNO30.452 31130302007蔗糖KNO30.589 01240402008葡萄糖蛋白胨0.365 8
表5 Plackett-Burman实验中各因素的显著性分析
Table 5 Significance analysis of various factors in Plackett-Burman experiment
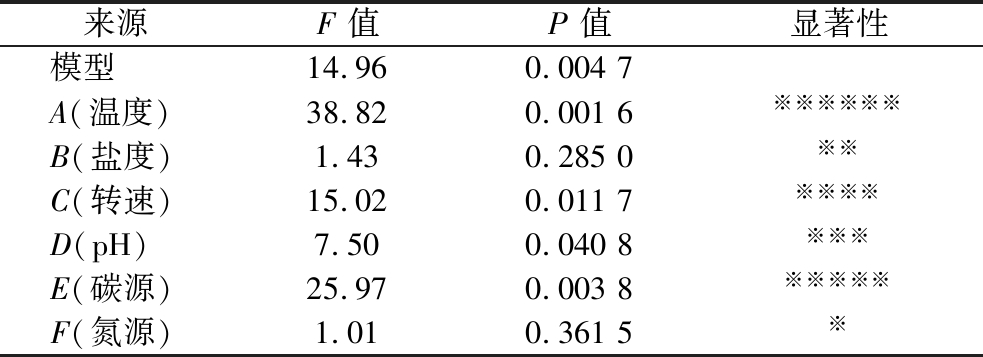
注:显著性从※※※※※※到※依次降低。
来源F 值P 值显著性模型14.960.004 7A(温度)38.820.001 6※※※※※※B(盐度)1.430.285 0※※C(转速)15.020.011 7※※※※D(pH)7.500.040 8※※※E(碳源)25.970.003 8※※※※※F(氮源)1.010.361 5※
2.6.3.2 Box-Behnken试验回归分析
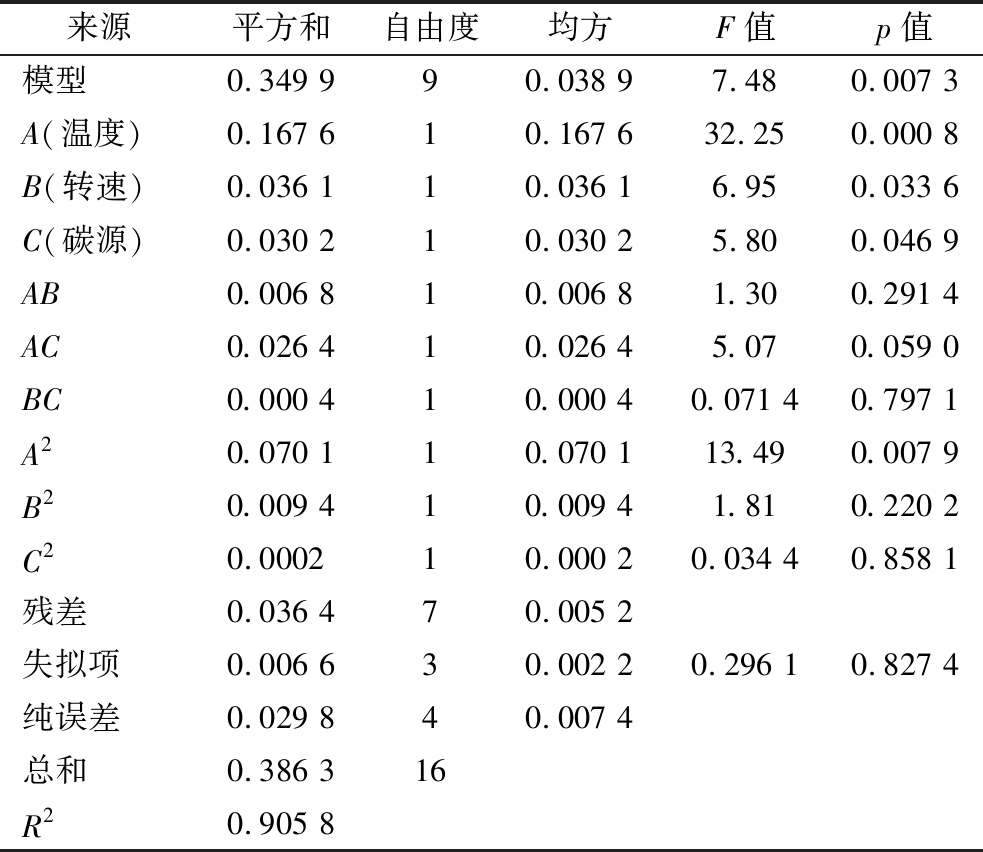
Box-Behnken试验设计的方案及实验结果见表6。回归分析结果见表7。该模型的P值<0.01,具有极高显著性;失拟项P值为0.827 4>0.05,不显著,实验误差小;相关系数R2为0.905 8,有90.58%的数据可以用该模型确定,该模型能较好拟合各因素与菌株生长之间的关系。方差分析结果表明,温度的一次项和二次项均对该菌株生长条件的回归模型影响极显著(P<0.01)。各因素对菌株A1生长的影响顺序为A>B>C。利用Design Expert 13.0 软件对该模型进行分析,得到回归方程:OD=-13.830 32+0.464 257A+0.048 253B+0.716 399C-0.000 329AB-0.016 238AC-0.000 385BC-0.005 161A2-0.000 076B2+0.006 516C2。各因素交互作用对菌株生长条件的曲面图和等高线图如图7所示。

a-温度与转速对菌株生长的响应图;b-转速与碳源对菌株生长的响应面图;c-温度与碳源对菌株生长的响应图;d-温度与转速对菌株生长的等高线图;e-温度与碳源对菌株生长的等高线图;f-转速与碳源对菌株生长的等高线图
图7 3D响应面图和等高线图
Fig.7 3D response surface maps and contour plots
表6 Box-Behnken试验设计及结果
Table 6 Box-Behnken experimental design and results
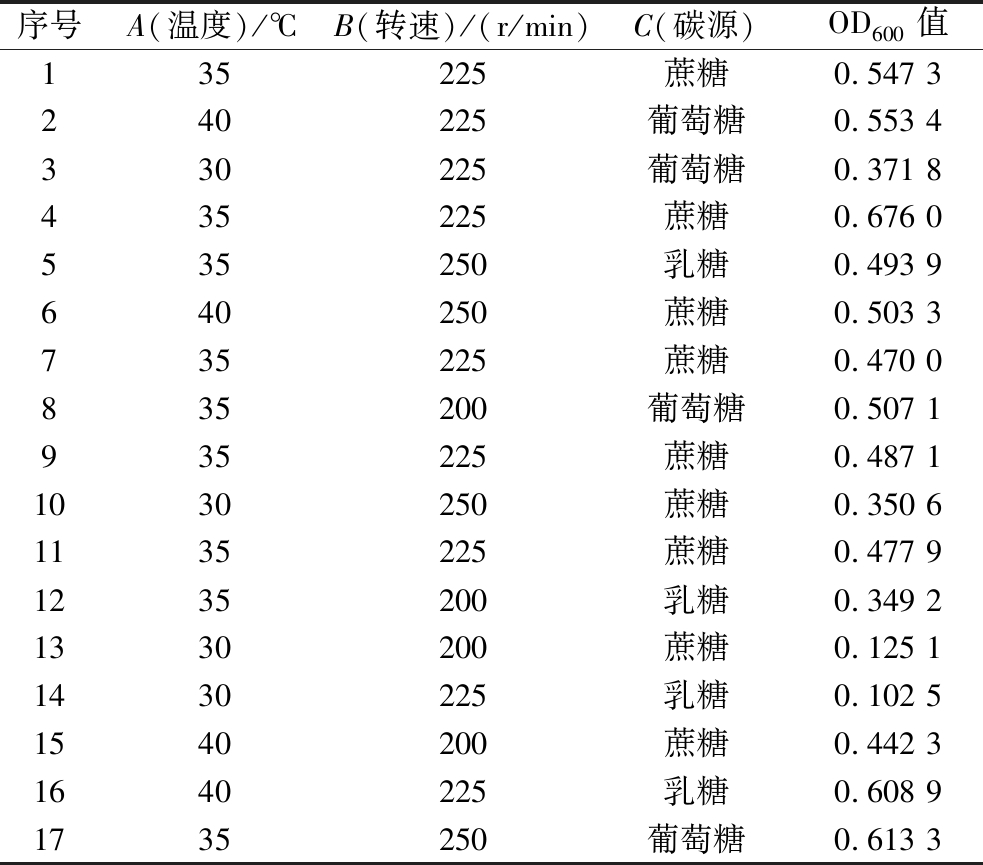
序号A(温度)/℃B(转速)/(r/min)C(碳源)OD600值135225蔗糖0.547 3240225葡萄糖0.553 4330225葡萄糖0.371 8435225蔗糖0.676 0535250乳糖0.493 9640250蔗糖0.503 3735225蔗糖0.470 0835200葡萄糖0.507 1935225蔗糖0.487 11030250蔗糖0.350 61135225蔗糖0.477 91235200乳糖0.349 21330200蔗糖0.125 11430225乳糖0.102 51540200蔗糖0.442 31640225乳糖0.608 91735250葡萄糖0.613 3
表7 Box-Behnken 试验回归方程和方差分析
Table 7 Box-Behnken test regression equation and analysis of variance
注:P<0.01为差异极显著;P<0.05为差异显著。
来源平方和自由度均方F值p值模型0.349 990.038 97.480.007 3A(温度)0.167 610.167 632.250.000 8B(转速)0.036 110.036 16.950.033 6C(碳源)0.030 210.030 25.800.046 9AB0.006 810.006 81.300.291 4AC0.026 410.026 45.070.059 0BC0.000 410.000 40.071 40.797 1A20.070 110.070 113.490.007 9B20.009 410.009 41.810.220 2C20.000210.000 20.034 40.858 1残差0.036 470.005 2失拟项0.006 630.002 20.296 10.827 4纯误差0.029 840.007 4总和0.386 316R20.905 8
2.6.3.3 响应面验证试验
通过Design-Expert13.0软件预测响应值最大值的各因素组合为温度39.999 9 ℃,转速229.32 r/min,碳源为葡萄糖,预测到最大值为OD600值0.535,为方便实际操作和验证模型的有效性,将最佳生长条件修正为温度40 ℃,转速229 r/min,碳源为葡萄糖。在此条件下,进行3次平行验证实验,得到实测值为0.579,较理论值的误差为8.224%,该模型预测结果可靠。
2.7 菌株的蛋白酶酶活测定结果
2.7.1 L-酪氨酸标准曲线
按照1.2.7节所述方法,绘制标准曲线,线性回归方程为y=0.013 69x+0.012 3,R2=0.995 2,线性拟合程度良好,可用于计算蛋白酶活力。
2.7.2 蛋白酶活力计算结果
吸取1%的种子液于添加1%葡萄糖和蛋白胨,pH值为9,盐度为30 g/L的发酵培养基中,并于40 ℃,229 r/min下培养24 h。获取粗酶液测定蛋白酶活力。根据蛋白酶活力计算公式和标准曲线,计算出在最佳生长条件下的酶活力为99.747 U/mL。ALIAS等[16]从马来西亚登嘉楼州拉温泉中分离筛选得到3株具有较高蛋白酶活力的枯草芽孢杆菌,酶活力分别为85.73、82.90和83.05 U/mL,赵龙妹等[17]筛选得到的菌株蛋白酶活力最高值57.77 U/mL,均比本实验菌株的酶活力低。
2.8 菌株的基因组测序及结果分析
2.8.1 A1基因组的基本特征
A1基因组的基本特征如表8所示。
表8 基因组的基本特征
Table 8 Basic characteristics of the genome

指标分析结果基因组总长/bp3 827 583前噬菌体/个13基因岛/个11基因岛总长度/bp93 769基因岛平均长度/bp8 524编码基因数量/个4 095编码基因总长度/bp3 414 150编码基因平均长度/bp834编码基因的N50长度/bp949 572编码基因的N90长度/bp319 524编码基因GC含量/%41.18tRNA数/个62rRNA数/个14sRNA/个10串联重复序列/个124
2.8.2 GO注释结果
在GO分类中,A1共有2 776个基因得到注释。其中在细胞学组件方面注释到最多的基因为细胞组分,有1 104个;生物学途径方面注释到最多的为代谢过程,有1 618个,表明该菌具有高度的代谢活性;分子功能共3 742个注释结果,其中结合(binding)、催化活性(catalytic activity)和转运活性(transporter activity)有关编码基因分别为1 366、1 575、273个,说明该菌在结合、转运和催化等方面也具有较强能力。
2.8.3 COG注释结果
注释到COG信息的基因数目为2 932个,占编码基因总数的71.60%。A1菌株基因组的COG注释结果如图8所示,富集在氨基酸的运输和代谢(amino acid transport and metabolism)的基因最多,为319个,表明 A1菌株具有丰富的氨基酸代谢基因。与转录(transcription)、一般功能预测(general function prediction only)、碳水化合物转运代谢(carbohydrate transport and metabolism)、辅酶运输和代谢(coenzyme transport and metabolism)、无机离子运输和代谢(inorganic ion transport and metabolism)有关的基因分别为305、240、237、192、176个。此外,A1菌株的基因组中含有编码运输各种底物,包括氨基酸、肽、糖、铁、核苷酸和其他小分子的MFS超家族转运蛋白(major facilitator superfamily,MFS)的32个编码基因。这些结果表明,菌株A1具有降解蛋白质和碳水化合物的良好潜力[18]。
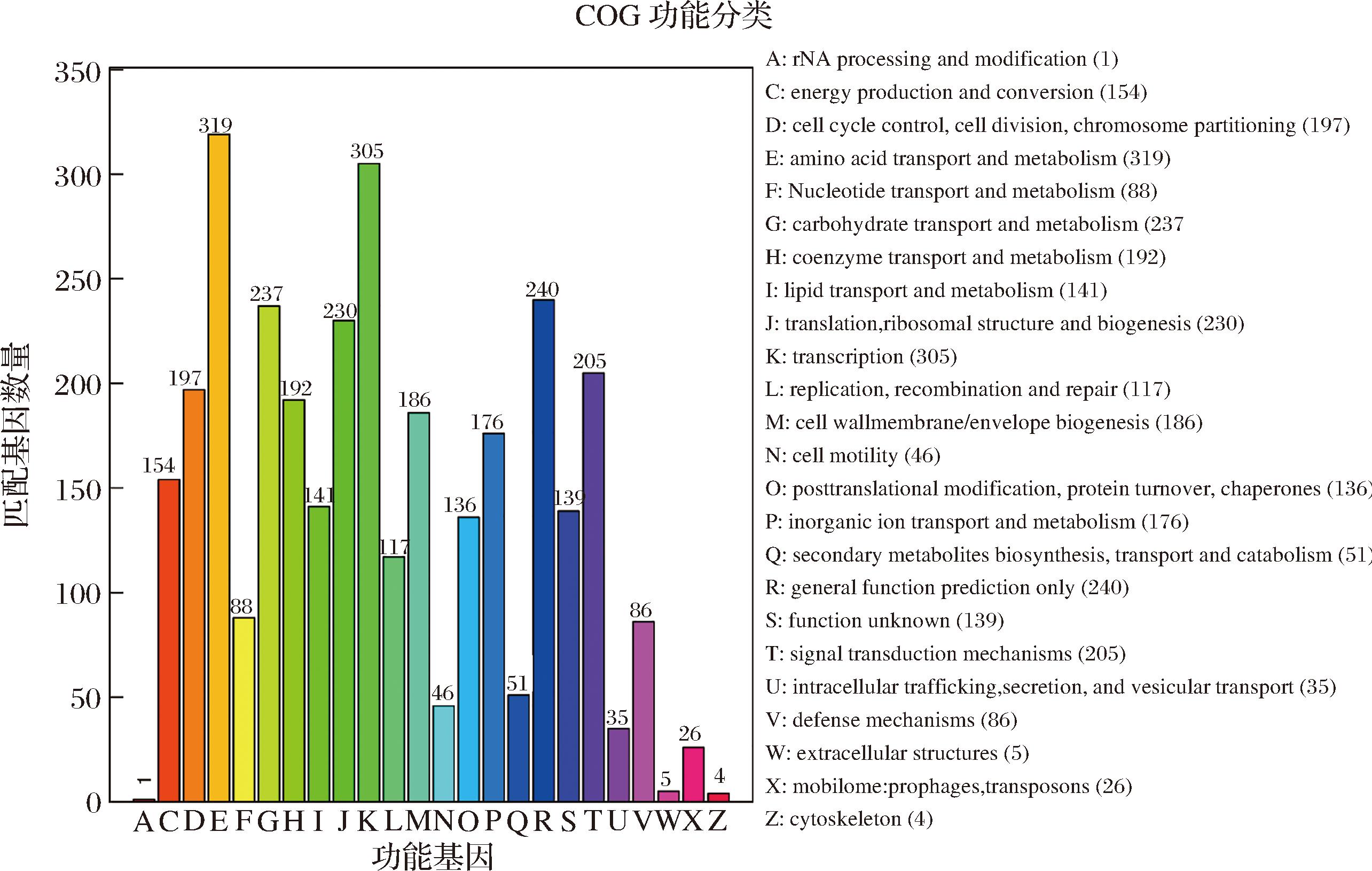
图8 A1的基因组COG注释结果
Fig.8 COG annotation results of the genome of strain A1
注:RNA processing and modification:RNA加工与修饰;energy production and conversion:能量产生与转换;cell cycle control, cell division, chromosome partitioning:细胞周期控制、细胞分裂、染色体分离;amino acid transport and metabolism:氨基酸运输与代谢; nucleotide transport and metabolism:核苷酸运输与代谢;carbohydrate transport and metabolis:碳水化合物运输与代谢;coenzyme transport and metabolis:辅酶运输与代谢:lipid transport and metabolism:脂质运输与代谢;translation, ribosomal structure and biogenesis:翻译、核糖体结构和生物发生;transcription:转录;replication, recombination and repair:复制、重组和修复;cell wall/membrane/envelope biogenesis:细胞壁/膜/外膜生物发生;cell motility:细胞运动;posttranslational modification, protein turnover,chaperones:翻译后修饰、蛋白质周转、分子伴侣;inorganic ion transport and metabolism:无机离子运输与代谢;secondary metabolites biosynthesis, transport and catabolism:次级代谢物的生物合成、运输和分解代谢;general function prediction only:仅一般功能预测;function unknown:功能未知;signal transduction mechanisms:信号转导机制;intracellular trafficking, secretion, and vesicular transport:细胞内运输、分泌和囊泡运输;defense mechanisms:防御机制;extracellular structures:细胞外结构;mobilome: prophages, transposons:动员组:噬菌体原,转座子;cytoskeleton:细胞骨架。
2.8.4 KEGG注释结果
A1的KEGG注释结果如图9所示,A1共有2 232个基因注释到KEGG,注释到新陈代谢的基因数量最多,主要富集在碳水化合物代谢(carbohydrate metabolism)和氨基酸代谢(amino acid metabolism),分别为208个和205个,表明A1菌株具有较强的营养物质的合成和代谢的能力。根据KEGG注释信息,A1中的蛋白编码基因主要富集在丙氨酸、天冬氨酸、谷氨酸、甘氨酸、丝氨酸、ABC转运体等代谢通路中。这些基因主要编码蛋白酶以及多种内肽酶和外肽酶,如丝氨酸蛋白酶AprX(serine protease AprX)、丝氨酸蛋白酶Do(serine protease Do)、主要胞内丝氨酸蛋白酶(major intracellular serine protease)、微量胞外丝氨酸蛋白酶Vpr(minor extracellular serine protease Vpr)、ATP依赖的Clp蛋白酶(ATP-dependent Clp protease)、ATP依赖的HslUV蛋白酶ATP结合亚基HslU(ATP-dependent HslUV protease ATP-binding subunit HslU)、枯草杆菌蛋白酶(subtilisin), 细胞壁相关蛋白酶(cell wall-associated protease)、细胞分裂蛋白酶FtsH(cell division protease FtsH)、Rhomboid蛋白酶Glup(rhomboid protease GluP)、蛋白酶Ⅳ(protease Ⅳ)、氨基肽酶(aminopeptidase)、Xaa-Pro氨基肽酶(Xaa-Pro aminopeptidase)、亮氨酸氨基肽酶(leucyl aminopeptidase)、丝氨酸型D-丙氨酸-D-丙氨酸羧肽酶(serine-type D-Ala-D-Ala carboxypeptidase)等。这些基因的存在表明菌株A1具有良好的蛋白质降解能力[19-20]。

图9 A1 的KEGG注释结果
Fig.9 KEGG annotation results for A1
注:cellular processes:细胞过程;transport and catabolism:运输与分解代谢;cell growth and death:细胞生长与死亡;cell communication:细胞通讯;cell motility:细胞运动;environmental information processing:环境信息处理;signaling molecules and interaction:信号分子与相互作用;signal transduction:信号转导;membrane transport:膜转运;genetic information processing:遗传信息处理;translation:翻译;transcription:转录;replication and repair:复制与修复;folding, sorting and degradation:折叠、分选和降解;human diseases:人类疾病;neurodegenerative diseases:神经退行性疾病;infectious disease: viral:感染性疾病:病毒性:infectious disease: parasitic:感染性疾病:寄生虫性;infectious disease: bacterial:感染性疾病:细菌性;immune diseases:免疫疾病;endocrine and metabolic diseases:内分泌与代谢疾病;drug resistance: antineoplastic:耐药性:抗肿瘤药物; drug resistance: antimicrobial:耐药性:抗微生物药物;cardiovascular disease:心血管疾病;cancer: specific types:癌症:特定类型;cancer: overview:癌症:概述;metabolism:代谢;xenobiotics biodegradation and metabolism:异生物质的生物降解与代谢;nucleotide metabolism:核苷酸代谢;metabolism of terpenoids and polyketides:萜类和多酮类代谢;metabolism of other amino acids:其他氨基酸代谢;metabolism of cofactors and vitamins:辅因子和维生素代谢;lipid metabolism:脂质代谢;glycan biosynthesis and metabolism:聚糖生物合成与代谢;energy metabolism:能量代谢;carbohydrate metabolism:碳水化合物代谢;biosynthesis of other secondary metabolites:其他次级代谢物的生物合成;amino acid metabolism:氨基酸代谢;organismal systems:生物系统;nervous system:神经系统;immune system:免疫系统;excretory system:排泄系统:environmental adaptation:环境适应;endocrine system:内分泌系统;digestive system:消化系统;aging:衰老。
3 结论与讨论
从山东省威海市麻子港海岸带海洋沉积物中采集的样品中筛选得到一株高产蛋白酶且具有耐盐特性的高地芽孢杆菌。经过16S rDNA分析、形态学、生理生化指标检测,鉴定该菌株为高地芽孢杆菌。通过单因素实验、Plackett-Burman试验和Box-Behnken试验,得出菌株A1的最适生长条件,菌株能在温度20~55 ℃,pH值5.0~10.0下生长,具有很好的耐热性和pH稳定性。且菌株A1在盐度10~80 g/L的条件下生长良好,160 g/L盐度下仍能生长。YOO等[21]从盐滩分离的耐盐菌的最高耐受温度为40 ℃,盐度为150 g/L,CHHETRI等[22]从水稻植物根部分离的中度耐盐细菌生长的pH值为5.0~8.0,万文结等[23]筛选得到的产碱性蛋白酶菌株在pH值为5和9时生长较差,而本文研究的菌株A1的最高耐受温度和盐度分别为55 ℃和160 g/L,pH值为5.0~10.0时仍能很好生长,与其相比,温度和盐度最大耐受限度更高,pH耐受范围更广,对环境的适应性更强。本耐盐菌适应期较短, 对数生长期与稳定期相对较长, 即耐盐菌在生产利用中更利于消耗有机物与各种无机离子,在处理废水时可快速发挥作用,持续并稳定地消耗污染物。该菌株在最佳生长条件下的酶活力为99.747 U/mL,孙佑赫等[24]筛选得到的产蛋白酶的菌株最高酶活力达50.07 U/ mL,低于本实验菌株。A1菌株环境适应性强、耐盐性好、安全性高且对蛋白质具有降解作用,可以为餐厨垃圾降解、高盐废水处理和微生物菌肥制作等提供生物学依据。
[1] 强济宇. 碱性蛋白酶耐盐芽孢杆菌DS5的筛选、鉴定和性质研究[D].太原:山西大学, 2023.QIANG J Y.Screening, identification and characterization of alkaline protease halotolerant bacillus DS5[D].Taiyuan:Shanxi University, 2023.
[2] 李文静, 李春生, 李来好, 等.鱼露中高产蛋白酶耐盐菌株的筛选、鉴定及产酶条件优化[J].食品与发酵工业, 2021, 47(23):134-142.LI W J, LI C S, LI L H, et al.Isolation, identification and enzyme-producing condition of a salt-tolerant strain with high protease production[J].Food and Fermentation Industries, 2021, 47(23):134-142.
[3] 赵谋明, 舒会, 崔春, 等.产耐盐蛋白酶深海细菌的分离鉴定[J].现代食品科技, 2015, 31(3):50-54.ZHAO M M, SHU H, CUI C, et al.The isolation and identification of deep-sea bacteria that produce salt-tolerant proteases[J].Modern Food Science and Technology, 2015, 31(3):50-54.
[4] 周佳, 陈研, 屈建航, 等.一株耐盐菌Halomonas sp.A20的分离及降解糖精钠废水的特性[J].微生物学通报, 2022, 49(3):942-955.ZHOU J, CHEN Y, QU J H, et al.Isolation of the salt-tolerant bacterium Halomonas sp.A20 and its degradation of saccharin sodium wastewater[J].Microbiology China, 2022, 49(3):942-955.
[5] 东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.DONG X Z,CAI M Y.Handbook of Systematic Identification of Common Bacteria[M].Beijing:Science Press, 2001.
[6] R.E. BUCHANAN,N.E. GIBBONS.伯杰细菌鉴定手册[M].第8版.北京:科学出版社,1984.BUCHANAN R E, GIBBONS N E.Bergey’s Manual of Determinative acteriology[M].8th Edition.Beijing:Science Press, 1984.
[7] 冯璨, 马香, 刘柱, 等.海南近海水域产碱性蛋白酶菌株的分离筛选及发酵条件优化[J].微生物学通报, 2022, 49(10):4291-4304.FENG C, MA X, LIU Z, et al.Isolation and screening of alkaline protease-producing strains from Hainan offshore areas and optimization of the fermentation conditions[J].Microbiology China, 2022, 49(10):4291-4304.
[8] 赵华, 任青霞, 张敏, 等.酒曲中高产蛋白酶菌株的筛选及其发酵培养基优化[J].中国酿造, 2023, 42(5):157-164.ZHAO H, REN Q X, ZHANG M, et al.Screening and fermentation medium optimization of high-yield protease-producing strain from Jiuqu[J].China Brewing, 2023, 42(5):157-164.
[9] 万明铼. 高产碱性蛋白酶菌株的筛选及酶学性质的研究[D].兰州:兰州理工大学, 2023.WAN M L.Screening of the bacterial strain producing high-yield alkaline protease strain and study on enzymatic properties[D].Lanzhou:Lanzhou University of Technology, 2023.
[10] 张雯姝. 产蛋白酶菌株的ARTP诱变及其降解蛋白质代谢通路分析[D].大连:辽宁师范大学, 2023.ZHANG W S.Mutation of protease-producing strain by ARTP and analysis of its metabolic pathway for degrading protein[D].Dalian:Liaoning Normal University, 2023.
[11] 孙倩. 高产碱性蛋白酶菌种的选育及发酵过程优化[D].郑州:河南工业大学, 2012.SUN Q. Breeding of high-yield alkaline protease strain and optimization of fermentation process[D]. Zhengzhou: Henan University of Technology, 2012.
[12] 冯曌卓,李海红,王玥,等.含盐废水中优势耐盐菌的筛选及生长条件优化[J].环境科学与技术, 2021,44(7):1-8.FENG Z Z, LI H H, WANG Y, et al.Screening and identification of dominant salt-tolerant bacteria and optimization of growth conditions[J].Environmental Science &Technology, 2021, 44(7):1-8.
[13] 马文花, 吴越, 李进胜, 等.一株耐盐细菌的分离初步鉴定及对医药废水降解特性的研究[J].大连民族学院学报, 2009, 11(3):204-208.MA W H, WU Y, LI J S, et al.Isolation and preliminary identification of a salt-tolerant strain and investigation of its role in medical wastewater degradation[J].Journal of Dalian Nationalities University, 2009, 11(3):204-208.
[14] 吴民熙, 刘清术, 刘前刚, 等.降解菜籽饼粗蛋白耐盐菌的筛选、鉴定及固体发酵的初步研究[J].天然产物研究与开发, 2014, 26(3):309-313.WU M X, LIU Q S, LIU Q G, et al.Screening, identification and preliminary study of solid-state fermentation of the crude protein-degradation salt-tolerant strains in rapeseed cake[J].Natural Product Research and Development, 2014, 26(3):309-313.
[15] 张程, 谭琴, 陈露露, 等.产蛋白酶菌株的筛选鉴定及产酶优化[J].包装学报, 2023, 15(1):62-70.ZHANG C, TAN Q, CHEN L L, et al.Screening and identification of protease-producing strain and optimization of enzyme production[J].Packaging Journal, 2023, 15(1):62-70.
[16] ALIAS M, CHE HARUN MOHAMMAD H, RAZALI NURUL A, et al.Production and characterisation of thermostable alkaline protease from Bacillus subtilis isolated from LA hot spring, Terengganu[J].Research Journal of Biotechnology, 2021, 16(7):84-91.
[17] 赵龙妹, 张玲.产蛋白酶芽孢杆菌的筛选鉴定及酶学特性分析[J].畜牧与兽医, 2021, 53(3):59-64.ZHAO L M, ZHANG L.Screening, identification and characterization of protease-producing Bacillus[J].Animal Husbandry &Veterinary Medicine, 2021, 53(3):59-64.
[18] 刘高强, 李新鹏, 刘文钊, 等.一株高效降解血液蛋白的枯草芽孢杆菌NWMCC0137全基因组测序及分析[J].江苏农业学报, 2023, 39(7):1460-1471.LIU G Q, LI X P, LIU W Z, et al.Whole genome sequencing and analysis of Bacillus subtilis NWMCC0137, an efficient blood protein degrading strain[J].Jiangsu Journal of Agricultural Sciences, 2023, 39(7):1460-1471.
[19] 贾仲昕, 赵佳男, 季芳, 等.芽孢杆菌蛋白酶基因的比较基因组学分析[J].广东农业科学, 2022, 49(5):110-117.JIA Z X, ZHAO J N, JI F, et al.Comparative genomics analysis of protease genes in Bacillus sp[J].Guangdong Agricultural Sciences, 2022, 49(5):110-117.
[20] 李淑娴, 姚玉峰.细菌体内的蛋白质降解[J].微生物学报, 2015, 55(5):521-528.LI S X, YAO Y F.Proteolysis in bacteria-a review[J].Acta Microbiologica Sinica, 2015, 55(5):521-528.
[21] YOO Y, LEE H, KHIM J S, et al.Halomonas getboli sp.nov., a halotolerant bacteria isolated from a salt flat[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(12):005634.
[22] CHHETRI G, KIM I, SEO T.Devosia oryzisoli sp.nov., a novel moderately halotolerant bacterium isolated from the roots of rice plants and genome mining revealed the biosynthesis potential as plant growth promoter[J].Antonie Van Leeuwenhoek, 2023, 116(3):231-242.
[23] 万文结, 薛芷筠, 张泽文, 等. 一株产碱性蛋白酶菌株的筛选鉴定及酶学特性研究[J]. 微生物学报, 2017, 57(5):737-747.WAN W J, XUE J X, ZHANG Z W, et al. Isolation and identification of an alkaline protease producing strain and study on enzymatic properties[J]. Acta Microbiologica Sinica, 2017, 57(5):737-747.
[24] 孙佑赫, 周开艳, 熊智. 松毛虫肠道产蛋白酶菌株的筛选鉴定及培养条件研究[J]. 中国农学通报, 2012, 28(16):18-21.SUN Y H, ZHOU K Y, XIONG Z. Screening and identification of protease-producing bacterium from intestinal canal of dendrolimu and liquid culture studies[J]. Chinese Agricultural Science Bulletin, 2012, 28(16):18-21.