黄酒是一种起源于中国的典型酒精饮料,通常在开放的环境下以大米为原料,经过浸米、蒸煮、发酵、压榨过滤、杀菌和陈化生产而成[1]。浸米工序对黄酒品质有重要影响,促进了大米吸水、酵母发酵和风味的形成[2]。然而,浸米工序带来的安全风险(生物胺)限制了黄酒行业的发展。有研究发现[3],浸米工序是造成黄酒高生物胺含量的主要原因(产生量≥170 mg/L),对黄酒酿造过程中生物胺的贡献率高达71%以上。
生物胺是低分子有机含氮化合物,主要通过微生物的氨基酸脱羧酶代谢产生,广泛存在于高蛋白质的发酵食品中[4]。适量的生物胺可参与机体的生理调节(细胞增殖分化、免疫应答、营养补给等),但高含量的生物胺将会对细胞产生毒害作用,甚至诱发肿瘤、癌症[5-6]。值得注意的是,酒精摄入会抑制胺氧化酶的代谢调节[7],所以欧盟国家严格限制了酒类饮料中的生物胺含量,如葡萄酒中其含量最高不得超过10 mg/L[8]。此外,高浓度的生物胺对发酵食品的感官特性也有较大影响[9]。
已有研究开发了一种循环浸米工艺[3],使浸米工序的生物胺积累维持在较低水平,且对黄酒酿造无较大影响。但在米浆水连续循环过程中,工厂管道中残留的微生物被携带入下一次循环,其中产胺微生物在生态位的竞争中逐渐成为优势微生物,导致生物胺的持续积累,以至于循环浸米只能进行3次。因此,微生物入侵成为了循环浸米工业化应用亟需解决的问题。
在近期研究报道中[10],微生物之间的交互作用给予了群落更强的抗干扰性及自我恢复能力,在抵御微生物入侵中发挥着协同作用。一方面,越多样化的群落社区对入侵的抵抗力就越大,可以适应多种生态位环境,或通过更完全地消耗入侵者所需的营养资源来抵御入侵[11]。另一方面,微生物群落之间会通过代谢分子介导彼此的生理变化,为其他物种创造生态位[12]。例如,在开菲尔的营养限制条件下,酿酒酵母(Sacchromyces cerevisiae)通过释放葡萄糖酸、氨基酸等营养物质促进霍氏乳杆菌(Lactobacillus hordei)的生长[13]。总之,稳定的微生物群落较单菌更适应环境的变化及营养物质的供应和分配。但如何构建一个高效抑制产胺微生物入侵的稳定群落仍是一个难题。
在此背景下,为了抵御产胺微生物的入侵,实现循环浸米的多批次循环。本研究开发了一种合成微生物组循环浸米工艺,为循环浸米工艺的工业化应用提供了新策略。首先评价了浸米过程中微生物与生物胺的相关性,在了解浸米过程中生物胺形成机理和贡献的基础上,对米浆水中核心微生物进行构建,并开发了新的循环浸米工艺,为循环浸米工业化应用创造了可能性。
1 材料与方法
1.1 材料与试剂
植物乳植杆菌(Lactiplantibacillus plantarum) JN01由本课题组筛选所得,在浸米阶段接种JN01可有效抑制生物胺的积累,其在循环浸米中的应用已在前期研究中报道[3];工厂米浆水由绍兴某酒厂提供,生物胺含量(86.05±5.16) mg/L,总酸含量(10.23±0.12) g/L;FastDNA®Spin kit for Soil试剂盒,美国MP Biomedicals公司;所有化学品均购自国药集团。
MRS培养基购自青岛海博公司;生物胺检测培养基及糖化液参考文献[14]。
1.2 仪器与设备
AW200SG厌氧培养箱,英国伊莱泰科公司;T100聚合酶链式反应仪,美国生命技术公司;VT4-8C涡旋空压机,中国英格索兰公司;安捷伦 A1100高效液相色谱仪,美国安捷伦公司;钴源辐照装置,无锡爱邦辐照公司。
1.3 实验方法
1.3.1 微生物的人工选择
在50 mL灭菌离心管中加入20 mL无菌水和10 g辐照灭菌大米(25 kGy)。第一代每个离心管接种200 μL工厂米浆水,共30管,20 ℃培养48 h。在每个培育期结束时,将生物胺含量最低的3管培养物汇集起来,作为下一代微群落(n=30)的接种物,微生物的接种量为200 μL,并重复15代。选择每代中3组生物胺含量最低的组于-80 ℃保存,用于最终群落分析及微生物的分离,具体操作见图1。
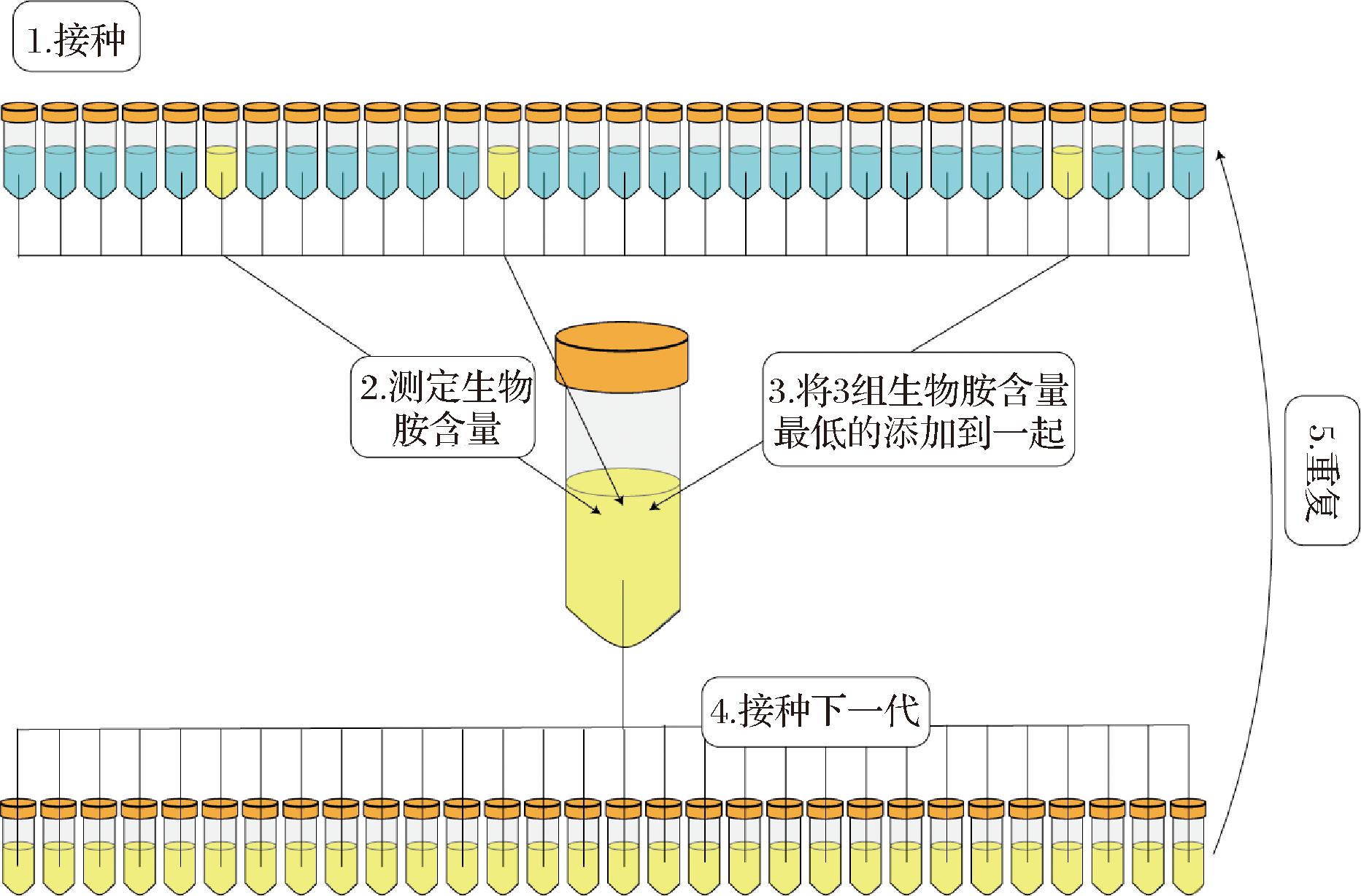
图1 用于人工选择微生物群落的方法
Fig.1 Method used for artificial selection of microbial communities
1.3.2 微生物组的构建
1.3.2.1 微生物的分离
将米浆水在MRS固体培养基上进行连续稀释,置于厌氧培养箱中,37 ℃培养48 h。将不同形态的单菌落在新的MRS固体培养基上划线纯化,将纯化后的菌株于MRS肉汤中,37 ℃培养24 h,以1%的接种量接种到生物胺检测培养基中,37 ℃培养72 h,观察颜色变化,显黄色为阴性,利用HPLC进一步验证阴性菌株的生物胺产生情况[3]。将不产生物胺的菌株于-81 ℃甘油保藏备用,并对菌株进行DNA分子鉴定[15]。
1.3.2.2 微生物的筛选
将菌株于MRS肉汤中,37 ℃培养24 h,以1%的接种量接种到糖化液中,37 ℃培养48 h后接种大米,接种量为9 mL/100 g大米。补加清水,使大米与水的料液比为1∶1.2(g∶mL),20 ℃培养72 h,测定总生物胺含量。同时以只接种L.plantarum JN01和不接种微生物的空白组为对照组。实验组及对照组都添加工厂米浆水,添加量9 mL/100g大米,以观察对米浆水中生物胺形成的抑制效果。
1.3.2.3 模拟工厂循环浸米
将菌株于MRS肉汤中,37 ℃培养24 h,以1%的接种量接种到糖化液中,37 ℃培养48 h,等比例接入大米中,总接种量为9 mL/100 g大米。补加清水,使大米与水的料液比为1∶1.2(g∶mL),室温静置72 h后收集所有米浆水。将收集的米浆水进行循环浸米,并补给一定量的清水到初始比例。每次循环持续2 d,一共循环4次。并且每次循环初期都补加工厂米浆水,补加量为1 mL/100 g大米,模拟工厂浸米环境以观察在循环浸米过程中对米浆水中生物胺形成的抑制效果。同时以只接种L.plantarum JN01和不接种微生物的空白组为对照组,按照上述条件进行循环浸米。测定循环过程中米浆水的总生物胺含量。
1.3.3 循环浸米工艺的优化
1.3.3.1 添加比例的优化
将活化后的菌液分别以1 mL/100 g大米~7 mL/100 g大米的接种量接入大米中,总接种量为9 mL/100 g大米。补加清水,使大米与水的料液比为1∶1.2(g∶mL),20 ℃静置72 h,收集米浆水测定生物胺含量。
1.3.3.2 循环浸米工艺条件的优化
取大米于烧杯中,将活化后的L. plantarum L-53、短促生乳杆菌(Levilactobacillus brevis)L-9、弯曲广泛乳杆菌(Latilactobacillus curvatus)MG-28分别按6 mL/100 g大米、2 mL/100 g大米、1 mL/100 g大米的比例进行接种。补给一定量的清水,使大米与水料液比为1∶1.2(g∶mL)。并每隔24 h补加清水,补加量为20 mL/100 g大米,总共补加3次,并用涡旋空压机加压混匀,室温发酵96 h结束初次浸米。收集全部米浆水,添加工厂米浆水以模拟工厂循环浸米中产胺微生物的入侵,添加量为10 mL/100 g大米。并每隔12 h补加清水,补加量为20 mL/100 g大米,总共补加3次,最后发酵12 h结束此次循环,后续循环浸米参照上述循环步骤。
1.3.4 细菌的群落结构分析
使用DNA试剂盒提取米浆水样品的基因组DNA。DNA纯化参照文献[16]所描述的方法。将提取的基因组DNA送至北京百迈客公司,对其16S rDNA的V3~V4高变区进行扩增并测序。数据质控后,OTUs序列采用Mothur法和SILVA132数据库进行注释。利用NCBI中的16S rDNA基因序列手工分析未分为物种的OTUs序列。
1.3.5 生物胺的测定
参考LIU等[3]所描述的方法测定生物胺含量。
1.4 数据处理
所有数据均重复进行3次,以平均值±标准差(SD)表示。采用单因素方差分析(ANOVA)计算P值,其余数据使用Origin 2022描绘。β-最近分类单元指数(β-NTI)和Bray-Curtis距离使用了R软件中的Picante包进行计算[17],并通过Origin 2022可视化。使用R软件进行数据间的相关性分析,XLSTAT 2016软件进行统计分析。
2 结果与分析
2.1 米浆水核心微生物的预测
一个群落可以在连续转移的选择过程中逐渐优化,人工选择一个可测量的理想性状被认为优于传统的富集实验,因为绕过了群落的瓶颈,减少了随机性[18]。此前,已有研究通过人工选择[19],成功实现了对高几丁质酶活微生物群落的定向选择。因此,本研究适当的改良了人工选择富集实验的步骤,如图1所示(详见1.3节),用于对米浆水中抑制生物胺形成的核心微生物群落进行定向富集。
2.1.1 人工选择富集实验中生物胺含量变化
在人工选择富集实验中,经过15次转移后,生物胺含量并没有预期持续下降(图2),总体呈先下降后上升趋势。第4代的平均生物胺含量最低可达31.97 mg/L,第14代的平均生物胺含量最高可达119.35 mg/L。研究发现,虽然接种了同一个微生物群落,但每代的生物胺含量并不固定,可能是传代过程中某些微生物的占比导致了生物胺的涨幅。于是对每代中生物胺含量最低的3组进行了群落结构的测定,以期结合生物胺产生情况分析造成生物胺升高或降低的核心微生物。
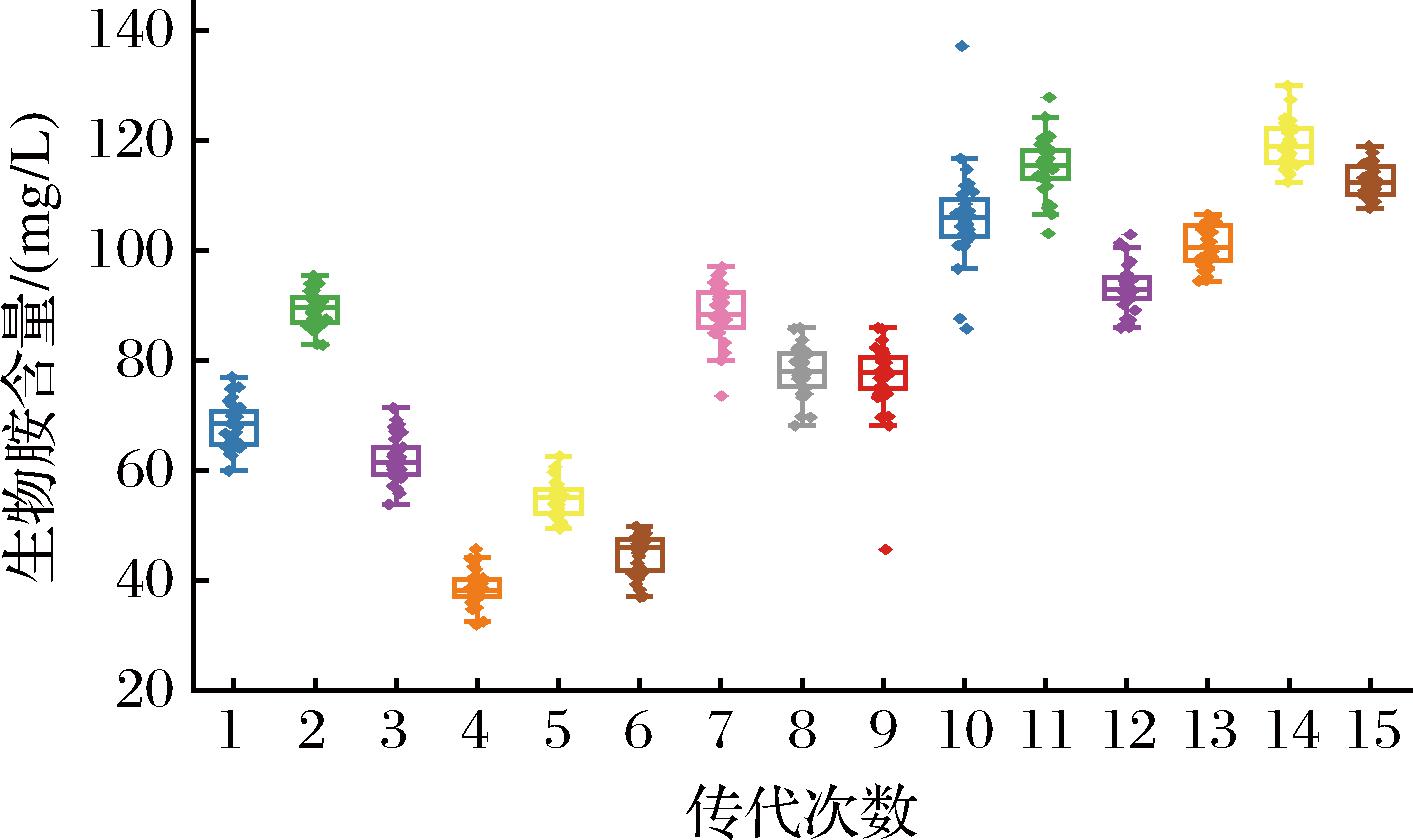
图2 人工富集筛选中生物胺含量变化(n=30)
Fig.2 Changes of biogenic amines in artificial selection experiment
2.1.2 人工选择富集实验中群落结构变化
对细菌的16S rRNA V3~V4区序列进行测序,探索在传代过程中的微生物的差异(图3)。在15代样品的细菌群落中,共鉴定出乳植物杆菌属(Lactiplantibacillus)、醋酸杆菌属(Acetobacter)、乳酪杆菌属(Lacticaseibacillus)、片球菌属(Pediococcus)、葡糖杆菌属(Gluconacetobacter)等4个属。在种水平上,L. plantarum、醋酸杆菌(Acetobacter sp.)、类干酪乳酪杆菌(Lacticaseibacillus paracasei)、产液葡糖酸醋杆菌(Gluconacetobacter liquefaciens)、有害片球菌(Pediococcus damnosus)、牛粪寡食乳杆菌(Paucilactobacillus vaccinostercus)、棒状腐败乳杆菌(Loigolactobacillus coryniformis)、L. brevis、巴氏醋杆菌(Acetobacter pasteurianus)、寡发酵寡食乳杆菌(Paucilactobacillus oligofermentans)、发酵粘液乳杆菌(Limosilactobacillus fermentum)、L. curvatus为主要优势微生物(以下出现的上述菌种省略属名)。L.plantarum、Acetobacter sp.、L.paracasei为最主要的微生物(总相对丰度>60%)。
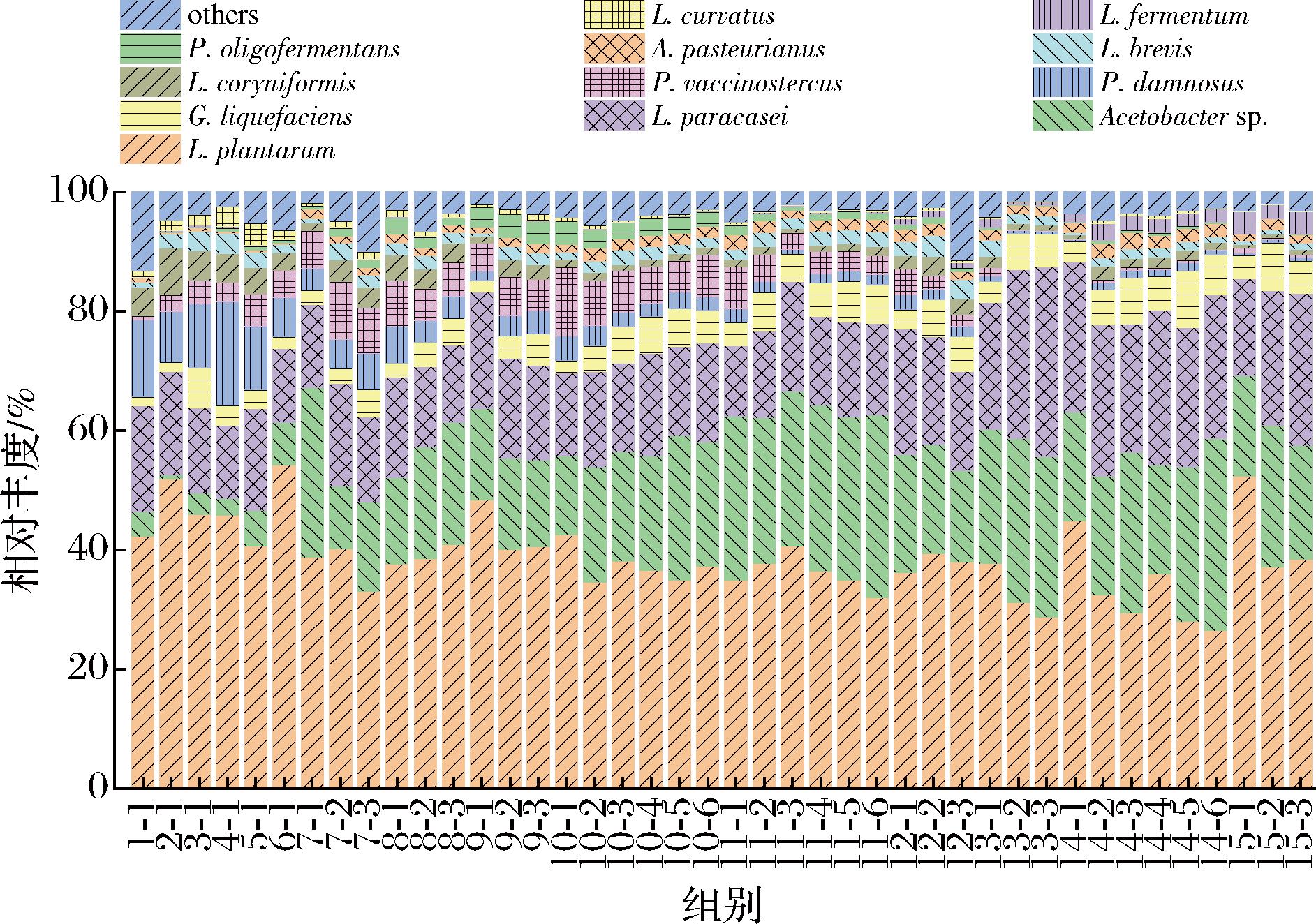
图3 人工富集筛选中细菌群落结构组成
Fig.3 Microbial community composition in artificial selection experiment
注:每代选取生物胺含量最低的3组,由于样品的缺失,1~6代只有1组样品;样品组别编号中“-”前代表传代次数,“-”后代表每代中生物胺含量最低的样品编号。
结果表明,乳酸菌在米浆水中占据绝对的优势地位,而不同代之间的物种差异及相互作用介导了生物胺的代谢。生态位的自然选择、营养物质的竞争和代谢物的产生广泛地影响着物种间的相互作用[20]。例如,异型乳酸菌在营养丰富及氧气充足的情况下生长代谢旺盛,而同型乳酸菌具有更强的抗酸胁迫能力[21]。而不同乳酸菌之间存在着交互作用,可以形成一个稳定高效的合成微生物群落,协同促进彼此的生长代谢活性[22]。推测大米在浸泡发酵过程存在多菌种共存和功能分工的特征。
2.1.3 人工富集实验中的群落构建分析
为了验证2.1.2节的推测,对富集筛选实验中不同传代次数样品的具体装配过程进行了量化,结果如图4所示。β-NTI可以用于量化微生物群落的随机系统发育和绝对系统发育的偏离程度。|β-NTI|>2说明微生物群落组装受确定性因素主导,其中当β-NTI>2时,样品微生物群落的遗传距离发散,受微生物的交互作用(变量选择、异质选择)影响;当β-NTI<-2时,样品微生物群落的遗传距离收敛,受环境选择(同质选择)主导。|β-NTI|<2说明微生物群落组装受随机过程影响,可以利用基于Bray-Curtis的Raup-Crick指数(RCbray)进一步对随机过程评估,可以分为同质扩散(|β-NTI|<2并且RCbray<-0.95)、未主导过程(|β-NTI|<2并且|RCbray|<0.95)、扩散限制(|β-NTI|<2并且RCbray>0.95)[17]。

a-β-NTI、生物胺含量;b-平均贡献率
图4 人工选择富集实验中细菌的群落构建
Fig.4 Bacteria community construction in artificial selection experiment
注:a图中每代数据的β-NTI值用小提琴图表示,小提琴宽窄代表样本点多少,生物胺含量为样本数据的平均值并以折线图呈现;b图为基于RCbray的群落组装过程。
随着传代次数的增加,微生物群落的构建逐渐由确定性过程主导,转变为由随机性过程主导。即以生物胺含量65 mg/L为界限,高生物胺含量组主要受到随机过程的影响,微生物群落结构由未主导过程(11.38%~60.16%)和扩散限制(13.82%~39.02%)主导。而低生物胺含量组,由异质选择(46.31%~97.56%)主导,受微生物的交互作用影响。因此,生物胺降低是微生物交互作用的结果,一定存在某几种微生物通过交互作用网络抑制了产胺微生物的代谢,抑制生物胺的产生。探究哪些微生物通过交互作用调控了生物胺的代谢,对于挖掘米浆水中的核心微生物组具有重要意义。
2.1.4 生物胺含量与微生物的相关性分析
为了探究优势微生物与生物胺代谢的相关性,利用Spearman相关性进一步分析人工选择富集实验中微生物的演替差异对生物胺的代谢影响(图5)。结果表明,L.plantarum、P.damnosus、L.curvatus、L.coryniformis、L.brevis、P.vaccinostercus与生物胺的含量呈负相关,虽然上述微生物促进了尸胺的产生,但样品中米浆水中尸胺浓度<1 mg/L,对于总生物胺的含量贡献较小;而Acetobacter sp.、A.pasteurianus、G.liquefaciens、L.paracasei、L.fermentum显著促进了腐胺、组胺、酪胺的形成(P<0.05),而腐胺和酪胺为米浆水中最主要的生物胺,对总生物胺含量的贡献可达90%以上。
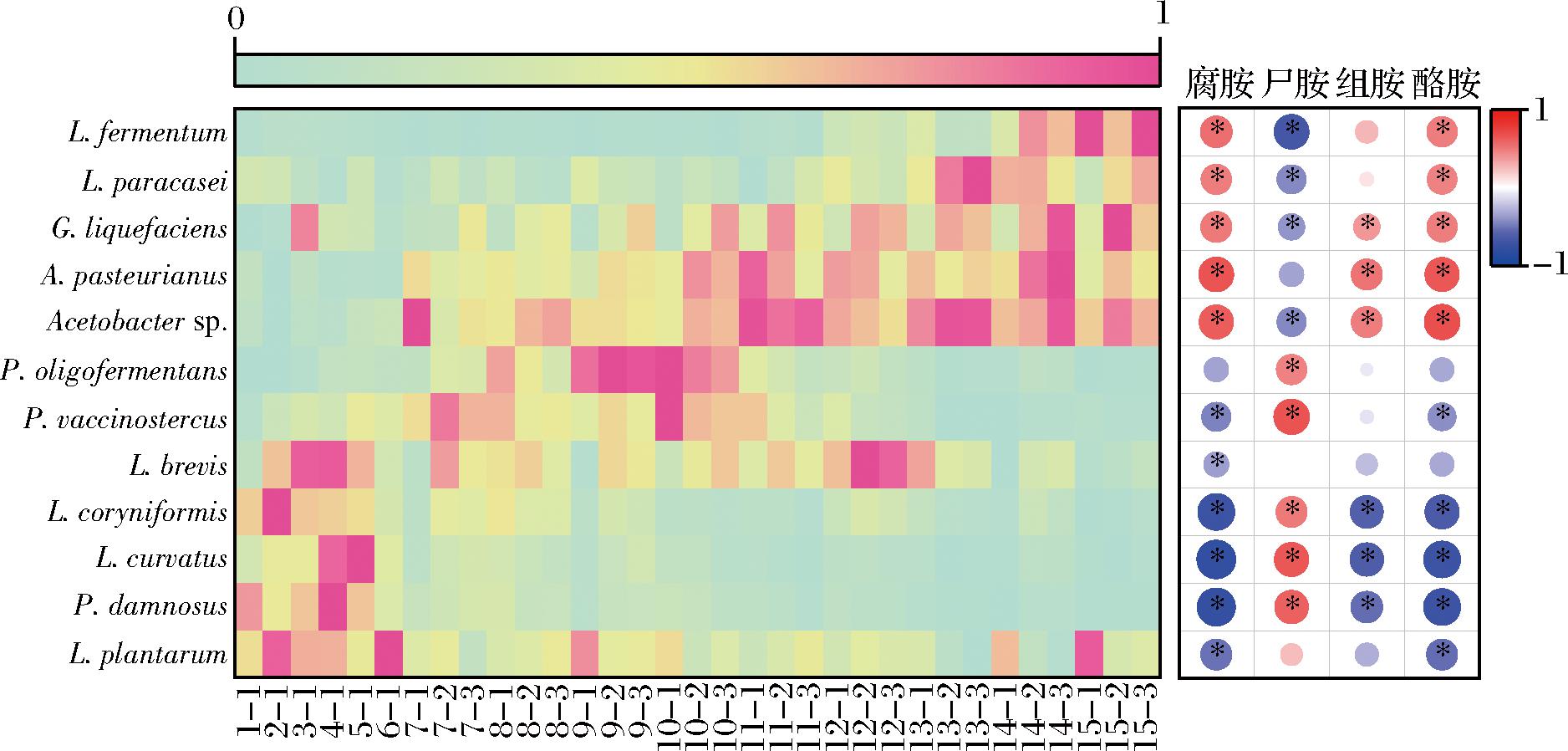
图5 人工选择富集实验中细菌与生物胺的相关性分析(P<0.05)
Fig.5 Correlation analysis between bacteria and biogenic amines in artificial selection experiment(P<0.05)
注:左侧为细菌在传代过程中的变化,颜色表示细菌的相对丰度归一化结果;右侧为细菌与生物胺的Spearman相关性,正相关为红色,负相关为蓝色。
对微生物群落结构的相对丰度归一化后绘制热图,发现微生物的演替规律与生物胺的变化具有显著相关。随着生物胺含量的增高,以L.plantarum、P.damnosus为代表的同型、兼性异型发酵乳酸菌的相对丰度逐渐下降,而以Acetobacter sp.为代表的异型发酵微生物相对丰度逐渐增加,这可能是Acetobacter sp.通过异型发酵产生CO2,促进了自身的扩散及生态位的竞争。测序结果表明,同型、兼型、异型发酵菌都可能参与大米浸泡过程,共同调解的生物胺的代谢。而现有的循环浸米工艺只接种了一种微生物,营养资源的竞争方式单一。对于不同资源水平的环境的适应能力较差,所以在循环的过程中逐渐走向衰败,诱导了生物胺含量的变化。
2.2 核心微生物组的构建
2.2.1 不产生物胺乳酸菌的分离
通过上述分析,推测米浆水中生物胺的降低是一个确定性过程(|β-NTI|>2),受微生物交互作用的影响,即不同属乳酸菌之间可能存在特定的相互作用模式,影响着物种的演替及生物胺的代谢。而基于Sperman秩相关性和冗沉分析(redundancy analysis,RDA),L.plantaurm、P.damnosus、L.curvatus、L.coryniformis、P.vaccinostercus、L.brevis是抑制生物胺产生的主要微生物。值得注意的是,L.plantaurm是抑制浸米过程中生物胺产生的最核心微生物,在米浆水中的相对丰度可达30%以上。结合在人工选择富集实验中第4代的生物胺含量最少的现象考虑,在第4代中以上述菌为目标进行微生物的分离纯化。利用显色法[23]和高效液相色谱对菌株产生物胺能力的进行了分析,经16S rRNA测序分析,全部为乳酸菌(表1)。但由于传统筛选的随机性或菌株不可培养性,经过大量筛选并没有得到不产生物胺的P.damnosus、P.vaccinostercus和L.coryniformis。
表1 筛选菌株及其生物胺代谢特性
Table 1 Strains screened and their biogenic amine metabolic properties
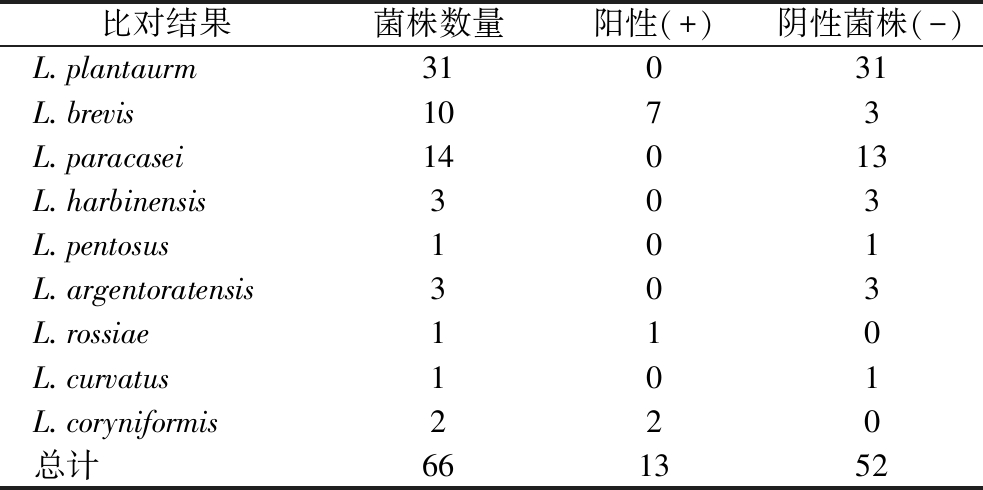
注:(+)/(-)代表产生/不产生物胺。
比对结果菌株数量阳性(+)阴性菌株(-)L.plantaurm31031L.brevis1073L.paracasei14013L.harbinensis303L.pentosus101L.argentoratensis303L.rossiae110L.curvatus101L.coryniformis220总计661352
2.2.2 核心微生物组的构建
对于乳酸菌群落的构建已有研究报道,罗意[24]构建了一个三元乳酸菌群落,并以L.brevis(具有氨基酸脱羧酶活力)作为入侵菌株研究群落结构的稳定,而L.brevis在传达过程中逐渐走向灭亡,表明三元乳酸菌群落具有较强的抗干扰性和恢复能力。可能原因是,三元乳酸菌群落中的微生物可通过负反馈作用促进/抑制彼此的生长,使群落结构更为稳定。同时,群落中物种的多种营养竞争策略(含同型、异型、兼型发酵乳酸菌),更能适应多变的生态位环境,提升了群落的稳定性。然而,以上研究都是在纯种培养体系中进行,循环浸米过程中产胺微生物的入侵是一个多菌种干扰的过程,三元乳酸菌群落对于复杂群落的干扰响应情况仍是未知。因此,基于2.1.4节的预测结果和筛选到的目标菌株(31株同型发酵的L.plantaurm、3株异型发酵的L.brevis、1株兼型发酵的L.curvatus)进行工厂浸米实验,以期筛选出每个属中高效抑制生物胺产生的微生物。然后,构建乳酸菌的三元相互作用群落,并评价其在循环浸米过程中对产胺微生物的抗入侵能力。
将上述35株目标菌株进行工厂浸米实验,研究其抑制生物胺产生的能力。浸泡48 h后对生物胺进行测定,其中,L.plantaurm L-53、L.brevis L-9、L.curvatus MG-28分别是各自属中抑制生物胺产生效果最好的菌株。而在总添加量相同时,L.plantaurm L-53、L.brevis L-9、L.curvatus MG-28等比例添加的群落较各自的单菌浸米组对于生物胺的抑制效果显著提升(图6),生物胺产生量仅为(38.54±2.40) mg/L。由此可知浸米过程中存在不同属乳酸菌的相互作用,促进了彼此的生长代谢,介导了生物胺的产生。此外,三元微生物组较于工厂应用菌株L.plantaurm JN01单菌浸米[(58.59±0.64) mg/L]生物胺降低了34.21%,主要表现为对于酪胺的抑制效果上升了42.33%。
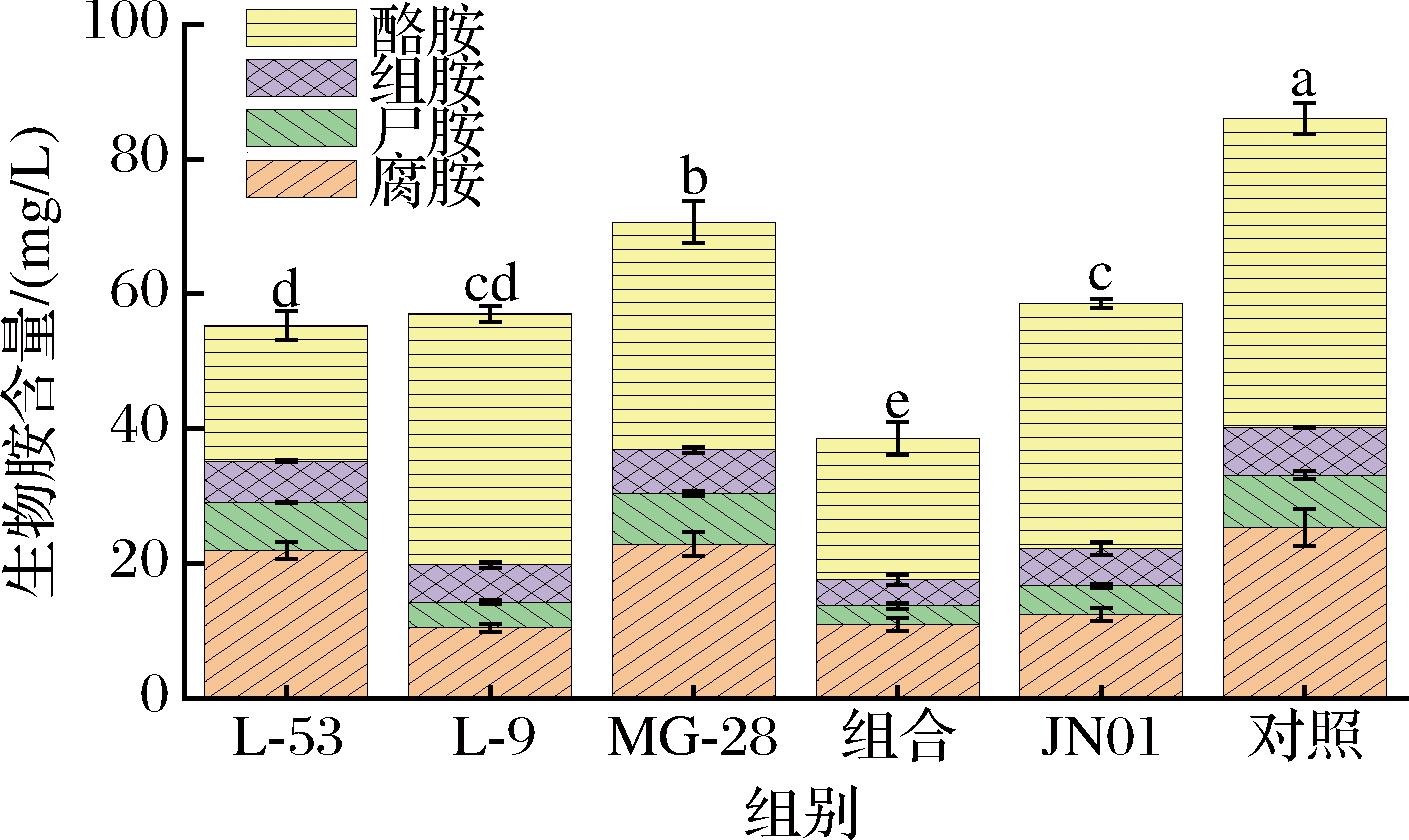
图6 模拟工厂环境浸米生物胺代谢情况
Fig.6 Simulating the biogenic amine metabolism in rice soaking systems within a factory environment
注:字母不同表示差异显著(P<0.05)(下同)。
2.2.3 模拟工厂环境循环浸米
将构建的群落应用到了循环浸米工艺中,并模拟了工厂循环浸米收集米浆水时管道残留的微生物入侵。在循环浸米过程中,生物胺含量呈上升趋势,而三元微生物组的上升速率明显低于对照组和L.plantaurm JN01组(图7)。三元微生物组在第4个循环后生物胺为(52.01±2.33) mg/L,较对照组降低了55.21%,且依然低于对照组的第1个循环[(57.94±1.84) mg/L],而L.plantaurm JN01组在第2个循环中已达到了(74.62±2.83) mg/L。对生物胺种类的研究发现,三元微生物组对于腐胺的抑制尤为明显,每次循环结束后生物胺的含量都低于5 mg/L,抑制效果最高达到了95.26%。
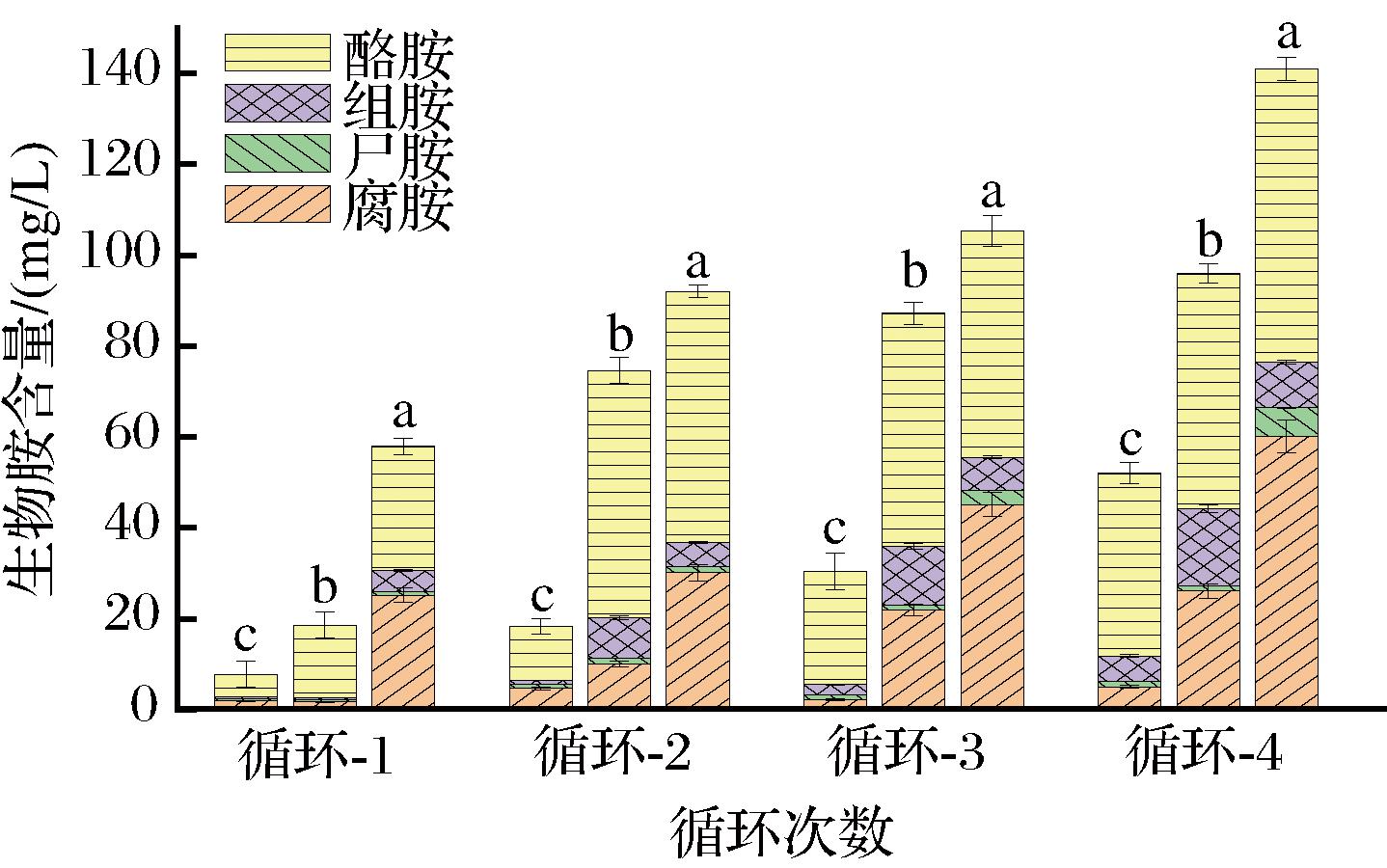
图7 模拟工厂循环浸米中生物胺代谢情况
Fig.7 Simulating the biogenic amine metabolism in cyclic soaking rice systems within a factory environment
注:每个循环中从左到右分别为三元微生物组、L.plantaurm JN01组、空白对照组。
2.3 循环浸米工艺的优化
2.3.1 添加比例的优化
乳酸菌构建的群落在循环浸米中的应用结果显示,三元乳酸菌群落对于抵御产胺微生物的入侵具有较好的效果,但随着循环次数的增加,生物胺含量依然缓慢增长。结合米浆水中各乳酸菌的相对丰度,推测是否三种微生物的相对比例影响着群落结构的稳定性。因此,本研究确定了一个总的添加比例9%[3],并优化了群落各微生物的相对比例,结果如图8所示。当L.plantaurm、L.brevis、L.curvatus以6∶2∶1添加时对生物胺的抑制效果最好,这也更接近于自然浸米系统中的群落构成,最终生物胺含量为(33.90±0.13) mg/L,相较于等比例添加[(46.10±1.39) mg/L]效果提升了26.46%。
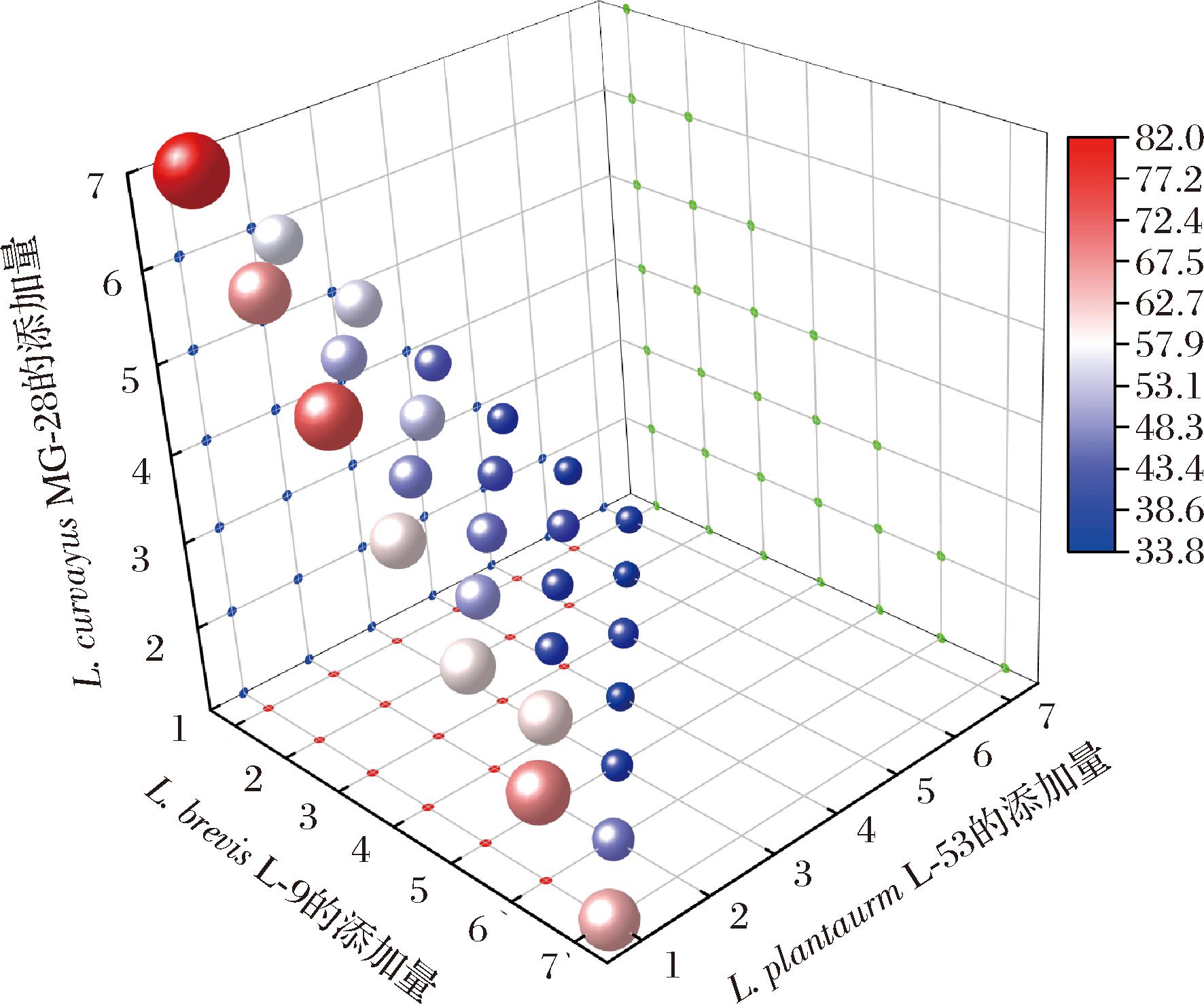
图8 不同添加比例对生物胺代谢的影响
Fig.8 Effect of different addition ratios on the metabolism of biogenic amines
注:颜色的变化及球体的大小代表着生物胺的高低。
2.3.2 工艺条件的优化
对添加比例优化后,三元乳酸菌群落的抗入侵能力有提升,但仍不能完全抑制生物胺的产生。已有研究发现[8],较高酸度可抑制生物胺的产生,这为循环浸米工艺的改良提供了思路。本研究将现有循环浸米工艺中在每次循环初期一次性补加清水改为在前一次循环的过程中分批次补加清水,并通过空气泵加压混匀以促进乳酸菌的充分发酵。按照上述步骤,可以使每次循环初期米浆水维持在高酸度水平,以抑制有害菌的代谢。利用改良后的循环浸米工艺进行了模拟工厂浸米。初次浸米72 h后总酸变化幅度较小,维持在11 g/L左右(图9)。且高初始酸度并没有影响乳酸菌的代谢,每次循环浸米结束后酸度都维持在11~12 g/L。
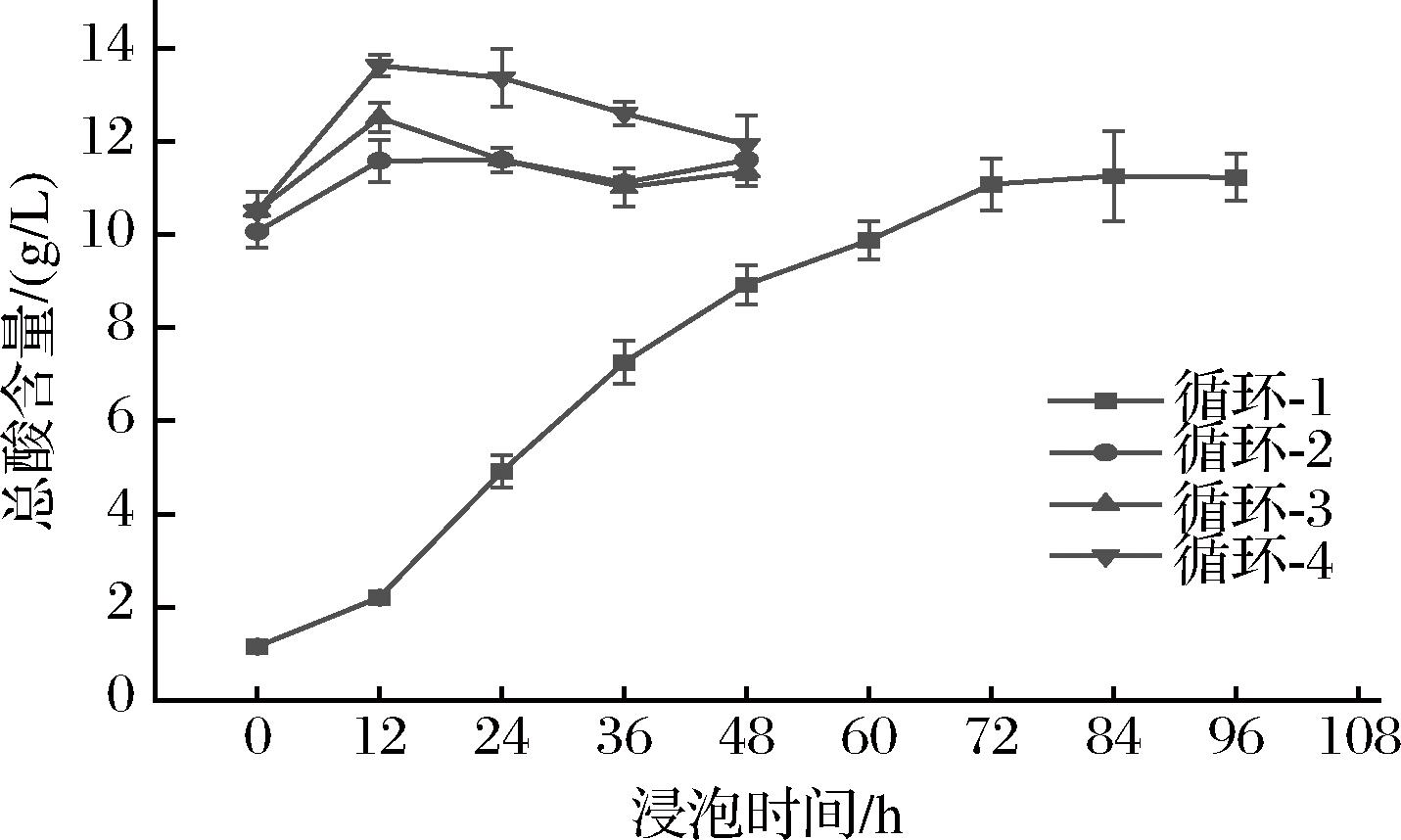
图9 改良循环浸米工艺的酸度变化
Fig.9 Changes in total acid during the improved cyclic soaking rice process
对每次循环结束后的生物胺进行了测定,发现生物胺依然能保持低水平(10 mg/L)。而L.plantaurm JN01单菌浸米组在循环过程中,生物胺含量持续增高,在第4个循环后生物胺含量达到了(62.03±3.02) mg/L。值得注意的是,工厂米浆水本身富含大量生物胺[(86.05±5.76) mg/L](图10)。使用改良后的循环浸米工艺,不但能抵御工厂米浆水中产胺微生物的入侵,还能降解携带入米浆水体系中的生物胺,实现生物胺的“零增长”。
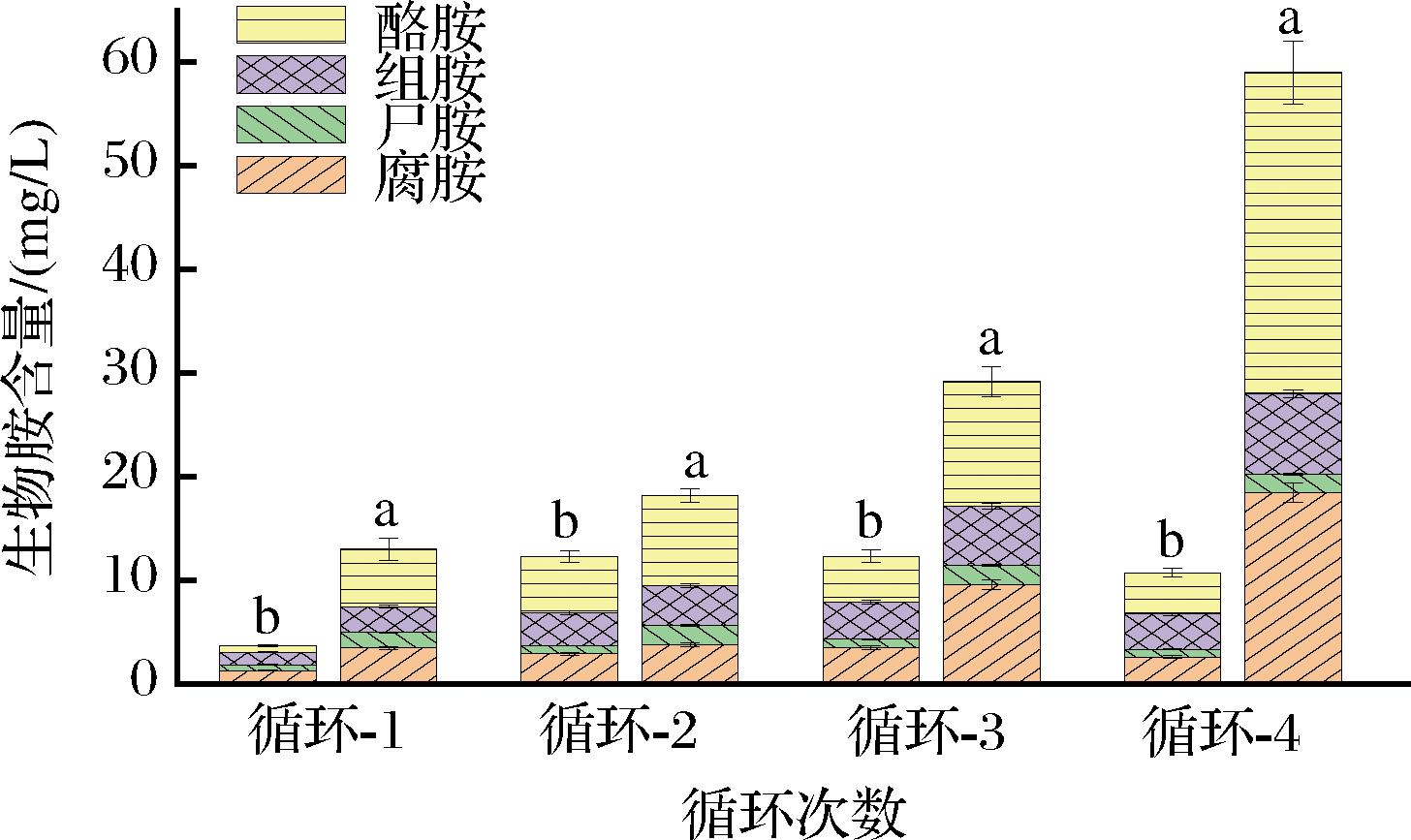
图10 改良循环浸米工艺的生物胺变化
Fig.10 Changes in biogenic amines during the improved cyclic soaking rice process
注:左侧为三元微生物组,右侧为L.plantaurm JN01组。
对每次循环结束后的细菌群落结构进行了测定(图11),研究发现不同循环米浆水中主要以L.plantaurm、L.brevis、L.curvatus为主,平均相对丰度占比可达90%以上,以L.plantaurm为最优势物种,占比>67.9%。但随着循环次数的增加,L.plantaurm的相对丰度逐渐下降,而L.brevis的相对丰度逐渐上升,到第4次循环丰度可达31.75%,结合生物胺产生情况,可以初步确定为接入的L.brevis L-9,而非工厂米浆水中的L.brevis。这也侧面反映了现有单菌循环浸米工艺的局限性,即循环浸米过程伴随着营养资源及生态位的变化,随着循环次数的增加,异型发酵的L.brevis的生长优势逐渐凸现。因此,本研究推测三元乳酸菌群落展现出高抗入侵能力,是利用多元化的营养竞争方式(L.plantaurm为同型、L.brevis为异型、L.curvatus为兼型异型),对限制性营养物质的进行垄断,通过更全面的消耗营养物质来限制入侵物种。
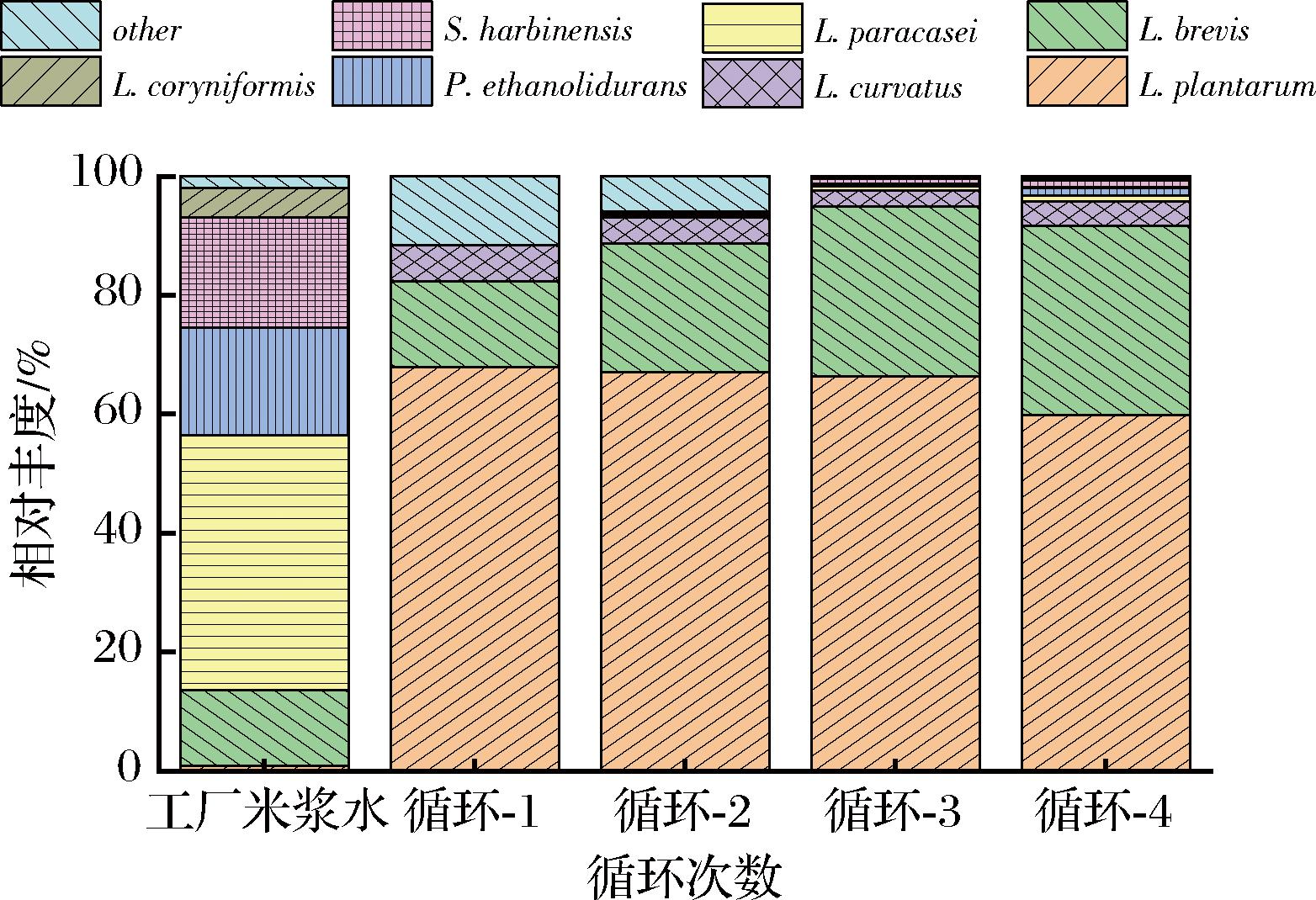
图11 改良循环浸米工艺的细菌群落结构变化
Fig.11 Structural changes in the bacterial community during the improved cyclic soaking rice process
3 结论与讨论
在人工选择富集实验中对微生物群落组装进行了分析,低生物胺含量组主要由异质选择主导,说明米浆水中生物胺的降低是微生物交互作用的结果。通过Sperman秩相关性和RDA分析,发现L.plantaurm、P.damnosus、L.curvatus、L.coryniformis、P.vaccinostercus、L.brevis与米浆水中生物胺的降低显著相关。基于预测结果,对米浆水中的核心微生物群落进行了筛选和构建。在模拟工厂浸米实验中,对生物胺形成的抑制效果达到了55.21%。4次循环浸米生物胺含量依然低于对照组1次循环[(57.94±1.84) mg/L],相较于L.plantaurm JN01效果提升了39.58%。随后优化了添加比例,发现L.plantaurm、L.brevis、L.curvatus以6∶2∶1添加时对生物胺的抑制效果最好,相较于等比例添加效果提升了26.46%。基于酸度对产胺微生物的影响,构建了一种新的循环浸米工艺。新工艺可以完全抑制产胺微生物的代谢,实现了生物胺的“零增长”,形成了以L.plantaurm、L.brevis、L.curvatus占主导的微生物体系,有效抑制了在循环过程中产胺微生物的入侵。新的循环浸米工艺技术解决了微生物入侵问题,为循环浸米工业化应用提供了有效思路。并且多菌种共酵的应用使强化接种工艺更加贴合传统自然发酵,在较小影响黄酒酿造工艺的同时提高了安全性,也为其他相关传统发酵食品的严格控制提供参考。
[1] WANG J, YU Y G, GAO X L, et al.Succession patterns of aroma components during brewing process of broomcorn millet (Panicum miliaceum L.) Huangjiu[J].Food Research International, 2022, 154:110982.
[2] YANG Y J, LI S, XIA Y J, et al.Effects of different lactic acid bacteria on the characteristic flavor profiles of Chinese rice wine[J].Journal of the Science of Food and Agriculture, 2024, 104(1):421-430.
[3] LIU S P, SUN H L, LIU C X, et al.Reducing biogenic amine in seriflux and Huangjiu by recycling of seriflux inoculated with Lactobacillus plantarum JN01[J].Food Research International, 2021, 150:110793.
[4] GARDINI F, ÖZOGUL Y, SUZZI G, et al.Technological factors affecting biogenic amine content in foods:A review[J].Frontiers in Microbiology, 2016, 7:1218.
[5] W JCIK W,
JCIK W,  UKASIEWICZ M, PUPPEL K.Biogenic amines:Formation, action and toxicity-a review[J].Journal of the Science of Food and Agriculture, 2021, 101(7):2634-2640.
UKASIEWICZ M, PUPPEL K.Biogenic amines:Formation, action and toxicity-a review[J].Journal of the Science of Food and Agriculture, 2021, 101(7):2634-2640.
[6] BENKERROUM N.Biogenic amines in dairy products:Origin, incidence, and control means[J].Comprehensive Reviews in Food Science and Food Safety, 2016, 15(4):801-826.
[7] GARC A-RUIZ A, GONZ
A-RUIZ A, GONZ LEZ-ROMPINELLI E M, BARTOLOMé B, et al.Potential of wine-associated lactic acid bacteria to degrade biogenic amines[J].International Journal of Food Microbiology, 2011, 148(2):115-120.
LEZ-ROMPINELLI E M, BARTOLOMé B, et al.Potential of wine-associated lactic acid bacteria to degrade biogenic amines[J].International Journal of Food Microbiology, 2011, 148(2):115-120.
[8] 刘彩霞. 乳杆菌酸化循环浸米工艺降低黄酒生物胺的研究[D].无锡:江南大学, 2020.LIU C X. Study on reducing biogenic amines in yellow rice wine by acidizing and circulating soaking rice by Lactobacillus[D]. Wuxi: Jiangnan University, 2020.
[9] PALOMINO-VASCO M, RODR GUEZ-C
GUEZ-C CERES M I, MORA-DIEZ N, et al.Biogenic amines profile in red wines regarding aging and storage conditions[J].Journal of Food Composition and Analysis, 2019, 83:103295.
CERES M I, MORA-DIEZ N, et al.Biogenic amines profile in red wines regarding aging and storage conditions[J].Journal of Food Composition and Analysis, 2019, 83:103295.
[10] XIA X L, LUO Y, ZHANG Q W, et al.Mixed starter culture regulates biogenic amines formation via decarboxylation and transamination during Chinese rice wine fermentation[J].Journal of Agricultural and Food Chemistry, 2018, 66(25):6348-6356.
[11] GRALKA M, SZABO R, STOCKER R, et al.Trophic interactions and the drivers of microbial community assembly[J].Current Biology:CB, 2020, 30(19):R1176-R1188.
[12] PHAM N P, LANDAUD S, LIEBEN P, et al.Transcription profiling reveals cooperative metabolic interactions in a microbial cheese-ripening community composed of Debaryomyces hansenii, Brevibacterium aurantiacum, and Hafnia alvei[J].Frontiers in Microbiology, 2019, 10:1901.
[13] XU D, BEHR J, GEIßLER A J, et al.Label-free quantitative proteomic analysis reveals the lifestyle of Lactobacillus hordei in the presence of Sacchromyces cerevisiae[J].International Journal of Food Microbiology, 2019, 294:18-26.
[14] 刘彩霞, 刘双平, 徐岳正, 等.乳酸菌强化技术降低黄酒浸米过程生物胺的积累[J].中国食品学报, 2021, 21(5):166-173.LIU C X, LIU S P, XU Y Z, et al.Reducing biogenic amines accumulation in rice socking process of Huangjiu based on enhancing technique of Lactobacillus[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(5):166-173.
[15] LIU S P, CHEN Q L, ZOU H J, et al.A metagenomic analysis of the relationship between microorganisms and flavor development in Shaoxing mechanized Huangjiu fermentation mashes[J].International Journal of Food Microbiology, 2019, 303:9-18.
[16] TANASE A M, MEREUTA I, CHICIUDEAN I, et al.Comparison of total DNA extraction methods for microbial community form polluted soil[J].Agriculture and Agricultural Science Procedia, 2015, 6:616-622.
[17] CHEN W D, REN K X, ISABWE A, et al.Stochastic processes shape microeukaryotic community assembly in a subtropical river across wet and dry seasons[J].Microbiome, 2019, 7(1):138.
[18] BLOUIN M, KARIMI B, MATHIEU J, et al.Levels and limits in artificial selection of communities[J].Ecology Letters, 2015, 18(10):1040-1048.
[19] WRIGHT R J, GIBSON M I, CHRISTIE-OLEZA J A.Understanding microbial community dynamics to improve optimal microbiome selection[J].Microbiome, 2019, 7(1):85.
[20] ROSENTHAL A Z, QI Y T, HORMOZ S, et al.Metabolic interactions between dynamic bacterial subpopulations[J].eLife, 2018, 7:e33099.
[21] ALFONZO A, URSO V, CORONA O, et al.Development of a method for the direct fermentation of semolina by selected sourdough lactic acid bacteria[J].International Journal of Food Microbiology, 2016, 239:65-78.
[22] SETTACHAIMONGKON S, ROBERT NOUT M J, ANTUNES FERNANDES E C, et al.Influence of different proteolytic strains of Streptococcus thermophilus in co-culture with Lactobacillus delbrueckii subsp.bulgaricus on the metabolite profile of set-yoghurt[J].International Journal of Food Microbiology, 2014, 177:29-36.
[23] JIN Y H, LEE J H, PARK Y K, et al.The occurrence of biogenic amines and determination of biogenic amine-producing lactic acid bacteria in Kkakdugi and Chonggak kimchi[J].Foods, 2019, 8(2):73.
[24] 罗意. 黄酒发酵核心微生物群落组装及生物胺调控机制[D].无锡:江南大学, 2022.LUO Y. Core microbial community assembly and biogenic amine regulation mechanism in yellow rice wine fermentation[D]. Wuxi: Jiangnan University, 2022.