溶藻弧菌是革兰氏阴性嗜盐菌,生长的适宜温度在17~35 ℃,对鱼、虾等水产养殖动物是条件致病菌,在水产动物免疫能力下降时较易受其感染[1],消费者若食用未煮熟的、被溶藻弧菌污染的水产品时也可能会发生食物中毒。HAIFA-HARYANI等[2]对马来半岛的养殖虾进行了采集检测,检出了溶藻弧菌;申艳琴等[3]对兰州市螺类水产品进行致病性弧菌检测,溶藻弧菌占比达30.29%。以上研究表明,市售水产品存在被溶藻弧菌污染的风险,有必要对水产品进行溶藻弧菌检测鉴定,有助于了解水产品中溶藻弧菌的污染程度,提示消费者应采取安全的加工熟制方式,减少或防止食品安全事件的发生。
目前研究发现从水产品或环境中分离的溶藻弧菌对万古霉素[4]、红霉素[4-5]、四环素[6]等抗生素具有耐药性。耐药基因的存在是细菌产生抗生素耐药的内在原因之一,胡梦华等[7]从分离到的多重耐药型溶藻弧菌中检测出链霉素耐药基因strA和strB、氯霉素耐药基因floR和氟喹诺酮类耐药基因qnrVC5,相应的菌株表现出了对链霉素、氯霉素和氟喹诺酮类药物环丙沙星等具有耐药性,但研究中还发现该溶藻弧菌对红霉素耐药却未检出相关耐药基因。通过检测耐药基因,从基因层面预测溶藻弧菌的耐药性及潜在的耐药可能,对水产养殖和临床医疗方面抗菌药物的选择具有重要的意义。
综上,本研究从市售水产品中检测分离到了14株溶藻弧菌对8类22种常见抗生素的耐药性以及8类21个耐药基因和1类整合子基因,分析了分离株的多重耐药性以及耐药基因,阐明溶藻弧菌分离株对抗生素耐药表型与耐药基因间的关联性,为防治食源性疾病、保障食品安全提供参考。
1 材料与方法
1.1 实验材料
1.1.1 菌株来源
2022年3~7月份,从杭州市3家农贸市场,按照随机采样原则,共采集105份水产品样品[具体样品信息如电子版增强出版附表1所示(https://doi.org/10.13995/j.cnki.11-1802/ts.039216)],采集后置于4 ℃保温箱中运回实验室进行菌株分离鉴定,从样品中共分离得到14株溶藻弧菌,如表1所示。
表1 十四株溶藻弧菌的来源
Table 1 Sources of 14 strains of V.alginolyticus

阳性菌株编号标签取样生物取样时间取样地点11-3中华对虾(Fenneropenaeus chinensis)2022-03-1321-8中华对虾(Fenneropenaeus chinensis)2022-05-2231-10中华对虾(Fenneropenaeus chinensis)2022-05-2242-5大黄鱼(Pseudosciaena crocea)2022-07-0652-7中华对虾(Fenneropenaeus chinensis)2022-07-0662-9花蛤(Venerupis variegata)2022-07-0672-10三文鱼(Oncorhynchus)2022-07-0682-11血蛤(Tegillarca granosa)2022-07-0693-1南美白对虾(Penaeus vannamei)2022-07-12103-8八爪鱼(Octopus vulgaris)2022-07-12113-10银鲳(Pampus argenteus)2022-07-12123-14刀额新对虾(Metapenaeus ensis)2022-07-12133-16刀额新对虾(Metapenaeus ensis)2022-07-12143-24中国枪乌贼(Loligo chinensis)2022-07-12浙江省杭州市(118°21′~120°30′ E, 29°11′~30°33′ N)
1.1.2 培养基与试剂
硫代硫酸盐-柠檬酸盐-胆盐-蔗糖(thiosulfate-citrate-bilesalts-sucroseagarmedium, TCBS)弧菌选择性培养基、胰蛋白胨大豆肉汤(tryptone soy broth, TSB)、30 g/L氯化钠胰蛋白胨大豆琼脂(NaCl tryptone soy agar, TSA)培养基、30 g/L氯化钠碱性蛋白胨水(NaCl alkaline peptone water, APW)、药敏纸片,杭州微生物试剂有限公司;DL 2000 Plus DNA Maker、Green Taq Mix,南京诺唯赞生物科技股份有限公司;Super Red核酸染料(10 000×H2O)、Biosharp琼脂糖,兰杰柯科技有限公司;细菌基因组DNA提取试剂盒,天根生化科技有限公司;引物和基因测序,杭州有康生物技术有限公司。
1.1.3 仪器与设备
LDZX-50KBS立式压力蒸汽灭菌锅,上海申安医疗器械厂;SIGMA3-30K高速冷冻离心机,德国Sigma公司;PowerPac核酸电泳仪,美国BIO-RAD公司;TC-XP基因扩增仪,上海博日科技有限公司;JS-680D全自动凝胶成像分析仪,上海培清科技有限公司;PX-150B-Z生化培养箱,上海博讯公司;SPECTRAMAX ID3多功能酶标仪,美国Molecular Devices公司;微量移液器,德国Eppendorf公司;ME204E电子天平,瑞士METTLER TOLEDO公司。
1.2 实验方法
1.2.1 药敏试验
以大肠杆菌ATCC 25922为质控菌,按照美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSI)推荐的药敏纸片扩散法(Kirby-Bauer氏法,K-B法)及CLSI M100 2021版判定标准测定菌株对22种抗生素耐药情况[8]。采用药敏纸片法,所有分离株经培养后稀释至0.5个麦氏浊度(OD625nm=0.08~0.1,细菌近似浓度为1.5×108 CFU/mL),各吸取100 μL分别均匀涂布于M-H琼脂平板上,在室温下静置2 min至菌液完全被吸收。用无菌镊子轻轻夹取药敏纸片分别紧密贴合于含菌M-H琼脂平板中,药敏纸片和平皿边缘的距离>15 mm,药敏纸片中心之间的距离>24 mm,将平板倒置于37 ℃恒温生化培养箱中培养24 h。待培养完成后,用游标卡尺测定抑菌圈直径(mm),最终结果以耐药(R)、中介(I)、敏感(S)表示,与抑菌圈直径对应关系见附表2,对3类及三类以上抗生素表现为耐药的菌株可判定为多重耐药菌株。
表2 不同分离株药敏试验结果
Table 2 Results of antibiotic sensitive tests of different isolated strains

抗菌药物抑菌圈大小判断标准/mm菌株编号敏感(S)中介(I)耐药(R)1234567891011121314β-内酰胺类(β-lactamides)头孢噻肟(cefotaxime,CTX)<2323-25>25SRRSSSSSRISRRI头孢他啶(ceftazidime,CAZ)<1818-20>20SIISSSSRIISIIS头孢吡肟(cefepime,FEP)<1919-24>24SIISSSSSIISIII头孢唑林(cefazolin,KZ)<2020-22>22SRRRRRRRRRRRRR哌拉西林/他唑巴坦(piperacillin/tazobactam,PTZ)<1818-20>20SSSSSIISSSSISS氨苄西林(ampicillin,AMP)<1414-16>16RRRRSRRRRRRRRR阿莫西林(amoxicillin,AML)<1414-17>17RRRRRRRRRRRRRR青霉素(penicillin,PEN)<2020-27>27RRRRRRRRRRRRRR美洛西林(mezlocillin,MEZ)<1818-20>20SIRSSSIISSIIIS美罗培南(meropenem,MEM)<1515-17>17SSSSSSSSSSSSSS氨曲南(amtreonam,AZT)<1818-20>20SIRRSIISIIISRI氨基糖苷类(aminoglycosides)卡那霉素(kanamycin,K)<1414-17>17SRIRSIIRIRIIRI阿米卡星(amikacin,AK)<1515-16>16SRRRSIRIRRIIRS庆大霉素(gentamicin,GM)<1313-14>14SIISSSSSSISSII大环内酯类(macrolides)阿奇霉素(azithromycin,AZM)<1212-13>13SSSSSISSSSSSSS四环素类(tetracyclines)四环素(tetracycline,TE)<1212-14>14SSSSSRSSISSSSS氯霉素类(chloramphenicol)氯霉素(chloramphenicol,CHL)<1313-17>17SSSSSISSSSSSSS氟苯尼考(florfenicol,FFC)<1313-17>17SSSSSRSSSSSSSS磺胺类(sulfonamides)复方新诺明(trimethoprim/sulfamethoxazole,SXT)<1111-15>15SISSSRSSSSSSSS喹诺酮类(quinolones)环丙沙星(ciprofloxacin,CIP)<1616-20>20SISSSRSSSSSSII诺氟沙星(norfloxacin,NOR)<1313-16>16SSSSSRSSSSSIIS利福霉素类(rifamycins)利福平(rifampicin,RD)<1717-19>19SRIISRSSIISSIR耐药合计/种 388731056664585类别统计/类 13221622221122
注:表中R为耐药,I为中介,S为敏感。
多重抗生素耐药性指数(multiple antimicrobial resistance index, MARI)采用Krumperman[9]的方法计算。MARI大于0.2通常表示受试细菌来源于经常使用抗生素的高风险污染地区,可能会对人体健康造成威胁[8]。其计算如公式(1)所示:
(1)
式中:M,MARI;a,某个分离株对抗生素有耐药性的抗生素数量;b,同一分离株测试过的总抗生素数量。
1.2.2 耐药基因定性分析
将溶藻弧菌菌液12 000 r/min离心2 min,弃上清液(细胞培养液),根据细菌基因组DNA提取试剂盒提取DNA并放置于冰上备用。采用PCR扩增法检测溶藻弧菌中存在的耐药基因,引物序列参考文献[10-22],由杭州有康生物技术有限公司合成,信息如附表3所示。PCR扩增体系(25 μL):包括ddH2O 10.5 μL,Green Taq Mix 12.5 μL,上下游引物(10 μmol/L)各0.5 μL,DNA模板1 μL。PCR反应条件:95 ℃预变性3 min,95 ℃变性15 s,退火温度如附表3所示,时长1 min,72 ℃延伸1 min,重复35个循环;最后72 ℃延伸5 min。随后,取PCR产物5 μL进行1.5%琼脂糖凝胶(含Gel Red核酸染料)电泳,电压120 V,时长30 min,完成后于全自动凝胶成像分析仪下检测结果,并将目的产物送杭州有康生物技术有限公司测序,结果经过Blast与NCBI数据库进行比对分析(http://www.ncbi.nlm.nih.gov/BLAST)。
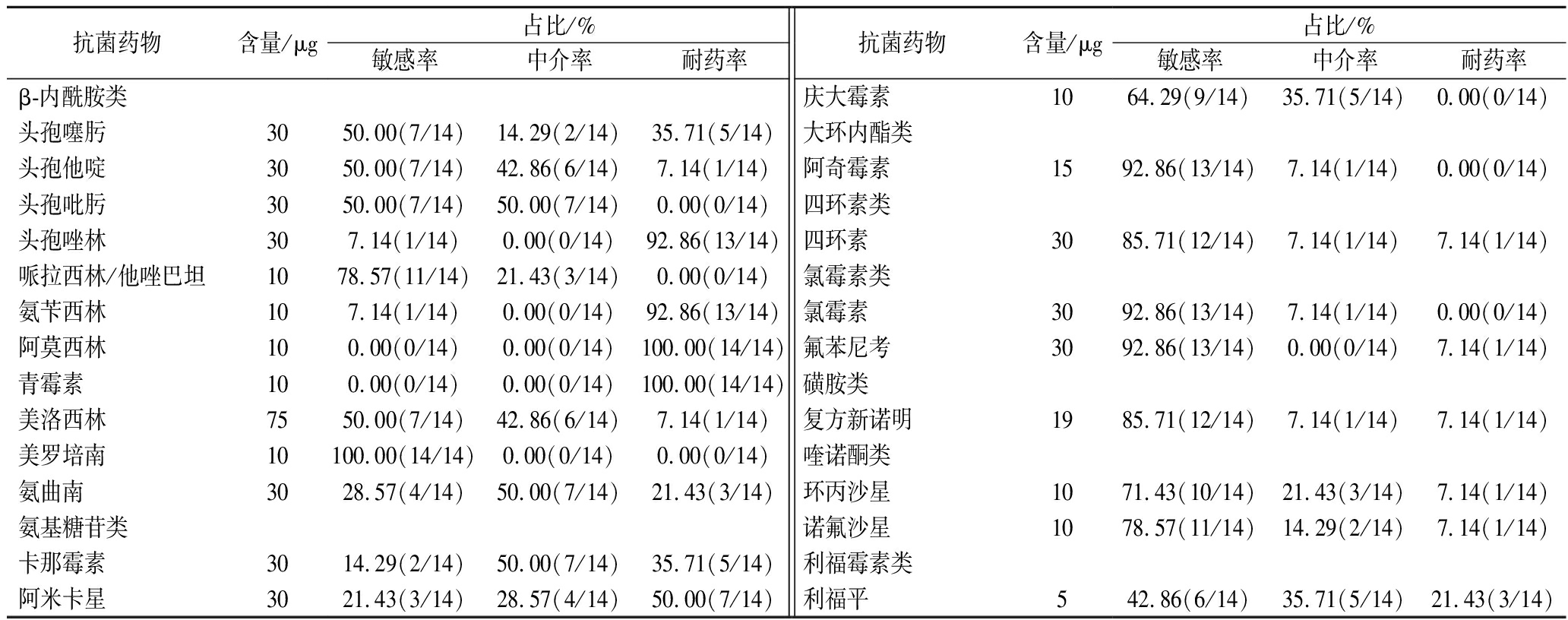
表3 溶藻弧菌分离株对22种抗菌药物敏感试验的结果
Table 3 Sensitivity test results of 14 strains of V.alginolyticus to 22 antibacterial agents
抗菌药物含量/μg占比/%敏感率中介率耐药率抗菌药物含量/μg占比/%敏感率中介率耐药率β-内酰胺类头孢噻肟3050.00(7/14)14.29(2/14)35.71(5/14)头孢他啶3050.00(7/14)42.86(6/14)7.14(1/14)头孢吡肟3050.00(7/14)50.00(7/14)0.00(0/14)头孢唑林307.14(1/14)0.00(0/14)92.86(13/14)哌拉西林/他唑巴坦1078.57(11/14)21.43(3/14)0.00(0/14)氨苄西林107.14(1/14)0.00(0/14)92.86(13/14)阿莫西林100.00(0/14)0.00(0/14)100.00(14/14)青霉素100.00(0/14)0.00(0/14)100.00(14/14)美洛西林7550.00(7/14)42.86(6/14)7.14(1/14)美罗培南10100.00(14/14)0.00(0/14)0.00(0/14)氨曲南3028.57(4/14)50.00(7/14)21.43(3/14)氨基糖苷类卡那霉素3014.29(2/14)50.00(7/14)35.71(5/14)阿米卡星3021.43(3/14)28.57(4/14)50.00(7/14)庆大霉素1064.29(9/14)35.71(5/14)0.00(0/14)大环内酯类阿奇霉素1592.86(13/14)7.14(1/14)0.00(0/14)四环素类四环素3085.71(12/14)7.14(1/14)7.14(1/14)氯霉素类氯霉素3092.86(13/14)7.14(1/14)0.00(0/14)氟苯尼考3092.86(13/14)0.00(0/14)7.14(1/14)磺胺类复方新诺明1985.71(12/14)7.14(1/14)7.14(1/14)喹诺酮类环丙沙星1071.43(10/14)21.43(3/14)7.14(1/14)诺氟沙星1078.57(11/14)14.29(2/14)7.14(1/14)利福霉素类利福平542.86(6/14)35.71(5/14)21.43(3/14)
1.2.3 统计学分析
药敏试验平行3次,使用 SPSS 18.0 软件进行统计学分析,以P<0.05为差异有统计学意义。
2 结果与分析
2.1 溶藻弧菌分离株的耐药表型分析
从杭州市105份市售水产品样品中共分离得到14株溶藻弧菌,检出率为13.3%,表2为溶藻弧菌药敏实验结果,发现8株溶藻弧菌对2类抗生素耐药,其中7株表现为对β-内酰胺类和氨基糖苷类耐药,14号分离株表现为β-内酰胺类和利福霉素类耐药。有2株溶藻弧菌对3类及以上抗生素耐药,2号分离株对3类抗生素(β-内酰胺类、氨基糖苷类和利福霉素类)具有耐药性,6号分离株对6类抗生素(β-内酰胺类、四环素类、氯霉素类、磺胺类、喹诺酮类和利福霉素类)耐药,分别来源于中华对虾和花蛤。表3列出了14株溶藻弧菌分离株对于8类共计22种常用抗生素产生耐药性的百分比。经检测,14株溶藻弧菌分离株至少对3种抗生素耐药。其中,全部菌株对阿莫西林和青霉素耐药,对美罗培南敏感;大多数菌株对于头孢唑林和氨苄西林耐药,耐药率达92.86%;少部分菌株对于头孢噻肟、氨曲南、卡那霉素、阿米卡星、利福平耐药;极少数菌株对头孢他啶、美洛西林、四环素、氟苯尼考、复方新诺明、环丙沙星、诺氟沙星耐药。所有菌株对头孢吡肟、哌拉西林/他唑巴坦、庆大霉素、阿奇霉素、氯霉素均不耐药但显示出一定的中介率。
2.2 溶藻弧菌分离株的多重耐药性分析
2.2.1 溶藻弧菌分离株的多重耐药指数
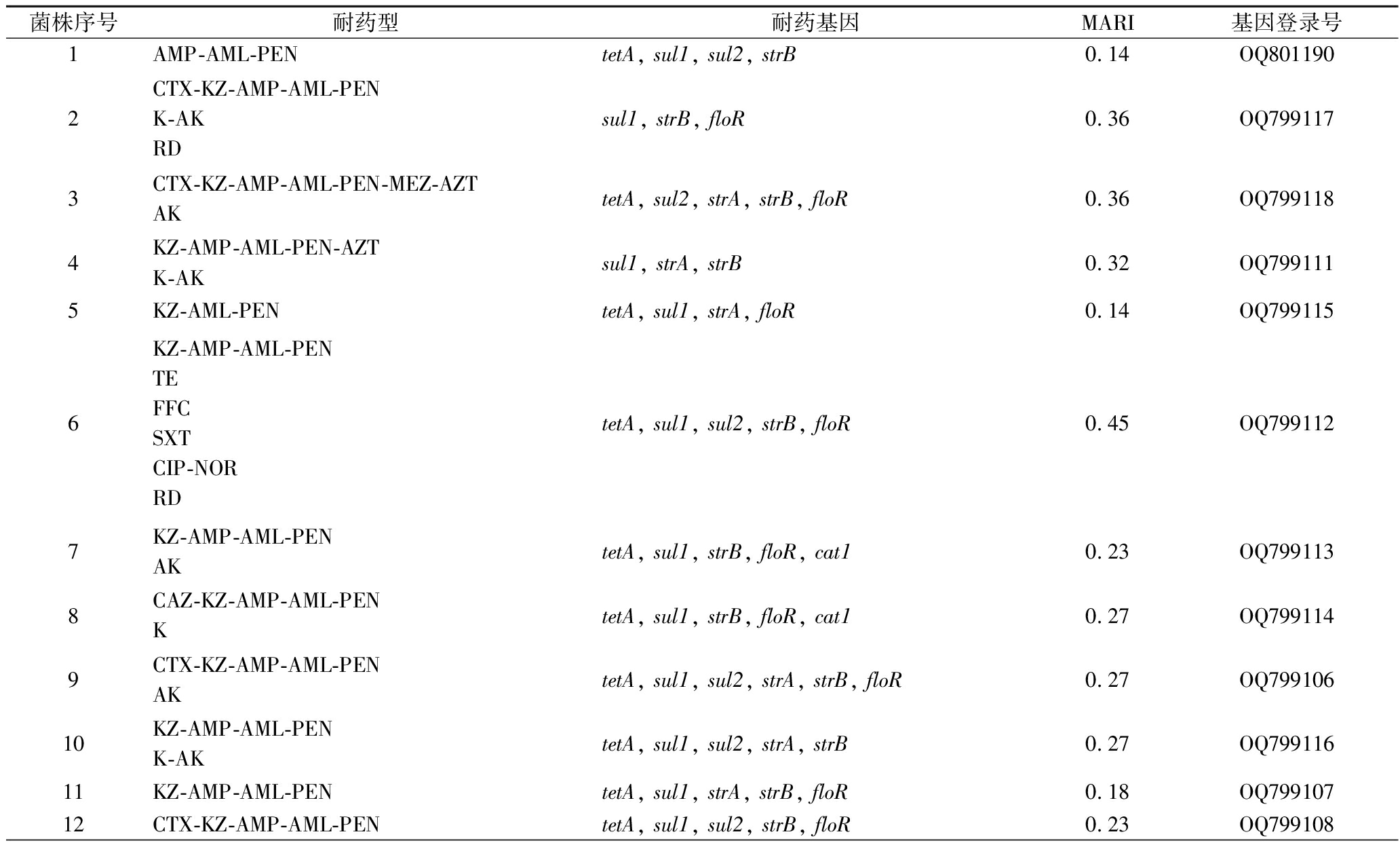
多重耐药性方面,14株溶藻弧菌出现了14种不同的耐药型(表4),其中3号、4号、7号、8号、9号、10号、13号和14号分离株对2类抗菌药物具备耐药性,2号分离株对3类抗菌药物耐药,分离自中华对虾;6号分离株对6类抗菌药物耐药,分离自花蛤。所有菌株的MARI指数为0.14~0.45。实验结果显示仅有1号、5号和11号分离株的MARI指数低于0.2,其余11个分离株的MARI指数均超过了0.2,这代表着本次实验分离到的菌株大多数都出自经常使用抗生素的高风险污染地区[8]。
表4 溶藻弧菌耐药型、耐药基因、多重耐药指数及基因登录号
Table 4 Resistance type, resistance genes, multiple antibiotic resistance index, and GenBank accession numbers of V.alginolyticus
菌株序号耐药型耐药基因 MARI基因登录号1AMP-AML-PENtetA, sul1, sul2, strB0.14OQ8011902CTX-KZ-AMP-AML-PENK-AKRDsul1, strB, floR0.36OQ7991173CTX-KZ-AMP-AML-PEN-MEZ-AZTAKtetA, sul2, strA, strB, floR0.36OQ7991184KZ-AMP-AML-PEN-AZTK-AKsul1, strA, strB0.32OQ7991115KZ-AML-PENtetA, sul1, strA, floR0.14OQ7991156KZ-AMP-AML-PENTEFFCSXTCIP-NORRDtetA, sul1, sul2, strB, floR0.45OQ7991127KZ-AMP-AML-PENAKtetA, sul1, strB, floR, cat10.23OQ7991138CAZ-KZ-AMP-AML-PENKtetA, sul1, strB, floR, cat10.27OQ7991149CTX-KZ-AMP-AML-PENAKtetA, sul1, sul2, strA, strB, floR0.27OQ79910610KZ-AMP-AML-PENK-AKtetA, sul1, sul2, strA, strB0.27OQ79911611KZ-AMP-AML-PENtetA, sul1, strA, strB, floR0.18OQ79910712CTX-KZ-AMP-AML-PENtetA, sul1, sul2, strB, floR0.23OQ799108
续表4

菌株序号耐药型耐药基因 MARI基因登录号13CTX-KZ-AMP-AML-PEN-AZTK-AKtetA, sul1, sul2, strA, strB, floR0.36OQ79910914KZ-AMP-AML-PENRDtetA, sul1, sul2, strA, strB, floR0.23OQ799110
2.2.2 溶藻弧菌分离株的耐药基因携带特征
本实验进行了8类21个耐药基因和整合酶基因的检测,共检出了4类7个耐药基因,在所有分离株中均未检出利福霉素类、喹诺酮类、大环内酯类、β-内酰胺类耐药基因和1类整合酶基因。酰胺醇类耐药基因cfr未检出,cat1和floR的检出率分别为14.28%(2/14)和78.57%(11/14);四环素类耐药基因tetB未检出,tetA的检出率为85.17%(12/14);磺胺类耐药基因sul3未检出,sul1和sul2的检出率分别为92.86%(13/14)和57.14%(8/14);氨基糖苷类耐药基因aacA和aadA未检出,strA和strB的检出率分别为57.14%(8/14)和92.86%(13/14)。结果表明从杭州市售水产品中分离得的溶藻弧菌菌株均同时携带3个及以上耐药基因,其中1号和5号分离株检出4个耐药基因,分离自中国对虾;3号、6号、7号、8号、10号、11号和12号分离株检出5个耐药基因,分别分离自中国对虾、花蛤、三文鱼、血蛤、八爪鱼、银鲳和刀额新对虾;9号、13号和14号分离株检出6个耐药基因,分别来自海产品中的南美白对虾、刀额新对虾和中国枪乌贼。
2.2.3 耐药表型与耐药基因的匹配性分析
通过头孢类抗菌药物的药敏实验可以发现,分离到的溶藻弧菌对头孢唑林有较高的耐药率(92.86%),少数分离株对于头孢噻肟和头孢他啶也出现了一定的耐药性(35.71%、7.14%),而头孢吡肟则表现出一定的中介率(50%),但尚未出现耐药性。青霉类抗菌药物的药敏试验的实验结果显示,所有的溶藻弧菌均对青霉素和阿莫西林具有耐药性,大多数分离株对氨苄西林耐药(92.86%),但大多数菌株对复合抗菌药物哌拉西林/他唑巴坦敏感(78.57%),对美洛西林表现出一定的中介率(42.86%),只有3号分离株对其表现出耐药性。对另两类β-内酰胺类抗菌药物,少数菌株对氨曲南耐药(21.43%),而所有分离株对美罗培南敏感。结合耐药基因的检测结果发现耐药性与耐药基因不完全匹配,如β-内酰胺类耐药基因blaCARB、blaTEM和blaCTX-M在所有菌株中均未检出,但分离株对部分β-内酰胺类抗菌药物具有耐药性。同样地利福霉素类药敏实验和耐药基因的检测具有类似的结果,2号、6号和14号对利福霉素类抗菌药物利福平表现出耐药但在所有菌株中均未检出利福霉素类耐药基因,耐药基因与耐药表型不符。
但是,也存在耐药性与耐药基因相匹配的情况。所有菌株中均检出了氨基糖苷类耐药基因,其中3号、4号、9号、10号、11号、13号和14号分离株同时携带了2个氨基糖苷类耐药基因,药敏实验结果显示部分菌株对卡那霉素和阿米卡星耐药(35.71%、50%),所有菌株对庆大霉素均不耐药。所有分离株均未检出大环内酯类耐药基因,相应的所有菌株对大环内酯类抗菌药物阿奇霉素均不具备耐药性,耐药基因与耐药表型相符。在喹诺酮类抗菌药物的试验中,仅11号分离株对环丙沙星和诺氟沙星耐药,另有少数菌株对环丙沙星和诺氟沙星表现出中介(21.43%、14.29%),其余分离株均对这2种药物敏感,而耐药基因检测结果显示,在所有菌株中未检出喹诺酮类耐药基因,耐药基因与耐药表型部分相符。
3 讨论
3.1 多重耐药性
药敏实验结果表明,超过半数的分离株对部分β-内酰胺类和氨基糖苷类抗菌药物耐药,可能是与这两类抗菌药物价格低廉、使用时间长且在水产养殖等相关方面应用广泛有关[23]。分离株对β-内酰胺类抗菌药物头孢唑林、氨苄西林、阿莫西林和青霉素表现出极高的耐药性,这与在上海[24]、山东[25-26]、重庆[27]等地的研究结果相似,说明环境微生物对部分早期的抗菌药物已经有较高的耐药性,这些药物在后续的防控或者医疗方面可能不再适用,需要开发新型药物以投入后续的应用。
溶藻弧菌分离株中有78.57%的MARI指数高于0.2,刘军军等[24]检测并计算2020—2021年上海市养殖水产及环境中副溶血性弧菌MARI指数超过0.2的达73.6%,与本文检测结果接近,但是STRATEV等[8]于2021—2022年对黑海水产品中副溶血性弧菌的研究,发现仅有33%的菌株MARI值达到或超过了0.2。说明国内外抗生素的使用情况存在差异,前些年国内抗生素滥用的现象较为普遍,尤其是一些应用广泛、价格低廉的抗菌素。有研究表明在我国范围内从水产品中分离得到微生物对特定抗菌药物具备高耐药性并非偶然,可能与这部分抗菌药物的长期、反复使用有关,抗生素可诱导细菌耐药的形成[28]。因此需要在加强对抗生素使用的监管的同时,加大对养殖户等相关人员的宣传科普力度,让更多人能够认识到抗生素滥用的危害从而自发拒绝滥用抗生素的行为,从源头上避免弧菌耐药性的产生。
3.2 耐药基因与表型对应性
本研究发现,从杭州水产品中分离得到的溶藻弧菌对β-内酰胺类抗菌药物的耐药性较高,其中对青霉类抗菌药物青霉素和阿莫西林的耐药性达到100%,92.86%菌株对氨苄西林耐药,但78.57%菌株对复合抗菌药物哌拉西林/他唑巴坦敏感,对美洛西林虽然表现出50%敏感率,但只有3号分离株对其表现出耐药性。在古小莉等[29]关于广州市售三文鱼的实验中得到了类似的结果,菌株对同类不同抗生素表现出的耐药性差异较大,可能是因为青霉素、阿莫西林和氨苄西林都属于早期抗生素,青霉素更是存在于天然环境中的,早在19世纪40年就已投入生产应用,阿莫西林于19世纪70年代投入生产使用,长期反复地使用使得环境中抗生素的浓度提高,可能会促使细菌耐药菌株在自然环境中比例更高。另外本研究发现分离株对一代头孢抗菌药物头孢唑林耐药率同样较高,达到92.86%,对第三代头孢抗菌素头孢噻肟和头孢他啶的耐药率分别为35.71%和7.14%,对第四代头孢抗菌素头孢吡肟暂时未检出耐药菌株,但出现了50%中介率。这与张铭琰等[23]的研究结果相似。实验发现分离株表现出相似的耐药趋势,可能是由于第一代头孢抗菌药物的使用时间最长,广泛重复地使用导致了溶藻弧菌耐药菌株比例增加。结合耐药基因的检测结果,发现耐药菌株未检出相应的耐药基因,考虑可能是其他耐药基因介导了溶藻弧菌对部分β-内酰胺类抗菌药物的耐药;也可能是由于非抗性遗传因素引起了菌株的抗性表型,如β-内酰胺类抗菌药物耐药的重要基质是青霉结合蛋白与抗菌剂的结合,可能是青霉结合蛋白发生了突变造成的耐药而非耐药基因导致[30],具体原因还需要更深入的研究。
另外,所有的菌株中均检出了氨基糖苷类耐药基因,但药敏实验结果显示部分菌株对卡那霉素和阿米卡星耐药(35.71%、50%),所有菌株对庆大霉素均不耐药,张西萌等[31]的研究发现,从北京进出口水产品中分离得的霍乱弧菌对卡那霉素、阿米卡星和庆大霉素的耐药率均低于6%;在吴书香等[26]的研究中,发现从山东青岛牡蛎和大黄鱼中分离出的菌株,有83.3%的菌株对卡那霉素耐药。上述研究与本文的结果有一定差异,推测可能与不同地区应用不同种类抗生素有关。
四环素类、磺胺类和氯霉素类抗菌药物的实验具有类似的实验结果,在易感菌株中检出了耐药基因,而在胡梦华等[7]的研究中,出现了类似的情况。有研究[32]显示,耐药基因可以通过质粒水平转移传播,可能实验中分离的溶藻弧菌耐药基因来源于其他耐药菌的质粒,而非自身突变并稳定遗传,但相关耐药基因不在细菌DNA的编码区,进而出现了携带基因却不表达的情况;也可能是因为这两类抗菌药物在药敏试验中都仅测试了一种抗生素,具有一定的局限性,值得后续深入研究。
在有关大环内酯类和喹诺酮类抗菌药物及耐药基因的研究中,发现所有菌株中均未检出这两类耐药基因,所有菌株对大环内酯类抗菌药物阿奇霉素不具备耐药性,只有6号分离株对喹诺酮类抗菌药物环丙沙星和诺氟沙星耐药,71.43%的菌株对这2种药物敏感,耐药基因与耐药表型部分相符。但阿奇霉素已于2005年在水产养殖方面被列为禁药,环丙沙星和诺氟沙星分别于2002年、2017年在水产养殖方面被禁用,这可能是大多数菌株对其敏感的原因之一,虽已禁用多年但在本次实验中还是能在水产品上分离出极少数的耐药菌,说明抗生素使用所造成的影响是延续性的,短期内无法消除。因此,加强对抗生素使用的监控,规范抗生素的使用就显得尤为重要。
4 结论
本文从杭州市105个市售水产品样品中分离出了14株溶藻弧菌,检出率为13.3%,对分离株进行了8类共22种抗菌药物的药敏试验并计算了多重耐药指数,同时进行了8类共21个相关耐药基因和1类整合子基因的检测。结果显示有2个分离株对3种抗生素耐药,MARI指数低于0.2,其余分离株均为4种及以上抗生素耐药,MARI指数高于0.2;耐药基因检测的结果显示所有分离株均同时携带3个及以上耐药基因,有3个分离株同时存在6个耐药基因,未检出1类整合子基因,抽检的水产品养殖或生活环境抗生素污染较为严重。
溶藻弧菌的致病性相较于副溶血性弧菌、霍乱弧菌等致病菌较弱,在监管的过程中可能会忽略对溶藻弧菌的防控,使环境中携带耐药基因的溶藻弧菌增多,对食品安全和人类健康具有不可忽视的风险和隐患。检测水产品中溶藻弧菌的耐药性和耐药基因,不仅有助于对水产品中溶藻弧菌的防控,也为我们了解环境中抗生素的污染情况提供了理论基础。
[1] 钟爱华, 王迎宾, 毕澜方, 等.溶藻弧菌体外刺激泥蚶(Tegillarca granosa)血细胞后早期转录组研究[J].海洋与湖沼, 2023, 54(1):286-295.
ZHONG A H, WANG Y B, BI L F, et al.The transcriptomic analysis of in vitro hemocytes of blood clam(Tegillarca granosa) challenged with Vibrio alginolyticus[J].Oceanologia et Limnologia Sinica, 2023, 54(1):286-295.
[2] HAIFA-HARYANI W O, AMATUL-SAMAHAH M A, AZZAM-SAYUTI M, et al.Prevalence, antibiotics resistance and plasmid profiling of Vibrio spp.isolated from cultured shrimp in peninsular Malaysia[J].Microorganisms, 2022, 10(9):1851.
[3] 申艳琴, 兰光, 张阳, 等.2019年兰州市螺类水产品中致病性弧菌污染状况调查[J].现代食品, 2021, 27(5):175-178.
SHEN Y Q, LAN G, ZHANG Y, et al.Investigation of pathogenic vibrios contamination status in gastropd in Lanzhou in 2019[J].Modern Food, 2021, 27(5):175-178.
[4] KUMARI P, PODDAR A, DAS S K.Characterization of multidrug resistance in Vibrio species isolated from marine invertebrates from Andaman Sea[J].3 Biotech, 2020, 10(10):456.
[5] SHAHIMI S, ELIAS A, ABD MUTALIB S, et al.Antibiotic resistance and determination of resistant genes among cockle (Anadara granosa) isolates of Vibrio alginolyticus[J].Environmental Science and Pollution Research International, 2021, 28(32):44002-44013.
[6] SAHA S, HALDER M, MOOKERJEE S, et al.Preponderance of multidrug-resistant, toxigenic, and thermotolerant enteropathogenic bacteria in raw and cooked seafood of Indo-Gangetic basin and associated health risks[J].Journal of Aquatic Food Product Technology, 2020, 29(9):838-849.
[7] 胡梦华, 马立才, 赵姝, 等.一株致病性溶藻弧菌的多重耐药与毒力基因分子分析[J].海洋渔业, 2015, 37(6):557-564.
HU M H, MA L C, ZHAO S, et al.Molecular analysis of multi-drug resistance and virulence genes in a pathogenic Vibrio alginolyticus[J].Marine Fisheries, 2015, 37(6):557-564.
[8] STRATEV D, FASULKOVA R, KRUMOVA-VALCHEVA G.Incidence, virulence genes and antimicrobial resistance of Vibrio parahaemolyticus isolated from seafood[J].Microbial Pathogenesis, 2023, 177:106050.
[9] KRUMPERMAN P H.Multiple antibiotic resistance indexing of Escherichia coli to identify high-risk sources of fecal contamination of foods[J].Applied and Environmental Microbiology, 1983, 46(1):165-170.
[10] 刘旭. 海水养殖源弧菌耐药性调查及qnrVC基因在弧菌中的流行情况研究[D].上海:上海海洋大学, 2016.
LIU X.Investigation on antimicrobial resistance and the prevalence of qnrVC gene in Vibrios from mariculture sources[D].Shanghai:Shanghai Ocean University, 2016.
[11] ROBICSEK A, STRAHILEVITZ J, SAHM D F, et al.Qnr prevalence in ceftazidime-resistant Enterobacteriaceae isolates from the United States[J].Antimicrobial Agents and Chemotherapy, 2006, 50(8):2872-2874.
[12] NG L K, MARTIN I, ALFA M, et al.Multiplex PCR for the detection of tetracycline resistant genes[J].Molecular and Cellular Probes, 2001, 15(4):209-215.
[13] XU J, XU Y, WANG H M, et al.Occurrence of antibiotics and antibiotic resistance genes in a sewage treatment plant and its effluent-receiving river[J].Chemosphere, 2015, 119:1379-1385.
[14] CHUANCHUEN R, PADUNGTOD P.Antimicrobial resistance genes in Salmonella enterica isolates from poultry and swine in Thailand[J].Journal of Veterinary Medical Science, 2009, 71(10):1349-1355.
[15] CHUANCHUEN R, KOOWATANANUKUL C, KHEMTONG S.Characterization of class 1 integrons with unusual 3′ conserved region from Salmonella enterica isolates[J].The Southeast Asian Journal of Tropical Medicine and Public Health, 2008, 39(3):419-424.
[16] OUOBA L I I, LEI V, JENSEN L B.Resistance of potential probiotic lactic acid bacteria and bifidobacteria of African and European origin to antimicrobials:Determination and transferability of the resistance genes to other bacteria[J].International Journal of Food Microbiology, 2008, 121(2):217-224.
[17] VAN T T H, CHIN J, CHAPMAN T, et al.Safety of raw meat and shellfish in Vietnam:An analysis of Escherichia coli isolations for antibiotic resistance and virulence genes[J].International Journal of Food Microbiology, 2008, 124(3):217-223.
[18] KEHRENBERG C, SCHWARZ S.Distribution of florfenicol resistance genes fexA and cfr among chloramphenicol-resistant Staphylococcus isolates[J].Antimicrobial Agents and Chemotherapy, 2006, 50(4):1156-1163.
[19] CHIOU J C, LI R C, CHEN S.CARB-17 family of β-lactamases mediates intrinsic resistance to penicillins in Vibrio parahaemolyticus[J].Antimicrobial Agents and Chemotherapy, 2015, 59(6):3593-3595.
[20] COLOMER-LLUCH M, JOFRE J, MUNIESA M.Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples[J].PLoS One, 2011, 6(3):e17549.
[21] HENRIQUES I S, FONSECA F, ALVES A, et al.Occurrence and diversity of integrons and β-lactamase genes among ampicillin-resistant isolates from estuarine waters[J].Research in Microbiology, 2006, 157(10):938-947.
[22] MAZEL D, DYCHINCO B, WEBB V A, et al.Antibiotic resistance in the ECOR collection:Integrons and identification of a novel aad gene[J].Antimicrobial Agents and Chemotherapy, 2000, 44(6):1568-1574.
[23] 张铭琰, 李雪, 刘海霞, 等.辽宁沿海地区溶藻弧菌的耐药性及分子分型[J].中国微生态学杂志, 2019, 31(1):48-51.
ZHANG M Y, LI X, LIU H X, et al.The drug resistance and molecular typing of Vibrio alginnolyficus strains in coastal area of Liaoning[J].Chinese Journal of Microecology, 2019, 31(1):48-51.
[24] 刘军军, 刘单阳, 李达容, 等.上海市养殖水产品及环境中副溶血性弧菌的流行、毒力及耐药性分析[J].食品与发酵工业, 2023, 49(4):250-257.
LIU J J, LIU D Y, LI D R, et al.The prevalence, virulence, and antibiotic resistance of Vibrio parahemolyticus in aquatic products and their breeding environment in Shanghai[J].Food and Fermentation Industries, 2023, 49(4):250-257.
[25] 张瑞卿. 山东省贝源弧菌流行病学调查及其毒力基因检测[D].泰安:山东农业大学, 2021.
ZHANG R Q.Epidemiological investigation of Vibrio from shellfish in Shandong province and detection of virulence genes[D].Tai’an:Shandong Agricultural University, 2021.
[26] 吴书香, 李丽倩, 姚琳, 等.海产源多重耐药共生菌的耐药表型及耐药基因分析[J].食品安全质量检测学报, 2023, 14(2):248-255.
WU S X, LI L Q, YAO L, et al.Analysis of antimicrobial resistance phenotype and genotype of multipleantimicrobial resistant commensal bacteria from marine products[J].Journal of Food Safety &Quality, 2023, 14(2):248-255.
[27] 赵力, 丁国英, 朱秋昊, 等.万州零售淡水鱼中副溶血性弧菌的污染状况与特征研究[J].食品与发酵工业, 2024, 50(16):242-248.
ZHAO L, DING G Y, ZHU Q H, et al.Study on the pollution status and characteristics of Vibrio parahaemolyticus in retail freshwater fish in Wanzhou[J].Food and Fermentation Industries, 2024, 50(16):242-248.
[28] 张凯, 张明明, 谢曦, 等.恩诺沙星诱导溶藻弧菌耐药和交叉耐药的研究[J].水产科学, 2022, 41(3):452-458.
ZHANG K, ZHANG M M, XIE X, et al.Analysis of drug-resistance and cross resistance in vitro of Vibrio alginolyticus induced by enrofloxacin[J].Fisheries Science, 2022, 41(3):452-458.
[29] 古小莉, 李惠青, 徐瑞文, 等.三文鱼来源副溶血性弧菌的血清分型、毒力基因及耐药性研究[J].中国食品卫生杂志, 2022, 34(6):1250-1256.
GU X L, LI H Q, XU R W, et al.Serotype, virulence and drug resistance of Vibrio parahaemolyticus from salmon[J].Chinese Journal of Food Hygiene, 2022, 34(6):1250-1256.
[30] 孙艳, 多丽波.耐碳青霉烯类肠杆菌科细菌耐药机制及实验室检测研究进展[J].国际检验医学杂志, 2020, 41(16):2011-2016.
SUN Y, DUO L B.Research progress on the mechanism of carbapenems Enterobacteriaceae drug resistance and its laboratory testing[J].International Journal of Laboratory Medicine, 2020, 41(16):2011-2016.
[31] 张西萌, 付溥博, 魏海燕, 等.北京进出口水产品中259株霍乱弧菌分离株的耐药性研究[J].食品安全质量检测学报, 2020, 11(14):4901-4906.
ZHANG X M, FU P B, WEI H Y, et al.Study on drug resistance of 259 Vibrio cholerae isolates from imported and exported aquatic products in Beijing[J].Journal of Food Safety &Quality, 2020, 11(14):4901-4906.
[32] 叶蕾. 广州市水产养殖品中耐药共生菌分布及耐药基因传播机制的研究[D].广州:华南理工大学, 2012.
YE L.Prevalence of antibiotic resistant commensal bacteria associated with aquaculture produces and the dissemination of antibiotic resistant gene from Guangzhou, China[D].Guangzhou:South China University of Technology, 2012.