木质素是自然界中含量丰富的三大生物质之一,占陆生植物干重的15%~40%,其降解产物极具成为生物质燃料的潜力。我国是农业生产大国,秸秆产量占世界总产量的1/3,但在实际农业生产中,大部分秸秆无法得到有效的降解或处理,成为了农业和工业生产过程中的主要废弃物[1]。这主要是因为木质素作为植物细胞骨架的主要成分,其结构为由苯丙烷单元通过醚键和碳碳键组成的具有三维空间结构的无定型芳香类化合物[2-3],这种特殊结构使其在自然界中难被降解,造成了严重的环境污染和资源浪费。因此,木质素高效环保降解方式的研究具有重要意义,一方面能减少工农业废弃物的堆积浪费,减轻环境负担;另一方面降解产生的沼气发酵有助于缓解能源问题。
自然界中木质素虽然难以降解,但是仍存在部分微生物对木质素有较好的分解作用,其中主要以真菌、细菌和放线菌为主[4]。真菌降解木质素的研究较多,但其遗传改造相对细菌难度较高,且大部分真菌为需氧菌,如在秸秆沼气发酵等厌氧或兼性厌氧环境,真菌的木质素降解作用难以发挥。细菌遗传改造简单,生长环境粗放,繁殖速度快,定向筛选需氧或厌氧菌均可持续稳定产酶,更适合工业化生产。通过优化发酵工艺及遗传改造可以高效降解特定的木质素结构,从而提高降解效率[5]。
枯草芽孢杆菌(Bacillus subtilis)是一种广泛存在于土壤中的厌氧细菌,具有良好的降解能力和多样性,其表达产物漆酶具有较好的木质素降解效果[6]。近年来,较多研究集中在B. subtilis全基因的挖掘与解析[7-12],却很少完整阐述B. subtilis在降解木质素中相关基因表述。本研究采用苯胺蓝脱色法结合酶活性评价,从自然界特殊环境中筛选出一株对木质素具有高效降解能力的Bacillus subtilis HB-011,并通过全基因分析获取其生物信息特征,同时对其酶学性质进行了表征,为木质素高效降解酶工程的开展提供了优质微生物菌种资源和一定理论依据。
1 材料与方法
1.1 实验材料
1.1.1 样品来源
样品采集于山东省烟台市和日照市等,黑龙江省伊春和齐齐哈尔市等,内蒙古自治区等地的腐熟秸秆堆、青贮黄贮饲料、秸秆沼气发酵池、牛羊马粪、林地腐朽木等处,共109份样品。
1.1.2 试剂及培养基
苯胺蓝、漆酶对照品、碱木质素,上海源叶生物科技有限公司;愈创木酚,天津大茂化学试剂厂;马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基,北京奥博星生物技术有限公司;LB培养基(g/L):胰蛋白胨10、酵母浸膏5、NaCl 10;发酵培养基(g/200 mL):碱性木质素1.0、牛肉膏1.0、NaCl 1.0、蛋白胨2.0,液体种子培养液。
1.1.3 仪器设备
ZHWY-2102C紫外分光光度计,上海菁华科技仪器公司;722S恒温培养振荡器,上海智城分析仪器制造有限公司;J-810型圆二色谱仪,日本JASCO公司。
1.2 筛选方法
初筛:将每个样品以2 g的量溶于200 mL无菌水中制得原菌液。将原菌液在37 ℃恒温培养24 h后用50 g/L生理盐水稀释至不同浓度,使用苯胺蓝脱色法在PDA培养中筛选出能产木质素降解酶的菌株[13],测量透明圈直径。将初筛的菌株在LB培养基进行分离纯化。
复筛:为进一步确定菌株的产物酶活性,将初筛菌液培养至OD>0.6后以1%接种量接种至50 mL发酵培养基中,37 ℃、120 r/min离心培养30 h后,4 ℃ 下8 000 r/min离心10 min,上清液即待测粗酶液。
1.3 漆酶酶活性测定方法
根据KUMARASAMY等[14]的方法稍作修改,在37 ℃下将2 mL,0.5 mmol/L ABTS(溶于0.1 mmol/L,pH值为5.0的醋酸-醋酸钠缓冲溶液)与1 mL粗酶液混合制备3 mL反应体系,在420 nm下测定吸光度的变化值。定义一个酶活力单位(U/L)为每分钟氧化1 μmol ABTS所需的酶量,酶活性按公式(1)计算:
(1)
式中:ΔA,时间(t)内的吸光度变化值;b,比色皿厚度,cm;t,反应时间,min;V1,反应体系的体积,L;V2,粗酶液的体积,L;ε,摩尔吸光系数,mol-1·cm-1,其中,ABTS的ε420=36 000 (mol-1·cm-1)。
1.4 木质素降解率的测定
将液体菌液接入产酶发酵培养基中,接种量为2%,培养温度为35 ℃,进行为期1周的降解实验,漆酶酶活性进行3组平行测定。细菌对木质素降解率的测定采用普鲁士法[15],测定木质素降解过程中浓度的变化情况,进行3组平行测定。
1.4.1 木质素标准曲线测定
取1.5 mL不同质量浓度(10~40 μg/mL)的碱木质素加入试管中,加入10 μL 8 mmol K3Fe和100 μL 0.1 mol/L FeCl3到试管中,混匀后在30 ℃反应5~10 min,在700 nm波长下测定吸光度,以去离子水作为空白对照,测定OD值并绘制木质浓度标准曲线(y=0.045 7x-0.032 1, R2=0.990 3)。
1.4.2 木质素浓度及降解率计算
液体发酵培养基稀释一定倍数后,按照上述方法测定其吸光度,将OD值带入木质素浓度标准曲线中,并按照公式(2)计算出木质素降解率:
木质素降解率![]()
(2)
式中:A0,木质素初始浓度,μg/mL;A1,某时刻培养基中剩余木质素浓度,μg/mL。
1.5 菌株鉴定
生理生化鉴定:使用LB对复筛获得的菌株在37 ℃恒温培养24 h进行活化后在PDA平板上划线涂布,再次在37 ℃恒温培养24 h后观察菌落形态并进行革兰氏染色,根据《常见细菌系统鉴定手册》[16]和《伯杰氏细菌鉴定手册》[17]进行生理生化试验鉴定。
根据KIM等[18]的方法进行细菌总DNA的提取。使用PCR通用引物(上游引物16S-7F:5′-AGAGTTTGATYMTGGCTCAG-3′,下游引物16S-1510R:5′-ACGGYTACCTTGTTACGACTT-3′)对纯化细菌进行扩增。
PCR体系为:DNA(70 ng/μL)模板2 μL;dNTP Mixture(2.5 mmol/L)2.5 μL;7F(20 μmol/L)1.5 μL;1510R(20 μmol/L)1.5 μL;10×ExTaq Buffer(Mg2+ pluse)5 μL;ExTaq DNA 聚合酶0.2 μL;补足ddH2O到50 μL。
PCR条件为:94 ℃预变性3 min;然后94 ℃变性1 min、55 ℃退火 1 min、72 ℃延伸 3 min,共30个循环;最后72 ℃延伸5 min。
随后对PCR产物进行1%琼脂糖凝胶电泳检测。选取符合预期要求的纯化PCR产物,送至生工生物工程(上海)股份有限公司测序。测序结果在GenBank(https://www.ncbi.nlm.nih.gov/genbank/)中进行blast序列比对,用Neighbor-Joining法构建系统发育树[18]。
1.6 全基因分析
本次测序委托上海美吉生物科技有限公司完成。样本质检合格后,采用二代测序技术通量测序平台完成测序分析,原始数据的接头去除及低质量序列等工作使用质量统计软件来进行优化。
1.6.1 基因组组装
基于二代测序数据,使用软件unicycler v0.4.8进行组装,组装过程中使用pilonjin v1.22软件进行序列校正,得到初步组装结果,再使用软件SOAPdenovo2对组装结果进一步校正,得最终组装结果[19-20]。
1.6.2 基因预测
通过GeneMarkS软件对基因组中的编码序列进行预测。利用tRNAscan-SE v2.0和Barrnap软件分别对基因组中tRNA和rRNA进行预测,获得基因组中tRNA和rRNA序列信息[21-23]。
1.6.3 基因组圈图的绘制
将测序得到的基因组序列、编码基因预测及非编码RNA等信息文件组装成GBK文件,通过CGView软件绘制单个样本基因组圈图,全面展示测序基因组特征[24]。
1.6.4 基因功能分析
预测的编码蛋白序列分别与Cluster of Orthologous Groups of Proteins(COG,https://www.ncbi.nlm.nih.gov/research/cog/)、Gene Ontology(GO,https://www.geneontology.org/)、Kyoto Encyclopedia of Genes and Genomes(KEGG,https://www.genome.jp/kegg/)、Carbohydrate-Active EnZymes Database(CAZY,http://www.cazy.org/)数据库中的蛋白序列进行blast比对和注释。预测的基因组序列分别Comprehensive Antibiotic Resistance Database(CARD,https://card.mcmaster.ca/)数据库进行序列比对,完成耐药基因分析。预测的氨基酸序列与致病毒力因子VFDB数据库进行比对,完成毒力基因注释结果[25-30]。
1.7 酶学研究
1.7.1 粗酶液的纯化
取适量液体产酶LB培养液在5 000 r/min下离心15 min得到的上清液为粗酶液,随后向其中缓缓加入适量(NH4)2SO4搅拌均匀,4 ℃静置过夜使其充分沉淀,以4 ℃、10 000 r/min冷冻离心15 min,上清液即为漆酶浓缩液。将预处理后的DEAE-A50葡聚糖凝胶装入层析柱,柱高15 cm,用NaCl缓冲液充分平衡层析柱,向A-50凝胶层析柱内加入1 mL漆酶浓缩液,充分结合后用缓冲液NaCl进行洗脱,收集到的洗脱组分即为纯化漆酶液。
1.7.2 动力学参数测定
以愈创木酚为底物并配制成0.11~0.99 mol/L的PBS,浓度梯度为0.11 mol/L,35 ℃下在不同的底物浓度下测定发酵漆酶酶活性,采用米氏方程拟合得出Vmax、Km与Kcat值。
1.7.3 稳定性测试
在最适温度下测定不同pH下漆酶酶活性,设置pH梯度为1,测定pH范围为2~12;在最适pH下测定不同温度下的漆酶酶活性,设置温度梯度为10 ℃,测定温度范围为15~85 ℃。
1.7.4 漆酶二级结构测定
使用圆二色谱仪漆酶标品以及纯化漆酶样品进行测定,样品蛋白浓度为6×10-6 mol/L。在25 ℃下,设置扫描范围190~250 nm。根据以下公式将数据转化为[θ](deg·cm2/dmol)后,使用CDpro软件包中Continill程序进行,计算按照公式(3)所示:
(3)
式中:θ,C的信号值;C,蛋白质浓度,mol/L;l,光程,cm;R,枯草芽孢杆菌漆酶平均氨基酸残基数,g/moL。B. subtilis漆酶平均氨基酸残基数为512 g/mol,分子质量53 kDa,质量浓度0.2 mg/mL,测量光程为1 mm。
1.8 统计分析
各试验均重复3次,数据表示为平均值±标准差。采用Origin 2023b软件进行绘图,Excel 2020软件进行数据处理。
2 结果与分析
2.1 复筛菌株酶活性初步测定
复筛及分离纯化的结果如图1所示,表明所得菌株分别在愈创木酚培养基、PDA苯胺蓝培养基中有不同的显色表现,从而初步得判断该菌株能产生多种降解木质素酶,对木质素的降解效果更佳。将获得的最优菌分离纯化,经过纯化化接入斜面保存,以便后续实验。
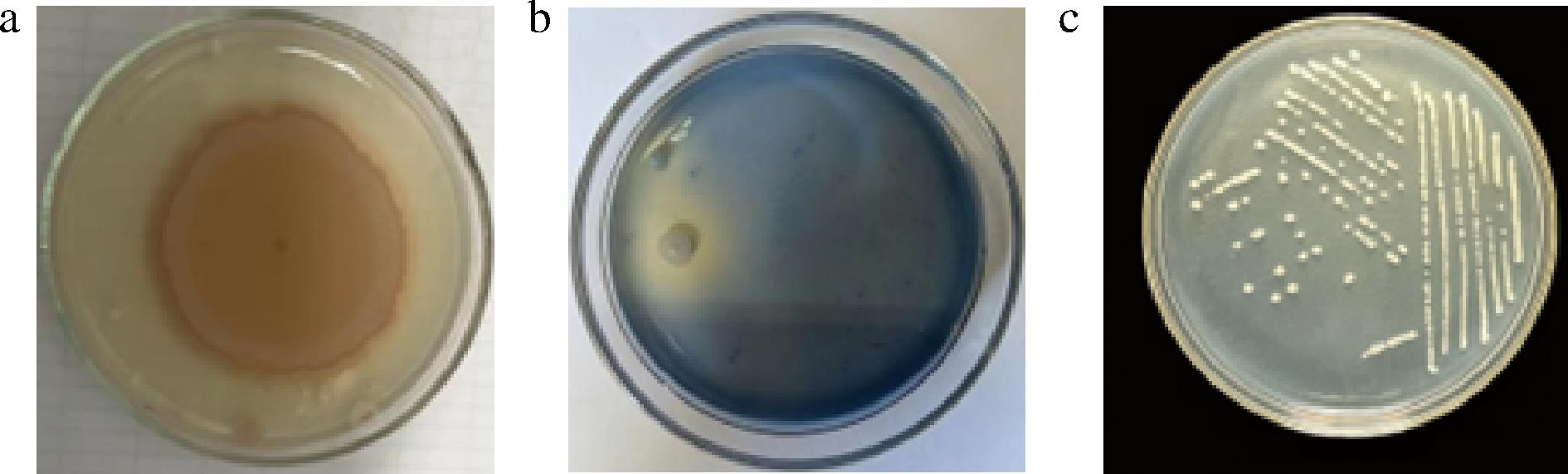
a-愈创木酚培养基;b-PDA苯胺蓝培养基;c-分离纯化
图1 复筛菌株酶活性初步测定结果
Fig.1 Preliminary results of enzyme activity of rescreened strains
2.2 菌株鉴定结果
2.2.1 油镜镜检
对纯化的菌株进行油镜镜检,如图2所示,油镜下复筛得到的菌株表面粗糙,是边缘整齐的圆形不透明菌落,呈黏着形态,大部分菌株为革兰阳性杆状细菌。
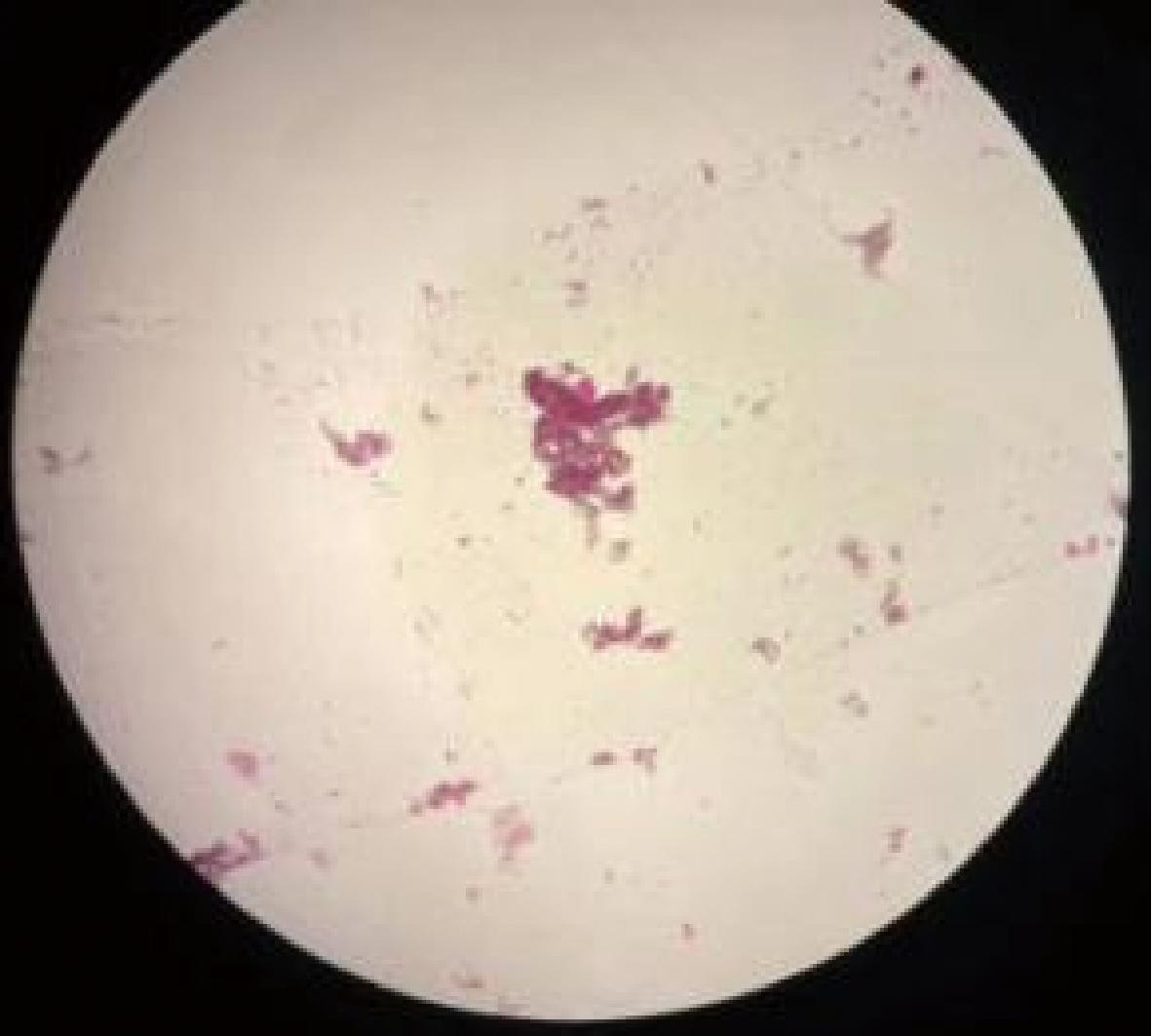
图2 复筛分离菌株油镜镜检图
Fig.2 Oil microscope image of the re-screened isolates
2.2.2 琼脂糖凝胶电泳及发育树的构建
对该菌株进行琼脂糖凝胶电泳实验并构建发育树。如图3所示,成功扩增的菌落PCR产物全长约15 000 bp。将测序结果与NCBI数据库进行核酸对比后得到的系统发育树,如图4所示,PCR产物与同属的地衣芽孢杆菌的最高同源性为97.1%。根据以上试验结果,初步确定分离株为B. subtilis,并命名为Bacillus subtilis HB-011。
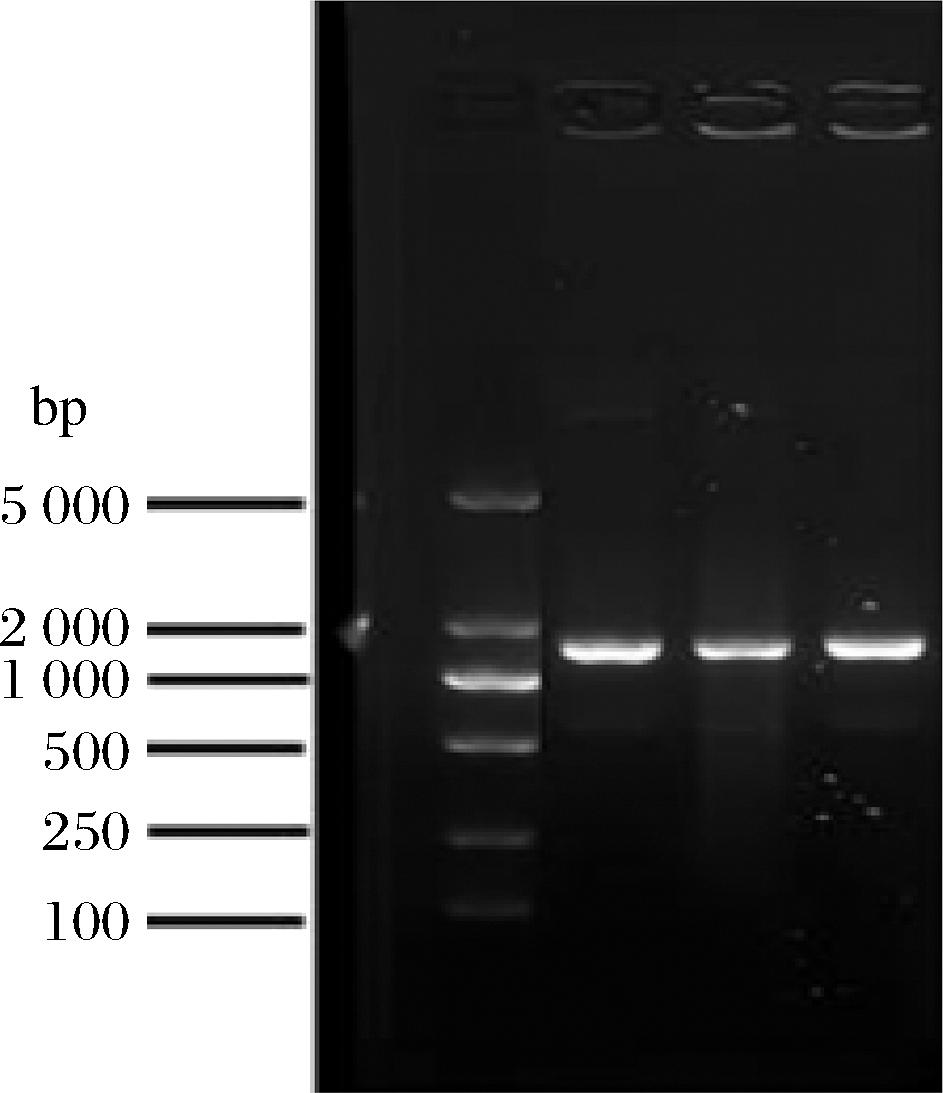
图3 PCR产物琼脂糖凝胶电泳结果图
Fig.3 PCR product agarose gel electrophoresis results
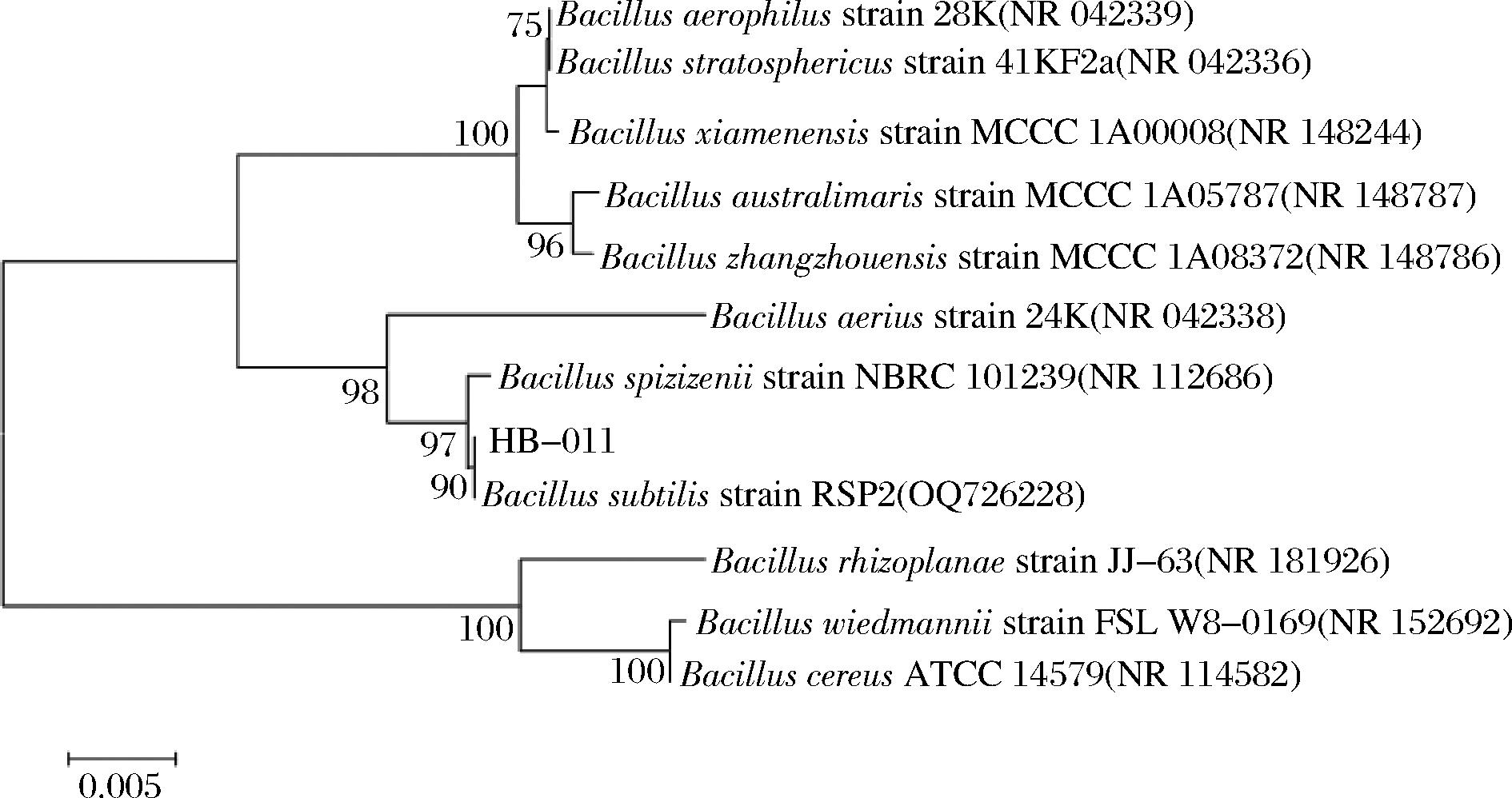
图4 PCR产物系统发育树的构建
Fig.4 Construction of phylogenetic tree of PCR products
2.3 Bacillus subtilis HB-011降解碱木质素分析
由图5可知,碱木质素降解过程中Bacillus subtilis HB-011所产漆酶在发酵第3天时酶活性最高6.58 U/L,在发酵过程中前3 d产酶速率增加后趋于稳定。可以推断出该细菌产漆酶的稳定性较好。
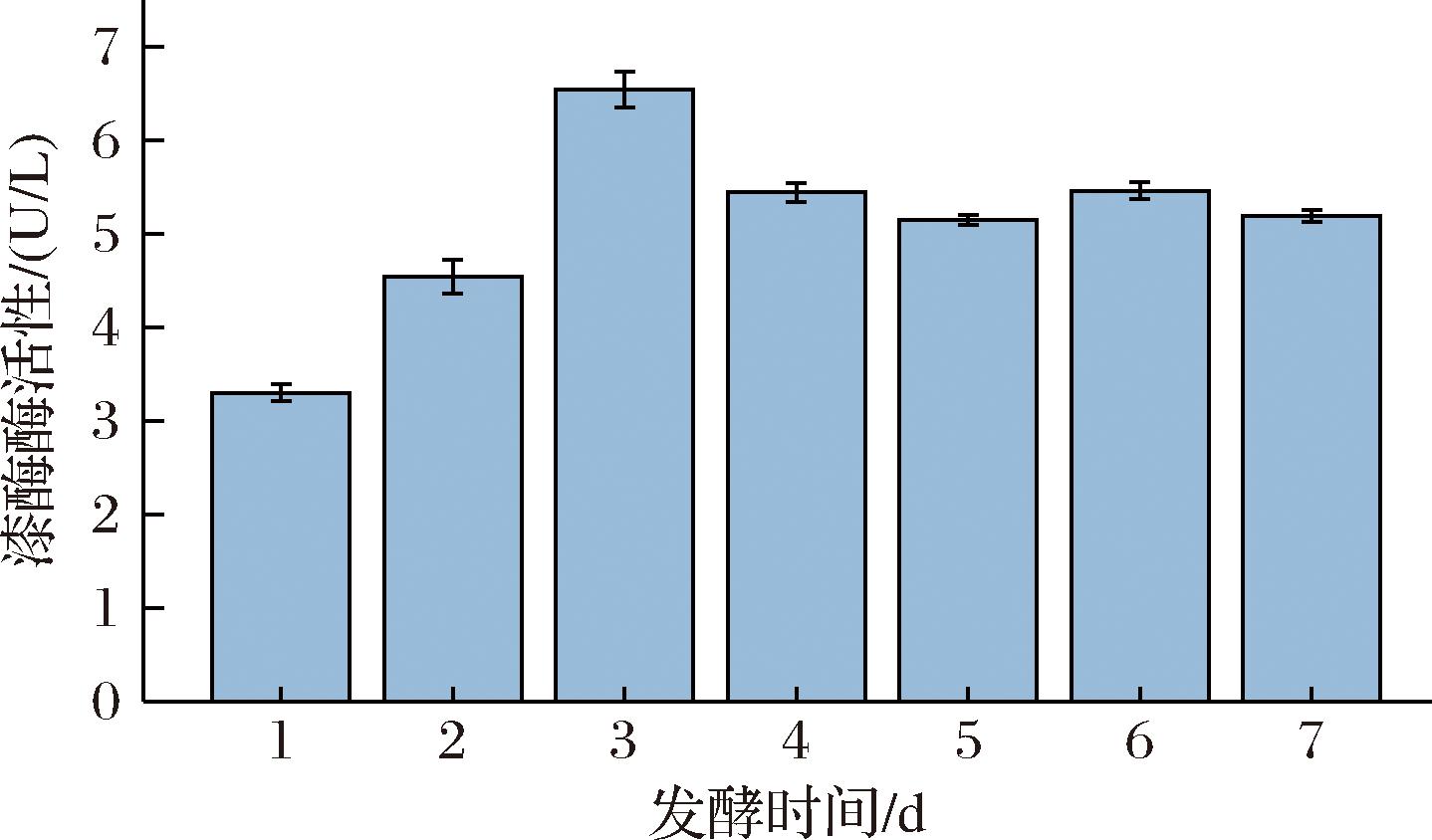
图5 木质素降解过程中漆酶酶活力变化情况
Fig.5 Changes in laccase enzyme activity during lignin degradation
由图6可知,经过发酵处理的碱木质素的质量浓度随发酵时间的延长产生了明显变化,发酵前3 d木质素降解速率明显增加,在发酵第3天时降解率达到20%,之后木质素质量浓度及降解速率变化缓慢。
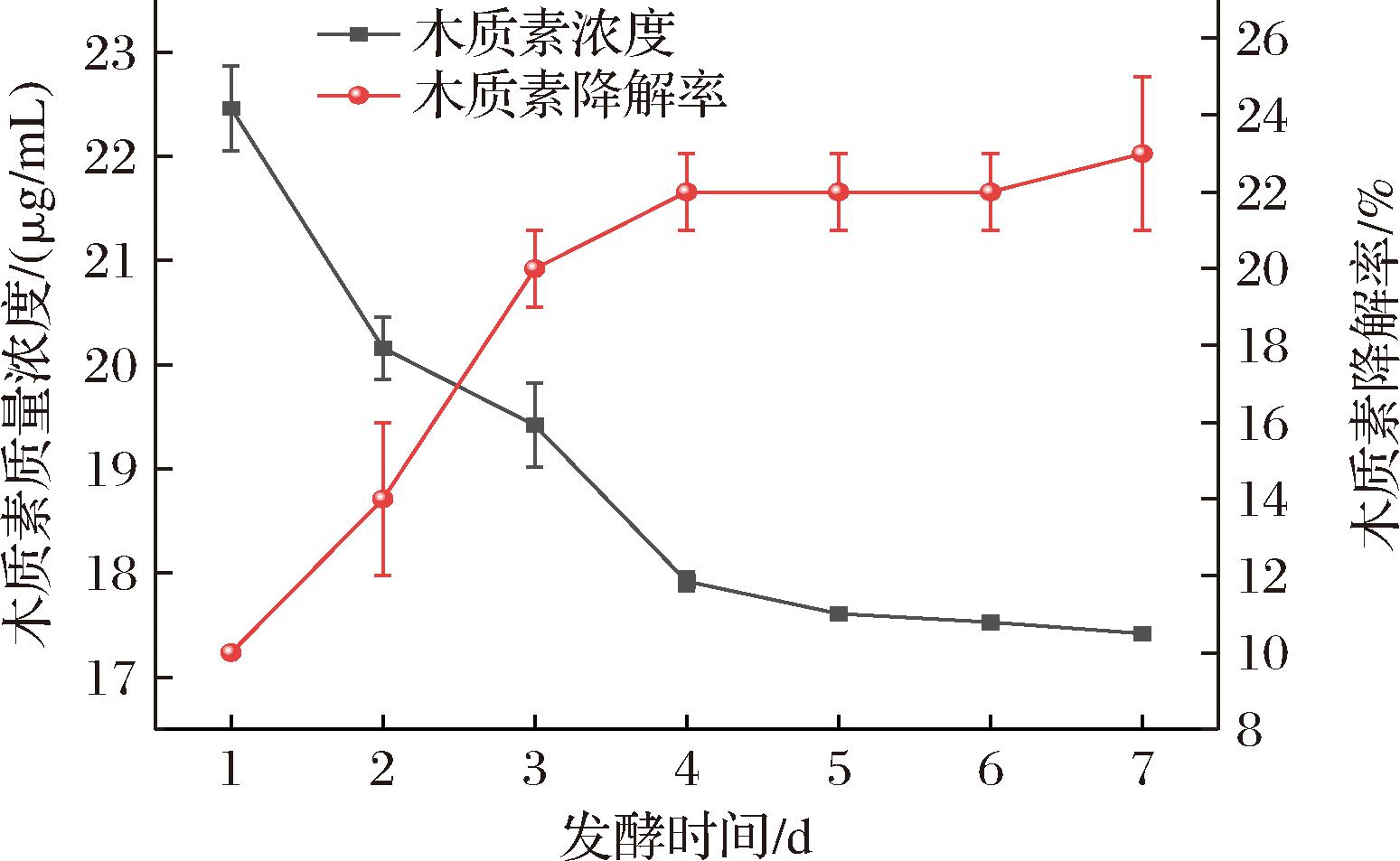
图6 木质素质量浓度及降解率变化情况
Fig.6 Variation in lignin concentration and degradation rate
2.4 Bacillus subtilis HB-011基因组分析
基因组分析结果(图7)显示,Bacillus subtilis HB-011的基因组全长为4 215 536 bp,CG含量43.51%。基因组预测编码了4 229个开放阅读框,预测基因的平均长度为878 bp,其中假基因11个,非编码RNA基因217个,主要包括30个rRNA、86个tRNA和101个其他ncRNA。

图7 Bacillus subtilis HB-011基因组特征分析
Fig.7 Genomic characterization of Bacillus subtilis HB-011
如图8所示,长度>500 bp的基因有1 544个,长度>1 000 bp的基因有1 560个。基因组特征显示重复序列43个,总长为16 091 bp,其占比为0.36%。
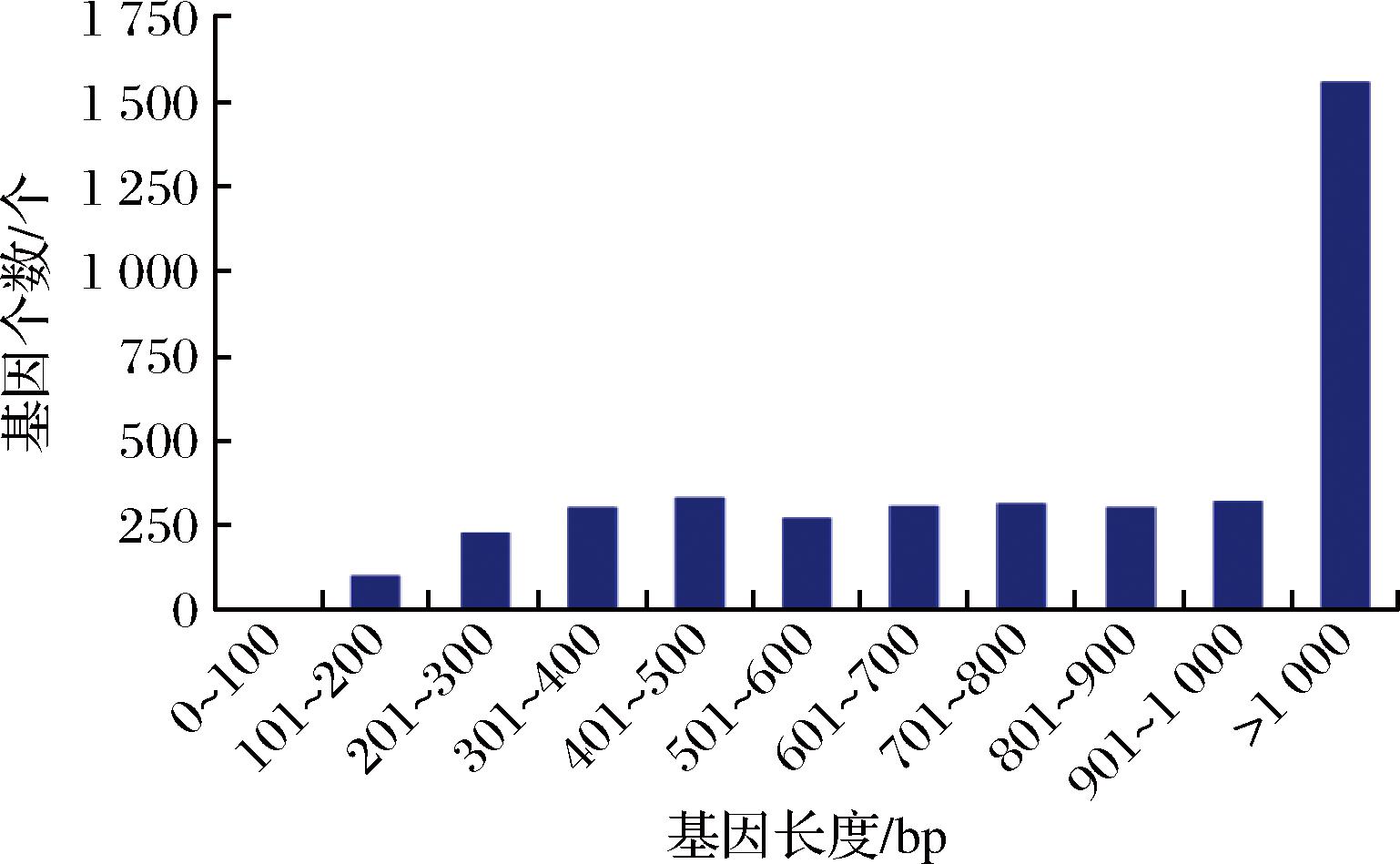
图8 基因编码长度统计结果
Fig.8 Statistical results of gene coding length
2.5 基因数据库注释
2.5.1 GO数据库注释
将Bacillus subtilis HB-011基因组中氨基酸序列与GO注释数据库进行对比分析,结果如图9所示,共有2 220个功能基因得到注释,其中504、1 388和1 115功能基因分别注释了分子功能、生物过程和细胞组分。
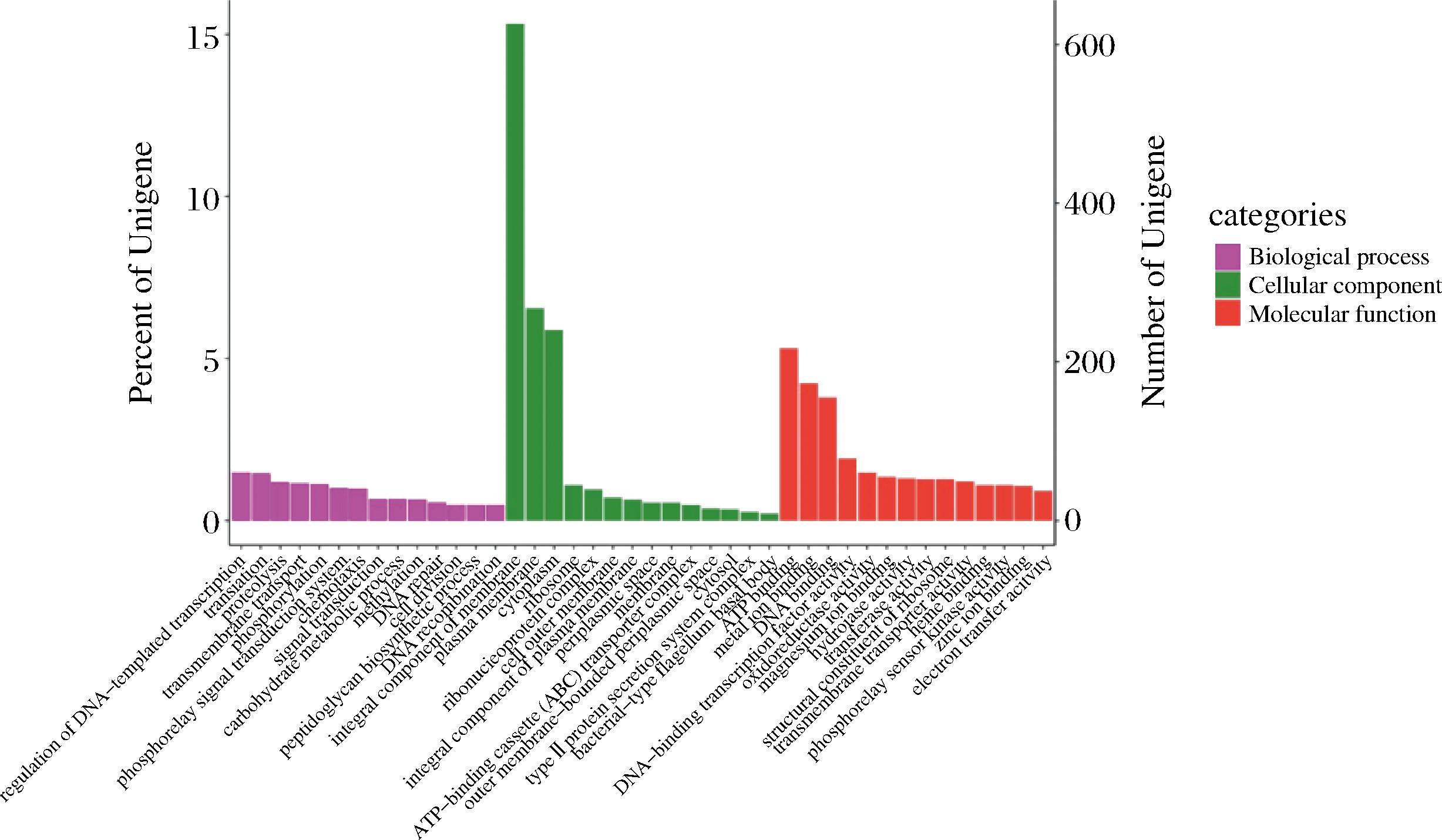
图9 Bacillus subtilis HB-011基因组基因功能GO数据库注释
Fig.9 Annotation of GO database on gene function of Bacillus subtilis HB-011 genome
注:regulation of DNA-templated transcription(DNA模板化转录的调控);translation(翻译);proteolysis(蛋白水解);ransmembrane transport(跨膜转运);phosphorylation(磷酸化);phosphorelay signal transduction system(磷酸转移信号转导系统);chemotaxis(趋化性);signal transduction(信号转导);carbohydrate metabolic process(碳水化合物代谢过程)methylation(甲基化);DNA repair(DNA修复);cell division(细胞分裂);peptidoglycan biosynthetic process(肽聚糖生物合成过程);DNA recombination(DNA重组)ntegral component of membrane(膜的积分组分);plasma membrane(质膜);cytoplasm(细胞质);ribosome(核糖体);ribonucleoprotein complex(核糖核蛋白复合物);cell outer membrane(细胞外膜);integral component of plasma membrane(质膜的整体成分);periplasmic space(细胞周质间隙);membrane(薄膜);ATP-binding cassette (ABC) transporter complex(ATP结合盒( ATP-binding cassette,ABC )转运蛋白复合体);outer membrane-bounded periplasmic space(外膜-有界周质空间);cytosol(细胞溶质);type II protein secretion system complex(Ⅱ型蛋白分泌系统复合体);bacterial-type flagellum basal body(细菌-型鞭毛基体);ATP binding(三磷酸腺苷结合);metal ion binding(金属离子结合);DNA binding(DNA结合);DNA-binding transcription factor activity(DNA结合转录因子活性);oxidoreductase activity(氧化还原酶)magnesium ion binding(镁离子结合);hydrolase activity(水解酶活性);transferase activity(转移酶活性);structural constituent of ribosome(核糖体的结构组成);transmembrane transporter activity(核糖体的结构组成);heme binding(血红素结合);phosphorelay sensor kinase activity(磷酸转移传感器激酶活性);zinc ion binding(锌离子结合);electron transfer activity(电子传递活性)。
在生物过程功能中调控DNA触发的转录、翻译和蛋白分解的功能基因分别有60、59和48个,调控跨膜运输的功能基因有46个,磷酸化调控基因和磷酸信号转导基因分别有47、41个。
在细胞组分中,膜组成相关的调控基因为628个,质膜的功能基因为268个,两者整体占比最多。
在分子功能中,ATP结合的相关基因有217个,金属离子结合的相关基因有173个,NDA结合的基因有155个,DNA结合转录因子活性的基因有78个,氧化还原酶活性的基因有60个,Mg2+结合相关基因55个,水解酶活性的基因为53个,转氨酶活性相关基因52个,血红素结合基因45个,磷酸酶活性相关基因45个,Zn2+结合相关基因44个。
2.5.2 COG数据库注释
在COG数据库检索Bacillus subtilis HB-011基因组的蛋白质编码基因,结果如图10所示,共有3 480个蛋白质编码基因得到注释。如图8所示,注释结果类别共有24个,其中比较高频的类别包括:细胞外结构(263个),氨基酸转运和代谢(319个),碳水化合物的运输和代谢(236个),辅酶的运输和代谢(186个),脂质运输和代谢(221个),翻译、核糖体结构和生物生成(258个),转录(247个),复制、重组和修复(153个),细胞壁/膜/包膜的生物生成(256个),翻译后修饰、蛋白质周转、合子(176个),无机离子传输和新陈代谢(217个),信号转导机制(343个)。

图10 Bacillus subtilis HB-011基因组基因功能COG数据库注释
Fig.10 Bacillus subtilis HB-011 genome gene function COG database annotation
注:A:RNA processing and modification(RNA加工和修饰);C:Energy production and conversion(能源生产与转换);D:Cell cycle control, cell division, chromosome partitioning(细胞周期控制、细胞分裂、染色体分裂);E:Amino acid transport and metabolism:(氨基酸转运和代谢);F:Nucleotide transport and metabolism(核苷酸转运与代谢);G:Carbohydrate transport and metabolism(碳水化合物转运与代谢);H:Coenzyme transport and metabolism(辅酶运输和代谢);I:Lipid transport and metabolism(脂质转运和代谢)J:Translation, ribosomal structure and biogenesis(翻译、核糖体结构和生物合成);K:Transcription(转录);L:Replication, recombination and repair(复制、重组和修复);M:Cell wall/membrane/envelope biogenesis(细胞壁膜/囊膜生物合成);N:Cell motility(细胞运动性)O:Posttranslational modification, protein turnover, chaperones(翻译后修饰,蛋白质周转,分子伴侣)P:inorganic ion transport and metabolism(无机离子转运与代谢):Q:Secondary metabolites biosynthesis, transport and catabolism(次级代谢产物的生物合成、转运和分解代谢);R:General function prediction only(仅进行一般函数预测);S:Function unknown(功能未知):T:Signal transduction mechanisms(信号传导机制);U:Intracellular trafficking, secretion, and vesicular transport(细胞内运输、分泌和囊泡运输);V:Defense mechanisms(防御机制);W:Extracellular structures(细胞外结构);X:Mobilome: prophages, transposons(转座组:原噬菌体、转座子);Z:Cytoskeleton(细胞骨架)。
2.5.3 KEGG数据库注释
KEGG是一个整合了基因组、化学和系统功能信息的数据库[26]。将Bacillus subtilis HB-011中的基因组数据与KEGG数据库进行对比分析,统计结果如图11所示。结果显示,共注释了42个代谢通路的相关功能基因,共有2 551个编码基因在KEGG pathway中富集到细胞过程、遗传信息处理、人类疾病、有机系统和环境信息的各个功能中。其中占比较大的注释基因分别为:糖蛋白的生物合成和代谢相关基因(837个),碳水化合物代谢相关基因(253个),氨基酸代谢相关基因(214个),信号传导相关基因(202个),膜运输相关基因(180个),辅因子和维生素的代谢相关基因(181个)。这些注释基因在遗传信息处理、人类疾病、有机系统和环境信息的各个功能中发挥着重要的作用。
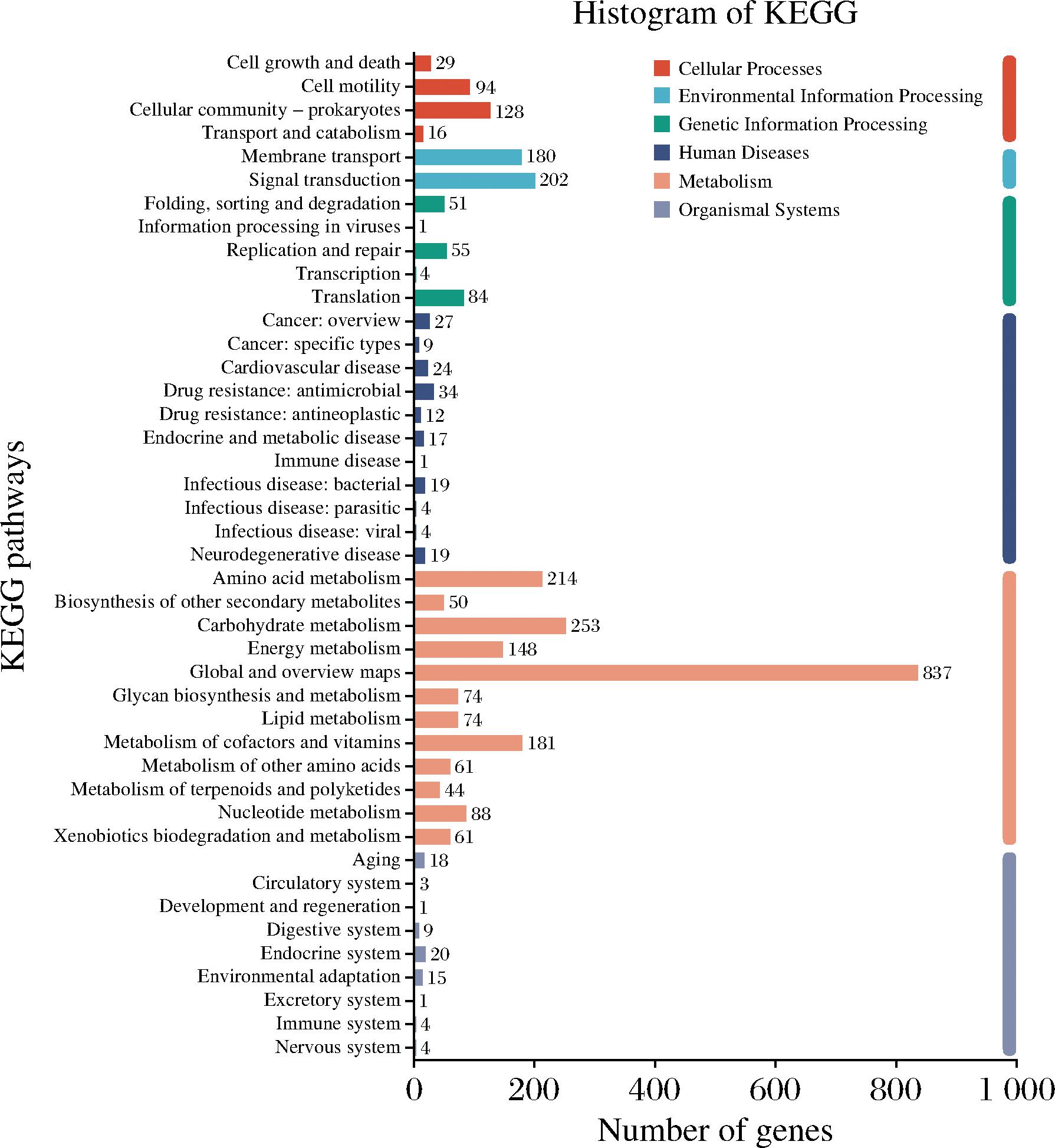
图11 Bacillus subtilis HB-011基因组基因功能KEGG数据库注释
Fig.11 Bacillus subtilis HB-011 genome gene function KEGG database annotation
注:Cell growth and death(细胞生长和死亡);Cell motility(细胞运动性);Cellular community-prokaryotes(细胞群落-原核生物);Transport and catabolism(运输和分解代谢);Membrane transport(膜转运);Signal transduction(信号转导);Folding, sorting and degradation(折叠、分类和退化);Information processing in viruses(病毒中的信息处理);Replication and repair(复制与修复);Transcription(转录);Translation(翻译);Cancer: overview(泛癌);Cancer: specific types(癌症);Cardiovascular disease(心血管疾病);Drug resistance: antimicrobial(耐药性:抗菌药物);Drug resistance: antineoplastic(耐药性:抗肿瘤);Endocrine and metabolic disease(内分泌与代谢病);Immune disease(免疫性疾病);Infectious disease: bacterial(感染性疾病:细菌性);Infectious disease: parasitic(传染病:寄生虫病);Infectious disease: viral(感染性疾病:病毒性);Neurodegenerative disease(神经退行性疾病);Amino acid metabolism(氨基酸代谢);Biosynthesis of other secondary metabolites(其他次级代谢产物的生物合成);Carbohydrate metabolism(碳水化合物代谢);Energy metabolism(能量代谢);Global and overview maps(全局通路图谱);Glycan biosynthesis and metabolism(聚糖的生物合成与代谢);Lipid metabolism(脂代谢);Metabolism of cofactors and vitamins(辅因子和维生素的代谢);Metabolism of other amino acids(其他氨基酸的代谢);Nucleotide metabolism(核苷酸代谢);Xenobiotics biodegradation and metabolism(异生菌的生物降解和代谢);Aging(老化);Circulatory system(循环系统);Development and regeneration(更新与发育);Digestive system(消化系统);Endocrine system(内分泌系统);Environmental adaptation(环境适应);Excretory system(排泄系统);Immune system(免疫系统);Nervous system(神经系统)。
2.5.4 CAZY数据库注释
在CAZY数据库中将Bacillus subtilis HB-011的基因数据进行对比,发现基因组中有99个编码基因的蛋白质结构属于CAZY家族,结果如图12所示。糖基转移酶是数量最多的酶,共有32个,占比为32.3%,这种酶在细胞内的糖代谢过程中起到了重要的调控作用。其次,糖苷水解酶有30个,占比为30.3%,这种酶在生物体内参与了多种生物化学反应,包括食物消化和能量代谢等过程。碳水化合物酯酶有19个,占比为19.2%。
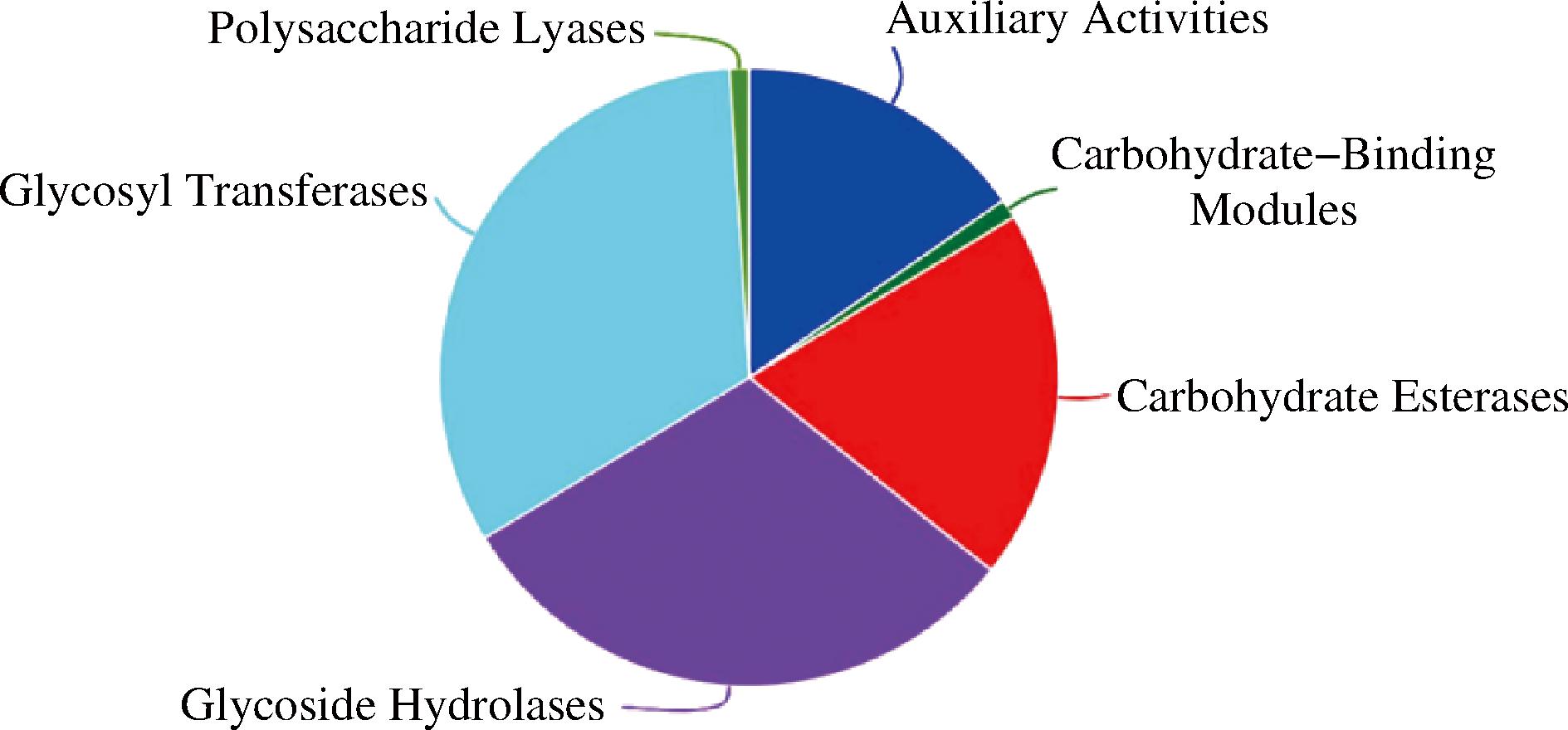
图12 Bacillus subtilis HB-011基因组基因功能CAZY数据库注释
Fig.12 Bacillus subtilis HB-011 genome gene function CAZY database annotation
注:Polysaccharide Lyases(多糖裂解酶);Glycosyl Transferases(糖基转移酶);Glycoside Hydrolases(糖苷水解酶);Auxiliary Activities(辅助功能反应);Carbohydrate-Binding Modules(糖类结合模块);Carbohy-drate Esterases(碳水化合物酯酶)。
2.5.5 Bacillus subtilis HB-011降解木质素机制解析
大多数芳香族化合物会通过常见的中间体儿茶酚或原儿茶酸酯对木质素进行有氧降解。在芳香族化合物的降解方面,β-酮己二酸途径作为一个汇聚途径,在细菌和真菌中普遍存在,整个过程中涉及儿茶酚和原儿茶酸2个分支。在儿茶酸分支中,愈创木酚通过去甲基化转化为邻苯二酚,而在原儿茶酸分支中,4-羟基苯甲醛转化为3,4-二羟基苯甲酸的过程中涉及到了特定的单加氧酶。
如表1和图13所示,β-芳香醚键作为木质素中最丰富的键类型,本研究发现其裂解可能与基因组中发现的谷胱甘肽转移酶和NAD依赖性酒精脱氢酶的基因有关。另外,3-甲基苯酚作为许多化合物的前体,在Bacillus subtilis HB-011中可能通过特定的代谢途径转化为其他化合物。木质素结构中的4-羟基肉桂酸经过特定的连接酶(5个基因)和β-氧化酶催化转化为4-羟基苯甲酸。在Bacillus subtilis HB-011中,4-羟基苯乙酮作为一个重要代谢产物,通过一系列反应转化为具有抗氧化活性和自由基清除能力的代谢物。
表1 Bacillus subtilis HB-011降解木质素代谢相关通路分析
Table 1 Analysis of pathways related to lignin degradation metabolism in Bacillus subtilis HB-011
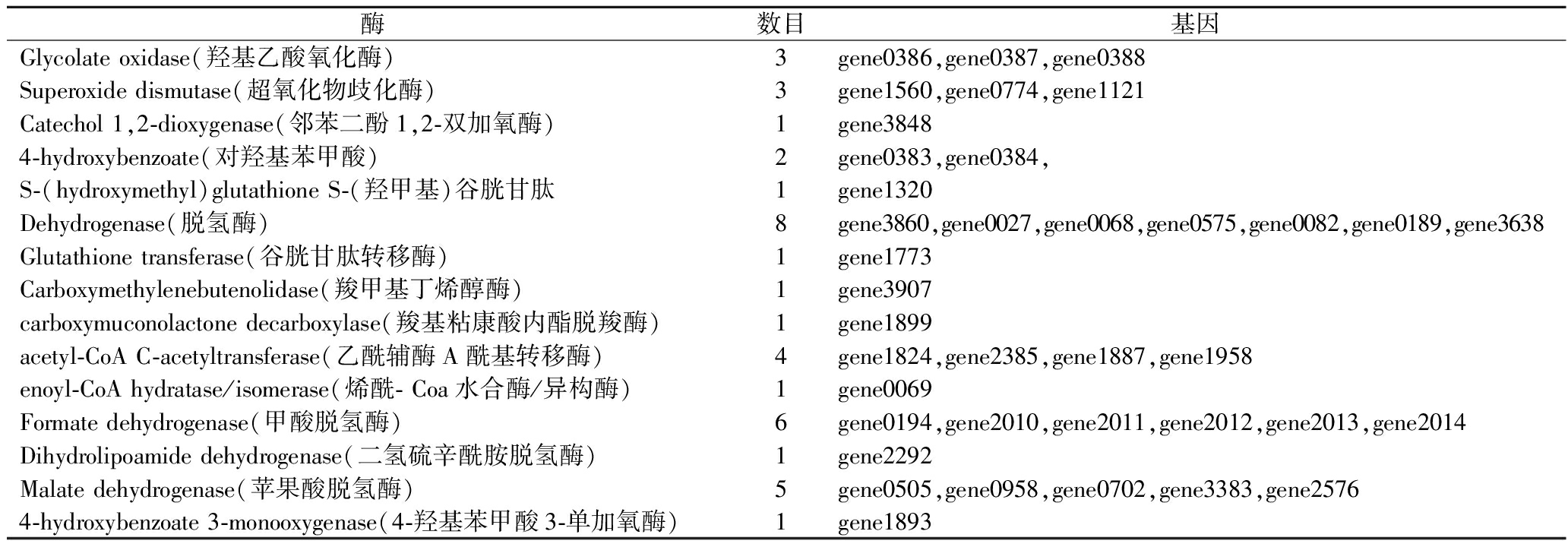
酶数目基因Glycolate oxidase(羟基乙酸氧化酶)3gene0386,gene0387,gene0388Superoxide dismutase(超氧化物歧化酶)3gene1560,gene0774,gene1121Catechol 1,2-dioxygenase(邻苯二酚1,2-双加氧酶)1gene38484-hydroxybenzoate(对羟基苯甲酸)2gene0383,gene0384,S-(hydroxymethyl)glutathione S-(羟甲基)谷胱甘肽1gene1320Dehydrogenase(脱氢酶)8gene3860,gene0027,gene0068,gene0575,gene0082,gene0189,gene3638Glutathione transferase(谷胱甘肽转移酶)1gene1773Carboxymethylenebutenolidase(羧甲基丁烯醇酶)1gene3907carboxymuconolactone decarboxylase(羧基粘康酸内酯脱羧酶)1gene1899acetyl-CoA C-acetyltransferase(乙酰辅酶A酰基转移酶)4gene1824,gene2385,gene1887,gene1958enoyl-CoA hydratase/isomerase(烯酰- Coa水合酶/异构酶)1gene0069Formate dehydrogenase(甲酸脱氢酶)6gene0194,gene2010,gene2011,gene2012,gene2013,gene2014Dihydrolipoamide dehydrogenase(二氢硫辛酰胺脱氢酶)1gene2292Malate dehydrogenase(苹果酸脱氢酶)5gene0505,gene0958,gene0702,gene3383,gene25764-hydroxybenzoate 3-monooxygenase(4-羟基苯甲酸3-单加氧酶)1gene1893
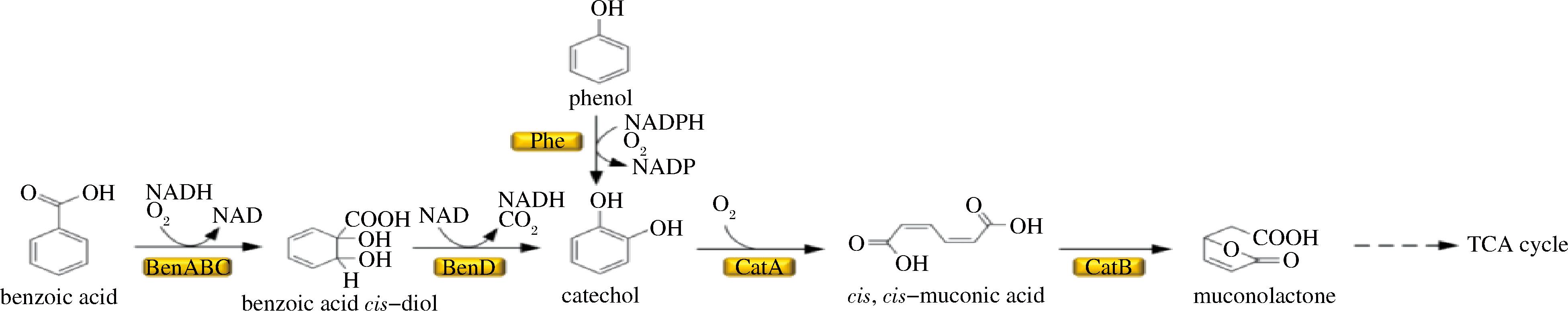
图13 芳香族化合物分解代谢途径[31]
Fig.13 Catabolic pathway of aromatic compounds
注:Benzoic acid:苯甲酸;BenABC:苯甲酸1,2-双加氧酶;Benzoic acid cis-diol:苯甲酸顺式-二醇;Bend:β-半乳糖苷酶;phe:苯丙氨酸羟化酶;Catechol:儿茶酚:CatA:过氧化氢酶;Cis,Cis-muconic acid:粘康酸;CatB:溶体组织蛋白酶B;Muconolactone:粘康酸内酯。
总体而言,木质素合成代谢通路及其相关代谢产物的研究对于理解植物生长发育、植物与环境互作以及木质素生物降解与利用具有重要意义。
2.6 酶学性质
2.6.1 酶促动力学
不同温度和pH对Bacillus subtilis HB-011酶与漆酶对照品酶活性的影响如图14所示,以愈创木酚为底物,漆酶对照品与发酵生产的漆酶酶促反应的最适pH值约为5.0,最适温度约为35 ℃;两者酶活性均随pH、温度的升高先上升后下降,在pH为2~6、温度为20~55 ℃条件下具有较好的稳定性。但是与漆酶对照品相比,发酵生产的漆酶具有更高的酶活性和稳定性。
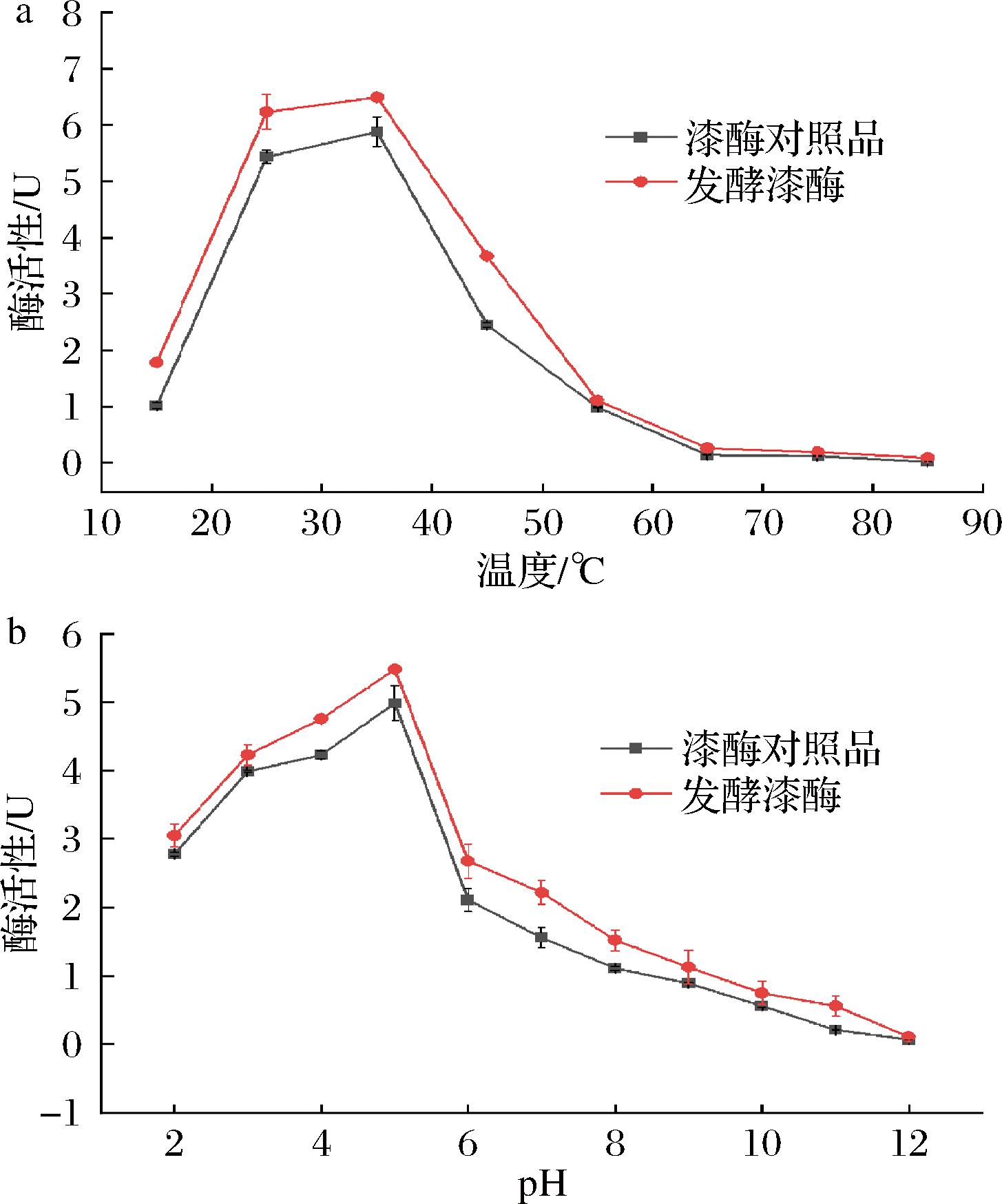
a-温度;b-pH
图14 不同温度和pH对Bacillus subtilis HB-011酶与漆酶对照品酶活性的影响
Fig.14 Effects of different temperatures and pH on the enzyme activities of Bacillus subtilis HB-011 enzyme and laccase controls
漆酶对照品与发酵生产的漆酶的酶促动力学参数拟合结果如图15和表2所示,漆酶对照品与发酵生产的漆酶的米氏常数Km值分别为0.231 5、0.189 5 mmol/L,这表明发酵生产的漆酶对底物愈创木酚的亲和力高于漆酶对照品;漆酶对照品与发酵生产的漆酶最大反应速率Vmax分别为0.016 3、0.016 9 μmol/min,两者催化速率相近;漆酶对照品与发酵生产的漆酶的Kcat分别为0.135 8、0.140 9 s-1,发酵漆酶在最有优条件下酶催化生成底物的速率略高于漆酶对照品。
表2 Bacillus subtilis HB-011酶与酶标准品的酶动力学参数拟合结果
Table 2 Fitting results of enzyme kinetic parameters of Bacillus subtilis HB-011 and enzyme standard

样品Km/(mmol/L)Vmax/(μmol/min)Kcat/s-1R2漆酶对照品0.231 50.016 30.135 80.96发酵漆酶0.189 50.016 90.140 90.95
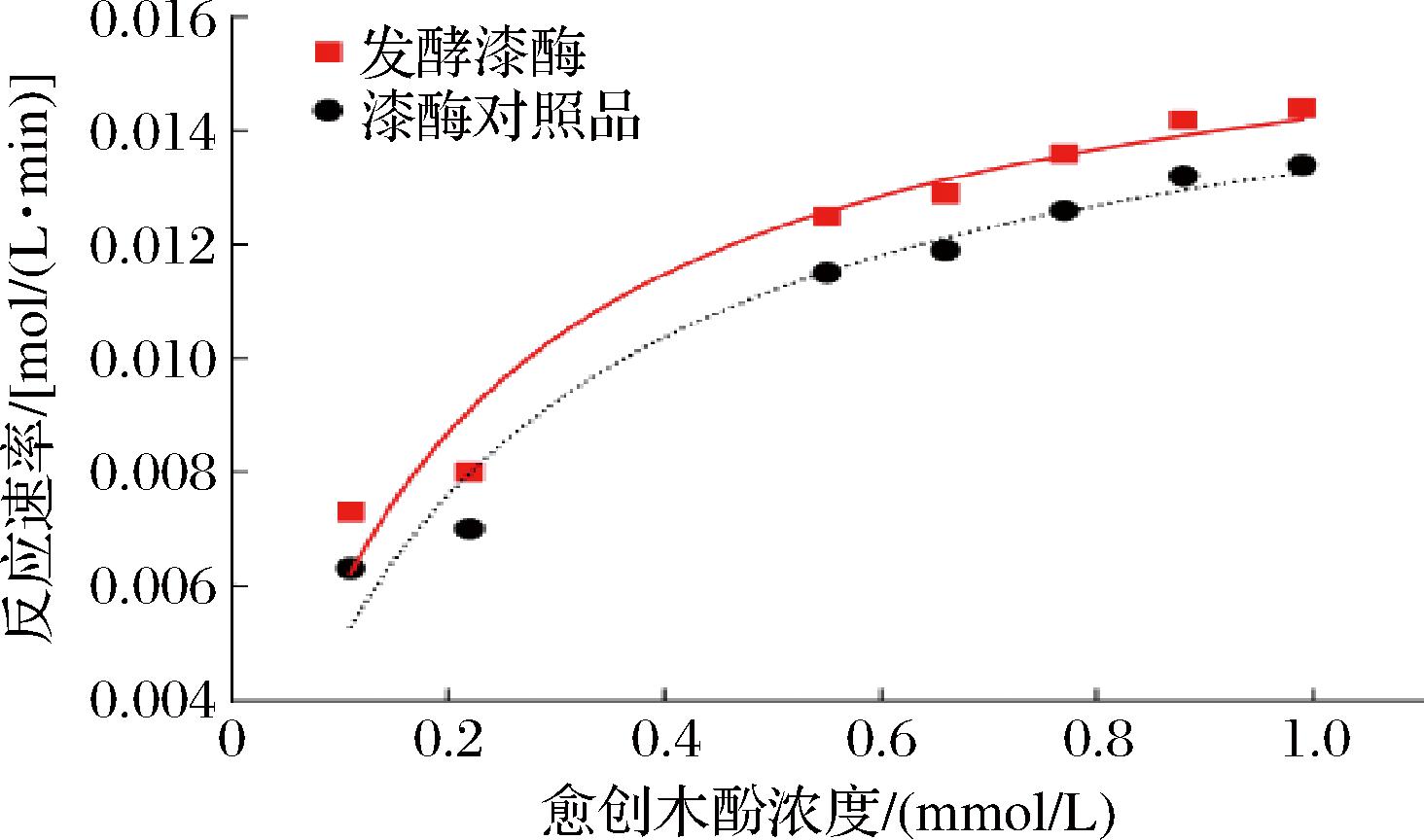
图15 发酵漆酶与漆酶对照品的酶动力学参数米氏方程拟合
Fig.15 Mie equation fitting of enzyme kinetic parameters of fermented laccase and laccase controls
2.6.2 酶的二级结构
如图16和表3所示,发酵漆酶与漆酶对照品都在206 nm附近有一正峰,具有较典型的β-转角;在216~218 nm有一负峰,在185~200 nm有一强的正峰,具有较典型的β-折叠;在198 nm附近有一负峰,在220 nm附近有一小而宽的正峰[32],为无规则蜷曲特征峰,而α-螺旋特征峰较弱。这与CDpro计算得的二级结构含量结果一致。β-折叠的增加也提高了发酵生产的漆酶的有序性,这可能是单因素试验时发酵漆酶热稳定性高于漆酶对照品的主要原因之一[33]。
表3 漆酶对照品与Bacillus subtilis HB-011发酵漆酶的二级结构含量 单位:%
Table 3 Secondary structure content of laccase control and Bacillus subtilis HB-011 fermentation laccase
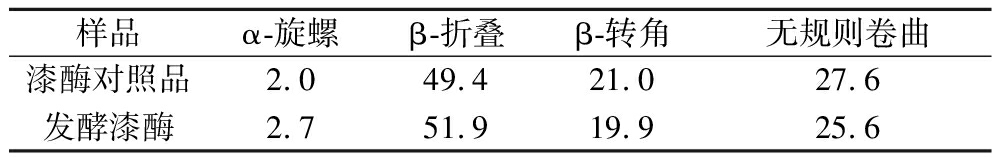
样品α-旋螺β-折叠β-转角无规则卷曲漆酶对照品2.049.421.027.6发酵漆酶2.751.919.925.6
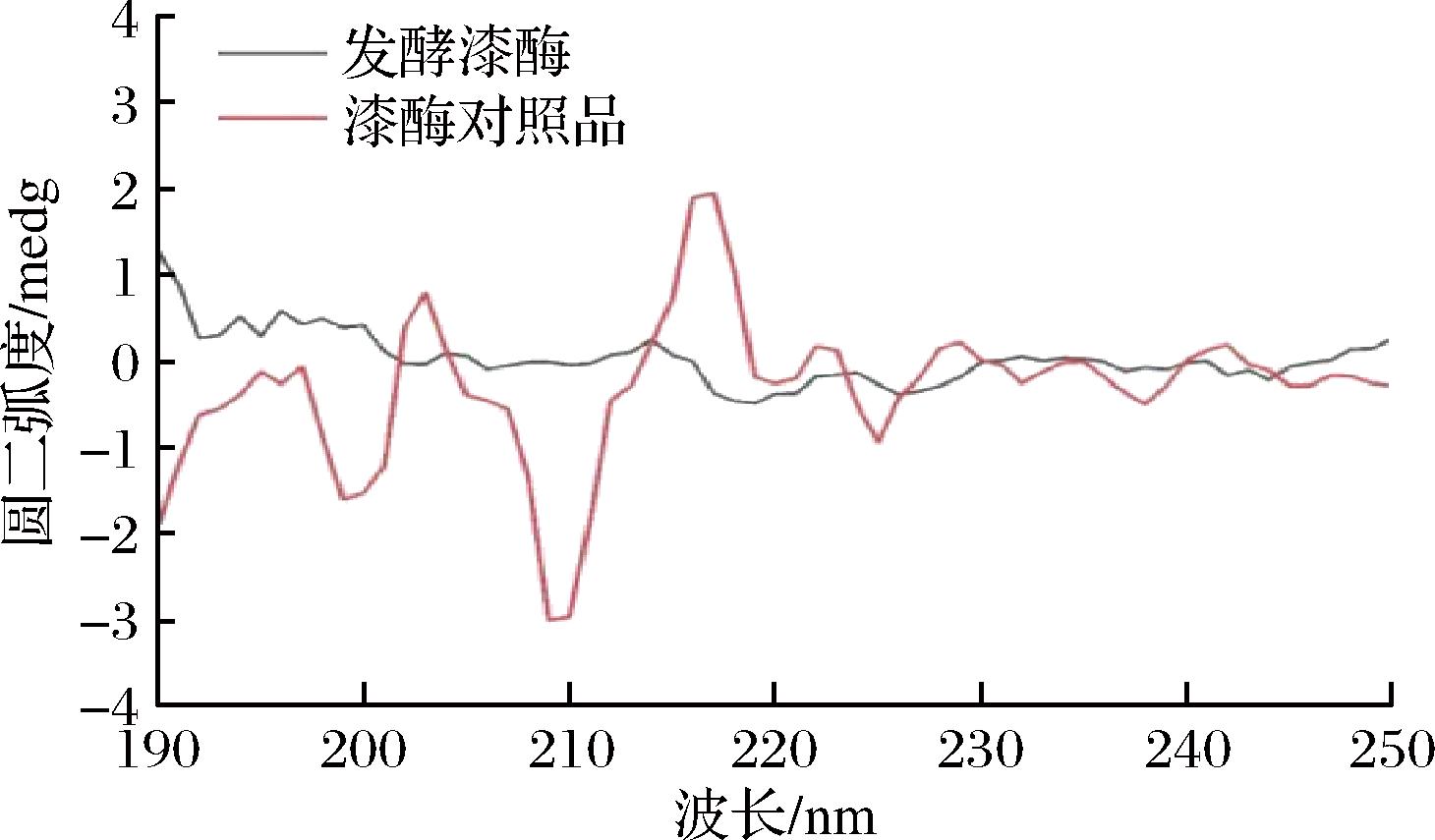
图16 漆酶对照品(固体)与Bacillus subtilis HB-011发酵漆酶(液体)的圆二色光谱图
Fig.16 Circular dichroic spectra of laccase standard (solid) and Bacillus subtilis HB-011 fermentation laccase (liquid)
3 结论与讨论
本研究筛选得到的木质素降解菌Bacillus subtilis HB-011可以产生漆酶、谷胱甘肽转移酶和NAD依赖性酒精脱氢酶等参与木质素降解的相关酶系,木质素的降解率达到了20%。Bacillus subtilis HB-011所产漆酶最适pH为5.0,最适温度为35 ℃;在pH值2~6、温度20~55 ℃条件漆酶活性最高值为6.58 U/L且具有较好的稳定性,其性能优于漆酶对照品,Bacillus subtilis HB-011酶在发酵过程产生的较高酶活性,从Bacillus subtilis HB-011全基因分析中发现的较多相邻关联功能基因(如gene0386,gene0387,gene0388;gene2010,gene2011,gene2012,gene2013,gene2014;gene0068,gene0069)所起的协同作用。本试验将为工业化生产提供优良菌种提供参考。
[1] 黄泰富, 于洪文, 黄利春, 等.提高玉米秸秆饲料化品质的食用菌筛选[J].中国饲料, 2024(3):34-39.
HUANG T F, YU H W, HUANG L C, et al.Screening of edible fungi for improving the feed quality of corn straw[J].China Feed, 2024(3):34-39.
[2] ZAKZESKI J, BRUIJNINCX P C A, JONGERIUS A L, et al.The catalytic valorization of lignin for the production of renewable chemicals[J].Chemical Reviews, 2010, 110(6):3552-3599.
[3] RAGAUSKAS A J, BECKHAM G T, BIDDY M J, et al.Lignin valorization:Improving lignin processing in the biorefinery[J].Science, 2014, 344(6185):1246843.
[4] THOLL D.Biosynthesis and biological functions of terpenoids in plants[J].Advances in Biochemical Engineering/Biotechnology, 2015, 148:63-106.
[5] BUGG T D, AHMAD M, HARDIMAN E M, et al.The emerging role for bacteria in lignin degradation and bio-product formation[J].Current Opinion in Biotechnology, 2011, 22(3):394-400.
[6] 温学鹏. 枯草芽孢杆菌降解纤维素的作用及纤维素酶基因过表达载体的构建[D].哈尔滨:东北农业大学, 2019.
WEN X P.Degradation of cellulose by Bacillus subtilis and construction of Bacillus subtilis overexpressing vector of cellulase gene[D].Harbin:Northeast Agricultural University, 2019.
[7] 刘高强, 李新鹏, 刘文钊, 等.一株高效降解血液蛋白的枯草芽孢杆菌NWMCC0137全基因组测序及分析[J].江苏农业学报, 2023, 39(7):1460-1471.
LIU G Q, LI X P, LIU W Z, et al.Whole genome sequencing and analysis of Bacillus subtilis NWMCC0137, an efficient blood protein degrading strain[J].Jiangsu Journal of Agricultural Sciences, 2023, 39(7):1460-1471.
[8] 赵羽, 王冰, 李燕.防病枯草芽孢杆菌GLB191的全基因组测序与比较基因组分析[C].中国植物病理学会2023年学术年会论文集.北京:中国农业科学技术出版社, 2023:669-670.
ZHAO Y, WANG B, LI Y.Whole genome sequencing and comparative genome analysis of disease-preventive Bacillus subtilis GLB191[C].Proceedings of the Annual Meeting of Chinese Society for Plant Pathology (2023).Beijing:China Agricultural Science and Technology Press, 2023.
[9] 王骞, 包慧芳, 丁荣荣, 等.生防菌Bacillus subtilis DNKAS全基因组测序及生物膜形成相关基因分析[J].草食家畜, 2022(5):56-64.
WANG Q, BAO H F, DING R R, et al.Whole genome sequencing and genes related to biofilm formation analysis of Bacillus subtilis DNKAS with biocontrol activity[J].Grass-Feeding Livestock, 2022(5):56-64.
[10] TOYMENTSEVA A A, MASCHER T, SHARIPOVA M R.Regulatory characteristics of Bacillus pumilus protease promoters[J].Current Microbiology, 2017, 74(5):550-559.
[11] CONTESINI F J, DE MELO R R, SATO H H.An overview of Bacillus proteases:From production to application[J].Critical Reviews in Biotechnology, 2018, 38(3):321-334.
[12] BENSON G.Tandem repeats finder:A program to analyze DNA sequences[J].Nucleic Acids Research, 1999, 27(2):573-580.
[13] 赵敏, 钱程.白腐菌木素氧化酶系的检测及其漆酶诱导产生的研究[J].中国造纸学报, 2005, 20(2):101-105.
ZHAO M, QIAN C.Study on the examination of oxidative system in white-rot fungi and its laccase production by induction[J].Transactions of China Pulp and Paper, 2005, 20(2):101-105.
[14] KUMARASAMY M, IN-HYUN N, YOUNG-MO K, et al. Decolorization of reactive dyes by a thermostable laccase produced by Ganoderma lucidum in solid state culture[J].Enzyme and Microbial Technology, 2007,40:1662-1672.
[15] ZHAO C, XIE S X, PU Y Q, et al.Synergistic enzymatic and microbial lignin conversion[J].Green Chemistry, 2016, 18(5):1306-1312.
[16] 东秀珠, 蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社, 2001.
DONG X Z, CAI M Y.Handbook of systematic identification of common bacteria[M].Beijing:Science Press, 2001.
[17] 布坎南R E, 吉本斯 N E.伯杰细菌鉴定手册[M].北京:科学出版社, 1994.
BUCHANAN R E, GIBBONS N E.Berger’s handbook of bacterial identification[M].Beijing:Science Press, 1994.
[18] KIM S B, YOON J H, KIM H, et al.A phylogenetic analysis of the genus Saccharomonospora conducted with 16S rRNA gene sequences[J].International Journal of Systematic Bacteriology, 1995, 45(2):351-356.
[19] SAITOU N, NEI M.The neighbor-joining method:A new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution, 1987, 4(4):406-425.
[20] LUO R B, LIU B H, XIE Y L, et al.SOAPdenovo2:An empirically improved memory-efficient short-read de novo assembler[J].GigaScience, 2012, 1(1):18.
[21] BESEMER J, LOMSADZE A, BORODOVSKY M.GeneMarkS:A self-training method for prediction of gene starts in microbial genomes.Implications for finding sequence motifs in regulatory regions[J].Nucleic Acids Research, 2001, 29(12):2607-2618.
[22] LAGESEN K, HALLIN P, RØDLAND E A, et al.RNAmmer:Consistent and rapid annotation of ribosomal RNA genes[J].Nucleic Acids Research, 2007, 35(9):3100-3108.
[23] CHAN P P, LOWE T M.tRNAscan-SE Searching for tRNA genes in genomic sequences[M].Clifton:Humana Press, 2019.
[24] STOTHARD P, WISHART D S.Circular genome visualization and exploration using CGView[J].Bioinformatics, 2005, 21(4):537-539.
[25] TATUSOV R L, KOONIN E V, LIPMAN D J.A genomic perspective on protein families[J].Science, 1997, 278(5338):631-637.
[26] CONSORTIUM T G O.The gene ontology resource:20 years and still GOing strong[J].Nucleic Acids Research, 2019, 47(D1):D330-D338.
[27] OGATA H, GOTO S, SATO K, et al.KEGG Kyoto encyclopedia of genes and genomes[J].Nucleic Acids Research, 1999, 27(1):29-34.
[28] LOMBARD V, GOLACONDA RAMULU H, DRULA E, et al.The carbohydrate-active enzymes database (CAZy) in 2013[J].Nucleic Acids Research, 2014, 42(D1):D490-D495.
[29] JIA B F, RAPHENYA A R, ALCOCK B, et al.CARD 2017:Expansion and model-centric curation of the comprehensive antibiotic resistance database[J].Nucleic Acids Research, 2017, 45(D1):D566-D573.
[30] LIU B, ZHENG D D, ZHOU S Y, et al.VFDB 2022:A general classification scheme for bacterial virulence factors[J].Nucleic Acids Research, 2022, 50(D1):D912-D917.
[31] BECKER J, KUHL M, KOHLSTEDT M, et al.Metabolic engineering of Corynebacterium glutamicum for the production of cis, cis-muconic acid from lignin[J].Microbial Cell Factories, 2018, 17(1):115.
[32] LAKKIS J, VILLOTA R.Effect of acylation on substructural properties of proteins:A study using fluorescence and circular dichroism[J].Journal of Agricultural and Food Chemistry, 1992, 40(4):553-560.
[33] 朱彩林, 吕祥, 夏小乐.盖子区域氨基酸的定点突变对T1脂肪酶酶学性质的影响[J].生物技术通报, 2020, 36(11):94-102.
ZHU C L, LYU X, XIA X L.Effect of site-directed mutagenesis of amino acids in lid region on the enzymatic properties of T1 lipase[J].Biotechnology Bulletin, 2020, 36(11):94-102.