格氏乳球菌(Lactococcus garvieae)是一种革兰氏阳性、非产气、非运动性的乳酸菌,属于乳链球菌属(Lactococcus),在自然界中广泛存在于发酵食品、水体以及动物体内[1]。在白酒酿造体系中,格氏乳球菌是一类重要的功能微生物[2],主要存在于大曲[3]、酒醅[4]、窖泥[5]等发酵环境。格氏乳球菌除了产生大量乳酸外,还会产生酯类等风味物质,既可以充分提升白酒风味、香气感官特征,又可以抑制发酵初期部分杂菌的生长。格氏乳球菌对维持酿造体系的生态环境起着重要作用[2],对丰富白酒的风味和口感至关重要[6]。
目前,随着生物信息学的蓬勃兴盛,对白酒环境微生物的研究也从传统生理生化实验转向基于基因水平揭示白酒酿造微生物组成、结构、功能、代谢机制等信息[7]。基因组学技术以其通量高、规模大等特点为研究复杂多样的微生物提供了新的手段[8]。对参与酿造的微生物基因组进行深度测序,成为架起酿造复杂微生物与风味成分之间的第一道桥梁[9]。
泛基因组的概念是TETTELIN等[10]在2005年提出的,其定义为一个物种全部基因组成。该物种所有样本共有的基因称为核心基因,一般与物种生物学功能和主要表型特征相关;仅在物种部分样本中存在的基因称为附属基因,一般与物种对特定环境的适应性或特有的生物学特征相关,反映了部分物种的特性;某一样本特有的基因称为个体特异性基因,揭示了该个体的特异性状。因此,通过分析L.garvieae泛基因组有助于了解该物种的群体特征及个体差异[11],为其遗传多样性和进化提供新的见解。
在此之前,已从白酒窖泥中分离到了4株L. garvieae,本文将对这4株L.garvieae进行全基因组测序和功能基因组注释。通过对这4株菌的基因组数据结合公共数据库中其他生态位的L.garvieae基因组数据,对L.garvieae进行了泛基因组分析,以期深入了解该菌株的基因组结构、功能特征以及代谢多样性,探讨其系统发育关系及进化机制。通过泛基因组分析,为L.garvieae进一步开发应用提供新的理论基础。
1 实验材料
1.1 实验所需格氏乳球菌信息收集
前期实验室从四川省宜宾市某浓香型酒厂A的窖泥中分离、鉴定得到1株L.garvieae,编号BRM002,本次从四川省绵阳市某浓香型酒厂B的窖泥中分离、鉴定得到3株L.garvieae,编号为J_1、J_3_15、J_3_6。
从NCBI Assembly公共数据库中下载获得78株L.garvieae基因组信息(截止2024年3月10日)。
1.2 主要软件与数据库
本中用到的主要软件与数据库如表1所示。
表1 本研究所用的主要软件及数据库
Table 1 Main software and database used in this study
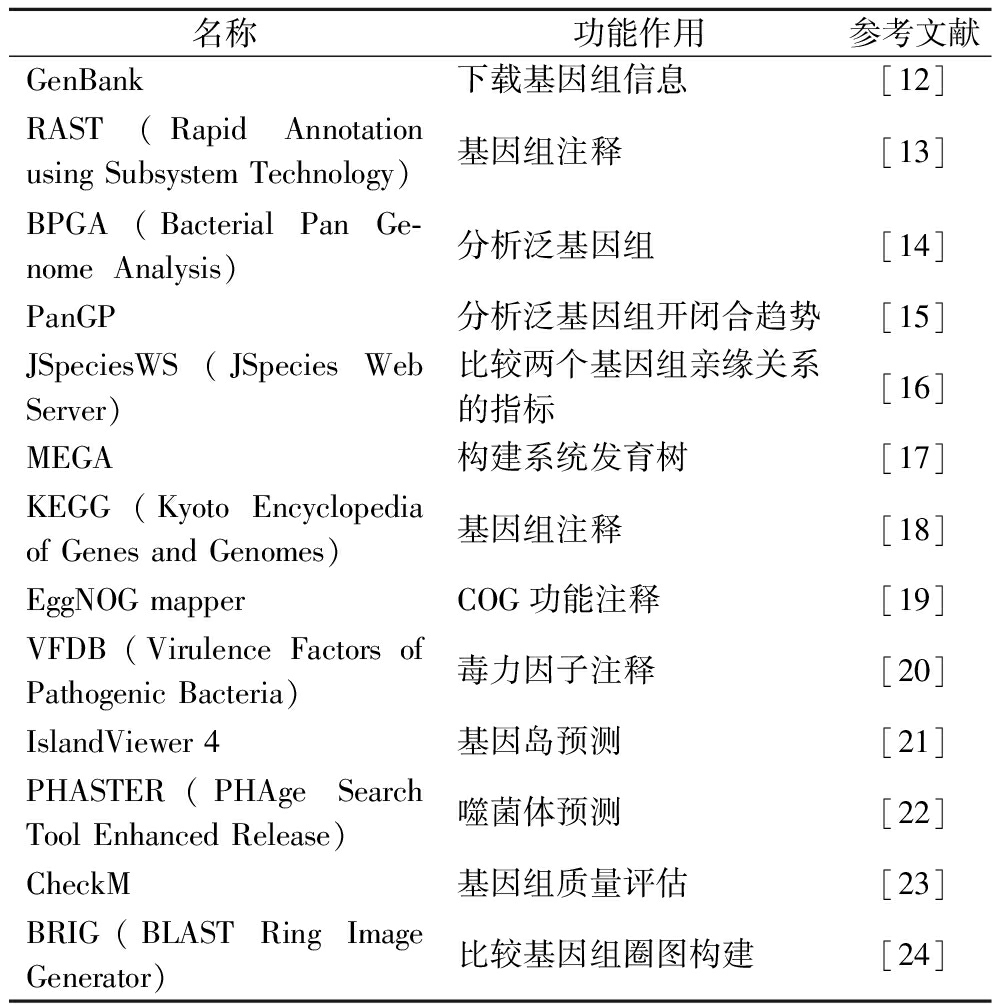
名称功能作用参考文献GenBank下载基因组信息[12]RAST (Rapid Annotation using Subsystem Technology)基因组注释[13]BPGA (Bacterial Pan Ge-nome Analysis)分析泛基因组[14]PanGP分析泛基因组开闭合趋势[15]JSpeciesWS (JSpecies Web Server)比较两个基因组亲缘关系的指标[16]MEGA构建系统发育树[17]KEGG (Kyoto Encyclopedia of Genes and Genomes)基因组注释[18]EggNOG mapperCOG功能注释[19]VFDB (Virulence Factors of Pathogenic Bacteria)毒力因子注释[20]IslandViewer 4基因岛预测[21]PHASTER(PHAge Search Tool Enhanced Release)噬菌体预测[22]CheckM基因组质量评估[23]BRIG(BLAST Ring Image Generator)比较基因组圈图构建[24]
2 实验方法
2.1 数据采集与基因注释
实验室共分离和鉴定出4株L.garvieae,其中BRM002在前期研究中已完成基因组测序和注释,本研究对剩下3株L.garvieae进行基因组测序。主要步骤包括:提取菌株DNA,采用全基因组鸟枪法策略,构建不同插入片段的文库,利用第二代测序技术,基于 Illumina NovaSeq测序平台,对文库进行双末端测序。对下机数据去除接头污染,进行质量过滤,将测序数据进行从头拼装并将结果进行评估和比较,使用 Pilon进行碱基校正,得到全基因组序列。BRM002、J_1、J_3_15、J_3_6基因组信息已上传至本实验室白酒微生物基因组数据库(https://mgdbaijiu.com/),编号分别为MGD-G145、MGD-G88、MGD-G108、MGD-G113。
另外从NCBI中下载获得78株基因组组装信息(截止2024年3月10日)。利用checkM[23]评估基因组质量,剔除其中基因组完整度<90%,污染度>10%的基因组(共4株)。通过JSpeciesWS[16]进行ANI计算,注意到有5株菌株EP01、A1、DCC43、FDAARGOS_893、feces_ps_bin.21与NCBI标准菌株ATCC49156的ANI值分别只有82.25%、80.85%、80.71%、81.1%、80.57%,明显低于定义同一物种的95%分界值,表明该菌株可能在NCBI中被错误分类,后续分析将去除这5株菌株。最终一共73株L.garvieae基因组数据用于后续分析(见电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.039507))。使用RAST[13]软件对所有下载的基因组核酸序列进行了注释,得到编码蛋白序列文件。利用BRIG完成比较基因组圈图构建。
2.2 泛基因组分析
使用BPGA软件对L.garvieae进行泛基因组分析与计算。同时利用PanGP软件绘制L.garvieae基因组的稀释曲线,分析泛基因组的开闭合趋势。
2.3 系统发育分析
提取L.garvieae基因组共有保守蛋白的基因序列(核心基因,相似度≥50%,覆盖度≥70%)。之后将串联的序列导入MEGA X[17],选取最大似然法构建系统发育树,探究不同菌株系统发育关系。最后用FigTree(http://tree.bio.ed.ac.uk/software/figtree/)对系统发育树作图进行美化。
2.4 基因功能注释
使用eggNOG-mapper对L.garvieae泛基因组进行蛋白质直向同源簇(COG)注释。采用KEGG自动注释服务器[25]对L.garvieae泛基因组进行在线的代谢通路注释,下载代谢通路的结果,再着重统计核心基因、附属基因和特异基因在各个代谢途径中数量和分布,并分析这些功能基因的具体作用机制。
2.5 毒力因子注释
使用白酒窖泥环境4株L.garvieae菌株的全基因组注释蛋白序列与毒力因子数据库(virulence factors of pathogenic bacteria database,VFDB)进行比对,以此来确定4株L.garvieae是否含有毒力基因,确立其窖泥发酵过程中的应用。
2.6 原噬菌体序列和基因组岛预测
对4株L.garvieae的基因岛和原噬菌体两种移动元件进行了预测。采用IslandViewer4软件预测了L.garvieae中的基因岛,该软件涉及3种方法:SIGI-HMM、IslandPath-DIMOB和IslandPick[21]。使用PHASTER软件识别L.garvieae基因组中完整和不完整的原噬菌体区域[22]。
3 结果与分析
3.1 泛基因组分析结果
根据统计,73个L.garvieae基因组大小在1.89~2.27 Mb,平均大小为2.09 Mb,蛋白编码基因的数量在1 702~2 307个,GC含量为38%~39.5%。基于L.garvieae J_3_6构建73株菌的比较基因组圈图,如图1所示。

图1 L.garvieae比较基因组圈图
Fig.1 Comparative genomic circle map of L.garvieae
对L.garvieae基因组进行直系同源基因簇的识别并构建泛基因组。73个基因组中的所有蛋白质编码基因被归类到6 348个基因簇。其中,在所有基因组中均存在的基因簇有1 182个,构成了L.garvieae的核心基因组,这些基因代表了L.garvieae最基础的代谢和生理特征。存在于两个或多个基因组中,但并非存在所有的73个基因组中的基因簇即附属基因组,共计3 316个。仅出现在单个基因组中的基因簇为菌株特异性基因,数目从0到298个不等。
L.garvieae的总基因簇数量与分析基因组个数拟合曲线如图2所示。泛基因组中包含的基因簇总数随新加入的基因组变化数目通过以下拟合公式描述:y=1 549.35x0.31+261.29(R2=0.999);核心基因组的数目变化通过以下拟合公式描述:y=480.94e-0.05x+1 201.11(R2=0.905)。
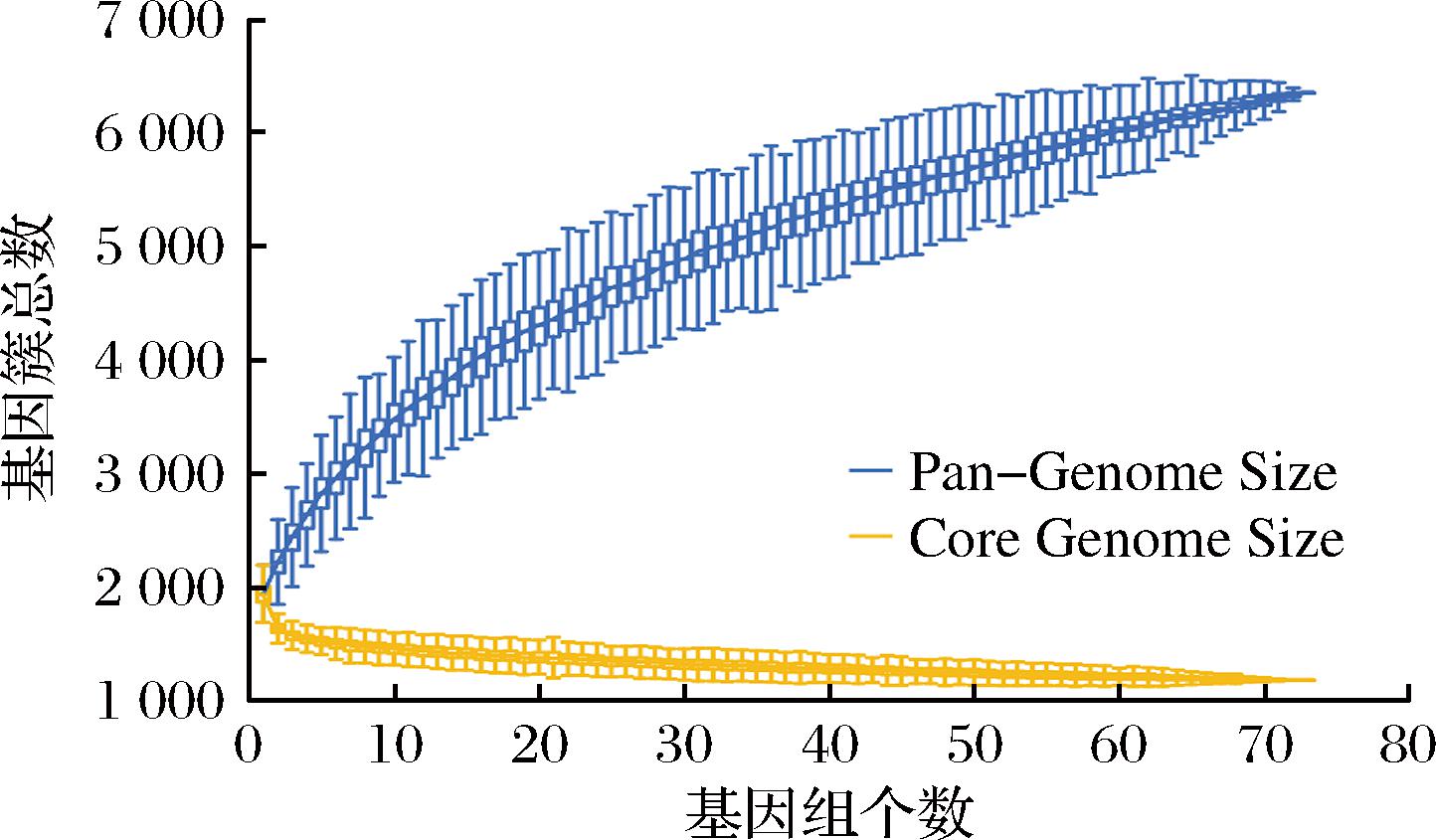
图2 L.garvieae泛基因组拟合特征曲线
Fig.2 L.garvieae pangenome fitting characteristic curve
由图2可知,泛基因组曲线的拟合指数为正,并且总基因簇呈现指数增加趋势,表明L.garvieae的泛基因组是开放的,即每个添加的基因组将会贡献新的基因簇,并增加泛基因组中的基因数量。泛基因组的开放意味着物种具有通过水平基因转移等方式获得外源基因的能力。
3.2 L.garvieae系统发育分析
为了分析L.garvieae菌株的系统发育关系,基于串联的核心基因比对构建了系统发育树,如图3所示。
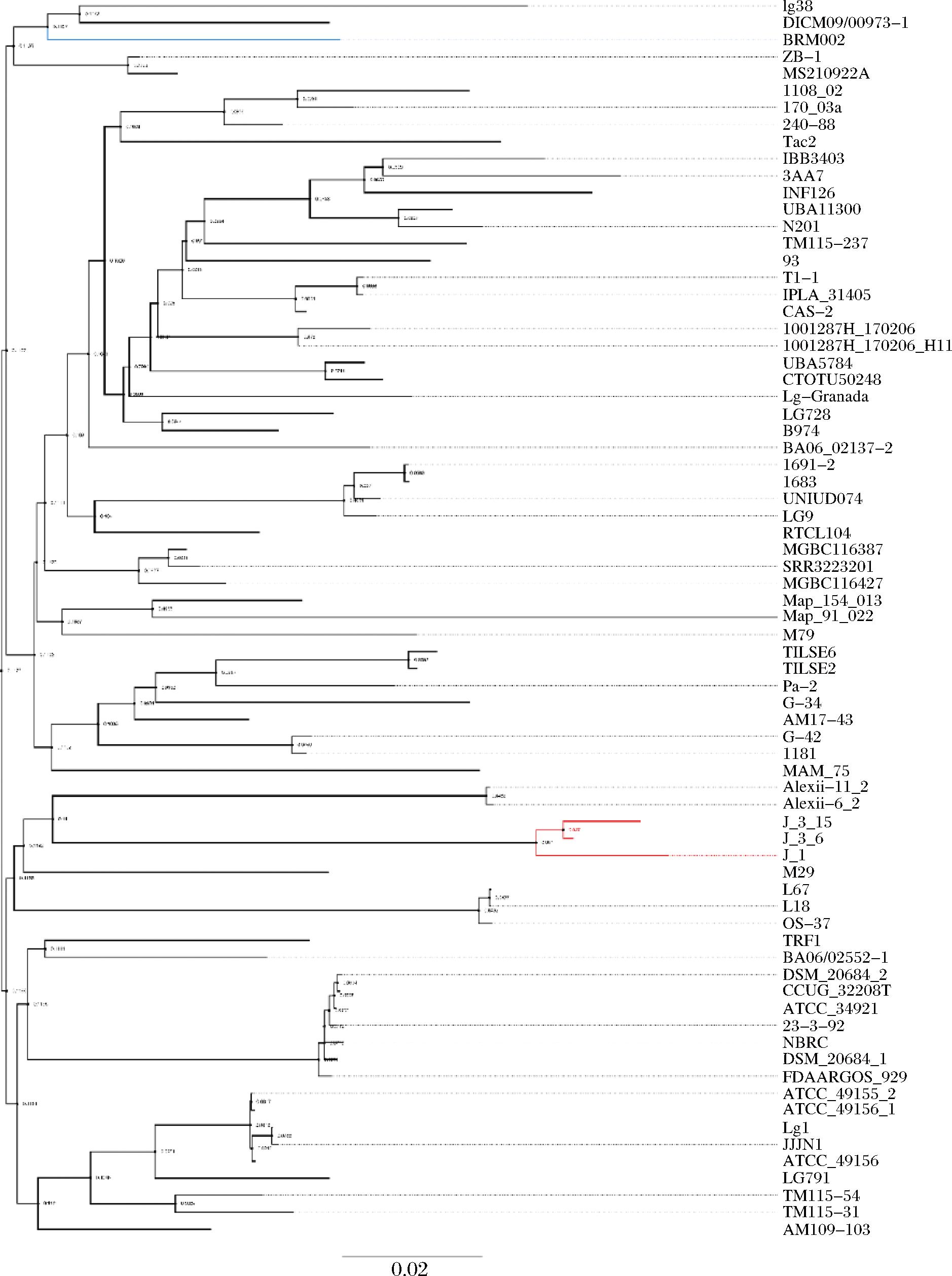
图3 L.garvieae系统发育树
Fig.3 L.garvieae phylogenetic trees
在系统发育树中,来自窖泥的菌株J_1、J_3_6和J_3_15汇聚在一条分支上。经JSpeciesWS计算,J_1与J_3_15的ANI值达到99.97%,J_3_6与J_3_15的ANI值也达到99.93%。从分离来源来看,菌株 J_1、J_3_6和J_3_15都来自白酒窖泥,它们的进化环境大致相似。实验室另外一株L.garvieae BRM002与L.garvieae lg38及L.garvieae DICM09/00973-1在1个进化分支,BRM002与lg38、DICM09/00973-1的ANI值分别为98.16%与98.06%,而L.garvieae lg38分离自日本豆腐中,L.garvieae DICM09/00973-1分离自生活环境中。与其相邻的另一支中的ZB-1分离自海水中常见的大黄鱼、MS210922A分离自日本琥珀鲳鱼,这表明L.garvieae广泛存在于各种生态系统中。BRM002与J_1、J_3_6和J_3_15分支较远,表明他们的基因组在进化上相距较远。虽然他们都同处于白酒窖泥环境下,但是可能由于地理环境的差异等因素导致物种在进化过程中发生分化和变异,显示出基因遗传差异。
3.3 L.garvieae功能注释
通过使用EggNOG mapper和KEGG数据库,对L.garvieae菌株进行了功能富集分析。通过 COG注释结果(图4),一般功能预测(R)(37.81%)是菌株核心基因组、附属基因组和特异性基因的最大类别,分别占12.66%、13.29%和11.85%。这个类别主要是针对功能尚未明确或者具有多种潜在功能的基因,但具体的功能还需要通过更深入的研究来确定。针对核心基因组而言,主要的功能性COG类别包括翻译、核糖体结构和生物合成(J)(9.57%),复制、重组和修复(L)(6.94%),氨基酸转运和代谢(E)(6.94%)、转录(K)(6.85%)、碳水化合物转运代谢(G)(6.28%)、无机离子转运代谢(P)(6.28%)、核苷酸转运和代谢(F)(5.16%)。这些类别在核心基因组中的占比较高,说明这些基因对于菌株的生命活动至关重要。
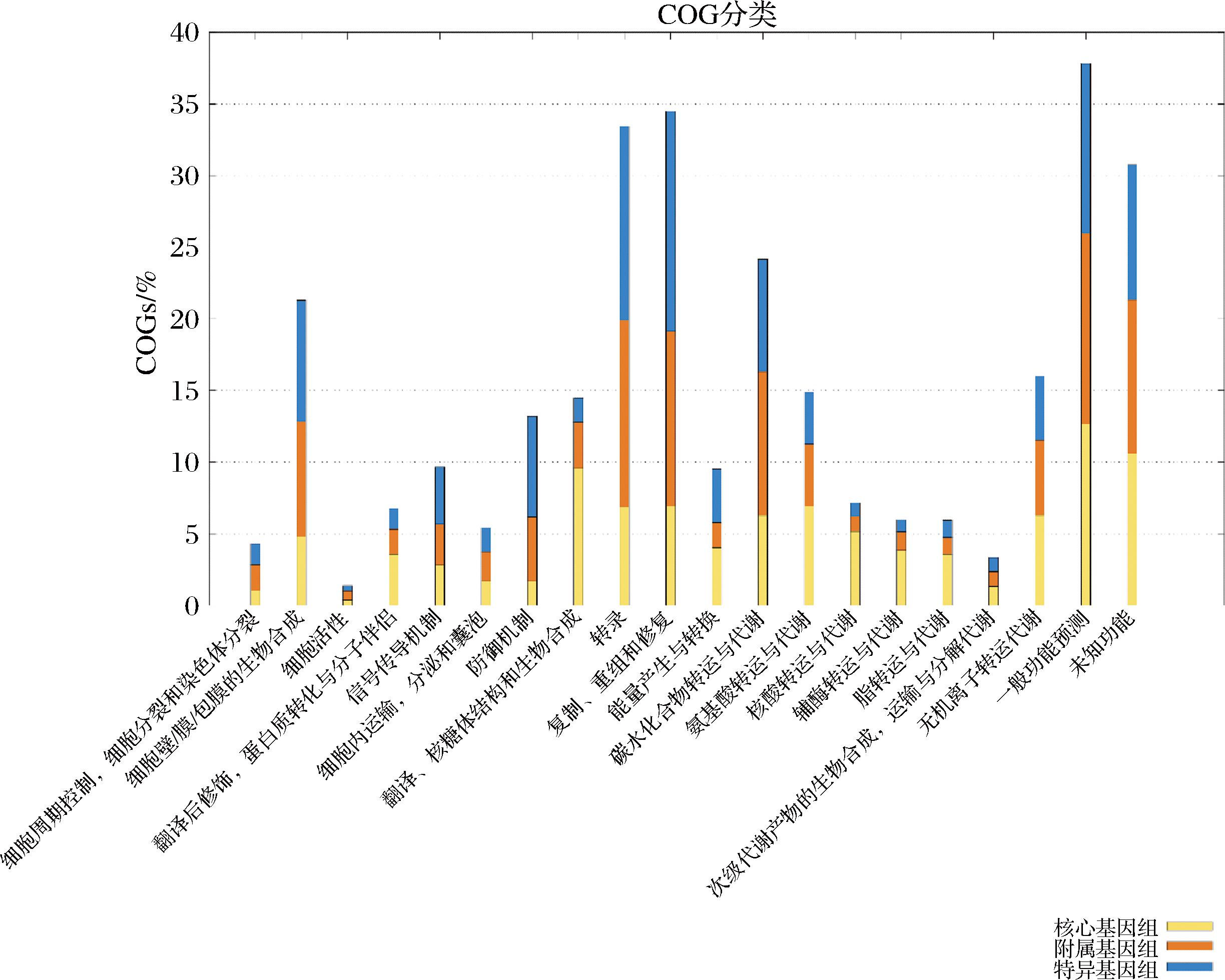
图4 L.garvieae菌株的核心基因组、附属基因组和菌株特异性基因之间的 COG 类别分布
Fig.4 Distribution of COG classes between the core genome, accessory genome, and strain-specific genes of the L.garvieae strain
对于附属基因组,COG注释显示,最大的类别是主要功能预测(R)(13.29%)、转录(K)(13.06%)和复制、重组和修复(L)(12.19%)。这表明这几个功能性COG类别的基因拥有较高的数量和重要性。在特异基因组中,COG注释中最大的类别是复制、重组和修复(L)(15.35%)、转录(K)(13.54%)和一般功能预测(R)(11.85%)。这说明这些特异基因组中的基因可能与菌株的特异功能和生物活动有关。结果显示,不同功能性COG类别在不同类别基因组中的占比存在差异,反映了菌株在基因功能分布上的特异性和多样性。
基于KEGG注释结果,L.garvieae基因组功能主要分布在碳水化合物代谢、核苷酸代谢、膜蛋白运输、氨基酸代谢中。KEGG代谢途径中基因数量的分布如图5所示。碳水化合物代谢途径中注释出的基因占比最高,其中核心基因组注释到124个、附属基因组注释到147个、特异基因组注释到53个。在核心基因组所有代谢通路中,嘌呤代谢(45)、嘧啶代谢(38)、碳代谢(38)通路注释到的基因个数最多,这些代谢都是生物体内的基础代谢过程,共同维持生物体内的代谢平衡和生命活动。
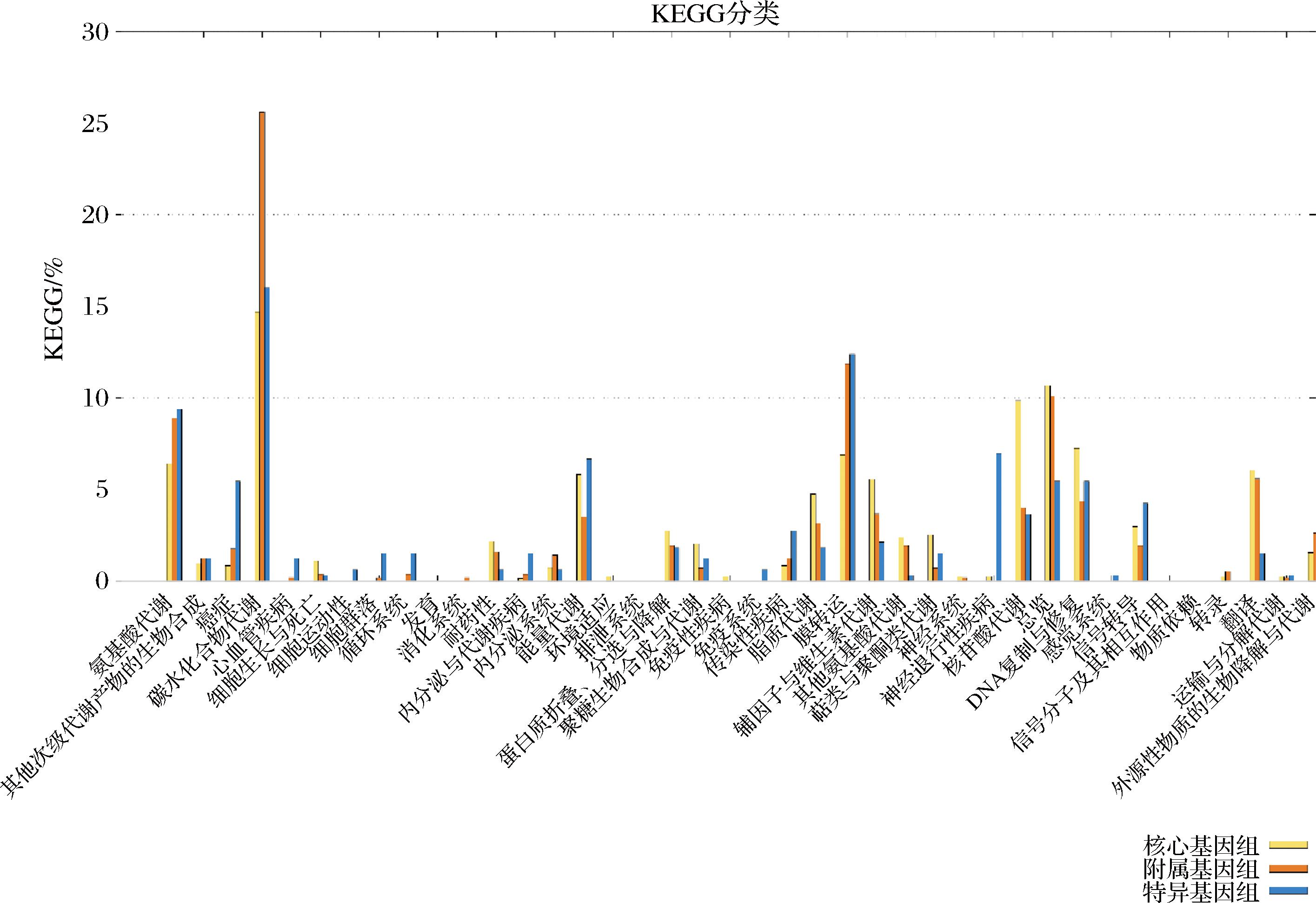
图5 L.garvieae菌株的核心基因组、附属基因组和菌株特异性基因之间的KEGG类别分布
Fig.5 Distribution of KEGG classes between the core genome, accessory genome, and strain-specific genes of the L.garvieae strain
基于4株白酒环境L.garvieae注释出的KO(KEGG Orthology)分析其基因组的表达产物。在白酒环境下,L.garvieae 能够产生淀粉酶、糖化酶、纤维素酶等酶类,同时为发酵产风味物质提供前体物质乳酸、乙酸、乙醇等,促进酯类物质产生。基于注释结果构建4株白酒环境L.garvieae的代谢途径(图6)。
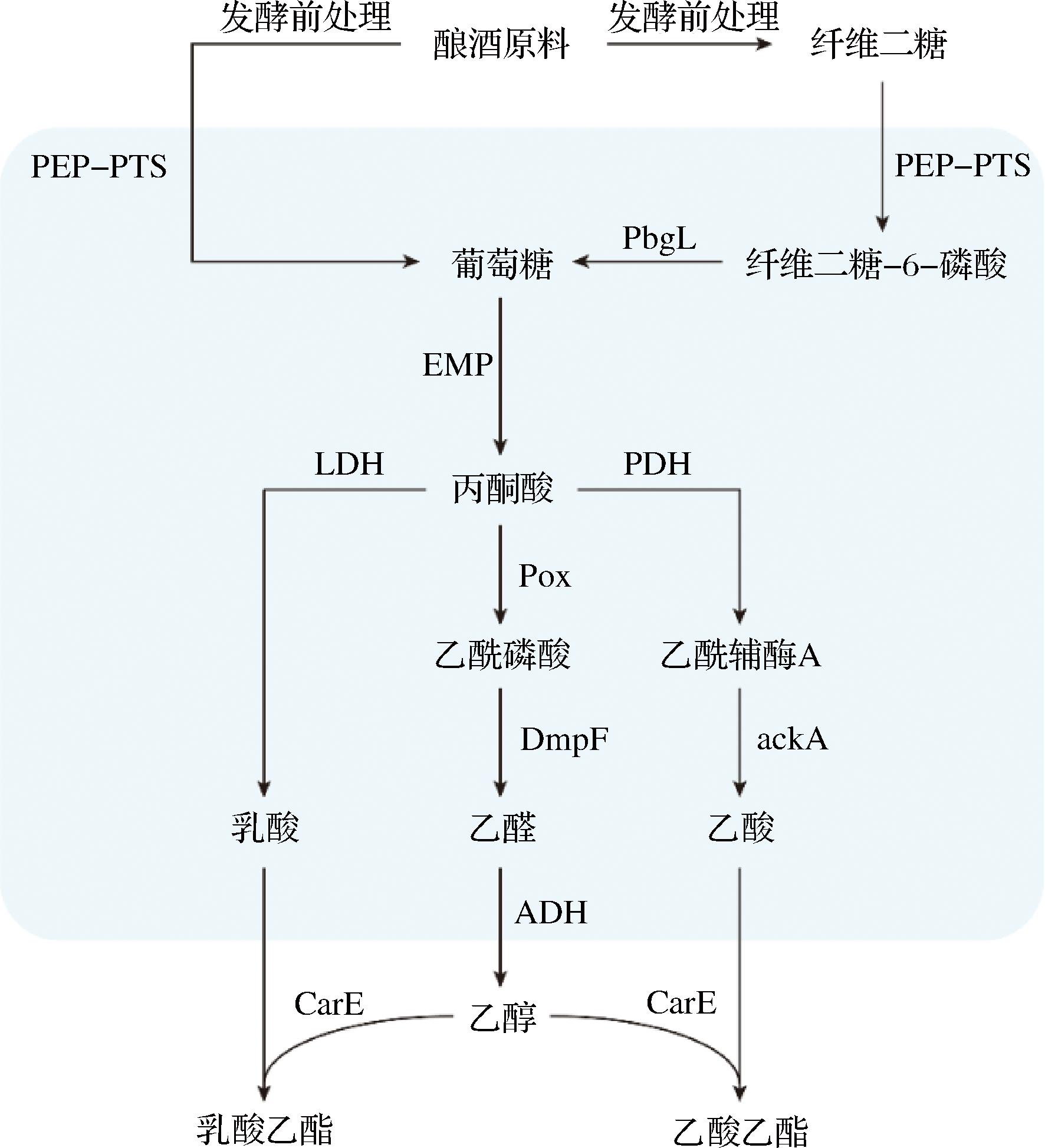
图6 L.garvieae的产酯代谢途径
Fig.6 The ester production pathway of L.garvieae
注:PEP-PTS:磷酸烯醇式丙酮酸-糖磷酸转移酶系统;PbgL:6-磷酸-β-葡萄糖苷酶;EMP:糖酵解途径;LDH:乳酸脱氢酶;PDH:丙酮酸脱氨酶;ackA:乙酸激酶;DmpF:乙醛脱氢酶;ADH:乙醇脱氢酶;Pox:丙酮酸氧化酶;CarE:羧酸酯酶。
L.garvieae中包含可诱导降解纤维二糖的6-磷酸-β-葡萄糖苷酶(EC:3.2.1.86)。纤维二糖可经由磷酸转移酶系统(pep-pts)转运入胞内并磷酸化为纤维二糖-6-磷酸,之后被6-磷酸-β-葡萄糖苷酶水解为等摩尔的葡萄糖和葡萄糖-6-磷酸。如图6所示。6-磷酸-β-葡萄糖苷酶在微生物利用纤维素及其降解产物方面占据重要地位。葡萄糖经糖酵解代谢成丙酮酸,并进一步转化为乳酸、乙酸、乙醇等。乳酸在乳酸脱氢酶(EC:1.1.1.27)的作用下合成,乳酸脱氢酶能够催化丙酮酸和NADH转化为乳酸和NAD+,在细胞内起着调节酸碱平衡、产生能量和废物处理等多种生理功能。乙酸则需要经过乙酸激酶(EC:2.7.2.1)合成,乙酸激酶将乙酰辅酶A通过磷酸化反应转换为乙酸,为细胞提供能量和提供生物合成所需的有机物。乙醇则需要经过磷酸乙酰转移酶(EC:2.3.1.8)、乙醛脱氢酶(EC:1.2.1.10)、乙醇脱氢酶(EC:1.1.2.8)来合成。乳酸、乙酸和乙醇分别通过转运蛋白运输到细胞外在羧酸酯酶(胞外酶)的作用下生成乳酸乙酯和乙酸乙酯(图6)。通过对纤维二糖代谢、酸类代谢、酯类合成路径的解析,反映了L.garvieae在白酒发酵中的重要作用,对于进一步开发L.garvieae应用提供了理论依据。
3.4 L.garvieae毒力因子分析
对筛选中白酒窖泥的4株L.garvieae的全基因组与VFDB数据库进行比对,进行菌株毒素安全性分析。J_1_9、J_3_6、J_3_15与BRM002分别注释出32、32、29、29个毒力基因,这些基因参与毒力因子的编码,包括黏附(fbp54、srtA、slrA和plr)、免疫逃逸(manA及Undetermined)、蛋白酶(htrA、tig)、毒性基因(Undetermined)、抗吞噬作用(cdsA)及调控(lisR)。详见表2。黏附相关基因可以促进黏附和生物膜形成。黏附基因slrA编码许多表面蛋白[26],这些表面蛋白被确定为重要的毒力因子,涉及细菌对宿主细胞上皮细胞的黏附,由识别黏附基质分子的微生物表面成分介导,从而促进宿主细胞黏附和组织定植[27]。此外,4株L.garvieae都只注释出唯一的毒素因子Hemolysin。这是一种形成孔的溶血素,是一种具有溶血活性的完整外膜蛋白。然而,溶血素表达需要完整的CylR2、CylA、CylB等8个蛋白,缺少任意一个都不能使梭菌溶血素表达。[28-29]。根据VFDB注释结果来看,L.garvieae不存在以上任意一个基因,VFDB注释对应的Hemolysin基因片段与WP_003358964.1高度相似,其表达产物与溶血素无关[30]。因此,可以说L.garvieae在自身基因水平上是安全的。但是,L.garvieae可能会受到外界环境因子以及宿主体内信号因子的调控,不排除外界环境诱导L.garvieae毒素基因表达的可能性。
表2 L.garvieae菌株毒力基因的注释结果
Table 2 Annotated results of virulence genes of L.garvieae strains
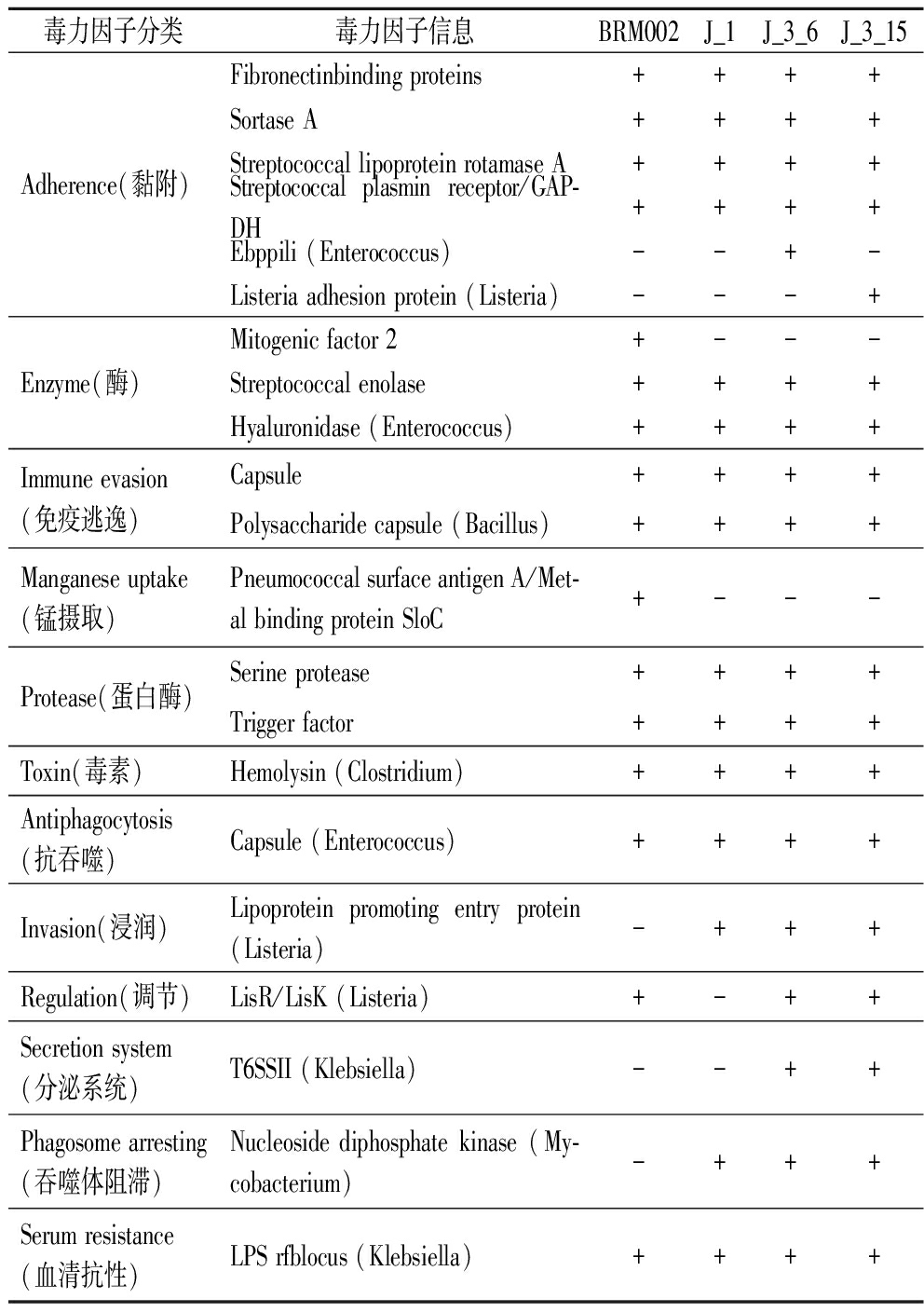
毒力因子分类毒力因子信息BRM002J_1J_3_6J_3_15Adherence(黏附)Fibronectinbinding proteins++++Sortase A++++Streptococcal lipoprotein rotamase A++++Streptococcal plasmin receptor/GAP-DH++++Ebppili (Enterococcus)--+-Listeria adhesion protein (Listeria)---+Enzyme(酶)Mitogenic factor 2+---Streptococcal enolase++++Hyaluronidase (Enterococcus)++++Immune evasion(免疫逃逸)Capsule++++Polysaccharide capsule (Bacillus)++++Manganese uptake(锰摄取)Pneumococcal surface antigen A/Met-al binding protein SloC+---Protease(蛋白酶)Serine protease++++Trigger factor++++Toxin(毒素)Hemolysin (Clostridium)++++Antiphagocytosis(抗吞噬)Capsule (Enterococcus)++++Invasion(浸润)Lipoprotein promoting entry protein (Listeria)-+++Regulation(调节)LisR/LisK (Listeria)+-++Secretion system(分泌系统)T6SSII (Klebsiella)--++Phagosome arresting(吞噬体阻滞)Nucleoside diphosphate kinase (My-cobacterium)-+++Serum resistance(血清抗性)LPS rfblocus (Klebsiella)++++
3.5 基因岛和原噬菌体预测
使用IslandViewer 4分别注释出4株L.garvieae的基因岛。通过与数据库对比,在BRM002、J_1、J_3_6、J_3_15的基因岛个数分别从6到8个不等。单个基因岛长度大小从3 896 bp(J_1)到59 556 bp不等(J_3_15),总长度从99 266 bp到152 243 bp不等。四株L.garvieae基因岛包含基因个数从106个到138个不等,其中BRM002基因岛中具有明确生物学意义的基因最多(51),J_1基因岛中具有明确生物学意义的基因最少(34),见表3。以上基因岛GC含量、二核苷酸偏好性以及密码子偏好性与其所在的基因组存在显著性差异。这些基因及其蛋白质产物在生物体内扮演着关键作用,涵盖了蛋白质编码与转运、细胞代谢、基因调控、DNA修复、细胞呼吸等重要生命活动,以及环境毒物抵抗等功能均有密切的联系。
表3 L.garvieae基因岛信息
Table 3 L.garvieae gene islands (GIs) information
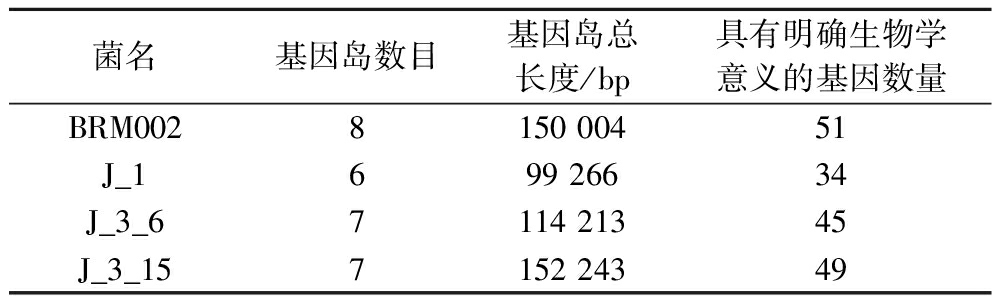
菌名基因岛数目基因岛总长度/bp具有明确生物学意义的基因数量BRM0028150 00451J_1699 26634J_3_67114 21345J_3_157152 24349
此外,使用PHASTER数据库与4株L.garvieae进行比对鉴定和注释出基因组内的噬菌体序列。检测到的原噬菌体数目见见电子版增强出版附表2(https://doi.org/10.13995/j.cnki.11-1802/ts.039507)。4株L.garvieae均未发现完整型原噬菌体序列,共有3个可疑型原噬菌体序列,13个不完整型原噬菌体序列。
可以观察到J_1、J_3_6、J_3_15都存在不完整的PHAGE_Lactoc_98201 (NC_031064)及PHAGE_Lactoc_PLgT_1 (NC_031016),见表4。以上噬菌体能够特异性地识别和感染乳球菌属(Lactococcus),通过转导过程促进与与其他微生物群落或L.garvieae细菌基因型之间的遗传交换从而导致水平基因转移。以上噬菌体可能在L.garvieae菌株的进化和多样性中发挥了重要作用。
表4 L.garvieae噬菌体信息
Table 4 Phage information of L.garvieae
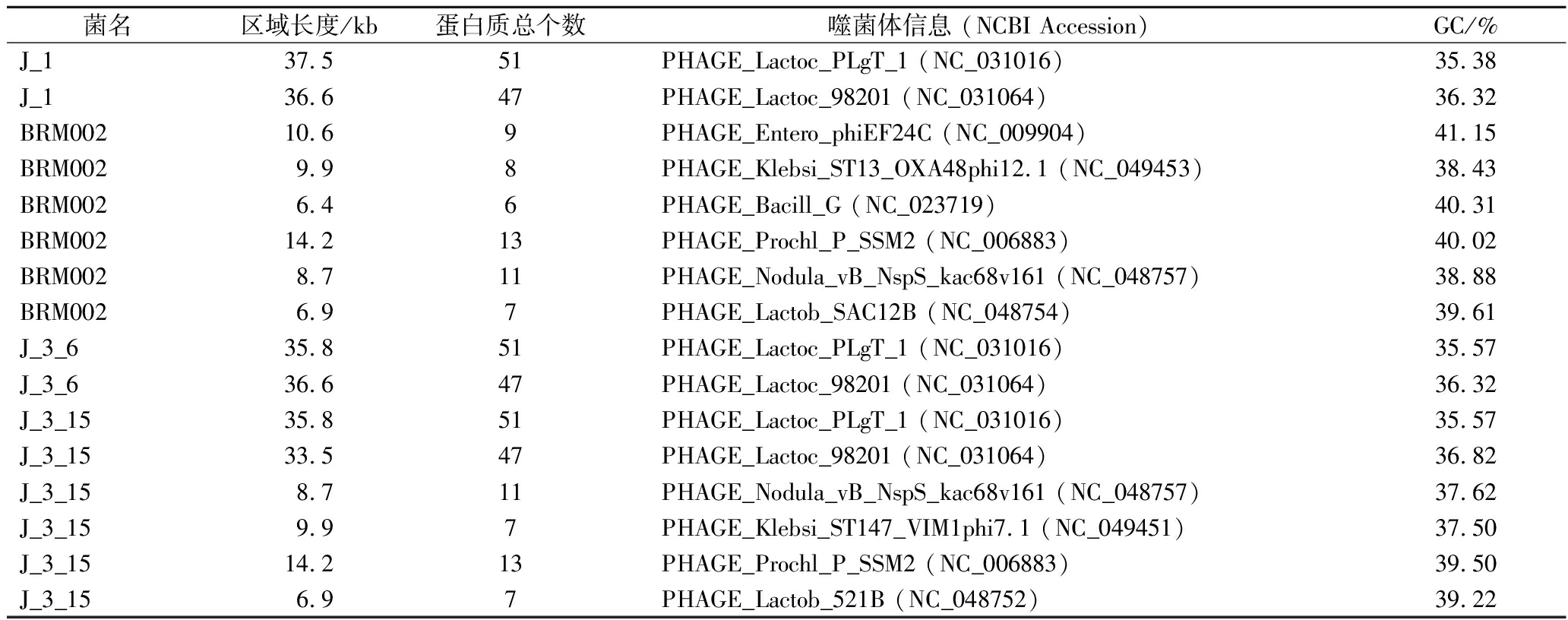
菌名区域长度/kb蛋白质总个数噬菌体信息 (NCBI Accession)GC/%J_137.551PHAGE_Lactoc_PLgT_1 (NC_031016)35.38J_136.647PHAGE_Lactoc_98201 (NC_031064)36.32BRM00210.69PHAGE_Entero_phiEF24C (NC_009904)41.15BRM0029.98PHAGE_Klebsi_ST13_OXA48phi12.1 (NC_049453)38.43BRM0026.46PHAGE_Bacill_G (NC_023719)40.31BRM00214.213PHAGE_Prochl_P_SSM2 (NC_006883)40.02BRM0028.711PHAGE_Nodula_vB_NspS_kac68v161 (NC_048757)38.88BRM0026.97PHAGE_Lactob_SAC12B (NC_048754)39.61J_3_635.851PHAGE_Lactoc_PLgT_1 (NC_031016)35.57J_3_636.647PHAGE_Lactoc_98201 (NC_031064)36.32J_3_1535.851PHAGE_Lactoc_PLgT_1 (NC_031016)35.57J_3_1533.547PHAGE_Lactoc_98201 (NC_031064)36.82J_3_158.711PHAGE_Nodula_vB_NspS_kac68v161 (NC_048757)37.62J_3_159.97PHAGE_Klebsi_ST147_VIM1phi7.1 (NC_049451)37.50J_3_1514.213PHAGE_Prochl_P_SSM2 (NC_006883)39.50J_3_156.97PHAGE_Lactob_521B (NC_048752)39.22
4 结论与讨论
本研究旨在构建L.garvieae的泛基因组,并对其核心基因组特征和窖泥环境下L.garvieae的潜在应用进行深入探究。根据分析,L.garvieae基因组中共发现6 347个泛基因簇,L.garvieae的泛基因组呈现出开放的特征,核心基因组主要涉及菌体的基础代谢功能。此外,结合COG和KEGG功能注释,表明L.garvieae产生淀粉酶、糖化酶、纤维素酶等酶类来催化微生物发酵过程,同时产生乳酸、乙酸、乙醇等风味前体物质,并且促进乳酸乙酯、乙酸乙酯等风味物质产生。详细阐述了L.garvieae在产酶及产风味物质方面的代谢合成路径,对于L.garvieae在白酒发酵中的应用提供了一定的理论依据。通过对这4株L.garvieae的毒力因子进行安全性评估,表明L.garvieae在自身基因水平上是安全的,外界环境诱导L.garvieae毒素基因表达的可能性有待进一步考证。
基于泛基因组对L.garvieae进行深入解析,结果揭示了该菌株的基因演化和功能潜力。通过对窖泥L.garvieae可移动遗传元件的挖掘,表明其在菌株基因组的进化和多样性中发挥的作用。研究结果对于理解L.garvieae在白酒行业中的开发具有一定的指导意义,为深入挖掘其在发酵工艺中的作用机制和潜在应用提供了一定理论基础。
[1] LIN Y S, KWEH K H, KOH T H, et al.Genomic analysis of Lactococcus garvieae isolates[J].Pathology, 2020, 52(6):700-707.
[2] 阎春悦, 利佳炜, 张茜, 等.不同香型白酒酿造过程乳酸菌的研究进展[J].酿酒科技, 2023(9):114-120.
YAN C Y, LI J W, ZHANG Q, et al.Research progress in lactic acid bacteria in the production of Baijiu of different flavor types[J].Liquor-Making Science &Technology, 2023(9):114-120.
[3] 吴正坤, 缪礼鸿, 张明春, 等.不同贮存期高温大曲中乳酸菌的多样性及其耐受性分析[J].中国酿造, 2018, 37(6):61-67.
WU Z K, MIAO L H, ZHANG M C, et al.Diversity and tolerance of lactic acid bacteria in high temperature Daqu at different storage periods[J].China Brewing, 2018, 37(6):61-67.
[4] 刘凡, 仇钰莹, 周新虎, 等.洋河浓香型白酒酒醅中产酸细菌与有机酸合成的相关性研究[J].食品与发酵工业, 2018, 44(12):22-29.
LIU F, QIU Y Y, ZHOU X H, et al.The correlation between organic acid producing bacteria and organic acids biosynthesis in fermented grains of Yanghe strong-aroma spirit[J].Food and Fermentation Industries, 2018, 44(12):22-29.
[5] 胡世伟, 王超凡, 王景春, 等.浓香型白酒窖泥微生物群落及其风味成分研究进展[J].中国酿造, 2023, 42(12):9-12.
HU S W, WANG C F, WANG J C, et al.Research progress of microbial community and flavor substances in strong-flavor Baijiu pit mud[J].China Brewing, 2023, 42(12):9-12.
[6] 陈聪, 邹伟, 汤秀娟, 等.产酯酶格氏乳球菌的筛选、鉴定与基因组注释[J].食品科学, 2024, 45(8):87-95.
CHEN C, ZOU W, TANG X J, et al.Screening, identification and genome annotation of esterase-producing Lactococcus garvieae[J].Food Science, 2024, 45(8):87-95.
[7] 骆红波, 孙优兰, 范奇高, 等.组学技术在白酒酿造微生物中的应用进展[J].中国酿造, 2023, 42(12):13-21.
LUO H B, SUN Y L, FAN Q G, et al.Progress on the application of omics technologies in Baijiu brewing microorganism[J].China Brewing, 2023, 42(12):13-21.
[8] ZHOU X G, REN L F, LI Y T, et al.The next-generation sequencing technology:A technology review and future perspective[J].Science China.Life Sciences, 2010, 53(1):44-57.
[9] 余晶晶, 黄永光.基因组学及其在白酒酿造基础研究领域中的应用[J].食品与发酵工业, 2021, 47(7):278-284.
YU J J, HUANG Y G.Genomics and its application in basic research field of Baijiu brewing[J].Food and Fermentation Industries, 2021, 47(7):278-284.
[10] TETTELIN H, MASIGNANI V, CIESLEWICZ M J, et al.Genome analysis of multiple pathogenic isolates of Streptococcus agalactiae:Implications for the microbial “pan-genome”[J].Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(39):13950-13955.
[11] 何苗, 赵雨晴, 李居行, 等.弯曲乳杆菌与清酒乳杆菌的比较基因组学分析[J].食品科学, 2024, 45(2):48-56.
HE M, ZHAO Y Q, LI J X, et al.Comparative genomic analysis of Latilactobacillus curvatus and L.sakei[J].Food Science, 2024, 45(2):48-56.
[12] TATUSOVA T, DICUCCIO M, BADRETDIN A, et al.NCBI prokaryotic genome annotation pipeline[J].Nucleic Acids Research, 2016, 44(14):6614-6624.
[13] AZIZ R K, BARTELS D, BEST A A, et al.The RAST Server:Rapid annotations using subsystems technology[J].BMC Genomics, 2008, 9:75.
[14] CHAUDHARI N M, GUPTA V K, DUTTA C.BPGA- an ultra-fast pan-genome analysis pipeline[J].Scientific Reports, 2016, 6:24373.
[15] ZHAO Y B, JIA X M, YANG J H, et al.PanGP:A tool for quickly analyzing bacterial pan-genome profile[J].Bioinformatics, 2014, 30(9):1297-1299.
[16] RICHTER M, ROSSELL -M
-M RA R, OLIVER GLÖCKNER F, et al.JSpeciesWS:A web server for prokaryotic species circumscription based on pairwise genome comparison[J].Bioinformatics, 2016, 32(6):929-931.
RA R, OLIVER GLÖCKNER F, et al.JSpeciesWS:A web server for prokaryotic species circumscription based on pairwise genome comparison[J].Bioinformatics, 2016, 32(6):929-931.
[17] KUMAR S, STECHER G, LI M, et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution, 2018, 35(6):1547-1549.
[18] KANEHISA M, SATO Y, KAWASHIMA M, et al.KEGG as a reference resource for gene and protein annotation[J].Nucleic Acids Research, 2016, 44(D1):D457-D462.
[19] HUERTA-CEPAS J, SZKLARCZYK D, HELLER D, et al.eggNOG 5.0:A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses[J].Nucleic Acids Research, 2019, 47(D1):D309-D314.
[20] CHEN L H, YANG J, YU J, et al.VFDB:A reference database for bacterial virulence factors[J].Nucleic Acids Research, 2005, 33(Database issue):D325-D328.
[21] BERTELLI C, LAIRD M R, WILLIAMS K P, et al.IslandViewer 4:Expanded prediction of genomic islands for larger-scale datasets[J].Nucleic Acids Research, 2017, 45(W1):W30-W35.
[22] ARNDT D, GRANT J R, MARCU A, et al.PHASTER:A better, faster version of the PHAST phage search tool[J].Nucleic Acids Research, 2016, 44(W1):W16-W21.
[23] PARKS D H, IMELFORT M, SKENNERTON C T, et al.CheckM:Assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J].Genome Research, 2015, 25(7):1043-1055.
[24] ALIKHAN N F, PETTY N K, BEN ZAKOUR N L, et al.BLAST ring image generator (BRIG):Simple prokaryote genome comparisons[J].BMC Genomics, 2011, 12:402.
[25] MORIYA Y, ITOH M, OKUDA S, et al.KAAS:An automatic genome annotation and pathway reconstruction server[J].Nucleic Acids Research, 2007, 35(Web Server issue):W182-W185.
[26] BOBER M, MÖRGELIN M, OLIN A I, et al.The membrane bound LRR lipoprotein Slr, and the cell wall-anchored M1 protein from Streptococcus pyogenes both interact with type I collagen[J].PLoS One, 2011, 6(5):e20345.
[27] XU S Y, LIU Y, GAO J, et al.Comparative genomic analysis of Streptococcus dysgalactiae subspecies dysgalactiae isolated from bovine mastitis in China[J].Frontiers in Microbiology, 2021, 12:751863.
[28] 贾伟娟, 宋丽丽, 张玲艳, 等.蜡样芽胞杆菌毒素的最新研究进展[J].中国抗生素杂志, 2022, 47(6):534-542.
JIA W J, SONG L L, ZHANG L Y, et al.Research progress of the toxins from Bacillus cereus[J].Chinese Journal of Antibiotics, 2022, 47(6):534-542.
[29] HAAS W, SHEPARD B D, GILMORE M S.Two-component regulator of Enterococcus faecalis cytolysin responds to quorum-sensing autoinduction[J].Nature, 2002, 415(6867):84-87.
[30] SEBAIHIA M, PECK M W, MINTON N P, et al.Genome sequence of a proteolytic (Group I) Clostridium botulinum strain Hall A and comparative analysis of the clostridial genomes[J].Genome Research, 2007, 17(7):1082-1092.