麦芽糖淀粉酶(maltogenic amylase,MAase),全称为1,4-α-D-葡聚糖α-麦芽糖基水解酶(1,4-α-D-glucan α-maltohydrolase, EC 3.2.1.133),与环糊精水解酶(cyclodextrin hydrolase, EC 3.2.1.54)和普鲁兰酶(neopullulanase, EC 3.2.1.135)被归属于糖苷水解酶GH13-20家族,是一种广泛应用于淀粉类食品的重要工业酶[1-2]。麦芽糖淀粉酶具有多底物特性和底物优先选择性,优先水解环状糊精,其次为淀粉等[3-4],它通过从淀粉分子末端水解析出麦芽糖或聚合度DP2~DP7的小分子寡糖来干扰淀粉重结晶以及淀粉粒与蛋白质大分子的缠绕,从而减少淀粉颗粒的回生率和重结晶率,达到抗老化的效果[5-6]。与乳化剂、增强剂等传统面包改良剂相比,麦芽糖淀粉酶在效率和安全性方面均被认为是更为理想的烘焙面制品改良剂,并已实现淀粉类食品中应用商业化[7]。
通常,麦芽糖淀粉酶作为一种配料被添加到面粉中,然后经历混合、发酵、揉合、二次发酵和烘烤完成面包制作。由于经过2次发酵后面团的pH值可达到4.0~6.0[8],耐酸性成为商业麦芽糖淀粉酶在淀粉食品工业应用中重要特性之一。目前大部分已报道的麦芽糖淀粉酶均来源于耐热微生物,例如来自嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)[9]、地衣芽孢杆菌(Bacillus licheniformis)[10]、枯草芽孢杆菌(Bacillus subtilis)[11]和栖热菌属(Thermus sp.)[12]的麦芽糖淀粉酶。尽管它们具有良好的热稳定特性,但其中多数对酸性条件敏感,导致其在低pH环境下快速失活,因此在利用低pH配方制作面包的工艺中添加该酶的有益效果有限[2]。目前,合理设计的酶耐酸性修饰策略主要包括催化中心改造和表面电荷修饰[13-14]。如LI等[15]使用序列比对和表面电荷策略来修饰β-葡聚糖酶提高耐酸性,突变体Q1E/I133L在pH 5.0和pH 6.0条件下可保持86.9%和100.5%的活性,显著高于野生型(38.2%和56.4%)。YANG等[16]将枯草芽孢杆菌来源的α-淀粉酶4个关键位置的组氨酸(His)突变为天冬氨酸(Asp)构建单点突变体和多点组合突变体,突变体H275/293/310D在pH 4.5时的比活性从108.2提高至216.2 U/mg,突变后氢键和盐桥的增加成功增强了突变体的酸稳定性。TURUNEN等[17]对里氏木霉(Trichoderma reesei)木聚糖酶催化亲核残基周围的丝氨酸(Ser)和苏氨酸(Thr)表面进行定点突变引入5个精氨酸(Arg)残基,将该酶的最适pH降低了0.5~1.0个pH单位。CHEN等[18]根据嗜酸普鲁兰芽孢杆菌(B. acidopullulyticus)普鲁兰酶的晶体结构,将可能改变催化残基去质子化常数(pKa)的残基突变为Arg获得突变体L627R,其最适pH值从5.0转变为4.0,相对活性提高至野生型的117%,成功改善了BaPul的耐酸性,提高了工业应用价值。上述试验验证了蛋白质工程技术在酶耐酸性改造方面的可行性,同时为本研究更加精准且高效地实现酶的耐酸性改造提供了宝贵的指导意义。
本研究针对当前麦芽糖淀粉酶作用条件受限的突出问题,以来源于类食品乳杆菌的麦芽糖淀粉酶(maltogenic amylase derived from Lactobacillus paralimentarius,LpaMA)基因作为出发基因,构建了E.coli BL21/pET28a-LpaMA重组表达体系,首先考察了该酶的基本酶学性质。其次,通过降低催化残基的pKa值和表面电荷改造的2种理性设计方法构建耐酸突变文库,成功筛选获得正向耐酸突变体,为麦芽糖淀粉酶在面制品工业中的应用奠定了基础。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
本研究所用大肠杆菌BL21(DE3)菌株和pET-28a(+)质粒,本实验室提供。LpaMA基因(NCBI No.WP_025085606.1)和引物由天霖生物科技(上海)有限公司合成。
1.1.2 试剂
所有限制性内切酶,宝生物工程(大连)有限公司;可溶性淀粉、3,5-二硝基水杨酸,上海阿拉丁生化科技股份有限公司;所有其他化学品均为商业上可以获得的最高等级。
1.2 实验方法
1.2.1 突变体菌株的构建
利用全质粒PCR技术,以获得的重组质粒pET28a-LpaMA为模板进行定点突变,获得突变体。PCR反应在50 μL体系中进行,反应条件为94 ℃预变性4 min;然后进入30个循环:98 ℃变性10 s,55 ℃退火5 s,72 ℃延伸5 min;最后72 ℃延伸10 min,4 ℃保温。PCR扩增产物用1%琼脂糖凝胶电泳进行检测,检测结束后,向10 μL扩增产物中加入0.5 μL甲基化模板消化酶(Dpn I),枪头吹吸进行混匀,于37 ℃条件下反应1.5 h,将Dpn I处理后的扩增产物转化大肠杆菌E.coli BL21(DE3),转化产物涂布于LB固体培养基,于37 ℃培养8~10 h,在LB固体培养基上挑取多个转化子,接入LB液体培养基培养,于37 ℃培养10 h后提取质粒,将此质粒进行序列测定,测序正确即获得含有编码突变体基因的重组大肠杆菌。
1.2.2 突变体蛋白的表达与纯化
将重组BL21/pET28a-LpaMA(6×His tag)及其突变体在含1%胰蛋白胨、0.5%酵母提取物和1% NaCl的液体LB培养基中,37 ℃、200 r/min培养至OD600达到0.6~0.8,然后用0.1 mmol/L异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG)17 ℃诱导20 h。将细胞重悬于100 mmol/L Tris-HCl缓冲液(pH 7.5)中,并在冰浴中超声波破碎。将上清液直接施加到Ni-NTA柱上,用500 mmol/L咪唑洗脱组合的重组LpaMA和突变体。通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)分析判断纯化蛋白的纯度,通过Bradford蛋白测定法测定蛋白浓度。
1.2.3 麦芽糖淀粉酶活性的测定
采用3,5-二硝基水杨酸法来精确测定麦芽糖淀粉酶的活性。在此方法中,使用10 g/L的可溶性淀粉作为底物。将500 μL底物与100 μL酶液混合后,反应进行10 min。随后,加入600 μL DNS溶液以终止反应。接着,将反应混合物置于沸水中进行显色反应,持续5 min。显色完成后,通过测定其在540 nm下的吸光度(OD540)来量化麦芽糖的产生量。麦芽糖淀粉酶的活性定义为:在一定条件下,每分钟催化产生1 μmol麦芽糖所需的酶量。
1.2.4 数据的处理与分析
选择源自栖热菌属的麦芽糖淀粉酶(PDB登录号:1SMA;分辨率:2.8 Å)的晶体结构为模板,运用Swiss-Model服务器(http://swissmodel.expasy.org)建立LpaMA及其突变株的三维模型。按照标准协议使用AMBER14软件包进行分子动力学模拟,使用最速下降和共轭梯度算法对系统进行最小化,然后以超过50 ps的恒定体积从0逐渐加热到333.15 K。平衡后,在恒压常温条件下对配合物进行无约束的70 ns分子动力学模拟。利用PyMOL软件进行可视化分析。利用origin 2021软件将初始速率与底物浓度数据非线性曲线拟合到Michaelis-Menten方程来计算麦芽糖淀粉酶对β-CD的Km和kcat值。本文涉及所有实验均采用3个独立数据样本,使用GraphPad Prism软件计算结果。
2 结果与分析
2.1 重组麦芽糖淀粉酶体系的构建与表达
本实验室前期筛选到一株具有潜在优良性质的类食品乳杆菌(Lactobacillus paralimentarius)麦芽糖淀粉酶LpaMA,其基因全长1 755 bp,编码584个氨基酸,并预计其相对分子质量为67.7 kDa。本研究以此为研究对象,将LpaMA基因通过pET-28a载体成功化转到E.coli BL21中,实现了重组LpaMA的可溶性表达。经过超声波破碎和镍柱亲和层析处理,获得带有6×His-tag的SDS-PAGE凝胶电泳纯LpaMA(图1)。
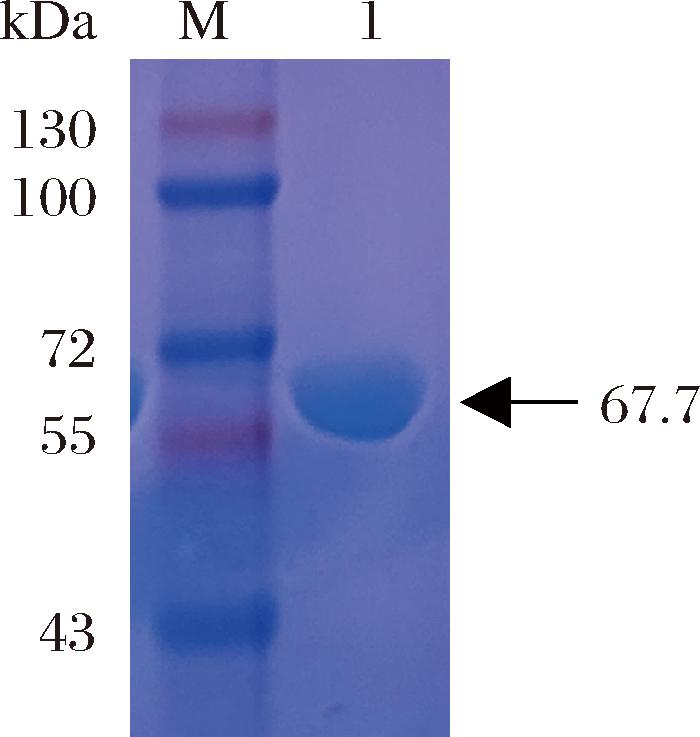
图1 LpaMA的SDS-PAGE分析
Fig.1 SDS-PAGE analysis of LpaMA
注:M-蛋白分子量标准;Lane 1-LpaMA纯酶。
重组LpaMA在50 ℃、pH 5.0的条件下表现出最高的酶活力,达到了274.79 U/mg(图2)。重组LpaMA在pH 4.0、4.5和5.0的条件下经过60 ℃热水浴孵育0.5 h后,其残余酶活力分别为初始活力40.44%、48.12%和53.82%。面团二次醒发后的pH值通常介于4.0~6.0之间,酸性环境会影响LpaMA的稳定性及水解活性的损失,进而减弱其改善面包品质的效果。因此,提升LpaMA在较低pH环境,特别是pH 4.0~5.0条件下的耐酸性,是当前亟待解决的问题。
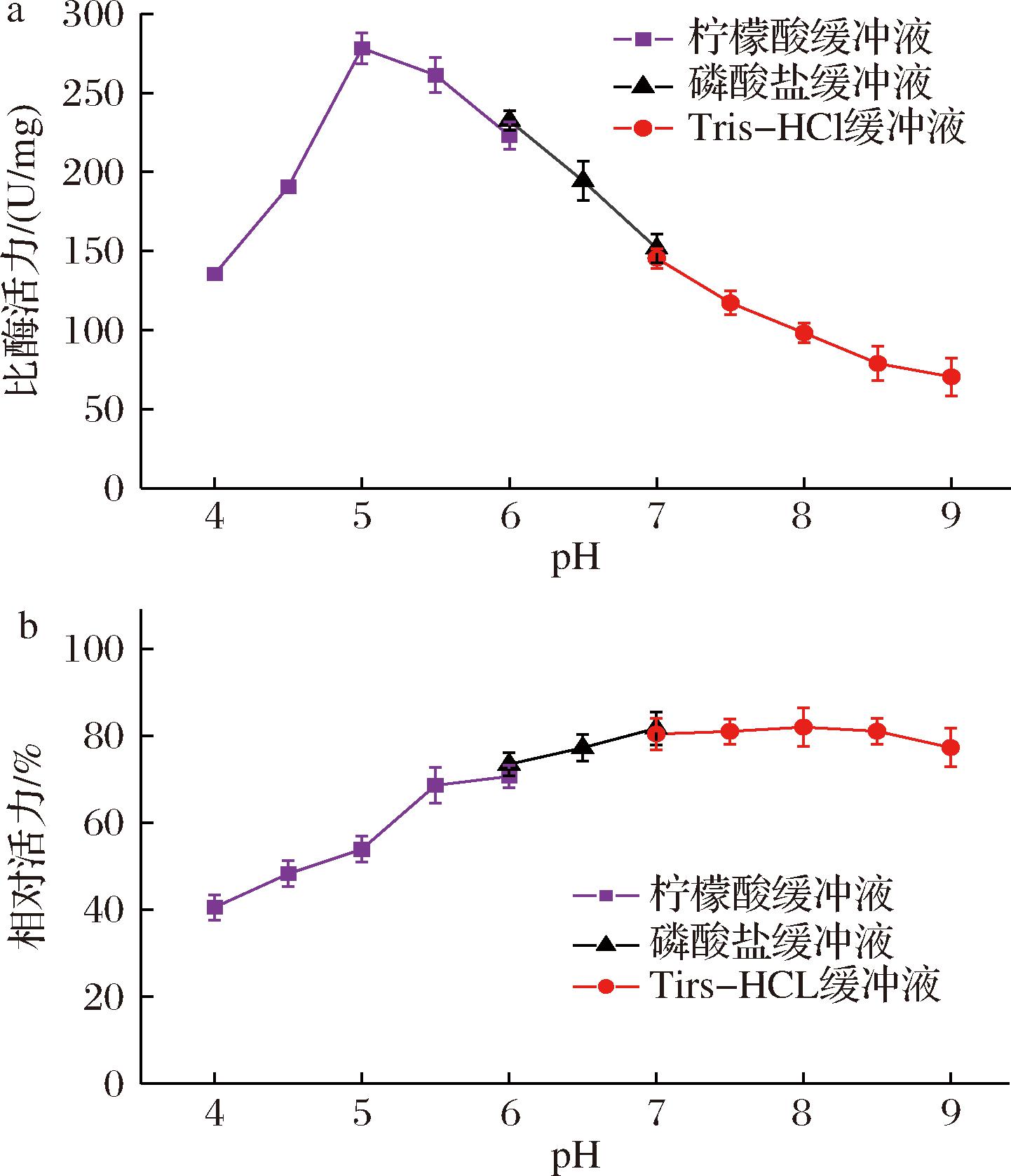
a-最适pH;b-pH稳定性
图2 LpaMA的最适pH及其pH稳定性
Fig.2 Optimal pH and pH stability of LpaMA
2.2 麦芽糖淀粉酶耐酸突变位点的选择与菌株的构建
近年来,通过关键残基的定点突变的蛋白质工程已成为提高酶活力、底物特异性和稳定性的有效手段。在调整酶的最适pH方面,主要策略聚焦于蛋白质的催化中心和表面电荷改造。一方面,针对位于first-shell与反应底物分子和产物或结合金属离子直接接触的氨基酸残基,由于其与底物有直接相互作用,对这些氨基酸残基的突变会对酶活力造成较大的影响。因此,本研究将与LpaMA活性中心的亲核残基(Asp339)和酸碱催化残基(Glu368)直接或间接形成一级氢键和二级氢键的位于second-shell或更远的氨基酸残基替换为带正电荷的精氨酸(Arg),其中大部分位点为不带电的氨基酸残基。该策略目的在于,通过改变活性中心裂隙周围的氨基酸,将带正电的氨基酸放置在距离亲核试剂一定距离的位置,稳定其去质子化形式,并导致其催化残基的pKa值降低[19-20]。
另一方面,针对表面电荷的改造,蛋白质表面的带电状态往往影响蛋白质在不同pH条件下的催化能力,一般最适pH偏酸性的酶表面具有更多的酸性氨基酸残基。因此,本研究为了优化酶的表面电荷分布,选取溶剂可及表面积≥50%且距离催化残基超过20 Å的碱性氨基酸残基(精氨酸Arg和赖氨酸Lys)作为目标位点引入酸性氨基酸天冬氨酸(Asp),以避免对活性位点口袋结构造成干扰。通过上述2种理性设计方法,构建了一个含35个突变位点的突变文库,各个耐酸突变位点在LpaMA的三维结构中的位置如图3所示。
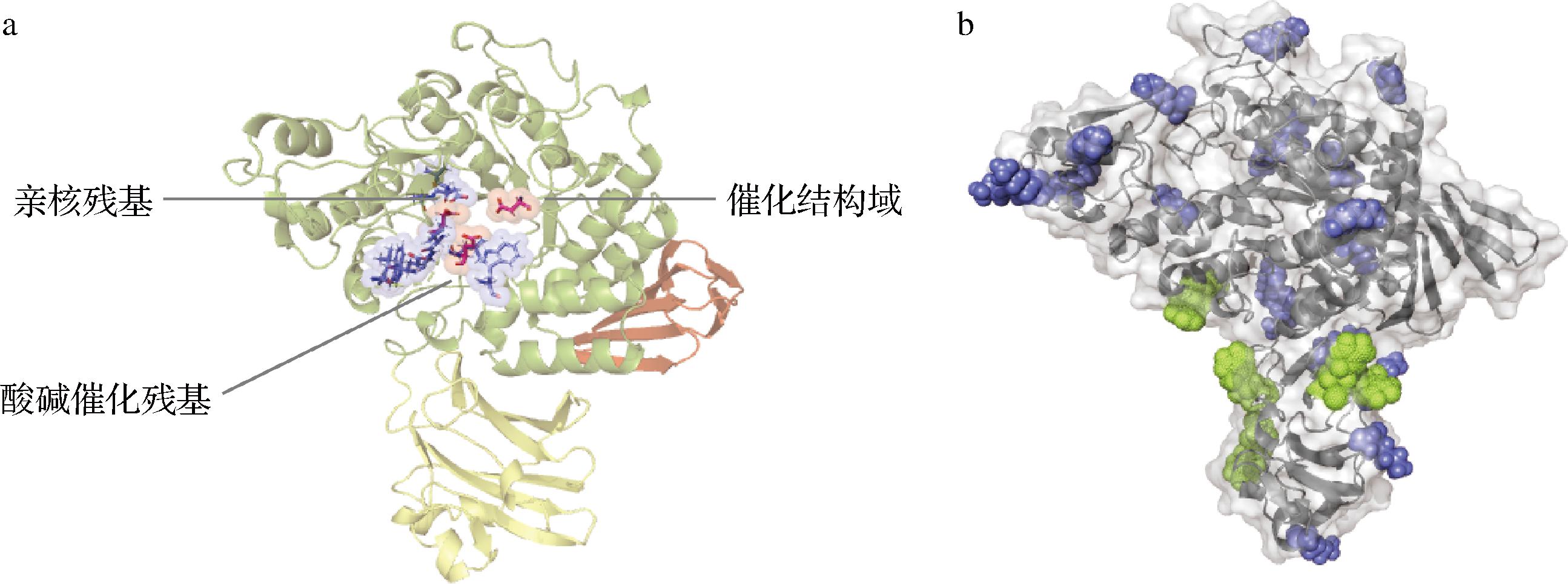
a-催化中心改造突变位点(蓝色棍棒模型分别代表L245R、D246R、A247R、V340R、A341R、E343R、V344R、W349R、L366R、W370R);b-表面电荷改造突变位点(绿色球状模型分别代表R10D、R32D、R62D、R120D、R350D、R354D;蓝色球状模型分别代表K20D、K36D、K61D、K109D、 K131D、K138D、K154D、K230D、K231D、K238D、K264D、K267D、K270D、K279D、K293D、K322D、K361D、K401D、K490D)
图3 耐酸突变位点在LpaMA的空间位置
Fig.3 Positions of acid-resistant mutation candidates in LpaMA
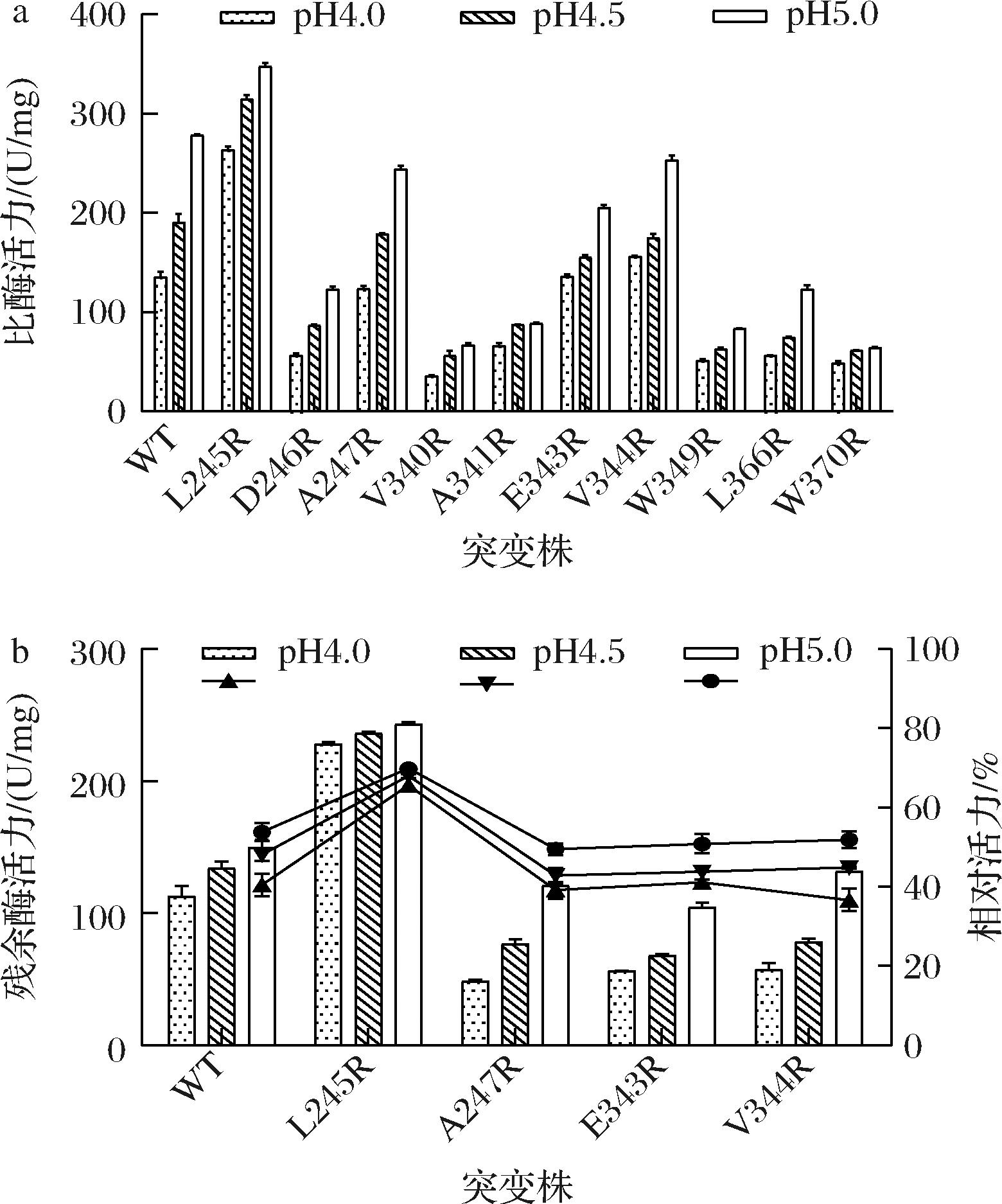
a-水解活力;b-pH稳定性
图4 活性中心改造突变体在酸性条件下的水解活力和pH稳定性
Fig.4 Hydrolytic activity and pH stability of the active site modified mutants under acidic conditions
注:柱形图代表比酶活力;折线图代表相对活力(下同)。
2.3 麦芽糖淀粉酶突变体酶学性质的测定
2.3.1 酶催化中心改造突变体
通过定点突变将带正电的精氨酸(Arg)放置在催化结构域周围,并对野生型及10个纯化突变酶在酸性条件(pH 4.0~5.0)下的比酶活力,以及在相应pH条件下60 ℃孵育30 min后的残余酶活力与相对活力进行了测定。如图4所示,突变体D246R、V340R、A341R、W349R、L366R和W370R的酶活力损失严重,A247R、E343R和V344R的整体水解活力虽然与野生型相当,但在酸性条件下的稳定性不足50%,出现了明显的下降。值得一提的是,突变体L245R在pH 4.0、4.5和5.0下的水解活力相较于野生型得到了显著的提升,分别为野生型的1.94倍、1.65倍和1.24倍。更重要的是,该突变体在各个pH下的稳定性也得到了相应的改善,其相对残余活力分别提高到65.60%、67.92%和69.90%。
2.3.2 酶表面电荷改造突变体
通过引入酸性氨基酸残基来改变LpaMA的表面电荷分布,前期预实验发现引入天冬氨酸(Asp)对耐酸性的正向提升效果比谷氨酸(Glu)更为显著,因此后续研究主要聚焦于天冬氨酸突变。首先,成功将LpaMA表面的6个精氨酸突变为天冬氨酸,并测定了其在酸性条件下的水解活力和pH稳定性变化情况,如图5-a和图5-b所示。综合分析水解活力和pH稳定性,6个突变体均出现了活力损失严重或者稳定性下降的问题。从该策略的负面突变结果来看,完全的反向电荷变化可能增加酶结构崩塌的风险并伴随着突变体稳定性的急剧下降。
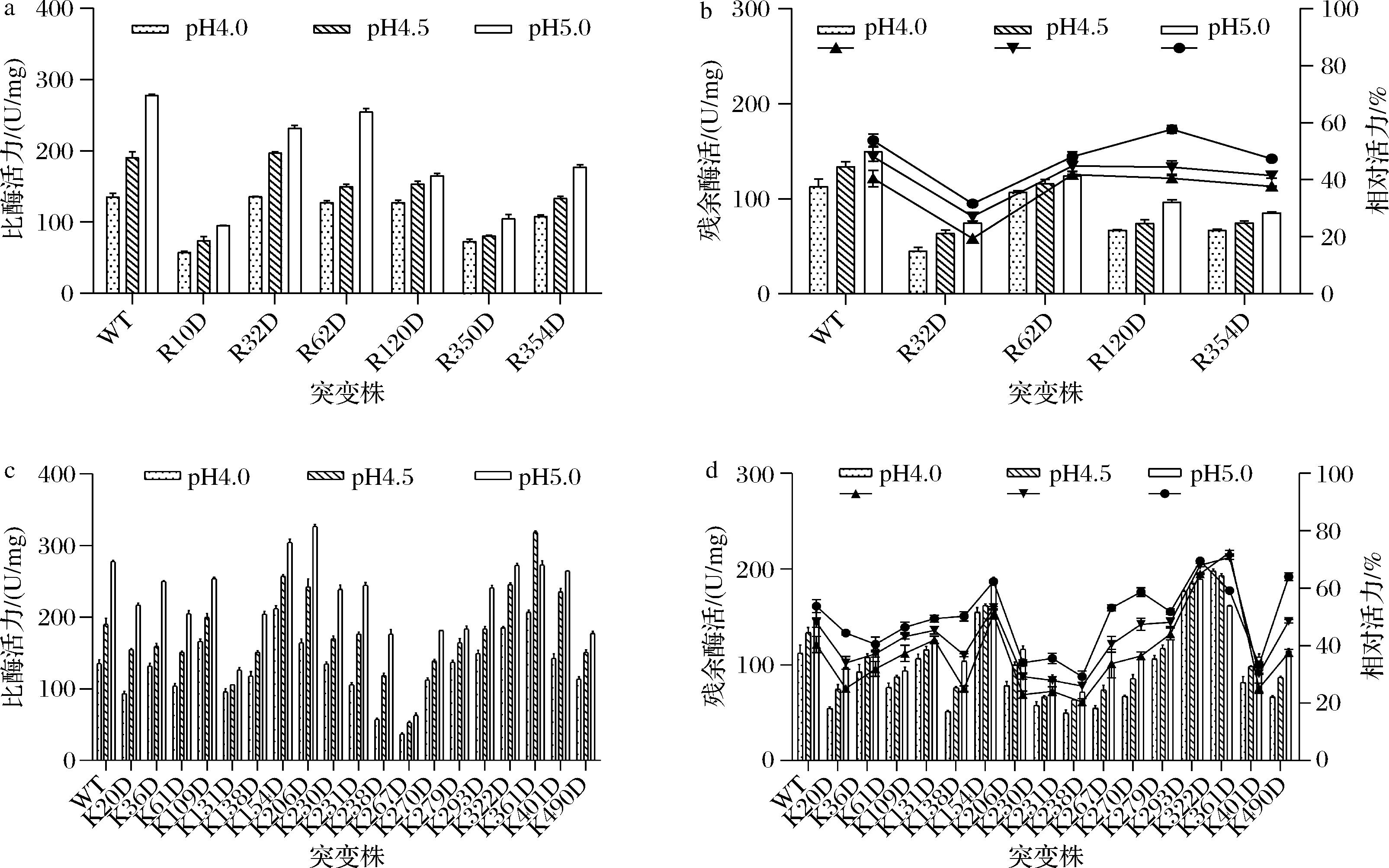
a-R/D突变株水解活力;b-R/D突变株pH稳定性;c-K/D突变株水解活力;d-K/D突变株pH稳定性
图5 表面电荷改造突变株在酸性条件下的水解活力和pH稳定性
Fig.5 Hydrolytic activity and pH stability of the surface charge modified mutants under acidic conditions
其次,将LpaMA表面的赖氨酸(Lys)突变为天冬氨酸,各个突变体在酸性条件(pH 4.0~5.0)下的水解活力和稳定性情况见图5-c和图5-d。除突变体K131D、K238D和K267D在酸性条件下的水解活力损失严重之外,其余大部分突变体在各个pH条件下的水解活力均呈现出不同程度的提升效果。突变体K154D、K206D、K322D、K361D和K401D在酸性条件(pH 4.0或pH 4.5)下的水解活力得到了显著提升。其中,K206D和K401D的酸稳定性较差,酶失活情况较为严重。突变体K154D、K322D和K361D则展现出了较好的酸稳定性,在pH 4.0条件下处理后,3个突变体的残余活力分别为50.92%、65.05%和70.73%,在pH 4.5条件下处理后,它们的残余活力分别为53.28%、68.06%和71.89%,而野生型WT在pH 4.0和pH 4.5下的残余活力分别为40.44%和48.12%,显示出这些突变体在酸稳定性方面的优势。值得一提的是,突变体K361D不仅表现出良好的酸稳定性,其最适pH从野生型的5.0降低到了4.5。此外,该突变体的最大水解活力达到了318.79 U/mg,相较于野生型WT在pH 5.0时的最大水解活力274.79 U/mg有了显著的提升。且野生型WT在pH 4.5时的比酶活力为190.65 U/mg,远低于突变体K361D的表现。
2.3.3 正向耐酸突变位点的组合突变
以上共筛选得到4个提高耐酸性的单点突变体(L245R、K154D、K322D和K361D),突变体在酸性条件下的酶活力损失情况得到了明显改善,特别是K361D突变体,其最适pH甚至降低至4.5,显示出良好的应用潜力。尽管以上有益突变位点在酶表面的分布不尽相同,但其普遍集中于活性中心的附近区域。有研究表明,这些表面残基位点对于底物进入活性位点、促进底物结合和催化过程具有重要影响。然而,单个突变位点的效应往往有限,需要通过突变叠加来发挥更为显著的作用。因此,本研究以上述所获正向耐酸单点突变体的基因序列为模板,进一步构建了6个双点突变体(K154D/K322D、K154D/K361D、K154D/L245R、L245R/K322D、L245R/K361D和K322D/K361D)。
各个组合突变体在pH 4.0~5.0条件下的初始酶活,以及在酸性条件下60 ℃温浴30 min后的残余酶活力考察情况如表1和表2所示。结果表明,K154D与L245R、K322D和K361D的叠加突变并未进一步增强LpaMA的酸稳定性,反而导致了酶活力的严重损失。K361D单点突变在降低LpaMA最适pH方面的积极贡献在与其他突变位点的组合中仍然得到了体现,特别是L245R/K361D和K322D/K361D这2个双点突变体,它们均在pH 4.5时达到了最大水解活力。其中,K322D/K361D突变体的表现尤为突出,其水解活力和酸稳定性均得到了显著提升,K322D/K361D突变体在pH 4.5时的水解活力达到了433.24 U/mg,为野生型酶的2.27倍。且该突变体在pH 4.0、4.5和5.0条件下的稳定性分别为71.50%、72.24%和69.68%,显示出了良好的应用前景。随后,以此突变体为新模板,引入三点突变体L245R/K322D/K361D,经诱导几乎无可溶性表达,尝试使用自诱导培养基和添加促溶标签也未能解决此问题。
表1 野生型和组合突变株在酸性条件下的水解活力 单位:U/mg
Table 1 Hydrolytic activity of wild type and combined mutants under acidic conditions
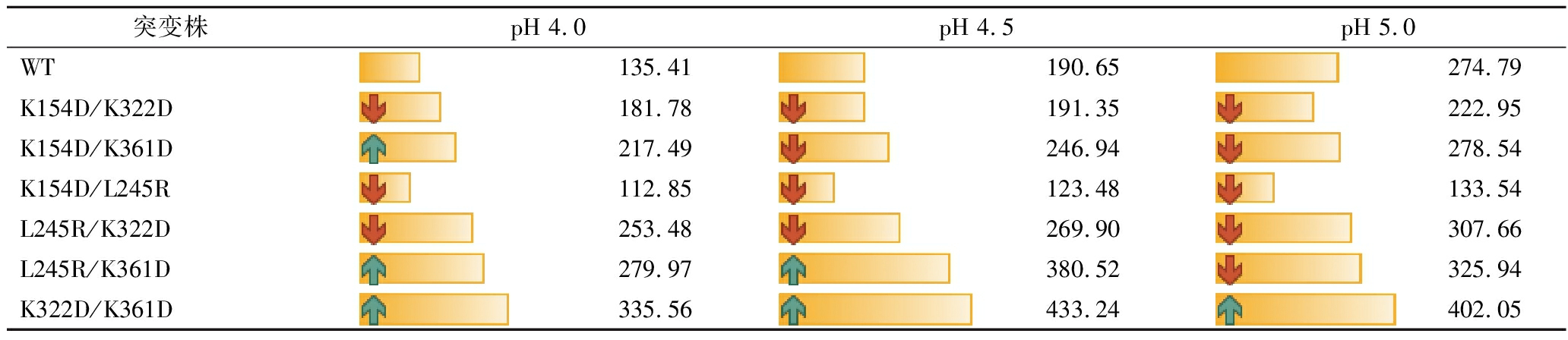
突变株pH 4.0pH 4.5pH 5.0WT135.41190.65274.79K154D/K322D181.78191.35222.95K154D/K361D217.49246.94278.54K154D/L245R112.85123.48133.54L245R/K322D253.48269.90307.66L245R/K361D279.97380.52325.94K322D/K361D335.56433.24402.05
注:绿色上箭头表示组合突变株的活力高于对应单点突变株;红色下箭头表示组合突变株的活力低于对应单点突变株。
表2 野生型和组合突变株在酸性条件下的稳定性 单位:%
Table 2 Stability of wild type and combined mutant strains under acidic conditions
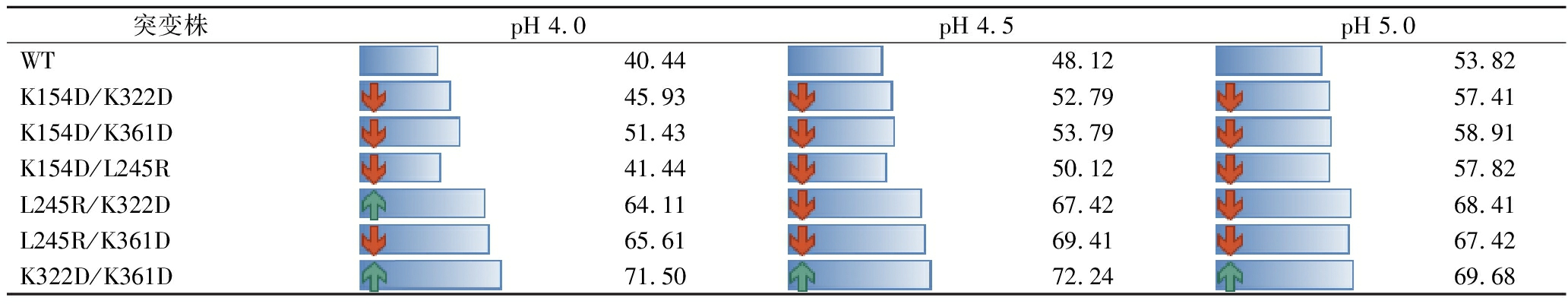
突变株pH 4.0pH 4.5pH 5.0WT40.4448.1253.82K154D/K322D45.9352.7957.41K154D/K361D51.4353.7958.91K154D/L245R41.4450.1257.82L245R/K322D64.1167.4268.41L245R/K361D65.6169.4167.42K322D/K361D71.5072.2469.68
注:绿色上箭头表示组合突变株的残余活力高于对应单点突变株;红色下箭头表示组合突变株的残余活力低于对应单点突变株。
2.3.4 正向耐酸突变体的机制解析
α-淀粉酶催化结构域的(α/β)8 TIM桶结构,形成蛋白质分子的核心,并包含3个活性位点残基Asp339-Glu368-Asp435(LpaMA),如图6-a所示。Asp339和Glu368被认为是2个催化基团,其中Asp339是催化亲核试剂,Glu368是酸碱催化剂,催化氢供体,Asp435被认为是通过与底物形成氢键并提高Glu368的pKa值来协助催化的过渡态稳定剂。SINNOTT等[21]对α-淀粉酶的催化机制进行了深入研究指出,为确保酶的高效催化活性,活性位点残基Glu368必须保持质子化状态,而催化亲核试剂Asp339则需维持去质子化状态。这意味着,质子供体Glu368在催化过程中需要保持其质子化特性以发挥催化作用,而亲核试剂Asp339则需要保持带负电的状态以达到最佳的催化效果。综上,α-淀粉酶在高pH环境下活性降低是由于Glu368的去质子化所致,而在低pH环境下,由于Asp339的质子化,酶的活性同样会受到影响。
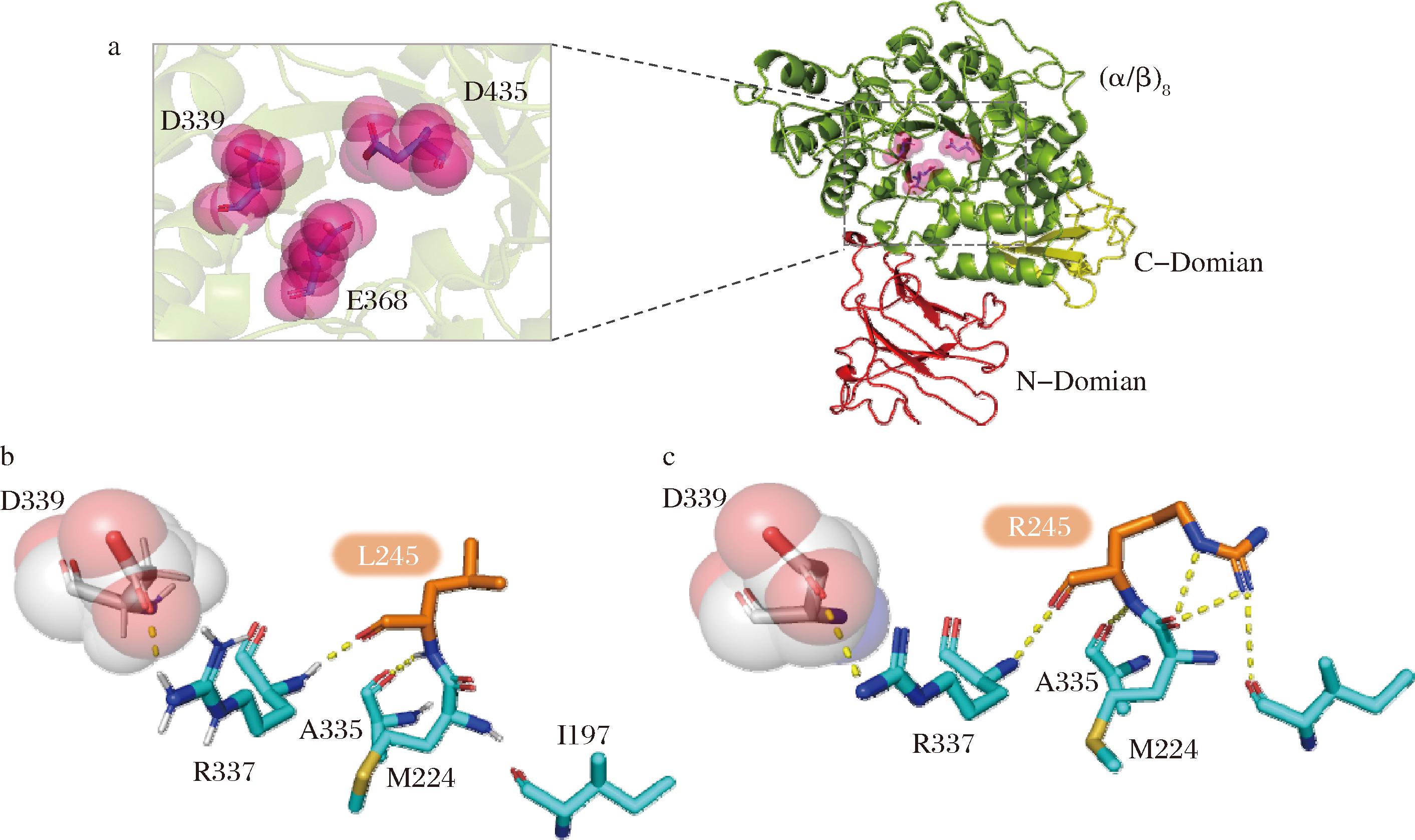
a-LpaMA催化结构域示意图;b-245位残基突变前结构;c-245位残基突变后结构
图6 LpaMA催化结构域示意图
Fig.6 Schematic diagram of the catalytic domain of LpaMA
对于酶催化中心改造中获得的正向突变体L245R,原本245位的Leu具有高疏水性,而疏水性残基在溶剂中暴露不利于蛋白质的稳定性[22],突变后带有亲水侧链的精氨酸Arg增加了其表面的亲水性。通过PROPK A3.0计算突变前后LpaMA活性中心亲核残基侧链的电离状态,发现由于Arg侧链胍基携带的正电荷,其对中性残基的取代使得亲核残基Asp339的pKa值从9.53降低到8.19,从而稳定其去质子化形式,增强了LpaMA在低pH下的稳定性。同时,新的氢键网络的形成也有助于附近区域的稳定,如Arg245处参与氢键网络构成:Arg245↔Met244和Arg245↔Ile197(图6-b和图6-c)。此外,采用分子动力学模拟,获得突变体L245R与LpaMA野生型在pH 5.0,70 ns时间内的运动轨迹和结构变化情况,如图7所示。突变体L245R和野生型的RMSF整体相差不大,说明位点的突变对LpaMA的关键区域结构并没有造成太大的影响。而野生型在40 ns以后的均方根偏差(root-mean-square deviation, RMSD)变化幅度高于突变体L245R,表明将带正电荷的Arg放置在在亲核残基Asp339周围降低了蛋白分子整体构像Cα的偏移幅度,这有利于LpaMA的pH稳定性。此外,麦芽糖淀粉酶分子通常呈现结构域交换二聚体状态,其中一个分子的N端结构域与相邻分子的(α/β)8桶结构域相互作用,使得活性位点位于又深又窄的底物结合区。而D246和L366等位点位于(α/β)8 TIM桶结构的β折叠上或附近,这些位点突变会影响折叠结构在反应介质中的运动,阻碍底物进入底物入口通道,从而导致酶活力下降严重。因此,催化残基附近的突变很可能影响催化机制并对催化速率产生总体负面影响。
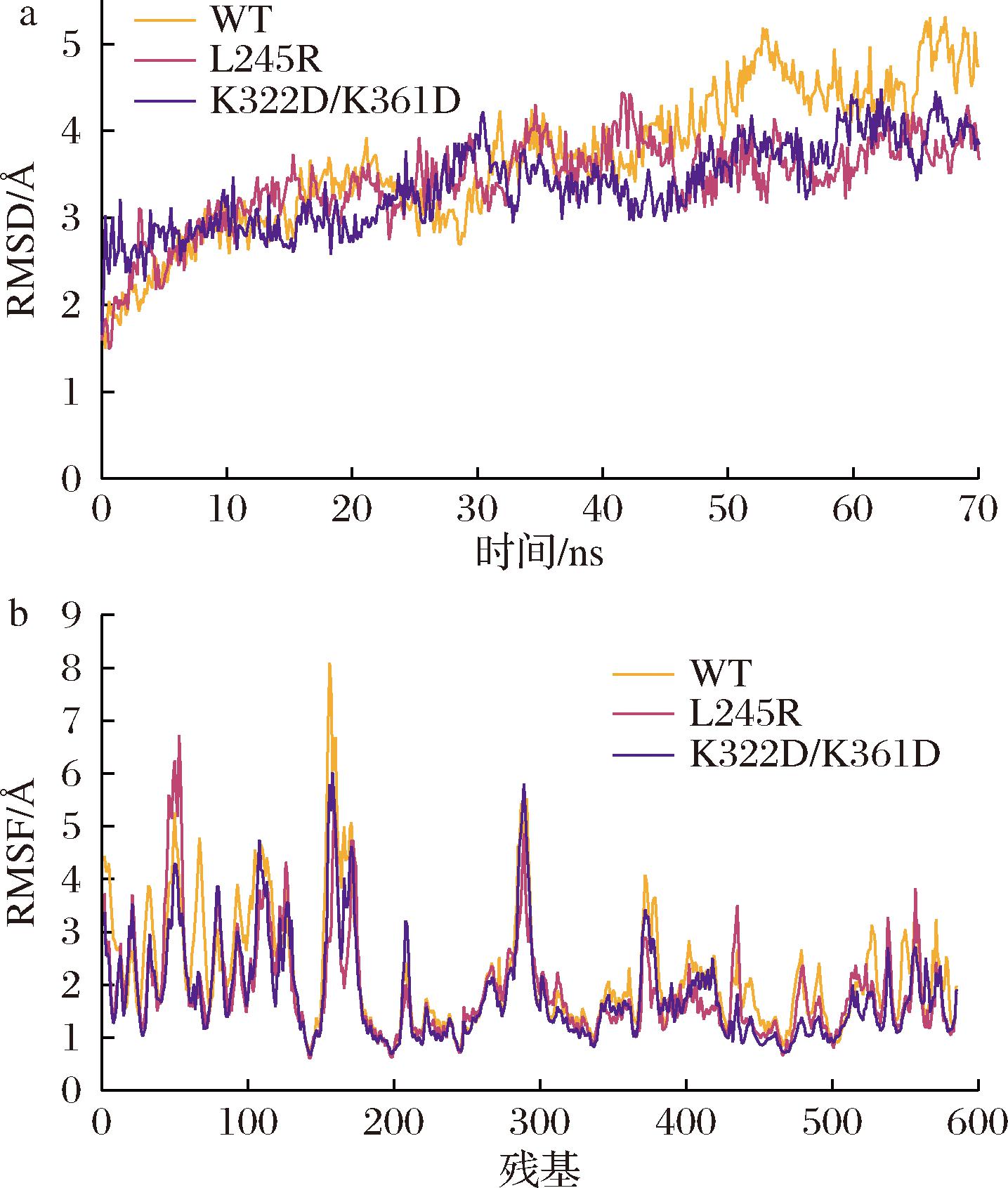
a-均方根偏差(RMSD);b-均方根波动(RMSF)
图7 70 ns内野生型和突变体的运动轨迹和结构变化
Fig.7 Motion trajectories and structural changes of the wild type and mutants within 70 ns
虽然pKa是影响酶pH谱的重要因素,但pH谱的变化并不总是与pKa的变化相关。据报道,除了pKa值外,表面电荷在确定酶pH谱方面也发挥着重要作用。一般来说,增加的净负电荷会将最佳pH值移至酸性区域,而增加的净正电荷会将最佳pH值移至碱性区域。本研究利用PyMOL计算蛋白质静电势能发现,酶表面电荷改造中所获得的正向突变体K154D、K322D和K361D的整体静电势能分别从-63.6 kcal/mol降低为-73.9、-73.3、-68.6 kcal/mol,表明这些突变增加了该酶的表面负电荷。进一步分析了MD模拟获得的突变体K322D/K361D的结构变化(图7),突变体K322D/K361D的RMSD变化幅度低于野生型,表明突变后酶的结构稳定性增加了,且K322D/K361D部分区域较高的均方根波动(root-mean-square fluctuation, RMSF)值也表明了该结构域残基的柔性增强。为了研究在酶表面引入酸性氨基酸对催化性能的影响,测定了突变体K322D/K361D和野生型的表观动力学参数,K322D/K361D对β-CD的催化效率kcat/Km提高到24.83 [mmol/(L·s)],约为WT的1.85倍[13.36 mmol/(L·s)],导致突变酶在低pH下表现较高的底物水解活性。综上,表面净电荷的变化可以改善酶在不同反应溶剂中的构象状态,有利于酶调整其柔性结构的能力,同时呈现新的pH适用范围。
3 结论
本研究通过对类食品乳杆菌来源的麦芽糖淀粉酶进行理性改造,旨在解决其在低pH环境下应用场景受限的问题。改变酶的酸耐受性以获得其在酸性pH值范围内的高活性,理想的做法是将酶的最适pH向酸性方向调节。然而,只有少数案例报道了通过蛋白质工程操纵酶的pH值,当无法改变pH时,增加酸稳定性也是有意义的。本研究基于降低催化中心残基pKa值和表面电荷改造策略,将带正电荷的精氨酸(Arg)放置在LpaMA亲核残基附近,以及将带负电荷的天冬氨酸(Asp)引入LpaMA蛋白质表面,考察了各个突变体的最适pH和酸稳定性,获得4个正向突变位点(L245R、K154D、K322D和K361D)。将正向单点突变位点进行组合突变,获得耐酸性提高的突变株K322D/K361D,其最适反应pH降低至4.5,最大淀粉水解活力提高至433.23 U/mg,为野生型的2.27倍(190.65 U/mg)。此外,K322D/K361D对β-CD的催化效率kcat/Km提高到24.83 [mmol/(L·s)],约为野生型的1.85倍[13.36 mmol/(L·s)],且其在酸性条件(pH 4.0~5.0)下的稳定性保持在68%以上,而野生型的酸稳定性不足53%。突变酶的表面静电势能和结构分析结果表明,Arg的引入增加了亲水性,并降低了亲核残基Asp339的pKa值,稳定了其去质子化形式,同时新的氢键网络也有助于增强区域稳定性。此外,Asp的引入降低了蛋白质表面静电势能,增强了区域残基的柔性,从而优化了酶的构象,提升了酶适应酸性环境的能力。尽管活性位点周围带电残基的分布以及酶总电荷的不同强度显著影响着酶的最适pH变化,但大多数消极的突变结果也表明完全反向的电荷变化可能增加酶结构崩塌的风险并伴随着突变体稳定性的急剧下降。因此,酶的整体状态仍然需要相反电荷离子的存在来维持平衡,特定的静电微环境是维持正确的蛋白质结构和酶的有效催化的关键。
综上,本研究通过理性改造麦芽糖淀粉酶,成功提高了其耐酸性能,为低pH配方发酵面制品的工业生产提供了有力的酶制剂支持,也为后续蛋白质工程在酶制剂领域的应用开辟了新的思路。
[1] LEE H S, KIM M S, CHO H S, et al.Cyclomaltodextrinase, neopullulanase, and maltogenic amylase are nearly indistinguishable from each other[J].Journal of Biological Chemistry, 2002, 277(24):21891-21897.
[2] JONES A, LAMSA M, FRANDSEN T P, et al.Directed evolution of a maltogenic α-amylase from Bacillus sp.TS-25[J].Journal of Biotechnology, 2008, 134(3-4):325-333.
[3] JI H Y, LI X X, JIANG T, et al.A novel amylolytic enzyme from Palaeococcus ferrophilus with malto-oligosaccharide forming ability belonging to subfamily GH13_20[J].Food Bioscience, 2022, 45:101498.
[4] ZHOU J, LI Z K, ZHANG H, et al.Novel maltogenic amylase CoMA from Corallococcus sp.strain EGB catalyzes the conversion of maltooligosaccharides and soluble starch to maltose[J].Applied and Environmental Microbiology, 2018, 84(14):e00152-18.
[5] CHANG R R, LI M, WANG Y F, et al.Retrogradation behavior of debranched starch with different degrees of polymerization[J].Food Chemistry, 2019, 297:125001.
[6] ZHANG W H, WANG J, GUO P P, et al.Study on the retrogradation behavior of starch by asymmetrical flow field-flow fractionation coupled with multiple detectors[J].Food Chemistry, 2019, 277:674-681.
[7] 郭玲玲, 张巍, 史铁嘉.酶制剂在面包品质改良方面的研究进展[J].农业科技与装备, 2010(7):37-39.
GUO L L, ZHANG W, SHI T J.Studies on the action mechanism of enzymic preparations to improve the baking quality of bread[J].Agricultural Science&Technology and Equipment, 2010(7):37-39.
[8] VAN KERREBROECK S, MAES D, DE VUYST L.Sourdoughs as a function of their species diversity and process conditions, a meta-analysis[J].Trends in Food Science &Technology, 2017, 68:152-159.
[9] CHA H J, YOON H G, KIM Y W, et al.Molecular and enzymatic characterization of a maltogenic amylase that hydrolyzes and transglycosylates acarbose[J].European Journal of Biochemistry, 1998, 253(1):251-262.
[10] KIM I C, CHA J H, KIM J R, et al.Catalytic properties of the cloned amylase from Bacillus licheniformis[J].Journal of Biological Chemistry, 1992, 267(31):22108-22114.
[11] CHO H Y, KIM Y W, KIM T J, et al.Molecular characterization of a dimeric intracellular maltogenic amylase of Bacillus subtilis SUH4-2[J].Biochimica et Biophysica Acta (BBA) - Protein Structure and Molecular Enzymology, 2000, 1478(2):333-340.
[12] KIM T J, KIM M J, KIM B C, et al.Modes of action of acarbose hydrolysis and transglycosylation catalyzed by a thermostable maltogenic amylase, the gene for which was cloned from a Thermus strain[J].Applied and Environmental Microbiology, 1999, 65(4):1644-1651.
[13] NIELSEN J E, MCCAMMON J A.Calculating pKa values in enzyme active sites[J].Protein Science, 2003, 12(9):1894-1901.
[14] JOSHI M D, SIDHU G, POT I, et al.Hydrogen bonding and catalysis:A novel explanation for how a single amino acid substitution can change the pH optimum of a glycosidase[J].Journal of Molecular Biology, 2000, 299(1):255-279.
[15] LI Z X, NIU C T, YANG X H, et al.Enhanced acidic resistance ability and catalytic properties of Bacillus 1, 3-1, 4-β-glucanases by sequence alignment and surface charge engineering[J].International Journal of Biological Macromolecules, 2021, 192:426-434.
[16] YANG H Q, LIU L, SHIN H D, et al.Structure-based engineering of histidine residues in the catalytic domain of α-amylase from Bacillus subtilis for improved protein stability and catalytic efficiency under acidic conditions[J].Journal of Biotechnology, 2013, 164(1):59-66.
[17] TURUNEN O, VUORIO M, FENEL F, et al.Engineering of multiple arginines into the Ser/Thr surface of Trichoderma reesei endo-1, 4-β-xylanase Ⅱ increases the thermotolerance and shifts the pH optimum towards alkaline pH[J].Protein Engineering, 2002, 15(2):141-145.
[18] CHEN A N, XU T T, GE Y, et al.Hydrogen-bond-based protein engineering for the acidic adaptation of Bacillus acidopullulyticus pullulanase[J].Enzyme and Microbial Technology, 2019, 124:79-83.
[19] LEE S J, LEE D W, CHOE E A, et al.Characterization of a thermoacidophilic L-arabinose isomerase from Alicyclobacillus acidocaldarius:Role of Lys-269 in pH optimum[J].Applied and Environmental Microbiology, 2005, 71(12):7888-7896.
[20] NIELSEN J E, BEIER L, OTZEN D, et al.Electrostatics in the active site of an α-amylase[J].European Journal of Biochemistry, 1999, 264(3):816-824.
[21] SINNOTT M L.Catalytic mechanism of enzymic glycosyl transfer[J].Chemical Reviews, 1990, 90(7):1171-1202.
[22] HARRIS T K, TURNER G J.Structural basis of perturbed pKa values of catalytic groups in enzyme active sites[J].IUBMB Life, 2002, 53(2):85-98.