硫酸软骨素(chondroitin sulfate, CS)作为糖胺聚糖中的一类酸性多糖,由葡萄糖醛酸(glucuronic acid, GlcA)和N-乙酰氨基半乳糖胺(N-acetylgalactosamine, GalNAc)通过β-1,3和β-1,4糖苷键交替连接,并经磺基转移酶修饰而成[1]。根据磺酸化修饰位点,CS主要分为CSA、CSC、CSD、CSE 4种结构。CS参与细胞分化、增殖、细胞识别和神经网络发育等生物学及病理过程,具有重要的生理功能。同时,它是软骨的重要基质,能为软骨细胞提供营养,并赋予软骨抗变形性能,被广泛应用于医疗和保健品[1]。目前,商业化CS的生产方式主要依赖于动物软骨组织提取,如猪鼻、牛气管、鱼翅等,其中CSA含量占比最高[2]。然而,不同动物来源、年龄以及不同提取部位导致CS批次间差异很大,且存在人畜交叉感染的风险[3]。因此,采用温和绿色的生物酶法合成结构均一、质量稳定的CS备受关注[4-9]。
软骨素4-O-磺基转移酶(chondroitin 4-O-sulfotransferase, C4ST)是合成CSA的关键酶,能够将3′-磷酸腺苷-5′-磷酸硫酸(3′-phosphoadenosine-5′-phosphosulfate, PAPS)中的磺酸基团转移到前体软骨素中GalNAc的4号位羟基,从而形成CSA[9]。目前已知有3种同工酶:C4ST-1、C4ST-2和C4ST-3。研究发现,C4ST-1的缺失会导致生物体内CS水平急剧下降,而C4ST-2和C4ST-3的缺失则未引起类似的效果,这表明C4ST-1在CS合成中具有至关重要的作用[10]。早期C4ST-1的异源表达研究主要在动物细胞中进行表达。近年来,研究人员已尝试在微生物中表达C4ST-1,并取得了一定进展。HE等[4]首次在大肠杆菌中表达人源C4ST-1,但该蛋白主要以包涵体的形式存在。为提高蛋白可溶性,TAKASHIMA等[5]通过低温诱导以及融合伴侣因子策略,在大肠杆菌中成功获得表达水平为18.9 mg/L的TF-C4ST-1。BADRI等[6]基于PROSS服务器在大肠杆菌中构建了人源C4ST-1组合突变体(K117R/H127E/S238Y/A245G),其转化率提高了3倍。LIU等[7]则通过蛋白质工程手段提升其催化效率和稳定性。鉴于糖基化对酶活性的影响[11],HE等[4]在毕赤酵母中表达了人源C4ST-1。
本研究团队前期在毕赤酵母中首次实现了小鼠来源C4ST-1的胞外分泌表达[8]。最近,针对C4ST-1表达量低及酶活性低的问题,本研究团队前期已经通过优化N端氨基酸序列、构建高通量筛选体系以及设计5′非翻译区(5′UTR)提高了C4ST-1表达量,同时采用共表达分子伴侣策略,最终在毕赤酵母中使C4ST-1酶活性表达到1 346.2 U/L[9]。为进一步提升C4ST-1酶活性,降低发酵成本。本研究继续在前期构建的重组毕赤酵母基础上对C4ST-1进行酶活性提升和发酵优化。首先考察3种毕赤酵母底盘细胞对C4ST-1表达的影响,其次通过筛选11种信号肽和9种促溶标签提升了C4ST-1酶活性。结合培养基组分分析,对培养基中碳源、氮源以及生物素成分进行分析,特别是昂贵组分酵母无氨基氮源(yeast nitrogen base without amino acids, YNB)的优化,得到C4ST-1适配性培养基BMG。最后,将重组菌在BMG培养基中进行5 L发酵罐发酵,发酵72 h时获得最高酶活性为5 040.7 U/L,显著提高了C4ST-1的发酵水平,对于推动CSA大规模生产具有重要意义。
1 材料与方法
1.1 实验材料
1.1.1 菌株及质粒
本研究使用的菌株及质粒见表1。
表1 本研究所使用的菌株和质粒
Table 1 Strains and plasmids used in this study

名称特征来源菌株Escherichia coli JM109克隆宿主实验室保藏Komagataella phaffii GS115表达宿主,His4,Mut+实验室保藏K.phaffii GS115-ΔVPS10-1K.phaffii GS115衍生菌株,ΔVPS10-1实验室保藏K.phaffii SMD1163K.phaffii GS115衍生菌株,Δpep4,Δprb1实验室保藏GS115K.phaffii GS115包含质粒pPIC9K-C4ST本研究ΔVPS10-1K.phaffii GS115-ΔVPS10-1包含质粒pPIC9K-C4ST本研究SMD1163K.phaffii SMD1163包含质粒pPIC9K-C4ST本研究Exg1pK.phaffii GS115包含质粒pPIC9K-Exg1p-C4ST本研究9AK.phaffii GS115包含质粒pPIC9K-9A-C4ST本研究α-amylaseK.phaffii GS115包含质粒pPIC9K-α-amylase-C4ST本研究Dane4K.phaffii GS115包含质粒pPIC9K-Dane4-C4ST本研究Dse4K.phaffii GS115包含质粒pPIC9K-Dse4-C4ST本研究IgG1K.phaffii GS115包含质粒pPIC9K-IgG1-C4ST本研究Fre2K.phaffii GS115包含质粒pPIC9K-Fre2-C4ST本研究OST1-αK.phaffii GS115包含质粒pPIC9K-OST1-α-C4ST本研究SP23K.phaffii GS115包含质粒pPIC9K-SP23-C4ST本研究Gas1K.phaffii GS115包含质粒pPIC9K-Gas1-C4ST本研究UNIK.phaffii GS115包含质粒pPIC9K-UNI-C4ST本研究SUMO StarK.phaffii GS115包含质粒pPIC9K-OST1-α-SUMO Star-C4ST本研究FLAGK.phaffii GS115包含质粒pPIC9K-OST1-α-FLAG-C4ST本研究GB1K.phaffii GS115包含质粒pPIC9K-OST1-α-GB1-C4ST本研究GSTK.phaffii GS115包含质粒pPIC9K-OST1-α-GST-C4ST本研究MBPK.phaffii GS115包含质粒pPIC9K-OST1-α-MBP-C4ST本研究TrxAK.phaffii GS115包含质粒pPIC9K-OST1-α-Trx A C4ST本研究SUMO Pro 3K.phaffii GS115包含质粒pPIC9K-OST1-α-SUMO Pro 3-C4ST本研究DEEP1K.phaffii GS115包含质粒pPIC9K-OST1-α-DEEP1-C4ST本研究NT∗K.phaffii GS115包含质粒pPIC9K-OST1-α-NT∗-C4ST本研究质粒pPIC9K分泌表达质粒,KanR,AmpR实验室保藏pPIC9K-C4STα信号肽连接带有融合标签SUMO的截短C4ST-1实验室保藏[9]pPIC9K-Exg1p-C4ST将质粒pPIC9K-C4ST的α信号肽替换成Exg1p本研究pPIC9K-9A-C4ST将质粒pPIC9K-C4ST的α信号肽替换成9A本研究pPIC9K-α-amylase-C4ST将质粒pPIC9K-C4ST的α信号肽替换成α-amylase本研究pPIC9K-Dane4-C4ST将质粒pPIC9K-C4ST的α信号肽替换成Dane4本研究质粒pPIC9K-Dse4-C4ST将质粒pPIC9K-C4ST的α信号肽替换成Dse4本研究
续表1
名称特征来源pPIC9K-IgG1-C4ST将质粒pPIC9K-C4ST的α信号肽替换成IgG1本研究pPIC9K-Fre2-C4ST将质粒pPIC9K-C4ST的α信号肽替换成Fre2本研究pPIC9K-OST1-α-C4ST将质粒pPIC9K-C4ST的α信号肽替换成OST1-α本研究pPIC9K-SP23-C4ST将质粒pPIC9K-C4ST的α信号肽替换成SP23本研究pPIC9K-Gas1-C4ST将质粒pPIC9K-C4ST的α信号肽替换成Gas1本研究pPIC9K-UNI-C4ST将质粒pPIC9K-C4ST的α信号肽替换成UNI本研究pPIC9K-OST1-α-SUMO Star-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成SUMO Star本研究pPIC9K-OST1-α-FLAG-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成FLAG本研究pPIC9K-OST1-α-GB1-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成GB1本研究pPIC9K-OST1-α-GST-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成GST本研究pPIC9K-OST1-α-MBP-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成MBP本研究pPIC9K-OST1-α-Trx A-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成Trx A本研究pPIC9K-OST1-α-SUMO Pro 3-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成SUMO Pro 3本研究pPIC9K-OST1-α-DEEP1-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成DEEP1本研究pPIC9K-OST1-α-NT∗-C4ST将质粒pPIC9K-OST1-α-C4ST的SUMO标签替换成NT∗本研究
1.1.2 主要试剂
质粒DNA抽提试剂盒、无缝克隆试剂盒、YNB、生物素、硫酸卡那霉素及遗传霉素,上海生工生物工程公司;胰蛋白胨、酵母提取物及脑心浸液,美国OXOID公司;大豆蛋白胨,北京沃凯生物科技有限公司;玉米浆、鱼蛋白胨,上海源叶生物科技有限公司;尿素,北京伊诺凯科技有限公司;酪蛋白胨、棉籽蛋白胨、牛肉蛋白胨、植物蛋白胨,上海麦克林生化科技有限公司;FP108酵母蛋白胨、FP103酵母蛋白胨、FP318胰蛋白胨、FP400大豆蛋白胨、FP220小麦蛋白胨,安琪酵母股份有限公司;限制性内切酶Sal I、蛋白质标准分子质量Marker、SDS-PAGE预制胶、DNA片段回收试剂盒,赛默飞世尔科技公司;PrimerSTAR Max DNA聚合酶,宝日医生物技术有限公司;磷酸盐、牛肉浸膏,国药集团试剂有限公司;其余试剂均为国产分析纯试剂。
1.1.3 仪器与设备
UV2450紫外分光光度计,日本Shimadzu公司;5L发酵罐,迪必尔上海生物工程有限公司;恒温摇床,上海知楚仪器有限公司;PCR仪、凝胶成像系统及电转化仪,美国Bio-Rad公司;电热恒温水浴锅,森信实验仪器有限公司;离心机,德国Eppendorf公司。
1.1.4 培养基
LB种子培养基(g/L):酵母提取物5,蛋白胨10,NaCl 10。
YPD培养基(g/L):酵母提取物10,蛋白胨20,葡萄糖20。
MD(minimal dextrose)培养基(g/L):葡萄糖20,YNB 13.4,生物素4×10-4,琼脂粉20。
BMGY(buffered glycerol-complex medium)培养基(g/L):酵母提取物10,胰蛋白胨20,K2HPO4·3H2O 3.6,KH2PO4 11.8,生物素4×10-4,YNB 13.4,甘油10。
BMMY(buffered methanol-complex medium)培养基(g/L):酵母提取物10,胰蛋白胨20,K2HPO4·3H2O 3.6,KH2PO4 11.8,生物素4×10-4,YNB 13.4。
BMG改良培养基(g/L):酵母提取物10,棉籽蛋白胨20,K2HPO4·3H2O 3.6,KH2PO4 11.8,生物素1×10-4,甘油40。
1.2 实验方法
1.2.1 表达质粒构建
使用表2引物以pPIC9K-C4ST为模板进行扩增,将α信号肽整体或部分替换成Exg1p、9A、α-amylase、Dane4、Dse4、IgG1、Fre2、OST1、SP23、Gas1、UNI信号肽。以Exg1p为例,利用引物对Exg1p-F/Exg1p-R以pPIC9K-C4ST质粒为模板进行PCR扩增,然后将其转入E.coli JM109感受态,测序成功后获得重组质粒pPIC9K-Exg1p-C4ST。其余10种信号肽同Exg1p。对于促溶标签SUMO Star、FLAG以及GB1,分别利用引物对SUMO Star-F/SUMO Star-R、FLAG-F/FLAG-R以及GB1-1-F/GB1-1-R,以pPIC9K-OST1-α-C4ST质粒为模板进行PCR扩增,其中GB1还需以PCR产物为模板,利用引物对GB1-2-F/GB1-2-R进行二段PCR。将PCR产物转入E.coli JM109感受态,测序成功后获得重组质粒pPIC9K-OST1-α-SUMO Star-C4ST、pPIC9K-OST1-α-FLAG-C4ST以及pPIC9K-OST1-α-GB1-C4ST。促溶标签GST、MBP和TrxA则按照无缝克隆试剂盒说明,通过同源重组整合至重组质粒pPIC9K-OST1-α-C4ST中。以GST为例,利用引物对GST-ZT-F/GST-ZT-R以pPIC9K-OST1-α-C4ST质粒为模板进行PCR扩增质粒骨架,利用引物对GST-F/GST-R以含有GST基因质粒为模板进行PCR扩增GST片段,按无缝克隆试剂盒说明进行相应操作后转入E.coli JM109感受态,测序成功后获得重组质粒pPIC9K-OST1-α-GST-C4ST。促溶标签SUMO Pro 3、DEEP1以及NT*基因由赛索飞生物科技有限公司根据毕赤酵母密码子偏好性合成,并克隆至质粒pPIC9K-OST1-α-C4ST中以替换原有的SUMO标签。
表2 本研究所用引物
Table 2 Primers used in this study

引物名称引物序列(5′-3′)Exg1p-FGATCACTTTGTTGTTCGCCTCTTTGTGCTCCGCTTACGTACACCATCATCACCATCACATGExg1p-RGAGGCGAACAACAAAGTGATCAAGTACAAGTTCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTG9A-FCAACTTTTCTTGTTGTTGGTTCTGTTGACCAATATAGTCAGTGGATACGTACACCATCATCACCATCACATG9A-RGAACCAACAACAAGAAAAGTTGTGGCACGTTGGAAGAGAATGACATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGα-amylase-FTGTTTTTGTACGGTTTGCAAGTTGCTGCTCCAGCTTTGGCTTACGTACACCATCATCACCATCACATGα-amylase-RTTGCAAACCGTACAAAAACAAAGACCACCAAGCAACCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGDan4-FCTATTTTGACATTATGTAAGGCCTGGGACTTGGAGGATGTCCAGGACGCCCCTAAGTACGTACACCATCATCACCATCACATGDan4-RCTTACATAATGTCAAAATAGAAGCAAAAGACAGCAAAGACTTCAAGAACATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGDse4-FCACCACAGCTTTTTCTGTTGTTGGTTCTGTTGACTAATATAGTCAGTGGGTACGTACACCATCATCACCATCACATGDse4-RCAACAGAAAAAGCTGTGGTGCTTTAGAAAAGAATGACATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGFre2-FGTTCTTATTGTTGACTGTTGCCGCCCAAGCCCATTTGGTTACTTTCCACTCTACCGAATACGTACACCATCATCACCATCACATGFre2-RCAACAGTCAACAATAAGAACAAAACGACTAGATCATTCAGATGGTTACGCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGGas1-FGTTTGTCCTGCGCATCTGACCTGACACCACCAATTGAAGTAACCGGAAACAAGTTTTTCTTCTACGTACACCATCATCACCATCACATGGas1-RGTCAGATGCGCAGGACAAACCCAAAAGAGTCAAAGCGGAAAGAATACTCAACATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGIgG1-FCTGTTCTTGGTTGCTACTGCAACTGGTGTTCATAGTCAGTACGTACACCATCATCACCATCACATGIgG1-RGCAGTAGCAACCAAGAACAGGATGATACAGGACCAACCCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGOST1-FGATTGTGGGATTGTTCCTATGTTTTTTCAACGTGTCTTCTGCTGCTCCAGTCAACACTACAACAGAAGATGOST1-RCATAGGAACAATCCCACAATCCAAGAGAACCAAACCTGCCTCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGSP23-FCATTGCTTCTTCTTTTTACGTTGGCCTTTGCTTACGTACACCATCATCACCATCACATGSP23-RCGTAAAAAGAAGAAGCAATGCACTTAATATTTTCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGUNI-FGTTGCTGCCTTTGGCAGGAGTTTCAGCTTACGTACACCATCATCACCATCACATGUNI-RCTCCTGCCAAAGGCAGCAACAGGGAGTATGCCAGCTTCATCCTTTTTCTGCTTCGAATAATTAGTTGTTTTTTGSUMOstar-FTTCTTGTACGACGGTATTGAAATTCAAGCTGATCAGACCCCTGAAGSUMOstar-RAATACCGTCGTACAAGAAAGTTAAGGAGTCCATTTCCTTACCCTGTCTTTTAGFLAG-FGATTACAAGGATGATGACGATAAAGCTGAGGCTGCCGCTAAGFLAG-RTTTATCGTCATCATCCTTGTAATCGTGATGGTGATGATGGTGTACGTAAGCGST-ZT-FGCTGAGGCTGCCGCTAAGGST-ZT-RGTGATGGTGATGATGGTGTACGTAAGCGST-FTACACCATCATCACCATCACATGTCCCCTATACTAGGTTATTGGAAAATTAAGGGGST-RTCCTTAGCGGCAGCCTCAGCTTTTGGAGGATGGTCGCCACMBP-FTACACCATCATCACCATCACATGAAAATCGAAGAAGGTAAACTGGTAATCTGGMBP-RTCCTTAGCGGCAGCCTCAGCAGTCTGCGCGTCTTTCAGGTrxA-FTACACCATCATCACCATCACATGAGCGATAAAATTATTCACCTGACTGACGTrxA-RTCCTTAGCGGCAGCCTCAGCGGCCAGGTTAGCGTCGAGGGB1-1-FAATGGAGTCGACGGTGAGTGGACTTACGATGATGCCACCAAAACGTTCACTGTCACAGAAGCTGAGGCTGCCGCTAAGGB1-1-RTTCAGTCGTGGTTTCGCCTTTCAATGTCTTTCCGTTAAGTATCAATTTATATGTATCCATGTGATGGTGATGATGGTGTACGTAAGCGB1-2-FACGGCTGAGAAGGTGTTCAAGCAGTACGCCAACGATAATGGAGTCGACGGTGAGTGGGB1-2-RTTGAACACCTTCTCAGCCGTAGCGGCATCAACAGCTTCAGTCGTGGTTTCGCCTTTC
注:下划线为同源臂序列。
1.2.2 重组菌株的构建
将测序正确的质粒经Sal I酶切(37 ℃,2 h)线性化,并纯化回收。随后电转化至相应毕赤酵母感受态细胞(条件参考伯乐电转仪说明书),30 ℃、220 r/min振荡培养40~60 min,4 500 r/min离心1 min,弃上清液至约100 μL,重悬后涂布MD平板,30 ℃培养2~3 d。挑取转化子接种含4 mg/mL G418的YPD平板,30 ℃培养2~3 d,筛选阳性克隆用于保藏或后续实验。
1.2.3 重组毕赤酵母菌摇瓶发酵
将单菌落接种于7.5 mL YPD液体培养基中,30 ℃、220 r/min振荡培养18 h,以10%接种量接种于45 mL BMGY培养基中,30 ℃、220 r/min继续振荡培养24 h,将发酵液在4 ℃、4 000 r/min条件下离心3 min,弃上清液;用25 mL 0.9%(质量分数)NaCl溶液清洗菌体3次;随后用BMMY培养基重悬并定容至50 mL,30 ℃、220 r/min诱导表达96 h,每24 h添加1%(体积分数)终体积的甲醇。发酵结束后,将发酵液在4 ℃、4 000 r/min离心15 min,收集发酵上清用于后续C4ST-1酶活性测定及SDS-PAGE分析。
1.2.4 C4ST-1酶活性的测定
C4ST-1酶活性测定如前所述[9],以对硝基苯酚(p-nitrophenol,PNP)的生成量作为酶活性的标志。在一定范围内,PNP的浓度与400 nm处的吸光度呈线性关系。在1.5 mL的反应体系中添加50 mmol/L对硝基硫酸苯酯(p-nitrosulfate,PNPS)、0.5 mmol/L 3′-磷酸腺苷-5′-磷酸(3′-adenosine phosphate-5′-phosphate,PAP)、2 mg/mL酰基磺酸转移酶(aryl sulfotransferase,AST Ⅳ)、20%(体积分数)甘油、2 mg/mL软骨素以及400 μL的C4ST-1发酵上清液,在37 ℃下反应2 h。沸水浴10 min以终止反应,然后在12 000 r/min下离心15 min,取上清液测定400 nm处的吸光度。C4ST-1的酶活单位定义为在特定反应条件(37 ℃,pH 7.4)下每小时产生1 μmol/L PNP所需的酶量。
1.2.5 重组毕赤酵母5 L罐发酵
将重组毕赤酵母在YPD平板上30 ℃培养2~3 d,挑取单菌落接种在含有50 mL YPD的250 mL摇瓶中,30 ℃、220 r/min培养24 h作为一级种子液。将一级种子液按10%(体积分数)的转接量转接至含有180 mL YPD的1 L摇瓶中,30 ℃、220 r/min培养24 h作为二级种子液。将二级种子液按照10%的接种量至含有1.8 L BMG改良培养基的5 L发酵罐中。以50%氨水控制pH 6.0,设定初始发酵温度、搅拌转速和通气量分别为30 ℃、200 r/min和2 L/min;随着发酵进行逐步调整转速至500 r/min,发酵过程中将溶氧控制在30%以下。若溶氧量在呈升高趋势且超过60%,则代表罐内甘油耗尽,可以进入甲醇诱导阶段。饥饿培养1 h后,通过逐级添加甲醇流速以及搅拌转速,将溶氧控制在30%以下。整个发酵期间每隔12 h进行取样,测定发酵液OD600和C4ST-1酶活性。
1.2.6 数据处理与分析
每个样品独立取样并分别测定,使用Excel进行数据统计与整理,使用Graphpad Prism 8.0以及Adobe Illustrator 2023对数据进行可视化处理。
2 结果与分析
2.1 不同底盘细胞对C4ST-1分泌表达的影响
毕赤酵母已成为常用的真核生物表达系统之一,其优越的翻译后修饰能力、强而严格的启动子、简便的培养条件,使其在分泌表达外源蛋白方面具有显著优势[12]。不同的毕赤酵母底盘细胞的甲醇利用能力、基因型、分泌能力以及工业生产特性均有所差异[12-14]。为此,对比了GS115、ΔVPS10-1和SMD1163 3种毕赤酵母底盘细胞对C4ST-1的表达能力。GS115是一种广泛应用于重组蛋白表达的经典毕赤酵母底盘细胞,凭借着其高效的诱导表达能力及良好的工业发酵适应性,正逐渐成为工业规模化生产的重要菌种[12]。ΔVPS10-1是基于GS115株,通过敲除液泡分选受体蛋白Vps10的结构域Ⅰ来降低液泡的错误分选[13];而SMD1163则在GS115的基础上通过突变pep4和prb1基因,导致蛋白水解酶A和蛋白水解酶B活性的丧失,从而保护外源蛋白免受降解[14]。将C4ST-1转入3种毕赤酵母底盘细胞,结果见图1。在0~24 h重组菌GS115、ΔVPS10-1和SMD1163的OD600基本一致。在甲醇诱导(24 h)开始后,随着培养时间的延长,重组菌GS115的OD600逐渐高于另外2株重组底盘细胞,这可能是由于基因敲除对细胞甲醇耐受性的影响,从而限制了菌体的生长。通过测定C4ST-1酶活性发现重组菌GS115酶活性明显高于另外2株重组菌,且经SDS-PAGE分析显示重组菌GS115条带较粗。因此,选择毕赤酵母GS115作为后续研究的底盘细胞。
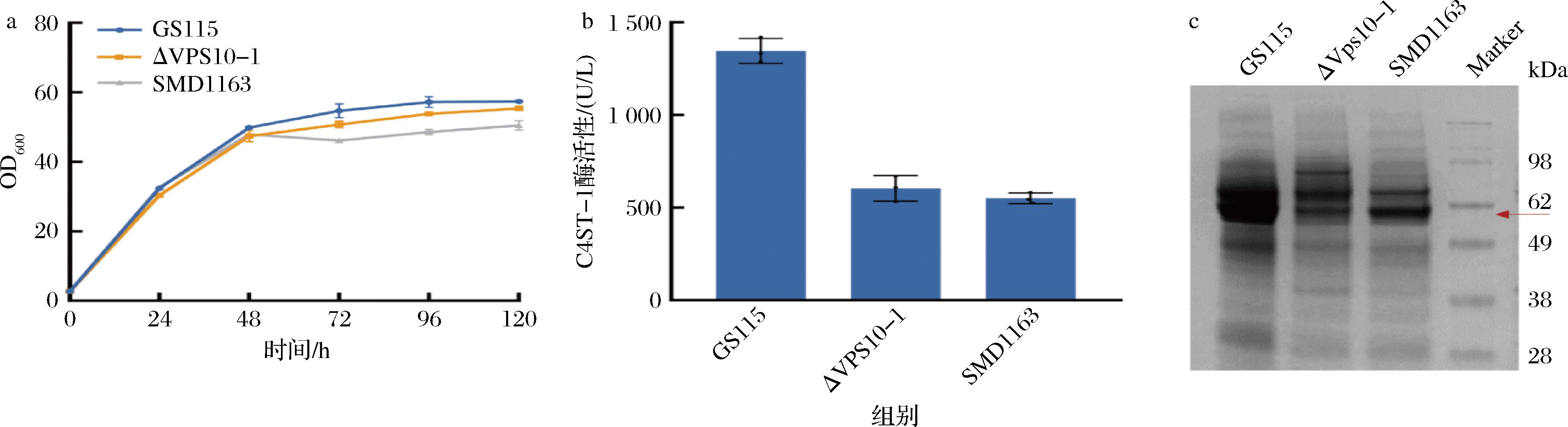
a-不同底盘细胞生长曲线测定;b-酶活性测定;c-SDS-PAGE分析
图1 不同底盘细胞对C4ST-1分泌表达的影响
Fig.1 Effect of different chassis cells on C4ST-1 secretory expression
2.2 不同信号肽对C4ST-1酶活性的影响
信号肽通过引导目标蛋白进入分泌途径在分泌表达中发挥关键作用,其序列特异性直接影响蛋白的分泌效率、正确折叠和活性。不同信号肽的引导机制和效率存在差异,针对特定目的蛋白选择合适信号肽以实现高效分泌表达[15-16]。基于此,筛选了11个在文献中报道效果较好的信号肽[15-21],分别命名为Exg1p(1.8 kDa,Gene ID:8198921)、9A(2.5 kDa,Gene ID:8197918)、α-amylase(2.2 kDa,Gene ID:64846086)、Dan4(3.3 kDa,Gene ID:8198555)、Dse4(2.5 kDa,Gene ID:8197918)、IgG1(2.1 kDa,Gene ID:674018)、Fre2(3.4 kDa,Gene ID:8200683)、OST1-α(10.4 kDa,OST1 Gene ID:853455)、sp23(1.8 kDa,Gene ID:8196664)、Gas1(3.4 kDa,Gene ID:8196459)以及UNI(1.6 kDa,Gene ID:34714176),按图2-a的方式进行构建。将重组菌在摇瓶中培养96 h后,发酵液离心收集上清液,测定C4ST-1酶活性。如图2-b所示,融合杂合信号肽OST1-α的C4ST-1酶活性最高为1 634.3 U/L,较原始菌株提高了21.4%。然而,融合其他信号肽的C4ST-1酶活性都表现降低,相应的SDS-PAGE如图2-c所示。因此,选择OST1-α作为后续研究中的信号肽。
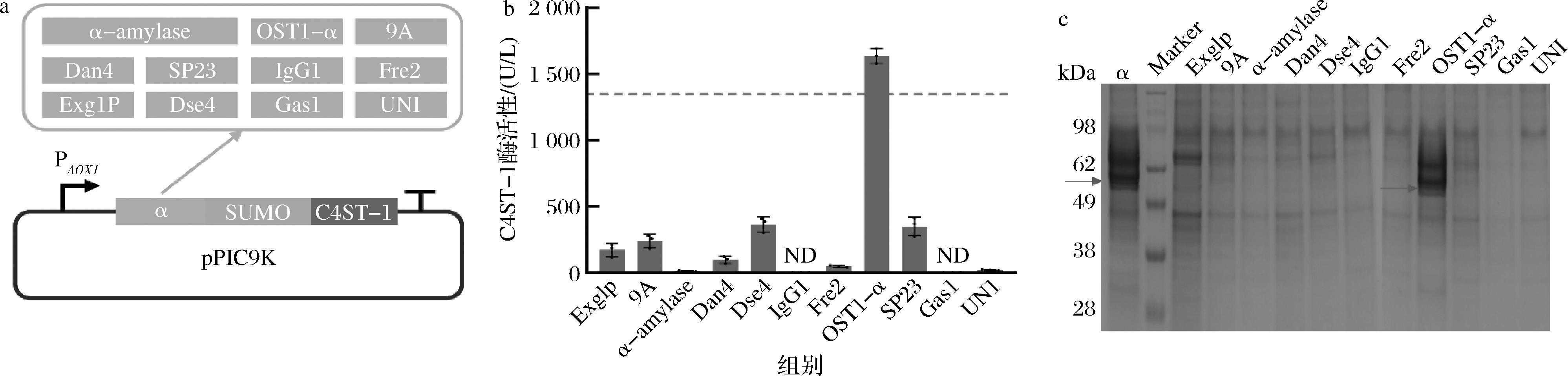
a-信号肽构建示意图;b-酶活性测定;c-SDS-PAGE分析
图2 不同信号肽融合对C4ST-1酶活性的影响
Fig.2 Effect of different signal peptide fusions on C4ST-1 enzyme activity
2.3 不同促溶标签对C4ST-1酶活性的影响
膜蛋白由于其强烈的疏水性,一直在可溶表达方面面临困难[22]。促溶标签是一类能够改善目标蛋白溶解性的短肽序列,通过改变目标蛋白的折叠方式或与宿主细胞内的分子机制相互作用,增强目标蛋白在细胞中的溶解度[23]。由此在2.2节的基础上又筛选了9种在真核系统中报道效果较好的促溶标签[23-27],分别为SUMO Star、FLAG、GST、MBP、Trx A、GB1、SUMO Pro 3、DEEP1和NT*,按图3-a的方式进行构建,结果见图3-b。经测定C4ST-1酶活性发现,融合SUMO Star、FLAG、GB1以及SUMO Pro 3标签表现为酶活性升高,其他融合标签显示酶活性降低,其中融合SUMO Pro 3标签酶活性最高为1 889.2 U/L,较原始菌株提高了40.3%。相应重组菌发酵上清液的SDS-PAGE如图3-c所示。因此,最终确定GS115为底盘细胞,OST1-α为信号肽,融合SUMO Pro 3标签的重组菌用于下一步培养基优化。
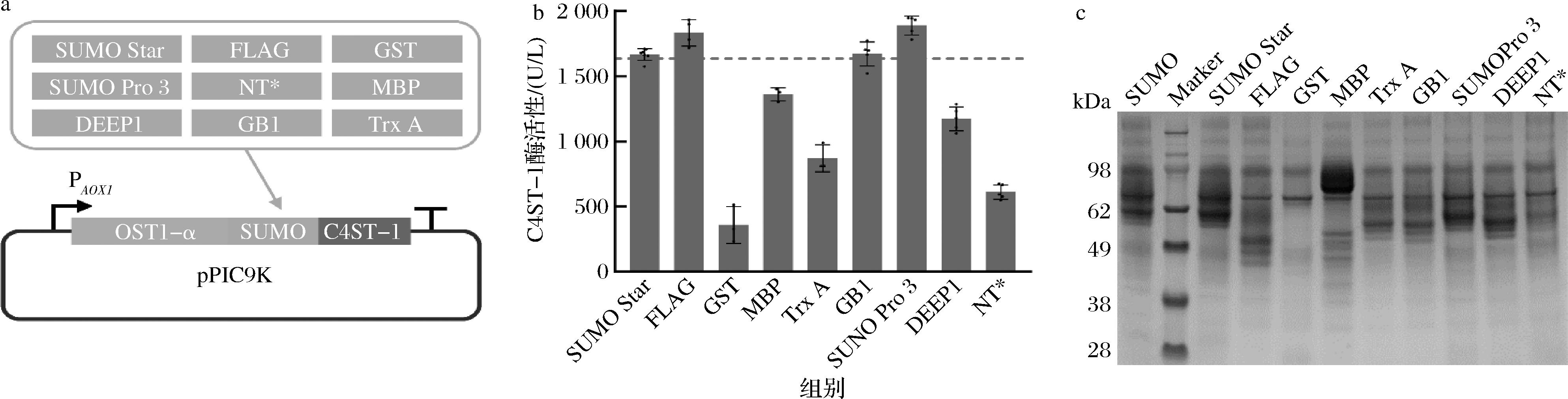
a-促溶标签构建示意图;b-酶活性测定;c-SDS-PAGE分析
图3 不同促溶标签融合对C4ST-1酶活性的影响
Fig.3 Effect of different solubility-enhancing tag fusions on C4ST-1 enzyme activity
2.4 碳源和微量元素优化
前期研究发现,重组C4ST-1毕赤酵母菌在BMGY培养基中能够正常分泌表达C4ST-1,而在BSM培养基中,由于离子成分的影响,酶活性几乎消失[9]。因此,本研究着眼于BMGY培养基优化以增强C4ST-1的生产效率。首先对培养基组分含量进行分析,见图4-a。BMGY培养基组分大致可分为4类:碳源、氮源、缓冲盐和微量元素(生物素)。其中,氮源包含胰蛋白胨、酵母粉和YNB,总含量占比63%。碳源和缓冲盐含量占比分别为15%和22%,微量元素生物素占比不到0.001%。其次对培养基组分价格进行分析,见图4-b。结果表明氮源占比最大,为总价格的97%。胰蛋白胨、酵母粉和YNB的价格占比分别为37%、9%和51%。基于培养基组分分析,对各组分进行优化,旨在实现降本增效。
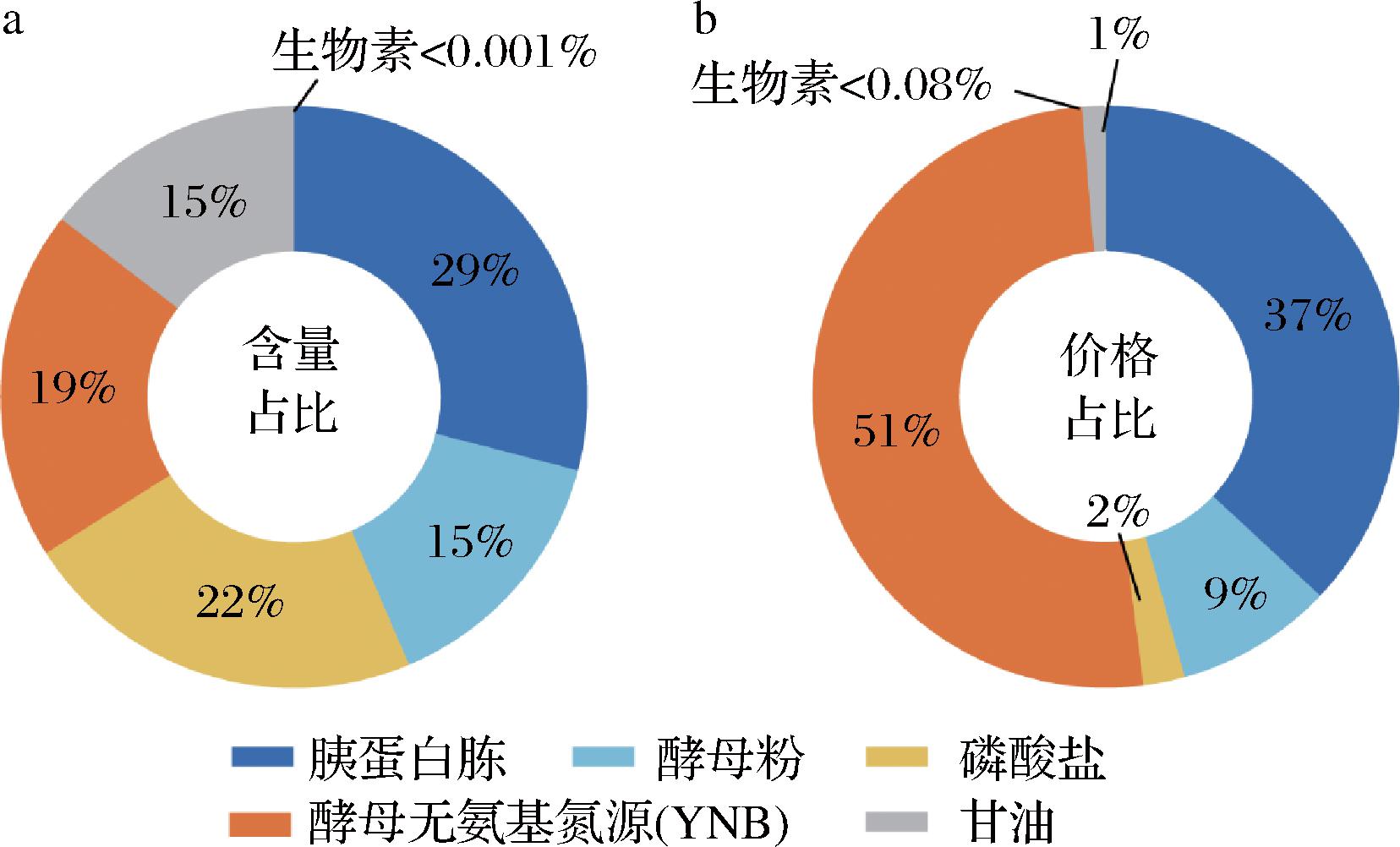
a-含量占比;b-价格占比
图4 BMGY培养基组分含量占比和价格占比
Fig.4 Content ratio and price ratio of BMGY medium components
首先比较了甘油、葡萄糖、山梨醇、甘露醇和蔗糖5种碳源对菌体生长和C4ST-1酶活性的影响,见图5-a。结果显示,以山梨醇、甘露醇和蔗糖为碳源OD600都明显下降,且C4ST-1酶活性降低。相比之下,以甘油和葡萄糖为碳源表现为OD600正常,但以葡萄糖为碳源测定的C4ST-1酶活性仅是甘油酶活性的49.8%。推测该结果可能是葡萄糖代谢产生的乙醇抑制了醇氧化酶1(alcohol oxidase 1, AOX1)活性,导致甲醇诱导强度下降,进而影响酶活性[28]。此外,尽管生物素在培养基中的含量占比较低,但作为一种水溶性维生素,它在糖异生、脂肪酸合成和氨基酸分解等代谢途径中发挥着重要作用[29]。过高或过低的生物素浓度都会打破细胞的代谢平衡,从而影响外源蛋白的表达。因此,探究了不同生物素浓度对C4ST-1酶活性的影响,结果如图5-b所示,当生物素质量浓度为1×10-4 g/L时,酶活性较初始提高了11.5%。因此选择甘油为碳源,生物素质量浓度为1×10-4 g/L。
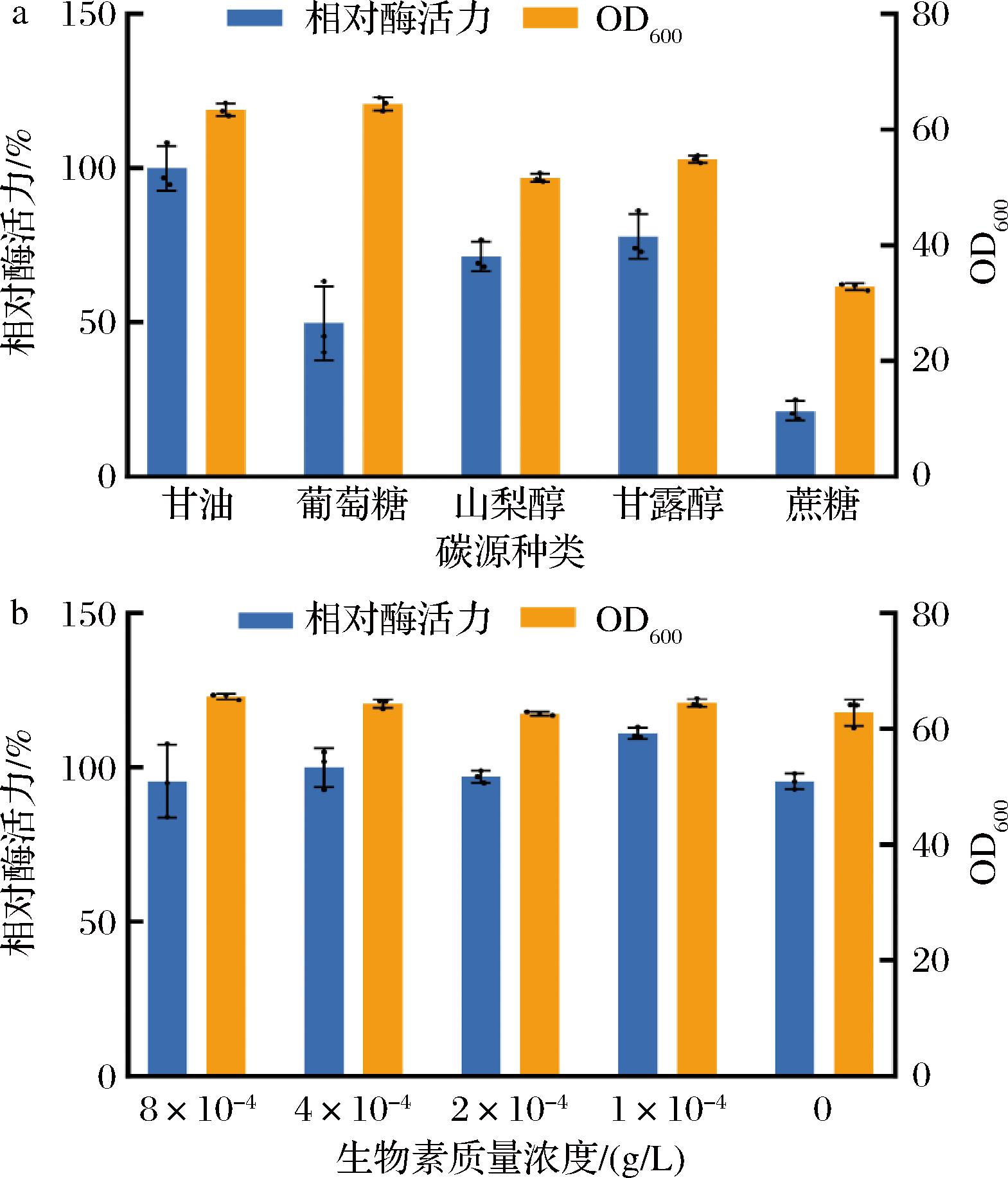
a-不同碳源对菌体生长及C4ST-1酶活性的影响;b-不同生物素质量浓度对菌体生长及C4ST-1酶活力的影响
图5 碳源和生物素优化
Fig.5 Optimization of carbon sources and biotin
2.5 氮源优化
基于2.4节组分分析,氮源中胰蛋白胨和YNB价格较高,限制了工业化生产。因此,首先筛选了常见的15种氮源等量替换20 g/L胰蛋白胨进行OD600和C4ST-1酶活性测定。如图6-a所示,相比胰蛋白胨,以便宜棉籽蛋白胨和酪蛋白胨为氮源,C4ST-1酶活性分别提高3.6%和3.3%,而其他碳源都表现为C4ST-1酶活性降低。最终,综合考虑价格因素后,选择棉籽蛋白胨作为后续氮源。此外,将20 g/L的胰蛋白胨替换为等量的尿素时,发现未检测到C4ST-1酶活性,推测可能是由于尿素代谢生成氨,高浓度氨抑制某些代谢通路的活性,进而影响了C4ST-1的表达效率。同时,进一步对YNB组分进行了详细分析,见图6-b,其主要成分为(NH4)2SO4(75%)、KH2PO4(15%)、MgSO4(7%)、NaCl(2%)和CaCl2(1%)。将这5种成分命名为“核心五盐”,其中(NH4)2SO4含量最高。采用逐级递减方式筛选非必要组分,即为原YNB分别替换为核心五盐[包含10 g/L (NH4)2SO4,2 g/L KH2PO4,1 g/L MgSO4,0.2 g/L NaCl以及0.2 g/L CaCl2]、10 g/L (NH4)2SO4、5 g/L (NH4)2SO4以及不添加YNB。结果如图6-c所示,随着组分的减少,酶活性呈现出升高的趋势,这也验证了高浓度的氨会影响C4ST-1的活性。特别地,在不额外添加YNB的情况下,酶活性较初始培养基提升了68.4%,而OD600没有受到影响。至此,将优化后的培养基命名为BMG培养基,采用该培养基进行摇瓶发酵,酶活性可达3 447.8 U/L,较初始培养基提高82.5%。
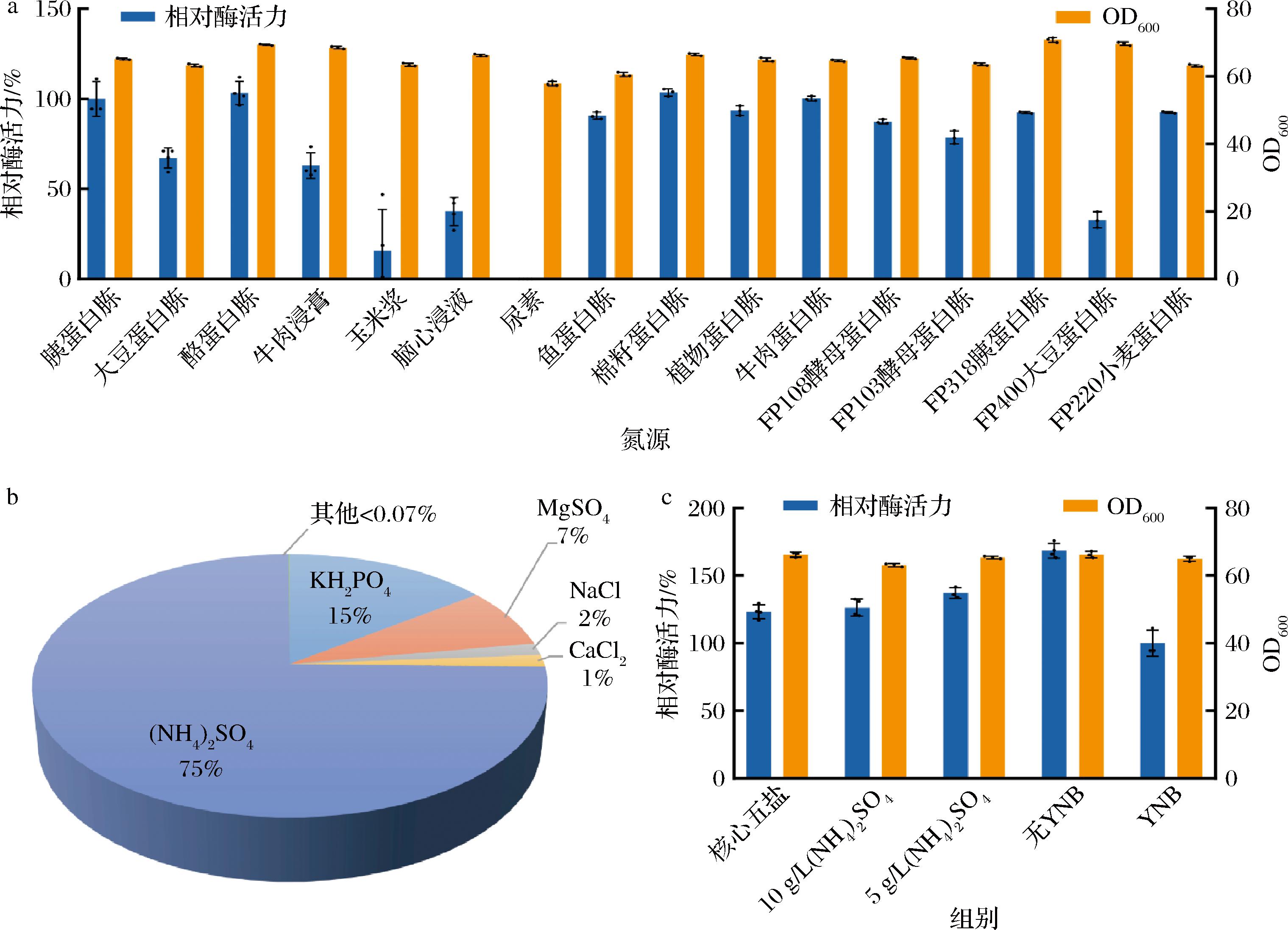
a-不同氮源对菌体生长及C4ST-1酶活性的影响;b-YNB组分分析;c-YNB组分对菌体生长及C4ST-1酶活性的影响
图6 氮源优化
Fig.6 Optimization of nitrogen sources
2.6 5L发酵罐放大培养
为评估重组菌的生产能力以及验证BMG培养基的工业化能力,采用5 L发酵罐进行补料分批发酵,结果如图7所示。在0~72 h内,随着发酵时间延长,酶活性和OD600都不断的上升。当发酵18 h时培养基中甘油耗尽,饥饿1 h后开始甲醇诱导。当发酵72 h时,OD600达到226.8,C4ST-1的酶活性达到最高值,为5 040.7 U/L。72 h之后,OD600缓慢增加到259.3稳定,而酶活性开始出现缓慢下降,推测可能是受C4ST-1酶稳定性影响。本研究通过对重组菌生产C4ST-1的放大培养分析,为后续大规模生物合成CSA奠定了坚实的基础。
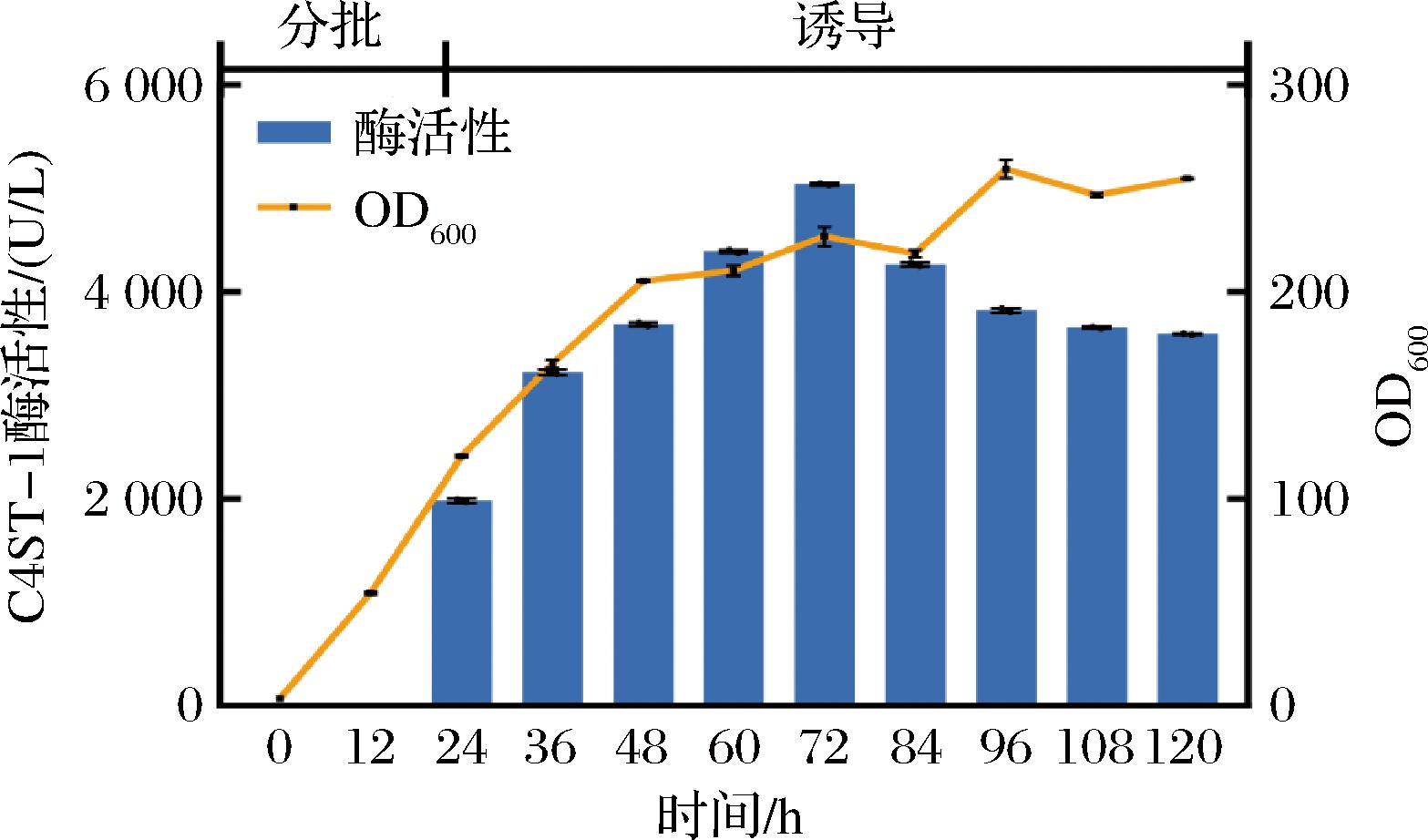
图7 补料分批发酵生产C4ST-1
Fig.7 Fed-batch fermentation for C4ST-1 production
3 结论
本研究在前期研究基础上,继续分别从重组菌构建和培养基组分分析2个方面对C4ST-1进行表达优化。首先通过对3种毕赤酵母底盘细胞、11种信号肽以及9种促溶标签的筛选,将酶活性提升至1 889.2 U/L,较原始菌株提高40.3%。其次,针对BMGY培养基进行组分优化,发现不添加昂贵的YNB时不仅未影响菌体生长,且C4ST-1酶活性较原始培养基提高68.4%。最后,结合其他培养基组分的筛选与优化,在5L发酵罐补料分批发酵72 h时C4ST-1酶活性达到最高为5 040.7 U/L。本研究打破了常规毕赤酵母通用型发酵培养基组分,且通过多种策略组合极大的提升了C4ST-1酶活性,对推动CSA的规模化生产具有重要的意义。针对发酵后期酶活性下降问题,后续可通过分子改造进一步提高C4ST-1的催化活性及其稳定性,亦或者对发酵过程进一步优化来提高整体发酵水平。
[1] 赵琳, 云振宇, 吴希, 等.硫酸软骨素钠的制备及抗骨关节炎活性研究进展[J].医药导报, 2024, 43(6):916-925.ZHAO L, YUN Z Y, WU X, et al.Research progress on preparation and anti-osteoarthritis activity of chondroitin sulfate sodium[J].Herald of Medicine, 2024, 43(6):916-925.
[2] MARTEL-PELLETIER J, FARRAN A, MONTELL E, et al.Discrepancies in composition and biological effects of different formulations of chondroitin sulfate[J].Molecules, 2015, 20(3):4277-4289.
[3] VOLPI N.Quality of different chondroitin sulfate preparations in relation to their therapeutic activity[J].Journal of Pharmacy and Pharmacology, 2009, 61(10):1271-1280.
[4] HE W Q, ZHU Y Y, SHIRKE A, et al.Expression of chondroitin-4-O-sulfotransferase in Escherichia coli and Pichia pastoris[J].Applied Microbiology and Biotechnology, 2017, 101(18):6919-6928.
[5] TAKASHIMA M, SUZUKI K, MOCHIZUKI H, et al.Expression of highly active chondroitin 4-O-sulfotransferase-1 in Escherichia coli by a trigger factor fusion protein expression system[J].Process Biochemistry, 2022, 115:146-151.
[6] BADRI A, WILLIAMS A, AWOFIRANYE A, et al.Complete biosynthesis of a sulfated chondroitin in Escherichia coli[J].Nature Communications, 2021, 12:1389.
[7] LIU H Y, WEI W Q, PANG Z X, et al.Protein engineering, cofactor engineering, and surface display engineering to achieve whole-cell catalytic production of chondroitin sulfate A[J].Biotechnology and Bioengineering, 2023, 120(7):1784-1796.
[8] ZHOU Z X, LI Q, HUANG H, et al.A microbial-enzymatic strategy for producing chondroitin sulfate glycosaminoglycans[J].Biotechnology and Bioengineering, 2018, 115(6):1561-1570.
[9] ZHANG W J, ZHANG P, WANG H, et al.Enhancing the expression of chondroitin 4-O-sulfotransferase for one-pot enzymatic synthesis of chondroitin sulfate A[J].Carbohydrate Polymers, 2024, 337:122158.
[10] KLÜPPEL M, WIGHT T N, CHAN C, et al.Maintenance of chondroitin sulfation balance by chondroitin-4-sulfotransferase 1 is required for chondrocyte development and growth factor signaling during cartilage morphogenesis[J].Development, 2005, 132(17):3989-4003.
[11] YUSA A, KITAJIMA K, HABUCHI O.N-linked oligosaccharides are required to produce and stabilize the active form of chondroitin 4-sulphotransferase-1[J].Biochemical Journal, 2005, 388(Pt 1):115-121.
[12] 李成成, 黄义德.毕赤酵母表达系统及其发酵策略[J].福建轻纺, 2023(10):31-36.LI C C, HUANG Y D.Pichia pastoris expression system and its fermentation strategy[J].The Light &Textile Industries of Fujian, 2023(10):31-36.
[13] LI J, SHAO Q N, XIANG Y L, et al.High-activity recombinant human carboxypeptidase B expression in Pichia pastoris through rational protein engineering and enhancing secretion from the Golgi apparatus to the plasma membrane[J].Biotechnology Journal, 2024, 19(5):2400098.
[14] GLEESON M A, WHITE C E, MEININGER D P, et al.Generation of protease-deficient strains and their use in heterologous protein expression[J].Methods in Molecular Biology, 1998, 103:81-94.
[15] DUAN G D, DING L M, WEI D S, et al.Screening endogenous signal peptides and protein folding factors to promote the secretory expression of heterologous proteins in Pichia pastoris[J].Journal of Biotechnology, 2019, 306:193-202.
[16] LIANG S L, LI C, YE Y R, et al.Endogenous signal peptides efficiently mediate the secretion of recombinant proteins in Pichia pastoris[J].Biotechnology Letters, 2013, 35(1):97-105.
[17] RUOHONEN L, HACKMAN P, LEHTOVAARA P, et al.Efficient secretion of Bacillus amyloliquefaciens α-amylase cells by its own signal peptide from Saccharomyces cerevisiae host[J].Gene, 1987, 59(2-3):161-170.
[18] CHUNG B H, NAM S W, KIM B M, et al.Highly efficient secretion of heterologous proteins from Saccharomyces cerevisiae using inulinase signal peptides[J].Biotechnology and Bioengineering, 1996, 49(4):473-479.
[19] MASSAHI A, ÇAL K P.Endogenous signal peptides in recombinant protein production by Pichia pastoris:From in-silico analysis to fermentation[J].Journal of Theoretical Biology, 2016, 408:22-33.
K P.Endogenous signal peptides in recombinant protein production by Pichia pastoris:From in-silico analysis to fermentation[J].Journal of Theoretical Biology, 2016, 408:22-33.
[20] AW R, MCKAY P F, SHATTOCK R J, et al.Expressing anti-HIV VRC01 antibody using the murine IgG1 secretion signal in Pichia pastoris[J].AMB Express, 2017, 7(1):70.
[21] BARRERO J J, CASLER J C, VALERO F, et al.An improved secretion signal enhances the secretion of model proteins from Pichia pastoris[J].Microbial Cell Factories, 2018, 17(1):161.
[22] GOVERDE C A, PACESA M, GOLDBACH N, et al.Computational design of soluble and functional membrane protein analogues[J].Nature, 2024, 631(8020):449-458.
[23] ESPOSITO D, CHATTERJEE D K.Enhancement of soluble protein expression through the use of fusion tags[J].Current Opinion in Biotechnology, 2006, 17(4):353-358.
[24] LIU L, SPURRIER J, BUTT T R, et al.Enhanced protein expression in the baculovirus/insect cell system using engineered SUMO fusions[J].Protein Expression and Purification, 2008, 62(1):21-28.
[25] KRONQVIST N, SARR M, LINDQVIST A, et al.Efficient protein production inspired by how spiders make silk[J].Nature Communications, 2017, 8:15504.
[26] ABELEIN A, CHEN G F, KITOKA K, et al.High-yield production of amyloid-β peptide enabled by a customized spider silk domain[J].Scientific Reports, 2020, 10(1):235.
[27] ZARZHITSKY S, JIANG A, E STANLEY E, et al.Harnessing synthetic biology to enhance heterologous protein expression[J].Protein Science, 2020, 29(8):1 698-1 706.
[28] INAN M, MEAGHER M M.Non-repressing carbon sources for alcohol oxidase (AOX1) promoter of Pichia pastoris[J].Journal of Bioscience and Bioengineering, 2001, 92(6):585-589.
[29] 潘林, 孙建义.生物素的生理功能及其分子作用机制[J].中国饲料, 2005(6):21-24.PAN L, SUN J Y.Physiological functions and molecular mechanisms of biotin[J].China Feed, 2005(6):21-24.