乳酸菌是革兰氏阳性细菌,能够将碳水化合物发酵成乳酸并产生多种代谢产物[1],在发酵过程中会产生肽、胞外多糖、细菌素、蛋白酶和其他重要的生物活性化合物[2]。随着对乳酸菌代谢产生的物质和降解多种难降解分子的研究越来越多[3],乳酸菌在食品工业,尤其是食品发酵领域中表现出了重要应用价值,例如,以白菜为原料,利用乳酸菌发酵产生乳酸等酸性物质,用于东北酸菜的工业化生产[4]。乳酸菌协同发酵可产生多种生物活性物质,改善发酵食品的感官品质,增强功能性,促进有益菌生长,降低发酵过程中潜在的有害物质的含量,助力新型发酵食品技术开发。这有利于环境保护和降低成本,同时推动农业产业升级。
不同微生物间的相互作用在调控产品品质和保障食品安全方面发挥着至关重要的作用。在发酵过程中,乳酸菌混合培养在抑制有害细菌生长的同时促进目标功能微生物活性的提升至关重要[5]。最近的研究发现,粪肠球菌CIRM-BIA2412、乳酸乳杆菌NCDO2125和乳酸乳杆菌CIRM-BIA244之间的相互作用与色氨酸、缬氨酸、苯丙氨酸、亮氨酸、异亮氨酸和肽的代谢有关[6]。有研究使用超高效液相色谱串联四极杆飞行时间质谱分析了酿酒酵母和植物乳植杆菌发酵的绿茶中的非挥发性成分,在乳酸菌和酵母菌共同培养的茶叶中,发现了2种独特的乳酸代谢产物,即D-苯基乳酸和4-羟基苯乳酸,它们具有生物活性和抗真菌特性,共同发酵对这2种化合物的生产具有协同作用[7]。越来越多的研究表明,发酵过程中产生的代谢物与乳酸菌之间存在密切关系[8-9]。CO2是氨基酸和核苷酸合成的前体,嗜热链球菌通过尿素酶的作用催化尿素分解为CO2,从而促进保加利亚乳杆菌的生长[10-11]。尽管对乳酸菌的协同作用关系进行了广泛研究,但乳酸菌混合培养的作用机制仍不清楚,需要进一步的探索来阐明。
本研究旨在探讨干酪乳酪杆菌HC 1378(Lacticaseibacillus casei,Lc)、植物乳植杆菌HC 1775(Lactiplantibacillus plantarum,Lpl)、鼠李糖乳酪杆菌HC 1509(Lacticaseibacillus rhamnosus GG,LGG)和嗜酸乳杆菌HSCC-LA011(Lactobacillus acidophilus,La)在混合体系中的相互作用关系,通过采用非靶向代谢组学对混合培养协同机理进行分析,为挖掘益生菌的功能提供重要的理论意义和实际应用价值。
1 材料与方法
1.1 材料与试剂
Lc、Lpl、LGG、La,郑州百益宝生物技术有限公司;BCA蛋白浓度测定试剂盒、乳酸脱氢酶(lactate dehydrogenase,LDH)检测试剂盒,北京索莱宝科技有限公司;RNA提取试剂盒、反转录试剂盒、SYBR Green Premix Pro Tap HS qPCR试剂盒,湖南艾科瑞生物工程有限公司。
1.2 仪器与设备
Q Exactive Plus组合型四极杆-静电场轨道阱质谱仪,美国Thermo Fisher Scientific;SMPss500-17805-2BF0酶标仪,上海闪普生物科技有限公司;NeOfuge 1600R高速冷冻离心机,上海力申科学仪器有限公司。
1.3 实验方法
1.3.1 生长曲线
乳酸菌在MRS肉汤中培养,在37 ℃下连续静置培养2次,每次24 h。单培养接种含菌量为3%(体积分数)的细胞悬浮液,共培养在MRS肉汤中将2株菌株按体积比1∶1的比例制备。通过测量波长600 nm处的光密度来监测细菌生长[12]。对照组由不含任何接种物的MRS培养基组成。
1.3.2 主要代谢物含量的测定
按照杨江威等[13]的方法稍作修改。取适量培养物,5 000 r/min离心10 min去除菌体。吸取0.5 mL上清液并适当稀释,取一定量稀释液加入等量钨酸溶液,室温静置,至出现明显絮状物,离心除去,60 ℃保温30 min。取5 mL待测液加入0.05 g Ca(OH)2,0.8 mL 4%(体积分数,下同)CuSO4溶液,混匀,沸水浴3 min,冰水浴冷却,5 000 r/min离心10 min,取上清液加入6 mL浓硫酸溶液,混匀,沸水浴5 min,冰水浴冷却,加入0.125 mL质量分数为0.5%的对羟基联苯溶液,静置10 min,沸水浴显色5 min,于565 nm处测吸光度并计算产生乳酸含量。
按照ABERNATHY等[14]的方法稍作修改。取1 mL培养物,加入等量蒸馏水、3 mL水合茚三酮溶液、0.1 mL抗坏血酸,混匀,沸水浴15 min,冷水冷却,用无水乙醇定容至20 mL,于570 nm处测吸光度并计算产生游离氨基氮含量。
1.3.3 无细胞上清液(cell free supernatant,CFS)代谢产物作用
将存在细菌间协同作用的一种培养物8 000 r/min离心10 min,取上清液制作CFS,将另一培养物同条件离心取菌体并用无菌PBS(pH 7.2)清洗2~3次,将CFS接种于清洗后的菌体中进行培养,同时将单独培养的菌液作为对照,测定细菌活菌数。
1.3.4 LDH活性及表达水平测定
制备CFS[15]。发酵24 h后,5 000 r/min离心2 min收集细菌细胞,得到细菌悬浮液。按照说明,使用LDH检测试剂盒测定LDH活性。以牛血清白蛋白(bovine serum albumin,BSA)为标准溶液,使用BCA蛋白检测试剂盒测定蛋白质浓度。发酵14 h后,制备细菌悬浮液并用100 μL质量浓度为100 mg/mL的溶菌酶溶液处理。使用SteadyPure通用RNA提取试剂盒提取细菌总RNA,并进行凝胶电泳分析。使用反转录试剂盒进行反转录。16S rRNA基因作为管家基因,ldh作为目标基因。使用SYBR Green Premix Pro Tap HS qPCR试剂盒进行qPCR,量化基因表达水平。
1.3.5 非靶向代谢组学
根据ELSAYED等[16]提出的方法稍作修改。移取100 μL样品,加入400 μL提取液[V(甲醇)∶V(乙腈)=1∶1],超声波充分混合,静置1 h,4 ℃、12 000 r/min离心15 min,取上清液上机检测。使用Vabquish超高液相色谱仪,通过Waters ACQUITY UPLC BEH Amide(2.1 mm×50 mm,1.7 μm)液相色谱柱对目标化合物进行色谱分离,流动相:25 mmol/L乙酸铵(A)和25 mmol/L氨水(B)。质谱参数:鞘气流速50 Arb,辅助气流速15 Arb,毛细管温度320 ℃,全质谱分辨率60 000,MS/MS分辨率15 000,碰撞能量SNCE 20/30/40,喷雾电压分别为3.8 kV(正)或-3.4 kV(负)。梯度洗脱:0~0.5 min,1% B;0.5~4 min,1%~99% B;4~4.5 min,99% B;4.5~4.55 min,99% B~1% B;4.55~6 min,1% B,柱温25 ℃,样品盘温度4 ℃,流速0.3 mL/min,进样量2 μL[17-18]。
1.3.6 数据分析
使用软件(ProteoWizard)处理原始色谱数据,进行峰识别、峰提取、峰对齐和积分等处理。进一步分析预处理的代谢物,使用SIMCA(V14.1)软件(https://www.sartorius.com)进行主成分分析(principle component analysis,PCA)与偏最小二乘法-判别分析(partial least squares method-discriminant analysis,PLS-DA),使用人类代谢组数据库HMDB、KEGG-COMPOUND、KEGG-PATHWAY(http://www.HMDB.ca;https://www.kegg.jp/kegg/compound/;https://www.kegg.jp/kegg/pathway.html)鉴定代谢物并分析代谢通路。使用MetaboAnalyst 4.0软件(http://www.MetaboAnalyst.ca)对差异代谢物进行途径富集分析。
2 结果与分析
2.1 乳酸菌单独及混合培养生长特性
为探究乳酸菌混合培养之间的相互作用关系,测定了乳酸菌及其混合培养的生长曲线,见图1。在延滞期,与Lpl+LGG相比,LGG的生长速度更快。在对数生长阶段,Lpl+LGG组表现出更高的生长速度,其生长曲线与LGG非常相似。10 h后,Lpl+LGG的数量明显多于单一培养的LGG和Lpl。同样,Lpl+La组的OD值也高于单一培养的Lpl和La。18 h后,各组都进入了稳定生长阶段,在这一阶段,Lpl+LGG组和Lpl+La组的OD值与各自的单一培养相比继续显著增加,其余各组均未观察到明显的促进生长趋势。
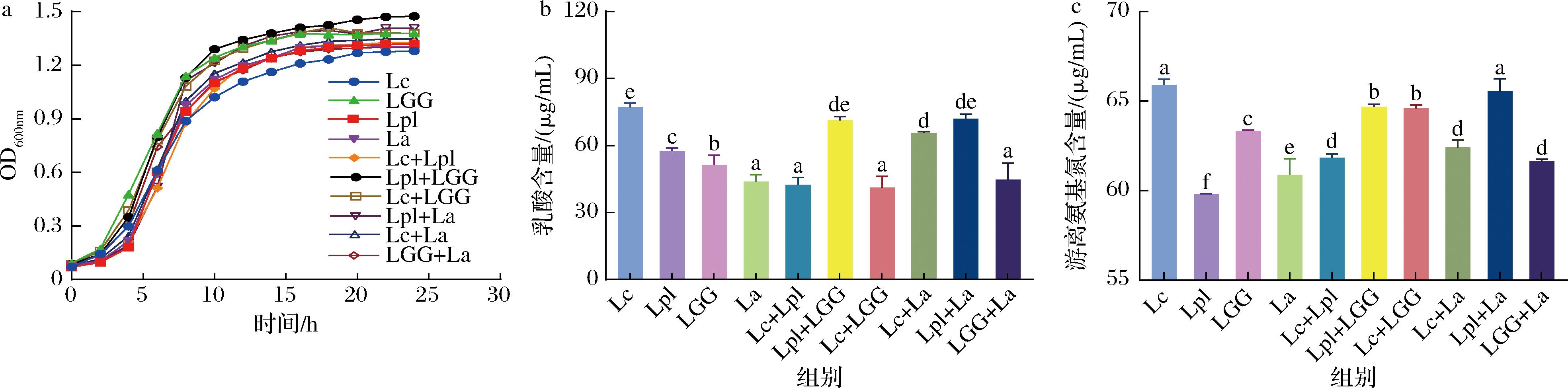
a-生长曲线;b-乳酸含量;c-游离氨基氮含量
图1 乳酸菌的生长曲线及主要代谢物产量
Fig.1 Growth curve and main metabolite production of lactic acid bacteria
注:不同字母表示组间有显著差异(P<0.05),相同字母表示组间没有显著差异(下同)。
通过测定乳酸和游离氨基氮含量探究乳酸菌代谢和发酵能力,Lpl+LGG的乳酸含量为71.20 μg/mL,与Lpl和LGG相比,分别显著增加了23.52%和38.68%(P<0.05)。同样,Lpl+LGG的游离氨基氮含量为64.68 μg/mL,与Lpl和LGG相比分别显著增加了8.14%和2.12%(P<0.05)。相反,与La混合培养时,Lpl+La的乳酸含量为72.07 μg/mL,游离氨基氮含量为65.56 μg/mL,均显著高于Lpl或La单独培养时的含量(P<0.05)。与单一菌株相比,其他共培养组的乳酸和游离氨基氮含量没有明显变化(P>0.05),可见Lpl和LGG以及Lpl和La之间存在潜在的协同作用。
2.2 CFS对协同效应的影响
通过添加CFS,研究代谢物对混合培养的影响。与图2所示,与单独培养相比,添加Lpl CFS可使LGG和La的存活细菌数分别显著增加32.83%和15.38%(P<0.05)。添加LGG CFS和La CFS后,Lpl含量也分别显著增加了23.70%和13.32%(P<0.05)。Lpl与LGG以及Lpl与La之间存在协同共生关系。添加CFS能显著增强协同效应,这表明协同作用与代谢物之间存在密切联系,代谢物对Lpl+LGG的协同作用有更明显的影响,这与CFS中差异代谢物有关,需进一步深入研究。
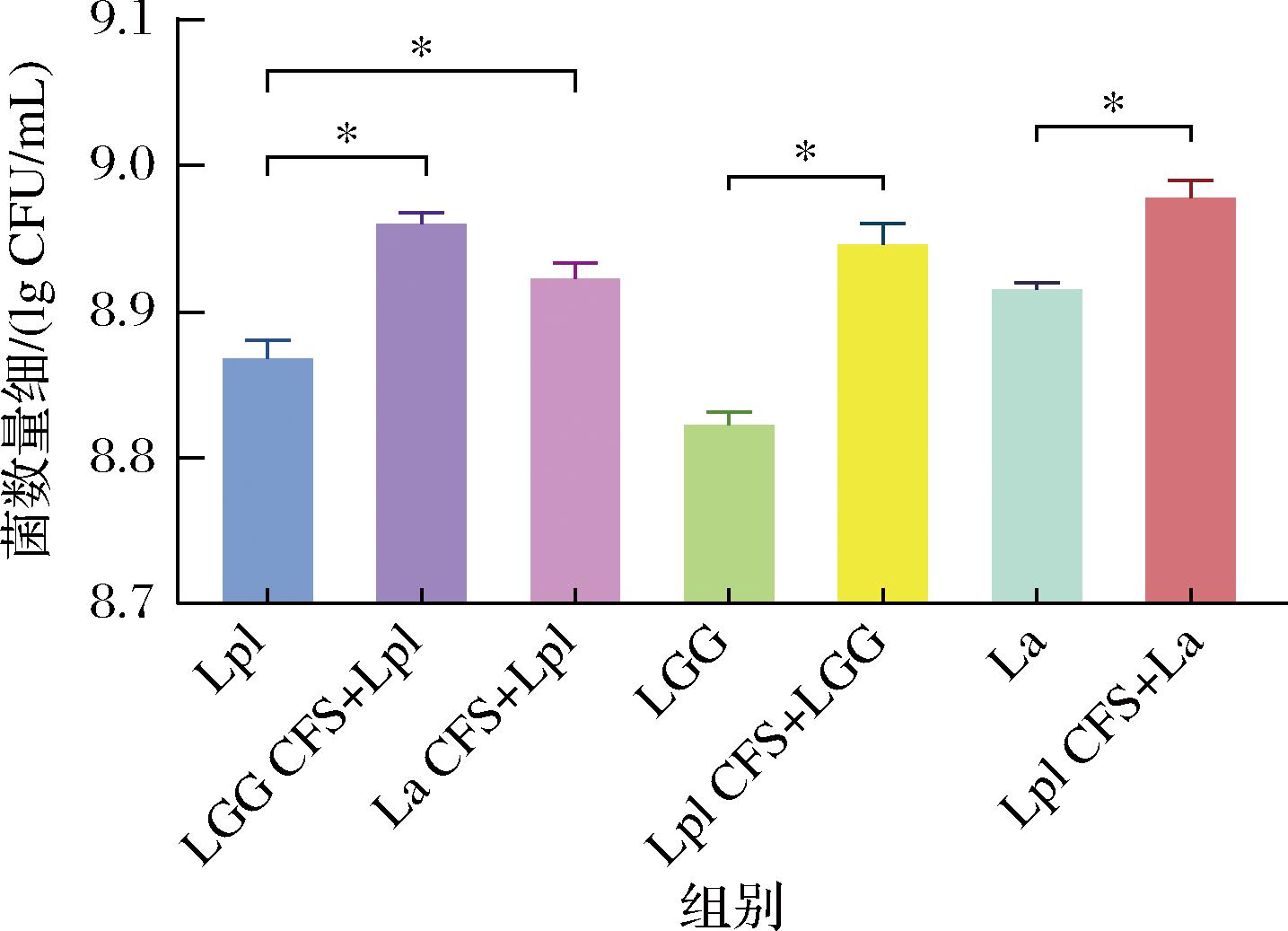
图2 添加CFS后的细菌数
Fig.2 Number of bacteria after adding cell-free supernatant
注:*表示组间有显著差异(P<0.05)(下同)。
2.3 LDH基因表达和酶活性的变化
为了研究LDH对乳酸菌协同作用的影响,评估了LDH活性及其基因的表达水平,如图3所示。细菌生长在发酵14 h后进入稳定期,与Lpl和LGG细菌相比,Lpl+LGG细菌的活力为78.46 U/mg prot(P<0.05),较Lpl、LGG分别显著提高了242.69%和113.33%。与Lpl和La细菌相比,Lpl+La细菌的活力为67.00 U/mg prot (P<0.05),较Lpl、La分别显著增加了192.61%和112.28%。以16S rRNA作为管家基因对ldh基因的表达进行了定量分析。与LGG组相比,Lpl+LGG组的ldh基因表达量显著升高了1.15倍,而Lpl+La组的ldh基因表达量相对于La组仅小幅升高了33%(图3)。Lpl+LGG组与其他组之间存在明显差异,表明ldh转录活性大幅上调。这一结果表明,通过ldh基因来调控细菌代谢可提高混合细菌培养物的代谢活性和酶效率。
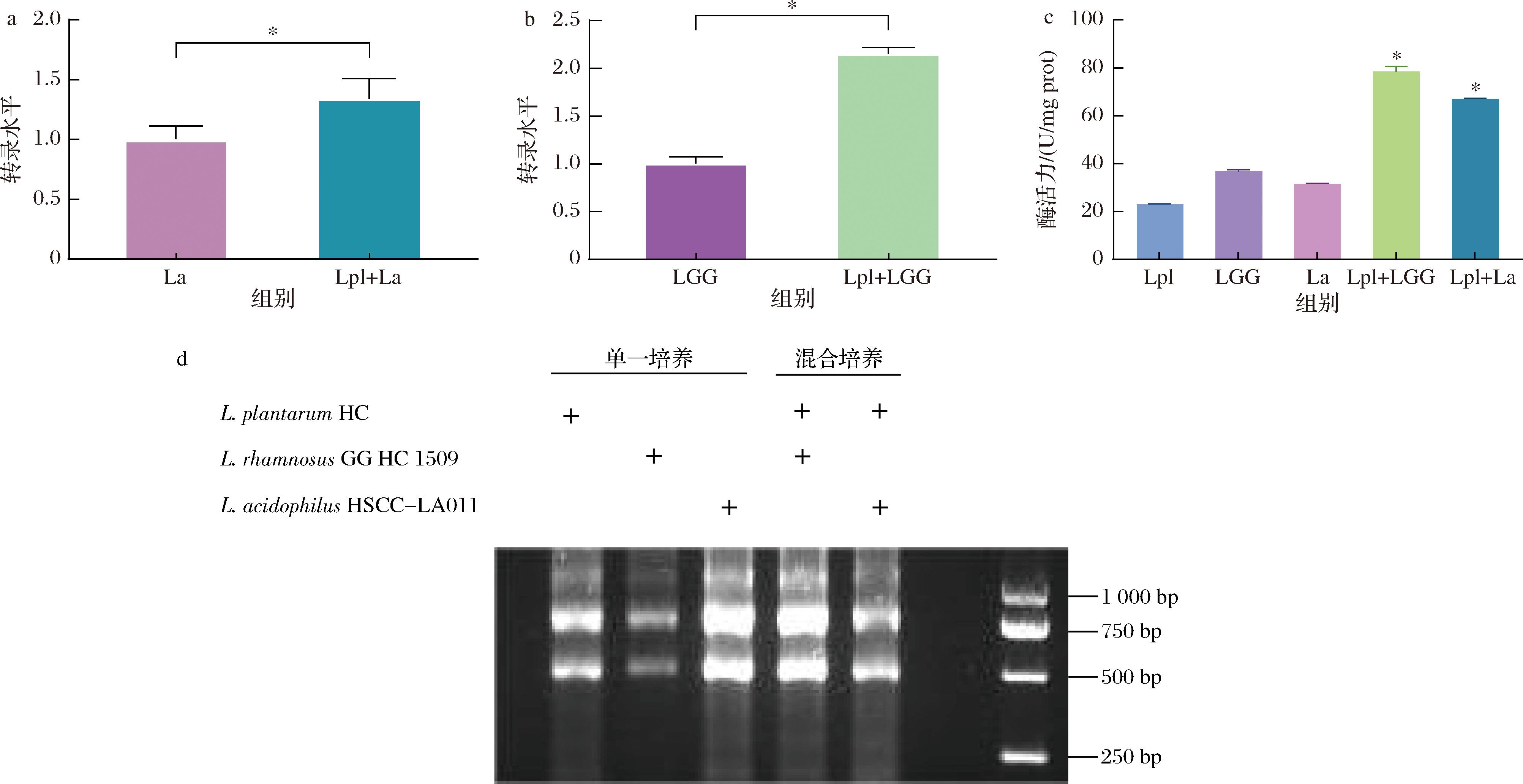
a-La与Lpl组ldh转录水平;b-LGG与Lpl组ldh转录水平;c-LDH活性;d-ldh电泳图
图3 乳酸菌LDH的活性及转录水平
Fig.3 Activity and transcriptional level of lactic dehydrogenase in lactic acid bacteria
LDH在乳酸菌代谢氨基酸等差异代谢产物的途径中发挥着至关重要的作用[19]。LDH可催化丙酮酸转化为乳酸,乳酸菌混合培养能通过上调ldh基因表达来增强其代谢能力,提高其乳酸生成的效率。有研究表明,共培养条件会影响乳酸菌的LuxS/AI-2群体感应系统,进而影响其生理功能。在Lpl和LGG混合培养中,LuxS/AI-2系统可能通过调节转录因子的活性,促进ldh基因的表达[20]。Lpl和LGG共同培养明显上调ldh基因表达,与转录因子、信号通路及代谢物的调节有关,仍需进一步研究。
2.4 非靶向代谢组学分析
2.4.1 质量控制(quality control,QC)分析和代谢物可视化
根据之前的分析,Lpl和LGG间的协同作用与代谢物之间存在很强的相关性,其中Lpl和LGG之间的协同作用影响尤为明显。采用了非靶向代谢组学研究Lpl和LGG单独培养和混合培养的代谢变化。如图4所示,对所有样品进行PCA,质控样品位于中间且分布密集,表明试验具有稳定性和可重复性。Lpl、LGG和Lpl+LGG样品之间有明显的分离趋势,它们之间的代谢结构和组成存在显著差异。Lpl+LGG样品的分布介于Lpl和LGG之间,但更接近后者。这表明在所有样本中LGG在代谢过程中起着主导作用。


a-所有样本的QC分析;b-所有代谢物热图;c-Lpl vs Lpl+LGG vs LGG 差异代谢物PCA图;d-Lpl vs Lpl+LGG vs LGG差异代谢物OPLS-DA图;e-Lpl vs Lpl+LGG 差异代谢物饼图;f-LGG vs Lpl+LGG 差异代谢物饼图;g-Lpl vs Lpl+LGG vs LGG 组相比差异代谢物热图;h-前100个差异代谢物的热图
图4 所有样本QC分析及所有代谢物结果
Fig.4 QC analysis of all samples and results of all metabolites
利用基于LC-MS的代谢组学鉴定了Lpl和LGG结合产生的代谢物及其代谢作用。所有代谢物热图都显示出不同培养物之间代谢组成和结构的显著差异。利用多元分析OPLS-DA模型[21]确定了项目中变量的重要性(variable importance in project,VIP),以识别不同品种或组织之间存在差异的代谢物。此外,根据单变量分析得出的P值对差异代谢物进行了进一步筛选[22]。PCA图显示了单菌培养和共培养之间的显著差异。OPLS-DA图显示模型R2X和R2Y值均接近1,表明准确反映了真实数据特征。Q2值接近于1,这意味着样本差异得到了有效解释,且没有过度拟合。
采用非靶向代谢组学技术研究了Lpl组与Lpl+LGG组和LGG组与Lpl+LGG组之间的差异代谢谱。饼图分别显示了共200和329个注释的差异代谢物(图4-d~图4-e)。这些代谢物主要属于其他(28.3%、27.3%)、氨基酸(25.3%、24.9%)、有机化合物(14.6%、17.9%)、脂质(7.6%、7.5%)、有机酸(4.3%、3.8%)、核苷(4.0%、2.7%)、糖类(3.6%、1.8%)、黄酮类(3.6%、3.0%)、萜类(2.6%、0.6%)、衍生物(1.3%、1.5%)、肽类(1.0%、1.2%)、二苯乙烯类(1%、0.3%)、碳水化合物(0.3%、0.6%)等。经过多维统计分析,VIP>1且单变量统计分析P<0.05的代谢物被确定为具有显著差异[23]。图4的代谢物图谱显示,Lpl和LGG之间以及Lpl+LGG和前100个物种之间存在显著差异。这些化合物包括氨基酸、有机酸、脂肪酸和碳水化合物等多种物质。研究发现,单一培养和混合培养的代谢物特征存在显著差异。
2.4.2 代谢物变化
聚类过程包括使用静态或动态方法将相似对象划分为若干子集,每个子集称为一个聚类[24]。图5中K值聚类分析时发现,同一聚类内的代谢物表达趋势高度相似,而不同聚类和样本之间的差异代谢物表达趋势相似度相对较低。通过比较Lpl和Lpl+LGG共发现了300个差异代谢物,包括164个上调代谢物和136个下调代谢物。从LGG和Lpl+LGG的比较中得到了329个差异代谢物,包括46个上调代谢物和283个下调代谢物。图5结果表明,大部分代谢物表现出明显的下调,表明细菌利用这些代谢物进行生长;同时,一些特定代谢物的表达水平升高,表明细菌在生长过程中产生了新的化合物。3个培养组都存在其他组未表达的代谢物:Lpl组仅有13种,LGG组有14种,Lpl+LGG组有2种。
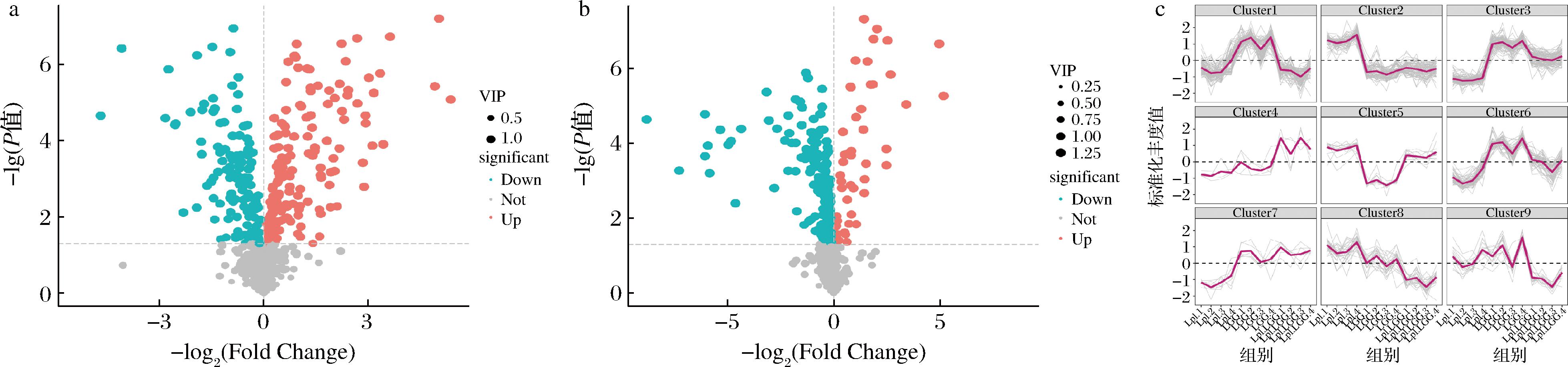
a-Lpl vs Lpl+LGG代谢物火山图;b-LGG vs Lpl+LGG代谢物火山图;c-k值聚类分析图
图5 代谢物变化情况
Fig.5 Metabolite changes
2.4.3 KEGG富集分析
利用KEGG深入了解生物和生态功能的分子方面,有助于全面了解其利用情况,不同代谢物之间的协调对于执行一系列生化反应以实现各自的生物功能至关重要[25]。采用皮尔逊相关分析法确定这些显著不同代谢物之间的相关系数,旨在研究它们之间的关系[14]。图6表明有机酸和氨基酸代谢物之间呈正相关,而类黄酮和糖代谢物之间呈负相关。利用KEGG数据库对确定的差异代谢物进行了代谢途径富集分析,显示共存在204条途径。采用P<0.05的显著性水平,通过筛选确定了前20条主要通路。表明在乳酸菌混合发酵后,代谢和生物系统(包括氨基酸代谢、碳水化合物代谢和其他生物合成途径)显著富集了不同含量的代谢物。

a-Lpl vs Lpl+LGG前20个富集途径分析图;b-LGG vs Lpl+LGG前20个富集途径分析图;c-代谢途径类型分析图;d-关联分析热图
图6 KEGG分析
Fig.6 KEGG analysis
将Lpl和LGG单独培养的代谢途径与二者混合培养的代谢途径进行了比较。在富集途径分析中,Lpl与Lpl+LGG中富集的前5条途径分别是次生代谢物的生物合成、植物次生代谢物的生物合成、核苷酸代谢、嘧啶代谢和氨基酸的生物合成。LGG与Lpl+LGG的前5条富集途径分别是代谢途径、核苷酸代谢、嘧啶代谢、ABC转运体以及半胱氨酸和蛋氨酸代谢。这些发现进一步说明了Lpl和LGG单独与混合培养之间代谢特征的显著差异。次生代谢物是通过次生代谢途径产生的特殊化合物,这些化合物主要包括天冬氨酸、丝氨酸、苹果酸和酪氨酸,它们的丰度都有显著增加,且在Lpl、LGG发酵过程中代谢产生,存在于CFS中,能够被乳酸菌生长代谢利用,与2.2节研究结果一致。已有研究表明,天门冬氨酸等代谢产物可以增强免疫功能、缓解焦虑和抑郁症状、恢复人体能量水平[26-28],在协同培养过程中,天冬氨酸、丝氨酸和酪氨酸是细胞合成蛋白质和其他生物大分子的重要原料,能够参与乳酸菌之间的氮代谢共享,其在CFS中的含量发生变化,为乳酸菌提供更丰富的营养物质,促进乳酸菌生长繁殖[29]。此外,苹果酸作为三羧酸循环的重要中间代谢物,参与细胞的能量代谢和物质代谢,与能量代谢和碳代谢的耦合有关,其变化能够影响乳酸菌的能量代谢状态,促进乳酸菌的生长[30]。
3 结论
植物乳杆菌HC 1775(Lactiplantibacillus plantarumHC 1775,Lpl)和鼠李糖乳杆菌GG HC 1509(Lacticaseibacillus rhamnosus GG HC 1509,LGG)之间存在协同作用关系,与单一培养条件相比,Lpl和LGG混合培养的菌数、乳酸和氨基氮代谢含量显著增加。此外,混合培养显著上调了ldh基因表达,增强了酶活性,影响了氨基酸和有机酸在CFS中的含量,显著提高了代谢产物丰度,导致代谢谱的显著差异。Lpl和LGG之间通过显著差异代谢物氨基酸类物质参与相关通路途径促进乳酸菌的生长,这是乳酸菌混合培养应对环境变化和优化代谢的一种途径。本研究虽揭示了Lpl和LGG短期混合培养的影响,但短期共培养实验条件相对简单,缺乏长期发酵过程中复杂的代谢物动态变化和菌群演替,无法模拟实际发酵过程中可能出现的营养限制、代谢产物积累以及环境胁迫等因素,未来的研究将结合宏基因组学技术,优化共培养比例,并对长期发酵过程进行动态监测,以更全面地理解菌群的协同作用及其在工业发酵中的应用潜力。
[1] GEORGE F, DANIEL C, THOMAS M, et al.Occurrence and dynamism of lactic acid bacteria in distinct ecological niches:A multifaceted functional health perspective[J].Frontiers in Microbiology, 2018, 9:2899.
[2] ABDUL HAKIM B N, XUAN N J, OSLAN S N H.A comprehensive review of bioactive compounds from lactic acid bacteria:Potential functions as functional food in dietetics and the food industry[J].Foods, 2023, 12(15):2850.
[3] RUIZ RODR GUEZ L G, MOHAMED F, BLECKWEDEL J, et al.Diversity and functional properties of lactic acid bacteria isolated from wild fruits and flowers present in northern Argentina[J].Frontiers in Microbiology, 2019, 10:1091.
GUEZ L G, MOHAMED F, BLECKWEDEL J, et al.Diversity and functional properties of lactic acid bacteria isolated from wild fruits and flowers present in northern Argentina[J].Frontiers in Microbiology, 2019, 10:1091.
[4] LIU S N, HAN Y, ZHOU Z J.Lactic acid bacteria in traditional fermented Chinese foods[J].Food Research International, 2011, 44(3):643-651.
[5] ZHANG K, ZHANG T T, GUO R R, et al.The regulation of key flavor of traditional fermented food by microbial metabolism:A review[J].Food Chemistry:X, 2023, 19:100871.
[6] WANG Y Q, WU J T, LYU M X, et al.Metabolism characteristics of lactic acid bacteria and the expanding applications in food industry[J].Frontiers in Bioengineering and Biotechnology, 2021, 9:612285.
[7] WANG R, SUN J C, LASSABLIERE B, et al.UPLC-Q-TOF-MS based metabolomics and chemometric analyses for green tea fermented with Saccharomyces boulardii CNCM I-745 and Lactiplantibacillus plantarum 299V[J].Current Research in Food Science, 2022, 5:471-478.
[8] HAN D, ZULEWSKA J, XIONG K, et al.Synergy between oligosaccharides and probiotics:From metabolic properties to beneficial effects[J].Critical Reviews in Food Science and Nutrition, 2024, 64(13):4078-4100.
[9] GADAGA T H, MUTUKUMIRA A N, NARVHUS J A.The growth and interaction of yeasts and lactic acid bacteria isolated from Zimbabwean naturally fermented milk in UHT milk[J].International Journal of Food Microbiology, 2001, 68(1-2):21-32.
[10] DE SOUZA OLIVEIRA R P, TORRES B R, PEREGO P, et al.Co-metabolic models of Streptococcus thermophilus in co-culture with Lactobacillus bulgaricus or Lactobacillus acidophilus[J].Biochemical Engineering Journal, 2012, 62:62-69.
[11] LIU E N, ZHENG H J, SHI T, et al.Relationship between Lactobacillus bulgaricus and Streptococcus thermophilus under whey conditions:Focus on amino acid formation[J].International Dairy Journal, 2016, 56:141-150.
[12] ROGERS A T, BULLARD K R, DOD A C, et al.Bacterial growth curve measurements with a multimode microplate reader[J].Bio-protocol, 2022, 12(9):e4410.
[13] 杨江威, 王越, 安家彦.对羟基联苯比色法测定葡萄酒发酵过程中的乳酸[J].大连工业大学学报, 2012, 31(6):402-404.YANG J W, WANG Y, AN J Y.Detection of lactic acid by p-hydroxybiphenyl colorimetry during wine fermentation[J].Journal of Dalian Polytechnic University, 2012, 31(6):402-404.
[14] ABERNATHY D G, SPEDDING G, STARCHER B.Analysis of protein and total usable nitrogen in beer and wine using a microwell ninhydrin assay[J].Journal of the Institute of Brewing, 2009, 115(2):122-127.
[15] E J J, ZHANG J Y, MA R Z, et al.Study of the internal mechanism of L-glutamate for improving the survival rate of Lactiplantibacillus plantarum LIP-1 after freeze-drying[J].Innovative Food Science &Emerging Technologies, 2023, 84:103253.
[16] ELSAYED Y, REFAAT J, ABDELMOHSEN U R, et al.Metabolomic profiling and biological investigation of the marine sponge-derived bacterium Rhodococcus sp.UA13[J].Phytochemical Analysis, 2018, 29(6):543-548.
[17] CHEN S, LIU H H, ZHAO X M, et al.Non-targeted metabolomics analysis reveals dynamic changes of volatile and non-volatile metabolites during oolong tea manufacture[J].Food Research International, 2020, 128:108778.
[18] WU S M, YU Q Y, SHEN S, et al.Non-targeted metabolomics and electronic tongue analysis reveal the effect of rolling time on the sensory quality and nonvolatile metabolites of congou black tea[J].LWT, 2022, 169:113971.
[19] LI X, CHEN Y G, ZHAO S, et al.Efficient production of optically pure L-lactic acid from food waste at ambient temperature by regulating key enzyme activity[J].Water Research, 2015, 70:148-157.
[20] 顾悦. 环境胁迫及酵母菌对乳酸菌LuxS/AI-2群体感应系统的影响[D].呼和浩特:内蒙古农业大学, 2017.GU Y.The effects of environmental stresses and yeast on LuxS/AI-2Quorum sensing system of lactic acid bacteria[D].Hohhot:Inner Mongolia Agricultural University, 2017.
[21] KANG C D, ZHANG Y Y, ZHANG M Y, et al.Screening of specific quantitative peptides of beef by LC-MS/MS coupled with OPLS-DA[J].Food Chemistry, 2022, 387:132932.
[22] SACCENTI E, HOEFSLOOT H C J, SMILDE A K, et al.Reflections on univariate and multivariate analysis of metabolomics data[J].Metabolomics, 2014, 10(3):361-374.
[23] YUAN Y X, WANG G, ZOU J H, et al.Study on comparative analysis of differential metabolites in Guanzhong dairy goat Semen before and after freezing[J].Theriogenology, 2023, 197:232-239.
[24] BECKONERT O, BOLLARD M E, EBBELS T M D, et al.NMR-based metabonomic toxicity classification:Hierarchical cluster analysis and k-nearest-neighbour approaches[J].Analytica Chimica Acta, 2003, 490(1-2):3-15.
[25] CHEN L, ZHANG Y H, ZOU Q, et al.Analysis of the chemical toxicity effects using the enrichment of Gene Ontology terms and KEGG pathways[J].Biochimica et Biophysica Acta (BBA) - General Subjects, 2016, 1860(11):2619-2626.
[26] RAVI P V, MAHARAJAN A, PATTABIRAMAN A, et al.Straightforward paper sensors for the detection of SSRI drugs using tyrosine functionalized GQDs:Fluorescence ‘turn-off’ turns on the crucial dosage monitoring[J].Diamond and Related Materials, 2023, 139:110407.
[27] ZHANG H, CHEN Y P, LI Y, et al.L-Threonine improves intestinal mucin synthesis and immune function of intrauterine growth-retarded weanling piglets[J].Nutrition, 2019, 59:182-187.
[28] ADEVA M M, SOUTO G, BLANCO N, et al.Ammonium metabolism in humans[J].Metabolism, 2012, 61(11):1495-1511.
[29] ZHAO L, ZHANG X T, LIU L J, et al.Effects of amino acids on growth and bacteriocin synthesis of Lactobacillus plantarum KLDS1.0391[J].Food Science, 2021, 42(18).
[30] 盖昱梓, 孙静娴, 黄刚, 等.苹果酸-乳酸发酵细菌乙醇胁迫应答机制研究进展[J].食品与发酵工业, 2021, 47(3):288-293.GAI Y Z, SUN J X, HUANG G, et al.The response mechanism of bacteria to ethanol stress in malolactic fermentation[J].Food and Fermentation Industries, 2021, 47(3):288-293.