人体每天产生的内源性嘌呤以及摄入的外源性嘌呤,在黄嘌呤氧化酶的氧化下会不断生成尿酸[1]。由于人类尿酸酶在进化早期逐步失活,人体缺乏代谢尿酸的酶,导致尿酸在体内不断累积,形成高尿酸血症(hyperuricemia,HUA)[2]。随着尿酸浓度不断升高,析出结晶,附着在关节上,便会引发痛风[3]。而目前常见的治疗高尿酸血症的方式是药物治疗,临床用药一般是别嘌呤醇和苯溴马隆,这些药物对肾脏的损伤很大,引发皮疹甚至会导致生命危险[4]。辅助治疗高尿酸血症的方法一般是控制饮食,减少海鲜、啤酒、动物内脏等的摄入,但可能会造成营养缺失且大多数患者无法长期控制[5]。因此,寻找新的安全的降尿酸方法成为目前研究的热点。
益生菌是指那些当摄入剂量达到有效水平时,能够给宿主健康带来益处的活性微生物。这些微生物主要通过改善肠道微生物平衡,从而对宿主产生积极的健康影响。乳脂乳球菌D2022可以通过增加肠道和全身代谢中短链脂肪酸的产生来减轻高尿酸血症,通过潜在的肠-肾-轴缓解高尿酸血症和抑制肾脏炎症[6]。凝结魏茨曼氏菌发酵的虾肉蛋白质饮料具有较高的黄嘌呤氧化酶抑制能力,从而具有降尿酸功效[7]。乳杆菌对尿酸的降解作用与细胞膜有关,细胞膜中的物质对尿酸起到吸附作用[8]。目前,多数研究表明乳酸菌通过降解嘌呤、核苷等物质抑制尿酸生成从而控制尿酸水平[9-11],而对直接降解尿酸机制的研究较少,且目前研究菌株多为乳酸菌。本研究菌株选用凝结魏茨曼氏菌,不仅具备调节肠道微生态平衡的益生作用,还有易培养、储存稳定性好的特性,值得进一步开发与应用。
益生菌要在体内定植,通常需具有良好的耐酸、耐胆盐能力。与常用的乳酸菌益生菌相比,凝结魏茨曼氏菌能够形成芽孢,这一特性赋予了该菌种对抗胃酸和胆盐的高耐受性。本研究拟筛选具有良好益生特性且具有直接降解尿酸能力的菌株。通过全基因组分析,初步解析其尿酸代谢机理,确定菌株尿酸代谢完整途径,为益生菌控制体内尿酸水平提供一种新方式。
1 材料与方法
1.1 材料与仪器
试验菌株:酸菜样品、健康人体肠道粪便、鸡源性粪便中分离纯化的132株菌株,包括嗜热链球菌、植物乳植杆菌、短左旋乳杆菌、棒状乳杆菌、干酪乳酪杆菌、副干酪乳酪杆菌、粪肠球菌、凝结魏茨曼氏菌和大肠杆菌等;标准菌株肺炎克雷伯菌(T35)、大肠杆菌(T21)、鼠伤寒沙门氏菌(T26)、鲍氏志贺氏菌(T28)、金黄色葡萄球菌(T22)、单核细胞增生李斯特菌(T42),均保存于内蒙古农业大学食品微生物资源开发与应用团队实验室。
尿酸,上海麦克林生化科技股份有限公司;尿酸标准品,成都德思特生物技术有限公司;猪胆盐,北京奥博星生物技术有限公司。
高速冷冻离心机,德国Sigma有限公司;1260InfinityⅡ型高效液相色谱仪,美国安捷伦公司;BioTek Epoch全波长酶标仪,美国BioTek公司;超声清洗机,宁波新芝生物科技股份有限公司。
1.2 降尿酸菌株的筛选及鉴定
1.2.1 尿酸平板初筛
挑取132株菌的单菌落于发酵培养基中进行增殖培养,调节OD600值为1.2,备用。利用牛津杯制备尿酸平板,平板下层倒入水琼脂,上层倒入含有尿酸的培养基。各取菌液200 μL接在牛津杯孔,静置于37 ℃培养箱培养,观察降解尿酸情况。
1.2.2 高效液相色谱法复筛
1.2.2.1 色谱条件
色谱柱:ZORBAX SB-C18柱(4.6 mm×25 mm,5 μm);流动相甲醇∶磷酸钠=10∶90(磷酸钠浓度为10 mmol/L,pH 4.7);流速0.5 mL/min;柱温25 ℃;波长243 nm;进样量20 μL;进样时间20 min[12]。
1.2.2.2 尿酸标准曲线的测定[13]
称取一定质量的尿酸标准品,配制浓度为0.07、0.11、0.15、0.22、0.30、0.59、1.19 mmol/L尿酸-中性磷酸钾溶液,利用HPLC进行色谱分析,记录标准溶液对应的色谱峰面积。以标准溶液的浓度为横坐标(X轴),色谱峰面积为纵坐标(Y轴),绘制标准曲线。
1.2.2.3 尿酸降解率的测定[14]
菌株通过液体活化至第3代,按照体积分数4%的接种量重新接种于新鲜MRS液体培养基中,37 ℃培养备用。取3 mL培养液于4 ℃、8 000 r/min离心6 min,菌体使用PBS重复洗涤3次,重悬于尿酸-中性磷酸钾溶液,静置培养4 d。所得菌悬液4 ℃、8 000 r/min离心6 min,取上清液与终止剂高氯酸按体积比9∶1混合均匀后,使用孔径0.22 μm的无菌过滤器进行过滤,滤后用于HPLC分析。根据HPLC色谱图与标准曲线,计算菌株的尿酸降解率。所有试验均设置3组平行试验。
1.2.3 降尿酸菌株的鉴定
采用高压灭菌后的牙签挑取单个菌落,作为模板,细菌通用引物27F-1492R扩增16S rDNA基因全长序列,由上海生工生物有限公司对序列进行测定。获得的16S rDNA序列与NCBI网站上已知序列进行比对,运用MEGA11.0软件构建系统发育树。
1.3 降尿酸菌株的特性研究
1.3.1 低pH耐受性测定
将活化菌株按10%的接种量接到pH值分别为2.0、3.0、4.0、5.0、6.5的MRS液体管中,连续培养24 h,每隔2 h测OD600值[15]。
1.3.2 胆盐耐受性测定
将活化菌株按10%的接种量接到胆盐质量分数为0.1%、0.2%、0.3%的MRS液体管中,连续培养24 h,每隔2 h测OD600值[15]。
1.3.3 抑菌性
实验于BSL-2实验室内完成,严格遵循生物安全操作规范:采用琼脂扩散法,参考呼静等[16]的方法测定抑菌性。使用双层平板,水琼脂培养基倒底层,无菌牛津杯置于凝固的水琼脂上;活化至对数期的指示菌用无菌生理盐水稀释到107 CFU/mL,取1 mL稀释后的菌悬液加入到50 mL、40 ℃左右的LB琼脂培养基中迅速混合均匀,向含有牛津杯培养皿中倒入30 mL混有指示菌的培养基,待凝固后拔出牛津杯[17]。将活化的凝结魏茨曼氏菌接种于MRS肉汤培养基增殖培养24 h,离心(4 ℃,8 000 r/min,6 min)后取上清液,0.22 μm水系滤膜过滤除菌,得到发酵上清液,蒸发浓缩到1/10。取200 μL发酵上清液进行抑菌试验,测定抑菌圈直径。实验废弃物经121 ℃高压灭菌30 min后,按医疗废物分类处置。研究全程符合《病原微生物实验室生物安全管理条例》。
1.4 全基因组测定
委托武汉希望组生物科技有限公司对离心收集到的菌泥进行测序。利用Qiagen试剂盒提取高质量的DNA,构建1D文库,利用Oxford Nanopore Technology测序仪PromethION对DNA进行单分子测序,获得原始测序数据。
1.5 生物信息学分析
下机后对原始数据进行质控,剔除低质量的数据。质控后的二代数据和三代数据用unicycler进行混合组装后,用pilon或者nextpolish结合二代测序数据进行矫正。矫正后的基因组使用自有脚本检测是否成环,切除冗余部分后,对已成环的序列用circlator将序列的原点移动到基因组的复制起始位点,从而得到最终的基因组序列。用prodial对编码基因进行预测,保留完整的CDS;提取基因组编码蛋白后,用Interproscan进行注释,提取TIGRFAMs、Pfam和GO数据库的注释信息;用blastp比对编码蛋白到KEGG、refseq和COG数据库,对菌株基因功能进行预测分析。
1.6 尿酸代谢相关基因分析
利用COG和KEGG数据库对与降尿酸潜力相关的基因进行注释。通过整合KEGG途径和基因信息,预测凝结魏茨曼氏菌B8的尿酸代谢途径。
1.7 菌株上清液、内容物对尿酸降解及尿酸酶活性测定
制备无细胞上清液:将活化的凝结魏茨曼氏菌B8于4 ℃、8 000 r/min离心6 min,收集上清液[18],通过0.22 μm滤膜得到不含菌体的液体即为无细胞上清液;制备细胞内容物:离心获得的菌体沉淀用PBS清洗2次,再次重悬于1 mL的PBS中,冰浴超声波破碎[15](超声波功率300 W,超声波时间10 min),超声波后的菌液4 ℃、8 000 r/min离心6 min,收集上清液经过0.22 μm的水系滤膜过滤后得到无细胞提取物[19]。分别吸取200 μL无细胞上清液和细胞内容物,涂布于尿酸筛选平板上,37 ℃静置培养4 d,观察其尿酸降解情况。
对细胞内容物进行尿酸酶活性测定:将样本稀释5倍,取30 μL用于检测,按照尿酸酶活性检测试剂盒说明书进行操作,于505 nm处测定吸光值,尿酸酶活性按公式(1)计算:
尿酸酶活性![]() 稀释倍数
稀释倍数
(1)
1.8 数据处理与分析
每组试验均重复3次,试验结果以“平均值±标准差”表示,数据采用Graphpad Prism 9软件进行方差分析和图形绘制。
2 结果与分析
2.1 降尿酸菌株的筛选
通过观察尿酸平板上透明圈的形成,初步筛选具有降尿酸降解潜力的微生物菌株[20]。由图1-a可知,在132株菌中仅有9株菌产生尿酸降解圈,具有降尿酸效果。利用高效液相色谱法进行进一步复筛,根据标准曲线得到线性方程为Y=15 347x+519.4(R2=0.999 9),分别计算得出菌株的降解率(图1-b)。结果表明有5株菌的尿酸降解率在20%以上。
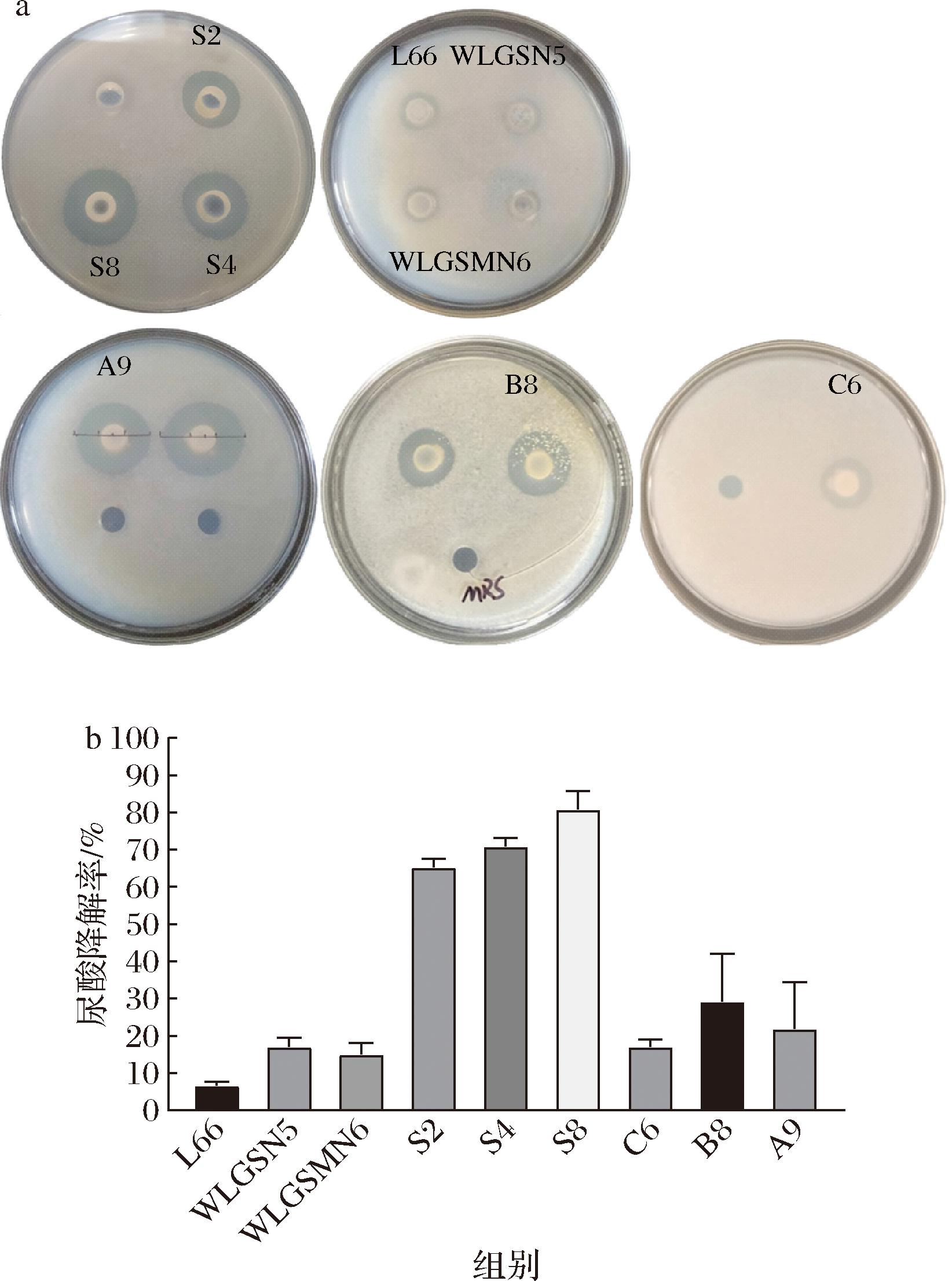
a-平板初筛;b-液相复筛
图1 尿酸降解菌株的筛选
Fig.1 Screening of uric acid-degrading strains
2.2 菌株的鉴定
以细菌单个菌落为模板,利用16S rDNA进行PCR扩增和测序。将各菌株16S rDNA测序结果在NCBI上进行BLAST比对,结果如表1所示。
表1 微生物鉴定结果
Table 1 The results of microbial identification
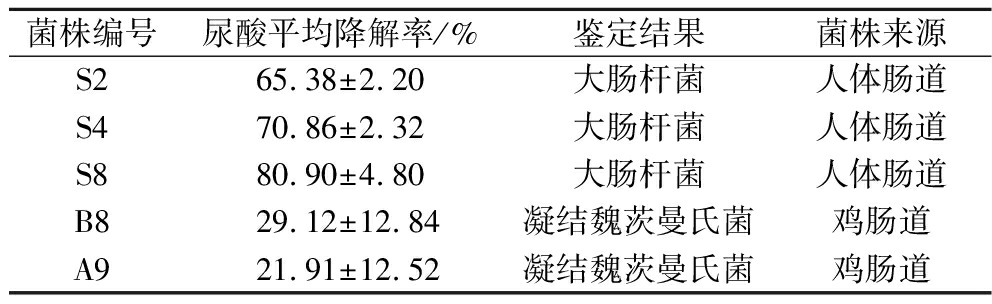
菌株编号尿酸平均降解率/%鉴定结果菌株来源S265.38±2.20大肠杆菌人体肠道S470.86±2.32大肠杆菌人体肠道S880.90±4.80大肠杆菌人体肠道B829.12±12.84凝结魏茨曼氏菌鸡肠道A921.91±12.52凝结魏茨曼氏菌鸡肠道
由于益生菌具有改善肠道健康、增强免疫系统、可以合成和提高营养物质的吸收利用率等功能[21],而凝结魏茨曼氏菌普遍认为是改善肠道健康的理想益生菌,且位于《可用于食品的菌种名单》。因此,本研究选用凝结魏茨曼氏菌B8、A9做后续研究。
2.3 菌株的特性研究
2.3.1 低pH耐受性
位于胃近端2/3(胃体)部位的壁细胞会分泌胃酸,导致胃腔内pH值下降,因此,益生菌预顺利通过胃部定植肠道就要具有良好的耐受低pH能力[22-23],低pH耐受性是评价益生菌的重要指标。如图2所示,在pH 5.0~6.5的条件下凝结魏茨曼氏菌B8、A9生长状况最好,在pH 2.0的条件下B8、A9仍具有活性(P<0.05)。过酸环境确实会抑制菌株生长,但相对而言B8、A9具有良好的耐酸能力,且B8的低pH耐受性强于A9。

a-B8低pH耐受性;b-A9低pH耐受性
图2 耐酸能力测定
Fig.2 Acid resistance evaluation test
注:*表示菌株在24 h不同pH值下差异显著(P<0.05);**表示P<0.01;***表示P<0.001;****表示P<0.000 1(下同)。
2.3.2 胆盐耐受性
胆汁是由肝脏分泌的,胆汁中的胆盐对脂肪的消化和吸收至关重要[24],而益生菌对胆盐的耐受性也是其在小肠中生存、繁殖的基本条件之一。图3中得出胆盐含量为0.1%时,菌株A9、B8生长较好,其OD600值均在0.4以上(P<0.000 1)。随着胆盐浓度升高,抑制了菌株的生长,但菌株仍然有活性(P<0.01)。表明凝结魏茨曼氏菌A9、B8可以基本适应人体肠道中胆盐浓度(胆盐的浓度范围约在0.03%~0.3%)[25-26],且B8的胆盐耐受性较A9更好。

a-B8胆盐耐受性;b-A9胆盐耐受性
图3 耐胆盐能力测定
Fig.3 Assessment of bile salt tolerance
2.3.3 抑菌性测定
凝结魏茨曼氏菌可以分泌如凝结素等抑菌物质来抑制致病微生物的生长,从而改善消化道微生态[27-28]。凝结魏茨曼氏菌B8、A9的代谢产物对6种常见的致病菌肺炎克雷伯菌(T35)、大肠杆菌(T21)、鼠伤寒沙门氏菌(T26)、鲍氏志贺氏菌(T28)、金黄色葡萄球菌(T22)和单核细胞增生李斯特菌(T42)均有较强的抑菌活性(表2)。其中,B8、A9对单增李斯特菌的抑菌活性最佳,抑菌圈均在20 mm以上;B8对以上6种致病微生物的抑菌性强于A9。综上可知B8具有更好的开发潜力。
表2 抑菌性测定
Table 2 Assay of bacteriostatic properties
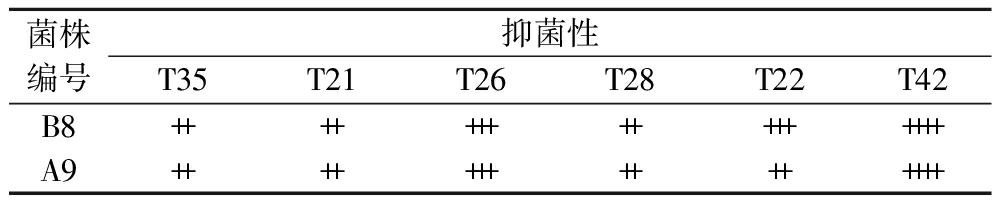
菌株编号抑菌性T35T21T26T28T22T42B8++++++++++++++++A9+++++++++++++++
注:8~10 mm弱抑菌性(+);10~15 mm较强抑菌性(++);15~20 mm强抑菌性(+++);>20 mm极强抑菌性(++++);无抑菌性(-)。
2.4 体外尿酸代谢机理解析
2.4.1 全基因组序列分析
采用circos绘制凝结魏茨曼氏菌B8基因组的圈图(图4-a)。该菌株的基因组由1个不包含质粒的完整环状染色体构成,其片段总长度为3 669 133 bp,GC含量为46.23%。利用prodial预测凝结魏茨曼氏菌B8基因组中的编码基因,共包含3 489个编码基因,占整个基因组的82.36%。

a-全基因组圈图;b-COG注释;c-GO注释;d-KEGG注释
图4 菌株的全基因组圈图、COG注释、GO注释和KEGG注释
Fig.4 Whole genome circle diagram of the strain, COG annotation, GO annotation, and KEGG annotation
利用Interproscan对具有生物学功能的蛋白编码基因进行生物学功能注释,通过与COG(Clusters of Orthologous Groups of proteins)数据库比对,可以对预测蛋白进行功能注释、归类以及蛋白进化分析[29]。凝结魏茨曼氏菌B8的COG功能注释分类图,如图4-b所示,凝结魏茨曼氏菌B8基因组的蛋白序列主要注释到碳水化合物运输和代谢(255个)、氨基酸运输和代谢(265个)、转录(220个)、翻译、核糖体结构和生物生成(212个)等功能中。
凝结魏茨曼氏菌B8的GO功能注释分类见图4-c,主要注释到生物学过程(1 696个)、分子功能(1 779个)和生物合成过程(664个)等分类中。
图4-d为凝结魏茨曼氏菌B8的KEGG数据库功能注释分布情况。凝结魏茨曼氏菌B8的基因组共注释了1 897个基因,这些编码基因功能主要注释于核苷酸代谢(89个)、碳水化合物代谢(262个)、氨基酸的代谢(185个)、膜运输(156个)和辅因子维生素的代谢(128个)等代谢途径中。其中核苷酸代谢中的嘌呤代谢与尿酸代谢直接相关,因此将进一步分析KEGG中与尿酸代谢有关的具体编码基因。
2.4.2 尿酸代谢相关基因分析
与KEGG数据库比较,将基因整合到KEGG代谢通路图上。含有基因数量最多的10条通路包括ko01100(567个)、ko01110(267个)、ko01130(203个)、ko01120(182个)、ko01230(116个)、ko01200(107个)、ko02010(103个,ABC 转运体)、ko02020(70个,双组分系统)、ko00230(62个,嘌呤代谢)、ko03010(54个,核糖体)。其中嘌呤代谢通路与尿酸代谢相关,尿酸代谢相关基因分析见表3。
表3 尿酸代谢相关基因分析
Table 3 Analysis of genes related to uric acid metabolism

基因ID基因名KEGG注释基因ID基因名KEGG注释B8_01870prs,PrsA,PRPS,prsA磷酸核糖焦磷酸激酶B8_00311holA,HolA,holADNA聚合酶Ⅲ亚基δB8_00386SpoT,relAGTP焦磷激酶B8_01448AdeC,ade腺嘌呤脱氨酶B8_01888hpt,TilS,hprT,hpt,HPRT1次黄嘌呤磷酸核糖转移酶B8_02014cysC,CysC,cysC腺苷酸硫酸激酶B8_01451purH,PurH,purH磷酸核糖基氨基咪唑甲酰胺甲基转移酶/IMP 环醇酶B8_01536guaA,guaA,GuaA1,guaA,GMPSGMP合成酶(谷氨酰胺水解酶)B8_00106Ndk,ndk,NME核苷二磷酸激酶B8_01177YjbM,E2.7.6.5X假定的GTP焦磷酸激酶B8_01998rpoA,RpoA,rpoADNA指导的RNA聚合酶亚基αB8_02013sat,MET3,sat,met3硫酸腺苷转氨酶B8_00460rdgB,RdgB,rdgBXTP/dITP二磷酸水解酶B8_02911YjbM,E2.7.6.5X假定的GTP焦磷激酶B8_00536pyk,PykF,PK,pyk丙酮酸激酶B8_01455purL,PurL1,purL,PFAS磷酸核糖酰甲酰基甘氨嘧啶合成酶B8_01458purC,PurC,purC磷酸核糖基氨基咪唑-琥珀酰甲酰胺合成酶B8_01994Adk,adk,AK腺苷酸激酶B8_01452purN,PurN,purN磷酸核糖基甘氨酰胺甲酰转移酶1B8_01450purD,PurD,purD磷酸核糖胺-甘氨酸连接酶B8_02667allB,AllB,allB尿囊素酶B8_03515PucG,pucG(S)-脲基甘氨酸-乙醛酸转氨酶B8_01966rpoC,RpoC,rpoCDNA引导的RNA聚合酶亚基β′B8_03516ArgE,allC尿囊酸脱氨酶B8_01460purK,PurK,purK5-(羧基氨基)咪唑核糖核苷酸合成酶B8_00235NrdA,E1.17.4.1A,nrdA,nrdE核糖核苷-二磷酸还原酶α链B8_01663rpoE,RpoE,rpoEDNA引导的RNA聚合酶亚基δB8_03259pnp,Pnp,pnp,PNPT1多核苷酸转移酶B8_01922Dck,dgk脱氧鸟苷激酶B8_02559ManB,pgm磷酸葡萄糖转化酶B8_03519uraD,PucL,PRHOX-NB,URAD2-氧代-4-羟基-4-羧基-5-脲基咪唑啉脱羧酶B8_03157gmk,Gmk,E2.7.4.8gmk鸟苷酸激酶B8_03248polC,polCDNA聚合酶Ⅲ亚基α% 2C革兰氏阳性型B8_01457purS,PurS,purL,PFAS磷酸核糖酰甲酰基甘氨嘧啶合成酶B8_02190NrdB,E1.17.4.1B,nrdB,nrdF核糖核苷-二磷酸还原酶β链B8_00166MutT,nudFADP核糖焦磷酸酶B8_01362deoB,DeoB,deoB磷酸戊二酸酶B8_01927dnaX,DnaX,dnaXDNA聚合酶Ⅲ亚基γ/tauB8_02188NrdA,E1.17.4.1A,nrdA,nrdE核糖核苷-二磷酸还原酶α链B8_00055xpt,xpt黄嘌呤磷酸核糖转移酶B8_01915guaB,GuaB,IMPDH,guaBIMP脱氢酶B8_03506NrdB,E1.17.4.1B,nrdB,nrdF核糖核苷-二磷酸还原酶 β链B8_01453purM,PurM,purM磷酸核糖酰甲酰基甘氨脒环连接酶B8_03158rpoZ,RpoZ,rpoZDNA引导的RNA聚合酶亚基ΩB8_00530polA,ExoIX,polADNA聚合酶ⅠB8_00541dnaE,DnaE,dnaEDNA聚合酶Ⅲ亚基αB8_01800purA,PurA,purA,ADSS腺苷琥珀酸合成酶B8_00044DnaQ,dnaQDNA聚合酶Ⅲ亚基εB8_01461purE,PurE,purE5-(羧基氨基)咪唑核糖核苷酸突变酶B8_01456purQ,PurL2,purL,PFAS磷酸核糖酰甲酰基甘氨嘧啶合成酶B8_01965rpoB,RpoB,rpoBDNA引导的RNA聚合酶亚基βB8_01454purF,PurF,purF,PPAT氨基磷酸核糖转移酶B8_01459purB,PurB,purB,ADSL腺苷琥珀酸酶B8_01921Dck,dck脱氧腺苷/脱氧胞苷激酶B8_01145UshA,cpdB2′% 2C3′-环核苷酸2′-磷酸二酯酶/3′-核苷酸酶B8_01364deoD,DeoD,deoD嘌呤核苷磷酸酶B8_01847holB,DnaX,holBDNA聚合酶Ⅲ亚基δ′B8_01827dnaN,DnaN,dnaNDNA聚合酶Ⅲ亚基βB8_01355guaC,GuaB,E1.7.1.7guaCGMP还原酶B8_03521uraH,HiuH,uraH,pucM,hiuH5-羟基异酸酯水解酶B8_00387apt,RpoD,APRT,apt腺嘌呤磷酸核糖转移酶B8_01281nrdD,NrdD,nrdD核糖核苷-三磷酸还原酶(甲酸盐)
将所有与降尿酸相关的基因整合绘制到KEGG代谢通路图上(图5)。从尿酸合成方向而言,5′-磷酸核糖和三磷酸腺苷(adenosine triphosphate,ATP)在磷酸核糖焦磷酸激酶作用下生成5-磷酸核糖-1-焦磷酸,继而生成次黄嘌呤核苷酸(inosincacid,inosinemonophosphate,IMP)[14,30-31]。由核苷酸生成尿酸大致分为4条途径:1)IMP在IMP脱氢酶的作用下生成黄嘌呤核苷酸(xanthosine monophosphate,XMP),XMP在GMP合成酶的作用下生成鸟嘌呤核苷酸(guanosine 5′-monophosphate,GMP),进一步生成鸟嘌呤,接着在鸟嘌呤脱氨酶的作用下生成黄嘌呤,进而生成尿酸。凝结魏茨曼氏菌B8缺乏直接将鸟嘌呤转化为黄嘌呤的鸟嘌呤脱氨酶,因此直接减少了黄嘌呤的产生,进而减少尿酸生成;2)IMP在5′-核苷酸酶的作用下生成黄嘌呤核苷,在嘌呤核苷磷酸酶作用下生成黄嘌呤,进而生成尿酸。凝结魏茨曼氏菌B8缺乏5′-核苷酸酶,从而减少黄嘌呤核苷的生成,间接减少尿酸的生成;3)IMP在GMP/IMP 5′-核苷酸酶的作用下生成肌苷,继而在嘌呤核苷磷酸酶作用下生成次黄嘌呤,进一步代谢为黄嘌呤和尿酸。凝结魏茨曼氏菌B8缺乏GMP/IMP 5′-核苷酸酶,间接减少尿酸的生成,同时次黄嘌呤磷酸核糖转移酶的存在将次黄嘌呤还原为IMP,进一步减少尿酸的生成;4)腺嘌呤核苷酸(adenosine nucleotides,AMP)在5′-核苷酸酶的作用下生成腺苷,在嘌呤核苷磷酸酶的作用下生成腺嘌呤,继而在腺嘌呤脱氨酶的作用下生成次黄嘌呤,进一步在黄嘌呤脱氢酶/氧化酶的作用下生成黄嘌呤,代谢为尿酸。凝结魏茨曼氏菌B8缺少5′-核苷酸酶无法生成腺苷,且缺少黄嘌呤脱氢酶/氧化酶无法生成黄嘌呤,进一步减少了尿酸的生成。

图5 嘌呤代谢通路图
Fig.5 Diagram of purine metabolic pathway
注:红色表示凝结魏茨曼氏菌B8全基因组中注释到的嘌呤代谢相关基因。
从尿酸代谢方向而言,凝结魏茨曼氏菌B8没有注释出尿酸氧化酶(此酶将尿酸盐代谢为5-羟基异酸酯),存在5-羟基异酸酯水解酶将5-羟基异酸酯代谢为S-羟基-2-氧代-4-脲基-2,5-二氢-1H-咪唑-5羧酸酯,自发代谢为(R)尿囊素,或者在2-氧代-4-羟基-4-羧基-5-脲基咪唑啉脱羧酶的作用下代谢为(S)尿囊素,(S)尿囊素在尿囊酸脱氨酶的作用下生成尿苷酸,在(S)-脲基甘氨酸-乙醛酸转氨酶的作用下代谢为甘氨酸,促进了尿酸的降解。
2.4.3 尿酸氧化酶活性的测定
凝结魏茨曼氏菌B8在仅有上清液存在(即代谢物)时,并不降解尿酸,在细胞内容物存在时,表现出良好的尿酸降解效果。因此推测降解尿酸的是细胞内容物中的胞内酶,由于其可以直接分解尿酸,推测可能存在尿酸氧化酶。测定细胞内容物中的尿酸氧化酶活性,发现可达(1.34±0.16) U/mL,进一步证明凝结魏茨曼氏菌B8存在尿酸酶可以直接降解尿酸,而全基因组测序功能注释时没有被注释到,进而推测B8含完整的尿酸代谢途径。
3 结论
本研究通过对132株不同来源的菌株进行筛选,获得具有较好尿酸降解能力的凝结魏茨曼氏菌B8和A9。研究发现,凝结魏茨曼氏菌B8耐酸性、耐胆盐能力以及抗菌性更强,具有更好的开发潜力。
通过全基因组分析发现,该菌株具有多个尿酸代谢的相关基因,在KEGG嘌呤代谢相关通路中共注释到62个,如尿囊素酶、磷酸核糖焦磷酸激酶、IMP脱氢酶、腺嘌呤磷酸核糖转移酶等,进一步证明了凝结魏茨曼氏菌B8具有潜在的降尿酸作用。通过结合嘌呤代谢途径分析,推测B8或含完整尿酸代谢途径,即:1)尿酸酶将尿酸盐代谢为5-羟基异酸酯,在5-羟基异酸酯水解酶作用下代谢为S-羟基-2-氧代-4-脲基-2,5-二氢-1H-咪唑-5羧酸酯,自发代谢为(R)尿囊素;2)尿酸酶将尿酸盐代谢为5-羟基异酸酯,继而代谢为S-羟基-2-氧代-4-脲基-2,5-二氢-1H-咪唑-5羧酸酯,在2-氧代-4-羟基-4-羧基-5-脲基咪唑啉脱羧酶的作用下代谢为(S)尿囊素,在尿囊酸脱氨酶的作用下生成尿苷酸,最后在(S)-脲基甘氨酸-乙醛酸转氨酶的作用下代谢为甘氨酸。从而凝结魏茨曼氏菌B8可以在人体内尿酸酶失活的情况下,替代尿酸酶的作用,在肠道中完成尿酸的代谢。
[1] WEN S J, ARAKAWA H, TAMAI I.Uric acid in health and disease:From physiological functions to pathogenic mechanisms[J].Pharmacology &Therapeutics, 2024, 256:108615.
[2] LIU Y Y, JARMAN J B, LOW Y S, et al.A widely distributed gene cluster compensates for uricase loss in hominids[J].Cell, 2023, 186(20):4472-4473.
[3] MIKULS T R.Gout[J].New England Journal of Medicine, 2022, 387(20):1877-1887.
[4] 郎文悦, 包海鹰, 贾淞贺, 等.粗毛纤孔菌提取物对高尿酸血症小鼠的降尿酸及肝肾保护作用[J].菌物学报, 2024, 43(10):144-162.LANG W Y, BAO H Y, JIA S H, et al.Effects of Inonotus hispidus fruiting body extracts on reducing uric acid and protecting liver and kidney of hyperuricemia mice[J].Mycosystema, 2024, 43(10):144-162.
[5] JURASCHEK S P, YOKOSE C, MCCORMICK N, et al.Effects of dietary patterns on serum urate:Results from a randomized trial of the effects of diet on hypertension[J].Arthritis &Rheumatology, 2021, 73(6):1014-1020.
[6] ZHAO X T, CAI P, XIONG S J, et al.Lacticaseibacillus rhamnosus NCUH061012 alleviates hyperuricemia via modulating gut microbiota and intestinal metabolites in mice[J].Food Bioscience, 2024, 58:103699.
[7] 罗学刚, 杨小涵, 秦济昆, 等.一种源于虾酱的具有降尿酸及抗氧化能力的凝结芽孢杆菌、方法及应用:中国,CN202210180130.2[P].2023-08-15.LUO X G, YANG X H, QIN J K, et al.A Bacillus coagulans derived from shrimp paste with uric acid-lowering and antioxidant capacity, method and application:China, CN202210180130.2[P].2023-08-15.
[8] 李婧, 姚彩青, 王俊国.高降尿酸活性乳酸菌的筛选及其作用机制[J].食品科学技术学报, 2024, 42(6):166-174.LI J, YAO C Q, WANG J G.Screening of lactic acid bacteria with high uric acid lowering activity and its mechanism of action[J].Journal of Food Science and Technology, 2024, 42(6):166-174.
[9] 任宇杰. 降解嘌呤核苷乳酸菌的筛选及高活性冻干菌粉制备研究[D].南京:南京农业大学, 2022.REN Y J.Study on the screening of purine nucleoside degrading lactic acid bacteria and preparation of highly active freeze-dried bacteria powder [D].Nanjing:Nanjing Agricultural University, 2022.
[10] 邹兴愿. 降解嘌呤乳酸菌的筛选及其分子机理初探[D].昆明:昆明理工大学, 2024.ZOU X Y.Screening of purine degrading lactic acid bacteria and its molecular mechanism[D].Kunming:Kunming University of Science and Technology, 2024.
[11] 刘慧敏. 乳酸菌嘌呤降解菌株的筛选、基因组特征及其应用研究[D].扬州:扬州大学, 2021.LIU H M.Screening, genomic characterisation and application of purine degrading lactic acid bacteria[D].Yangzhou:Yangzhou University, 2021.
[12] LU L H, LIU T T, LIU X L, et al.Screening and identification of purine degrading Lactobacillus fermentum 9-4 from Chinese fermented rice-flour noodles[J].Food Science and Human Wellness, 2022, 11(5):1402-1408.
[13] 王诗琪. 肉源性乳酸菌降嘌呤机制研究[D].呼和浩特:内蒙古农业大学, 2023.WANG S Q.Mechanism of purine reduction by meat-derived lactic acid bacteria[D].Hohhot:Inner Mongolia Agricultural University, 2023.
[14] 金方. 降血尿酸益生菌株的筛选和对高尿酸血症模型大鼠作用机理的探索[D].上海:上海交通大学, 2018.JIN F.Isolation of hypouricemic probiotics andexploration the mechanism of hyperuricemic effects on model rats [D].Shanghai:Shanghai Jiao Tong University, 2018.
[15] 吴世芳. 传统发酵牦牛乳中降尿酸乳酸菌的筛选及功能评价[D].兰州:甘肃农业大学, 2023.WU S F.Screening and functional evaluation of uric acid-loweringlactic acid bacteria in traditional fermented yak milk [D].Lanzhou:Gansu Agricultural University, 2023.
[16] 呼静, 崔鹏月, 双全.高效降尿酸乳酸菌的筛选及其益生特性研究[J].食品与发酵工业, 2022, 48(16):150-155.HU J, CUI P Y, SHUANG Q.Screening of lactic acid bacteria with high ability of uric acid-lowering and evaluation of its probiotic properties[J].Food and Fermentation Industries, 2022, 48(16):150-155.
[17] 赵雪婷. 鼠李糖乳酪杆菌NCUH061012及其发酵胡萝卜缓解高尿酸血症作用与机制研究[D].南昌:南昌大学, 2024.ZHAO X T.The effect and mechanism of alleviating hyperuricemia by Lacticaseibacillus rhamnosus NCUH061012 and fermented carrots [D].Nanchang:Nanchang University, 2024.
[18] 王学颖, 刘艳丽, 何苗, 等 副干酪乳杆菌FP02对高尿酸血症大鼠血清尿酸水平的影响[J].食品科学, 2024, 45(14):114-120.WANG X Y, LIU Y L, HE M, et al.Effect of Lactobacillus paracasei FP02 on the serum uric acid level in hyperuricemic rats[J].Food Science, 2024, 45(14):114-120.
[19] 王家彬, 潘力.降解核苷作用乳酸菌的筛选及其潜在降尿酸功能[J].食品科学, 2022, 43(10):199-206.WANG J B, PAN L.Screening and uric acid-lowering potential of nucleoside-degrading lactic acid bacteria [J].Food Science, 2022, 43(10):199-206.
[20] WU Y, YE Z, FENG P Y, et al.Limosilactobacillus fermentum JL-3 isolated from “Jiangshui” ameliorates hyperuricemia by degrading uric acid[J].Gut Microbes, 2021, 13(1):1897211.
[21] CHANDRASEKARAN P, WEISKIRCHEN S, WEISKIRCHEN R.Effects of probiotics on gut microbiota:An overview[J].International Journal of Molecular Sciences, 2024, 25(11):6022.
[22] XIE A Q, JI H H, LIU Z Y, et al.Modified prebiotic-based “shield” armed probiotics with enhanced resistance of gastrointestinal stresses and prolonged intestinal retention for synergistic alleviation of colitis[J].ACS Nano, 2023, 17(15):14775-14791.
[23] 李利, 孔丽, 张宁, 等.耐酸耐胆盐乳酸菌的鉴定及筛选[J].食品科学, 2015, 36(21):123-128.LI L, KONG L, ZHANG N, et al.Identification and selection of lactic acid bacteria resistant to acid and bile salt[J].Food Science, 2015, 36(21):123-128.
[24] HSU C L, SCHNABL B.The gut-liver axis and gut microbiota in health and liver disease[J].Nature Reviews Microbiology, 2023, 21(11):719-733.
[25] 张汝京, 胡亚凡, 海丹, 等.具有抑制肠道致病菌和黏附Caco-2细胞作用的益生性乳酸菌的筛选及鉴定[J].食品工业科技, 2019, 40(20):133-139;153.ZHANG R J, HU Y F, HAI D, et al.Screening and identification of probiotic Lactobacillus strains inhibiting intestinal pathogens and adhering to caco-2 cells[J].Science and Technology of Food Industry, 2019, 40(20):133-139;153.
[26] 汪晓辉, 于平, 励建荣.泡菜、传统腊肠中降胆固醇乳酸菌的筛选及鉴定[J].微生物学报, 2009, 49(11):1438-1444.WANG X H, YU P, LI J R.Isolation and identification of cholesterol-reducing lactic acid bacteria from indigenously fermented pickles and dried-sausage[J].Acta Microbiologica Sinica, 2009, 49(11):1438-1444.
[27] 董惠钧, 姜俊云, 郑立军, 等.新型微生态益生菌凝结芽孢杆菌研究进展[J].食品科学, 2010, 31(1):292-294.DONG H J, JIANG J Y, ZHENG L J, et al.Research advances of novel microecologic probiotics Bacillus coagulans[J].Food Science, 2010, 31(1):292-294.
[28] 王一帆, 岑丛楠, 刘福奇, 等.凝结芽孢杆菌的益生特性及机制研究进展[J].食品工业科技, 2023, 44(1):458-464.WANG Y F, CEN C N, LIU F Q, et al.Research progress on the probiotic characteristics and mechanism of Bacillus coagulans[J].Science and Technology of Food Industry, 2023, 44(1):458-464.
[29] XIONG J, FENG L X, YOUSAF M, et al.Assessment of uric acid-lowering activity, safety and stress tolerance of Lactiplantibacillus plantarum MC14 based on whole gene sequencing and phenotyping experiments[J].Food Bioscience, 2024, 61:104536.
[30] SCHULTZ A C, NYGAARD P, SAXILD H H.Functional analysis of 14 genes that constitute the purine catabolic pathway in Bacillus subtilis and evidence for a novel regulon controlled by the PucR transcription activator[J].Journal of Bacteriology, 2001, 183(11):3293-3302.
[31] 苗超, 张宇, 杨鑫焱, 等.降尿酸乳酸菌菌株的筛选及其全基因组分析[J].中国食品学报, 2024, 24(7):88-99.MIAO C, ZHANG Y, YANG X Y, et al.Screening and whole genome sequencing analysis of lactic acid bacteria with lowering uric acid[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(7):88-99.