在食品发酵工业中,氨基酸作为一类关键的有机氮源,在微生物的生命活动进程中扮演着举足轻重的角色。其不仅对微生物的生长代谢过程产生显著影响,还与发酵过程中若干风味化合物的生物合成存在紧密关联[1]。鉴于此,氨基酸的含量状况已然成为食品发酵工业领域内一项极为重要的衡量指标。在实际检测与分析中,氨基酸含量水平一般借助“氨基酸态氮”这一参数予以表征。所谓氨基酸态氮,具体是指以氨基酸形态存在的氮元素。在发酵食品体系里,氨基酸态氮的形成机制主要源于蛋白质在蛋白酶以及肽酶的相继作用下逐步降解,进而生成小肽和氨基酸,以氨基酸态氮的形式存在[2-3],氨基酸态氮含量可以评估发酵程度和发酵食品的等级,并作为评价产品质量是否合格的一个重要理化指标[4]。在发酵食品行业,少数不良厂商为提升产品的鲜味以及氨基酸态氮含量,会违规添加味精(谷氨酸钠)。然而,这种做法存在损害消费者健康的潜在风险,并且借助氨基酸谱测定技术能够有效检测出来[5]。基于此,在不额外添加氨基酸或味精的情况下,实现发酵食品中氨基酸含量的提升,已成为当下亟待攻克的关键难题。
研究发现,向发酵过程中引入适量氨基酸生产菌株,或者加入外肽酶生产菌,都不失为有效提升食品中氨基酸含量的可行途径。但天然氨基酸生产菌株比如谷氨酸棒杆菌[6]对盐的耐受性较差,需氧量较高,不适合应用于高盐、低氧的发酵食品生产环境。而源自发酵食品的外肽酶生产菌,可以耐受含盐量较高的发酵食品生产环境,是提高发酵食品“氨基酸态氮”的理想候选功能菌株。理论上,氨肽酶[7-8]和羧肽酶[9-10]分别从多肽链的氨基端和羧基端依次切断肽键,释放游离氨基酸。兼具氨肽酶和羧肽酶活性的菌株,能够显著提高蛋白质的水解速度,避免了同时添加单一氨肽酶生产菌株和羧肽酶生产菌株可能导致的营养竞争和相互影响,便于操作,节省人力物力,更适用于工厂发酵。
因此,本研究拟以富含蛋白质的传统发酵食品酸鱼、酸肉、虾酱为主要研究材料,筛选出兼具氨肽酶活性和羧肽酶活性的菌株,并通过形态学、分子生物学鉴定以及安全性和益生性评价,确定一株外肽酶活性最高且安全性和益生性较好的菌株。从而为提高发酵食品中氨基酸含量提供优良菌株,为发酵食品提质减害提供技术支持。
1 材料与方法
1.1 材料与仪器
酸鱼,贵州省黎平县筑绿康农业发展贸易有限公司;酸肉,贵州省黔东南苗族侗族自治州;大连虾酱、蜢虾酱、鲜虾酱,辽宁省大连市仟和农贸市场;Tris、50×TAE缓冲液、TE缓冲液,生工生物工程(上海)股份有限公司;对硝基苯胺、L-亮胺酰对硝基苯胺(L-leucine-p-nitroanilide,LNA)和Z-Phe-Leu,上海麦克林生化科技股份有限公司;L-亮氨酸、水合茚三酮、氯化镉,阿拉丁试剂(上海)有限公司;2×Taq Master Mix(Dye Plus),诺唯赞(南京)生物科技股份有限公司;Genview原装琼脂糖,GEN-VIEW SCIENTIFIC公司;其他均为分析纯试剂。
Jipad-20拍打式无菌均质器,宁波海曙亿恒仪器有限公司;Leica DM2500光学显微镜,德国徕卡显微系统;S1000 PCR仪、ChemiDoc MP System 凝胶成像仪,美国BIO-RAD公司;Scan4000全自动超高清菌落计数器,法国Interscience公司;Micro 17R 冷冻高速离心机,美国Thermo Scientific公司;INFINITE 200多功能酶标仪,瑞士TECAN公司。
1.2 实验方法
1.2.1 培养基配制及菌株分离
LB液体培养基(g/L):胰蛋白胨10.0,酵母粉5.0,NaCl 10.0,pH 7.0。
MRS液体培养基(g/L):蛋白胨10.0,牛肉浸粉8.0,酵母粉4.0,葡萄糖20.0,K2HPO4 2.0,柠檬酸氢二铵2.0,乙酸钠5.0,MgSO4 0.2,MnSO4 0.04,吐温80 1.0。
YPD液体培养基(g/L):蛋白胨20.0,葡萄糖20.0,酵母浸粉10.0。
固体培养基则在液体培养基基础上添加2%(质量分数)琼脂粉。
所有培养基均经121 ℃高压灭菌15 min备用。分别取10.0 g酸肉、酸鱼、虾酱样品置于均质袋中,加入50 mL无菌水进行均质,800 r/min离心5 min以去除样品残渣。将相对混浊的上清液以5 000 r/min再次离心5 min,重悬于1 mL生理盐水,进行梯度稀释,取稀释梯度为10-6~10-3梯度的菌悬液分别涂布到LB平板、MRS平板、YPD平板上,放置于37 ℃或30 ℃培养24 h,选取菌落数为30~300的平板,挑取不同形态的单菌落于相应平板划线并分离纯化3代,直至得到纯种单菌落。将单菌落转接至相应液体培养基中,培养48 h,备用[11]。
1.2.2 肽酶生产菌株筛选
1.2.2.1 菌株发酵液的制备
将1.2.1节中的培养液,5 000 r/min离心5 min收集上清液,0.22 μm滤膜过滤,所得发酵液即样品粗酶液。
1.2.2.2 氨肽酶生产菌株的酶活力测定
利用LNA法[12-13],测定所得分离菌株发酵液中氨肽酶的活性。配制40 μg/mL对硝基苯胺贮液,在此基础上配制一系列质量浓度梯度的标准溶液(2.0、4.0、6.0、8.0、10.0、12.0、14.0、16.0、18.0、20.0、22.0、24.0、26.0、28.0、30.0、32.0、34.0、36.0、38.0、40.0 μg/mL),分别测定不同质量浓度标准溶液在405 nm波长处的吸光度,绘制对硝基苯胺标准曲线。实验组操作如下:精确吸取0.5 mL样品酶液,将其与6 mL pH 9.0的Tris - HCl缓冲液进行充分且均匀的混合,将其置于恒温58 ℃的环境中,持续加热5 min。待加热环节结束,随即加入0.5 mL浓度为26 mmol/L 的LNA乙醇溶液。而后展开水浴反应,时长严格控制为10 min。待反应进程完毕,立即将反应液从水浴装置中取出,迅速转移至冰浴环境维持5 min,以确保反应彻底终止。最终,测定反应液在405 nm波长下的吸光值。与之相对,空白对照组是以等量的无水乙醇完全替换样品酶液。
1.2.2.3 羧肽酶生产菌株的酶活力测定
采用镉-茚三酮法[14-15],测定所得分离菌株粗酶液样品中的羧肽酶活性。依次配制一系列不同浓度的L-亮氨酸标准溶液,浓度依次设定为20、40、60、80、100、120、140 μmol/L。取各浓度的标准溶液1 mL,与2 mL镉-茚三酮试剂充分混合,混合液置于84 ℃的恒温水浴环境中,持续加热5 min。待加热过程结束,自然冷却混合液,直至其温度降至室温状态。利用酶标仪测定其在507 nm波长下的吸光值,绘制L-亮氨酸标准溶液曲线。分别取等量的酶与2 mL的Z-Phe-Leu溶液混合,室温反应20 min,取50 μL样液稀释20倍,加入2 mL的镉-茚三酮试剂,84 ℃水浴条件下加热5 min后立即冰浴以停止反应。测定反应液在507 nm波长下的吸光值。
1.2.3 酶活力定义
1.2.3.1 氨肽酶活力定义
一个酶活力单位(U)被定义为:在58 ℃的反应环境下,每分钟能够催化底物水解,进而生成1 μg对硝基苯胺(p-nitroaniline,p-NA)所需要的酶量。按公式(1)计算氨肽酶活性:
(1)
式中:W,酶活力,U/mL;ΔOD,反应液测定前后变化值;7,反应体系总体积,L;D,样品稀释倍数;K,标准曲线斜率;10,反应时间,min。
1.2.3.2 羧肽酶活力定义
在室温条件下,每分钟使底物Z-Phe-Leu降解产生1 μg亮氨酸所需酶量定义为一个酶活力单位(U)。按公式(2)计算羧肽酶活性:
(2)
式中:W,酶活力,U/mL;x,L-亮氨酸浓度,μmoL/L;y,酶所分解Z-Phe-Leu的总体积,L;t,反应时间,min;f,稀释倍数。
1.2.4 肽酶生产菌株的鉴定
1.2.4.1 菌株形态学鉴定
利用普通光学显微镜观察菌体形态。
1.2.4.2 菌株分子生物学(16S rDNA)鉴定
菌株基因组DNA的提取采用玻璃珠破碎法[16]。利用16S rDNA通用引物27F:5′-AGAGTTTGATCCTGGCTCAG-3′和1492R:5′-GGTTACCTTGTTACG-ACTT-3′[17-18]以菌株的基因组DNA为模板进行PCR扩增。PCR反应体系构建如下:在总体积为50 μL的体系中,包含2×Taq Master Mix 25 μL;上游引物(浓度为10 μmol/L)与下游引物(浓度为10 μmol/L)各添加2 μL;基因组模板(质量浓度为50 ng/μL)加入1 μL;余量20 μL为灭菌水。PCR扩增遵循以下条件:起始阶段,95 ℃预变性处理3 min;随后进入循环阶段,每个循环依次为95 ℃变性15 s,50 ℃退火15 s,72 ℃延伸1 min,如此循环共进行35 次;扩增结束后,4 ℃保温15 min。琼脂糖凝胶电泳分析则选取PCR扩增产物中的5 μL开展,而剩下的45 μL PCR扩增产物将送至生工生物工程(上海)股份有限公司测序。菌株的测序结果通过NCBI(http:// www.ncbi.nlm.nih.gov/)网站上的BLAST程序与GenBank中核酸数据库进行对比分析。
1.2.5 安全性评价
1.2.5.1 溶血性实验
将冻存菌株活化2代以上,挑取单菌落,在含有5%脱纤维羊血的哥伦比亚培养基平板上进行划线接种。接种完成后,将平板放置于37 ℃恒温培养箱中,静置培养24 h。培养结束后,仔细观察平板上菌落周围是否出现溶血圈现象。本次实验设置了阳性与阴性对照,其中选用金黄色葡萄球菌ATCC 25923作为溶血阳性对照菌株,益生菌大肠杆菌Nissle 1917[19]作为溶血性阴性对照菌株。为确保实验结果的可靠性与重复性,整个实验过程独立重复进行3次[20]。
1.2.5.2 抗生素敏感性实验
抗生素敏感性试验测定采用纸片扩散法[21]。将无溶血圈的菌株,在37 ℃、200 r/min条件下液体培养至对数生长期,分别吸取100 μL菌液,均匀涂布于LB固体培养基上。随后,在平板上依次放置10种抗生素药敏纸片,具体包括链霉素(10 μg)、四环素(30 μg)、头孢氨苄(30 μg)、头孢唑林(30 μg)、阿米卡星(30 μg)、米诺环素(30 μg)、庆大霉素(10 μg)、万古霉素(30 μg)、青霉素(10 μg)以及红霉素(15 μg)。需注意,各药敏纸片间的间距应严格控制在≥24 mm。将涂布与放置好纸片的平板置于37 ℃中,静置培养24 h。培养结束后,精确测量各药敏纸片周围抑菌圈的直径,并依据测量结果进行统计分析,判断其耐药性,划分标准为:抗性R(抑菌圈直径≤14 mm)、中期I(抑菌圈直径在14~20 mm之间)以及敏感S(抑菌圈直径≥20 mm),实验重复3次进行。
1.2.6 抗氧化实验
1.2.6.1 DPPH自由基清除率
抗氧化能力是菌株益生性实验中非常重要的评价指标,参照RIZZELLO等[22]的方法,进行测定。首先将菌株悬浮液按适当比例稀释,然后吸取1 mL稀释菌液与0.2 mmol/L V(DPPH)∶V(无水乙醇溶液)=1∶1混合,并在室温条件下避光反应30 min。以稀释菌液加无水乙醇作为空白,1×PBS加0.2 mmol/L DPPH-无水乙醇溶液作为对照。在4 000 r/min条件下离心10 min,得到上清液。用酶标仪测定其在517 nm处的吸光度值。实验重复3次进行。按公式(3)计算清除率:
DPPH自由基清除率![]()
(3)
式中:Ai,1 mL待测稀释菌悬液加1 mL DPPH-无水乙醇溶液的吸光度值;Aj,1 mL待测稀释菌悬液加1 mL无水乙醇的吸光度值;Ac,1 mL 1×PBS溶液加1 mL DPPH-无水乙醇溶液的吸光度值。
1.2.6.2 ABTS阳离子自由基清除率
参照文献[23]的方法,并稍作修改。将筛选出的菌株同时进行ABTS阳离子自由基清除试验。首先配制14 mmol/L的ABTS溶液,同时配制5 mmol/L过硫酸钾溶液,将上述试剂按体积比1∶1比例混合,将混合溶液在室温条件下反应避光16 h,待后续使用。吸取100 μL培养至对数生长期的待测菌液添加至900 μL的前期配制好的ABTS-过硫酸钾混合溶液中,在室温条件下避光培养15 min。以ABTS-过硫酸钾混合溶液作为对照。在4 000 r/min条件下离心10 min,得到上清液。用酶标仪测定其在734 nm处的吸光度。实验重复3次进行。按公式(4)计算清除率:
ABTS阳离子自由基清除率![]()
(4)
式中:As,100 μL待测菌悬液加900 μL ABTS溶液的吸光度值;Ac,1 mL的ABTS-过硫酸钾混合溶液的吸光度值。
2 结果与分析
2.1 肽酶生产菌株筛选
本实验分别从酸鱼、酸肉、虾酱中筛选出8、22、29株共59株菌,分别为19株芽孢杆菌、22株乳酸菌、18株酵母菌。采用LNA法测定氨肽酶活力[24],镉-茚三酮法测定羧肽酶活力[25]。酶活力越高代表菌株产生的外肽酶对蛋白水解能力越强,且产酶能力越强。经过菌株发酵筛选,发酵48 h后菌株的氨肽酶活力范围在0~800 U/mL,羧肽酶活力范围在0~90 U/mL。如表1所示。
表1 氨肽酶和羧肽酶生产菌株的酶活力结果 单位:U/mL Table 1 Enzyme activity results for aminopeptidase and carboxypeptidase producing strains
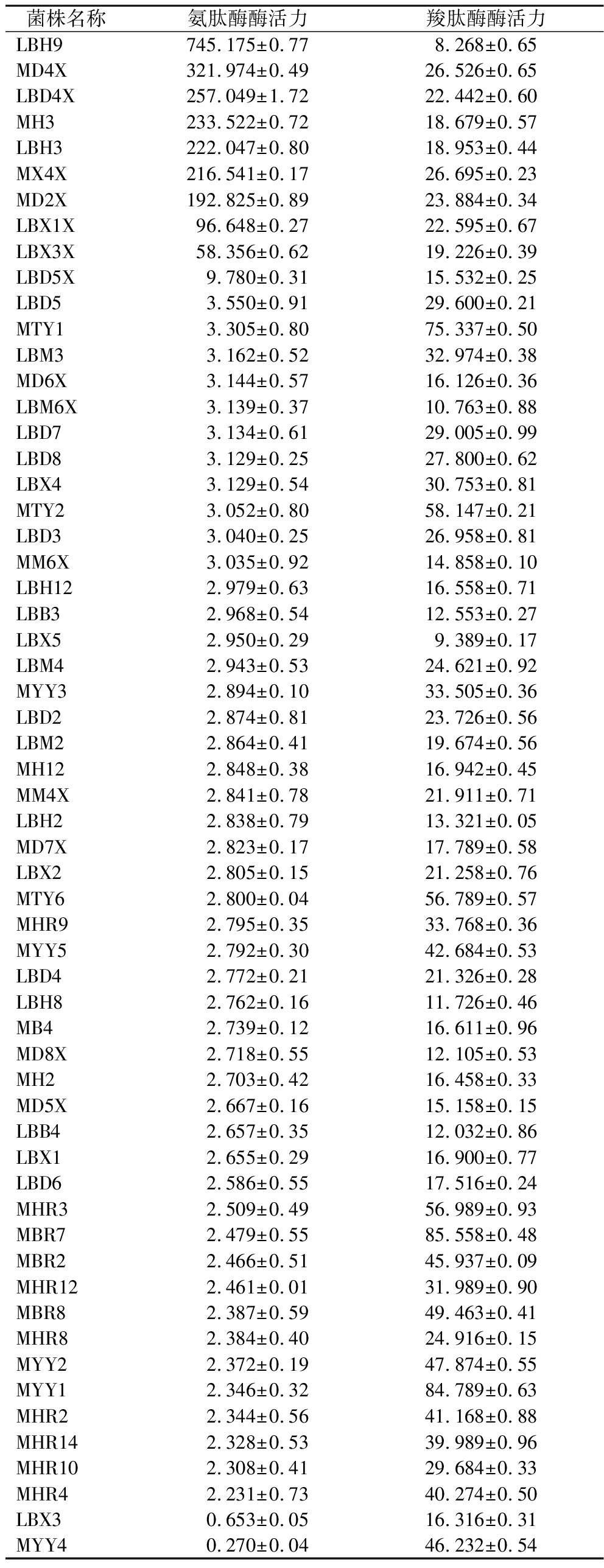
菌株名称氨肽酶酶活力羧肽酶酶活力LBH9745.175±0.778.268±0.65MD4X321.974±0.4926.526±0.65LBD4X257.049±1.7222.442±0.60MH3233.522±0.7218.679±0.57LBH3222.047±0.8018.953±0.44MX4X216.541±0.1726.695±0.23MD2X192.825±0.8923.884±0.34LBX1X96.648±0.2722.595±0.67LBX3X58.356±0.6219.226±0.39LBD5X9.780±0.3115.532±0.25LBD53.550±0.9129.600±0.21MTY13.305±0.8075.337±0.50LBM33.162±0.5232.974±0.38MD6X3.144±0.5716.126±0.36LBM6X3.139±0.3710.763±0.88LBD73.134±0.6129.005±0.99LBD83.129±0.2527.800±0.62LBX43.129±0.5430.753±0.81MTY23.052±0.8058.147±0.21LBD33.040±0.2526.958±0.81MM6X3.035±0.9214.858±0.10LBH122.979±0.6316.558±0.71LBB32.968±0.5412.553±0.27LBX52.950±0.299.389±0.17LBM42.943±0.5324.621±0.92MYY32.894±0.1033.505±0.36LBD22.874±0.8123.726±0.56LBM22.864±0.4119.674±0.56MH122.848±0.3816.942±0.45MM4X2.841±0.7821.911±0.71LBH22.838±0.7913.321±0.05MD7X2.823±0.1717.789±0.58LBX22.805±0.1521.258±0.76MTY62.800±0.0456.789±0.57MHR92.795±0.3533.768±0.36MYY52.792±0.3042.684±0.53LBD42.772±0.2121.326±0.28LBH82.762±0.1611.726±0.46MB42.739±0.1216.611±0.96MD8X2.718±0.5512.105±0.53MH22.703±0.4216.458±0.33MD5X2.667±0.1615.158±0.15LBB42.657±0.3512.032±0.86LBX12.655±0.2916.900±0.77LBD62.586±0.5517.516±0.24MHR32.509±0.4956.989±0.93MBR72.479±0.5585.558±0.48MBR22.466±0.5145.937±0.09MHR122.461±0.0131.989±0.90MBR82.387±0.5949.463±0.41MHR82.384±0.4024.916±0.15MYY22.372±0.1947.874±0.55MYY12.346±0.3284.789±0.63MHR22.344±0.5641.168±0.88MHR142.328±0.5339.989±0.96MHR102.308±0.4129.684±0.33MHR42.231±0.7340.274±0.50LBX30.653±0.0516.316±0.31MYY40.270±0.0446.232±0.54
根据氨肽酶活力和羧肽酶活力测定结果最终选取2种酶活力总和最高的6株菌株(菌株编号为:LBH9、MD4X、LBD4X、MH3、LBH3、MX4X),如图1所示。诸多研究致力于氨基肽酶相关探索。例如,XI等[26]开展了关于枯草芽孢杆菌zj016氨基肽酶基因的研究工作,他们将该基因在毕赤酵母中进行异源表达。在甲醇诱导96 h后,所获得的酶活力水平达到28.4 U/mL。此外,孟广超[27]以米曲霉菌株CICC 2066为研究对象,通过发酵实验发现,在发酵进行至48 h时,氨肽酶的酶活力可达到150 U/mL,但研究中未发现其同时具有一定水平的产羧肽酶能力。相比较,LBH9菌株氨肽酶酶活力显著高于实验中其他菌株,其酶活力值为745.175 U/mL,且同时具有产羧肽酶能力,其酶活力值为8.268 U/mL。LBH9菌株具有更高的外肽酶活力,有助于提高发酵调味品鲜味。MD4X菌株氨肽酶酶活力仅次于LBH9菌株,其酶活力值为321.974 U/mL,但具有比LBH9菌株强的产羧肽酶能力,其酶活力值为26.526 U/mL,也适用于发酵食品。
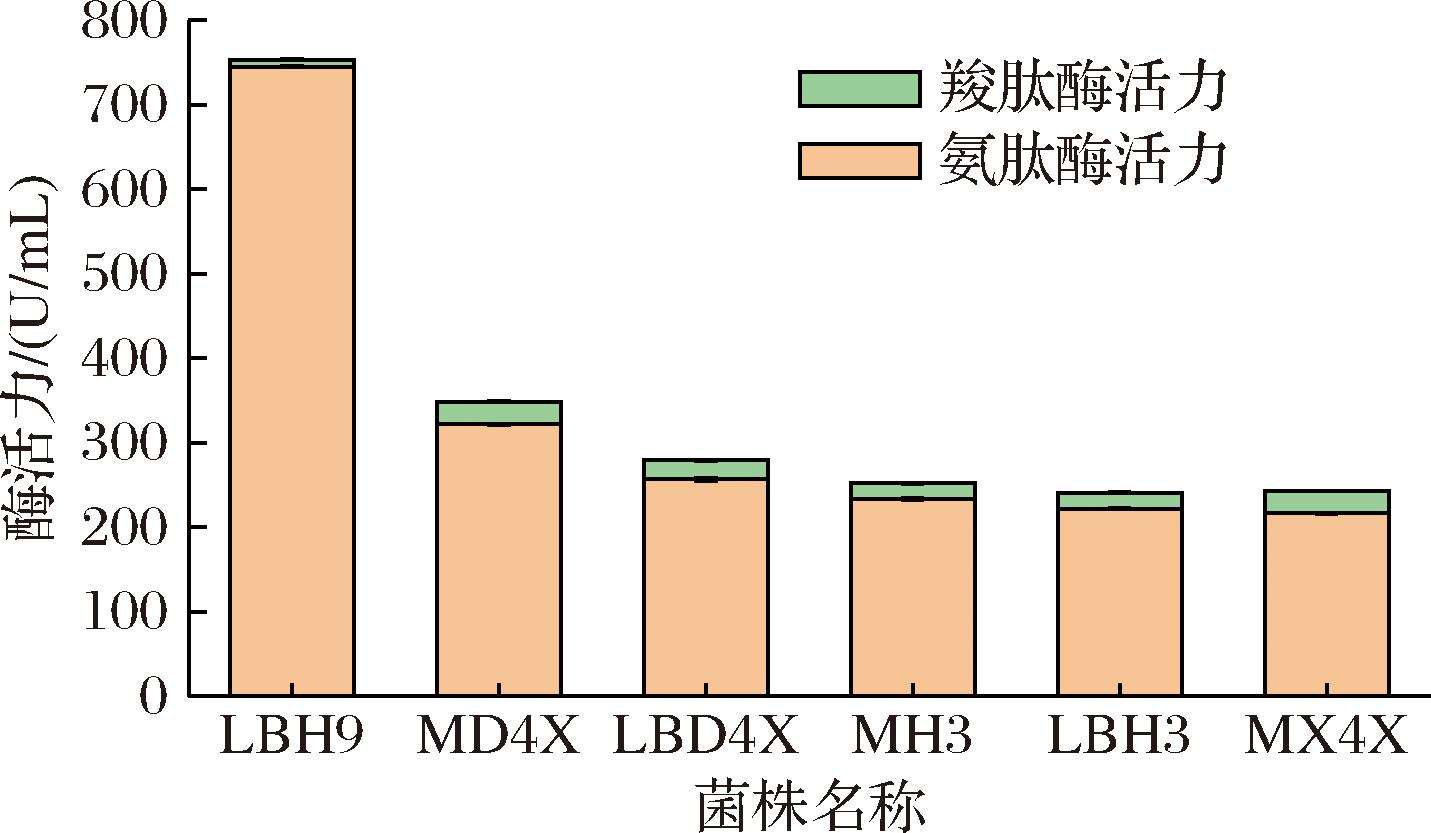
图1 氨肽酶和羧肽酶生产菌株的酶活力结果
Fig.1 Enzyme activity results for aminopeptidase and carboxypeptidase producing strains
2.2 形态学鉴定
对上述6株具有外肽酶活性的菌株进行菌株形态观察和显微镜检。如图2所示,通过形态学观察可知,所有菌株在平板上具有典型的芽孢杆菌形态,菌株的菌落较大,呈现出乳白色,均不透明,表面光滑且边缘整齐,用接种环挑取时黏稠拉丝。通过形态学鉴定,菌株的菌体呈杆状或短杆状,大多以单个形式存在,可以初步判断为芽孢杆菌。

图2 外肽酶生产菌株形态学及镜检(40×)观察
Fig.2 Morphological and microscopic (40×) observations of exopeptidase-producing strains
2.3 分子生物学鉴定
菌株的分子生物学鉴定结果如表2所示。
表2 细菌16S rDNA序列分析结果
Table 2 Results of 16S rDNA sequence analysis for bacteria

菌株编号来源鉴定结果同源性/%参考菌株LBH9酸肉B.subtilis99.93OP696631.1MD4X大连虾酱B.subtilis100OR335071.1LBD4X大连虾酱B.subtilis100OR335071.1MH3酸肉B.tequilensis99.93OP435775.1LBH3酸肉B.vallismortis100OP175973.1MX4X鲜虾酱B.subtilis100OR261238.1
本课题组从传统发酵食品酸鱼、酸肉、虾酱中分离得到59株菌株,筛选到6株具有高产外肽酶活性菌株,经16S rDNA分子学鉴定发现均属于芽孢杆菌属(Bacillus sp.),其中包括4株枯草芽孢杆菌(Bacillus subtilis)、1株死谷芽孢杆菌(Bacillus vallismortis)和1株特基拉芽胞杆菌(Bacillus tequilensis)。枯草芽孢杆菌因具有低致病性和强大蛋白质分泌能力以及较强的发酵能力而被广泛应用于各个领域[28]。据报道,WAGNER等[29]将来源于枯草芽孢杆菌的氨肽酶成功在毕赤酵母GS115中表达,重组氨肽酶经过糖基化修饰之后,将其纯化测定酶学性质发现其最适温度为40 ℃,最适pH值为8.5,最适作用底物为精氨酸对硝基苯胺。向军[30]、周慧娉等[31]都从发酵豆豉中筛选出产氨肽酶菌株,鉴定出的菌株均为芽孢杆菌属。因此,本文筛选的菌株与已报道的食源性氨肽酶产生菌株在属水平上相对一致。羧肽酶不仅能广泛应用于多肽的氨基酸测序和制备游离氨基酸过程中,还对去除食品中多肽的苦味起到有利作用。羧肽酶大多数被发现于动物、高等植物以及丝状真菌中。因此,本实验从酸鱼、酸肉、虾酱中筛选出的兼具氨肽酶和羧肽酶生产菌株可用于后续发酵食品中。
2.4 安全性评价
2.4.1 溶血性实验
研究表明溶血素与菌株的致病性和毒性相联系[32],结果如图3所示,将6株菌株和对照组菌株在含有5%(体积分数)哥伦比亚血平板上37 ℃培养24 h,对照组金黄色葡萄球菌ATCC 25923出现β-溶血圈,B.subtilis LBH9、B.tequilensis MH3出现β-溶血圈,其他菌株均无溶血圈产生,说明B.subtilis LBH9、B.tequilensis MH3产生溶血素,因此将B.subtilis LBH9、B.tequilensis MH3排除,其余菌株进行后续的试验研究。
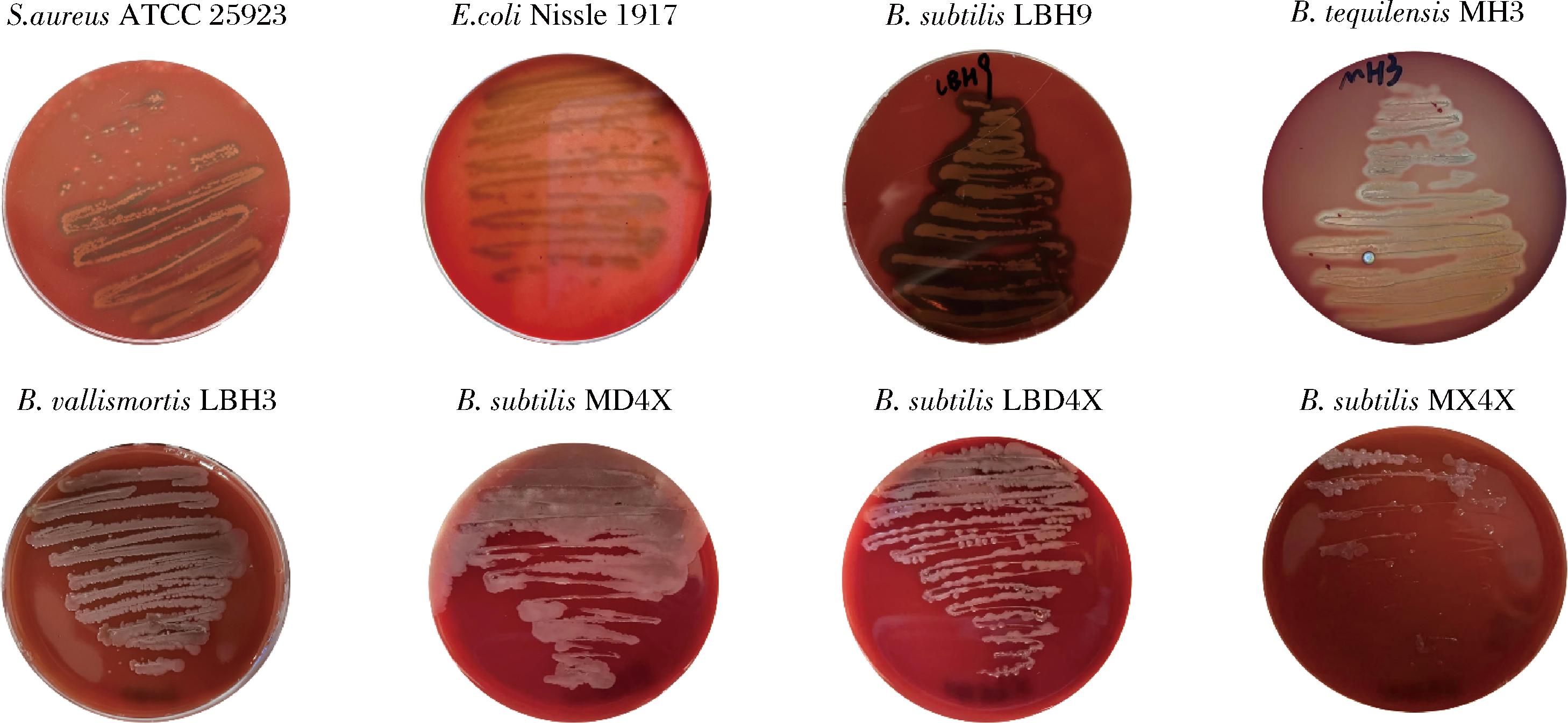
图3 分离菌株的溶血性
Fig.3 Hemolytic properties of isolated strains
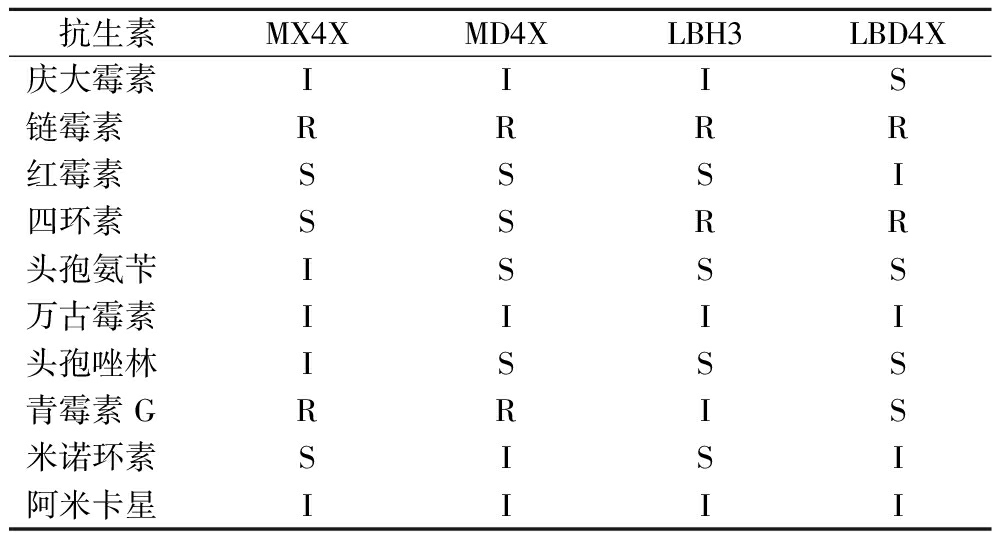
2.4.2 抗生素敏感性实验
随着抗生素的滥用,引起的耐药性问题引发的危害已经越来越严重[33],因此菌株的抗生素敏感试验至关重要。将通过溶血性试验的4株菌株采用药敏纸片法进行抗生素敏感性试验,结果如图4所示。颜色强度对应抑菌圈直径从大(粉色)到小(蓝色),单位为mm。抗生素敏感性评价等级如表3所示。外肽酶生产菌株B.subtilis MD4X对抗生素链霉素和青霉素有耐药性抗性,对4种抗生素庆大霉素、万古霉素、米诺环素、阿米卡星中度敏感,对4种抗生素红霉素、四环素、头孢氨苄、头孢唑林敏感。可以看出该株菌具有抗生素敏感性,说明其安全性较高,可用于后续试验研究。
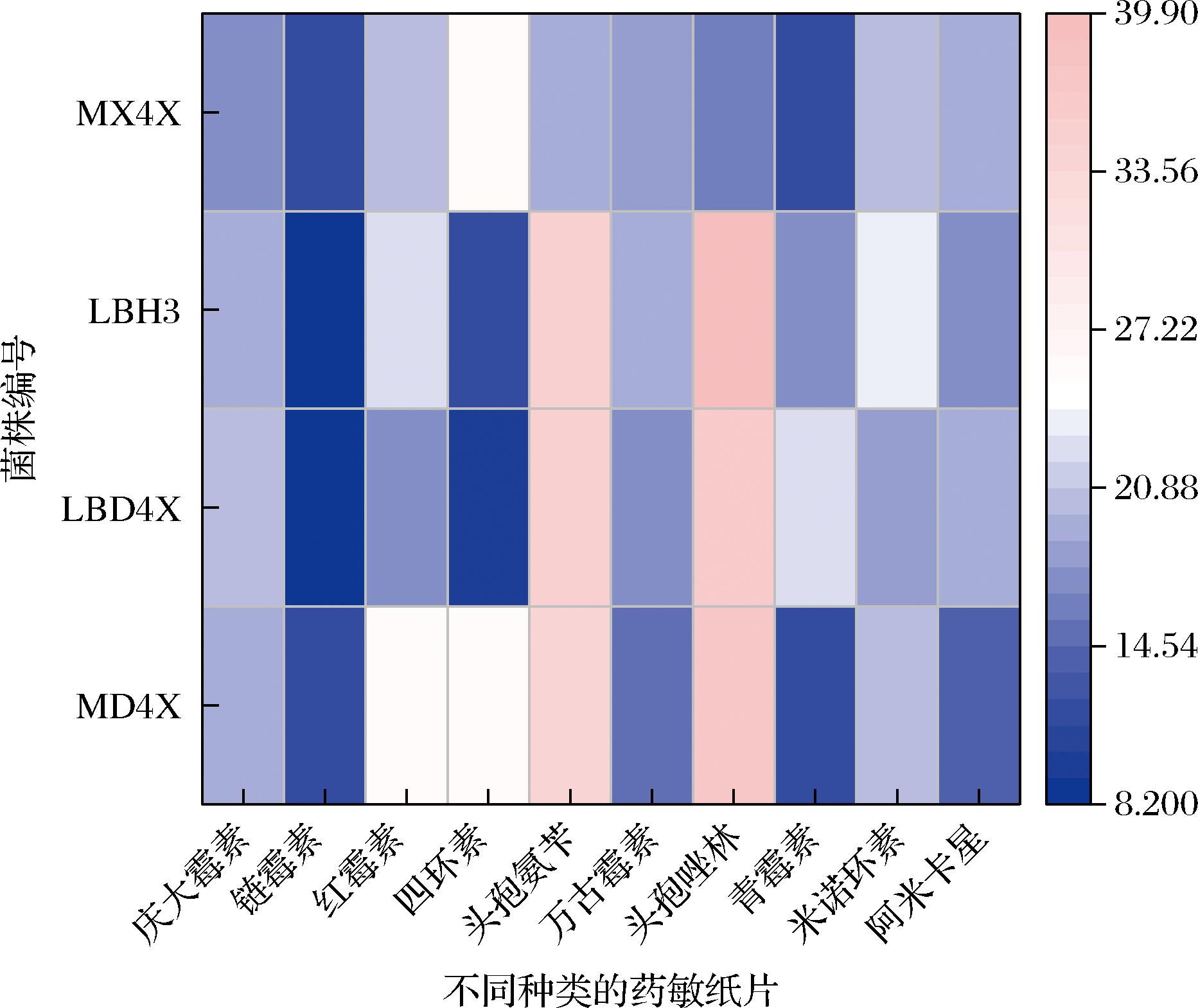
图4 氨肽酶和羧肽酶生产菌株的药敏纸片试验结果
Fig.4 Results of drug-sensitive paper test of aminopeptidase and carboxypeptidase producing strains of bacteria
表3 氨肽酶和羧肽酶生产菌株抗生素敏感性等级评价
Table 3 Evaluation of antibiotic susceptibility classes of aminopeptidase- and carboxypeptidase-producing bacterial strains
抗生素MX4XMD4XLBH3LBD4X庆大霉素IIIS链霉素RRRR红霉素SSSI四环素SSRR头孢氨苄ISSS万古霉素IIII头孢唑林ISSS青霉素GRRIS米诺环素SISI阿米卡星IIII
注:R(≤14 mm)耐药、I(14~20 mm)中度敏感、S(≥20 mm)敏感。
2.5 抗氧化实验
对通过溶血性试验的4株菌进行了DPPH、ABTS阳离子自由基清除试验。从图5-a可以看出,菌株的DPPH自由基清除率范围在20%~30%,B.subtilis MD4X的DPPH自由基清除率为24.32%。如图5-b所示,菌株的ABTS阳离子自由基清除率范围在60%~80%,菌株都表现出较高的ABTS阳离子自由基清除能力,B.subtilis MD4X的ABTS阳离子自由基清除率为77.59%。综上所述,可以初步判断B.subtilis MD4X具有较强的抗氧化活性。
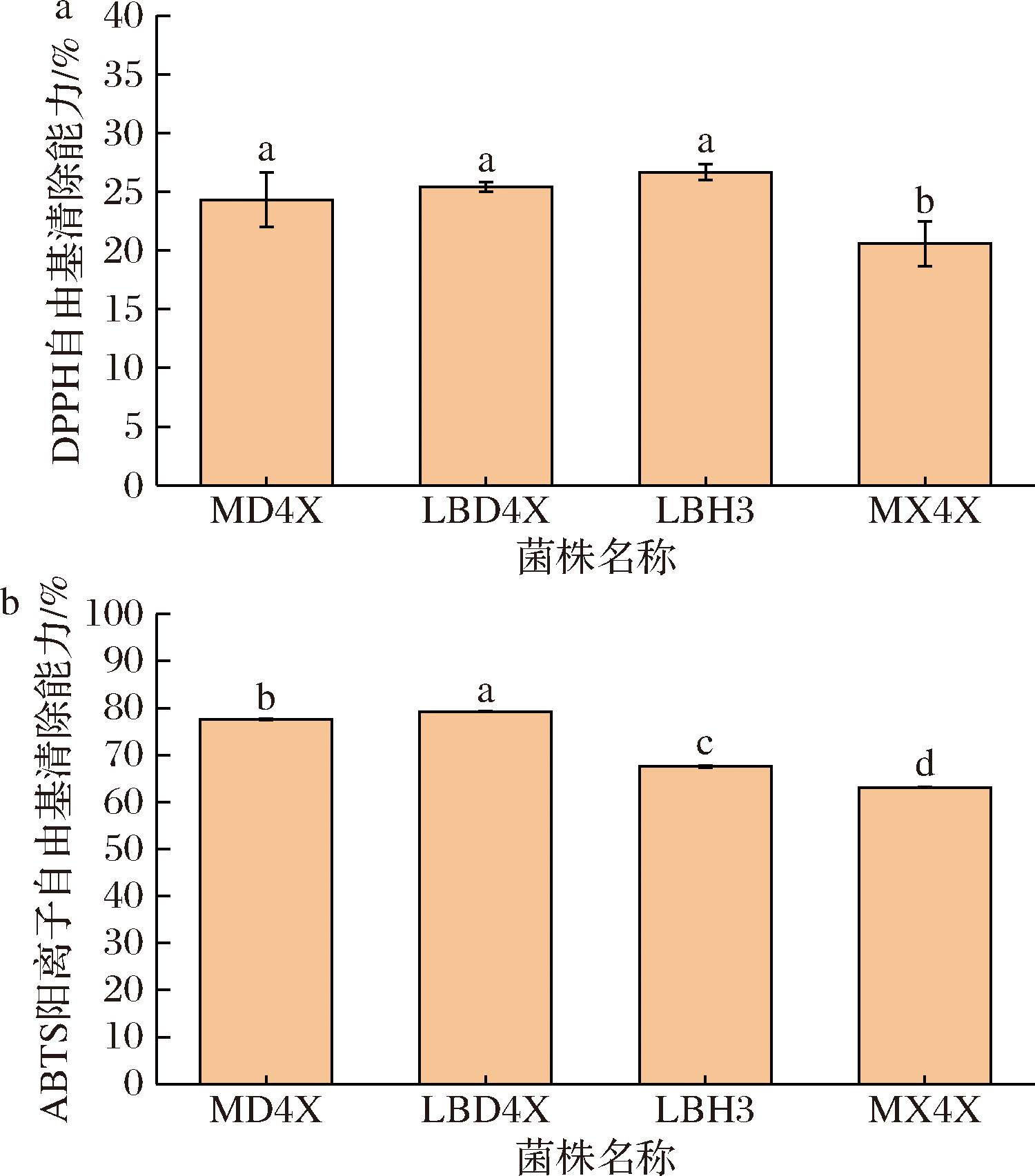
a-DPPH自由基清除能力;b-ABTS阳离子自由基清除能力
图5 氨肽酶和羧肽酶生产菌株抗氧化实验
Fig.5 Antioxidant experiments with aminopeptidase- and carboxypeptidase-producing bacterial strains
注:不同小写字母表示差异显著(P<0.05)。
3 结论
本研究从传统发酵食品酸鱼、酸肉、虾酱中分离得到芽孢杆菌19株、乳酸菌22株、酵母菌18株,经氨肽酶活力和羧肽酶活力测定,形态学及分子生物学鉴定,得到6株兼具2种酶活力且外肽酶活力总和大于240 U/mL的高产外肽酶菌株,其中包括枯草芽孢杆菌(B.subtilis)、死谷芽孢杆菌(B.vallismortis)和特基拉芽胞杆菌(B.tequilensis)3种不同菌属,极大地丰富了酸鱼、酸肉、虾酱中外肽酶生产菌株的菌株库。B.subtilis LBH9具有最高外肽酶活力,但表现出溶血现象,不具备安全性能。而B.subtilis MD4X氨肽酶活性为321.974 U/mL,羧肽酶活性为26.526 U/mL,并且未表现出溶血现象且具有抗生素敏感性,同时具有良好的抗氧化能力,其DPPH自由基、ABTS阳离子自由基清除率分别为24.32%和77.59%。因此,B.subtilis MD4X可作为发酵剂应用于发酵食品中以提高氨基酸含量和改善产品风味。
[1] 周影, 魏启舜, 王琳, 等.地衣芽孢杆菌ZSZ6高效降解羽毛产氨基酸条件的优化[J].江西农业学报, 2020, 32(6):115-120.ZHOU Y, WEI Q S, WANG L, et al.Optimization of conditions for efficient degradation of feather and amino acid production by Bacillus licheniformis ZSZ6[J].Acta Agriculturae Jiangxi, 2020, 32(6):115-120
[2] 丁奇, 马立利, 郎爽, 等.不同调味品中氨基酸态氮、总酸含量的分析及比较研究[J].分析仪器, 2021(3):70-74.DING Q, MA L L, LANG S, et al.Analysis and comparison of amino acid nitrogen and total acid in different condiments[J].Analytical Instrumentation, 2021(3):70-74.
[3] CHEN Z Y, FENG Y Z, CUI C, et al.Effects of koji-making with mixed strains on physicochemical and sensory properties of Chinese-type soy sauce[J].Journal of the Science of Food and Agriculture, 2015, 95(10):2145-2154.
[4] 张素芳, 任晓蕾, 郭宝松, 等.一株暹罗芽孢杆菌及其应用:中国, 202210968288[P].2024-05-07.ZHANG S F, REN X L, GUO B S, et al.A strain of Bacillus siamensis and its application:China, 202210968288[P].2024-05-07.
[5] KINOSHITA S, UDAKA S, SHIMONO M.Studies on the amino acid fermentation.Part 1.Production of L-glutamic acid by various microorganisms[J].Journal of General and Applied Microbiology, 2004, 50(6):331-343.
[6] 王震. 谷氨酸棒状杆菌发酵生产L-精氨酸的溶氧条件优化[J].安徽农业科学, 2019, 47(7):120-123.WANG Z.Optimization of dissolved oxygen conditions for Corynebacterium glutamicum producting L-arginine[J].Journal of Anhui Agricultural Sciences, 2019, 47(7):120-123.
[7] 郭宝松, 张玉姣, 代艺伟, 等.氨肽酶生产菌株Bacillus subtilis LBJ4-5的全基因组测序及基因功能分析[J].食品工业科技, 2024, 45(24):32-41.GUO B S, ZHANG Y J, DAI Y W, et al.Whole-genome sequencing and sequence analysis of an aminopeptidase-producing Bacillus subtilis LBJ4-5[J].Science and Technology of Food Industry, 2024, 45(24):32-41.
[8] 黄甜, 孟勇, 张秀红, 等.氨肽酶在食品中的研究进展[J].现代食品, 2022, 28(19):20-22.HUANG T, MENG Y, ZHANG X H, et al.Research progress of aminopeptidase in food[J].Modern Food, 2022, 28(19):20-22.
[9] 荣雅利, 周婷婷, 施文正, 等.南极磷虾羧肽酶的分离纯化及酶学性质[J].上海海洋大学学报, 2020, 29(4):593-601.RONG Y L, ZHOU T T, SHI W Z, et al.Purification and enzymatic properties of carboxypeptidase from Euphausia superba[J].Journal of Shanghai Ocean University, 2020, 29(4):593-601.
[10] 叶茂, 邓毛程, 林凯旋, 等.黑曲霉固态发酵产羧肽酶条件优化及在酱油酿造中的应用[J].中国酿造, 2021, 40(7):83-88.YE M, DENG M C, LIN K X, et al.Optimization of solid-state fermentation conditions for carboxypeptidase production by Aspergillus niger and its application in soy sauce brewing[J].China Brewing, 2021, 40(7):83-88.
[11] 任晓蕾, 鲍捷, 郭宝松, 等.东北传统大酱中产抗菌物质菌株的筛选及安全性研究[J].中国食品学报, 2024, 24(7):100-110.REN X L, BAO J, GUO B S, et al.Screening and safety study of antimicrobial substance producing strains in traditional soybean paste in Northeast China[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(7):100-110.
[12] 李冬琪. 皖北豆酱中氨肽酶产生菌的分离鉴定及其发酵效果研究[D].合肥:安徽农业大学, 2022.LI D Q.Isolation and identification of aminopeptidase producing bacteria from Wanbei soybean sauce and its fermentation effect[D].Hefei:Anhui Agricultural University, 2022.
[13] 梁二宾. 氨肽酶产生菌的鉴定、酶的分离纯化及性质研究[D].广州:华南理工大学, 2014.LIANG E B.Identification of aminopeptidase-producing strain, purification and characteristics of the enzyme[D].Guangzhou:South China University of Technology, 2014.
[14] 单雨瑶. 羧肽酶Y的酵母异源表达及其在固相酶法肽合成中的应用[D].天津:天津大学, 2018.SHAN Y Y.Expression of recombinant carboxypeptidase Y in Pichia pastoris and its application in kinetically controlled solid phase enzymatic peptide synthesis (SPEPS) of the antioxidant dipeptide Tyr-Ala[D].Tianjin:Tianjin University, 2018.
[15] 马蕾. 沸石咪唑酯骨架化合物负载羧肽酶A降解赭曲霉毒素A的性能研究[D].镇江:江苏科技大学, 2020.MA L.Carboxypeptidase a immobilization with zeolite imidazolate framework for enhancement of ochratoxin a degradation ability[J].Zhenjiang:Jiangsu University of Science and Technology, 2020.
[16] 徐文欢, 吴若菡, 李采婵, 等.传统虾酱中酵母菌分离鉴定及碳源利用特性[J].中国食品学报, 2021, 21(4):303-309.XU W H, WU R H, LI C C, et al.Separation and identification of yeast from traditional shrimp paste and their carbon utilization characteristics[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(4):303-309.
[17] 朱涵明月, 潘远智.蓝花楹内生拮抗细菌筛选及对茎腐病的防治研究[J].南京林业大学学报(自然科学版), 2017, 41(6):61-67.ZHU H M Y, PAN Y Z.Screening for endophytic antagonistic bacterium of Jacaranda acutifolia and biocontrol effect against stem rot[J].Journal of Nanjing Forestry University (Natural Science Edition), 2017, 41(6):61-67.
[18] WANG J X, ZHANG S F, TAN H D, et al.PCR-based strategy for construction of multi-site-saturation mutagenic expression library[J].Journal of Microbiological Methods, 2007, 71(3):225-230.
[19] 赵琳琳. 益生菌大肠杆菌Nissle 1917的新型基因表达平台构建与应用[D].无锡:江南大学, 2022.ZHAO L L.Construction and application of a novel probiotic Escherichia coli Nissle 1917 gene expression platform[D].Wuxi:Jiangnan University, 2022.
[20] HUMPHRIES R, BOBENCHIK A M, HINDLER J A, et al.Overview of changes to the clinical and laboratory standards institute Performance standards for antimicrobial susceptibility testing, M100, 31 st edition[J].Journal of Clinical Microbiology, 2021, 59(12):e0021321.
[21] OSKOUEE S, FEGHHI S A H, SOLEIMANI N.Antibiotic susceptibility variations of Methicillin-resistant Staphylococcus aureus after gamma irradiation[J].International Journal of Radiation Biology, 2020, 96(3):390-393.
[22] RIZZELLO C G, LORUSSO A, RUSSO V, et al.Improving the antioxidant properties of quinoa flour through fermentation with selected autochthonous lactic acid bacteria[J].International Journal of Food Microbiology, 2017, 241:252-261.
[23] MOGHADAM M, SALAMI M, MOHAMMADIAN M, et al.Development of antioxidant edible films based on mung bean protein enriched with pomegranate peel[J].Food Hydrocolloids, 2020, 104:105735.
[24] LI X C, OUYANG X J, CAI R X, et al.3′, 8″-dimerization enhances the antioxidant capacity of flavonoids:Evidence from acacetin and isoginkgetin[J].Molecules, 2019, 24(11):2039.
[25] DOI E, SHIBATA D, MATOBA T.Modified colorimetric ninhydrin methods for peptidase assay[J].Analytical Biochemistry, 1981, 118(1):173-184.
[26] XI H X, TIAN Y P, ZHOU N D, et al.Characterization of an N-glycosylated Bacillus subtilis leucine aminopeptidase expressed in Pichia pastoris[J].Journal of Basic Microbiology, 2015, 55(2):236-246.
[27] 孟广超. 基于ARTP技术筛选高产氨肽酶米曲霉菌株及发酵和酶解条件优化的研究[D].郑州:郑州大学, 2021.MENG G C.Creening of aminopeptidase producing strains based on ARTP technology and optimization of fermentation and enzymolysis conditions[D].Zhengzhou:Zhengzhou University, 2021.
[28] XIANG M J, KANG Q, ZHANG D W.Advances on systems metabolic engineering of Bacillus subtilis as a chassis cell[J].Synthetic and Systems Biotechnology, 2020, 5(4):245-251.
[29] WAGNER F W, RAY L E, AJABNOOR M A, et al.Bacillus subtilis aminopeptidase:Purification, characterization and some enzymatic properties[J].Archives of Biochemistry and Biophysics, 1979, 197(1):63-72.
[30] 向军. 产氨肽酶甲基营养型芽孢杆菌的鉴定、发酵优化及酶学性质研究[D].广州:华南理工大学, 2012.XIANG J.Identification, fermentation optimization and determination of enzymatic characterization for aminopeptidase production of Bacillus methylotrophicus CC[D].Guangzhou:South China University of Technology, 2012.
[31] 周慧娉, 李静, 邓毛程, 等.亮氨酸氨肽酶产生菌筛选与特性的研究[J].中国酿造, 2017, 36(2):98-101.ZHOU H P, LI J, DENG M C, et al.Screening and characteristics of leucine aminopeptidase-producing strain[J].China Brewing, 2017, 36(2):98-101.
[32] JASIN MANSUR F, TAKAHARA S, YAMAMOTO M, et al.Purification and characterization of hemolysin from periodontopathogenic bacterium Eikenella corrodens strain 1073[J].Bioscience, Biotechnology, and Biochemistry, 2017, 81(6):1246-1253.
[33] WRIGHT G D.Antibiotic adjuvants:Rescuing antibiotics from resistance[J].Trends in Microbiology, 2016, 24(11):862-871.