近年来,伴随着生活水平的提升,由于高糖高热量饮食导致罹患过度肥胖、高血糖血脂等疾病的人数急速增加,为了能让人们在享受美味的同时保持健康,低热量代糖引起了学界的广泛关注。作为一种稀少糖,D-阿洛酮糖具有蔗糖70%的甜度,但其热量却仅为蔗糖的10%,并且口感与蔗糖极为类似,是理想的蔗糖替代品[1]。与此同时,D-阿洛酮糖还具有多种益生功能:减少小肠对葡萄糖的吸收,抑制血糖水平的升高[2];减少体内脂肪合成、促进脂肪酸氧化、增加能量消耗,从而控制体重[3-4]。因此,D-阿洛酮糖作为低热量功能糖在食品、医药等行业具有极强的应用潜力[5-6]。
D-阿洛酮糖3-差向异构酶(D-allulose 3-epimerase,DAE)[7-8]是目前工业制备D-阿洛酮糖的主要用酶,能够以D-果糖为底物生成D-阿洛酮糖。然而,DAE仍面临诸多性能上的挑战,例如催化活力不足、热稳定性不佳等[9]。因此,亟需对DAE进行分子改造以提升其催化活性及稳定性,以满足旺盛的市场需求。定向进化作为一种蛋白质工程改造的有效策略,在提升酶性能甚至获得新功能方面具有显著优势[10]。有效的定向进化方法往往离不开高通量筛选方法的应用,基于转录因子的生物传感器可将输入信号转换为易于检测的输出信号(如荧光、抗生素耐药性或生长表型),可用于感知小分子以及代谢产物的产量[11-12],为加速目标突变体的筛选提供了一种前景广阔的解决方案[13]。例如,XU等[14]利用转录因子ArgR和sacB作为输出报告模块,设计了精氨酸响应生物传感器,该筛选系统通过监测细胞内依赖于精氨酸的细胞生长表型来筛选高产精氨酸的菌株。近年来,建立在转录因子PsiR及其同源启动子pPsiA上的D-阿洛酮糖特异型生物传感器将绿色荧光蛋白基因的表达与大肠杆菌中DAE活力相关联起来[15-16]。将该生物传感器与荧光激活细胞分选技术(fluorescence-activated cell sorting,FACS)相结合,能够将筛选通量进一步提高至108个细胞/h[17]。
本研究中,为提高Clostridium cellulolyticum H10来源的DAE(CcDAE)的比活力,通过易错PCR(error-prone PCR,epPCR)构建了CcDAE的突变体文库,并通过优化D-阿洛酮糖特异型生物传感器及筛选条件,对构建的突变体文库进行超高通量初筛。同时,结合96孔微孔板筛选对初筛获得的潜在优势突变体进行了高通量复筛。通过以上建库、初筛、复筛的一系列步骤以期获得比活力高的DAE突变体,为D-阿洛酮糖的工业化制备提供技术保障。
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
克隆宿主Escherichia coli JM109,表达宿主E.coli BL21(DE3),重组菌E.coli BL21 (DE3)/pET-20b (+)-ccdae以及质粒pRSFDuet-psir-PpsiA-mEmerald-T7 term均来源自本实验室。
1.1.2 试剂及培养基
4-羟乙基哌嗪乙磺酸[4-(2-Hydroxyethyl) piperazine-1-ethanesulfonic acid),HEPES],上海维塔化学试剂有限公司;CoCl2、NaH2PO4、Na2HPO4、NaCl、NaOH、咪唑、乙腈,国药集团;镍柱填料,美国GE公司;D-阿洛酮糖液相标品,日本GlyCarbo公司;高保真DNA聚合酶、快速PCR酶、一步克隆试剂盒和限制性内切酶,宝日医生物技术(北京)有限公司;卡那霉素(kanamycin,Kana)、四环素(tetracycline,Tet),上海生工生物工程股份有限公司;质粒小提试剂盒、普通琼脂糖凝胶DNA回收试剂盒,天根生化有限公司。
HEPES缓冲液:20.0 mmol/L HEPES,0.1 mmol/L CoCl2,pH 7.5。
纯化缓冲A液:50.0 mmol/L NaH2PO4,50.0 mmol/L Na2HPO4,0.5 mol/L NaCl,用NaOH调节pH至7.5。
纯化缓冲B液:300.0 mmol/L咪唑,用纯化缓冲A液溶解,此为B液母液。洗脱过程中再以不同体积比例与纯化缓冲A液混合使用,此为纯化缓冲B液。
LB培养基(g/L):5.0酵母粉,10.0 NaCl,10.0胰蛋白胨。对于固体培养基,需在液体LB培养基中加入15.0琼脂粉。
TB培养基(g/L):5.0甘油,12.0胰蛋白胨,24.0酵母粉,2.3 KH2PO4,12.54 K2HPO4。
1.2 仪器与设备
Waters e2695高效液相色谱,上海沃特世科技有限公司;ProFlex型PCR仪,Thermo Fisher Scientific;高速冷冻离心机,美国Beckman Coulter公司;水浴恒温振荡器,上海知楚仪器有限公司;紫外可见分光光度计,北京普析通用仪器公司;多功能凝胶图像分析系统,上海天能科技公司;荧光酶标仪Spark,瑞士Tecan公司; FACSArica Ⅲ流式细胞仪,美国BD公司。
1.3 实验方法
1.3.1 筛选质粒的构建
在实验室前期构建的PsiR生物传感器基础上,使用引物DAE-正向引物/DAE-反向引物(5′TAG-AGAAAAGAATTCAAAAGATCTAAAGAGGAGAAAGG-ATC3′/5′TCCTTTCTCCTCTTTAGATCTTTTGAATTCT-TTTCTCTATC3′)扩增ccdae片段,并将上述扩增片段通过Overlap PCR方法[18]再进一步连接到pRSFDuet-psir-PpsiA-mEmerald-T7 term上,获得pRSFDuet-psir-PpsiA-mEmerald-ccdae-T7 term。
1.3.2 DAE突变文库的构建及筛选
利用引物ep-正向引物/ep-反向引物(5′TCTAAAGAGGAGAAAGGATC3′/5′TGGTGGTGGTGGTGCT-CGAG3′),通过epPCR[19]构建CcDAE突变体文库,构建完成后将文库转入E.coli BL21 (DE3),然后将转化后的重组菌接种至含20 mmol/L D-果糖以及30 μg/mL Kana的LB培养基中过夜培养10~12 h,离心收集适量菌体,用鞘液(PBS,pH 7.4)重悬菌体并稀释OD600值至约0.3。最后,使用流式细胞仪对样本进行荧光分析和分拣。将荧光值高于野生型的所有细胞中,荧光值大小约为前0.5%的细胞通过流式分选仪分拣出来,收集到200 μL LB培养基中,37 ℃、200 r/min培养2 h,再涂布至含有30 μg/mL Kana的LB固体培养基上,置于37 ℃恒温培养10~12 h。然后挑取单菌落到含有200 μL LB培养基(30 μg/mL Kana)的96孔微孔板中,并按照BOSSHART等[20]建立的微孔板筛选方法对其进行复筛,通过核糖醇脱氢酶(ribitol dehydrogenase,KpRD)催化D-阿洛酮糖还原成蒜糖醇的过程中伴随着NADH的氧化,通过监测340 nm波长下吸光值变化以检测D-阿洛酮糖产量。
1.3.3 DAE的酶活力测定
1.3.3.1 DAE的摇瓶发酵
挑取单菌落或从甘油管中取20 μL接种至10 mL含相应抗生素的LB培养基中,在37 ℃、200 r/min下过夜培养10~12 h,然后取2.5 mL上述菌液转接至50 mL含相应抗生素TB发酵培养基中,在37 ℃、200 r/min下培养1.5~2 h后,加入诱导剂(0.2 μg/mL Tet),随后将发酵液转移至25 ℃,200 r/min下继续发酵24 h。结束发酵的发酵液,取适量菌液离心获得菌体,使用HEPES缓冲液重悬菌体,获得OD600为5的重悬液,然后通过高压匀浆机破碎细胞,再次离心并分离破壁上清液,该破壁上清液为粗酶液,计算如公式(1)所示:
粗酶总活力![]()
(1)
1.3.3.2 DAE纯化
将发酵液离心获得菌体,使用HEPES缓冲液重悬菌体,并使用高压匀浆机破碎细胞,再次经过离心获得破壁上清液,使用0.22 μm水相滤膜过滤,将过滤后的酶液慢速通过镍离子亲和层析柱,经优化洗脱浓度,为了洗脱杂蛋白,先用含有90 mmol/L咪唑的纯化缓冲液冲洗约10个柱体积,再为了洗脱目的蛋白,使用含有120 mmol/L咪唑的纯化缓冲B液再冲洗10个柱体积。收集10个柱体积的洗脱目的蛋白的缓冲液,再利用截留分子质量为10 kDa的超滤管对上述洗脱液进行浓缩,浓缩过程中加入HEPES缓冲液,以实现对纯化缓冲液的置换。浓缩及置换完成后,为了测量蛋白比活力,使用Bradford蛋白浓度测定试剂盒来测定纯化酶液的蛋白浓度。
酶活力测定:底物为800 μL 100 g/L D-果糖(20 mmol/L HEPES,pH 7.5,0.1 mmol/L Co2+),取200 μL酶液与底物混合后于60 ℃下精准反应10 min后,再放入持续沸腾的热水中水浴10 min以终止反应。底物D-果糖需提前在反应温度下预热10 min。
高效液相色谱检测条件:将煮沸的反应液离心取上清液,用纯水进行适当稀释,再次离心后,使用0.22 μm的水系滤膜过滤除杂。Waters e2695高效液相色谱(示差检测器),ShodexTM AsahipakTM NH2P-50 4E色谱柱,柱温和检测温度均为35 ℃,流动相为V(乙腈)∶V(水)=75∶25,流速0.8 mL/min。
酶活力单位定义:反应条件为60 ℃与pH 7.5时,每分钟内生成1 μmol D-阿洛酮糖所用的酶量为1个酶活力单位(U)。
1.3.3.3 DAE的最适温度、最适pH和热稳定性测定
为了测定CcDAE野生型(wild type,WT)和突变体催化的最适温度,分别在50、55、60、65、70、75 ℃下测定酶活力,除反应温度外的其余反应条件均与1.3.3.2节中DAE活力测定方法保持一致。将最适温度下的比酶活力定义为100%,通过计算不同温度条件下的相对酶活力,确定WT与突变体的最适温度。
为了测定WT与突变体催化的最适pH,在pH不同(6.5、7.0、7.5、8.0、8.5)的HEPES缓冲液中,在对应的最适反应温度下测定了WT和突变体的酶活力,其余反应条件和操作均与1.3.3.2节中DAE活力测定方法保持一致。将最适pH下的比酶活力定义为100%,通过计算对应条件下的相对酶活力变化,确定WT与突变体的最适pH。
为测定WT以及突变体催化的热稳定性,分别将酶液置于60 ℃的水浴中长时间孵育,定点取样并按1.3.3.2节酶活力测定方法测定残余酶活力。
1.3.3.4 DAE的动力学参数测定
为了测定CcDAE WT及其突变体的动力学参数,在60 ℃、pH 7.5条件下,以不同浓度的D-果糖(8~2 400 mmol/L)为底物,精确反应10 min后,测量产生的D-阿洛酮糖量。根据结果计算了不同条件下酶的比活力,并在数据处理软件中根据Michaelis-Menten方程进行了非线性拟合,根据拟合参数计算反应动力学参数。
1.3.3.5 分子动力学模拟
以CcDAEase (PDB ID:3VNK)为模板通过AlphaFold3同源建模。以ff14SB作为蛋白质力场,TIP3P作为水分子力场,在330 K下用AMBER 16对CcDAEase 野生型及突变体进行分子动力学模拟,共模拟80 ns。使用cpptraj对模拟轨迹进行分析,并分别统计野生型与不同突变体的均方根波动(root mean square fluctuation,RMSF)值以及关键氨基酸残基之间的距离。
2 结果与分析
2.1 PsiR生物传感器和DAE突变体文库的构建
在实验室前期研究中,根据已有的PsiR生物传感器原理[15-16],通过组装psir、PpsiA等元件获得了pRSFDuet-psir-PpsiA-mEmerald质粒(图1)。后来出于将该生物传感器应用在DAE定向进化中的需求,本研究将ccdae片段同样组装至该质粒上,获得了质粒pRSFDuet-psir-PpsiA-mEmerald-ccdae。在采用epPCR构建DAE突变体文库的过程中,通过优化epPCR体系中的Mn2+浓度、更换DNA聚合酶等方法构建流程,成功构建了突变数量多、类型丰富的DAE突变体文库,以便进行后续筛选。
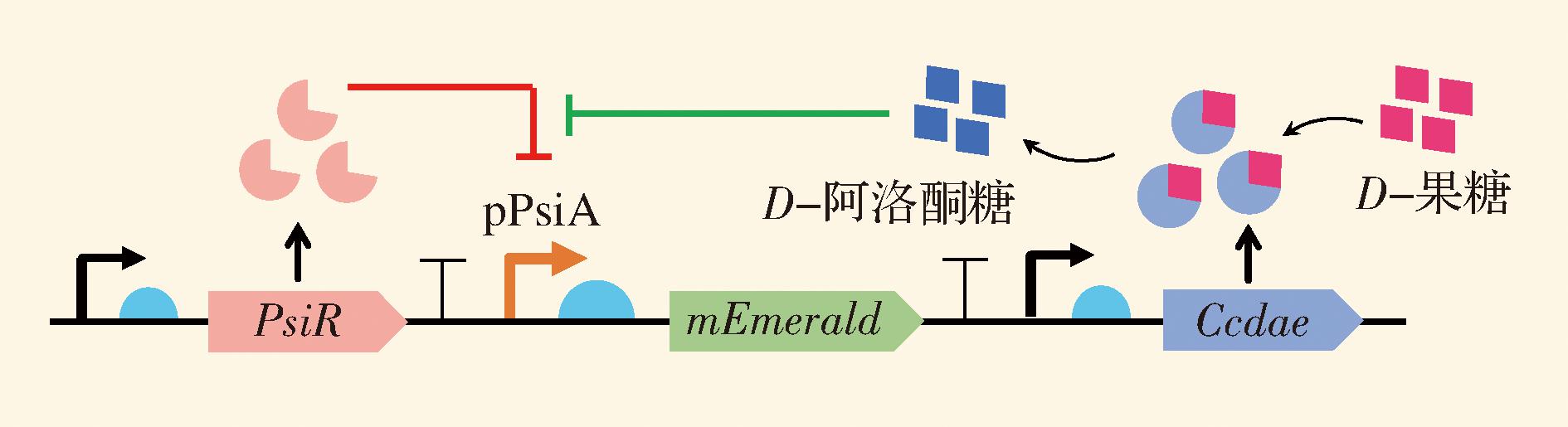
图1 基于PsiR生物传感器的筛选质粒示意图
Fig.1 Schematic diagram of the screening plasmid based on PsiR biosensor
2.2 DAE优势突变体的筛选
DAE突变体文库构建完成后,先将FACS技术和生物传感器相结合,由于理论上荧光亮度越高则酶活力越高,所以通过荧光亮度分析了约40 000个克隆,并分选收集荧光值高于WT的所有细胞中,荧光值大小约为前0.5%的细胞以作为潜在优势突变体,最终共收集到了约200个细胞。潜在优势突变体文库经1.3.2节的方法孵育培养后涂布至琼脂平板上,再将单菌落挑至96孔微孔板中培养,通过BOSSHART等[20]建立的微孔板筛选方法对其进行高通量复筛。在复筛结束后,选取在340 nm波长下ΔA值大于WT的前3个突变体进行摇瓶发酵,经过摇瓶发酵测得粗酶液活力比WT提高了的最优突变体,经测序,确定其突变位点为F147L(表1)。在确定位点后,为了更好更具针对性地对该突变体进行表征,将该突变体基因与载体pET-20b(+)重组构建表达载体pET-20b-ccdae F147L,并将其转入E.coli BL21 (DE3)中以便对其进行酶学性质表征。
表1 DAE WT和突变体F147L的粗酶液活力
Table 1 Rough enzyme activity of DAE WT and mutant F147L
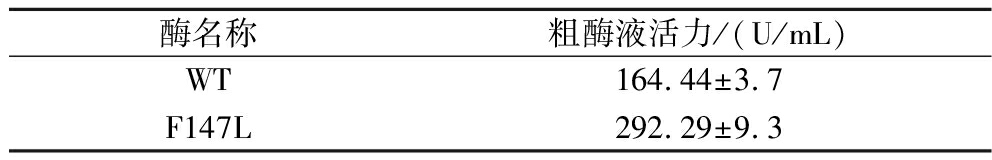
酶名称粗酶液活力/(U/mL)WT164.44±3.7F147L292.29±9.3
2.3 DAE WT和突变体F147L的酶学性质测定
在对WT和突变体F147L进行重组表达后,通过优化组氨酸亲和层析过程中洗脱蛋白所用的咪唑浓度以及纯化流程,对WT和突变体F147L进行了纯化并成功获得了纯度较高的目的蛋白(图2)。
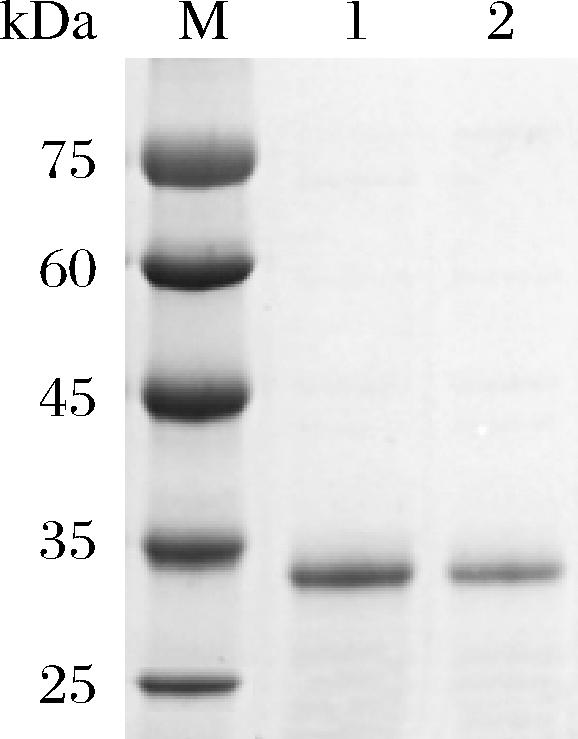
M-Marker;1-DAE WT纯化蛋白;2-F147L纯化蛋白。
图2 DAE WT和突变体F147L纯化蛋白的SDS-PAGE结果(稀释15倍后)
Fig.2 SDS-PAGE of purified DAE WT and mutant F147L (diluted 15 times)
按照1.3.3.3节的方法对WT和突变体F147L进行了最适温度和最适pH的测定,并绘制了相对酶活力-温度和相对酶活力-pH曲线。如图3-a和图3-b所示,发现WT的最适温度为65 ℃,突变体F147L的最适温度为60 ℃;而WT和突变体F147L的最适pH均为7.5。
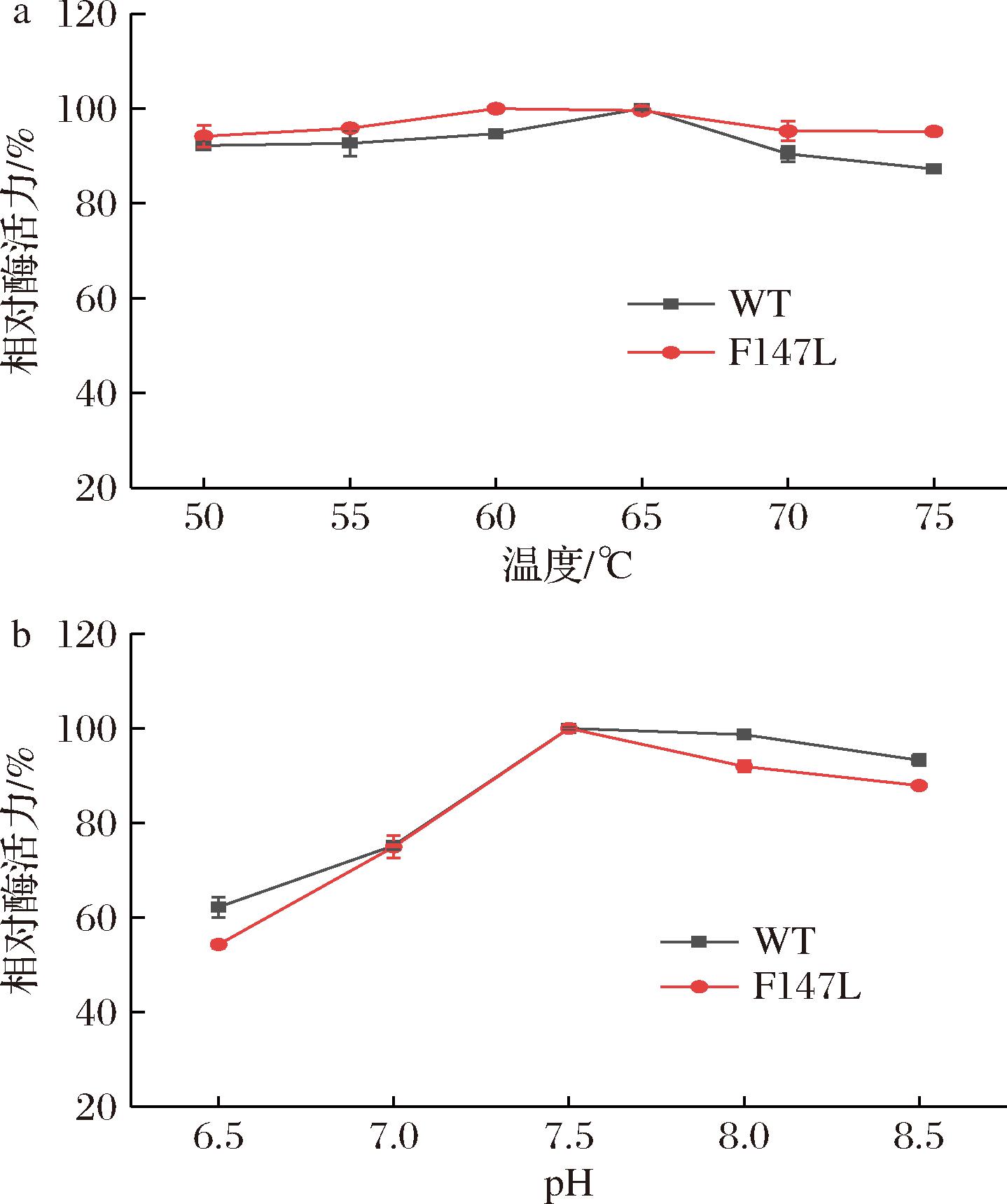
a-最适温度;b-最适pH
图3 DAE WT和突变体F147L的最适温度与最适pH曲线
Fig.3 Optimum temperature and optimum pH curves of DAE WT and mutant F147L
分别在WT和突变体F147L的最适温度下比较二者的比活力,如表2所示,在60 ℃和65 ℃下,突变体F147L的比活力分别比WT提高了26.21%和19.03%,均表现出了明显的比活力提高。
表2 60 ℃和65 ℃下DAE WT和突变体F147L的比活力
Table 2 Specific activity of DAE WT and mutant F147L
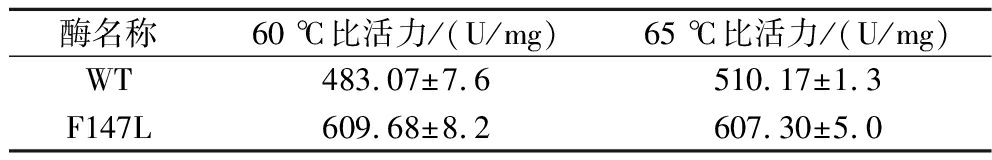
酶名称60 ℃比活力/(U/mg)65 ℃比活力/(U/mg)WT483.07±7.6510.17±1.3F147L609.68±8.2607.30±5.0
同时,对WT和突变体F147L进行了热稳定性的表征。将2种酶分别置于60 ℃下持续孵育,每隔固定时间取样测量残余酶活力,并绘制了相对酶活力-时间曲线。如图4所示,发现突变体F147L的热稳定性略低于WT,推测原因可能为F147L位点的突变影响了酶整体构象的稳定,从而导致了热稳定性的下降。
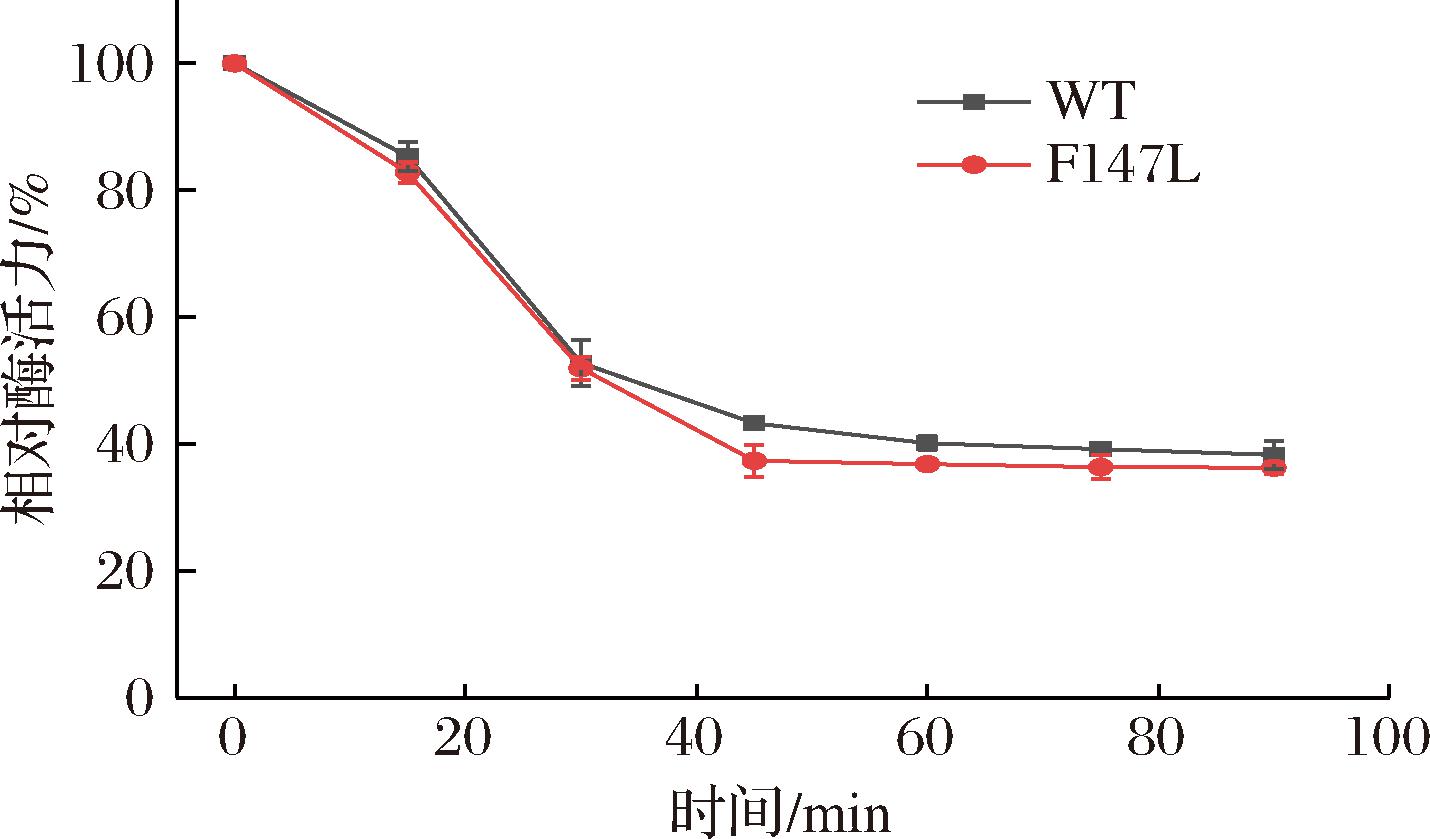
图4 DAE WT和突变体F147L的热稳定性曲线
Fig.4 Thermal stability curves of DAE WT and mutant F147L
为了进一步表征突变体的催化活力变化,通过测定不同底物浓度下的酶活力,在数据处理时拟合Michaelis-Menten方程得到了CcDAE WT和F147L的动力学参数,结果见表3。和WT相比,F147L对底物的亲和力提升,Km值为174.09 mmol/L;与此同时F147L的kcat值为3 641.48 min-1,高于WT,证实突变体F147L的催化效率得到了提升。
表3 DAE WT和突变体F147L的动力学参数
Table 3 Kinetic parameters of DAE WT and mutant F147L

酶名称Km/(mmol/L)kcat/(min-1)kcat/Km/[L/(mmol·min)]WT208.19±5.73 108.08±18.214.93±1.8F147L174.09±12.33 641.48±15.720.92±1.5
2.4 DAE突变体F147L的构效关系分析
为解析优势突变体的构效关系,对其进行分子动力学分析。RMSF值表示的是一定时间内蛋白结构中各原子相对于平均位置的变化幅度,即原子灵活度,在一定程度上可以表征蛋白质稳定性的变化[21]。如图5所示,可以发现WT的α4-β4间的loop区域上111~121位残基(红框内)RMSF值较高,说明该区域具有较大的柔性,而F147L的引入赋予了该区域更强的刚性,同时也给246~261位残基区域(橙框内)带来了更大的波动,使该区域柔性增大。
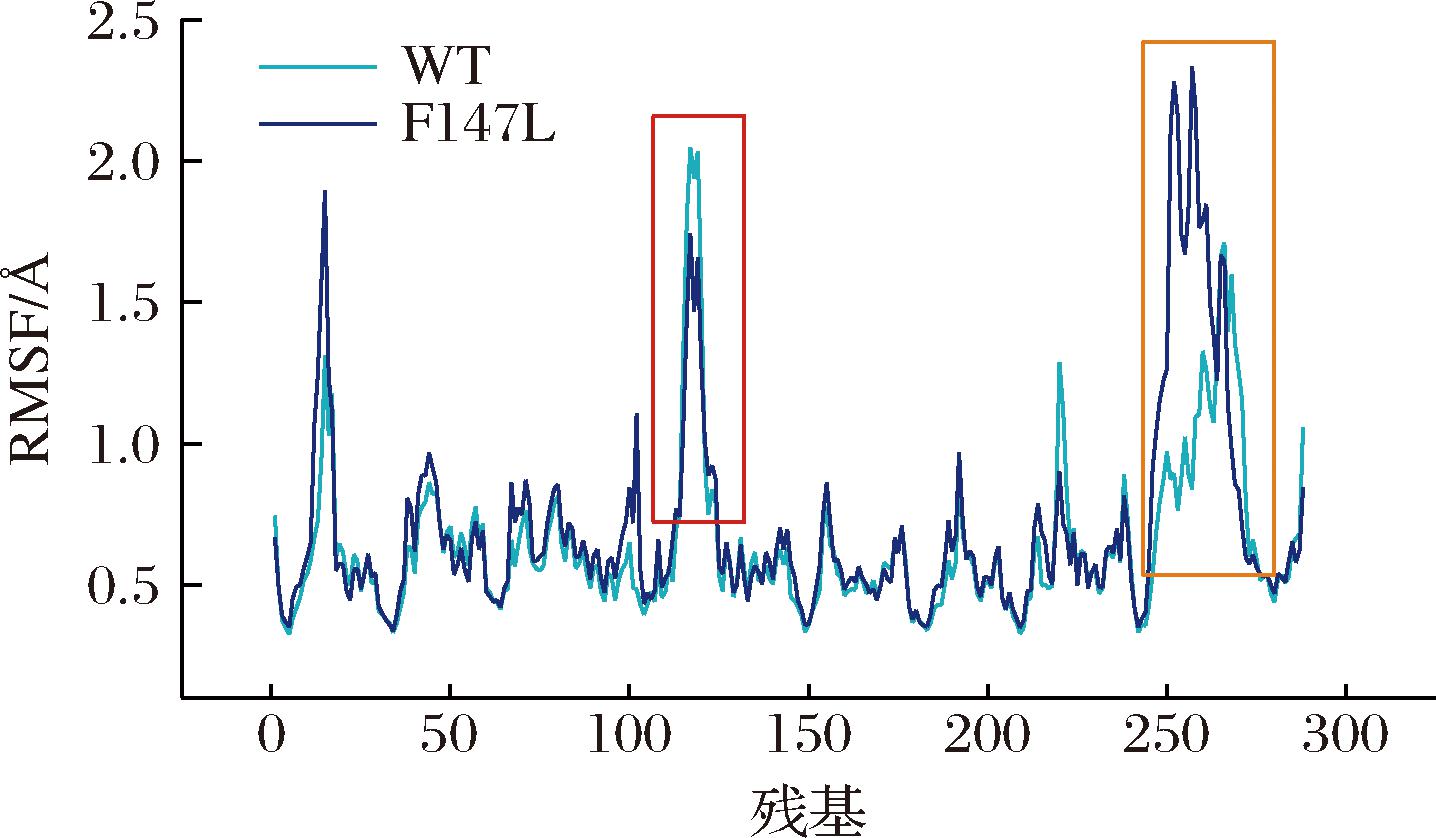
图5 DAE WT和突变体F147L的RMSF值分析图
Fig.5 RMSF analysis of residues of DAE WT and mutant F147L
对结合蛋白晶体结构(图6)进行进一步分析,发现106~121位残基(绿色)和246~261位残基(橙色)共同参与构成结合口袋入口。其中112位点的色氨酸与底物间存在疏水作用力,是底物结合位点之一。推测147位点的苯丙氨酸突变为亮氨酸,导致与147位点在空间上相邻的106~121位残基区域的空间位阻减小,进而降低了111和121位残基区域的氨基酸骨架波动,底物更易与112位点结合,F147L的Km值降低也印证了这一推测,进而使催化效率得到提高。考虑到106~121位残基和246~261位残基都位于结合口袋入口,进一步推测是106~121位区域刚性增强在一定程度上增加了246~261位区域的氨基酸骨架波动性,因此导致了F147L的热稳定性相比WT略有下降。在后续研究中,可以在突变体F147L的基础上进行进一步迭代优势突变位点,获得在酶活力提高的同时,热稳定性同样也能得到保障的优质CcDAE突变体。

图6 DAE WT和突变体F147L的结构分析图
Fig.6 Structural analysis of DAE WT and mutant F147L
注:DAE的106~121位残基区域(绿色)和246~261位残基区域(橙色)共同参与构成结合口袋入口;W112和D-果糖间形成疏水作用力(红色)。
3 结论
本研究成功将转录因子PsiR构成的D-阿洛酮糖特异型生物传感器应用于CcDAE的定向进化中。在建立突变体文库后,通过一轮FACS以及一轮96孔微孔板筛选,最终获得了在60 ℃下比活力相较CcDAE WT提高了26.21%的优势突变体F147L。本研究展示了转录因子型生物传感器在酶定向进化中的重要作用,为D-阿洛酮糖的工业制备提供了技术保障,也为DAE的分子改造提供了新思路。
[1] CHEN D, CHEN J J, LIU X Y, et al.Biochemical identification of a hyperthermostable L-ribulose 3-epimerase from Labedella endophytica and its application for D-allulose bioconversion[J].International Journal of Biological Macromolecules, 2021, 189:214-222.
[2] KIMURA T, KANASAKI A, HAYASHI N, et al.D-Allulose enhances postprandial fat oxidation in healthy humans[J].Nutrition, 2017, 43-44:16-20.
[3] OCHIAI M, ONISHI K, YAMADA T, et al.D-Psicose increases energy expenditure and decreases body fat accumulation in rats fed a high-sucrose diet[J].International Journal of Food Sciences and Nutrition, 2014, 65(2):245-250.
[4] NAGATA Y, KANASAKI A, TAMARU S, et al.D-Psicose, an epimer of D-fructose, favorably alters lipid metabolism in Sprague-Dawley rats[J].Journal of Agricultural and Food Chemistry, 2015, 63(12):3168-3176.
[5] SUNA S, YAMAGUCHI F, KIMURA S, et al.Preventive effect of D-psicose, one of rare ketohexoses, on di-(2-ethylhexyl) phthalate (DEHP)-induced testicular injury in rat[J].Toxicology Letters, 2007, 173(2):107-117.
[6] KIM S E, KIM S J, KIM H J, et al.D-Psicose, a sugar substitute, suppresses body fat deposition by altering networks of inflammatory response and lipid metabolism in C57BL/6J-ob/ob mice[J].Journal of Functional Foods, 2017, 28:265-274.
[7] IZUMORI K, KHAN A R, OKAYA H, et al.A new enzyme, D-ketohexose 3-epimerase, from Pseudomonas sp.ST-24[J].Bioscience, Biotechnology, and Biochemistry, 1993, 57(6):1037-1039.
[8] KIM H J, HYUN E K, KIM Y S, et al.Characterization of an Agrobacterium tumefaciens D-psicose 3-epimerase that converts D-fructose to D-psicose[J].Applied and Environmental Microbiology, 2006, 72(2):981-985.
[9] ZHANG W, WEI M J, SUN X X, et al.Fine-tuning of carbon flux and artificial promoters in Bacillus subtilis enables high-level biosynthesis of D-allulose[J].Journal of Agricultural and Food Chemistry, 2022, 70(43):13935-13944.
[10] HENDEL S J, SHOULDERS M D.Directed evolution in mammalian cells[J].Nature Methods, 2021, 18(4):346-357.
[11] MAHR R, VON BOESELAGER R F, WIECHERT J, et al.Screening of an Escherichia coli promoter library for a phenylalanine biosensor[J].Applied Microbiology and Biotechnology, 2016, 100(15):6739-6753.
[12] ROGERS J K, TAYLOR N D, CHURCH G M.Biosensor-based engineering of biosynthetic pathways[J].Current Opinion in Biotechnology, 2016, 42:84-91.
[13] DELLA CORTE D, VAN BEEK H L, SYBERG F, et al.Engineering and application of a biosensor with focused ligand specificity[J].Nature Communications, 2020, 11:4851.
[14] XU M J, LIU P P, CHEN J M, et al.Development of a novel biosensor-driven mutation and selection system via in situ growth of Corynebacterium crenatum for the production of L-arginine[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:175.
[15] ARMETTA J, BERTHOME R, CROS A, et al.Biosensor-based enzyme engineering approach applied to psicose biosynthesis[J].Synthetic Biology, 2019, 4(1):ysz028.
[16] LI C, GAO X, QI H B, et al.Substantial improvement of an epimerase for the synthesis of D-allulose by biosensor-based high-throughput microdroplet screening[J].Angewandte Chemie International Edition, 2023, 62(10):e202216721.
[17] LIU Z Z, CHEN S, WU J.Advances in ultrahigh-throughput screening technologies for protein evolution[J].Trends in Biotechnology, 2023, 41(9):1168-1181.
[18] BRYKSIN A V, MATSUMURA I.Overlap extension PCR cloning:A simple and reliable way to create recombinant plasmids[J].BioTechniques, 2010, 48(6):463-465.
[19] WILSON D S, KEEFE A D.Random mutagenesis by PCR[J].Current Protocols in Molecular Biology, 2001, Chapter 8:Unit 8.3.
[20] BOSSHART A, HEE C S, BECHTOLD M, et al.Directed divergent evolution of a thermostable D-tagatose epimerase towards improved activity for two hexose substrates[J].ChemBioChem, 2015, 16(4):592-601.
[21] LIU Z M, FU X D, YUAN M X, et al.Surface charged amino acid-based strategy for rational engineering of kinetic stability and specific activity of enzymes:Linking experiments with computational modeling[J].International Journal of Biological Macromolecules, 2021, 182:228-236.