羧肽酶是一类水解多肽链的羧基端氨基酸残基的外肽酶,在细菌、真菌、植物和动物中均有发现[1-2]。按其活性中心的特点可以分成丝氨酸羧肽酶、天冬氨酸羧肽酶以及半胱氨酸羧肽酶。羧肽酶Y是一种丝氨酸羧肽酶,它的活性中心由丝氨酸、天冬氨酸和组氨酸(Ser-Asp-His)组成[3]。羧肽酶Y不仅底物广泛,而且具有其他羧肽酶不具备的从多肽链羧基端降解脯氨酸残基的能力[4]。因此,它在多肽氨基酸分析和食品脱苦方面有着广泛的应用[5-6]。目前研究较多的羧肽酶Y来源于酿酒酵母[1],而对其他物种,特别是丝状真菌来源的羧肽酶Y的研究相对较少。
雅致放射毛霉(Actinomucor elegans)应用于中国传统食品腐乳的制作过程中,经长期高蛋白环境的驯化,具有了强大胞外蛋白分泌能力。在腐乳发酵过程中,其丰富的胞外蛋白酶系将豆腐中的蛋白质分解成短肽和氨基酸[7]。大豆蛋白的水解物常常具有苦味,有研究发现,雅致放射毛霉中的羧肽酶可以应用于大豆蛋白水解物的脱苦[7-8]。
毕赤酵母表达系统具有操作简单、能够进行高密度发酵、没有内毒素、能够进行翻译后修饰等诸多优点[9],使其广泛应用于来源真核基因的异源表达。外源蛋白在毕赤酵母中的表达量受到多种因素的影响,如信号肽、启动子、外源基因的拷贝数以及外源蛋白的分泌途径等[10-13]。有研究者通过提高抗生素浓度的筛选方法[14],筛选得到不同拷贝数的重组菌株,从中得到高效表达菌株,实现了外源蛋白的高效分泌表达,还有研究者通过在重组菌中共表达分泌促进因子binding protein(BIP)、protein disulfide isomerase (PDI)、endoplasmic oxidoreductin 1(ERO1),将蛋白的分泌量提升到原来的1.9倍、2.2倍和2.3倍[15]。
前期研究中,在雅致放射毛霉的基因组中克隆了一种属于羧肽酶Y家族新型的蛋白酶的基因,进行密码子优化后,在毕赤酵母GS115中进行异源表达[17]。本研究构建含有不同启动子的重组菌,通过提高抗生素浓度的筛选方法,获得高效分泌表达菌株。
1 材料与方法
1.1 菌种、质粒和培养基
Escherichia coli JM109购自Novagen公司;表达宿主Pichia Pastoris GS115、质粒pPICZα、pGAPZα购自美国Invitrogen公司;Actinomucor elegans PEP001由本实验室保存;pPIC9K-proCPY质粒由安徽通用生物公司合成;pGAPZ-BIP、pGAPZ-PDI、pGAPZ-ERO1由江南大学金坚教授馈赠。
大肠杆菌生长培养基为low salt(LB)培养基:溶解5 g酵母提取物,10 g蛋白胨,5 g NaCl于900 mL水中,完全溶解后定容到1 L(配置固体培养基时加入 20 g/L琼脂粉);毕赤酵母菌株生长培养基为YPD培养基;表达培养基为BMGY和BMMY,两者均按照Invitrogen公司毕赤酵母表达手册配制。
1.2 试剂与仪器
N-苄氧羰酰基-L-苯丙氨酸-L-酪氨酸(CBZ-Phe-Tyr)购自上海Peptides International公司;T4连接酶、高保真DNA聚合酶Prime Star、限制性内切酶EcoR I、Not I、Sac I、Dpn I均购自日本宝生物工程有限公司;蛋白酶K购自上海生物工程公司;其他试剂均为常用分析纯试剂。
1.3 表达载体pPICZα-proCPY的构建
从本实验室保存的菌株Actinomucor elegans PEP001中反转录得到羧肽酶Y的基因[16],对反转录得到的基因密码子优化之后由安徽通用生物公司进行基因合成。用限制性内切酶EcoR I和Not I从合成质粒pPIC9K-proCPY上将CPY基因切下,通过双酶切连接的方式连接到毕赤酵母表达载体pPICZα上。转化大肠杆菌JM109,涂布于含有25 μg/mL的Low Salt LB培养基平板筛选转化子。
1.4 重组菌株pPICZα-proCPY/GS115的筛选及表达
将测序正确的质粒pPICZα-proCPY用限制性内切酶Sac I线性化,将线性化质粒电转化到毕赤酵母GS115中,涂布于含有100 μg/mL Zeocin的YPD平板,28 ℃培养2~3 d,筛选转化子。将转化子用无菌移液枪头分别点在含有600、1 000、1 800 μg/mL Zeocin的YPD平板上,置于28 ℃培养箱培养2~3 d。根据菌株所能耐受抗生素浓度的上限,将重组菌株依次命名为Zα-100、Zα-600、Zα-1000、Zα-1800。
将重组菌株转接到YPD培养基中,过夜培养后,按1%的接种量转入BMGY培养基中,28 ℃,250 r/min培养到OD600为6时,室温5 000 r/min离心5 min收集菌体,将菌体全部转移到BMMY培养基中,每隔24 h补加1%的甲醇进行诱导,诱导144 h后取样。采用Bradford法[17]检测上清液中的蛋白浓度,SDS-PAGE法检测CPY酶原的表达。
1.5 表达载体pGAPZα-proCPY的构建
将CPY基因通过酶切连接的方式连接到毕赤酵母表达载体pGAPZα上,转化大肠杆菌,在含有25 μg/mL Zeocin的Low Salt LB培养基平板上筛选转化子。
1.6 重组菌株pGAPZα-proCPY/GS115的筛选及表达
将测序正确的质粒用限制性内切酶Avr Ⅱ线性化之后,电转化到毕赤酵母中,在含有100 μg/mL的YPD平板筛选转化子,通过提高抗生素浓度的方法筛选得到重组菌株GAP-100、GAP-600、GAP-1000和GAP-1800。
将重组菌转接到YPD试管中,过夜培养后,按1%的接种量转入含有50 mL YPD培养基的250 mL锥形瓶中,28 ℃,250 r/min培养144 h后取样,采用Bradford法检测胞外总蛋白浓度,SDS-PAGE法检测CPY酶原的表达。
1.7 共表达BIP重组菌株的构建及表达
设计引物G418 up和G418 down如表1所示,以pPIC9K为模板PCR得到G418的抗性基因,将PCR产物纯化后作为引物,以pGAPZ-BIP质粒(BIP为毕赤酵母来源。Gene Bank登录号,XM_002490982.1)为模板进行PCR,用限制性内切酶Dpn I消除模板之后转化大肠杆菌JM109,在含有50 μg/mL卡那霉素的LB平板上筛选转化子。将抗性替换的质粒pGAPG-BIP用限制性内切酶Avr Ⅱ线性化,转入筛选得到的重组菌Zα-600、Zα-1000和Zα-1800中,涂布于含有2 mg/mL G418的YPD平板, 28℃培养2~3 d,筛选转化子。挑选转化子进行表达,用SDS-PAGE检测羧肽酶Y酶原的表达。用软件Image Lab 6.0分析CPY酶原条带的灰度值,并与重组菌株Zα-600 CPY酶原条带灰度值比较。
表1 PCR引物
Table 1 PCR pries
1.8 共表达PDI-ERO1重组菌株的构建及表达
按照质粒pGAPG-BIP的构建方法,将质粒pGAPZ-PDI、pGAPZ-ERO1(PDI为毕赤酵母来源,Gene Bank登录号:AJ302014.1,ERO1为毕赤酵母来源,Gene Bank登录号:XM_002489600.1)上的抗性基因替换成G418的抗性基因,得到质粒pGAPG-PDI和pGAPG-ERO1。用限制性内切酶Avr Ⅱ线性化后转入重组菌Zα-600、Zα-1000、Zα-1800中,得到菌株Zα-600-PDI、Zα-1000-PDI、Zα-1800-PDI及Zα-600-ERO1、Zα-1000-ERO1、Zα-1800-ERO1。挑选转化子进行甲醇诱导表达,用SDS-PAGE检测CPY酶原的表达。用软件Image Lab 6.0分析CPY酶原条带的灰度值,并与Zα-600 CPY酶原条带灰度值进行比较。
2 结果与讨论
2.1 表达载体pPICZα-proCPY的构建
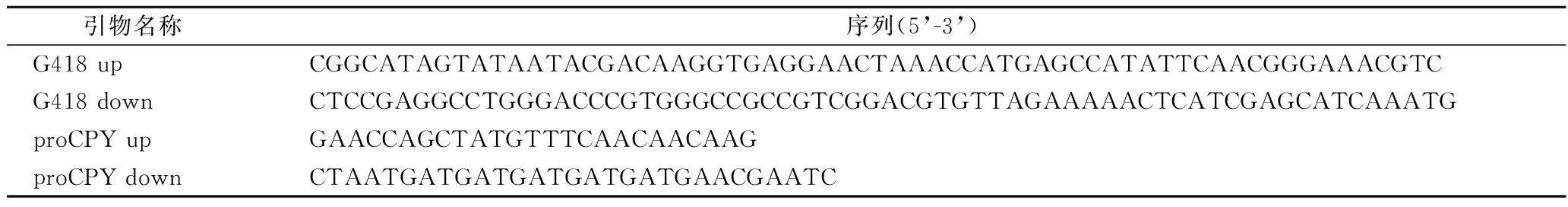
利用双酶切连接的方法将经过密码子优化的羧肽酶Y(CPY)基因克隆到表达载体pPICZα上,转化大肠杆菌JM109。将得到的转化子进行菌落PCR鉴定,鉴定结果如图1所示,有一条约1 500 bp的条带,与预计的1 494 bp相符,将验证正确的菌株进行测序,测序序列完全正确的菌株提取质粒进行下一步实验操作。
M-5 000 bp DNA Marker; 1-PCR产物
图1 pPICZα-proCPY PCR验证
Fig.1 pPICZα-proCPY verification
2.2 重组表达菌株pPICZα-proCPY/GS115的构建及菌株的筛选
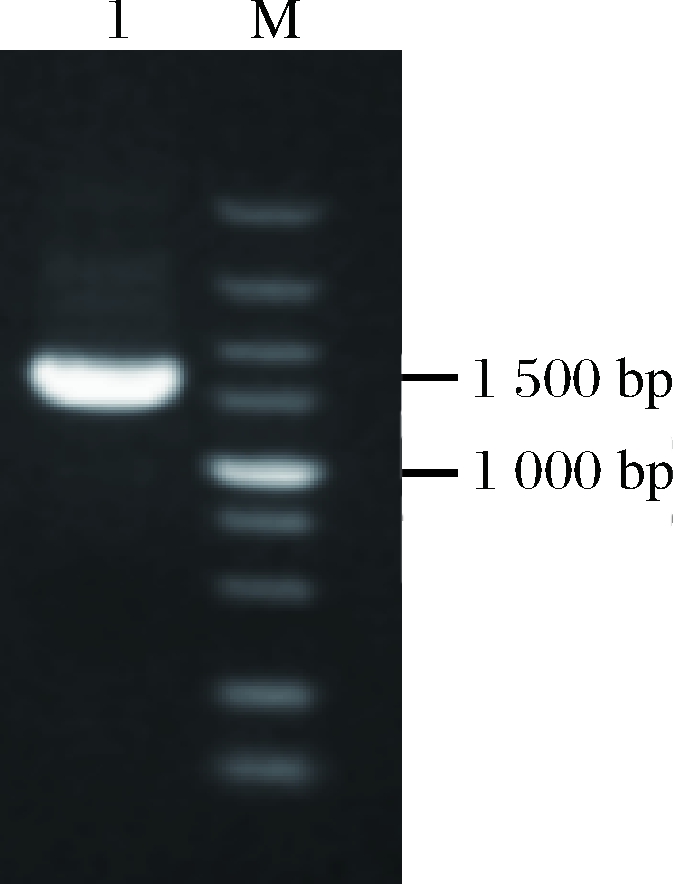
将质粒pPICZα-proCPY,用限制性内切酶Sac I线性化后电转化毕赤酵母中。将得到的转化子分别点在含有不同抗生素浓度的YPD平板上,根据菌株所能耐受的最高抗生素浓度,得到重组菌株Zα-100、Zα-600、Zα-1000、Zα-1800。将转化子进行表达,经诱导144 h后发酵液上清液经SDS-PAGE测试后,分析结果如图2所示,重组CPY酶原分子量在60 ku左右,与理论值58.8 ku相符,表明CPY酶原成功实现了分泌表达;通过不同菌株目的蛋白条带的比较,可以看出Zα-600的表达效果要明显强于其他重组菌株,Bradford法测定发酵液上清中胞外总蛋白含量达到了(275±20) mg/L,是现有报道雅致放射毛霉羧肽酶Y在毕赤酵母中表达量[16]的1.82倍。
M-标准蛋白Marker; 1-Zα-100; 2~4-Zα-600; 5~7-Zα-1000;
8~9-Zα-1800
图2 重组菌株分泌表达CPY酶原
Fig.2 Recombinant strains secrete CPY zymogen
2.3 表达载体pGAPZα-proCPY的构建
按照质粒pPICZα-proCPY的构建方法得到质粒pGAPZα-proCPY,转化大肠杆菌JM109。将转化子进行菌落PCR鉴定,鉴定结果如图3所示,有一条约1500 bp的条带,与预计的1494 bp相符,将验证正确的菌株进行测序,测序序列完全正确的菌株提取质粒进行下一步实验操作。
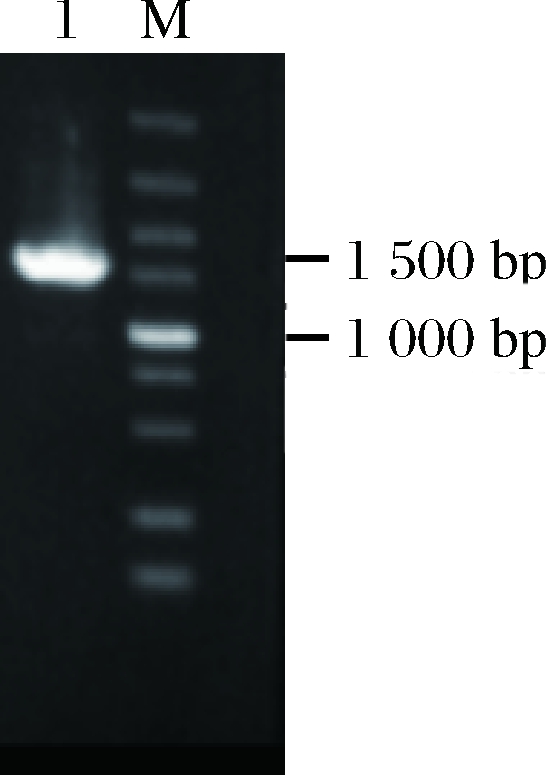
M-5 000 bp DNA Marker; 1-PCR产物
图3 pGAPZα-proCPY PCR验证
Fig.3 pGAPZα-proCPY PCR verification
2.4 重组表达菌株pGAPZα-proCPY/GS115的构建及菌株的筛选
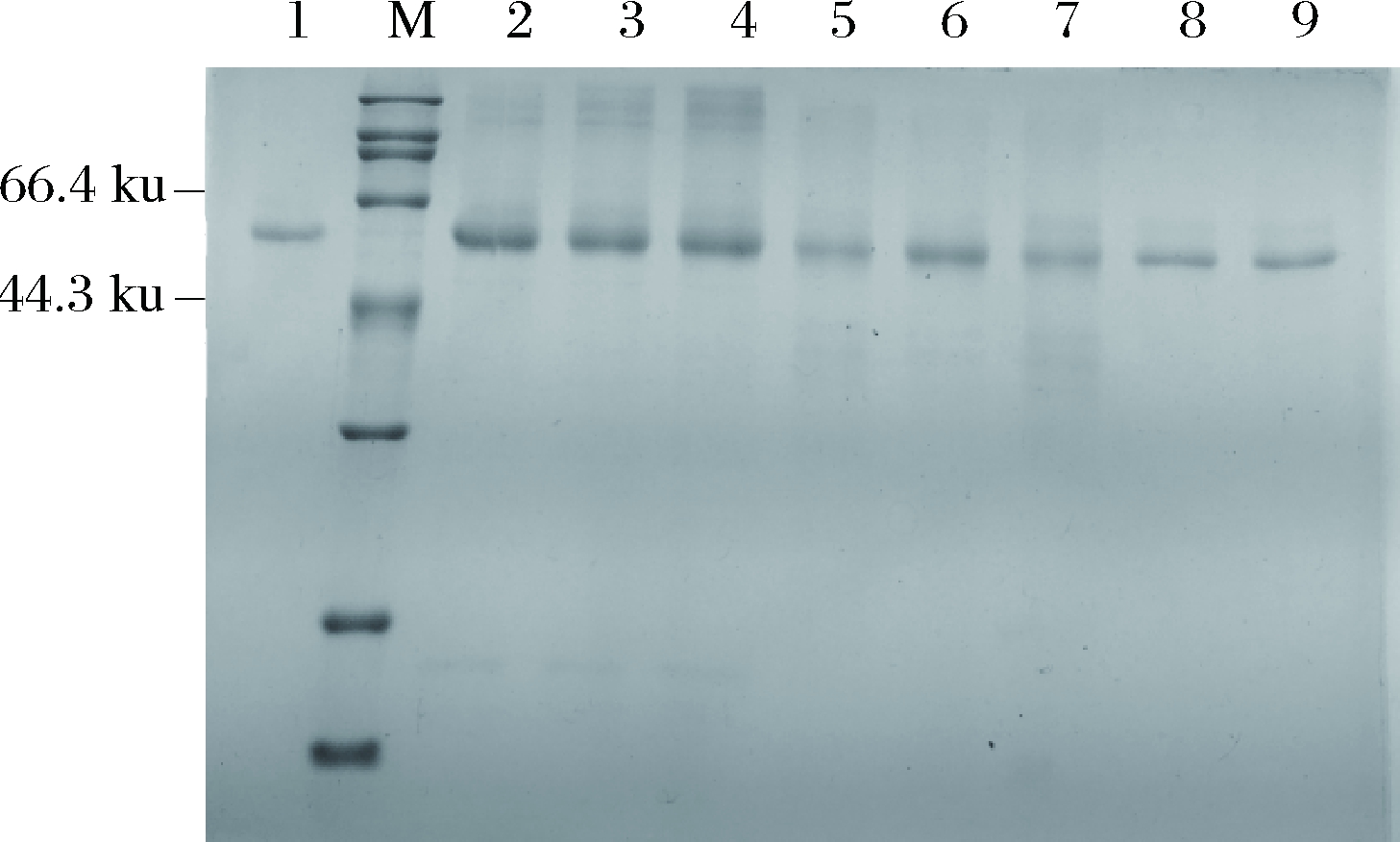
将质粒pGAPZα-proCPY用限制性内切酶Avr Ⅱ线性化后电转化毕赤酵母。通过提高抗生素浓度的方式筛选重组菌株,得到重组菌GAP-100、GAP-600、GAP-1000、GAP-1800。将转化子在YPD进行表达,144 h后发酵液上清经SDS-PAGE分析结果如图4-A所示,重组蛋白的分子量大小在60 ku左右与CPY酶原理论值58.8 ku相符,说明CPY酶原实现了成功表达。通过用软件Image Lab 6.0对蛋白条带灰度值进行分析,结果如图4-B所示,在所筛选的重组菌中GAP-600-1的分泌表达效果最好,但是其CPY酶原的分泌量只有重组菌Zα-600的64%。
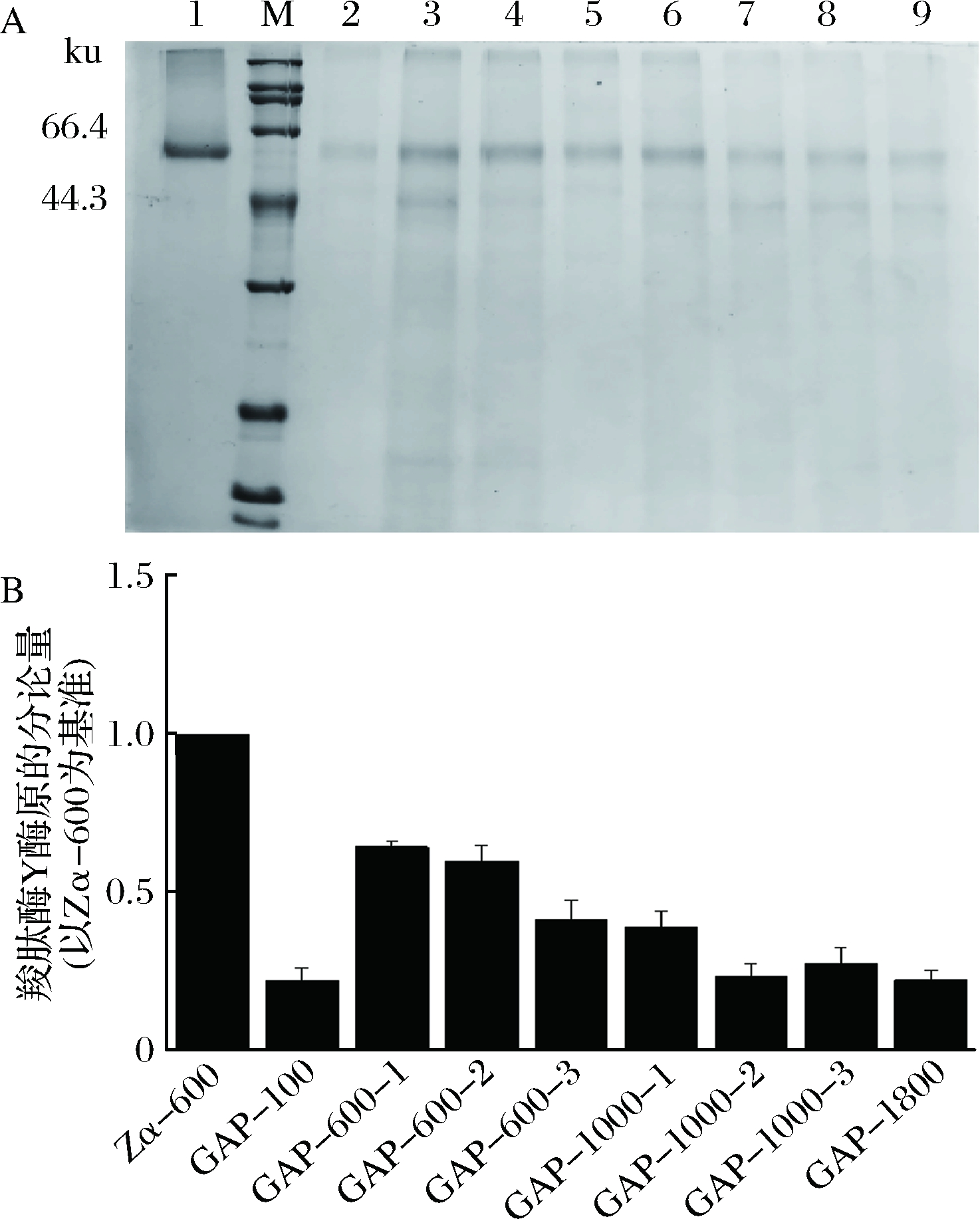
A-SDS-PAGE,M:标准蛋白Marker; 1-Zα-600;2:GAP-100;
3~6:GAP-600-; 7~8:GAP-1000; 9:GAP-1800;B:灰度值分析
图4 重组菌株pGAPZα-proCPY/GS115分泌表达CPY
酶原
Fig.4 Recombinant strains pGAPZα-proCPY/GS115
secrete CPY zymogen
2.5 共表达BIP重组菌株的构建及筛选
前期实验结果表明重组菌株pPICZα-proCPY/GS115分泌表达CPY酶原效果pGAPZα-proCPY/GS115要好,因而本实验选择在重组菌株pPICZα-proCPY/GS115中共表达分子伴侣BIP。有研究表明在高拷贝菌株中共表达分子伴侣的效果要比在低拷贝菌株中的效果好[14],因而将带有G418抗性基因的质粒pGAPG-BIP用限制性内切酶Avr Ⅱ线性化后,转入重组菌Zα-600、Zα-1000、Zα-1800中,在含有2 mg/mL G418的YPD平板上得到重组菌Zα-600-BIP、Zα-1000-BIP、Zα-1800-BIP。分别随机挑选转化子进行表达,经甲醇诱导144 h后发酵上清液经SDS-PAGE分析结果如图5-A所示,共表达分子伴侣BIP后,重组菌中CPY酶原表达量都有所提升,重组菌Zα-1800-BIP提升尤其明显。CPY酶原蛋白条带灰度值分析结果如图5-B所示,重组菌Zα-1800-BIP CPY酶原表达量是重组菌Zα-600的1.87倍,其胞外总蛋白的分泌量达到了(525±15) mg/L。共表达的BIP为胞内表达,不分泌到胞外,因此SDS-PAGE中无显示。
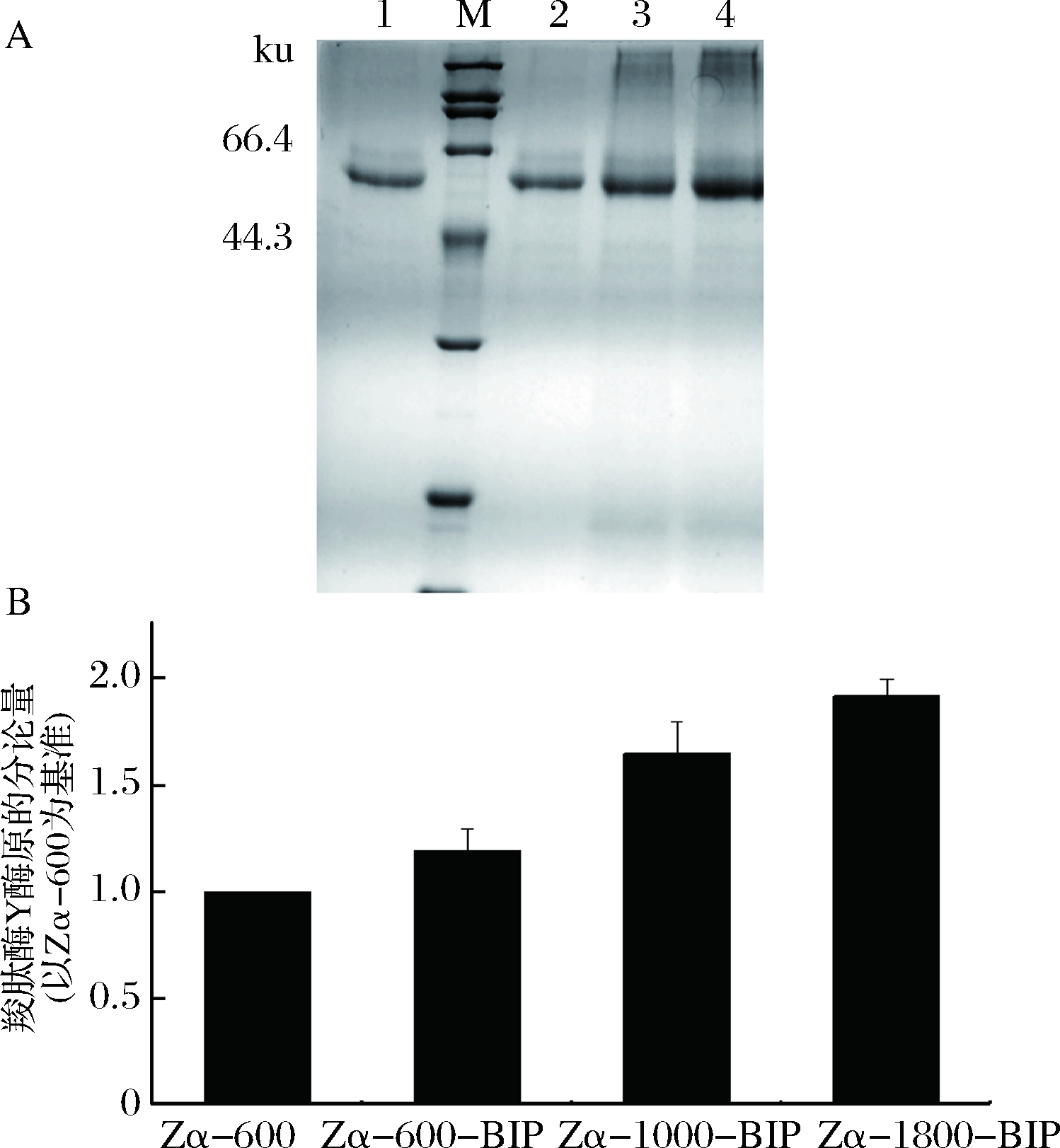
A-SDS-PAGE, M-标准蛋白Marker; 1-Zα-600;
2-Zα-600-BIP; 3-Zα-1000-BIP;4-Zα-1800-BIP
图5 共表达BIP重组菌株分泌表达CPY酶原
Fig.5 Co-expressing BIP recombinant strains secret
CPY zymogen
研究报道BIP具有辅助蛋白质折叠,并且促进初生肽链向内质网运输的作用[15]。高拷贝菌株中CPY基因的转录水平高,但是缺少足够的伴侣蛋白帮助其初生肽链向内质网运输以及初步折叠,影响了CPY酶原分泌表达,拷贝数越高的重组菌株影响越严重,当在重组菌株中加强BIP(毕赤酵母来源)表达之后,更多的BIP可以促进CPY酶原向内质网运输、并且辅助CPY酶原的折叠。有足够的BIP来辅助CPY酶原的折叠和运输之后,高拷贝重组菌Zα-1800的CPY酶原分泌量有了极大的提升,超过了Zα-600共表达BIP的效果。
2.6共表达PDI重组菌株的构建及筛选
重组菌株Zα-600、Zα-1000、Zα-1800转入质粒pGAPG-PDI,在含有2 mg/mL G418的YPD平板上得到重组菌株Zα-600-PDI、Zα-1000-PDI、Zα-1800-PDI。
随机挑选转化子进行表达,经甲醇诱导144 h后发酵液上清液经SDS-PAGE检测结果如图6-A所示。从SDS-PAGE图可以看出共表达PDI的重组菌中CPY酶原较重组菌Zα-600均有提升,重组菌Zα-1800-PDI提升尤其明显。CPY酶原条带的灰度值分析结果如图6-B所示,重组菌Zα-1800-PDI CPY酶原表达量是重组菌Zα-600的1.71倍。共表达的PDI为胞内表达,不分泌到胞外,SDS-PAGE图上无显示。
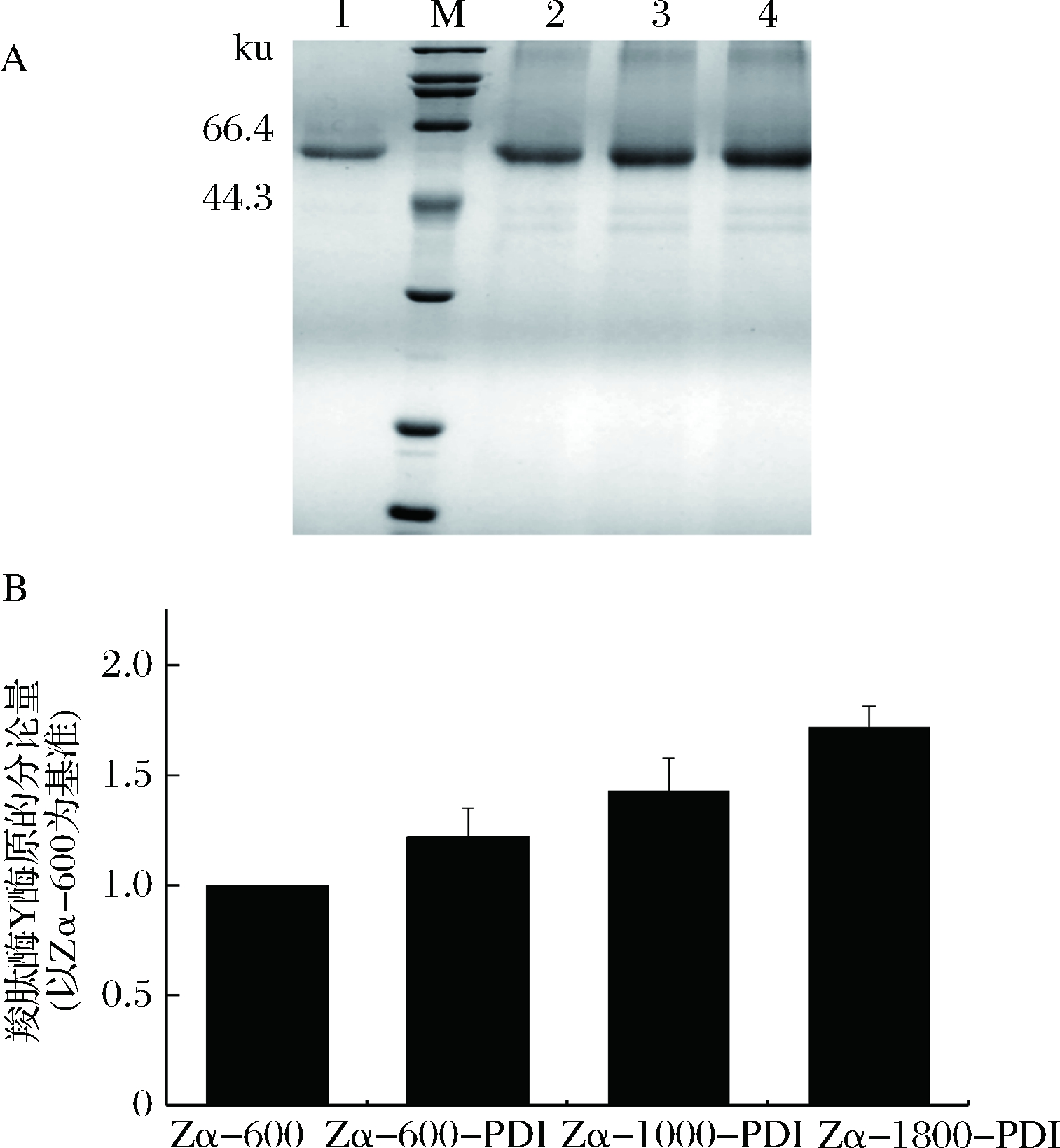
A-SDS-PAGE; M-标准蛋白Marker; 1-Zα-600;
2-Zα-600-PDI; 3-Zα-1000-PDI; 4-Zα-1800-PDI;B-灰度值分析
图6 共表达PDI重组菌株分泌表达CPY酶原
Fig.6 Co-expressing PDI recombinant strains secret CPY zymogen
有研究者通过在重组菌株中共表达PDI,将蛋白的分泌表达量提升到了原来的2.2倍[16],本研究构建重组菌Zα-600-PDI、Zα-1000-PDI、Zα-1800-PDI,分泌表达结果显示,共表达PDI对CPY酶原的分泌有较大的促进作用,在拷贝数越高的菌株中作用越明显。PDI具有辅助二硫键的形成和分子伴侣的作用[14]。在高拷贝菌株中CPY基因的转录水平很高,但是CPY酶原因缺少足够的分子伴侣辅助折叠,导致无法分泌胞外,共表达PDI之后,更多的PDI可以辅助蛋白的折叠,因而有更多的CPY酶原可以分泌到胞外去。
2.7 共表达ERO1重组菌株的构建及筛选
在重组菌株Zα-600、Zα-1000、Zα-1800转入质粒pGAPG-ERO1,在含有2 mg/mL G418的YPD平板上得到重组菌Zα-600- ERO1、Zα-1000- ERO1、Zα-1800- ERO1。
随机挑选转化子进行表达,经甲醇诱导144 h后发酵液上清液经SDS-PAGE检测结果如图7所示,共表达ERO1的重组菌中CPY酶原较重组菌Zα-600均有略微的提升。CPY酶原条带的灰度值分析结果如图7-B,重组菌Zα-1800-ERO1 CPY酶原表达量是重组菌Zα-600的1.26倍。共表达的ERO1为胞内表达,不分泌到胞外,SDS-PAGE图无显示。
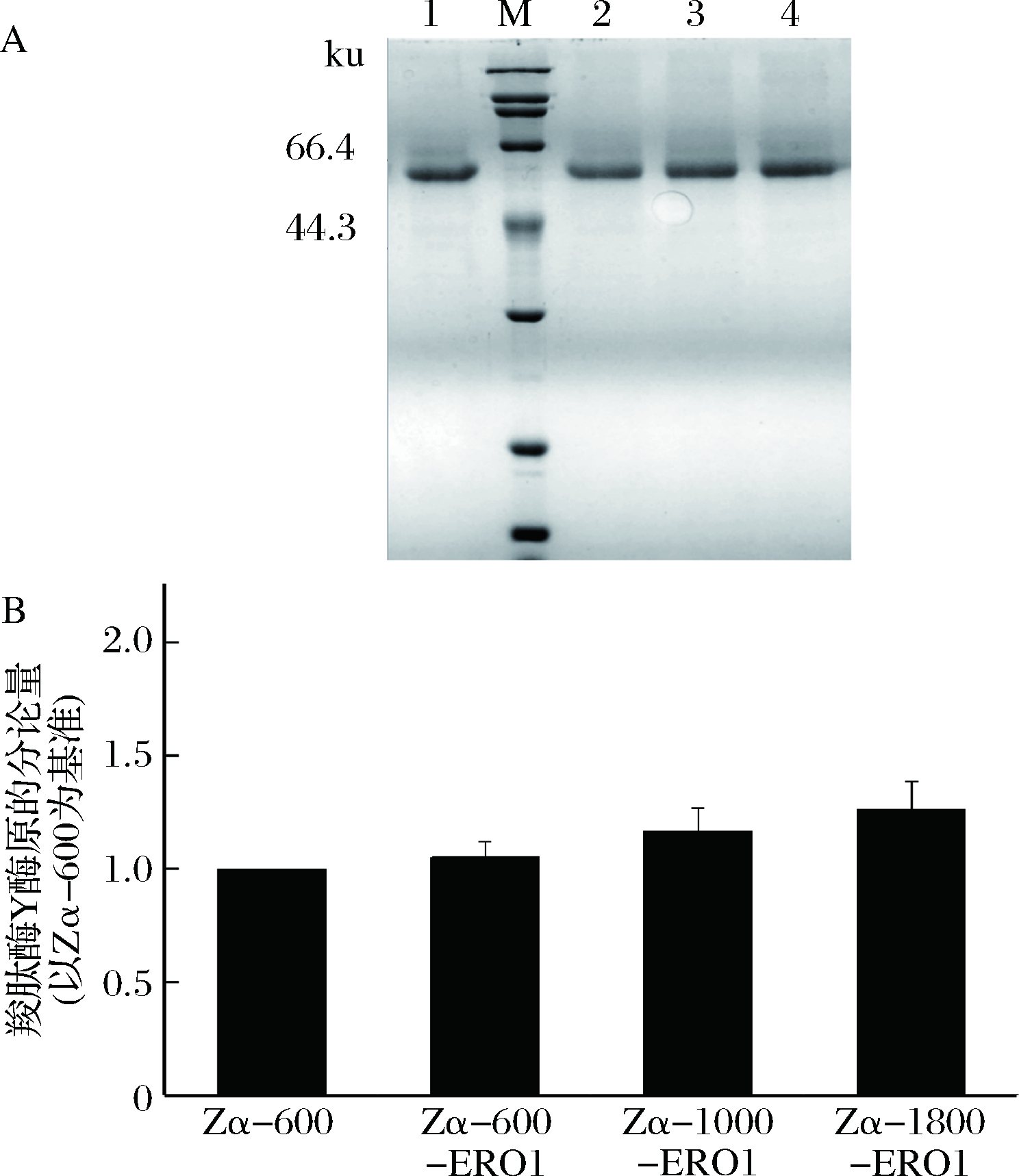
A-SDS-PAGE; M-标准蛋白Marker; 1- Zα-600; 2-Zα-600-
ERO1;3-Zα-1000-ERO1; 4-Zα-1800-ERO1;B-灰度值分析
图7 共表达ERO1重组菌株分泌表达CPY酶原
Fig.7 Co-expressing ERO1 recombinant strains secret CPY zymogen
ERO1在PDI辅助二硫键作用过程中起着帮助PDI再生的作用[14]。有研究者发现当单独共表达ERO1时,蛋白的分泌量提升到了原来的2.3倍,比共表达PDI的效果还要好[15]。本研究结果显示在毕赤酵母中分泌表达雅致放射毛霉羧肽酶Y,分子伴侣ERO1中的作用有限。推测可能形成二硫键并不是限制CPY酶原分泌表达过程中的重要步骤。
在毕赤酵母中分泌表达雅致放射毛霉羧肽酶Y所得到的是具有前导肽的酶原,在体外可以在胰蛋白酶[16]的作用下,切除前导肽得到成熟的羧肽酶Y。
3 结论
本文在毕赤酵母中异源表达雅致放射毛霉羧肽酶Y分别构建了含有AOX1启动子和GAP启动子的重组菌,筛选得到高效分泌CPY酶原的重组菌Zα-600。通过筛选得到的具有不同拷贝数的Zα系列重组菌中,共表达分子伴侣BIP、PDI、ERO1,表达效果最佳的重组菌Zα-1800-BIP将CPY酶原的分泌量提升到了Zα-600的1.82倍,胞外总的蛋白量达到了(525±15) mg/L。
参考文献
[1] YUX H, ZHAI C, ZHONG X, et al. High-level expression and characterization of carboxypeptidase Y from Saccharomyces cerevisiae in Pichia pastoris GS115 [J]. Biotechnology Letters, 2015, 37(1):161-167.
[2] 吴静,闵柔,邬敏辰等.羧肽酶研究进展[J].食品与生物技术学报, 2012, 31(8):793-801.
[3] HB V D H, KIELLANDBRANDT M C, WINTHER J R. Review: biosynthesis and function of yeast vacuolar proteases[J]. Yeast, 1996, 12(1):1-16.
[4] JUNG G, UENO H, HAYASHI R. Carboxypeptidase Y: structural basis for protein sorting and catalytic triad[J]. Journal of Biochemistry, 1999, 126(1):1-6.
[5] COOL D R, HARDIMAN A. C-terminal sequencing of peptide hormones using carboxypeptidase Y and SELDI-TOF mass spectrometry[J]. Biotechniques, 2004, 36(1):32-34.
[6] HAYASHI R, MOORE S, STEIN W H. Carboxypeptidase from yeast. Large scale preparation and the application to COOH-terminal analysis of peptides and proteins[J]. Journal of Biological Chemistry, 1973, 248(7):2296-302.
[7] 付静.A.elegans、A.oryzae和R.oligosporus肽酶系统及其脱苦机理的比较研究[D].广州:华南理工大学博士学位论文, 2011.
[8] FU J, LI L, YANG X Q. Specificity of carboxypeptidases from Actinomucor elegans and their debittering effect on soybean protein hydrolysates [J]. Applied Biochemistry & Biotechnology, 2011, 165(5-6):1 201-1 210.
[9] 郭雨刚.毕赤酵母表达系统及其应用[D].合肥:中国科学技术大学, 2012.
[10] AHMAD M, HIRZ M, PICHLER H, et al. Protein expression in Pichia pastoris: recent achievements and perspectives for heterologous protein production[J]. Applied Microbiology & Biotechnology, 2014, 98(12):5 5301-5 317.
[11] DAMASCENO L M, HUANG C J, BATT C A. Protein secretion in Pichia pastoris and advances in protein production[J]. Applied Microbiology & Biotechnology, 2012, 93(1):31-39.
[12] SHIN S Y, BAE Y H, KIM S K, et al. Effects of signal sequences and folding accessory proteins on extracellular expression of carboxypeptidase Y in recombinant Saccharomyces cerevisiae[J]. Bioprocess & Biosystems Engineering, 2014, 37(6):1 065.
[13] DALY R, HEARN M T W. Expression of heterologous proteins in Pichia pastoris : a useful experimental tool in protein engineering and production[J]. Journal of Molecular Recognition, 2005, 18(2):119-138.
[14] LEONARDO M, DAMASCENO, CHUNG-JR H, et al. Protein secretion in Pichia pastoris and advances in protein production.[J]. Applied Microbiology and Biotechnology, 2012, 93(1):31-39.
[15] YANG J, LU Z, CHEN J, et al. Effect of cooperation of chaperones and gene dosage on the expression of porcine PGLYRP-1 in Pichia pastoris[J]. Applied Microbiology & Biotechnology, 2016, 100(12):5 453-5 465.
[16] GUAN B, CHEN F, SU S, et al. Effects of co‐overexpression of secretion helper factors on the secretion of a HSA fusion protein (IL2‐HSA) in Pichia pastoris[J]. Yeast, 2016, 33(11):587-600.
[17] 纪海兵,刘中美,郭军玲,等.雅致放射毛霉羧肽酶Y的基因克隆、表达与酶学性质研究[J].现代食品科技, 2017(1):81-86.
[18] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry,1976, 72(1-2):248-254.