副溶血弧菌是一种常见的嗜盐性革兰氏阴性菌,适宜生长2.5%~3%的盐溶液中,主要分布于海水及鱼、虾、蟹、贝等海产食品中,是引起海鲜类食物中毒的主要原因[1-3]。据报道,在亚洲,尤其是在中国、日本、韩国,因为生吃海鲜而引起的副溶血弧菌食物中毒事件频频发生[4-5]。2003-2005年广东省水产品污染状况调查显示,431份样品中有156份检出副溶血性弧菌,总检出率为36.19%[6]。2008年上海的调查数据显示,副溶血弧菌是导致夏秋季腹泻的主因,门诊腹泻患者肛拭检测副溶血性弧菌阳性率为2.95%,远高于沙门菌(0.53%),位居首位[7]。2016年唐山市调查显示,采集的204份海产品中,副溶血性弧菌阳性检出率为32.84%[8]。副溶血弧菌携带tdh、trh毒力基因,人感染后会导致急性肠胃炎,临床症状为腹泻、头痛、呕吐、恶心、低烧[9]。
由于抗生素的长期、不合理使用以及缺乏有效监管,病原菌的耐药性越来越强,且出现多重耐药性问题。近年来,多种“超级细菌”不断被发现,引起了国内外的广泛关注[10]。2010年8月《柳叶刀》报道了英国卡迪夫大学医学院蒂莫西教授发现的携带新德里一号金属酶(NDM-1)基因的细菌,这是继MRSA之后发现的又一超级耐药细菌[11]。耐药感染性疾病已成为当前临床感染性疾病死亡的主要原因[12]。海产养殖过程中抗生素的滥用也导致了副溶血弧菌耐药性的增强,使得抗生素的药敏性大大下降甚至消失,从而导致临床治疗副溶血弧菌感染病症难度变大,成本增加。
生物膜(biofilm)是单一或混合的微生物聚集、附着于物体表面上生长繁殖,并包被在自身分泌的胞外聚合物中而形成的类膜结构的聚合体[13-14]。生物膜能够使细菌免受宿主的获得性免疫应答以及吞噬细胞的捕食,使其耐药性提高10~1 000倍[15]。本研究对广州市售海产品中的副溶血弧菌进行分离鉴定,并分析了其耐药性及生物膜形成能力,旨在为海产品的质量安全控制以及防治副溶血弧菌感染提供理论依据和参考数据。
1 材料与方法
1.1 材料
1.1.1 标准菌株与材料
副溶血弧菌(Vibrio parahaemolyticus)ATCC 17802、大肠杆菌(Escherichia coli)ATCC 25922由本实验室保藏。鱼、虾、蛤、蚝等海产品采集于广州市区超市及农贸市场。氧化酶试纸、副溶血弧菌生化检测试剂盒购于广东环凯微生物科技有限公司。细菌基因组DNA提取试剂盒购于北京索莱宝科技有限公司。
1.1.2 药敏纸片
20种常用抗生素的药敏检测纸片,包括青霉素(PEN,10 μg)、氨苄西林(AMP,10 μg)、氨苄西林/舒巴坦(AMS,10 μg)、美罗培南(MEM,10 μg)、多粘菌素B(PB,300 μg)、万古霉素(VAN,30 μg)、头孢噻吩(CEP,30 μg)、头孢他啶(CAZ,30 μg)、庆大霉素(GEN,10 μg)、卡那霉素(KAN,30 μg)、链霉素(STR,10 μg)、四环素(TET,30 μg)、强力霉素(DOX,30 μg)、氯霉素(CHL,30 μg)、乙酰螺旋霉素(ASP,30 μg)、红霉素(ERY,15 μg)、环丙沙星(CIP,5 μg)、萘啶酸(NA,30 μg)、利福平(RIF,5 μg)、复方新诺明(SXT,23.75/1.25 μg),购于杭州市微生物试剂有限公司。
1.1.3 溶液及培养基
质量浓度为1 g/L结晶紫溶液;体积分数为33%冰乙酸溶液:量取100 mL冰乙酸,加入到200 mL去离子水中。
含质量浓度为30 g/L NaCl的胰蛋白胨大豆肉汤培养基(TSB):胰蛋白胨15 g、大豆蛋白胨5 g、NaCl 30 g、去离子水1 L。用1 mol /L NaOH 溶液调节pH 为7.0~7.4; 121 ℃,0.15 MPa下高压灭菌15 min。
含30 g/L NaCl 的胰蛋白胨大豆肉汤琼脂培养基(TSA):在上述TSB 配方的基础上加入1.5% 的琼脂,加热融化后灭菌。
含30 g/L NaCl的碱性蛋白胨水、TCBS培养基、3%氯化钠三糖铁琼脂、MR-VP培养基、MHB培养基均购于广东环凯微生物微生物科技有限公司。
1.2 方法
1.2.1 样品采集及处理
从广州市各区的14个超市及农贸市场采集市售的鱼、虾、蛤、蚝等海产品,用无菌采样袋装好后放入冰盒装中返校,确保在2 h内处理样品。鱼取表面组织、腮、肠;虾(甲壳类)用自来水洗净后取整个动物,包括肠和腮;蛤和蚝(贝类)用自来水冲洗干净后取内容物。取样时均为无菌操作方式。
1.2.2 副溶血弧菌的分离鉴定
菌株分离鉴定:按照GB 4789.7—2013中副溶血弧菌检验方法,样品在质量浓度为30 g/L氯化钠碱性蛋白胨水中增菌后,划线分离于TCBS平板中,挑取可疑单菌落进行革兰氏染色镜检、氧化酶试验、3%氯化钠三糖铁琼脂试验、嗜盐性试验,并利用副溶血弧菌生化检测试剂盒进行生化鉴定,包括30 g/L氯化钠甘露醇、3%氯化钠赖氨酸脱羧酶、ONPG和MR-VP试验。
16S rDNA测序:生化试验阳性菌株进行16S rDNA同源性比较分析。菌株在TSB培养液中培养过夜后,用细菌基因组DNA提取试剂盒提取基因组DNA,于-20 ℃中保存备用。基因组DNA进行PCR扩增,所用引物为27F:5’-AGAGTTTGATCCTGGCTCAG-3’ 和1495R: 5’-CTACGGCTACCTTGTTACGA-3’。扩增体系为:无菌水10.5 μL,混合引物1 μL,taq PCR Master Mix 12.5 μL,DNA模板1 μL。其中混合引物为1 μL 27f、1 μL 1492r和8 μL无菌水。PCR反应条件:95 ℃预变性5 min(95 ℃变性1 min,54 ℃复性1 min,72 ℃延伸2 min),30个循环,72 ℃保温10 min。扩增后的DNA经检验送至上海美吉生物医药科技有限公司进行测序。将16S rDNA 基因测序结果通过BLAST在GenBank中进行同源比对分析。
1.2.3 菌株的耐药性分析
将副溶血弧菌分离菌株、ATCC 17802菌株和质控菌株大肠埃希菌ATCC 25922分别接种于TSB和LB培养基中,于120 r/min的摇床内37 ℃活化12 h。培养液于4 000 r/min离心10 min,收集菌体,用生理盐水10倍稀释至浓度为108 CFU/mL。
取100 μL菌液在MH琼脂平板中,涂布均匀。干燥数分钟后,用无菌镊子夹取20种常用抗生素纸片轻轻放置在平板中,纸片间距不小于24 mm,距离平板边缘不小于15 mm。平行实验3次。将平板倒置于培养箱中,37 ℃下培养18 h后,用标准直尺测量抑菌圈大小,根据参照美国临床实验室标准化协会(Clinical and Laboratory Standards Institute, CLSI)抗菌药物敏感性试验执行标准推荐的纸片扩散法及判定标准[16]、杭州微生物试剂有限公司提供的产品说明书整理出的标准(表1)判定结果。
1.2.4 结晶紫染色法测定生物膜形成量
将副溶血弧菌TSB培养液离心后收集菌体用TSB培养基10倍稀释至浓度为106 CFU/mL;分别在96孔板中每孔加入200 μL菌液;封盖后用保鲜膜包好,在37 ℃下静置培养24 h;将孔板中的培养液去除,使用多道移液枪在每孔中加入250 μL无菌生理盐水清洗3遍,去除浮游菌; 60 ℃干燥15 min;在干燥后的孔板中加入200 μL质量浓度为1 g/L的结晶紫溶液,染色10 min;弃去结晶紫溶液,使用无菌生理盐水清洗3遍,继续干燥15 min;在每孔中加入200 μL、33%冰乙酸,静置10 min后,使用Multiskan FC酶标仪(Thermo, USA)测量OD595值[17]。
表1 副溶血弧菌对20种抗生素的耐药标准
Table 1 Criteria for antibiotic resistance of Vibrio parahaemolyticus against 20 antibiotics
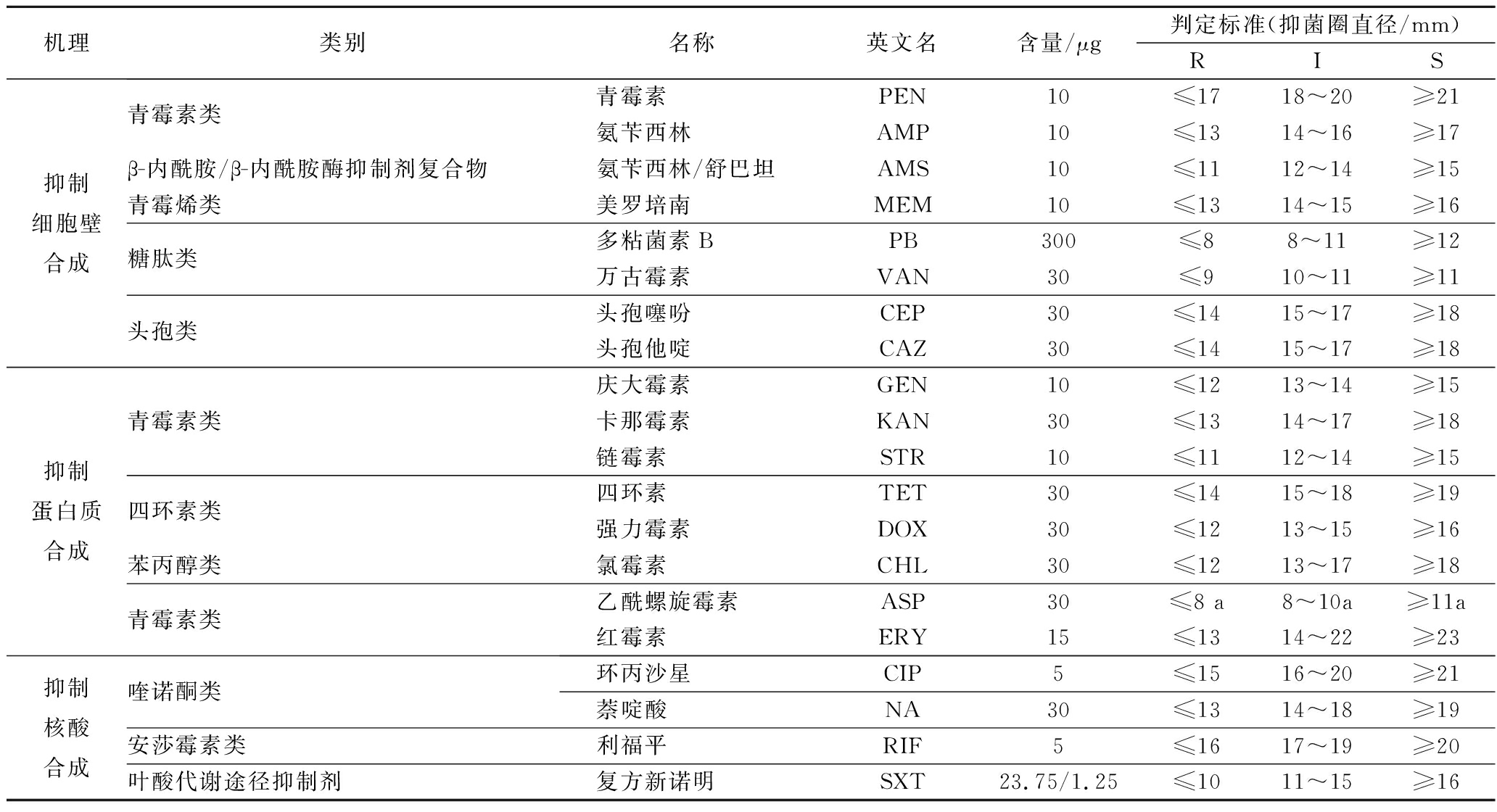
注: R为耐药;I为中介敏感;S为敏感。a为尚未在CLSI标准和相关文献中找到的判定标准,是本实验室暂定的判定值。下同。
2 结果与分析
2.1 海产中副溶血弧菌的分离鉴定
从广州市区的超市及农贸市场采集市售海产品,随即进行样品处理,增菌18 h后进行TCBS平板划线分离、革兰氏染色镜检、氧化酶试验、质量浓度为30 g/L的氯化钠三糖铁试验、嗜盐性试验。结果表明,分离菌株在TCBS培养基中菌落为绿色(图1),镜检为短弧状的杆菌(图2)。
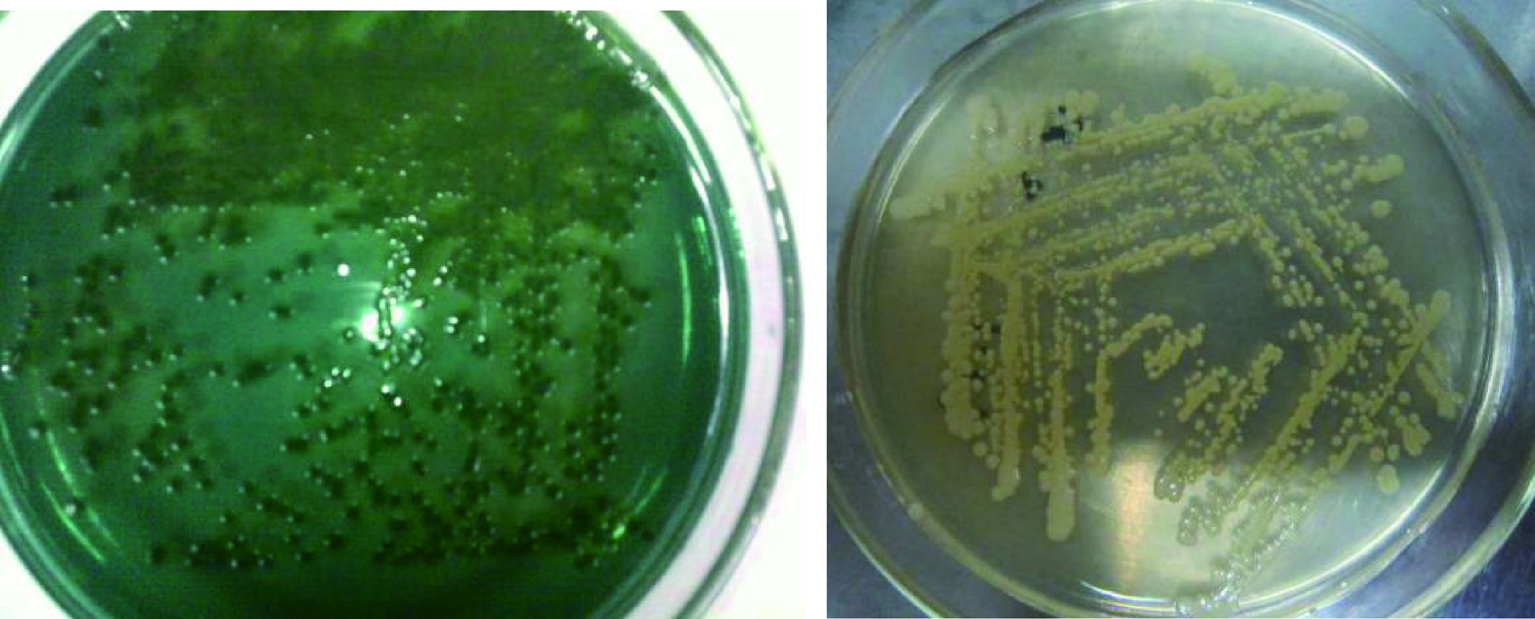
图1 副溶血弧菌在TCBS、TSA平板上的菌落形态
Fig.1 Colony morphology of Vibrio parahaemolyticus on TCBS and TSA plates
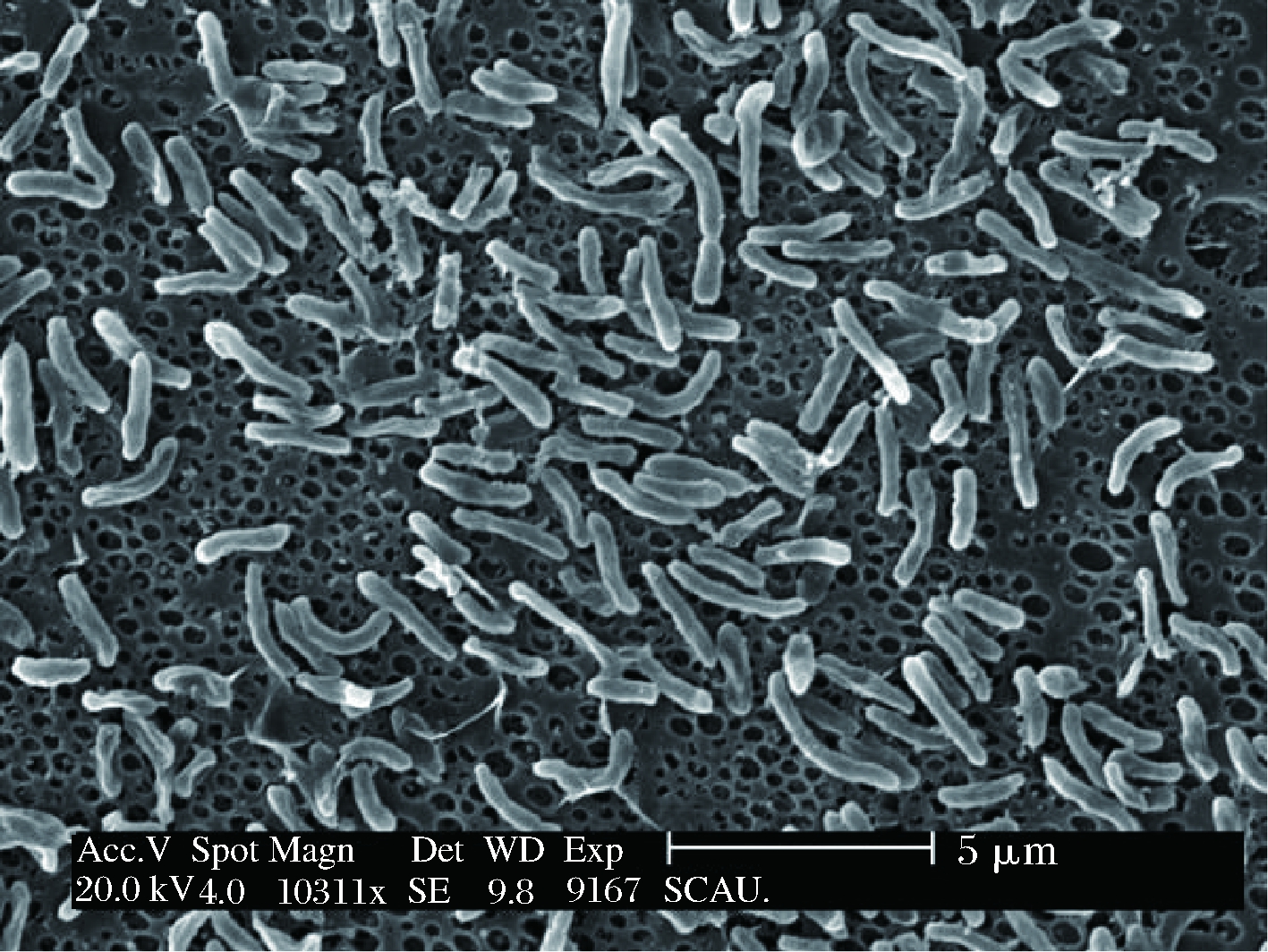
图2 副溶血弧菌分离菌株扫描电镜图
Fig.2 SEM picture of the isolated strain of Vibrioparahaemolyticus
氧化酶试验呈阳性,具有嗜盐性。对8株菌株进行生理生化实验和16S rDNA同源性比较,鉴定为副溶血弧菌菌株(表2)。
2.2 分离菌株的耐药性分析
测定副溶血弧菌分离菌株和ATCC 17802菌株对常用20种抗生素的药敏结果如表3、图3所示。结果表明,8株分离菌株对青霉素类、β-内酰胺类、青霉烯类的抗生素抗性很强,对氨基糖苷类、四环素类、苯丙醇类、大环内酯类、喹诺酮类、安莎霉素类、叶酸代谢途径抑制剂等抗生素较为敏感。其中对青霉素、万古霉素2种抗生素均显示耐药性;对氨苄西林、氨苄西林/舒巴坦、头孢噻吩耐药的菌株也达到7株,对美罗培南、利福平耐药的菌株分别有6株和5株。而对多粘菌素B、头孢他啶、庆大霉素、氯霉素、红霉素、环丙沙星、萘啶酸等抗生素均敏感,对卡那霉素、氯霉素、四环素、强力霉素、复方新诺明具有耐药性的菌株比较少,有1~2株,说明这些抗生素表现出对副溶血弧菌分离菌株较强的抑制作用。此外,只有8号菌株的耐药性弱于ATCC 17802菌株,大部分分离菌株耐抗生素的种类更多。
表2 分离菌株16S rDNA同源性比对分析
Table 2 16S rDNA sequence analysis of the isolated strains of Vibrio parahaemolyticus

表3 分离菌株的耐药性测定
Table 3 Drug resistances of the isolated strains ofVibrio parahaemolyticus
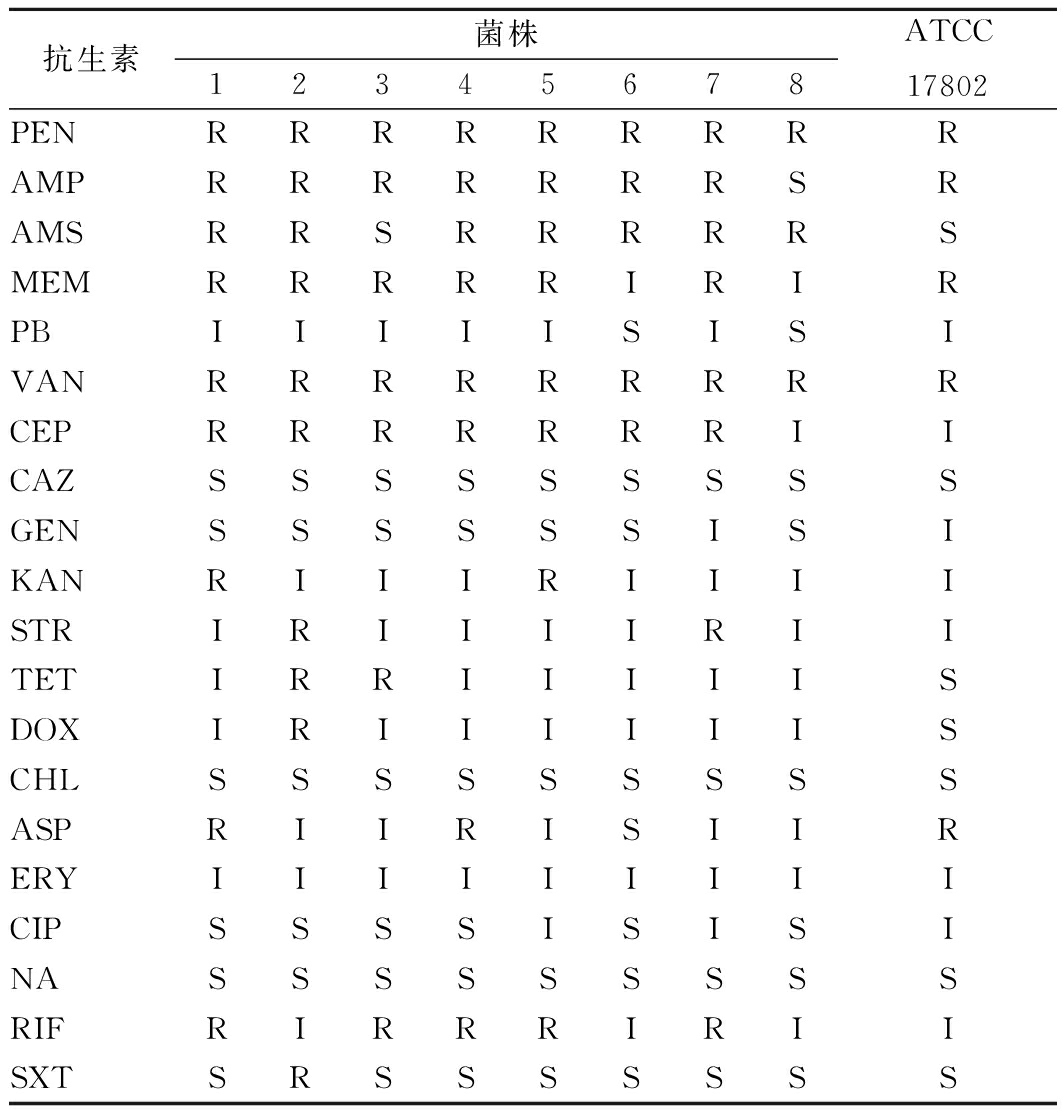
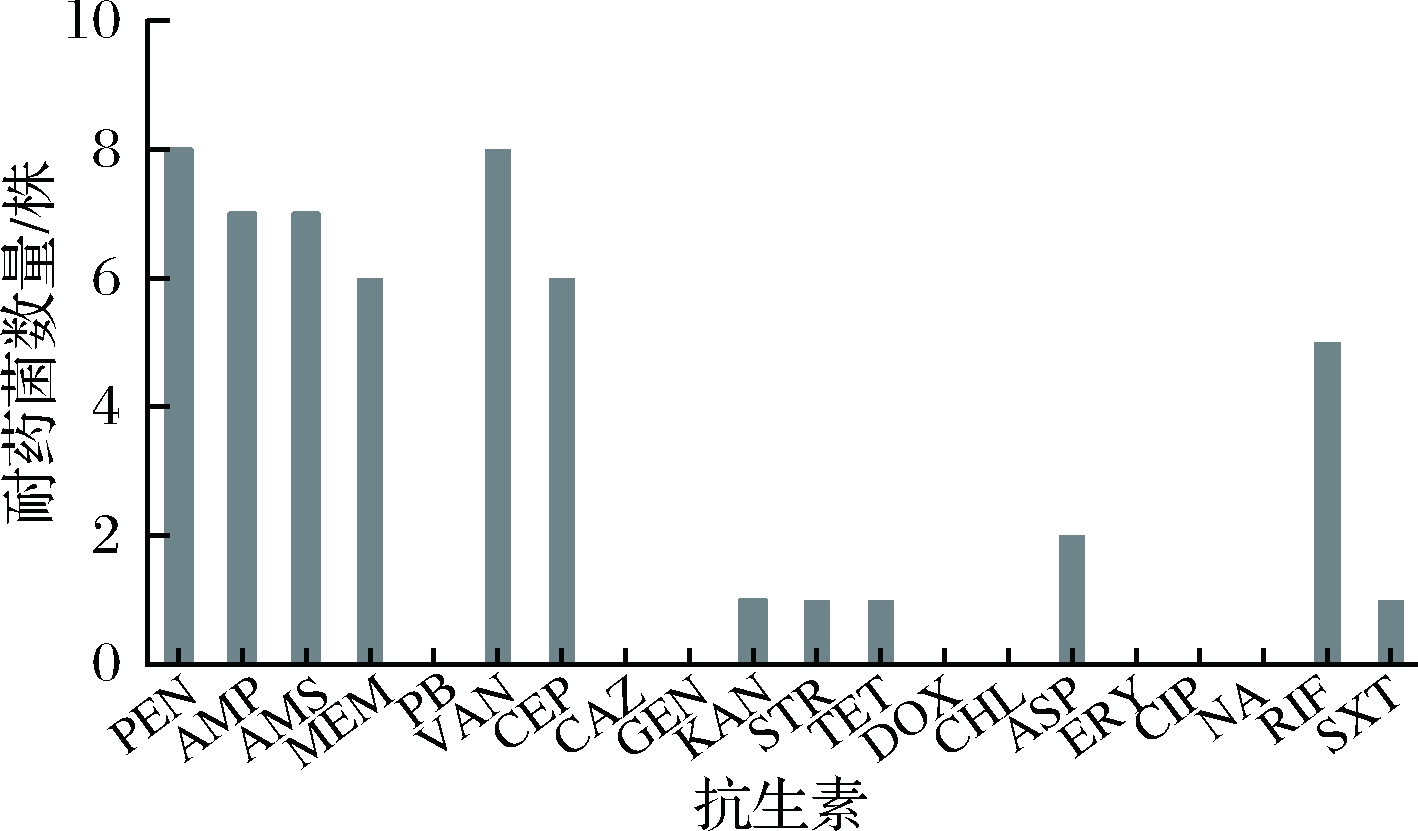
图3 不同抗生素的耐药性菌株数量分析
Fig.3 The numbers of the resistant strains against different antibiotics
2.3 菌株的多重耐药性分析
通过分析发现,所有菌株都存在多重耐药性,且耐抗生素种类都在3~10种,多重耐药性主要发生于青霉素类、β-内酰胺类、青霉烯类之间(图4)。由此可见副溶血弧菌分离菌株多重耐药性很强。同时,ATCC 17802菌株也具有多重耐药性,耐抗生素种类为5种。其中来源于鲈鱼、虾体内的分离菌株多重耐药性很强,2号菌株耐药性最强,耐抗生素种类高达10种,1、4、5、7号菌株耐抗生素8种以上,均比ATCC 17802菌株多。仅有来源于蛤的8号菌株耐3种抗生素,多重耐药性相对ATCC 17802菌株较弱。在对多重耐药谱分析中发现,来源于虾体内的4、5号菌株耐抗生素都是8种,且两者只有1种抗生素差别;来源于鱼的1、2、6、7号菌株的多重耐药无论数量还是种数差异都比较大(表4)。
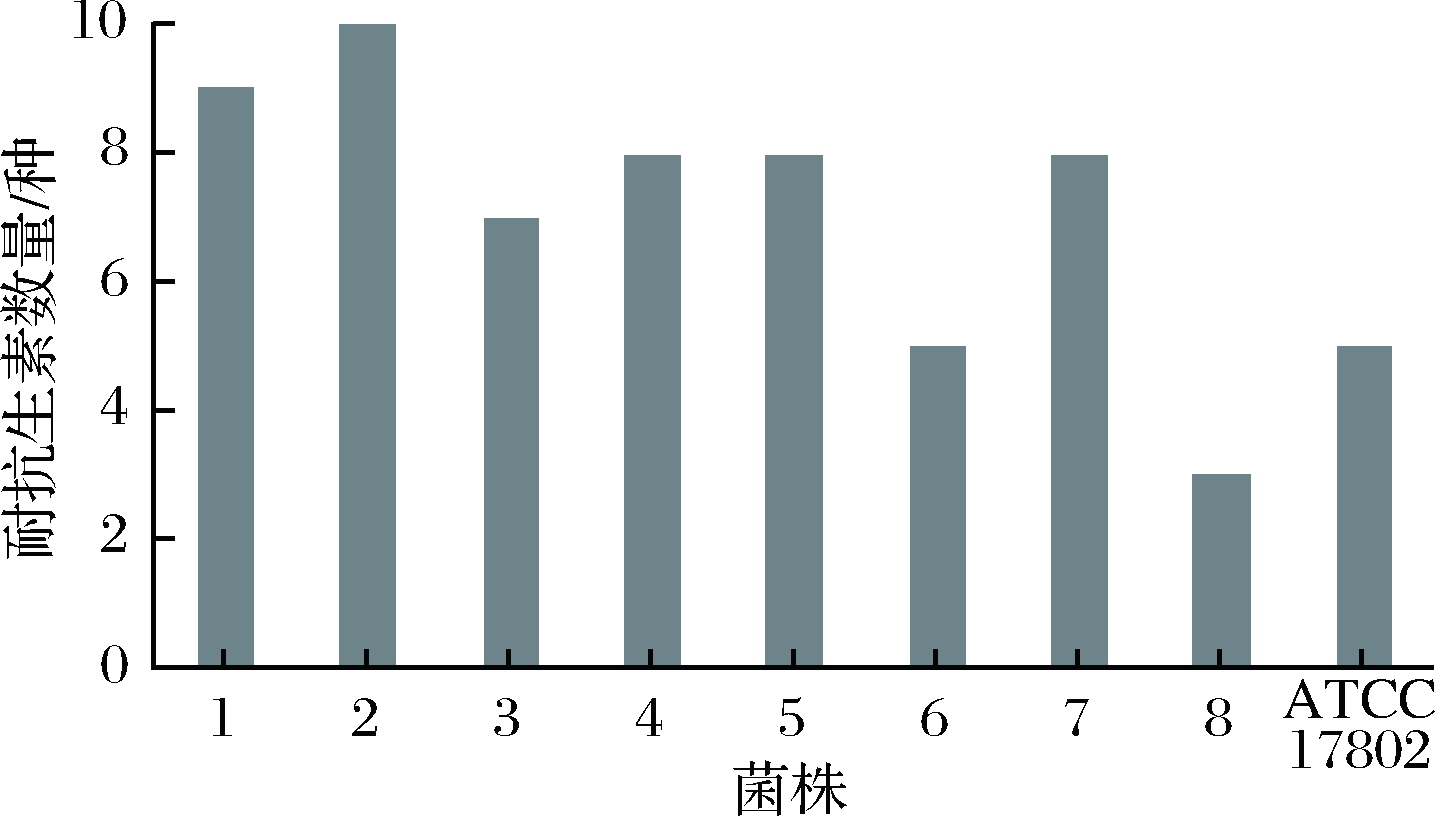
图4 副溶血弧菌分离菌株的多重耐药性分析
Fig.4 Multi-drug resistances of the isolated strains of Vibrio
parahaemolyticus
2.4 副溶血弧菌生物膜形成能力分析
副溶血弧菌标准菌株和8株分离菌株的生物膜形成能力如图5所示。所有菌株都具有一定的生物膜形成能力,但生物膜形成量有一定的差异。其中1、2、3、4、5、7号菌株形成生物膜的能力比较强,尤其是1、2、3、4号菌株;6、8号菌株生物膜成膜能力较弱。仅有8号菌株的生物膜形成能力弱于标准菌株。
表4 副溶血弧菌分离菌株的多重耐药谱
Table 4 Multidrug resistance spectrum of Vibrioparahaemolyticus strains

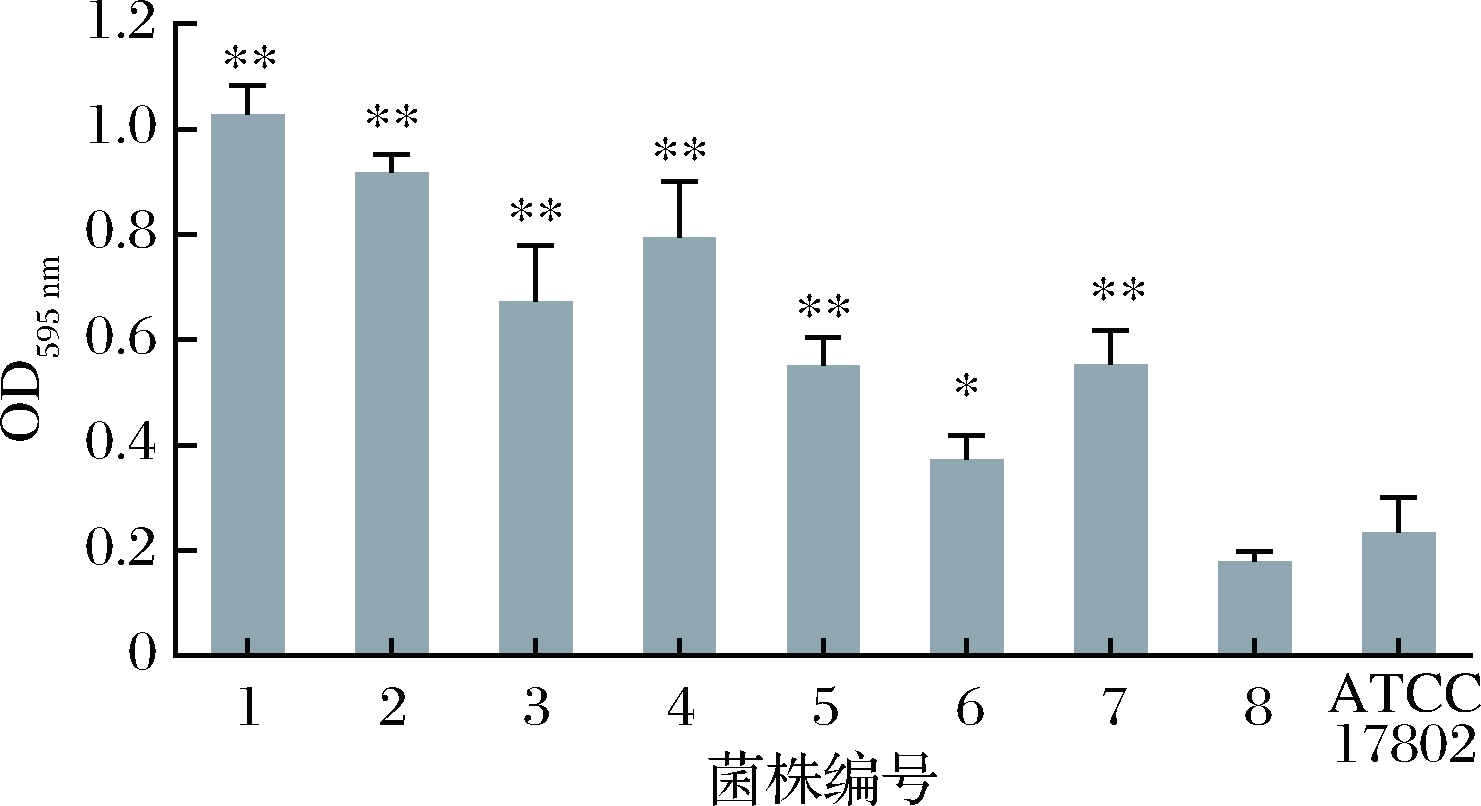
图5 副溶血弧菌生物膜形成能力
Fig.5 The biofilm formation ability of Vibrio parahaemoly-ticus strains
注:**表示与ATCC 17802菌株对比有极显著差异(p<0.01)。*表
示与ATCC 17802有显著差异(p<0.05),没有标识为无显著差异。
3 结论与讨论
3.1 菌株耐药性差异分析
副溶血弧菌耐药性具有时空特性,不同地区的菌株具有不同的耐药性。这是由于每个地区使用抗生素种类、剂量和菌群特异性导致的。本研究的结果表明,市售海产品中8株副溶血弧菌分离菌株对青霉素、万古霉素2种抗生素显示完全耐药;对美罗培南、氨苄西林、氨苄西林/舒巴坦、头孢噻吩、利福平这5种抗生素具有非常普遍的耐药性,耐药菌株在5株以上;对卡那霉素、氯霉素、四环素、强力霉素、复方新诺明5种抗生素耐药性很低,对多粘菌素B、头孢他啶、庆大霉素、氯霉素、红霉素、环丙沙星、萘啶酸等抗生素均敏感。同为广州市海产品中副溶血弧菌分离菌株,冼钰茵等[18]的研究表明,其对万古霉素、氨苄西林、链霉素的耐药率分别为100%、77.01%和85.06%,对利福平、庆大霉素、红霉素、头孢噻吩、四环素、复方新诺明、环丙沙星的耐药率在5%及以下。这与本实验结果具有比较高的一致性,但又在头孢噻吩、利福平这2种抗生素上存在区别。说明同地区的副溶血弧菌耐药性也不尽相同。而由于不同地区抗生素使用数量和剂量的差别、菌群差别,在国内的其他地区或国外地区,副溶血弧菌耐药性差异更大。卢奕等[19]研究表明,上海市水产品中副溶血弧菌对卡那霉素、多粘菌素B、环丙沙星的耐药性很高,对氨苄西林/舒巴坦、头孢噻吩、万古霉素、利福平的耐药性非常低,这与本实验的结果不同。
分离菌株对同一类抗生素的耐药性不同。如万古霉素和多粘菌素B都是糖肽类抗生素,头孢噻吩和头孢他啶都是头孢类抗生素,由于前者是第一代药物,后者是第三代药物,副溶血弧菌对前者耐药性明显比后者强。不同来源的副溶血弧菌菌株的耐药性也不同,本研究发现,来源于鱼和虾的7株菌多重耐药性强,耐抗生素种类为5~10种。相比较而言,来源于蛤的8号菌株,多重耐药性较弱,耐抗生素为3种。
3.2 细菌生物膜与耐药性
细菌生物膜是细菌生存的一种重要机制,它增强了细菌对不良环境的耐受力,尤其是耐药性。生物膜阻碍隔绝抗生素的进入,同时为膜内细胞的生存及相互交流提供稳定环境[20-21]。同时,生物膜外排泵系统[22]、特定基因变化引起的应激反应[23]、群体感应[24]、膜内细菌代谢活性下降[25]也是其耐药机制。本研究分析副溶血弧菌药敏实验结果及其生物膜形成能力发现,两者之间存在正相关。其中,1、2、3、4、5、7号菌株生物膜形成能力比较强,其多重耐药性也非常显著,耐抗生素种类为7~10种;6号和8号菌株生物膜形成能力比较差,耐抗生素种类分别为5种和3种。与标准菌株ATCC 17802相比,6号和8号菌株在形成生物膜能力差异相对小,其多重耐药性的差异也不大,而其他菌株生物膜形成能力极显著(p<0.01)强于标准菌株,其多重耐药性也十分显著。有研究表明生物膜细菌对次氯酸的抗性比浮游菌强15~3 000倍[26],致病性大肠杆菌对环丙沙星及氟苯尼考的耐药性与生物膜形成能力呈正相关[27]。
3.3 副溶血弧菌耐药机制
副溶血弧菌作为常见的食源性致病菌,威胁着人类的健康,其耐药性一直受到国内外专家学者的广泛关注。已有研究表明,副溶血弧菌耐药机制主要有:(1)可移动基因元件。质粒介导的获得耐药基因盒的整合子能够在细菌间直接进行基因水平转移。已有研究表明,副溶血弧菌存在质粒介导的Blaper-1、Blacmy-2、Blaveb-1 3种超广谱β-内酰胺酶(ESBL),能够破坏抗生素,尤其是头孢类抗生素[28-30]。(2)药物作用靶位改变。革兰氏阴性菌在β-内酰胺类抗生素作用下,其青霉素结合蛋白位点发生突变而导致与抗生素亲和力发生改变产生耐药性。(3)细胞膜渗透性改变。由于一些细菌细胞膜的膜孔蛋白少、通道小,导致抗生素(如β-内酰胺类)很难进入细胞内,这是细菌的“固有耐药性[31]。(4)外排机制是细菌形成耐药性及多重耐药性的重要因素。副溶血弧菌的外排泵主要有Vmr A、Nor M和Vme AB,其能够将大分子物质排出细胞外,帮助其产生多重耐药性[31-32]。本研究通过对市售海产品中副溶血弧菌的耐药性及其生物膜形成能力的分析,可以为后续的副溶血弧菌耐药性的深入研究提供参考资料,也为海产品的质量安全控制、防治副溶血弧菌感染提供理论依据,具有积极的现实意义。
参考文献
[1] WANG Wen, LI Min, LI Yan-bin. Intervention strategies for reducing Vibrio parahaemolyticus in seafood: A review[J]. Journal of Food Science, 2015,80(1):R10-R19.
[2] BROBERG C A, CALDER T J,ORTH K. Vibrio parahaemolyticus cell biology and pathogenicity determinants[J]. Microbes and Infection, 2011,13(12-13):992-1 001.
[3] YEUNG P S M, BOOR K J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections[J]. Foodborne Pathogens and Disease, 2004,1(2):74-88.
[4] HAN N, MIZAN M F R, JAHID I K, et al. Biofilm formation by Vibrio parahaemolyticus on food and food contact surfaces increases with rise in temperature[J]. Food Control, 2016,70:161-166.
[5] NEWTON A E, GARRETT N, STROIKA S G, et al. Increase in Vibrio parahaemolyticus infections associated with consumption of atlantic coast shellfish-2013[J]. MMWR-Morbidity and Mortality Weekly Report, 2014,63(15):335-336.
[6] 严纪文, 马聪, 朱海明, 等. 2003~2005年广东省水产品中副溶血性弧菌的主动监测及其基因指纹图谱库的建立[J]. 中国卫生检验杂志, 2006,16(4):387-391.
[7] 刘弘, 陆屹, 高围溦, 等. 2008年上海市食源性疾病监测[J]. 中国食品卫生杂志, 2011,23(2):126-131.
[8] 秦磊, 王建红, 高静, 等. 河北省唐山市2016年海产品中副溶血性弧菌监测结果分析[J]. 医学动物防制, 2018(3):205-209.
[9] XU De-feng, WANG Ya-ling, SUN Li-jun, et al. Inhibitory activity of a novel antibacterial peptide AMPNT-6 from Bacillus subtilis against Vibrio parahaemolyticus in shrimp[J]. Food Control, 2013,30(1):58-61.
[10] MOELLERING R C. NDM-1-A cause for worldwide concern[J]. New England Journal of Medicine, 2010,363(25):2 377-2 379.
[11] KUMARASAMY K K, TOLEMAN M A, WALSH T R, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK: a molecular, biological, and epidemiological study[J]. Lancet Infectious Diseases, 2010,10(9):597-602.
[12] 朱永官, 欧阳纬莹, 吴楠, 等. 抗生素耐药性的来源与控制对策[J]. 中国科学院院刊, 2015,30(4):509-516.
[13] PANTANELLA F, VALENTI P, NATALIZI T, et al. Analytical techniques to study microbial biofilm on abiotic surfaces: pros and cons of the main techniques currently in use[J]. Ann Ig, 2013,25(1):31-42.
[14] HALLSTOODLEY L, COSTERTON J W, STOODLEY P. Bacterial biofilms: from the natural environment to infectious diseases[J]. Nature Reviews Microbiology, 2004,2(2):95-108.
[15] EHRLICH G D, AHMED A, EARL J, et al. The distributed genome hypothesis as a rubric for understanding evolution in situ during chronic bacterial biofilm infectious processes[J]. Fems Immunology and Medical Microbiology, 2010,59(3):269-279.
[16] Clinical and Laboratory Standards Institute(CLSI). CLSI document M45-ACISBN 1-56238-607-7 methods for antimicrobial dilution disk dusceptibility desting of infrequently isolated for fastidious bacteria;Approved Guideline[S]. Wayne, Pennsylvania: Clinical and Laboratory Standards Institute, 2012.
[17] 魏宇清, 谢婷, 刘欢, 等. 香辛料提取物对副溶血弧菌生物膜的抑制作用[J]. 食品工业科技, 2017,38(4):101-105.
[18] 冼钰茵, 余翀, 阮荣勇, 等. 广州市售水产品副溶血弧菌和溶藻弧菌的耐药性评估[J]. 安徽农业科学, 2017,45(28):74-77.
[19] 卢奕, 陈玮祎, 刘海泉, 等. 上海市售水产品中副溶血性弧菌耐药性分析[J]. 食品工业科技, 2016,37(19):271-275.
[20] ANONYMOUS. 20th meeting of the european-tissue-repair-society, gent, BELGIUM, september 15-17, 2010 [J]. Wound Repair and Regeneration, 2010,18(6):A77-A559.
[21] DAVEY M E, O′TOOLE G A. Microbial biofilms: from ecology to molecular genetics[J]. Microbiology and Molecular Biology Reviews, 2000,64(4):847-867.
[22] MAH T T. Biofilm-specific antibiotic resistance[J]. Future Microbiology, 2012,7(9):1 061-1 072.
[23] HØIBY N, BJAMSHOLT T, GIVSKOV M, et al. Antibiotic resistance of bacterial biofilms[J]. International Journal of Antimicrobial Agents, 2010,35(4):322-332.
[24] JOLIVET-GOUGEON A, BONNAURE-MALLET M. Biofilms as a mechanism of bacterial resistance[J]. Drug Discovery Today Technologies, 2014,11:49-56.
[25] YOON S S, HENNIGAN R F, HILLIARD G M, et al. Pseudomonas aeruginosa anaerobic respiration in biofilms: Relationships to cystic fibrosis pathogenesis[J]. Developmental Cell, 2002,3(4):593-603.
[26] LECHEVALLIER M W, CAWTHON C D, LEE R G. Inactivation of biofilm bacteria[J]. Appl Environ Microbiol, 1988,54(10):2 492-2 499.
[27] 陈传荣, 韩敏敏, 张乃嘉, 等. 腹泻仔猪源致病性大肠杆菌生物膜与耐药性及毒力的相关性[J]. 微生物学通报, 2016,43(10):2 234-2 241.
[28] WU Jun, XIE Lian-yan, ZHANG Fang-fang, et al. Molecular characterization of ISCR1-Mediated blaPER-1 in a Non-O1, Non-O139 vibrio cholerae strain from China[J]. Antimicrobial Agents and Chemotherapy, 2015,59(7):4 293-4 295.
[29] LI Rui-chao, LIN Da-chuan, CHEN Kai-chao, et al. First detection of AmpC beta-Lactamase blaCMY-2 on a conjugative IncA/C Plasmid in a Vibrio parahaemolyticus isolate of food origin[J]. Antimicrobial Agents and Chemotherapy, 2015,59(7):4 106-4 111.
[30] 叶楝巍. 畜禽肉和活虾中弧菌鉴定及第三代头孢耐药机制初探[D]. 南京:南京农业大学, 2016.
[31] 叶灵琼. 副溶血弧菌耐药谱及其对头孢类药物抗性分析[D]. 上海:上海交通大学, 2011.
[32] MAKINO K, OSHIMA K, KUROKAWA K, et al. Genome sequence of Vibrio parahaemolyticus: a pathogenic mechanism distinct from that of V cholerae[J]. Lancet, 2003,361(9 359):743-749.