发酵普洱茶是以云南地理标志范围内的大叶种晒青毛茶为原料,并在地理标志范围内经过人工渥堆自然接种发酵而制成的[1],过程中大量的微生物参与了茶叶物质转化,形成了香气独特、汤色红浓、滋味醇厚的感官特征,有降胆固醇和降血脂的保健功效[2-3],产品形式主要有散茶或紧压茶。
目前,普洱茶微生物的研究主要集中于其渥堆过程中微生物多样性分析,从20世纪70年代开始,研究人员已开始关注发酵普洱茶中的微生物种类,近几年来基于高通量测序、宏基因组学的分析技术,在发酵普洱茶微生物多样性分析和功能研究方面已开展了大量的研究工作,并取得了显著成果[4]。本文通过传统可培养方法和基于高通量测序的微生物多样性分析技术跟踪分析了从晒青毛茶至发酵成熟整个过程的真菌微生物多样性情况,并采用多相鉴定方法准确鉴定了分离菌种的分类学地位,系统的展现了普洱茶整个发酵过程中真菌物种的消亡规律,为发酵普洱茶微生物安全性评价及功能分析奠定了良好的基础。
1 材料与方法
1.1 样品的采集与处理
2017年4~6月,于云南省勐海县百中堂茶庄(经纬度E100°27′13′′,N 21°58′57′′;海拔1 136 m)共采集到10份普洱茶样品,其中样品G2-G10为跟踪同一个发酵茶堆的9个不同发酵时期的采样点,BH为发酵车间环境取样,样品具体信息见表1。
表1 样品信息
Table 1 Sample information

样品采集方法:分别从普洱茶发酵堆上层、中层、下层的四周和中央各选取5个位点,利用灭菌取样钎在每个位点处取约200 g发酵茶样品,所有位点样品置于取样袋中充分混匀(共约3 kg),样品置于采样箱冰袋保鲜,运至实验室后,将样品分成2等份,1份4 ℃保存用于可培养方法分离,1份于-80 ℃保存用于基因组提取进行高通量测序;发酵环境取样为选取发酵车间东南西北及中央共5个位点,每个位点放置孟加拉红琼脂平板3块(含氯霉素0.1 g/L),静置30 min后,封口置于自封袋中带回实验室。
1.2 试剂与仪器
察氏酵母提取粉琼脂原料(CYA),麦芽提取粉琼脂(MEA),马铃薯葡萄糖琼脂培养基(PDA),孟加拉红培养基,均购自北京陆桥技术股份有限公司;真菌DNA提取试剂盒,磁珠组织DNA提取试剂盒购自美国Omega Bio-Tek公司;PCR MasterMix,DL 2000 Marker购自北京全式金生物技术有限公司;PCR引物由生工生物工程(上海)股份有限公司合成。
高速冷冻离心机(5424R),德国Eppendorf公司;PCR仪(TRIO48),德国Biometra公司;微量核酸蛋白分析仪(BioDrop-μLite),英国Biochrom有限公司;电泳仪(EC250-90),美国BIO-RAD公司;恒温培养箱(GHP-9160),上海一恒科学仪器有限公司;光学显微镜(尼康80i),日本Nikon公司;测序由北京诺赛基因组研究中心有限公司完成;Illumina Miseq高通量测序由深圳华大基因科技有限公司完成。
1.3 可培养分析方法
1.3.1 样品可培养真菌的分离纯化
四分法缩分后准确称取发酵茶样品25 g,置于225 mL灭菌生理盐水中,30 ℃ 150 r/min振荡1 h,制成10倍稀释液(10-1),取1 mL稀释液于9 mL灭菌生理盐水的试管中,依次稀释后制成 10-2、10-3、10-4、10-5、10-6等浓度梯度稀释液,选取10-3、10-4、10-5、10-6四个稀释度,用移液枪吹吸混匀后分别吸取100 μL稀释液涂布于孟加拉红平板培养基上(含氯霉素0.1 g/L),每个稀释度3个平行,置于30 ℃、45℃培养箱内倒置培养,从第2天开始根据菌落表型特征(包括大小、形状、颜色、质地、有无渗透液、有无可溶性色素等)依次挑取各种特征菌落于PDA平板上(含氯霉素0.1 g/L)进行划线分纯[5],并参照国标GB4789进行菌株计数[6],共计5 d。分纯后转接于PDA试管斜面进行保存。
发酵车间环境菌株分离:将环境取样的孟加拉红琼脂平皿置于30 ℃培养箱内倒置培养,从第2天开始根据菌落表型特征对平板上的单菌落进行分离并计数,共5 d。
1.3.2 样品中可培养真菌的多相鉴定
形态学鉴定:将分纯得到的霉菌三点接种于CYA、MEA或PDA培养基上,25 ℃培养3~14 d,酵母菌划线接种于MEA平板上,28 ℃培养3~5 d,利用单反相机拍摄菌落照片,观察菌落直径、颜色、质地、边缘是否完整、长势强弱、表面特征、反面颜色、是否产生渗出液和可溶性色素等形态特征;利用光学显微镜进行显微形态的观察,拍照并测量菌株产孢结构大小,观察产孢结构的发生方式,菌丝颜色及表面粗糙程度,孢子的颜色、大小及形状,是否产生厚垣孢子以及产生方式,是否产生有性生殖结构以及有性孢子的大小形状等特征。
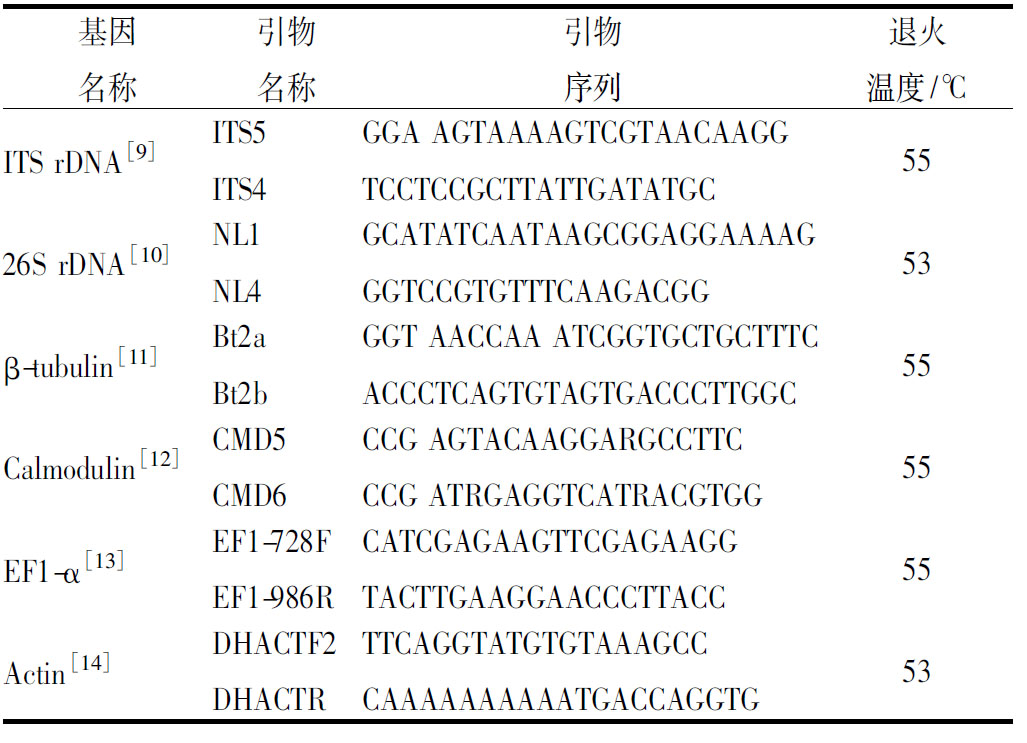
分子生物学鉴定:利用真菌基因组提取试剂盒提取分离菌株的DNA,再利用核糖体翻译间隔序列(ITS)、核糖体rDNA大亚基(26S D1/D2)、β微管蛋白基因(β-tubulin)、钙调蛋白基因(calmodulin)、肌动蛋白基因(actin)等分子标记引物进行丝状真菌和酵母菌的核酸序列扩增及测序(引物信息见表2),测序序列上传至NCBI核酸数据库,将测序结果在GenBank数据库中进行比对分析,下载与目标菌株相近种的模式菌株序列,采用ClustalX 1.83进行多序列比对[7],再用MEGA 5.0进行邻接法(neighbor joining)聚类系统发育及分子进化分析[8]。
表2 引物信息
Table 2 Primer Information
1.4 基于高通量测序的真菌多样性分析
1.4.1 DNA提取及高通量测序
将样品充分混匀,每份样品随机取样3次(每次2 g)分别用液氮充分研磨,通过磁珠组织DNA提取试剂盒提取样品基因组,利用微量核酸蛋白分析仪检测DNA浓度及纯度,将3个重复提取的基因组DNA混合,利用Illumina Miseq 2×250 bp paired-end高通量测序平台对真菌翻译间隔序列ITS1区进行双末端测序。
1.4.2 高通量测序数据分析
下机数据经过滤,除掉低质量的reads,使用软件FLASH将双末端测序的reads通过之间的overlap关系拼接成Tags[15-16];利用软件USEARCH在97%相似度下将拼接好的Tags聚类,得到OTU(Operational Taxonomic Units)的代表序列[17],通过RDP classifer软件将OTU代表序列与真菌ITS数据库(UNITE)比对进行物种注释[18];基于OTU和物种注释结果对样品中真菌物种丰度与多样性进行分析。根据各样品中物种丰度差异,利用软件QIIME采用迭代算法对样品进行聚类分析并计算样品间距离[19],以判断各样品物种组成的相似性。
2 结果与分析
2.1 样品中可培养真菌的分离与鉴定
可培养方法共分离到真菌纯培养物107株,其中霉菌75株,酵母菌32株,通过形态学与核酸序列分析可知霉菌为7个属共12种(包含潜在新种4种),酵母菌为4个属共4种;根据分离平板所对应的稀释度估算分离菌种在每克样品中的数量,菌株分离鉴定信息见表3,菌落形态及显微形态见图1。
表3 菌种分离及鉴定信息
Table 3 Isolation and identification information of strains


图1 分离菌种的菌落形态及显微形态
Fig.1 Colony morphology and microscopic morphology of isolated strains 注:照片A1至P2对应的菌种信息见表3
根据表3可知,发酵茶中数量达到103CFU/g的菌种共11种,包括8种霉菌:琉球曲霉(Aspergillus luchuensis)、新黑曲霉(Aspergillus neoniger)、烟曲霉(Aspergillus fumigatus)、伞枝横梗霉(Lichtheimia corymbifera)、青霉属(Penicillium sp.)、枝孢菌属(Cladosporium sp.)、微小根毛霉(Rhizomucor pusillus)、间座壳属(Diaporthe sp.);3种酵母:食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)、法布里德巴利酵母(Debaryomyces fabryi)、布兰克假丝酵母(Candida blankii)。其中琉球曲霉(Aspergillus luchuensis)、新黑曲霉(Aspergillus neoniger)、食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)、布兰克假丝酵母(Candida blankii)在发酵前期及后期均大量分离到,属于主发酵菌种;法布里德巴利酵母(Debaryomyces fabryi)、烟曲霉(Aspergillus fumigatus)、枝孢菌属(Cladosporium sp.)、青霉属(Penicillium sp.)主要分离于发酵前期样品;间座壳属(Diaporthe sp.)属于植物病原菌,主要分离于晒青毛茶;黄色类蝴蝶银耳(Papiliotrema flavescens)只在晒青毛茶中分离到,米根霉(Rhizopus oryzae)只在环境样品中分离到。另外,共发现新种4种:包括青霉属2种、枝孢菌1种、间座壳属1种。
2.2 样品高通量测序真菌多样性分析
2.2.1 样品检测及OTU统计分析
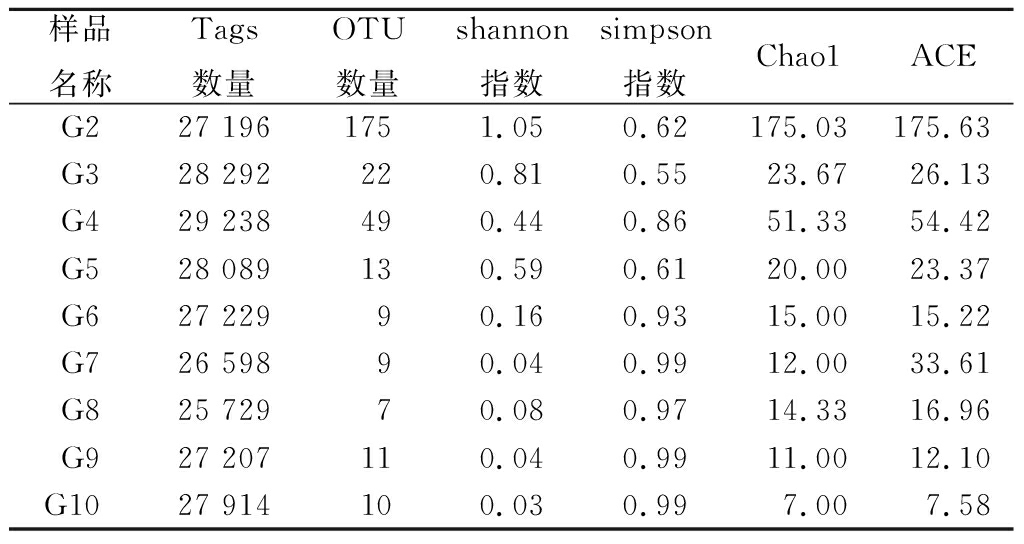
利用软件USEARCH在97%相似度下将测序得到的Tags聚类,9个样品共产生189个OTU,每个样品测序得到的Tags数量、OTU数量及香浓指数见表4。通过shannon和simpson指数反映出普洱茶发酵过程真菌物种多样性,Chao1和ACE指数反映出普洱茶发酵过程中真菌物种丰度,数值越大丰度越高[20],由表4可知发酵前期样品真菌多样性较高,其中晒青毛茶(G2)中微生物数量最多,香浓指数最高。
表4 各样品测序结果Tags数量、OTU数量及香浓指数
Table 4 The number of Tags, OTU and the fragrance index of the sequencing results of each sample
2.2.2 基于ITS1序列分析各样品中真菌属级单元多样性及丰度
将上述聚类得到的189个OTU的代表序列与真菌ITS数据库(UNITE)比对,进行物种注释,189个OTU归属于69个属(详见表5),各样品中属水平的多样性分布见图2。由表5和图2可知,发酵普洱茶样品中真菌主要以子囊菌门(Ascomycota)为主,担子菌门(Basidiomycota)和接合菌门(Zygomycota)物种多样性和丰度均较低,大部分真菌主要分布在发酵前期。各样品中丰度大于1%的属级分类单元包括:芽生葡萄孢酵母属(Blastobotrys)、假丝酵母属(Candida)、德巴利酵母属(Debaryomyces)、枝孢菌属(Cladosporium)、曲霉属(Aspergillus)、青霉属(Penicillium),其中芽生葡萄孢酵母属(Blastobotrys)和假丝酵母属(Candida)真菌在普洱茶整个发酵过程均能分析到,将二者的ITS1代表序列进行NCBI Blast比对,被分别鉴定为“食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)”和“布兰克假丝酵母(Candida blankii)”,其中“食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)”丰度高达70%以上,为绝对优势种,与可培养方法结果一致;德巴利酵母菌属(Debaryomyces)在发酵前期较优势,发酵后期少量分离到;枝孢菌属(Cladosporium)主要存在于晒青毛茶中,丰度达到13%;根毛霉属(Rhizomucor)真菌在发酵前期和后期均分离到,发酵中期第1次翻堆时丰度较高;样品G2(晒青毛茶)真菌多样性最高,G3(发酵3 d)、G4(第1次翻堆)真菌多样性较高。
表5 各样品中真菌属级分类单元的丰度 单位:%
Table 5 Abundance of fungi classification units in each sample

续表5

注:“-”表示样品中未检测到该属真菌。
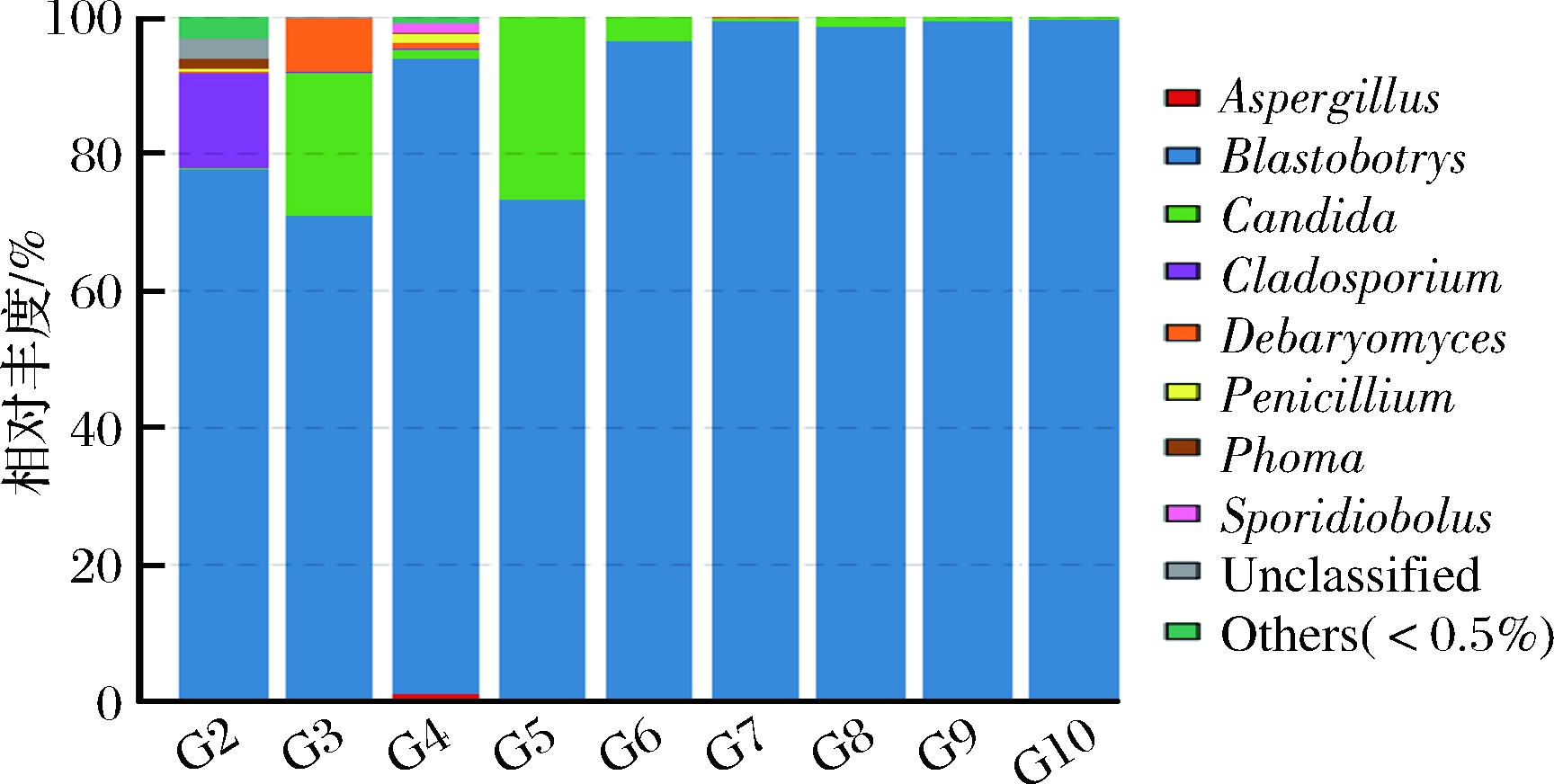
图2 真菌属级分类单元在各样品中分布
Fig.2 The taxonomic composition distribution in samples at genus-level
2.2.3 样品间物种组成聚类分析
各样品间物种组成聚类分析结果详见图3,样品间越靠近,枝长越短,说明2个样品的物种组成越相似,由图3可知样品G2(晒青毛茶)与其他发酵阶段的样品中真菌多样性相差最远,发酵阶段前期样品G3、G4、G5、G6聚为一支,较相近;发酵后期G7、G8、G9、G10聚为一支,较相近。
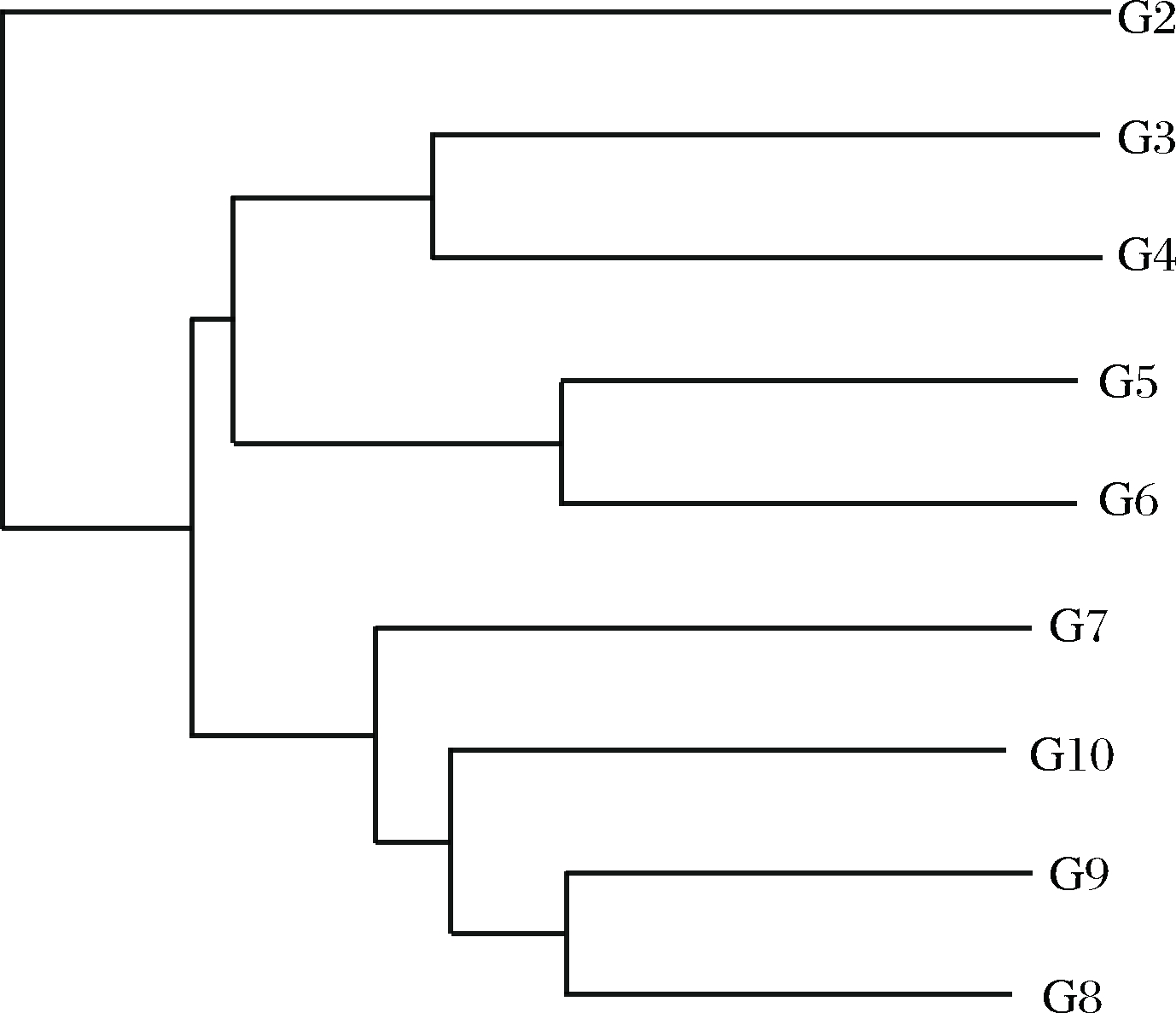
图3 样品聚类分析图
Fig.3 Clustering results of samples
3 讨论
本研究通过可培养方法从勐海百中堂茶庄普洱茶整个发酵过程分离到可培养真菌107株,共11个属,16个种,优势种包括琉球曲霉(Aspergillus luchuensis)、新黑曲霉(Aspergillus neoniger)、烟曲霉(Aspergillus fumigatus)、青霉菌(Penicillium sp.)(潜在新种)、枝孢菌(Cladosporium sp.)(潜在新种)、微小根毛霉(Rhizomucor pusillus)、伞枝横梗霉(Lichtheimia corymbifera)、食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)、法布里德巴利酵母(Debaryomyces fabryi)、布兰克假丝酵母(Candida blankii)。与传统可培养方法比,高通量测序更全面地展现了勐海发酵普洱茶样品的真菌群落结构,揭示发酵过程真菌微生物主要为子囊菌,担子菌和接合菌次之,高通量测序共分析到真菌69个属(包括子囊菌54属,担子菌13属,接合菌2属),共189个OTU,包含了可培养方法所有分离菌种对应的属级分类单元,根据庞雄飞等的优势属划分原则[21],可知“芽生葡萄孢酵母属(Blastobotrys)”为整个发酵过程优势属;“假丝酵母属(Candida)”在发酵3 d和15 d的样品中丰度达到20%,为前期优势属;“枝孢霉属(Cladosporium)”在晒青毛茶样品中为优势属,丰度达到13.79%,3种优势属均与可培养方法结果相吻合。由高通量测序结果可知晒青毛茶中微生物多样性最高,且大部分菌种丰度均小于1%,其中部分种可能只是以死菌的形式存在于毛茶中,而导致可培养方法没有分离得到。另外可培养方法显示曲霉菌为普洱茶发酵过程优势菌,但高通量技术分析结果显示曲霉菌丰度较低,且在后期没有分析到该属真菌,可能是曲霉菌后期凋亡较快,基因组降解严重而导致的。综合考虑,高通量技术更能全面宏观的反映普洱茶发酵过程微生物多样性及物种消亡规律,而可培养方法分离到一些丰度较高的优势菌种,有利于菌种的进一步开发研究。
综合2种方法研究结果可知,发酵前期真菌多样性较后期高,其中法布里德巴利酵母(Debaryomyces fabryi)、枝孢菌(Cladosporium sp.)、青霉菌(Penicillium)主要分布于普洱茶发酵前期,法布里德巴利酵母主要产酯酶,枝孢菌和青霉菌可产纤维素酶、脂肪酶可水解毛茶基质纤维素[22];曲霉菌(Aspergillus)主要分布于普洱茶发酵前期及中期,代谢产生有机酸以及多酚氧化酶、糖化酶、果胶酶、纤维素酶、单宁酶等酶类,是前中期茶叶粗纤维组织软化、促进大分子物质转化的重要菌种[22-23];布兰克假丝酵母(Candida blankii)、伞枝横梗霉(Lichtheimia corymbifera)、微小根毛霉(Rhizomucor pusillus)主要分布于普洱茶发酵中期,该3个菌种均为耐高温菌种[24-26],推测其在普洱茶发酵中期的高温环境下发挥重要作用;食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)属于整个发酵过程优势种,其可培养数量可达到107CFU/g,发酵后期丰度达到99%以上,该菌种耐高温,发酵可产生蛋白酶、酯酶、没食子酸脱羧酶[27],对普洱茶发酵过程的物质转变及风味形成具有重要的贡献[28]。此外,研究结果显示优势种属在晒青毛茶原料中均有分布,少部分分离自发酵环境,推测普洱茶发酵的优势真菌主要来源于晒青毛茶原料。
本研究分离到大量的曲霉属黑色组真菌(Aspergillus section Nigri),其与普洱茶渥堆过程中茶堆表面可见的大量黑色曲霉菌的表观现象相吻合,多相鉴定结果显示这些菌种应为琉球曲霉(Aspergillus luchuensis)和新黑曲霉(Aspergillus neoniger),二者在普洱茶中均为首次报道,与已有报道的普洱茶发酵过程中优势种为黑曲霉(Aspergillus niger)[29-30]、塔宾曲霉(Aspergillus tubingensis)[31]结果均不一致,因为已有的文献研究中只应用了真菌通用的ITS rDNA序列进行分析,而“琉球曲霉、新黑曲霉、黑曲霉、塔宾曲霉”四者在ITS水平序列相似性为100%,无法区分开,需结合微管蛋白基因(β-tubulin)和钙调蛋白基因(calmodulin)才可区分四者。另外,法布里德巴利酵母(Debaryomyces fabryi)在普洱茶中属于首次报道,其需通过26S rDNA序列结合肌动蛋白基因(actin)才可以准确鉴定至种水平,否则容易误鉴定为“汉逊德巴利酵母(Debaryomyces hansenii)”。
发酵普洱茶安全问题一直备受关注,大量研究报道普洱茶易受黄曲霉毒素污染[32-33],本研究未分析到产黄曲霉毒素的菌种,但本研究分离到的烟曲霉(Aspergillus fumigatus)是公认的临床致病菌,其主要导致侵袭性肺曲霉病[34],且该菌种可产生烟曲霉素、胶霉毒素等致病真菌毒素[25],已有大量文献报道该菌种存在于发酵普洱茶中[35],本研究显示在发酵7 d的样品中分离率达到每克104 CFU/g的菌落数,建议发酵过程中参与翻堆工作的茶厂工人做好相关防护,发酵完成的普洱熟茶应进行相关的霉菌毒素检测。另外,微小根毛霉、伞枝横梗霉等菌种为机会致病菌,可引起接合菌病[22,25],免疫低下者应进行相关防护。而优势菌“食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)”、“布兰克假丝酵母(Candida blankii)”、“琉球曲霉(Aspergillus luchuensis)”均未见产毒素和致病性报道[24,36]。
普洱茶发酵过程中,大量的微生物参与了物质转化,对这些微生物分类学名称的准确鉴定是发酵普洱茶微生物安全性评价及功能性分析的重要基础,弄清楚这些微生物的功能关系是普洱茶自然开放式发酵工艺改进的前提条件。本文分析到的普洱茶真菌微生物的具体功能及安全性有待进一步研究。
参考文献
[1] 中华人民共和国国家质量监督检验检疫总局.GB/T 22111—2008,地理标志产品普洱茶[S].北京:中国标准出版社,2008:2-5.
[2] 周红杰,秘鸣,韩俊,等.普洱茶的功效及品质形成机理研究进展[J].茶叶,2003,29(2):75-77.
[3] 赵龙飞,周红杰,安文杰.云南普洱茶保健功效的研究[J].食品研究与开发,2005,26(2):114-118.
[4] 王辉,李亚莉,苏丹,等.宏基因组学在普洱茶微生物研究中的应用[J].食品安全质量检测学报,2015(6):2 195-2 220.
[5] 白飞荣,刘洋,李辉,等.香山黄栌枯萎病叶片真菌的分离与鉴定[J].生物学通报,2014,49(5):51-53.
[6] 中华人民共和国国家卫生和计划生育委员会.GB 4789.15—2016,食品安全国家标准:食品微生物学检验,霉菌和酵母计数[S].北京:中国标准出版社,2016:3-5.
[7] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The clustalx windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4 876-4 882.
[8] TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution, 2011,28(10): 2 731-2 739.
[9] WHITE T J,BRUNS T D,LEE S B,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M].Cambridge:Academic Press inc,1990:315-322.
[10] O'DONNELL K.Fusarium and its near relatives fungal holomorph mitotic meiotic & pleomorphic speciation in fungal systematic[M].California: International,1993:225-233.
[11] DONALDSON G C,BALL L A,AXELROOD P E,et al.Primer sets developed to amplify conserved genes from filamentous ascomycetes are useful in differentiating Fusarium species associated with conifers[J].Applied & Environmental Microbiology,1995,61(4):1 331-1 340.
[12] HONG S B,GO S J,SHIN H D,et al.Polyphasic taxonomy of Aspergillus fumigatus and related species[J].Mycologia, 2005,97(6):1 316-1 329.
[13] CARBONE I,KOHN L M.A method for designing primer sets for speciation studies in filamentous ascomycetes[J]. Mycologia,1999,91(3):553-556.
[14] NGUYEN H V,GAILLARDIN C,NEUVéGLISE C.Differentiation of Debaryomyces hansenii and Candida famata by rRNA gene intergenic spacer fingerprinting and reassessment of phylogenetic relationships among D.hansenii, C.famata, D.fabryi, C.flareri (=D.subglobosus) and D.prosopidis[J].Fems Yeast Research,2009,9(4):641-662.
[15] FADROSH D W,BING M,GAJER P,et al.An improved dual-indexing approach for multiplexed 16S rRNA gene sequencing on the Illumina MiSeq platform[J].Microbiome,2014,2(1):6.
![]() T,SALZBERG S L.Flash:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics,2011,27(21):2 957.
T,SALZBERG S L.Flash:fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics,2011,27(21):2 957.
[17] EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013, 10(10):996-998.
[18] ABARENKOV K,NILSSON R H,LARSSON K H,et al.The UNITE database for molecular identification of fungi-recent updates and future perspectives[J].New Phytologist,2010,186(2):281-285.
[19] CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[20] SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur: open-source, platform-independent, community -supported software for describing and comparing microbial communities[J].Applied&Environmental Microbiology,2009, 75(23):7 537-7 541.
[21] 庞雄飞,尤民生.昆虫群落生态学[M].北京:中国农业出版社,1996:77-103.
[22] 周红杰,龚加顺.普洱茶与微生物[M].昆明:云南科技出版社,2010:12-163.
[23] 董文明,谭超,付晓萍,等.5种成品普洱茶中微生物的分离及其产酶特性研究[J].食品科技,2013(6):22-25.
[24] KURTZMAN C P, FELL J W, BOEKHOUT T. The yeasts: A taxonomic study[M]. Amsterdam,Elsevier,2011:962-1 031.
[25] PITT J I,HOCKING A D.Fungi and food spoilage[M].Springer Dordrecht Heidelberg London New York,Springer,2009:1-524.
[26] 杨瑞娟,吕杰,严亮,等.普洱茶渥堆发酵中嗜热真菌的分离和鉴定[J].茶叶科学,2011,31(4):371-378.
[27] MEIER A K,WORCH S,BÖER E,et al.Agdc1p-a gallic acid decarboxylase involved in the degradation of tannic acid in the yeast Blastobotrys (Arxula) adeninivorans[J].Frontiers in Microbiology,2017,8:1-16.
[28] MICHIHARU A,NAOHIRO T,YOSHITO I,et al.Characteristic fungi observed in the fermentation process for Puer tea[J]. International Journal of Food Microbiology,2008,124:199-203.
[29] 马燕,赵明,周玲,等.普洱茶样中“金花”微生物的分离鉴定[J].茶叶通讯,2011,38(2):17-20.
[30] 方祥,陈栋,李晶晶,等.普洱茶不同贮藏时期微生物种群的鉴定[J].现代食品科技,2008,24(2):105-108.
[31] 赵振军,童华荣,周黎,等.普洱茶中真菌合成洛伐他汀的能力分析[J].食品科学,2012,33(12):173-176.
[32] 陈宗懋.普洱茶中的黄曲霉素问题[J].茶博览,2017(9):76-78.
[33] 杨质高.检测方法不同致普洱茶黄曲霉毒素检测结果差异悬殊[J].普洱,2017(10):94-96.
[34] VILLAR C C,DONGARIBAGTZOGLOU A.Immune defence mechanisms and immunoenhancement strategies in oropharyngeal candidiasis[J].Expert Reviews in Molecular Medicine,2008,10(10):e29.
[35] 胡捷.不同季节普洱茶渥堆过程动态分析[D].广州:华南理工大学,2013:16-36.
[36] MOGENSEN J M,VARGA J,THRANE U,et al. Aspergillus acidus from Puerh tea and black tea does not produce ochratoxin A and fumonisin B2[J].International Journal of Food Microbiology,2009,132(2/3):141-144.