脂肪酶(Lipase, EC3.1.1.3,甘油酯水解酶)是一类特殊的酯键水解酶,作为生物催化剂具有底物专一性好,立体选择性强和反应条件温和,产品易分离等优点[1]。因此,利用脂肪酶作为催化剂进行高选择性和高效地有机合成是当今合成化学的热点,更因为其对环境友好和节约能源等特点,被人们称为“绿色化学”而备受瞩目。枯草杆菌(Bacillus subtilis)脂肪酶LipA为分泌性蛋白,具有高度专一的立体选择性,是迄今为止发现的分子量最小的脂肪酶之一,这些特点使枯草杆菌脂肪酶近年来受到关注。然而,在酶蛋白的合成表达过程中,细胞能量供应不足或产生较多有毒中间代谢产物,都会影响酶的持续高效合成[2]。因此本研究从整个代谢网络入手,从代谢层次深入认知各种代谢物与目标产物间的关系。
代谢通量分析(metabolic flux analysis,MFA)又称代谢流分析[3],是一种计算流经各种代谢途径的物质流的技术,用于描述不同途径的相互作用和围绕代谢分支点的物质流分布,在代谢工程中占有重要地位。通过计算不同途径或不同条件下的代谢通量分布,可以表征细胞的代谢能力[4],发现遗传修饰对细胞代谢状态的影响,从而为进一步更加合理地遗传改造提供理论依据。近年来,MFA在燃料乙醇、1,3-丙二醇、有机酸(乳酸、丁二酸等)、维生素、氨基酸(谷氨酸、赖氨酸等)和抗生素等重要发酵产品,新药开发以及环境生物技术方面都取得重要进展。但是将MFA应用于蛋白合成过程的仅有少数研究。CALIK[5]在1999年发表了第1篇关于蛋白合成代谢通量分析的研究,考察了B. licheniformis 的EMP途径、PP途径、TCA循环和氨基酸合成途径等,建立了B. licheniformis的中间代谢途径模型。SONG[6]等基于代谢流分析开发了双阶段供养策略,提高了B. subtilis脂肪酶产量。
本文通过建立代谢流量平衡模型[7],分析了诱导前后重组大肠杆菌合成脂肪酶过程中代谢通量分布,确定影响酶合成的关键节点、关键途径,为脂肪酶高表达和高比活发酵奠定基础。
1 材料与方法
1.1 脂肪酶LipA重组菌的构建
根据B. subtilis 168染色体上lip A基因核酸序列,设计PCR引物lip A-up (5′-CCGGAATTCATGGCAAAGAAAGACGAACAC-3′)和lip A-down (5′- CCCAAGCTTTGCTTGTGCTTGACG-3′),在lipA-up和lip A-down中分别引入Sal Ⅱ和Hind Ⅲ酶切位点。以B. subtilis 168基因组为模板,PCR扩增约1 kb的lip A基因片段。PCR产物与表达载体pET22b,分别用Eco RI和Hind Ⅲ双酶切后进行连接反应。连接产物转化E. coli Top10,在含有100 μg/mL氨苄青霉素的LB平板上随机挑取单菌落进行验证,筛选转化子。经提取质粒、酶切电泳以及PCR检测,证明得到了含有lipA的重组质粒pET22b-lipA。重组质粒再转化表达宿主E. coli BL21,获得重组菌BL21- pET22b-lipA。
1.2 培养基及培养方法
种子活化:接种1环斜面活化菌株接入装有50 mL LB培养基(LB培养基g/L:蛋白胨 10,酵母粉5,NaCl 10)的三角瓶中,37 ℃、200 r/min培养12 h。
一级种子培养:将种子以5%的接种量接入250 mL装有50 mL M9培养基(M9培养基g/L: Na2HPO4·7H2O 17.096, KH2PO4 3, NaCl 0.5, NH4Cl 1, MgSO4 0.493, CaCl2 0.011 1, 葡萄糖4, 自然pH)的三角瓶中, 37 ℃、200 r/min发酵6 h。
二级种子培养:将一级种子以5%的接种量接入250 mL装有50 mL M9培养基的三角瓶中,37 ℃、200 r/min发酵10 h。
5 L发酵罐培养:5 L发酵罐装液量为3 L,将5%二级种子培养液接入含有M9培养基的发酵罐,用2 mol/L NaOH控制发酵过程pH为7.0,于37 ℃,搅拌转速200 r/min,通气量1.0 vvm条件下培养;待发酵液OD600达到0.6~0.8时,加终浓度为0.8 mmol/L IPTG诱导菌体产酶,在30 ℃条件下产酶。
1.3 分析方法
葡萄糖的测定[8]:通过SBA-40C生物传感仪测定。
生物量的测定[9]:采用紫外分光光度计测定OD600下吸光值,去离子水做空白,并记录数据。
脂肪酶活力测定方法[10]:以对硝基苯酚棕榈酸酯(p-nitrophenylpalmitate,pNPP)为底物的分光光度法测定脂肪酶酶活。
有机酸浓度的测定[6]:采用高效液相色谱法,色谱柱:Sepax C18(250 mm×4.6 mm, 5μm);流动相:C:10 mmol/L磷酸(pH=2.14);D:乙腈。检测波长:210 nm;进样量:20 μL;柱温:25 ℃。
氨基酸浓度的测定[6]:发酵液上清通过衍生化,采用高效液相色谱法,色谱柱:Sepax AA专用柱(250 mm×4.6 mm,5 μm);流动相:A:0.1 mol/L,pH 6.5醋酸钠溶液(含7% 乙腈);B:乙腈(含25% 水)。流速:1.0 mL/min;检测波长:254 nm;进样量:20 μL;柱温:36 ℃。
菌体干重的测定[11]:取5mL发酵液,离心8 000 r/min,5 min收集菌体,用蒸馏水洗涤2~3次,离心后菌体60 ℃烘干至恒重,称重。
蛋白含量的测定[12]:采用Bradford法测定蛋白质浓度,标准蛋白为牛血清蛋白(BSA)。
1.4 代谢通量分析方法
根据E.coli的生化反应网络和KEGG等数据库资源[13],结合E.coli的生长代谢特性,建立了E.coli代谢合成脂肪酶的反应网络(图1)及其代谢通量平衡模型,包括主要的碳骨架代谢途径和氨基酸生物合成途径。在建立网络模型时做了以下假设和简化处理:1)细胞中的中间代谢物处于拟稳态,即瞬间浓度变化为0;2)只考虑主要中心碳代谢反应的物流平衡[14];3)无分支点的几个连续反应简化为一个反应式,所有反应包括生物量合成都按照固定比例进行;4)能量供需平衡,即合成菌体与产物所需的能量和EMP、HMP及TCA循环产生的能量总数相等。E.coli中NADH和FADH2都可以通过电子传递链生成ATP[15];5)葡萄糖进入细胞均需消耗ATP;6)脂肪酶LipA的反应方程是根据其氨基酸组成确定的[16],蛋白质合成所需要的能量为4.306 μmol ATP/μmol氨基酸[17],LipA的蛋白浓度通过SDS-PAGE电泳分析得到;7)在代谢过程中,由于一部分碳源用于细胞的维持、产能、CO2释放以及分泌其它一些未知的代谢产物等,造成碳源的不平衡现象,该部分通量在代谢网络通量的计算时,并入生物量通量(VBiomass)中,生物量反应方程根据文献[18]建立。模型中包含的反应如表1,涉及到的简称如表2所示。
代谢通量分析根据代谢路径中各反应的计量关系以及底物、产物的通量和细胞组成等确定代谢网络的通量分布。对于胞内代谢物采用拟稳态假设:假设细胞内的中间代谢物均处于拟稳态[19],即其整个浓度变化速率为0;对于胞外代谢产物则通过检测发酵液中该产物的比生产速度得到。于是可以得到Av=rmet,其中A(49×50)为化学计量系数矩阵,v(49×1)为代谢通量向量,rmet为代谢物净积累速率向量(50×1)[20]。整个代谢网络包含49个代谢反应,其中中间代谢物为45个,则自由度为4。该代谢网络中可测速率有5个,即葡萄糖消耗速率以及乳酸、乙酸、lipA和生物量的生成速率。考虑到氨基酸是脂肪酶组成的重要组分可能存在胞外分泌,因此也检测了胞外20种基本氨基酸浓度变化。由于可测定的速率大于自由度,则该体系为超定系统,可用最小二乘法求得简单解,利用MATLAB 7.1软件求得代谢分布。在此代谢网络中以100 mmol/(g·DCW·h)的葡萄糖的碳摩尔量为计算基准,所有代谢通量v的单位均为mmol/(g·DCW·h)[21]。
表1 代谢模型中包含的反应
Table 1 Reaction contained in the metabolic models

表2 代谢模型中的简称
Table 2 Abbreviations in metabolic models

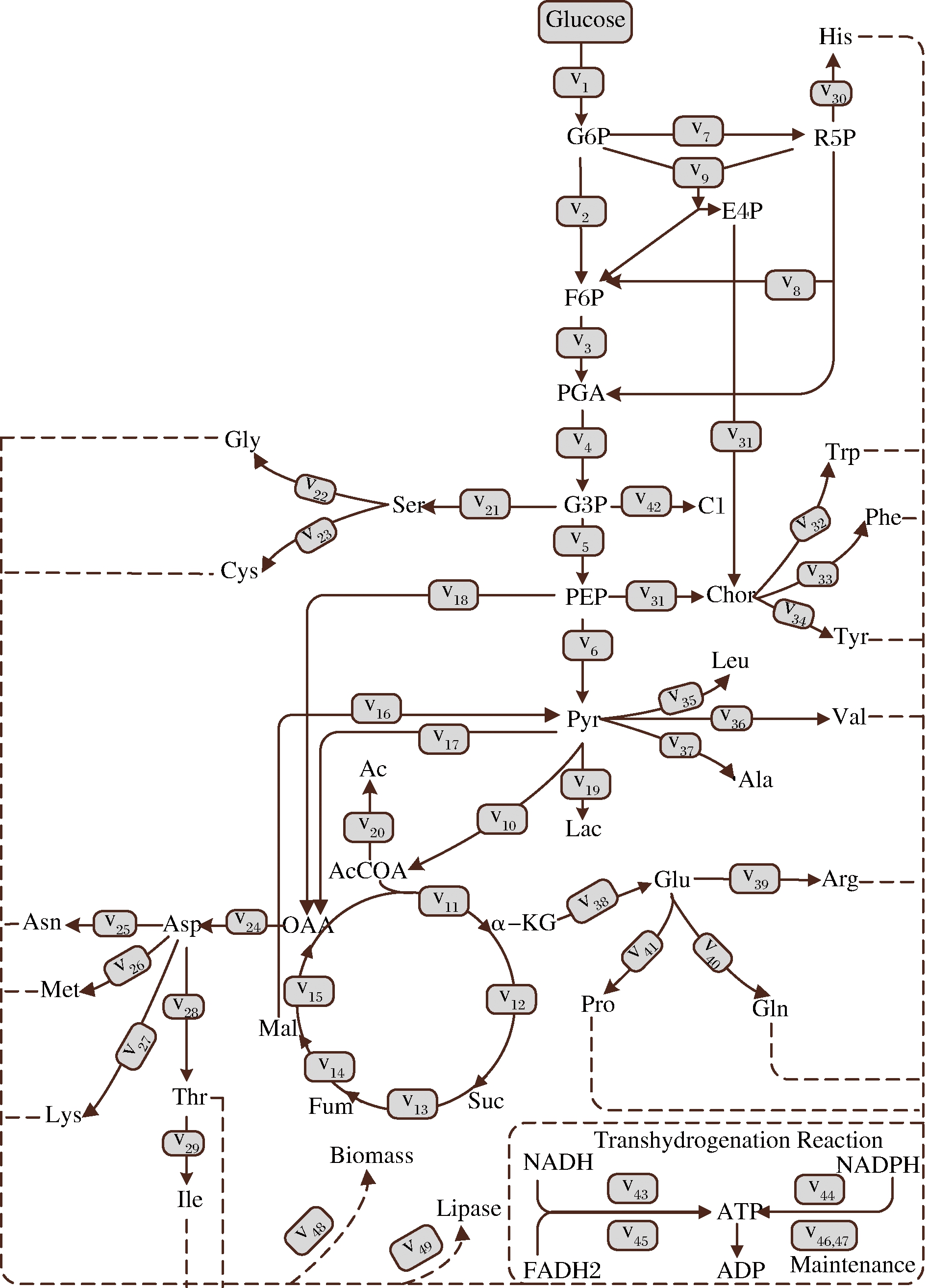
图1 重组大肠杆菌产脂肪酶LipA代谢反应网络
Fig.1 Metabolic reaction network for LipA production in Recombinant E. coli
2 结果与讨论
2.1 5 L罐中重组大肠杆菌产LipA的发酵性能研究
在发酵过程中(图2),菌体没有明显延迟期,在0~4 h葡萄糖的消耗速率一直维持在0.46 g/(L·h)。3 h时,OD600达到0.6~0.8,加入诱导剂IPTG进行诱导产酶,此时葡萄糖的消耗速率开始降低,可能是由于IPTG的毒性造成的。加入诱导剂后,E.coli开始大量产酶,5 h时比活达到最高值18.11 U/mg。随着发酵的进行,脂肪酶比活开始降低,8 h时比活降至14.14 U/mg。
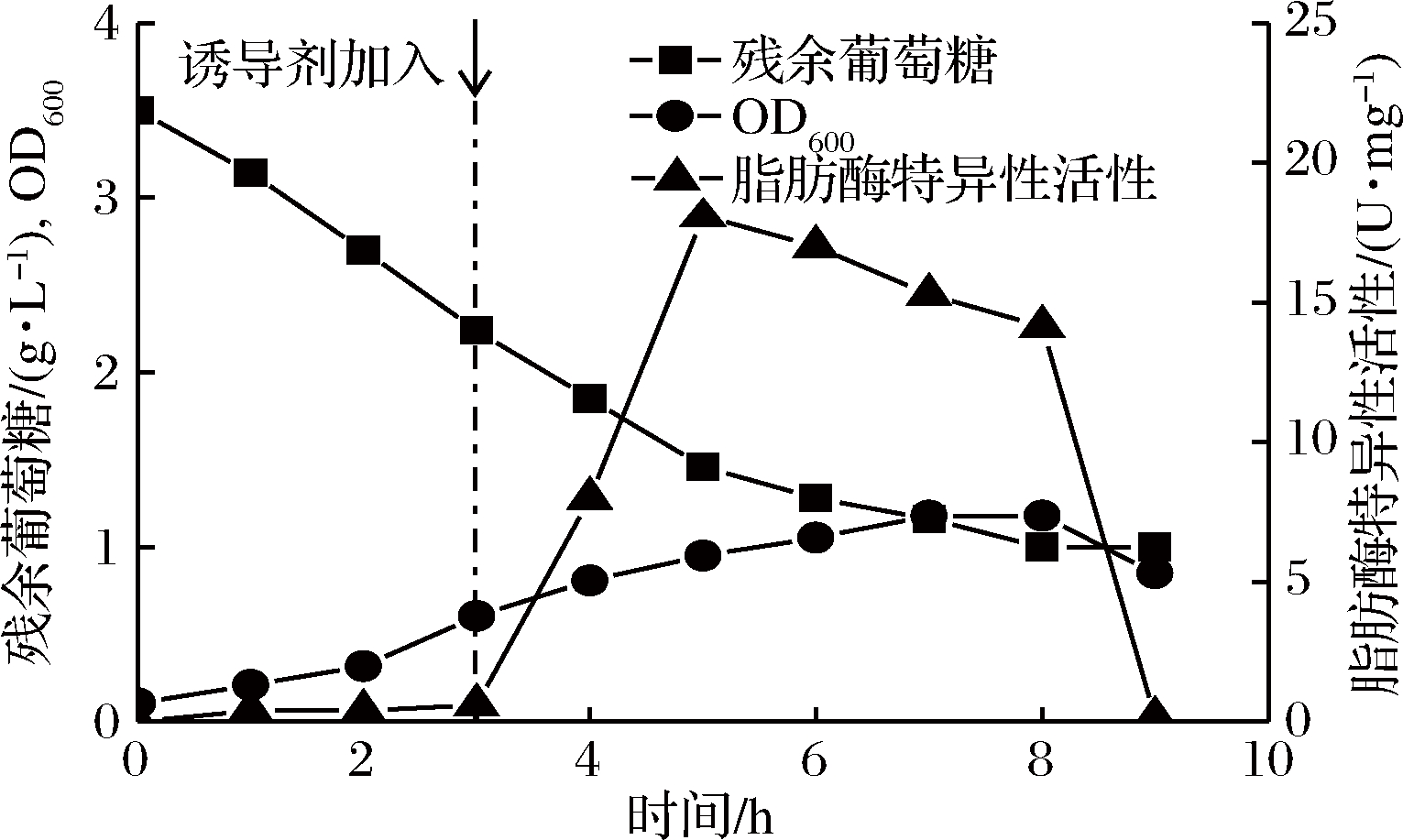
图2 E.coli在5 L罐中发酵产酶曲线
Fig.2 Enzyme production curve of E.coli fermentation in 5 L fermenter
采用SDS-PAGE对菌体胞内蛋白进行分析,结果如图3所示。LipA脂肪酶的分子质量为19.1 kDa。1~3 h时,在14.3~20.1 kDa之间无特征条带出现,说明未诱导时宿主菌E.coli BL21不产LipA。发酵3 h加入诱导剂后,LipA开始表达。随着诱导时间的延长,LipA的表达量在7 h时达到最大(LipA占总蛋白含量为3%),然而到了9 h时各组分蛋白条带不清晰且LipA无条带。可能是因为在对数后期和平稳期时细胞内蛋白酶的积累,分解了总蛋白,因此导致了包含脂肪酶在内的蛋白含量的下降[24]。
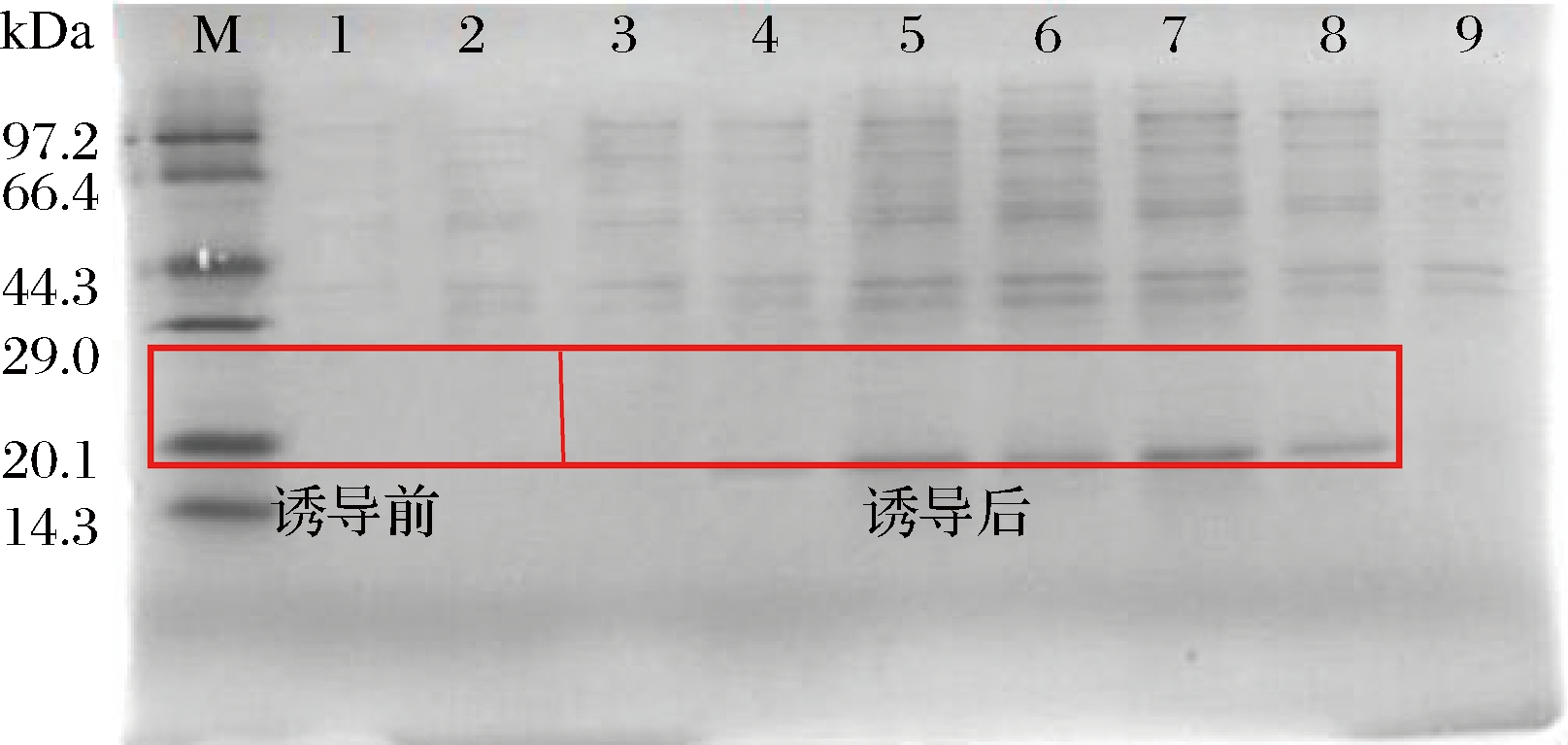
M-protein marker;1~9-E. coli BL21 pET2 2b-lipA 1~9 h发酵粗酶液
图3 重组大肠杆菌诱导产脂肪酶的SDS-PAGE分析图
Fig.3 SDS-PAGE analysis of LipA production induced by recombinant E. coli
2.2 诱导对有机酸及氨基酸的影响
发酵液中有机酸和氨基酸等杂酸含量是微生物发酵过程的另一个重要指标,通过对其分析,能够从另一个角度更加全面的了解微生物的发酵过程。可以检测到的副产物如图4,图5所示。通过与20种基本氨基酸标品色谱图对照,显示发酵液能检测到精氨酸(Arg)、缬氨酸(Val)、亮氨酸(Leu)和脯氨酸(Pro)共4种氨基酸。氨基酸浓度的变化随着发酵的进行呈现上下波动的趋势。其中,浓度最高的是缬氨酸,最高浓度达到22 mmol/L。其他氨基酸的浓度都基本在5 mmol/L以下。分析为氨基酸在发酵过程中不断生成又不断消耗造成的,并且在发酵中后期,可能由于营养物质匮乏,缬氨酸被用作为氮源促进菌体生长[26],所以在6 h时缬氨酸浓度会降低。同时发酵中后期,由于呼吸作用细胞内的ROS会积累,过量的ROS会对菌体造成损伤[28]。亮氨酸和脯氨酸具有抵御胞内ROS的作用,菌体分泌亮氨酸和脯氨酸防止ROS对菌体造成损伤[27],可能因此造成了亮氨酸和脯氨酸浓度增加。此外,本研究还测定了发酵液中的12种有机酸,然而在培养基中只检测到了乳酸(Lac)。乳酸在发酵前期浓度较低,3 h时浓度为0.33 mmol/L,在4 h时达到最大浓度5.10 mmol/L,5 h时浓度开始降低至0.27 mmol/L,在发酵末期9 h时乳酸浓度又回升至2.26 mmol/L。图2可以看出,在4 h时,菌体生物量已经达到较高水平且增长变缓,溶氧下降,因此导致乳酸浓度升高。但是随着发酵的进行,碳源也被迅速消耗,导致生成的乳酸被用作成碳源维持细胞生长[25],所以乳酸浓度出现了降低。而发酵末期,发酵液溶氧效果差,细胞进行无氧呼吸,又导致了乳酸的增加。至于是否IPTG诱导导致乳酸在诱导前后出现较大变化还有待进一步研究。
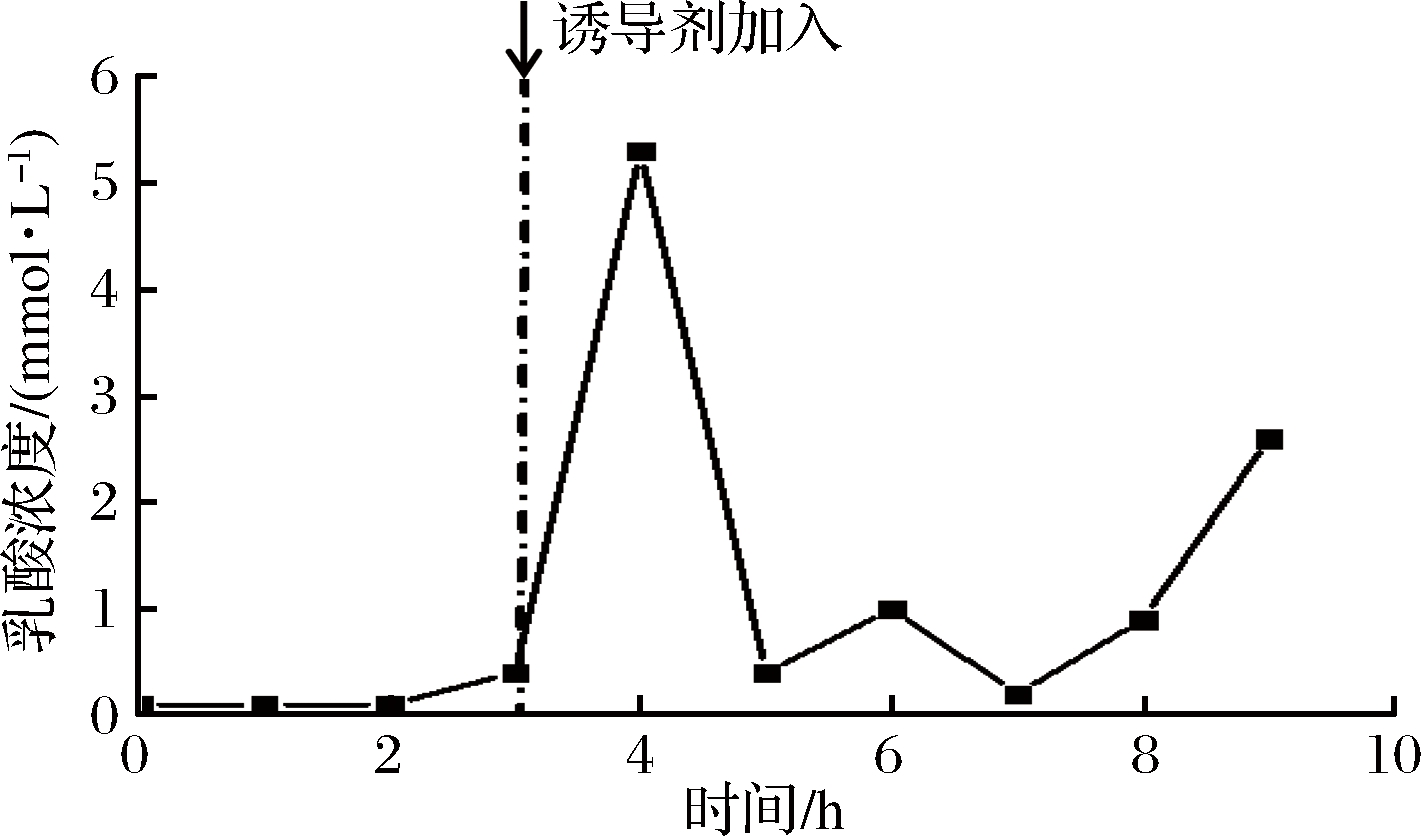
图4 LipA发酵过程中有机酸浓度曲线
Fig.4 Concentration curve of organic acid produced byE.coli f in LipA fermentation
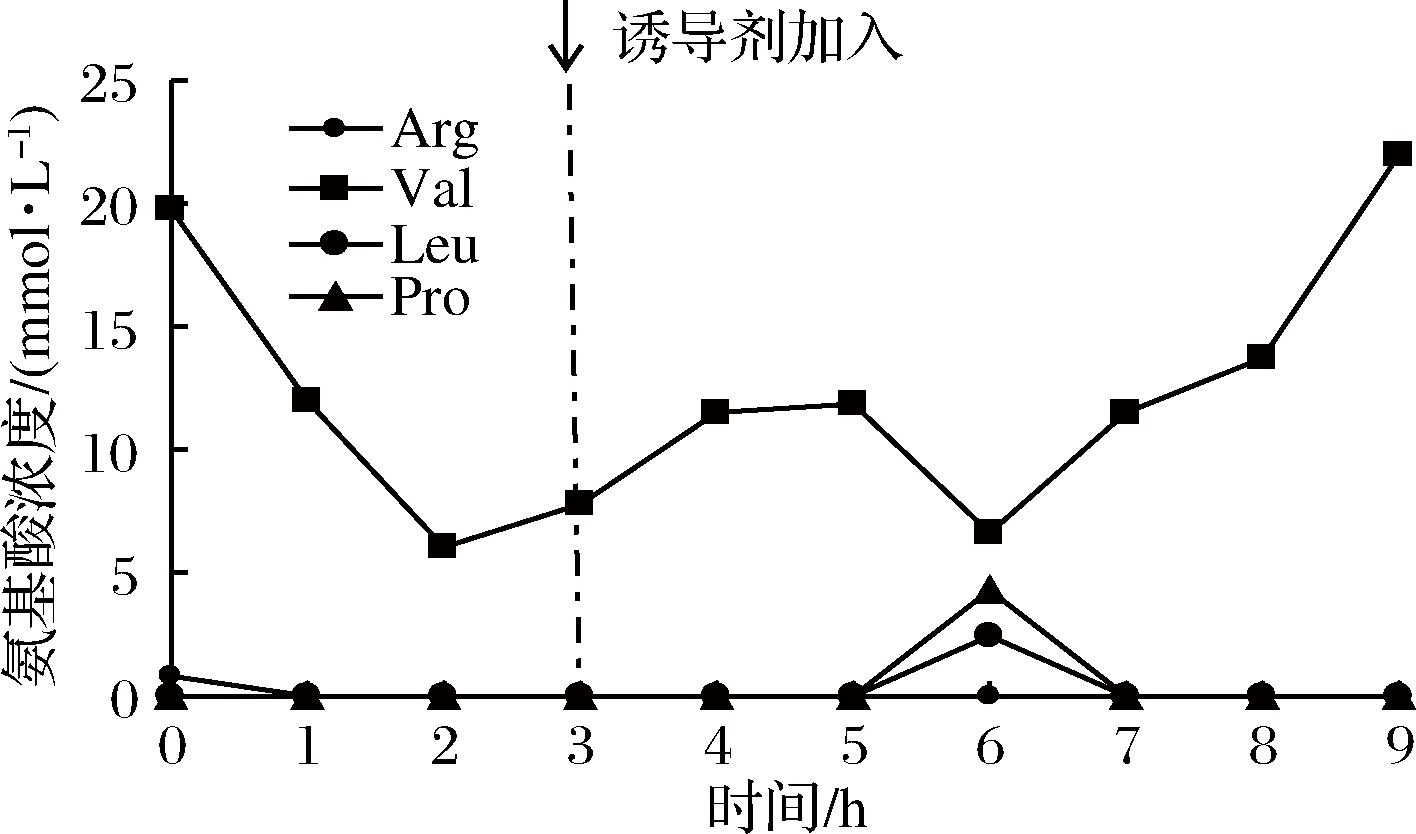
图5 LipA发酵过程中氨基酸浓度曲线
Fig.5 Concentration curve of amino acid produced byE.coli in LipA fermentation
2.3 诱导对代谢流迁移的影响
对诱导前1 h(发酵时间2 h)和诱导后1 h(发酵时间4 h)的数据进行代谢通量分析。在3 h时,重组大肠杆菌开始诱导产酶,此时细胞内的代谢流量分布开始发生显著变化,因此对诱导前后时间点进行代谢流分析,如图6所示。

图6 诱导前后的代谢通量分布图
Fig.6 Distribution of metabolic flux before and after induction
注:反应方程编号:发酵2 h代谢通量/发酵4 h代谢通量
乳酸合成途径(r19)的代谢通量在诱导前后变化最为明显。经诱导产酶后,r19代谢通量由0显著增加至137.09,碳流通过EMP途径生成乳酸,分流了部分合成生物量的碳流,降低了生物量合成的速率,造成这种现象的原因是指数生长后期,菌体大量生长,发酵体系中的溶氧不能满足菌体生长的要求,部分菌体进行无氧呼吸,导致乳酸途径的通量增加。这与图2中OD600的变化速率相符。本研究也与SONG等的研究相符,SONG等指出在指数生长期提供较高的溶氧,有利于脂肪酶的合成[6]。在组成脂肪酶的181个氨基酸中,亮氨酸占比10.4%,缬氨酸占4.9%,脯氨酸占比2.2%,精氨酸占比3.3%,亮氨酸和缬氨酸占比相对较高,且从图6代谢通量图中可以看出,缬氨酸代谢通量(r36)由0增加至60.68,而脯氨酸(r41)、精氨酸(r39)、亮氨酸(r35)代谢通量无变化。缬氨酸的通量的增加可能是由于该时刻脂肪酶大量合成,对缬氨酸的需求增加所致。同时代谢通量分析的数据也解释了6 h时缬氨酸浓度出现下降的原因,消耗的缬氨酸可能被用于脂肪酶合成。但是代谢通量的数据并不能解释胞内亮氨酸和脯氨酸在发酵6 h后出现增加。推测可能在发酵中后期,由于有氧呼吸作用造成细胞内的ROS的积累,过量的ROS会对菌体造成损伤[28]。亮氨酸和脯氨酸具有抵御胞内ROS的作用,菌体分泌亮氨酸和脯氨酸防止ROS对菌体造成损伤[27],因此可能会造成了亮氨酸和脯氨酸浓度增加。
诱导后,磷酸戊糖途径、TCA循环和ATP合成的通量出现了下降。磷酸戊糖途径中的R5P(r7)通量下降了43.4%,造成诱导生物量积累速度减缓。作为能量供应环节的TCA循环通量(r11-r15)和ATP合成通量(r43-r46)降低明显。酶蛋白的表达和折叠需要ATP作为能供应[22-23],诱导后ATP供应不足,可能是E.coli中不能高效表达脂肪酶的一个原因。
3 结论
通过对E.coli产脂肪酶的发酵特性研究,发现在诱导2 h后(发酵时间5h)胞内脂肪酶比活达到最高18.11 U/mg。通过建立重组E.coli产脂肪酶LipA的代谢网络模型,分析诱导前后的代谢流迁移发现,经诱导产酶后,乳酸代谢通量增加,碳流通过EMP途径生成乳酸,分流了部分合成生物量的碳流,降低了生物量合成的速率;缬氨酸通量的增加满足了脂肪酶的合成需求;ATP通量的减少限制了脂肪酶的高效合成。因此乳酸和ATP是E. coli合成脂肪酶合成的关键节点。本研究为提高生产脂肪酶菌种的发酵条件优化和代谢途径改造提供了理论依据。
参考文献
[1] 孙宏丹,孟秀香,贾莉,等. 微生物脂肪酶及其相关研究进展[J]. 大连医科大学学报, 2001, 23(4):292-295.
[2] BANEYX F, MUJACIC M. Recombinant protein folding and misfolding in Escherichia coli[J]. Nature Biotechnology, 2004, 22(11):1 399.
[3] ANTONIEWICZ M R. Methods and advances in metabolic flux analysis: a mini-review[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(3):317-325.
[4] GEBRESELASSIE N A, ANTONIEWICZ M R. (13)C-metabolic flux analysis of co-cultures: A novel approach[J]. Metabolic Engineering, 2015, 31:132.
[5] CALIK P, CALIK G, TAKAC S, et al. Metabolic flux analysis for serine alkaline protease fermentation by Bacillus licheniformis in a defined medium: Effects of the oxygen transfer rate [J]. Biotechnology and Bioengineering. 1999, 64(2): 151-167.
[6] SONG P, CHEN C, TIAN Q, et al. Two-stage oxygen supply strategy for enhanced lipase production by Bacillus subtilis, based on metabolic flux analysis[J]. Biochemical Engineering Journal, 2013, 71(1):1-10.
[7] 黄金,徐庆阳,温廷益,等. 不同溶氧条件下L-苏氨酸生物合成菌株的代谢流量分析[J]. 微生物学报, 2008, 48(8):1 056-1 060.
[8] 任婷月,周万里,张利群,等. 一种检测葡萄糖氧化酶活力的新方法[J]. 食品与发酵工业, 2015, 41(1):212-215.
[9] 阎博,赵林,谭欣,等. 浊度法快速测定培养液中的生物量[J]. 天津化工, 2003, 17(1):45-46.
[10] 宋萍,戚小灵,胡燚,等. 响应面法优化枯草芽孢杆菌产脂肪酶的合成培养基[J]. 中国生物工程杂志, 2010, 30(8):100-105.
[11] 乔宇,丁宏标,闫俊艳,等. 重组大肠杆菌产普鲁兰酶的高密度发酵工艺研究[J]. 生物技术进展, 2012, 02(3):195-2009.
[12] ZUO S S, LUNDAHL P. A Micro-bradford membrane protein assay[J]. Analytical Biochemistry, 2000, 284(1):162.
[13] RAMESH K G, SUBAZINI T K, ASHOK S. ECO-MP: e coli-metabolic pathway-development of genome-scale metabolic pathway database for Escherichia coli[J]. Trends in Bioinformatics, 2014, 7(1):7-12.
[14] 申铁,金杰军,乙引,等. 大肠杆菌基因缺失菌株中碳中心代谢系统的通量平衡分析和通量变异分析[J]. 计算生物学, 2013, 3(3):15-19.
[15] MAHADEVAN I, GHOSH I. Analysis of E.coli promoter structures using neural networks[J]. Nucleic Acids Research, 1994, 22(11):2 158-2 165.
[16] BANEYX F AND MUJACIC M. Recombinant protein folding and misfolding in Escherichia coli[J]. Nature Biotechnology. 2004, 22(11): 1 399-1 408.
[17] KROEMER J O, WITTMANN C, SCHROEDER H, et al. Metabolic pathway analysis for rational design of L-methionine production by Escherichia coli and Corynebacterium glutamicum[J]. Metabolic Engineering. 2006, 8(4): 353-369.
[18] LI M, HO P Y, YAO S J, et al. Effect of lpdA gene knockout on the metabolism in Escherichia coli based on enzyme activities, intracellular metabolite concentrations and metabolic flux analysis by C-13-labeling experiments [J]. Journal of Biotechnology, 2006, 122(2): 254-266.
[19] 张慧敏. 酵母在不同培养环境下中间代谢途径代谢调控过程的研究[D]. 杭州:浙江大学, 2005.
[20] KRÖMER J O, WITTMANN C, SCHRÖDER H, et al. Metabolic pathway analysis for rational design of L-methionine production by Escherichia coli and Corynebacterium glutamicum[J]. Metabolic Engineering, 2006, 8(4):353-369.
[21] 陈飞,冯小海,吴波,等. 丙酸杆菌的两种固定化细胞反应器发酵生产丙酸及其代谢通量分析[J]. 化工学报, 2011, 62(4):1 034-1 041.
[22] DOBSON C M. Review article protein folding and misfolding[J]. Nature, 2003(6 968):884-890.
[23] GOLDBERG M E. The second translation of the genetic message: protein folding and assembly[J]. Trends in Biochemical Sciences, 1985, 10(10):388-391.
[24] 赵树欣,梁慧珍,程丽娟,等. 固定化丙酸菌发酵底物的研究[J]. 中国食品添加剂, 2005(1):7-11.
[25] 沈雯. 瘤胃pH下乳酸作为枯草芽孢杆菌可利用碳源的研究[D]. 北京:北京林业大学, 2016
[26] 潘自皓. 产转氨酶大肠杆菌高密度培养的研究[D]. 南京:南京工业大学, 2006
[27] ANGCAJAS A B, HIRAI N, KANESHIRO K, et al. Diversity of amino acid signaling pathways on autophagy regulation: A novel pathway for arginine[J]. Biochemical & Biophysical Research Communications, 2014, 446(1):8-14
[28] NANOU K, ROUKAS T, PAPADAKIS E. Oxidative stress and morphological changes in Blakeslea trispora, induced by enhanced aeration during carotene production in a bubble column reactor[J]. Biochemical Engineering Journal, 2011, 54(3):172-177.