植物乳杆菌是一类革兰氏阳性、厌氧或兼性厌氧菌,属于同型发酵乳酸菌,是我国卫生部颁布的可用于食品的益生菌之一[1]。该菌主要应用于发酵食品,其良好的益生特性及保健功能已被大众广泛接受和认可,是近年来研究较多的一种益生乳酸菌。植物乳杆菌P-8(Lactobacillus plantarum P-8)是张和平教授2005年从内蒙古巴彦淖尔市乌拉特中旗草原上牧民家庭自然发酵牛乳样品中分离筛选出的1株性能优良的益生菌,研究表明,该菌株具有改善脂质代谢、降低血脂、保护肝脏、提高机体免疫力,改善肠道菌群等益生特性[2-3]。在益生菌发酵乳制品方面,只有产品中的活菌数达到106~107 CFU/mL才能充分发挥其益生作用[4],因此,发酵乳中的活菌数直接影响相关产品品质。然而传统的平板计数法存在操作繁琐,检测周期长,检测结果滞后等缺点[5],开发一种能够快速准确的检测益生菌活菌数的方法显得尤为重要。叠氮溴化丙锭(propidiummonoazide, PMA)是一种对核酸具有高度亲和力的光敏染料,能够渗透进入具有不完整细胞膜的死菌细胞,在光诱导下交联死菌DNA,产生稳定的共价交联沉淀物,使DNA分子永久修饰,从而抑制DNA扩增[6],同时反应体系中未结合的PMA在光照下与水分子相互作用生成羟胺化合物,失去交联活性。利用PMA选择性交联样品中死菌和活菌细胞的机理,与荧光定量PCR检测技术相结合,可快速检测发酵乳中益生菌活菌数[7]。
本研究利用PMA-qPCR检测方法,以L. plantarum P-8为例,通过热灭活方式获得膜损伤细胞,对影响PMA作用的浓度、曝光时间和暗孵育时间进行优化,进一步完善发酵乳产品中L. plantarum P-8活菌数的检测方法。
1 材料与方法
1.1 材料
1.1.1 实验菌株
L. plantarum P-8分离自牧民家庭自然发酵牛乳样品,由内蒙古农业大学乳品生物技术与工程教育部重点实验室乳酸菌菌种库提供。
YF-L904由嗜热链球菌(Streptococcus thermophiles)和保加利亚乳杆菌(Lactobacillus bulgaricus)组成,购自科·汉森(中国)有限公司。
1.1.2 试剂
PMA(1mg,上海吉逸生物科技有限公司):溶解于1 000 μL ddH2O,得到1 mg/mL储备液,于-20 ℃避光保存。MRS培养基(广州环凯微生物科技有限公司)、LB培养基(广州环凯微生物科技有限公司),无水乙醇(上海沪宇生物科技有限公司),TE缓冲液、5×TBE电泳缓冲液贮液、Taq DNA聚合酶、10×EasyTaq Buffer、dNTPs、电泳缓冲液、琼脂糖、核酸染料GELVIEW、6×DNA Loading Buffer、DL2000 DNA Marker(天根生化科技有限公司)。
1.1.3 实验主要试剂盒(表1)
表1 实验主要的试剂盒
Table 1 The experiment mainly laboratory kits

载体:pEASY®-Blunt Simple Cloning Kit
1.1.4 主要仪器设备(表2)
表2 实验主要的仪器
Table 2 The experiment mainly instrument

1.2 方法
1.2.1 L. plantarum P-8活菌悬液的制备
取L. plantarum P-8菌液接种于5 mL MRS液体培养基(121 ℃灭菌15 min),置37 ℃培养24 h,按2%的接种量接入MRS培养液中活化至3代,在37 ℃下培养达到指数生长期,采用平板计数法确定活菌数,用0.85%的生理盐水重悬3次,得到浓度约为1×108 CFU/mL的菌悬液。
1.2.2 L. plantarum P-8热致死菌悬液的制备
为建立死菌对照组,设计热致死实验进行L. plantarum P-8死菌悬液的制备。吸取稀释后的1 mL菌悬液于1.5 mL离心管中,采用80 ℃水浴处理0、10、20、30、40、50、60、70、80、90、100 s后,立即置于冰上冷却,利用平板计数法测定各管中活菌数,每组设置3个平行,通过该处理,使细胞壁与细胞膜有不同程度损伤而得到L. plantarum P-8的热致死菌悬液。
1.2.3 PMA质量浓度的优化
分别取500 μL活菌悬液及热致死菌悬液于1.5 mL离心管中,加入PMA(初始质量浓度1 mg/mL)液,使PMA的终质量浓度分别为0、10、20、30、40、50、60、70 μg/mL(每个浓度n=3),将样本放置于LED光敏反应仪(V-MIX)中,开启混匀功能,黑暗混匀孵育10 min,使得PMA透过受损的细胞膜进入细胞内。开启LED光敏反应仪的光源,同时开启混匀功能,曝光 20 min,以完成PMA与细菌DNA的结合,同时完全钝化游离的PMA分子以避免“假阴性”结果。10 000 r/min离心10 min,取沉淀,使用细菌基因组DNA提取试剂盒提取基因组DNA,离心取上清进行qPCR分析,确定PMA影响活菌DNA扩增的工作浓度以及抑制死菌DNA扩增的最低工作浓度。
1.2.4 PMA暗孵育时间的优化
分别取500 μL活菌悬液及热致死菌悬液于1.5 mL离心管中,以1.2.3确定的PMA工作浓度处理,将样本放置于LED光敏反应仪(V-MIX)中,开启混匀功能,暗孵育0、5、10、15、20 min(每个时间段n=3)。开启LED光敏反应仪的光源,同时开启混匀功能,曝光 20 min,10 000 r/min离心10 min,取沉淀,使用细菌基因组DNA提取试剂盒提取样品基因组DNA,离心取上清进行qPCR分析。
1.2.5 PMA曝光时间的优化
分别取500 μL活菌悬液及热致死菌悬液于1.5 mL离心管中,以1.2.3确定的PMA工作浓度处理,将样本放置于LED光敏反应仪(V-MIX)中,开启混匀功能,以1.2.4确定的时间暗孵育处理,将离心管盖打开,管口朝上,开启LED光敏反应仪的光源,同时开启混匀功能,曝光0、5、10、15、20、25、30 min(每个时间段n=3),10 000 r/min离心10 min,取沉淀,使用细菌基因组DNA提取试剂盒提取基因组DNA,离心取上清进行qPCR分析。对照组为未经PMA处理的活菌悬液及热致死悬液。
1.2.6 PMA对活细胞/热致死细胞混合样品的影响
取2份浓度约为1×108 CFU/mL的活菌悬液,1份按照1.2.2确定的方法得到热致死菌悬液,在含有500 μL热致死菌悬液的1.5 mL离心管中分别加入浓度为1×108、1×107、1×106、1×105、1×104 CFU/mL的500 μL活菌悬液,用1.2.3、1.2.4、1.2.5确定的PMA条件处理悬浮液。实验对照组如下:(1)取1 mL上述5种浓度的活菌悬液,用1.2.3、1.2.4、1.2.5确定的PMA条件处理活菌悬液;(2)取1 mL浓度为1×108 CFU/mL活菌悬液,不使用PMA相关条件处理;(3)取1 mL浓度为1×108 CFU/mL的热致死菌悬液,不使用或使用PMA相关条件处理。此外,采用相同的实验条件将不同浓度的热致死菌悬液(1×108、1×107、1×106、1×105、1×104 CFU/mL)添加至活菌悬液中,并采用最佳PMA条件处理,设计对照组为:(1)取1 mL上述5种浓度的热致死菌悬液,用最佳PMA条件处理;(2)取1 mL浓度为1×108 CFU/mL活菌悬液,用最佳PMA条件处理;(3)取1 mL浓度为1×108 CFU/mL的活菌悬液或热致死菌悬液,不使用PMA相关条件处理。提取DNA进行荧光定量PCR检测。
1.2.7 L. plantarum P-8基因组DNA的提取
使用细菌基因组DNA提取试剂盒提取L. plantarum P-8基因组DNA。
1.2.8 L.plantarum P-8标准品的制备及标准曲线的建立
取活化后的L.plantarum P-8菌悬液,提取DNA,PCR后根据琼脂糖凝胶电泳检测结果,选取目标条带清晰的PCR扩增产物,切胶回收,用纯化试剂盒纯化。将纯化的片段连接到pEASY®-Blunt Simple Cloning Vector,连接产物转化至感受态细胞Trans1-T1,用含氨苄青霉素、X-Gal和IPTG的LB培养基37 ℃、24 h培养,挑选白色菌斑接种于LB/Amp+的液体培养基中,37 ℃摇床培养,PCR鉴定阳性克隆后,用试剂盒提取质粒DNA,确定质粒DNA浓度,通过公式换算成基因的拷贝数,质粒DNA拷贝数(拷贝/μL)=DNA浓度/(片段大小×660)×NA (NA为阿伏伽德罗常数:6.02×1023)。将线性质粒的8个梯度作为模板进行荧光定量PCR反应,根据荧光值的变化规律,系统将自动生成标准曲线。
使用Step One-Plus Real-Time PCR System(Applied Biosystems,Carlsbad,CA)在48孔板中进行qPCR测定。参照张佳超等[8]合成扩增L.plantarum P-8的特异性引物。
L.plantarum P-8正向引物序列:5′-ACTAACGGGAGGAGTGAT 3′;
L.plantarum P-8反向引物序列:5′-ATAGTTCTCAAATCGGGAC-3′,长度为292 bp。qPCR反应体系20 μL:模板DNA 2 μL,SYBRR Premix Ex TaqTM II 10 μL,上游引物0.4 μL,下游引物 0.4 μL,ROX Reference Dye II 0.4 μL,ddH2O 6.8 μL。qPCR反应条件为:预变性95 ℃ 5 min,变性95 ℃ 1 min,低温退火62 ℃ 1 min,引物延伸72 ℃ 7 min,40个循环[8]。用无菌超纯水代替阴性对照中的DNA,以检测可能的污染。
1.2.9 实际样本的检测
将益生菌L. plantarum P-8以接种量为2×106 CFU/g分别与酸乳基础发酵剂(YF-L904)复配发酵牛乳,发酵结束(pH=4.5)于4 ℃冷藏24 h后,采用传统的平板计数、qPCR以及PMA-qPCR方法分别检测发酵乳贮藏期间(0、7、14、21 d)活菌数变化。为保证发酵乳中存在的PCR抑制剂不会影响荧光定量PCR的结果,取1 mL发酵乳于无菌离心管中,5 500 r/min离心2 min,去掉发酵乳盐离子和杂质,菌体用生理盐水洗涤2次后用1 mL生理盐水重悬。
1.3 数据统计
本研究涉及实验至少重复3次,各指标均为3组平行,结果表示为![]() 采用SPSS 21.0软件对实验数据进行统计学分析,2组数据间的比较采用独立样本T检验进行分析,显著性水平设定为0.05。采用Origin 2017软件做柱状图和点线图。
采用SPSS 21.0软件对实验数据进行统计学分析,2组数据间的比较采用独立样本T检验进行分析,显著性水平设定为0.05。采用Origin 2017软件做柱状图和点线图。
2 结果与分析
2.1 热致死时间的确定
建立L. plantarum P-8生长曲线,结果显示L. plantarum P-8接种于MRS培养基中,在3.5 h后进入指数生长期,3.5~12 h呈指数生长特性,12 h后进入稳定期,使用培养10 h后的L.plantarum P-8来确定PMA的交联条件;针对80 ℃不同热处理时间的菌悬液梯度稀释进行平板计数,37 ℃培养72 h后,计数结果如图1所示。
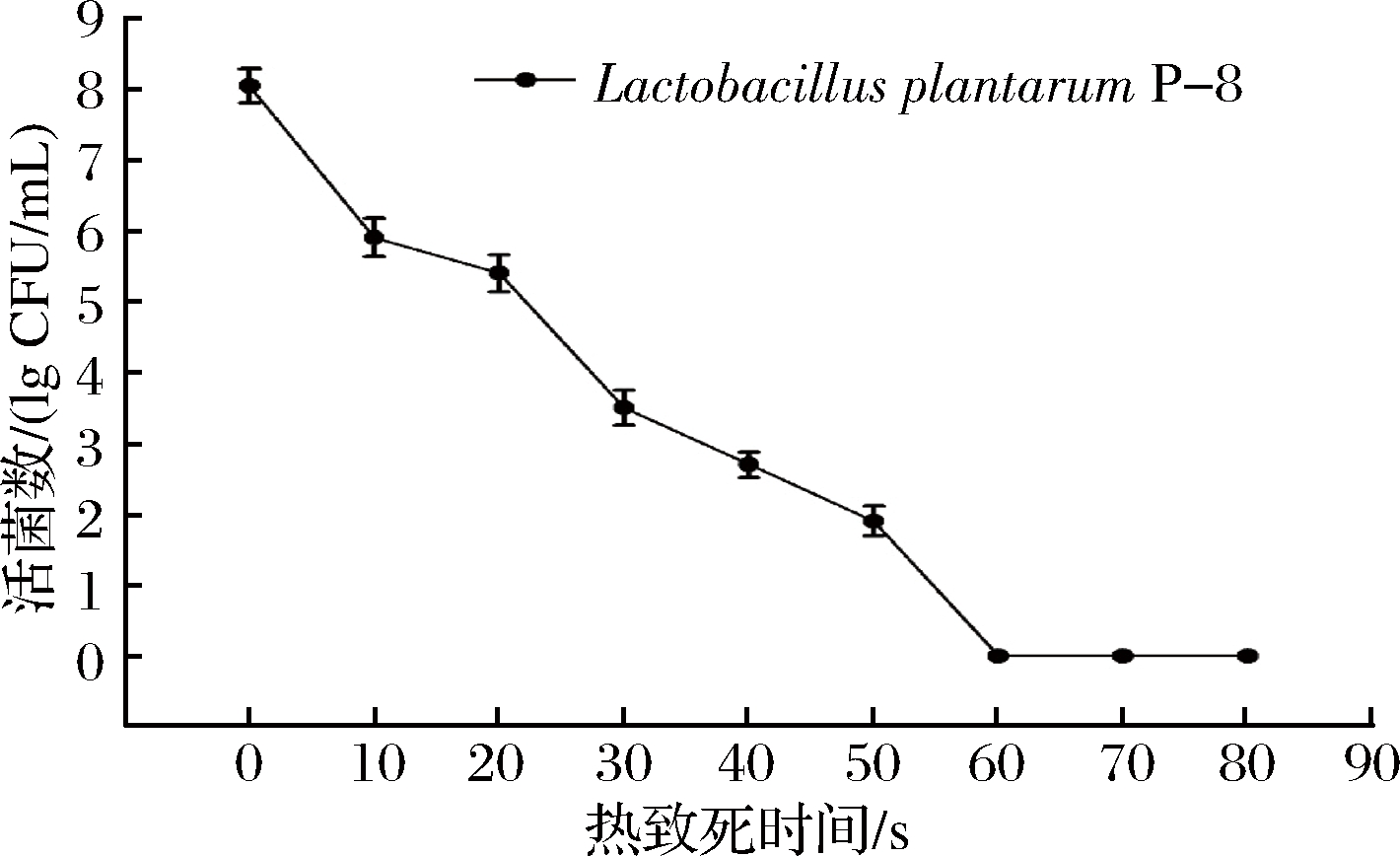
图1 热致死时间的筛选
Fig.1 Screen of heat-killed time
由图1可知,浓度为1×108 CFU/mL的L. plantarum P-8菌悬液,经过80 ℃ 60 s水浴处理后,计数平板中未出现菌落,该实验组即为L. plantarum P-8的热损伤死菌。因此,本实验采用80 ℃、60 s热处理获得L. plantarum P-8死菌悬液。
2.2 PMA质量浓度的优化
应用qPCR检测方法验证不同PMA浓度对L. plantarum P-8热致死菌悬液及其活菌悬液作用的变化,结果如图2和图3所示。由图2可知,在L. plantarum P-8热致死菌qPCR中,随着PMA工作浓度增加,L. plantarum P-8的qPCR阳性计数显著降低(P<0.05),当PMA质量浓度为40 μg/mL时,热致死菌qPCR阳性计数结果为6.5×103 CFU/mL(Ct=30.1),PMA对热灭活菌qPCR抑制率达到99.9%;当PMA质量浓度为70 μg/mL时,PMA对热致死菌qPCR抑制达到最大,但较质量浓度为40 μg/mL的PMA相比,差异不显著(P>0.05)。
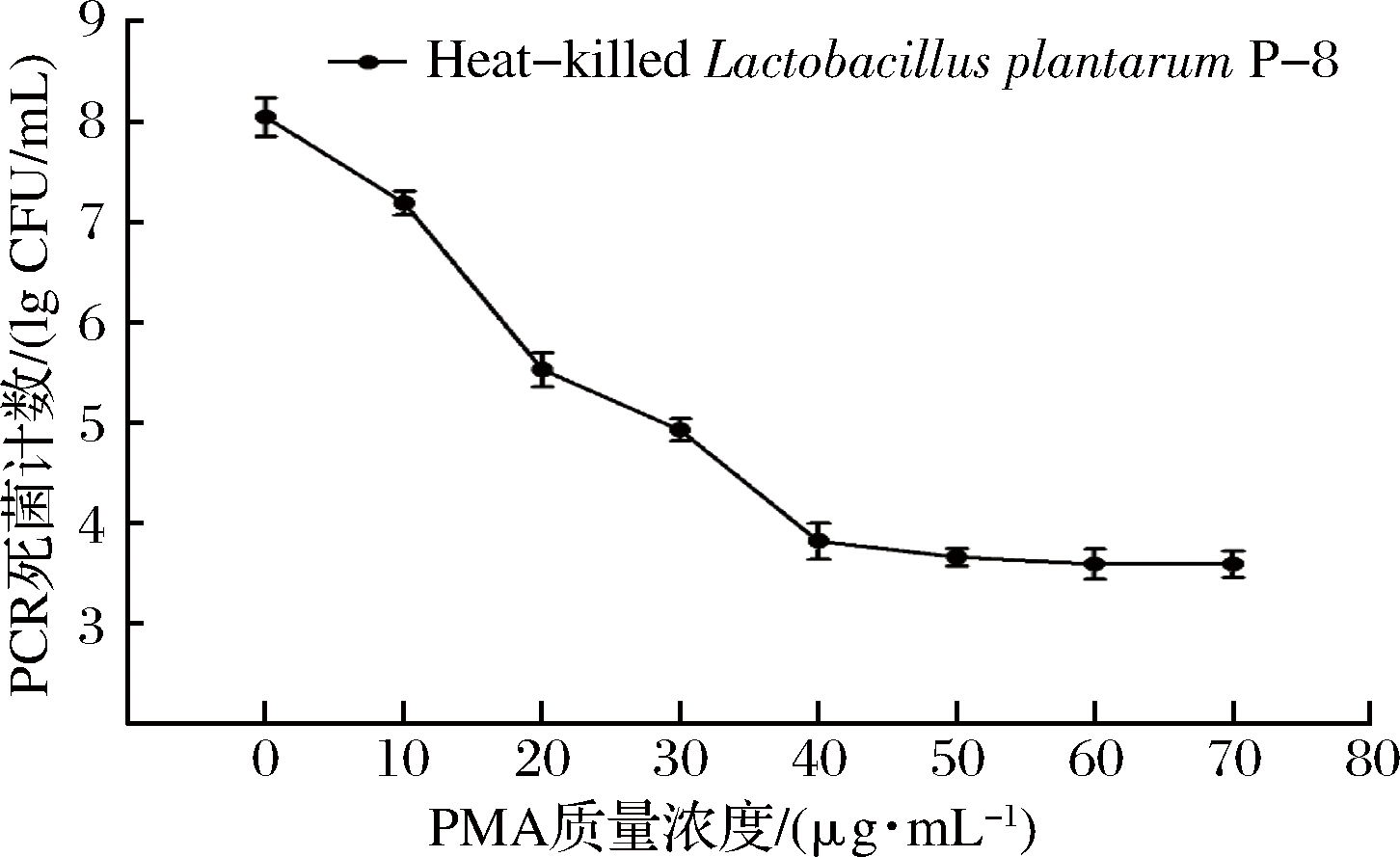
图2 不同质量浓度PMA对热灭活菌的影响
Fig.2 Effect of PMA concentration on dead bacteria
由图3可知,在L. plantarum P-8活菌qPCR中,当PMA质量浓度≤40 μg/mL时,其活菌qPCR阳性计数结果约为1.1×108 CFU/mL(Ct=14.8),PMA对活菌qPCR抑制率低于0.1%,当PMA质量浓度高于40 μg/mL时明显抑制活细胞检出率,且浓度越高,抑制作用越强。因此,当PMA质量浓度为40 μg/mL时,既可有效抑制热致死菌DNA扩增,同时并不影响活菌DNA的PCR扩增。本结论与王力均等[9]利用PMA处理副干酪乳杆菌抑制其死菌DNA的PCR扩增所得实验结果(最佳PMA浓度5 μg/mL)有所偏差,这可能是由于不同种属微生物之间细胞壁、细胞膜的结构、通透性不同引起的。此外,本实验中L. plantarum P-8菌悬液与王力均等所用菌悬液相比浓度较高,可能是引起PMA工作浓度较高的原因之一。PMA的工作浓度是影响活菌检测的重要因素,浓度过低,无法排除死菌DNA的干扰,导致“假阳性”结果;浓度过高,PMA透过活菌细胞膜与活菌DNA结合,造成“假阴性”结果。PMA工作浓度的选择受到多种因素的制约,包括(1)细菌不同的灭活方式造成其细胞膜损伤程度不同,对PMA的敏感度不同,如紫外照射灭活细菌能够破坏其核酸结构却无法使细胞膜完全受损,影响PMA与死菌DNA发生交联反应[10];(2)不同种属微生物之间细胞壁、细胞膜的结构、通透性不同,PMA最优工作浓度可能不同。因此,实验过程中必须对PMA的浓度进行优化,保证PMA与全部死菌DNA结合形成稳定的共价交联沉淀物,抑制死菌DNA扩增。

图3 不同质量浓度PMA对活菌的影响
Fig.3 Effect of PMA concentration on live bacteria
2.3 PMA暗孵育时间的优化
应用qPCR检测方法验证不同暗孵育时间对L. plantarum P-8热致死菌悬液作用的变化,结果如图4所示。由图4可知,将通过PMA处理后的L. plantarum P-8热致死菌悬液黑暗混匀孵育,0~10 min,随着暗孵育时间的增加,该菌的qPCR阳性计数由1.1×108 CFU/mL(Ct=14.8)降至6.1×103 CFU/mL(Ct=30.3),10 min后,各个时间点Ct值差异不显著(P>0.05)。因此,确定处理L. plantarum P-8热致死菌悬液的最佳暗孵育时间为10 min。暗处理阶段实现PMA进入热致死死细胞,暗孵育时间决定PMA嵌入DNA双链的多少,合理的暗孵育时间在保证PMA进入全部死菌细胞内的基础上,阻止其渗透活细胞。

图4 不同暗孵育时间对热灭活菌的影响
Fig.4 Effect of dark incubation time on dead bacteria
2.4 PMA曝光时间的优化
应用qPCR检测方法验证不同曝光时间对L. plantarum P-8热致死菌悬液作用的变化,结果如图5所示。由图5可知,将暗孵育10 min后的L. plantarum P-8热致死菌悬液(已加入PMA)暴露于强光下,0~15 min,随着光照时间的增加,该菌的qPCR阳性计数显著降低(P<0.05),由光照前的1.1×108 CFU/mL(Ct=14.8)降至6.9×105 CFU/mL(Ct=21.1),当光照时间为20 min时,L. plantarum P-8的qPCR阳性计数结果为6.5×103 CFU/mL(Ct=30.1),此后,各个时间点Ct值差异不显著(P>0.05)。通过上述分析可知,为确保PMA与热致死菌交联反应有效,选择20 min的光照用于随后的实验中。该实验结论与SHAO等[11]所研究的完全抑制死菌DNA扩增的最佳曝光时间为20 min一致。PMA在暗孵育条件下,进入热致死菌细胞内,曝光时间决定PMA与DNA的结合程度,从而影响qPCR检测方法的准确性。若光照时间过短,无法保证PMA与全部死菌DNA共价交联,未结合的死菌DNA参与后续PCR扩增,导致“假阳性”结果;然而光照时间过长,照射产生的高热量造成活菌的热致死,导致qPCR检测结果偏低。曝光时间的长短受到多种因素的影响,包括实际样品溶液浓度、光源类型以及光照强度等,此外,由于不同种属微生物其物理特征及菌落状态不同,也会引起最佳曝光时间的差异[12]。因此,为保证PMA与全部死菌DNA结合,同时未结合的PMA与水分子反应形成羟胺化合物,避免其与裂解后的活菌DNA结合[13],实验过程中须对曝光时间进行优化。

图5 不同曝光时间对热灭活菌的影响
Fig.5 Effect of light exposure time on dead bacteria
2.5 PMA对活细胞/热致死细胞混合样品的影响
应用qPCR检测方法验证PMA对L. plantarum P-8活细胞/热致死细胞混合样品作用的变化,结果如图6和图7所示。由图6可知,当不同浓度的活细胞加入标准浓度的死细胞中时,经过PMA处理,随着活细胞浓度的增加,Ct值呈下降趋势,与未加死细胞的活菌悬液相比,Ct值差异不显著(P>0.05),表明PMA对活菌DNA扩增无影响。当样品中全是死细胞时,不经PMA处理的样品与PMA处理后的样品Ct值差异性显著(P<0.05),ΔCt=16.09,表明PMA能够渗入膜损伤细胞,抑制其PCR扩增。由图7可知,当不同浓度的死细胞加入标准浓度的活细胞中时,经过PMA处理,随着死细胞浓度的增加,Ct值保持稳定,与未加活细胞的热致死菌悬液相比,Ct值差异性显著(P<0.05),当样品中全是活细胞时,不经PMA处理的样品与PMA处理后的样品相比,Ct值差异不显著(P>0.05)。以上结果表明:使用PMA处理活细胞/热致死细胞混合液,PMA能够与死菌细胞结合,产生稳定的共价交联产物,从而有效抑制其后续DNA扩增。活细胞中的细胞膜能够将PMA阻隔在外,保证其DNA扩增的顺利进行。

图6 PMA对活细胞/热致死细胞混合样品的影响(活细胞浓度依次增加)
Fig.6 Effect of PMA on mixed samples of live/dead bacteria (increase of living bacteria concentration)

图7 PMA对活细胞/热致死细胞混合样品的影响(热致死细胞浓度依次增加)
Fig.7 Effect of PMA on mixed samples of live/dead bacteria(increase of dead bacteria concentration)
2.6 L.plantarum P-8标准品的制备及标准曲线的建立
使用质粒提取试剂盒提取重组质粒DNA得到L.plantarum P-8质粒DNA,质粒DNA质量浓度为130.9 ng/μL。将质粒DNA经10倍梯度稀释制作标准品溶液,进行荧光定量PCR扩增,形成以Ct值为纵坐标,质粒拷贝数的对数为横坐标的标准曲线,如图8所示,图中表明Ct值与质粒拷贝数的对数存在良好的线性关系,线性回归方程为y=-3.631x+44.021,相关系数(R2)为0.992 9,最低检测限为103 CFU/mL。
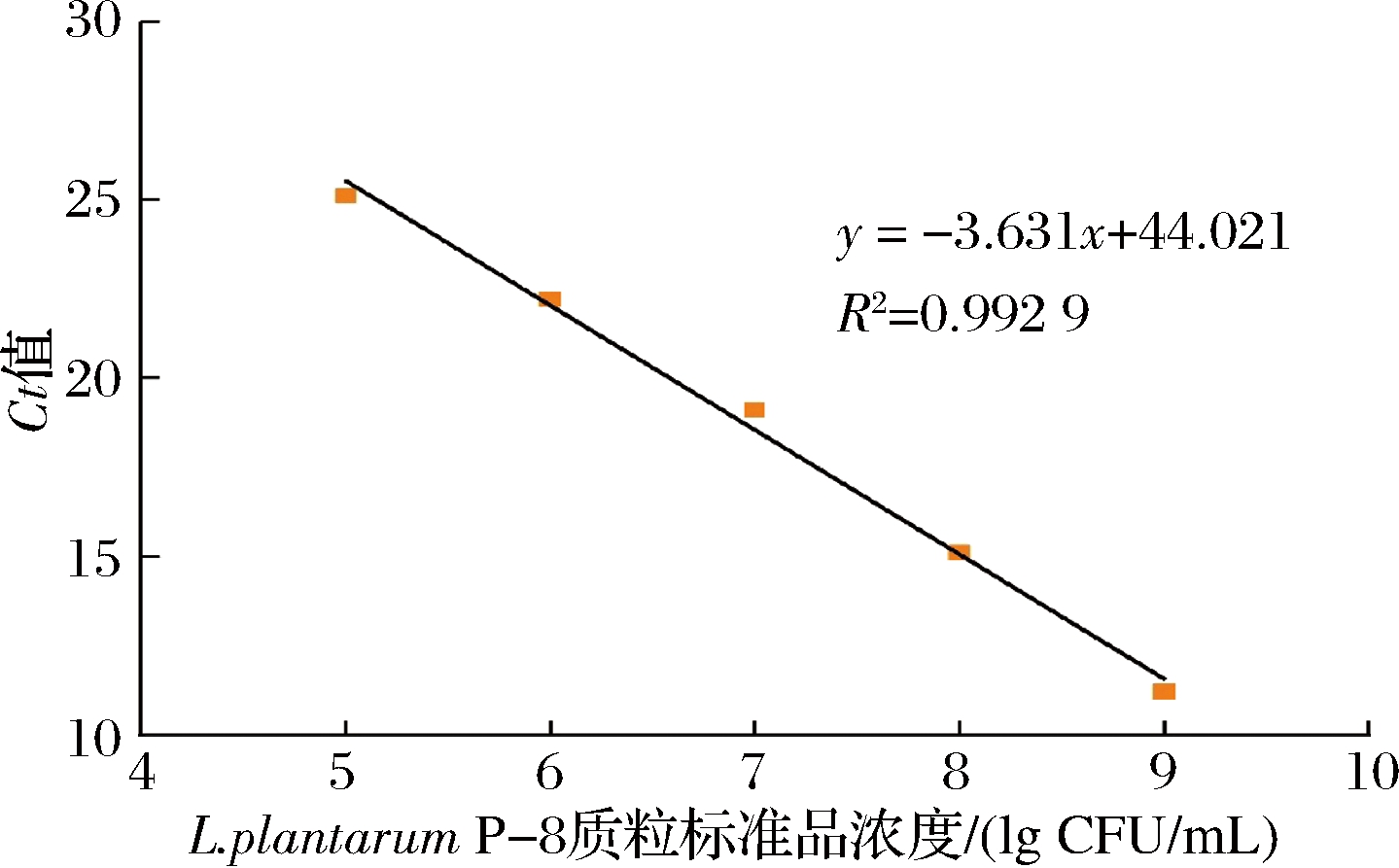
图8 L.plantarum P-8标准曲线
Fig.8 L.plantarum P-8 standard curve
2.7 实际样品的检测
采用传统的平板计数、qPCR以及PMA-qPCR的方法分别检测发酵乳贮藏期间(0、7、14、21 d)L.plantarum P-8活菌数的变化,结果如表3所示。
由表3可知,在实际样品中,经过PMA处理的样品得到的计数结果均明显小于未经过PMA处理组,说明使用PMA处理实际样品后,PMA能够通过渗透作用进入死菌细胞,抑制死菌细胞PCR扩增,消除了死菌DNA对检测结果的影响。采用PMA-qPCR方法检测活菌数所得结果略高于传统的平板计数,但无显著性差异(P>0.05),可能是因为该方法检测出的活菌数包括活的非可培养态(VBNC)[14]菌体,VBNC菌体虽然使用常规的培养方法无法检测出来,但仍然保持代谢活性,在适宜的条件下,可能进行复苏。同时,在具体的发酵乳检测过程中,受样品成分如乳蛋白等的影响,检测结果存在一定的误差。
表3 L.plantarum P-8定量检测结果
Tablet 3 The result of the detection of the L.plantarum P-8

注:NTa表示样品未经PMA处理,Tb表示样品经过PMA处理。
3 结论
在活菌计数方面,众多科研工作者在改进传统计数方法的同时,研究出更多精确省时的方法。MASCO等通过qPCR方法(依据16SrRNA基因序列设计引物,使用SYBR Green I染料法)检测发酵乳中的乳酸菌,包括嗜热链球菌、德氏乳杆菌、双歧杆菌、鼠李糖乳杆菌以及干酪乳杆菌。结果表明,荧光定量PCR方法能够准确定量发酵乳中的乳酸菌,最低检测限为103 CFU/mL[15-17]。FALENTIN,SHEU等采用PMA-qPCR、EMA-qPCR方法对发酵乳中的干酪乳杆菌和双歧杆菌做定量分析,检测结果与传统的平板计数无显著性差异(P<0.05)[18-20]。目前,就国内外研究而言,PMA-qPCR方法主要用于食源性致病菌(大肠杆菌、沙门氏菌)的检测。从益生菌领域来看,qPCR与其他技术结合实现对植物乳杆菌活菌数的检测在国内尚未见报道。
本研究将PMA与荧光定量PCR相结合,去除死菌DNA对L.plantarum P-8活菌检测的干扰,降低假阳性结果出现的可能。在具体的操作过程中,从前期PMA的预处理,到DNA的提取以及最后利用qPCR的准确定量,整个过程快速、高效,在最大程度上提高工作效率。该方法在食品领域有巨大的潜在应用前景,为发酵乳产品中的益生菌进行大规模、多元化、快速检测提供了新的参考方法。
参考文献
[1] PEREZCANO F J,DONG H,YAQOOB P. In vitro immunomodulatory activity of Lactobacillus fermentum CECT5716 and Lactobacillus salivarius CECT5713: two probiotic strains isolated from human breast milk[J].Immunobiology,2010,215(12):996-1 004.
[2] WANG Z,BAO Y,ZHANG Y,et al.Effect of soymilk fermented with Lactobacillus plantarum P-8 on lipid metabolism and fecal microbiota in experimental hyperlipidemic rats[J].Food Biophysics,2013,8(1):43-49.
[3] BAO Y,WANG Z,ZHANG Y,et al.Effect of Lactobacillus plantarum, P-8 on lipid metabolism in hyperlipidemic rat model[J]. European Journal of Lipid Science & Technology,2012,114(11):1 230-1 236.
[4] 王记成.基于转录组学和蛋白质组学对益生菌Lactobacillus casei Zhang在牛乳和豆乳中生长机理的研究[D].呼和浩特:内蒙古农业大学, 2012.
[5] MASTERS C I,SHALLCROSS J A,MACKEY B M.Effect of stress treatments on the detection of Listeria monocytogenes and enterotoxigenic Escherichia coli by the polymerase chain reaction[J].Journal of Applied Microbiology,1994,77(1):73-79.
[6] NOCKER A,CAMPER A K.Novel approaches toward preferential detection of viable cells using nucleic acid amplification techniques[J].Fems Microbiology Letters,2009,291(2):137-142.
[7] CHANG B,TAGURI T,SUGIYAMA K,et al.Comparison of ethidium monoazide and propidium monoazide for the selective detection of viable Legionella cells[J].Japanese Journal of Infectious Diseases,2010,63(2):119.
[8] 张佳超,王丽凤,高鹏飞.一株可以在人肠道中定植并繁殖的益生菌及其检测方法:中国,102747012 A[P]. 2012-05-15.
[9] 王力均,谭强来,朱江.等.应用PMA-qPCR方法快速准确检测发酵乳制品中副干酪乳杆菌活菌的研究[J].中国微生态学杂志, 2013,25(1):1-4.
[10] NOCKER A,SOSSA K E,CAMPER A K.Molecular monitoring of disinfection efficacy using propidium monoazide in combination with quantitative PCR[J].Journal of Microbiological Methods,2007,70(2):252-260.
[11] SHAO Y,WANG Z,BAO Q,et al.Application of propidium monoazide quantitative real-time PCR to quantify the viability of Lactobacillus delbrueckii ssp. bulgaricus[J].Journal of Dairy Science,2016,99(12):9 570.
[12] NOCKER A,CAMPER AK.Selective removal of DNA from dead cells of mixed bacterial communities by use of ethidium monoazide[J].Applied and Environmental Microbiology, 2006,72(3):1 997-2 004.
[13] TAKASHI S,KEN-ICHIRO I,TIAN Q,et al.Method to detect only live bacteria during PCR amplification[J].Journal of Clinical Microbiology,2008,7:2 305-2 313.
[14] SALMA M,ROUSSEAUX S,SEQUEIRA-LE G A,et al.Characterization of the viable but nonculturable (VBNC) state in Saccharomyces cerevisiae[J].Plos One,2013,8(10):e77 600.
[15] FURET J P,QUéNéE P,TAILLIEZ P.Molecular quantification of lactic acid bacteria in fermented milk products using real-time quantitative PCR.[J].International Journal of Food Microbiology,2004,97(2):197-207.
[16] MASCO L,VANHOUTTE T,TEMMERMAN R,et al.Evaluation of real-time PCR targeting the 16S rRNA and recA, genes for the enumeration of bifidobacteria in probiotic products[J].International Journal of Food Microbiology, 2007,113(3):351-357.
[17] SHEU S J,HWANG W Z,CHIANG Y C,et al.Use of tuf gene-based primers for the PCR detection of probiotic Bifidobacterium species and enumeration of bifidobacteria in fermented milk by cultural and quantitative real-time PCR methods[J].Journal of Food Science,2010, 75(8):521-527.
[18] SHEU S J,HWANG W Z,CHEN H C,et al.Development and use of tuf gene-based primers for the multiplex PCR detection of Lactobacillus acidophilus, Lactobacillus casei group, Lactobacillus delbrueckii, and Bifidobacterium longum in commercial dairy products[J].Journal of Food Protection,2009,72(1):93-100.
[19] GARC A-CAYUELA T,TABASCO R,PEL
A-CAYUELA T,TABASCO R,PEL EZ C,et al.Simultaneous detection and enumeration of viable lactic acid bacteria and bifidobacteria in fermented milk by using propidium monoazide and real-time PCR[J].International Dairy Journal,2009,19(6):405-409.
EZ C,et al.Simultaneous detection and enumeration of viable lactic acid bacteria and bifidobacteria in fermented milk by using propidium monoazide and real-time PCR[J].International Dairy Journal,2009,19(6):405-409.
[20] FALENTIN H,POSTOLLEC F,PARAYRE S,et al.Specific metabolic activity of ripening bacteria quantified by real-time reverse transcription PCR throughout emmental cheese manufacture [J].International Journal of Food Microbiology,2010,144:10-19.