国内外大量研究表明,乳酸菌(lactic acid bacteria, LAB)具有调节宿主肠道微生态平衡、降低胆固醇、调节免疫功能及延缓衰老等生理功能[1]。耐久肠球菌(Enterococcus durans)隶属于硬杆菌门(Firmicutes),杆菌纲(Bacilli),乳酸杆菌目(Lactobacillales),肠球菌科(Enterococcaceae),肠球菌属(Enterococcus)。该菌是动物肠道微生物的重要组分,且常见于发酵食品,如酸奶和奶酪中[2]。不同来源的耐久肠球菌的生物学特性和生理功能各不相同[3-4]。例如,黄坚等[5]从牦牛发酵酸奶中筛选到耐久肠球菌SWUN5857菌株,发现该菌株能够适应小鼠胃肠环境,有效提高小鼠体质量,促进小鼠胸腺和脾脏发育,增强小鼠免疫功能。LIU等[6]从内蒙古天然发酵奶油中分离到耐久肠球菌KLDS6.0930菌株,该菌则具有耐胆盐,吸收胆固醇等作用。
耐低温LAB不仅能够有效抑制腐败菌在低温下的生长和繁殖,而且,在食品发酵过程中因其低温适应性,有助于降低生产能耗,节约企业成本。因此,耐低温LAB的筛选对于食品加工与贮藏具有重要意义。然而,大多数LAB属于中温型微生物,一般最适生长温度为30~40 ℃;在低于10 ℃的温度下,LAB生长微弱,甚至不生长[7]。迄今为止,国内外有关嗜冷LAB的研究已有报道。例如,LI等[8]报道了3株在15 ℃生长的植物乳杆菌(Lactobacillus plantarum) LZ95、CY2和CY3菌株,其中,LZ95菌株在de Man-Rogosa-Sharpe(MRS)培养基中培养48 h后,OD620nm值增加了10倍。LI等[9]从藏牦牛瘤胃中分离到具有纤维素分解能力的粪肠球菌(Enterococcus faecalis) JF85和Y83菌株,发现它们能够在15 ~55 ℃的温度下生长。
本研究基于前期研究基础,聚焦于1株分离自中国传统发酵食品且具有良好的体外抗氧化性的耐久肠球菌C11菌株(E. durans C11)[10],分析该菌株在不同环境胁迫条件下的耐受性,并运用比较基因组学方法探讨E. durans的低温适应机制。
1 材料与方法
1.1 材料
1.1.1 菌株
耐久肠球菌C11菌株分离自我国传统发酵食品——乳饼[10],由本实验室筛选、鉴定并保藏。
1.1.2 培养基、主要试剂及仪器
MRS培养基,北京陆桥技术有限公司;磷酸盐缓冲液(phosphate-buffered saline,PBS) (pH 7.4)、λ/HindIII DNA Marker (MD202)、6×Loading buffer,天根生化科技有限公司;Takara MiniBEST细菌基因组DNA提取试剂盒,上海皓嘉科技发展有限公司;胆盐和琼脂,国药集团化学试剂有限公司;胃蛋白酶(活性1∶3 000)、胰酶(活性1∶250)、微孔过滤膜(0.22 μm)、NaCl,上海生工生物工程有限公司;多功能酶标仪,BioTek公司;雷磁pHS-3C pH仪,上海雷磁仪器厂;Research® plus pipette移液器、台式高速低温冷冻离心机5417R、高速冷冻离心机5424、PCR仪,Eppendorf公司;凝胶成像系统Molecular Imager® Gel DocTM XR+ System,Bio-Rad公司;全自动生长曲线分析仪,Bioscreen公司。
1.2 实验方法
1.2.1 E. durans C11菌株的培养
参考XU等[10]的方法,E. durans C11菌株以体积分数1%接种于MRS液体培养基中,置于37 ℃厌氧培养至对数生长期,连续传代培养2代,得到新鲜培养物。4 000 r/min离心10 min,用无菌PBS洗涤菌体3次(避免带入接种物中的酸性物质从而改变pH)。用适量的PBS悬浮菌体沉淀作为种子液备用。
1.2.2 温度、酸、渗透压的耐受性分析
按照1%体积分数的接种量将新鲜种子液接种到MRS液体培养基中,在不同温度(10、15、20、25、37、45 ℃)培养72 h,采用全自动生长曲线分析仪实时检测培养物在600 nm波长处的吸光度值(OD600 nm),3次重复。
按照1%体积分数的接种量将新鲜种子液分别接种到不同起始pH值(2.0、3.0、4.0、5.0、6.0、7.0)、不同NaCl质量分数(0、5.0%、8.0%、10.0%、12.0%、15.0%)、以及不同胆盐质量分数(0、0.05%、0.10%、0.20%、0.30%)的MRS液体培养基中,于37 ℃培养24 h,实时检测培养物的OD600 nm值,3次重复。
1.2.3 人工胃液及肠液的耐受性分析
参照ZHANG等[11]的方法制备人工模拟胃液(pH 2.0)和人工模拟肠液(pH 6.8)。按照10%体积分数将新鲜种子液分别接种于经膜过滤除菌的1.0 mL人工胃液和人工肠液中,充分混匀,分别于37 ℃孵育180 min和240 min后取样,10倍梯度稀释,以标准平板法计数,计算细菌的存活率,实验3次重复。
1.2.4 细菌基因组DNA的制备
采用Takara MiniBEST细菌基因组DNA提取试剂盒,按照试剂盒说明书步骤提取E. durans C11菌株的基因组DNA。采用0.7%琼脂糖凝胶电泳检测DNA样品,凝胶成像系统拍照记录实验结果,多功能酶标仪测定DNA样品的浓度和纯度(A260/A280)。
1.2.5 基因组DNA序列的测定、质控和装配
运用Illumina HiSeq Xten测序平台,由北京诺禾致源生物信息科技有限公司测定E. durans C11全基因组序列。采用FASTQC软件[12]分析原始序列reads的质量。采用SOAP denovo软件(version 2.04)(http://soap.genomics.org.cn/)进行序列装配。
1.2.6 基因组注释
采用Glimmer (Version 3.0) [13]、tRNA_scan-SE (Version 1.3.1) [14]、RNAmmer (Version 1.2)软件[15]分别预测编码基因、tRNA、rRNA基因。采用Prophage Finder预测噬菌体序列(http://phast.wishartlab.com/)。采用CRISPRFinder软件[16]预测CRISPR (clustered regularly interspaced short palindromic repeats)位点。采用PGAP[17]、CD-Search [18]、SignalP 4.1 Serve[19]、TMHMM[20] 软件分别预测假基因、Pfam域的基因、信号肽、跨膜结构域。通过比对毒力因子数据库VFDB(virulence factors database) (http://www.mgc.ac.cn/VFs/)、耐药基因数据库ARDB (antibiotic resistance genes database) (http://arpcard.mcmaster.ca) 分别预测毒力、耐药基因(Identity>70%, E<1e-10)。
1.2.7 比较基因组分析
截止到2018年12月20日,从美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)基因组数据库(http://www.ncbi.nlm.nih.gov/genome)中检索到17株E. durans菌株的基因组序列,其中3株菌为全基因组序列,14株菌为基因组草图,及E.durans C11共18株耐久肠球菌用于比较基因组分析(表1)。采用BLAST(basic local alignment search tool)软件比对分析菌株特异基因(阈值e-10)。采用CD-HIT[21]、Muscle软件[22]分析同源基因并对其进行多序列比对。采用PhyML软件[23]以邻接算法构建系统发育树,并且进行1 000次bootstrap校验,使用Evolview软件绘制系统发育树[24]。
2 结果与讨论
2.1 不同温度对E. durans C11菌株生长的影响
本研究测定了E. durans C11菌株在不同温度下(10~45 ℃)的生长曲线,结果如图1所示。在45 ℃高温条件下,E. durans C11在MRS培养基(pH 6.8)中几乎不能生长;与最适生长温度(37 ℃)相比,在25 ℃条件下,E. durans C11仍生长较快,约6 h进入对数期,21 h达到对数生长中期,30 h到达稳定期;在20 ℃和15 ℃条件下,该菌株约6 h进入对数期,27 h到达对数生长中期,42 h到达稳定期;在10 ℃条件下,菌株生长缓慢,约12 h进入对数期,40 h到达对数生长中期,54 h到达稳定期。上述结果表明,E. durans C11菌株在MRS培养基中能够适应10~25 ℃的低温条件。
表1 18株耐久肠球菌及其基因组序列
Table 1 The 18 E. durans strains and their genome information

菌株来源分离日期基因组大小/Mb备注NCBI序列号E. durans C11奶豆腐2014-072.99本研究RQWF00000000E. durans KLDS6.0933发酵乳2009-02-023.07[25]NZ_CP012366.1E. durans KLDS 6.0930发酵乳2009-02-023.07[6]NZ_CP012384.1E. durans BDGP3成熟果蝇粪便2015-04-062.99[26]NZ_CP022930.1E. durans ATCC 6056_v1--3.15-NZ_KE136370.1E. durans ATCC 6056_v2--3.17-NZ_KE136516.1E. durans IPLA 655奶酪-3.06[2]NZ_AOSM01000009.1E. durans IQ23奶酪20113.13-NZ_LKPF01000001E. durans NBRC 100479--3.02-NZ_BCQB01000001.1E. durans F0321E 104牛粪2005-03-212.93-NZ_MJEE01000001.1E. durans FDAARGOS_396智人粪便3.40-NZ_PDEB01000004.1E. durans CVM N59549F雏鸡20142.96-NZ_PTWL01000008.1E. durans 4EA1鸡胸肉-3.34-NZ_LEPB01000004.1E. durans NCTC8130--3.36-NZ_UGIP01000002.1E. durans NCTC8129--3.26-NZ_UGIF01000002.1E. durans AF11-32-H智人粪便2013-09-013.08-NZ_QSAN01000001.1E. durans P16 CL A28-20052.89-NZ_RKNR01000001.1E. durans 18S干酪2007-03-032.76-NCVP01000049.1
注:“-”未知。
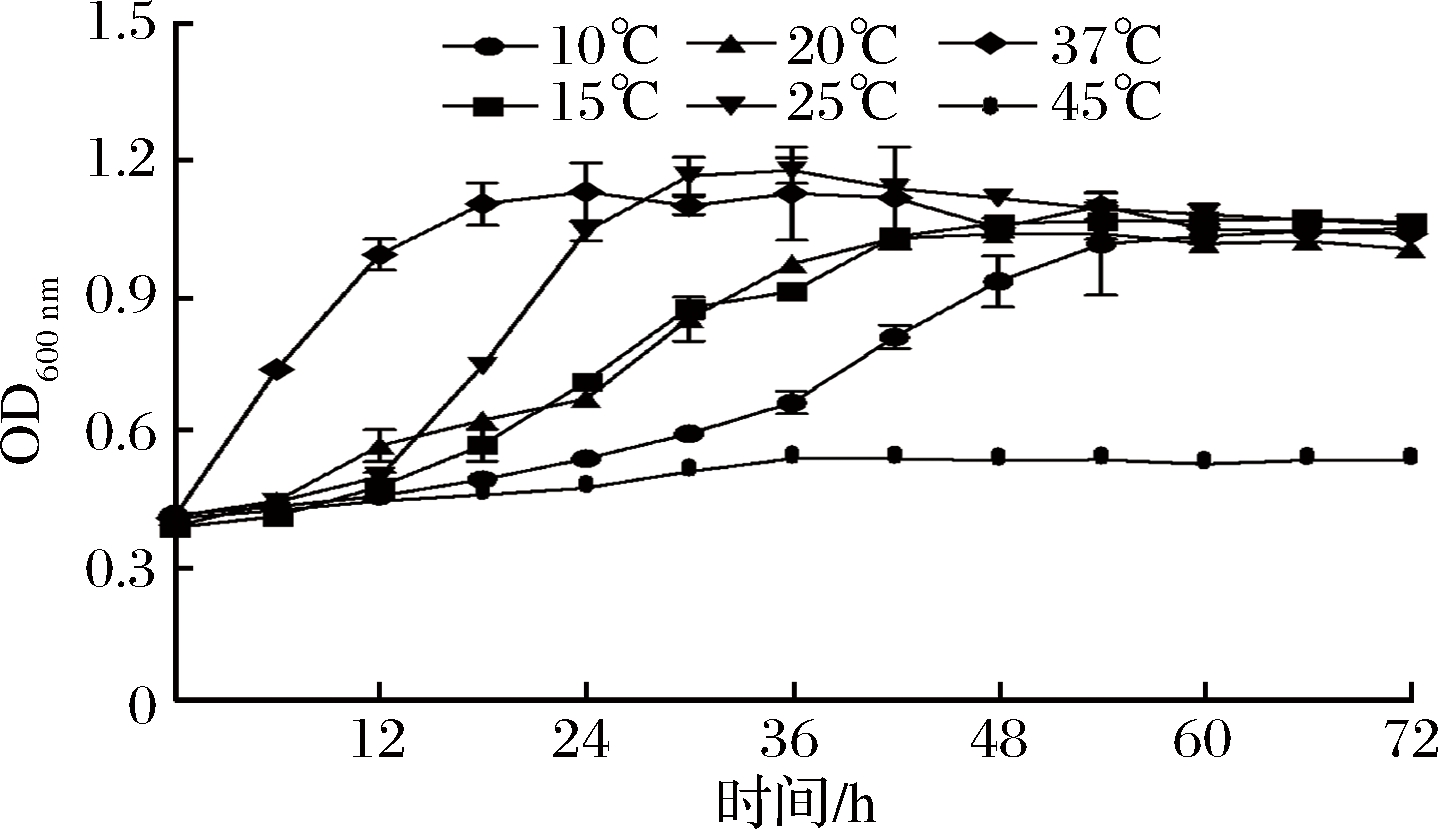
图1 温度对耐久肠球菌C11菌株生长的影响
Fig.1 The effect of temperatures on the growth ofE. durans C11
2.2 不同起始pH值对E. durans C11菌株生长的影响
如图2所示,当MRS培养基的pH为2.0~4.0时,E. durans C11菌株生长缓慢甚至不生长,其OD600nm值无明显改变。当pH为5.0~6.0时,E. durans C11菌株能够生长,但与最适生长pH (6.8~7.0)相比,酸性培养条件(pH 5.0~6.0)显著抑制了E. durans C11的生长。
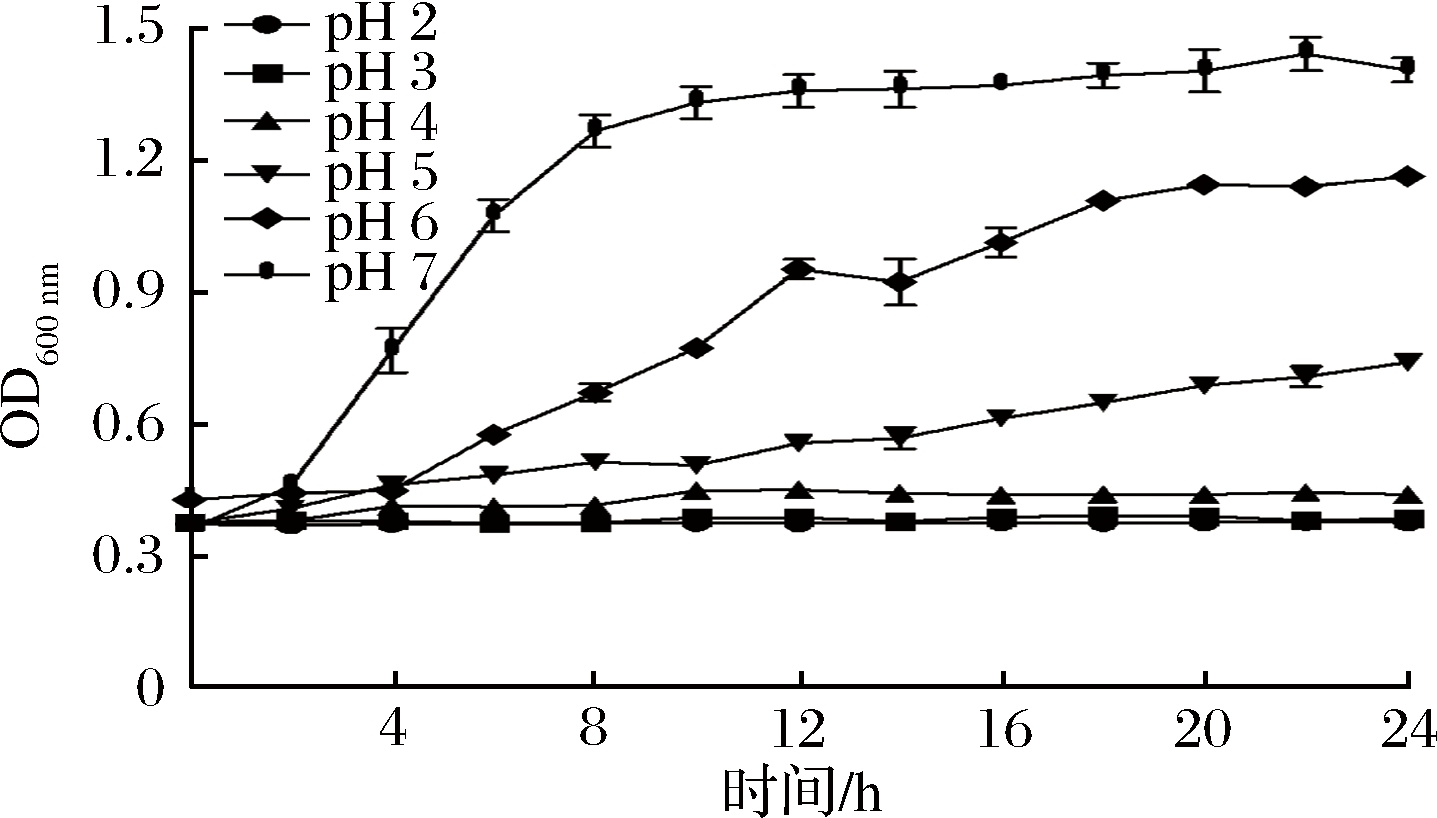
图2 不同起始pH值对耐久肠球菌C11生长的影响
Fig.2 The effect of different initial pH on the growth ofE. durans C11
2.3 E. durans C11菌株对NaCl的耐受性
与没有添加NaCl的MRS培养基相比,添加5.0%的NaCl显著抑制了E. durans C11菌株的生长,约6 h进入对数期,16 h到达对数中期,24 h到达稳定期;当MRS培养基中添加的NaCl质量分数大于8.0%时(8.0%~15.0%),E. durans C11菌株几乎不能生长(图3)。
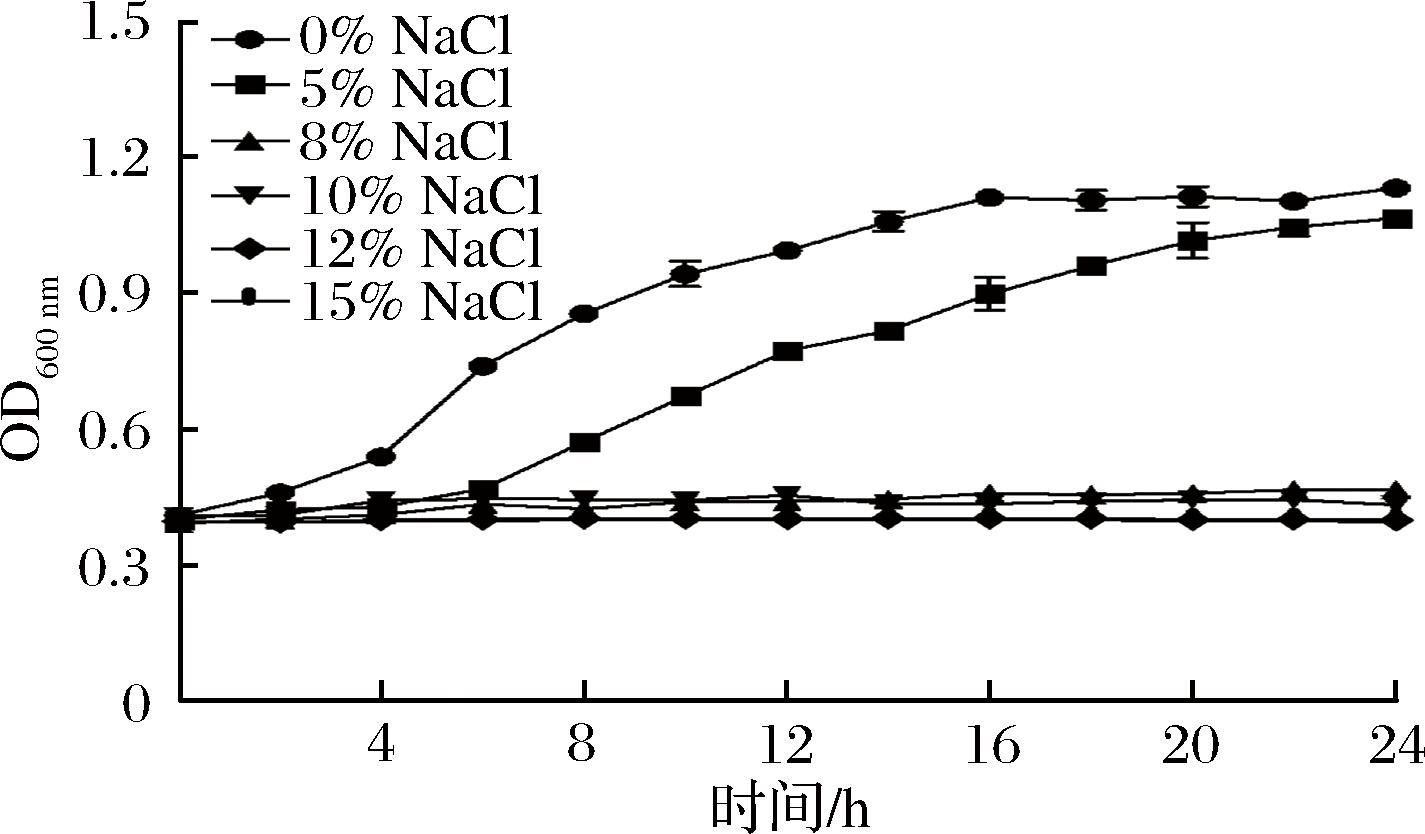
图3 耐久肠球菌C11对不同质量分数NaCl的耐受性
Fig.3 Tolerance of E. durans C11 to different concentrations of NaCl
2.4 E. durans C11菌株对胆盐的耐受性
如图4所示,当胆盐质量分数大于0.05%时,E. durans C11菌株不能生长,表明该菌株对胆盐(0.05%~0.3%)无耐受性。
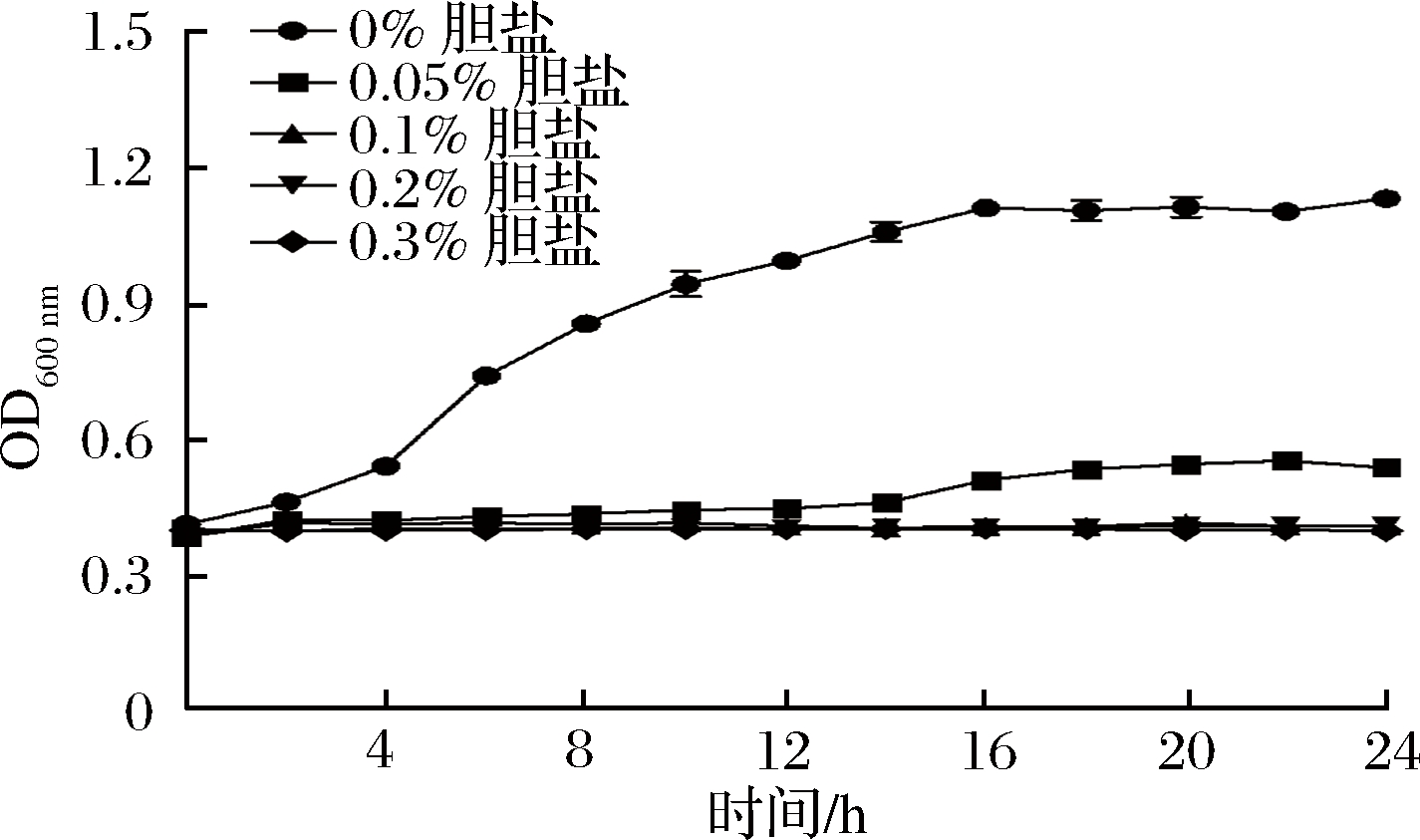
图4 耐久肠球菌C11对不同质量分数胆盐的耐受性
Fig.4 Tolerance of E. durans C11 to different concentrations of bile salt
2.5 E. durans C11菌株对人工胃肠液的耐受性
E. durans C11菌株对人工胃液(pH 2.0)、人工肠液(pH 6.8)的耐受性如表2所示。在人工胃液中处理180 min后,E. durans C11菌株的存活率为52.5%;而在人工肠液中处理240 min后,E. durans C11的存活率为85.1%,表明E. durans C11菌株对人工肠液的耐受性较强。
表2 耐久肠球菌C11对人工胃液及肠液的耐受性
Table 2 Tolerance of E. durans C11 to artificialgastric and intestinal fluids

菌株人工胃液(lg CFU/mL)1 min180 min存活率/%人工肠液(lg CFU/mL)1 min240 min存活率/%E. durans C112.552.2752.55.715.6485.1
2.6 E. durans C11基因组草图特征
运用Illumina Hiseq二代测序技术,本研究测定获得了E. durans C11的全基因组草图,共获得3 126 666条reads,序列全长为2 988 204 bp,GC含量为37.68%。序列装配得到111个Scaffolds,测序深度为151×。通过基因组序列分析,预测到2 986 个基因,包括2 715个蛋白编码基因,77个RNA基因,194个假基因。E. durans C11基因组中比对到Pfam域、信号肽、跨膜螺旋结构的基因分别有2 278、137、713个。另外,鉴定到3个CRISPR重复序列,其中包含1个未知的CRISPR序列,位于scaffold 18的16 679~16 757 bp之间,长度为24 bp;2个已知的CRISPR序列,分别位于scaffold 30 的 3 942~4 175 bp和scaffold 53的16 419 ~16 618 bp之间,长度分别为24、36 bp,分别编码假设蛋白和复制蛋白(replication protein, RepA)。还鉴定到1个完整的原噬菌体基因簇,位于scaffold 3的580~16 832 bp之间,序列全长为16.2 kb,共编码25个蛋白,其中20个编码原噬菌体相关蛋白,5个编码假设蛋白(表3)。
表3 E. durans C11基因组测序数据
Table 3 E. durans C11 genome statistics
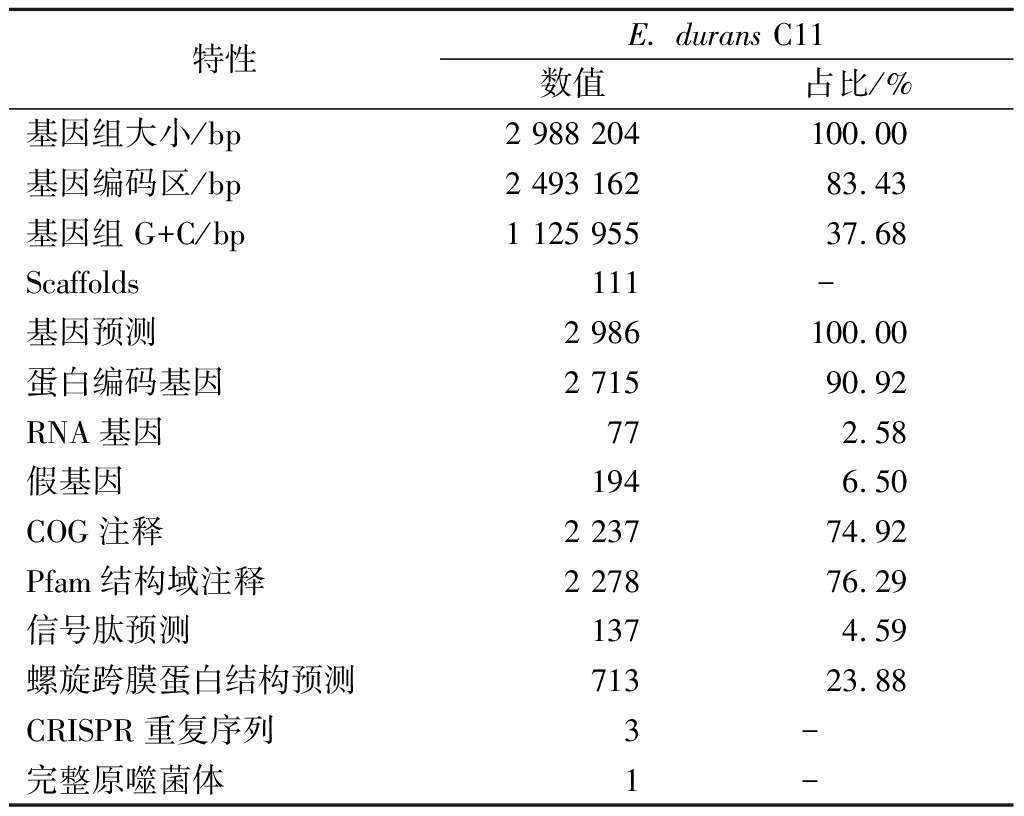
特性E. durans C11数值占比/%基因组大小/bp2 988 204100.00基因编码区/bp2 493 16283.43基因组G+C/bp1 125 95537.68Scaffolds111- 基因预测2 986100.00蛋白编码基因2 71590.92RNA基因772.58假基因1946.50COG注释2 23774.92Pfam结构域注释2 27876.29信号肽预测1374.59螺旋跨膜蛋白结构预测71323.88CRISPR重复序列3- 完整原噬菌体1-
注:“-”:未知
E. durans C11基因组中还鉴定到2个耐药基因(tetm、baca),分别与四环素(tetracycline)、杆菌肽(bacitracin)抗性相关。此外,还发现了17个编码黏附素、产胞外酶、膜表面蛋白、荚膜多糖等相关基因(efaA、eno、gapA、tuf、bsh、GroEL、bopD、srt2/srt1/bee1/bee3、pilF、pilB、hasC),可能与耐久肠球菌对宿主细胞的黏附、定殖、侵染等相关[27]。
2.7 比较基因组分析
2.7.1 E. durans C11基因组预测蛋白质编码基因的COG功能分类
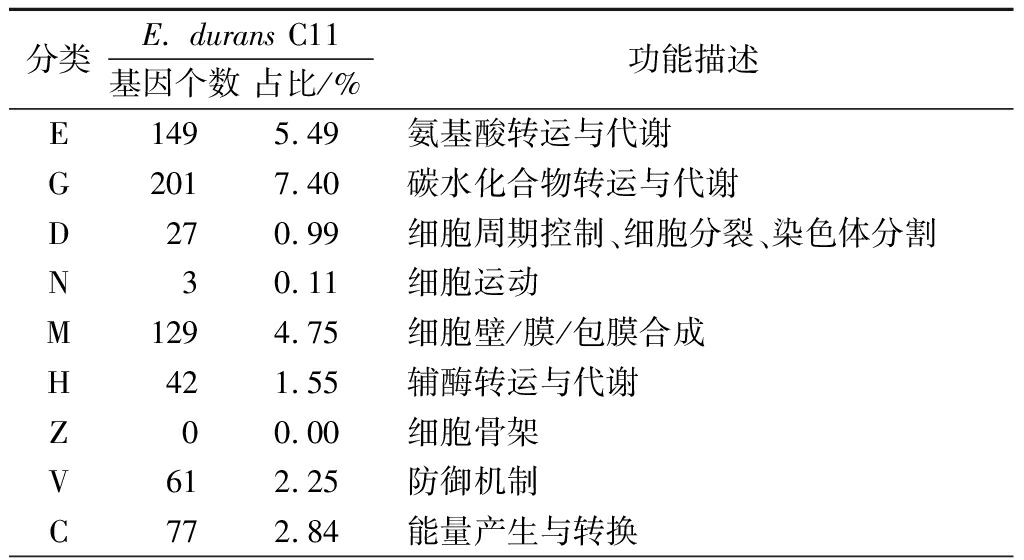
比较基因组分析揭示了18株E. durans菌株的基因组中48 112个蛋白质编码基因,它们在C11菌株基因组中的COG功能分类如表4所示。
表4 E. durans C11基因组预测蛋白质编码基因的COG功能分类
Table 4 The number of genes classified into the 24 COGfunctional categories in E. durans C11 genome
分类E. durans C11基因个数占比/%功能描述E1495.49氨基酸转运与代谢G2017.40碳水化合物转运与代谢D270.99细胞周期控制、细胞分裂、染色体分割N30.11细胞运动M1294.75细胞壁/膜/包膜合成H421.55辅酶转运与代谢Z00.00细胞骨架V612.25防御机制C772.84能量产生与转换
续表4
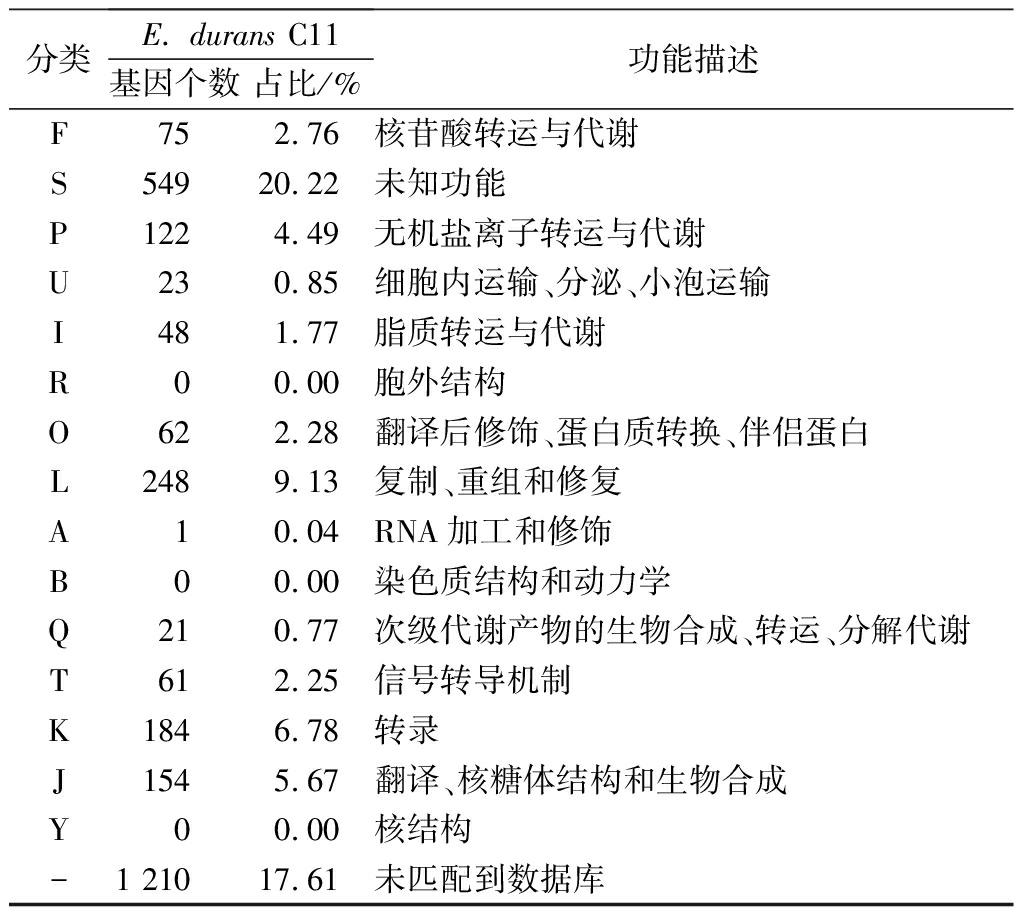
分类E. durans C11基因个数占比/%功能描述F752.76核苷酸转运与代谢S54920.22未知功能P1224.49无机盐离子转运与代谢U230.85细胞内运输、分泌、小泡运输I481.77脂质转运与代谢R00.00胞外结构O622.28翻译后修饰、蛋白质转换、伴侣蛋白L2489.13复制、重组和修复A10.04RNA加工和修饰B00.00染色质结构和动力学Q210.77次级代谢产物的生物合成、转运、分解代谢T612.25信号转导机制K1846.78转录J1545.67翻译、核糖体结构和生物合成Y00.00核结构-1 21017.61未匹配到数据库
E. durans C11基因组中约2 237个蛋白质编码基因比对到COG数据库,分属24个功能分类(表4)。其中,编码未知功能(S),复制、重组和修复(L),碳水化合物转运与代谢(G),转录(K),翻译、核糖体结构与生物合成(J)基因的占比位居前5位,分别占所有蛋白编码基因的20.22%、9.13%、7.40%、6.78%、5.67%。
2.7.2 E. durans基因组系统发育树
基于18株E. durans基因组的同源基因构建了系统发育树(图5)。由图5可见,18株E. durans的基因组聚为3簇,分别为cluster α、 β、γ。其中,cluster γ又聚为2个亚类,分别为Subcluster I和Subcluster II。在Subcluster II中,E. durans C11与E. durans NCTC8130、E. durans 18S菌株具有相近的系统发育关系,其中,E. durans 18S分离自奶酪,而E. durans NCTC8130的来源未知,它们的生物学特性均未有报道。
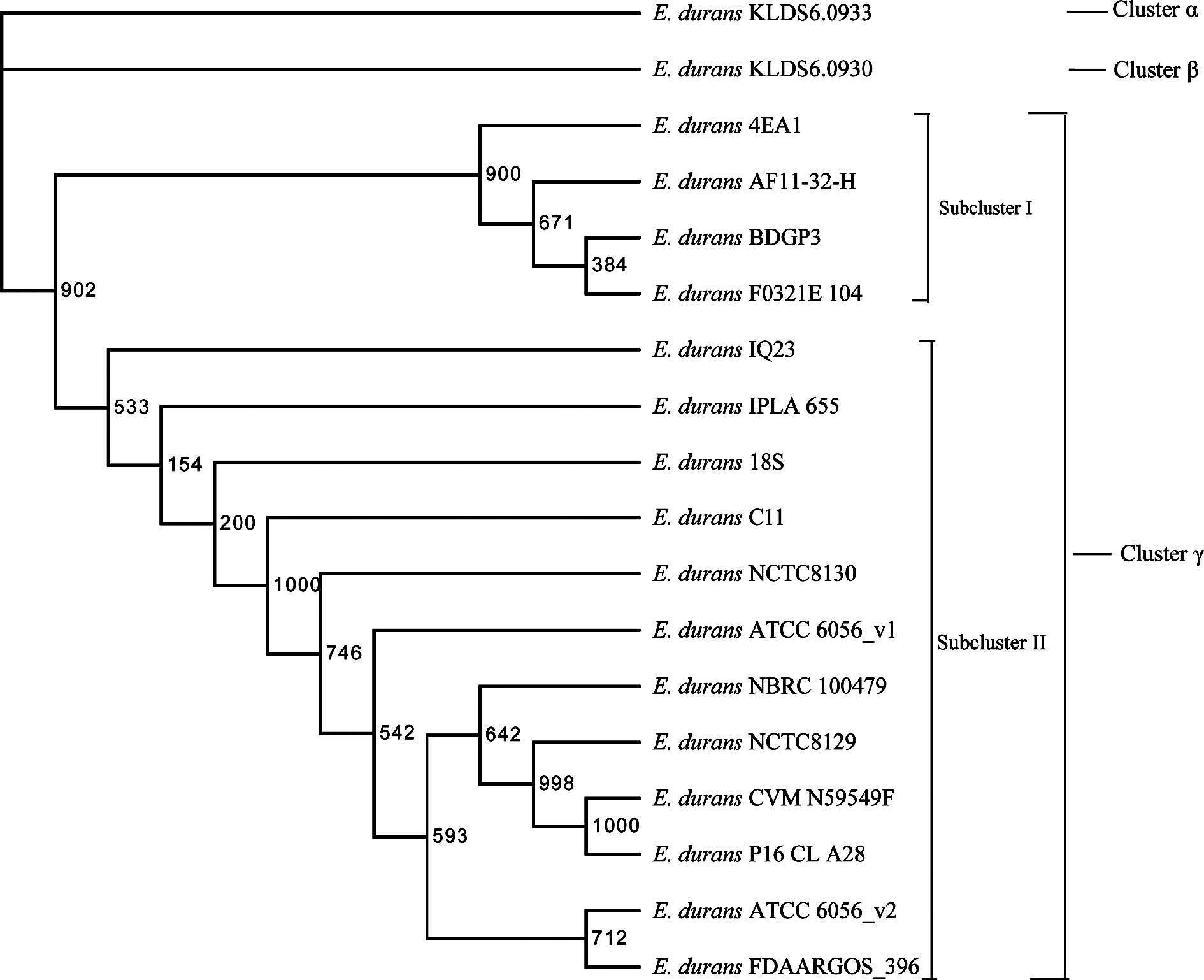
图5 18株耐久肠球菌基因组的系统发育树
Fig.5 A phylogenetic tree of the 18 E. durans genomes
2.7.3 E. durans C11菌株的特异基因
比较基因组分析揭示了E. durans C11基因组中33个菌株特异基因,其中,26个编码假设蛋白,7个分别编码螺旋转角螺旋域蛋白(helix-turn-helix domain-containing protein) (EIA52_00535)、环内酯自诱导肽(cyclic lactone autoinducer peptide) (EIA52_00555)、I型限制性内切酶R亚基(type I restriction endonuclease subunit R) (EIA52_06120)、DUF3037域结合蛋白(DUF3037 domain-containing protein) (EIA52_12720)、切除酶(excisionase) (EIA52_13810)、CPBP家族膜内金属蛋白酶(CPBP family intramembrane metalloprotease) (EIA52_13980, EIA52_13990),它们与E. durans C11的低温适应性关系机制有待进一步的研究。
2.8 E. durans C11可能的低温适应机制
2.8.1 调节细胞膜脂组成
当环境温度下降时细胞膜的流动性降低,细胞难以发挥正常生理功能。然而,耐低温菌通过改变膜脂组成,如提高不饱和脂肪酸比例来调节细胞膜的流动性[28]。在大肠杆菌(Escherichia coli)中,参与不饱和脂肪酸合成的2个关键酶为3-羟基癸脂酰ACP脱水异构酶(3-hydroxydecanoyl-ACP dehydratase/isomerase, FabA)和3-酮脂酰ACP合成酶 I(3-Ketoacyl-ACP synthase I, FabB)[29]。研究发现,革兰氏阳性菌粪肠球菌(Enterococcus faecalis)基因组中含有fabZ1 (EIA52_09905)和fabF1 (EIA52_10800)基因,分别与大肠杆菌fabA和fabB序列高度同源[30]。在本研究中,从E. durans C11基因组中鉴定到fabZ1和fabF1基因,它们与E. faecalis中的fabZ1和fabF1有较高的氨基酸序列同源性(97%, 91%),推测E. durans C11可能通过FabZ1-FabF1途径合成不饱和脂肪酸,以增加细胞膜的流动性,减少低温对细胞的损伤。
2.8.2 增加细胞内相容性溶质(compatible solutes)
据文献报道,相容性溶质(如甘氨酸、甜菜碱、甘油、海藻糖、甘露醇和山梨醇等)能够在细胞质内积累到很高浓度,并保护细胞免受低温、高温、干燥等应激损害[31]。在本研究中,通过比较基因组分析,在E. durans C11基因组中鉴定到编码甘氨酸/甜菜碱/胆碱转运(glycine betaine/carnitine transport)的基因簇(OpuABCD) (EIA52_01405, EIA52_12825, EIA52_12830, EIA52_12835, EIA52_12840)。研究发现,在15 ℃培养条件下,枯草芽孢杆菌(Bacillus subtilis)利用甜菜碱转运蛋白OpuA、OpuC和OpuD将甜菜碱摄入细胞,或利用前体胆碱合成甜菜碱,从而保护细胞免受低温损伤[32]。
2.8.3 改变蛋白质的氨基酸组成
KREIL等[33]研究发现,当温度降低时,蛋白质中谷氨酰胺(Gln)、丝氨酸(Ser)、苏氨酸(Thr)、组氨酸(His)的含量增加。另外,嗜冷酶通过疏水性脯氨酸(Pro)含量的减少来提高酶的柔韧性[34],使酶的活性中心更容易与底物接近,使之在低耗能时发挥作用。本研究中E. durans C11在低温条件下表达蛋白质中Gln、Ser、Thr、His和Pro的含量变化情况有待蛋白质组学的进一步研究。
2.8.4 冷激蛋白和热激蛋白
冷激蛋白(cold shock proteins, Csps)参与细胞内多种代谢途径,如转录、翻译、蛋白质折叠,以及细胞膜流动性的调控等[35]。热激蛋白(heat shock proteins, Hsps)不仅在细菌的热应激,而且在冷应激中也发挥作用,原因在于许多Hsps是分子伴侣,在低温及其他应激条件下同样被诱导表达[36]。研究发现,当温度发生变化时,分子伴侣可以在原位调控蛋白质暴露的活性区域之间的相互作用,使蛋白折叠成为正确的构象[37]。在本研究中,比较基因组分析揭示了E. durans C11基因组4个分子伴侣:GrpE (EIA52_03480)、DnaJ (EIA52_03490)、DnaK (EIA52_03485)、hslO (EIA52_12115);2个Hsps (EIA52_03735, EIA52_12085);1个Hsps转录调节因子hrcA (EIA52_03475);6个Csps (EIA52_03155, EIA52_04230, EIA52_04370, EIA52_09445, EIA52_13120, EIA52_11270)。此外,在E. durans C11基因组中还鉴定到1个DEAD-box ATP-dependent RNA解旋酶编码基因(CshB,EIA52_09480),与DEAD-box RNA解旋酶CsdA(Psyc_1082)氨基酸序列同源性为32%。当温度下降时,CsdA促进mRNA的解旋,调控微生物基因表达[38]。
2.8.5 应激相关调节子
转录组学研究发现,在低温条件下,副溶血性弧菌(Vibrio parahaemolyticus) CHN25的应激相关调节子编码基因表达上调,例如LysR、GntR、MerR家族转录调节子、3’-5’-环磷酸腺苷(cAMP)受体蛋白(CRP)。在本研究中,在E. durans C11基因组中鉴定到CRP(EIA52_03380, EIA52_06660, EIA52_08790, EIA52_13485)、LYSR(EIA52_00465, EIA52_09350, EIA52_09710, EIA52_12995)、GntR(EIA52_00775, EIA52_00995, EIA52_03005, EIA52_04360, EIA52_05535, EIA52_11320)、MerR(EIA52_02670, EIA52_03215, EIA52_03870) 家族转录调节子等编码基因[39]。CRP是一种参与糖代谢的调节因子,在大肠杆菌的冷适应中发挥重要作用。据研究报道,细菌对于零下温度的适应涵盖了能量的合成、翻译、运输等方面[40]。例如,低温嗜冷杆菌(Psychrobacter cryohalolentis) K5菌株能够在-4 ℃下诱导一些重要调控子的表达,如调节能量需求的F1/F0 ATP合成酶,在低温下排除有害物质的外膜外排系统蛋白(TolC),加快翻译过程的延伸因子Ts(EF-Ts)等,这些基因在E. durans C11基因组中均被鉴定到(EIA52_00860, EIA52_06915, EIA52_06625),可能与其低温适应性相关。
3 结论
本研究发现耐久肠球菌C11菌株能够在15~25 ℃温度条件下生长。该菌株的基因组草图已被测定,全长2 988 204 bp,GC含量37.68%,GenBank序列登录号为RQWF00000000。比较基因组学分析揭示了E. durans C11的33个菌株特异性基因,以及大量参与细胞膜脂组成、相容性溶质吸收或合成、环境胁迫应激调控等相关基因,可能与其低温适应相关。转录组学和蛋白质组学的进一步分析将有助于阐释耐久肠球菌低温适应的分子调控网络。
[1] GUEIMOND M,FRIAS R,OUWEHAND A C.Assuring the continued safety of lactic acid bacteria used as probiotics[J]. Biologia,2006,61(6):755-760.
[2] LADERO V,LINARES D M,DEL R B,et al.Draft genome sequence of the tyramine producer Enterococcus durans strain IPLA 655[J].Genome Announcements,2013,1(3):e00 265-13.
[3] AVRAMHANANEL,LIRAZ,JULIA,et al.E. durans strain M4-5 isolated from human colonic flora attenuates intestinal inflammation[J].Diseases of the Colon and Rectum,2010,53(12):1 676-1 686.
[4] RAMAKRISHNAN V,GOVEAS L,CONCEPTA,et al.Kinetic modeling, production and characterization of an acidic lipase produced by Enterococcus durans NCIM5427 from fish waste[J].Journal of Food Science and Technology,2015, 52(3):1 328-1 338.
[5] 黄坚,童京京,岳华,等.牦牛发酵酸奶中耐久肠球菌的筛选鉴定和益生特性[J].食品科学,2017,38(12):43-49.
[6] LIU Fei,LI Bailiang,DU Jincheng,et al.Complete genome sequence of Enterococcus durans KLDS6.0930,a strain with probiotic properties[J].Journal of Biotechnology,2016,217:49-50.
[7] 隋馨瑶,王莹,张卫兵,等.低温乳酸菌的研究与应用现状[J].食品与发酵科技,2017,53(4):83-87.
[8] LI Ping,GU Qiang.Complete genome sequence of Lactobacillus plantarum LZ95,a potential probiotic strain producing bacteriocins and B-group vitamin riboflavin[J].Journal of Biotechnology,2016,229:1-2.
[9] LI Junfeng,YUAN Xianjun,DESTA S T,et al. Characterization of Enterococcus faecalis JF85 and Enterococcus faecium Y83 isolated from Tibetan yak (Bos grunniens) for ensiling Pennisetum sinese[J].Bioresource Technology,2018,257:76.
[10] XU Shuang,LIU Taigang,RADJI C A,et al.Isolation, identification, and evaluation of new lactic acid bacteria strains with both cellular antioxidant and bile salt hydrolase activities in vitro[J].Journal of Food Protection,2016,79(11):1 919-1 928.
[11] ZHANG Bei,WANG Yanping,TAN Zhongfang,et al.Screening of probiotic activities of Lactobacilli strains isolated from traditional Tibetan Qula,a raw yak milk cheese[J].Asian-Australasian Journal of Animal Sciences,2016,29(10):1 490-1 499.
[12] BROWN J,PIRRUNG M,MCCUE L A.FQC Dashboard. integrates FastQC results into a web-based,interactive,and extensible FASTQ quality control tool[J].Bioinformatics,2017,33(19):3 137-3 139.
[13] DELCHER A L,BRATKE K A,POWERS E C,et al.Identifying bacterial genes and endosymbiont DNA with Glimmer[J]. Bioinformatics,2007,23(6):673-679.
[14] LOWE T M,EDDY S R.tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Res,1997,25(5):955-964.
[15] LAGESEN K,HALLIN P,RODLAND E A,et al.RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Res,2007,35(9):3 100-3 108.
[16] GRISSA I,VERGNAUD G,POURCEL C.CRISPRFinder: a web tool to identify clustered regularly interspaced short palindromic repeats[J].Nucleic Acids Research,2007,35:52-57.
[17] TATUSOVA T,DICUCCIO M,BADRETDIN A,et al.NCBI prokaryotic genome annotation pipeline[J].Nucleic Acids Research,2016,44(14):6 614-6 624.
[18] MARCHLER-BAUER A,DERBYSHIRE M K,GONZALES N R,et al.CDD:NCBI's conserved domain database[J].Nucleic Acids Research,2015,43:D222-226.
[19] PETERSEN T N,BRUNAK S,V HEIJNE G,et al.SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods,2011,8(10):785-786.
[20] KROGH A,LARSSON B,VON H G,et al.Predicting transmembrane protein topology with a hidden Markov model: application to complete genomes[J].Journal of Molecular Biology,2001,305(3):567-580.
[21] FU Limin,NIU Beifang,ZHU Zhengwei,et al.CD-HIT:accelerated for clustering the next-generation sequencing data[J]. Bioinformatics,2012,28(23):3 150-3 152.
[22] EDGAR R C.MUSCLE:multiple sequence alignment with high accuracy and high throughput[J].Nucleic Acids Research, 2004,32(5):1 792-1 797.
[23] GUINDON S,GASCUEL O.A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood[J]. Systematic Biology,2003,52(5):696.
[24] ZHANG Huangkai,GAO Shenghan,LERCHER M J,et al.EvolView,an online tool for visualizing, annotating and managing phylogenetic trees[J].Nucleic Acids Research,2012,40:569-572.
[25] LI Bailiang,EVIVIE S E,JIN D,et al.Complete genome sequence of Enterococcus durans KLDS6.0933, a potential probiotic strain with high cholesterol removal ability[J].Gut Pathogens,2018,10(1):32.
[26] WAN K H,YU C,PARK S,et al.Complete genome sequence of Enterococcus durans Oregon-R-modENCODE strain BDGP3,a lactic acid bacterium found in the Drosophila melanogaster gut[J].Genome Announcements,2017,5(40):e01 041-01 017.
[27] 祝进,白永凤,陆军,等.粪肠球菌毒力基因及耐药性分析[J].放射免疫学杂志,2012,25(3):276-279.
[28] 辛玉华,周宇光,东秀珠.低温细菌与古菌的生物多样性及其冷适应机制[J].生物多样性,2013,21(4):468-480.
[29] 余永红,马建荣,王海洪.细菌脂肪酸合成多样性的研究进展[J].微生物学杂志,2016,36(4):76-83.
[30] CASANUEVA A,TUFFIN M,CARY C,et al.Molecular adaptations to psychrophily:the impact of 'omic' technologies[J]. Trends In Microbiology,2010,18(8):374-381.
[31] WELSH D T.Ecological significance of compatible solute accumulation by micro-organisms:from single cells to global climate[J].FEMS Microbiology Reviews,2000,24(3):263-290.
[32] TAMARA H,ERHARD B.Protection of Bacillus subtilis against cold stress via compatible-solute acquisition[J].Journal of Bacteriology,2011,193(7):1 552-1 562.
[33] KREIL D P,OUZOUNIS C A.Identification of thermophilic species by the amino acid compositions deduced from their genomes[J].Nucleic Acids Research,2001,29(7):1 608-1 615.
[34] KASANA R C,GULATI A.Cellulases from psychrophilic microorganisms:a review[J].Journal of basic Microbiology,2011, 51(6):572-579.
[35] PHADTARE S.Recent developments in bacterial cold-shock response[J].Current Issues in Molecular Biology,2004, 6(2):125-136.
[36] CHATTOPADHYAY M K.Mechanism of bacterial adaptation to low temperature[J].Journal of Biosciences (Bangalore),2006, 31(1):157-165.
[37] 刘芳明.3株南极海洋石油烃低温降解菌(Shewanella sp.NJ49、Pseudoalteromonas sp.NJ289和Planococus sp.NJ41)基因组学及比较研究[D].青岛:中国科学院研究生院(海洋研究所),2016.
[38] KUHN E.Toward understanding life under subzero conditions:the significance of exploring psychrophilic "cold-shock" proteins[J].Astrobiology,2012,12(11):1 078-1 086.
[39] ZHU Chunhua,SUN Boyi,LIU Taigang,et al.Genomic and transcriptomic analyses reveal distinct biological functions for cold shock proteins (VpaCspA and VpaCspD) in Vibrio parahaemolyticus CHN25 during low-temperature survival[J].BMC Genomics,2017,18(1):436.
[40] BAKERMANS C,TOLLAKSEN S L,GIOMETTI C S,et al. Proteomic analysis of Psychrobacter cryohalolentis K5 during growth at subzero temperatures[J].Extremophiles:Life under Extreme Conditions,2007,11(2):343-354.