甘薯粉丝(条)是以甘薯淀粉为主要原料,经和浆(打糊)、成型(漏粉)、冷却等程序制成的条状或丝状食品。甘薯淀粉中支链淀粉含量较高,具有独特的高黏性,加工成的甘薯粉丝(条)有较强的弹性,筋道、耐煮,透明度较高。因其具有久煮不烂、清香可口、食法多样的特点,成为中国大部分地区群众喜爱和常见的食品。中国地理标志保护产品,如筠连粉条、周礼粉条、铜仁红薯粉丝、卢龙粉丝等均是以甘薯为生产原料。
由于市场上甘薯淀粉与玉米淀粉、木薯淀粉价格差异较大,一些企业或个人受经济利益驱使,使用掺入玉米淀粉或木薯淀粉的甘薯淀粉为原料生产粉丝,冒充纯甘薯淀粉粉丝,使广大消费者利益受到损害。康维民等[1]根据玉米淀粉与甘薯淀粉遇碘制剂呈现不同的显色反应原理,用分光光度计测定混合淀粉试液的吸光值,并通过数据拟合回归方程确定混合淀粉中玉米淀粉的含量;侯汉学等[2]发现甘薯淀粉中玉米淀粉含量与平均粒径、糊化峰黏度有较强的相关性,可以根据回归方程确定甘薯淀粉中玉米淀粉的含量,以解决粉条加工中甘薯淀粉不纯、混掺玉米淀粉等问题,但上述方法都是针对原料淀粉,对甘薯粉丝(条)成品是否掺假无法检测。杜连起[3]利用粉条膨润度、煮沸损失和耐煮性的差异,结合外观和口感的变化检验甘薯粉条中是否掺假,但该方法需要专业人员对粉丝进行感官评定,且操作复杂,并不适合快速检测。
近红外光谱(near infrared spectra, NIRS)分析技术具有快速、效率高、成本低、无污染、无需前处理等优点[4],被广泛应用于成分检测[5-7]、产地鉴别[8-10]、品质鉴定[11-12]、产品分级[13-14]、掺假识别[15-17]等方面。LU等[18]尝试用NIRS预测甘薯淀粉的热力学性质以及甘薯粉丝的品质,结果显示,使用NIRS可以较为准确地预测甘薯淀粉的糊化起始温度、顶点温度、温程以及粉丝的耐煮性,同时对甘薯淀粉糊化终点温度、糊化焓及甘薯粉丝的煮沸损失和膨润度均有一定的预测能力。DING等[19]成功地使用NIRS对紫薯淀粉以及掺假紫薯淀粉进行了鉴别,并建立了紫薯淀粉花青素含量及总抗氧化活性定量检测模型。许多研究表明,支持向量机(support vector machine, SVM)在解决小样本、非线性样品分类及高维模式识别中表现出特有的优势,并对样本异常值及噪音具有很强的鲁棒性[20]。有研究将SVM应用于羊肉掺假[21]、淀粉的分类[22]和品质预测[23],均取得了良好的效果。目前,基于NIRS及SVM的甘薯粉丝成品掺假快速判别及定量检测方法尚未见报导。本实验以甘薯粉丝为对象,分别采用9种不同厂家生产的甘薯淀粉为原料,掺入一定比例的木薯淀粉或玉米淀粉制成粉丝样品,探讨采用NIRS结合支持SVM对甘薯粉丝掺假进行快速检测的可行性,考察不同预处理方式对定性判别及定量分析模型的影响,并通过化学计量学方法对模型进行优化,以期为准确、快速鉴别甘薯粉丝的真伪及掺假情况提供参考。
1 材料与方法
1.1 材料
共收集9种不同厂家生产的甘薯淀粉为原料,分别用代号a、b、c、d、e、f、g、h、i代表。其中8种来自大型超市,并经显微形态检查符合甘薯淀粉的一般特征;1种直接采集自合作企业。玉米淀粉、木薯淀粉,山东成武华丰淀粉有限责任公司。
1.2 方法
1.2.1 甘薯粉丝样品的制作
向甘薯淀粉中掺入一定比例的玉米淀粉和(或)木薯淀粉,混合均匀后,参照NY/T 982—2006《甘薯粉丝加工技术规范》中的方法,采用漏粉法加工甘薯粉丝,具体操作如下:称取10 g(以干基计)淀粉与85 g温水(30 ℃左右)于容器中搅拌混合,然后在沸水中不断搅拌90 s后形成芡糊。将芡糊与190 g(以干基计)淀粉一起转移至和面机中进行和面。最初5 min内缓缓加入75 g温水(30 ℃左右),以150 r/min的速度继续搅拌10 min制得粉团。采用圆孔直径为2.5 cm的漏瓢将粉团漏成细线,在沸水(位于漏瓢下方35 cm处)中熟化20 s后立即捞至自来水中冷却5 min,沥干,保鲜膜包覆后放入4 ℃冰箱中冷藏,24 h后取出理丝。将理丝后的粉丝置于40 ℃电热恒温鼓风干燥箱中干燥8 h,密封,放置在干燥箱中备用。
本实验选取了9个不同厂家生产的甘薯淀粉作为原料,共制得样品168个,其中纯甘薯淀粉样品36个,甘薯+玉米淀粉样品36个,甘薯+木薯淀粉样品36个,甘薯+玉米+木薯淀粉样品52个,纯玉米淀粉及纯木薯淀粉样品各4个。从中随机取出126个样品作为校正集,其余42个样品作为验证集(详见表1)。实验拟建立定性判别及定量分析2种模型,对于定性判别模型,仅需考虑样品掺假与不掺假(即100%甘薯淀粉样品)2种情况。
表1 甘薯粉丝样品的制备及样品划分
Table 1 Preparation and partition of sweet potato starchnoodle samples
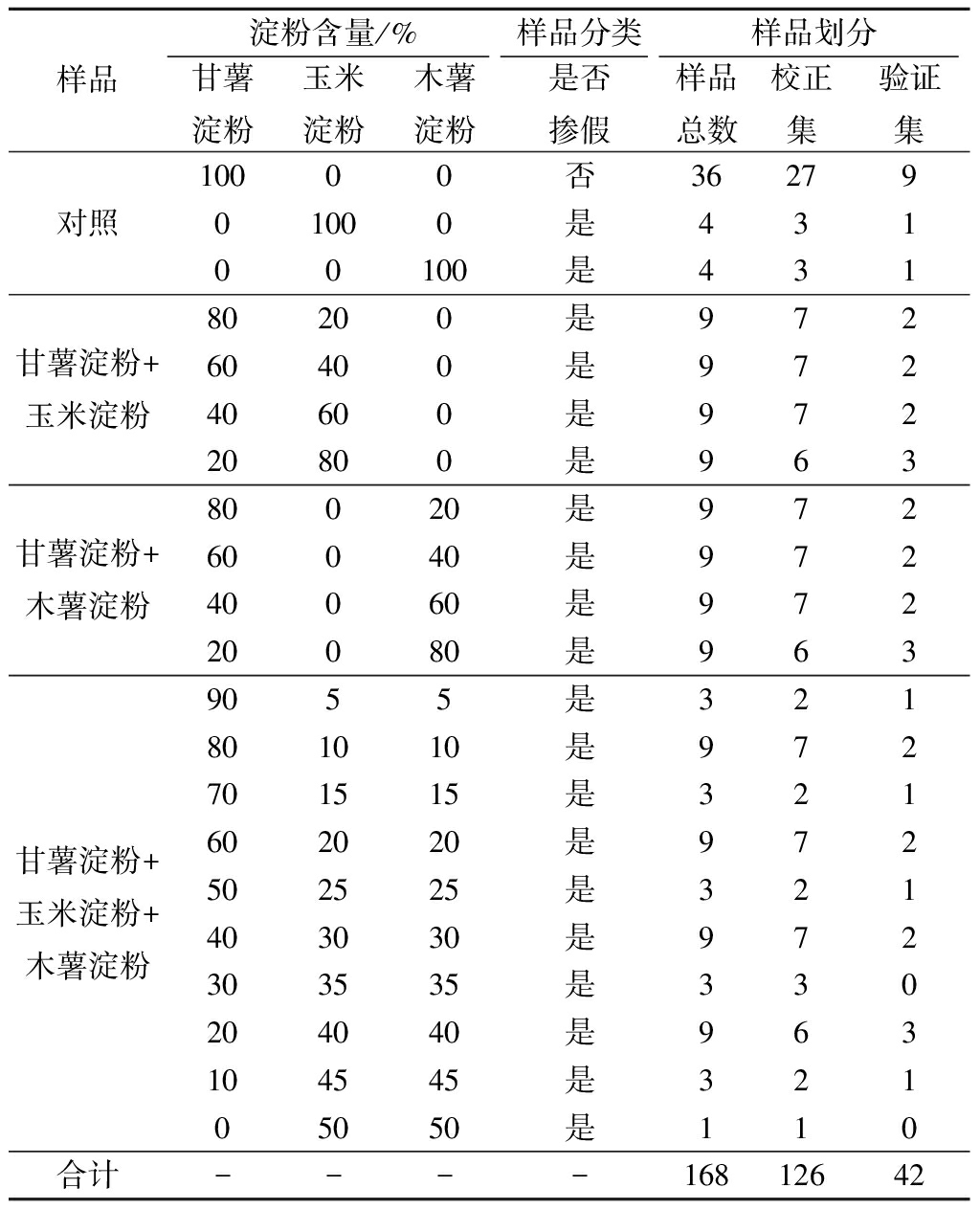
样品淀粉含量/%样品分类样品划分甘薯淀粉玉米淀粉木薯淀粉是否掺假样品总数校正集验证集对照10000否3627901000是43100100是431甘薯淀粉+玉米淀粉80200是97260400是97240600是97220800是963甘薯淀粉+木薯淀粉80020是97260040是97240060是97220080是963甘薯淀粉+玉米淀粉+木薯淀粉9055是321801010是972701515是321602020是972502525是321403030是972303535是330204040是963104545是32105050是110合计----16812642
1.2.2 近红外光谱采集
采用德国Bruker公司MPA近红外光谱仪。将粉丝样品粉碎后过100目筛,放入石英样品杯中,采用PbS检测器漫反射方式采集近红外光谱,光谱扫描范围12 000~4 000 cm-1,分辨率4 cm-1,扫描次数16次,采用仪器自带的OPUS 7.0采集并处理光谱。
1.2.3 数据处理及建模
采用Matlab 2016a(美国MathWorks公司)软件。在构建模型前,光谱须经过适当的预处理,提高光谱的分辨率和灵敏度[24]。实验中采用标准正态变量变换(standard normal variate transformation,SNV)、一阶导数(1st Der)、二阶导数(2nd Der)、SNV+1st Der及SNV+2nd Der五种方法进行光谱预处理,以考察不同光谱预处理方法对模型预测能力的影响。
SVM判别模型采用Lib-SVM工具箱[25]的c-SVM模型建立,核函数采用径向基核函数(radial basis function, RBF),最优模型参数(惩罚参数c和RBF核参数g)通过网格搜索法[26]获得。为了对比SVM判别模型的预测效果,同时构建了马氏距离判别模型[27]进行对比。根据表1校正集与验证集的划分情况,校正集126个样品中有不掺假甘薯粉丝(100%甘薯淀粉粉丝)27个、掺假甘薯粉丝99个,验证集42个样品中有不掺假甘薯粉丝(100%甘薯淀粉粉丝)9个、掺假甘薯粉丝33个,以此构建定性判别模型,并根据判别准确率评价模型的分类能力。判别准确率越高,说明模型的判别分析能力越强。
SVM定量分析模型采用Lib-SVM工具箱的epsilon-SVR模型建立,核函数采用RBF核函数,网格搜索获得最佳模型参数。利用模型对预测集样品的预测均方差(root mean square error of prediction, RMSEP)、预测值与实测值间的相关系数r进行对模型的预测性能考察。RMSEP主要用于评价模型对于外部样本的预测能力,其值越小表明预测能力越高,反之则越低;相关系数r用于衡量验证集样本的预测值和实测值之间的相关程度,r越接近于1,表明相关程度越好。同时,为了进一步增强SVM模型的预测能力,实验采用了前向区间支持向量机(forward interval support vector machine, fi-SVM)变量筛选算法[28]对光谱波长进行筛选。
2 结果与分析
2.1 甘薯粉丝与掺假甘薯粉丝红外光谱比较及主成分分析
图1是甘薯粉丝与掺假甘薯粉丝的近红外光谱图。近红外光谱主要反映的是分子中化学键振动的倍频和合频信息,由于近红外光谱的特性,在12 000~4 000 cm-1波数范围内,甘薯粉丝与掺假甘薯粉丝近红外光谱的峰形和位置都非常相似,无法通过图谱直观地进行区分与检测。甘薯粉丝是由甘薯淀粉经打芡、和面、漏粉、煮熟、冷却等一系列工序加工而成,因此甘薯粉丝的近红外光谱与以往文献报导中淀粉的近红外光谱极为相似[29],光谱图中几处明显的波峰及其对应的化学键振动分别为:7 100 cm~6 500 cm-1处较宽的波峰对应的是O—H或N—H伸缩振动,5 350 cm-1附近为O—H合频振动,4 960 cm-1附近为N—H合频振动,4 440 cm-1附近为C—H合频振动,3 900 cm-1 附近为C—N—C伸缩振动的一级倍频以及C—H、C—C和C—O—C伸缩振动[23]。
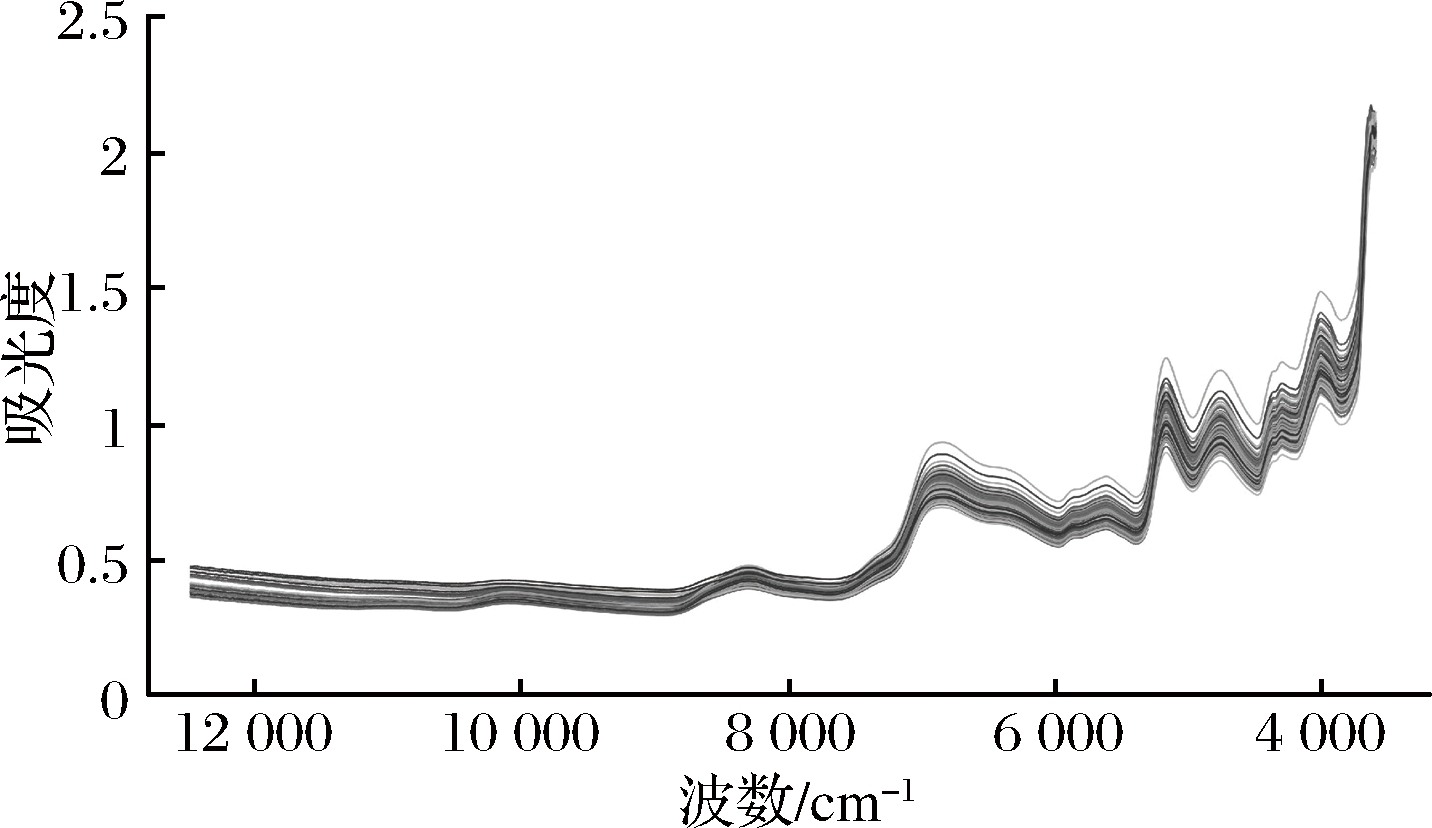
图1 甘薯粉丝与掺假甘薯粉丝近红外光谱图
Fig.1 Near infrared spectra of sweet potato starch noodles and adulterated sweet potato starch noodles
对样品光谱进行主成分(principal component analysis, PCA)提取,并以前3个主成分得分作图,结果见图2。

图2 粉丝样品前3个主成分得分图
Fig.2 Score plot for the first three principal components of starch noodle samples
由图2可以看出,由于选取了9个不同厂家的甘薯淀粉作为原料,同一厂家甘薯淀粉制作的粉丝相对聚集,不同厂家甘薯淀粉粉丝分散在不同的区域内,掺假的粉丝样品分散在其周围,无明显的聚集或集中区域,无法直观地通过主成分加以区分。
2.2 甘薯粉丝掺假SVM定性判别模型
选择适当的光谱预处理方法对建立预测能力强、稳健性好的分析模型至关重要[24]。实验中采用SNV、1st Der、2nd Der、SNV+1st Der及SNV+2nd Der五种方法进行光谱预处理,提取前20个主成分构建SVM判别模型和马氏距离判别模型,对校正集和验证集样品进行判别分析,结果见表2。
表2 不同光谱处理方法对SVM判别模型和马氏距离判别模型的影响
Table 2 Effects of different spectra preprocessing methodson SVM and mahalanobis distance discriminant models
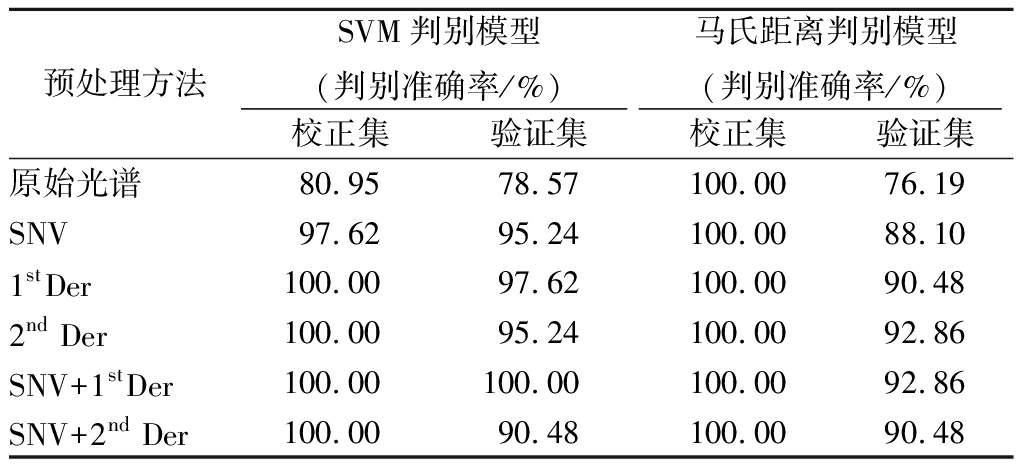
预处理方法SVM判别模型(判别准确率/%)马氏距离判别模型(判别准确率/%)校正集验证集校正集验证集原始光谱80.9578.57100.0076.19SNV97.6295.24100.0088.101stDer100.0097.62100.0090.482nd Der100.0095.24100.0092.86SNV+1stDer100.00100.00100.0092.86SNV+2nd Der100.0090.48100.0090.48
注:SNV,标准正态变量变换;1stDer,一阶导数;2nd Der,二阶导数。下同。
可以看出,以上5种光谱预处理方式均可以提升SVM判别模型及马氏距离判别模型的判别准确率。马氏距离判别模型对于校正集数据的判别准确率较高,但对于验证集数据,原始光谱模型的判别准确率仅为76.19%,经过适当的光谱预处理后判别准确率最高可达到92.86%。原始光谱SVM判别模型对验证集的判别准确率为78.57%,经过SNV+1stDer预处理后,模型对于校正集和验证集的判别准确率均达到100%。在相同的光谱预处理方式下,对于验证集SVM模型的判别准确率均高于马氏距离判别模型,这可能是因为SVM对非线性数据有着更好的处理能力,由于实验选取了9个不同厂家的甘薯淀粉为原料,制成的粉丝样品组成更为复杂,SVM对于非线性数据拟合具有更好的鲁棒性和容错性[30]。以往的文献中,使用近红外光谱预测高淀粉含量原料的品质时,例如用近红外及SVM预测大米[31]或大麦[32]的淀粉或蛋白质含量,也发现类似的结果。
2.3 甘薯粉丝掺假SVM定量分析模型
2.3.1 光谱预处理
采用SNV、1st Der、2nd Der、SNV+1st Der及SNV+2nd Der五种方法进行光谱预处理,分别建立木薯淀粉和玉米淀粉含量的SVM预测模型,采用内部交叉验证法确定模型的主成分数并对验证集进行验证,结果见表3。
表3 不同光谱预处理方法对SVM定量分析模型影响
Table 3 Effects of different spectra preprocessing methodson SVM quantitative analysis models
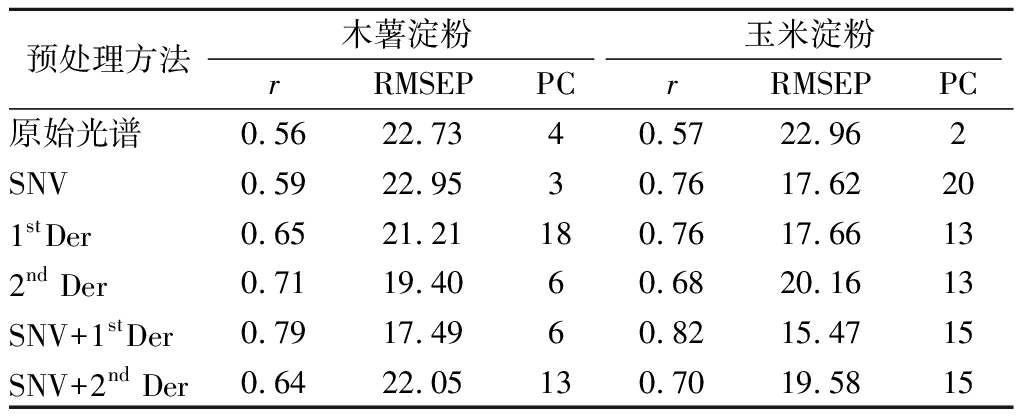
预处理方法木薯淀粉玉米淀粉rRMSEPPCrRMSEPPC原始光谱0.5622.7340.5722.962SNV0.5922.9530.7617.62201stDer0.6521.21180.7617.66132nd Der0.7119.4060.6820.1613SNV+1stDer0.7917.4960.8215.4715SNV+2nd Der0.6422.05130.7019.5815
注:r,相关系数;RMSEP,预测均方误差;PC,主成分数。下同
可以看出,相对于原始光谱模型,经过适当的光谱预处理后,模型的预测能力均有所提升。原始光谱经SNV+1stDer预处理后,木薯淀粉SVM模型的r和RMSEP分别达到0.79和17.49,玉米淀粉SVM模型的r和RMSEP分别达到0.82和15.47,优于其他方法。这可能是因为SNV消除了固体颗粒大小、表面散射以及光程变化对NIR漫反射光谱的影响,导数处理消除了基线和背景干扰,提高了光谱分辨率和灵敏度[24],但导数处理同时会引入噪声,导数阶数越高,光谱信噪比越低[33]。因此,选择SNV+1stDer作为定量分析模型的光谱预处理方法。
2.3.2 基于fi-SVM的光谱变量筛选
采用前向区间支持向量机(fi-SVM)对光谱波长进行筛选,以进一步提高SVM定量分析模型的预测精度。将全光谱划分为10~100个子区间(间隔为10),筛选出各子区间划分情况下最优fi-SVM预测模型,结果见表4。
可以看出,总体上,模型的预测效果随着子区间划分数量的增加而提升,但并不是子区间划分越多越好。对于木薯淀粉,当光谱划分为80个子区间时fi-SVM模型(记为80-fi-SVM)预测精度最高,模型的r和RMSEP分别为0.92和11.20;对于玉米淀粉,当光谱划分为70个子区间时fi-SVM模型(记为70-fi-SVM)的预测精度最高,模型的r和RMSEP分别达到0.96和7.49。因此,分别选择80和70子区间划分作为木薯淀粉和玉米淀粉fi-SVM模型的最优区间划分。
表4 不同光谱子区间划分下fi-SVM最优模型参数
Table 4 Optimal fi-SVM model parameters under differentspectral subinterval partitions
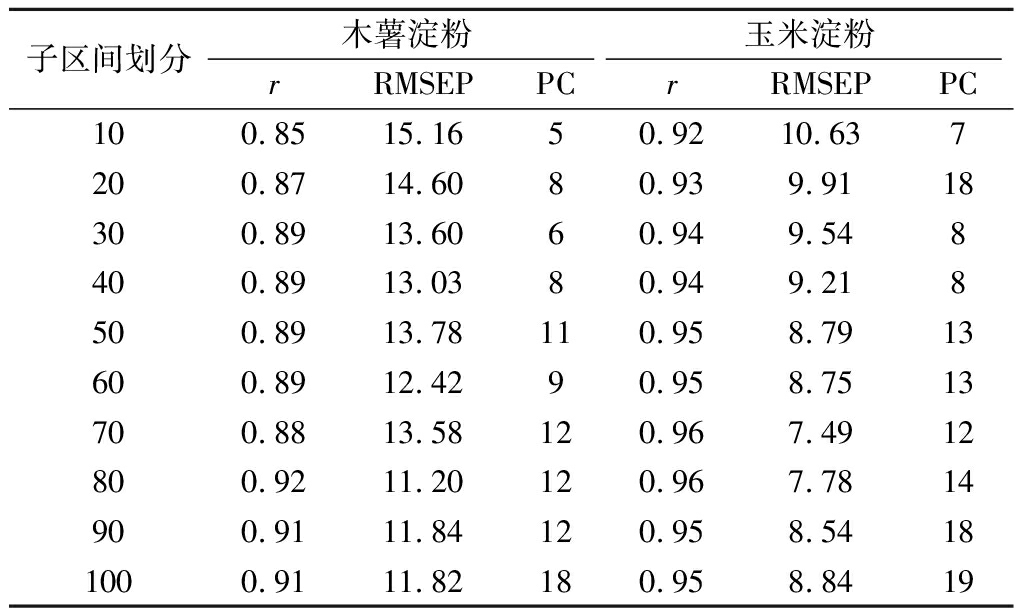
子区间划分木薯淀粉玉米淀粉rRMSEPPCrRMSEPPC100.8515.1650.9210.637200.8714.6080.939.9118300.8913.6060.949.548400.8913.0380.949.218500.8913.78110.958.7913600.8912.4290.958.7513700.8813.58120.967.4912800.9211.20120.967.7814900.9111.84120.958.54181000.9111.82180.958.8419
木薯淀粉80-fi-SVM模型和玉米淀粉70-fi-SVM模型在变量筛选过程中,模型的r和RMSEP值随固定子区间数量的增加,变化趋势见图3。
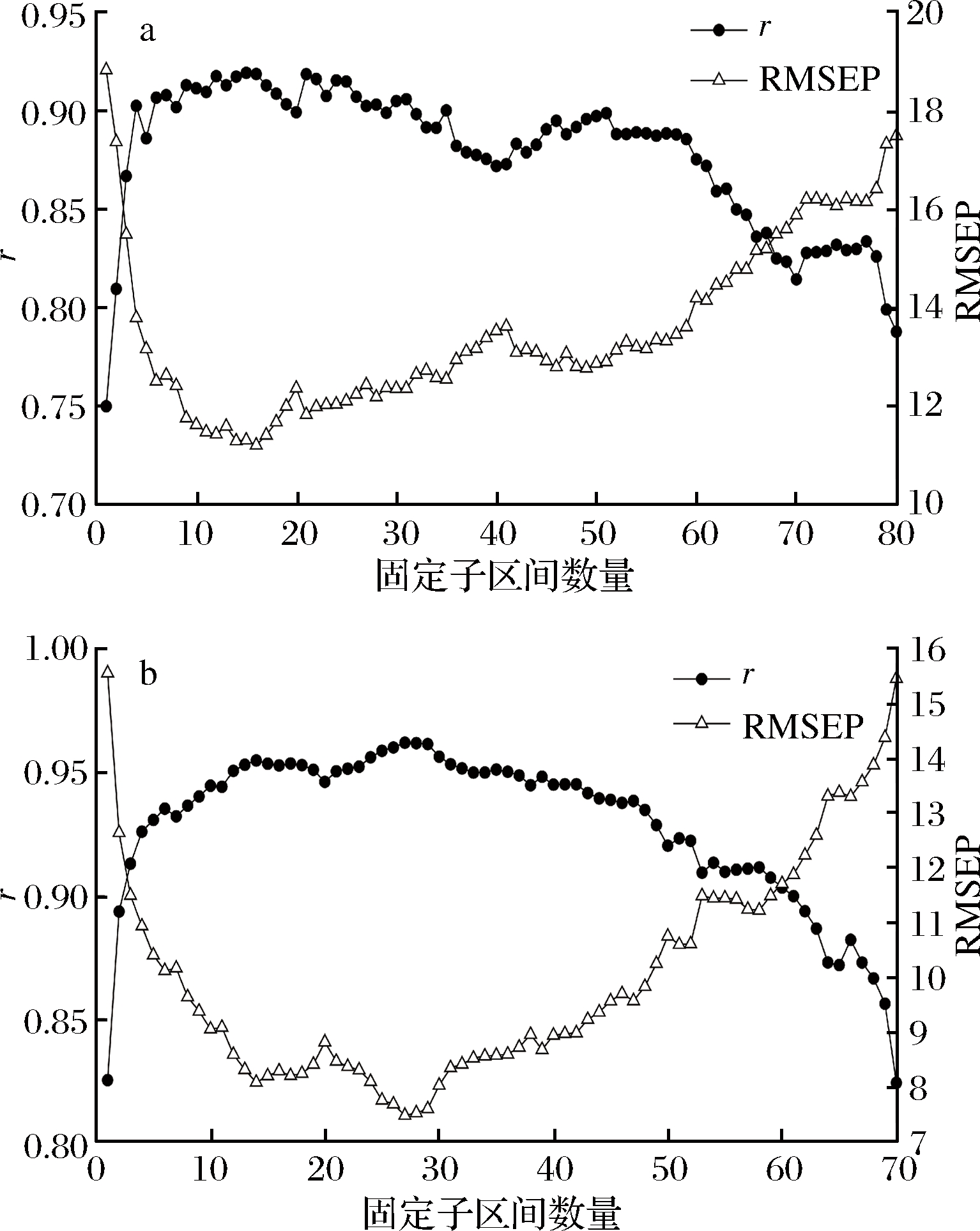
图3 木薯淀粉80-fi-SVM模型(a)和玉米淀粉70-fi-SVM模型(b)r和RMSEP值变化情况
Fig.3 Changes in r and RMSEP values in 80-fi-SVM(a)and 70-fi-SVM(b) model for determination of cassava starch and corn starch content
可以看出,在fi-SVM变量筛选过程中,模型的预测精度随算法的运行呈先升高后降低的趋势,这是因为在fi-SVM算法运行之初,筛选出的固定子区间数量较少,用于模型校正的光谱变量较少,有效信息不足,模型预测能力较低。随着fi-SVM算法的运行,固定子区间数量增加,模型中的有效信息不断积累,模型预测效果不断提升,表现为r上升,RMSEP下降。当固定子区间积累到一定数量时,RMSEP达到最小。随后RMSEP随着固定子区间数量的增加不断升高,这是因为当固定子区间数量增加到一定程度后,引入了过多无用和干扰信息导致模型预测效果下降,表现为r下降,RMSEP升高。
对于木薯淀粉80-fi-SVM模型,当固定子区间数量为16时,模型的RMSEP值最低,对应的光谱范围为[12 385.3~12 281.2 cm-1,10 418.2~10 310.2 cm-1,9 299.6~9 191.6 cm-1,8 964.0~8 520.4 cm-1,7 174.3~7 066.3 cm-1,5 943.9~5 835.9 cm-1,5 608.3~5 500.3 cm-1,4 937.2~4 829.2 cm-1,4 601.6~4 381.7 cm-1,4 266.0~4 046.1 cm-1,3 818.6~3 710.6 cm-1](相邻子区间合并书写,下同);对于玉米淀粉70-fi-SVM模型,当固定子区间数量为27时,模型的RMSEP最低,对应的光谱范围为[12 246.5~12 126.9 cm-1,11 486.6~11 363.2 cm-1,10 977.5~10 854.0 cm-1,9 704.6~9 581.2 cm-1,9 450.0~9 326.6 cm-1,8 304.4~8 181.0 cm-1,8 049.9~7 926.4 cm-1,7 413.4~7 290.0 cm-1,7 031.6~6 908.2 cm-1,6 395.2~6 144.4 cm-1,6 013.3~5 762.6 cm-1,5 631.4~4 998.9 cm-1,4 867.7~4 617.0 cm-1,4 485.9~3 598.7 cm-1]。
将筛选出的最优模型光谱子区间与全光谱区间对比作图,以灰色区域表示筛选出的光谱子区间,结果见图4。对于木薯淀粉(图4-a),最优模型使用的光谱子区间为16个,相对于全光谱模型,所用光谱变量减少了80.00%,但模型的r提升了16.46%,RMSEP下降了35.96%;对于玉米淀粉(图4-b),最优模型使用的光谱子区间为27个,相对于全光谱模型,所用光谱变量减少了61.43%,但模型的r提升了17.07%,RMSEP下降了51.58%。可见,采用fi-SVM进行变量筛选后,模型使用的光谱变量数量大幅降低,但预测能力显著提升。

图4 木薯淀粉80-fi-SVM最优模型(a)和玉米淀粉70-fi-SVM最优模型(b)光谱子区间筛选结果
Fig.4 Spectral subintervals (marked with gray bars) selected in 80-fi-SVM and 70-fi-SVM optimal model for determination of cassava starch content(a) and corn starch(b) content
2.3.3 模型验证
使用未参与建模的验证集数据对上步构建最优模型进行验证,结果见图5。对于木薯淀粉80-fi-SVM模型,预测值与真实值相关系数r达到0.92;对于玉米淀粉70-fi-SVM模型,预测值与真实值相关系数r达到0.96,且预测值与真实值在显著水平α=0.01下无显著差异,说明模型有较强的预测能力。需要强调的是,不能使用定量分析模型的结果代替定性分析,即对于纯甘薯淀粉粉丝,由于定量分析模型存在一定误差,可能会出现预测结果不为0的情况,从而可能引起误判。在实际使用中,可以先用上文中已建立的SVM定性判别模型进行定性分析,再使用fi-SVM定量分析模型进行含量预测。
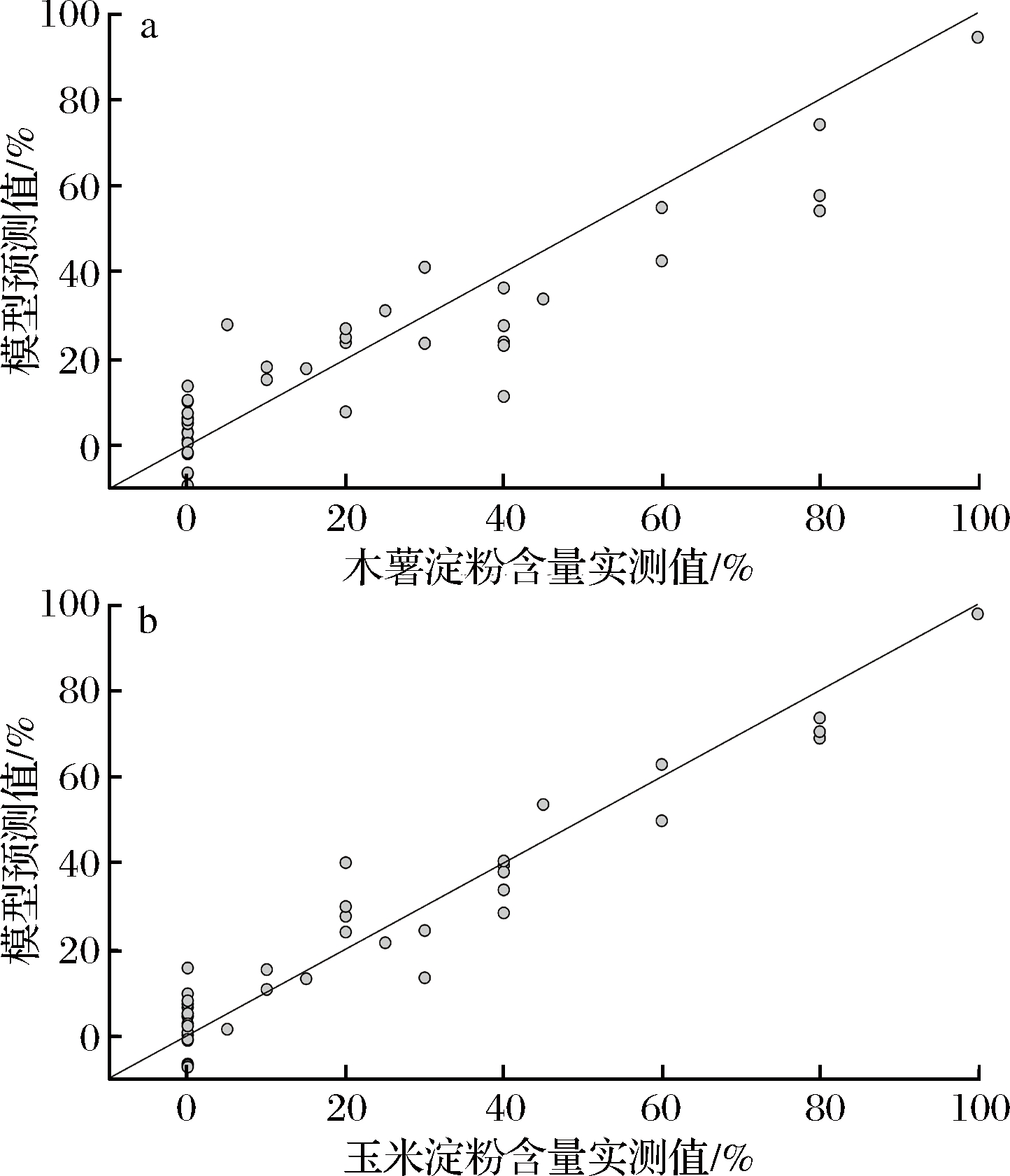
图5 验证集木薯淀粉含量(a)和玉米淀粉含量(b)
预测值与真实值比较
Fig.5 Relationship between predicted values and actual values of cassava starch content(a) and corn starch content(b) in validation set
3 结论与讨论
实验建立了基于近红外光谱及支持向量机的甘薯粉丝掺假定性判别及定量分析快速检测模型。对于甘薯粉丝掺假SVM定性判别模型,采用SNV+1st Der光谱预处理后,模型判别准确率可达100%。对于甘薯粉丝掺假SVM定量分析模型,采用SNV+1st Der光谱预处理及fi-SVM筛选光谱变量后,木薯淀粉80-fi-SVM最优模型的r和RMSEP分别达到0.92和11.20,玉米淀粉70-fi-SVM最优模型的r和RMSEP分别达到0.96和7.49,达到了较好的预测精度。在实际使用中,应该先用定性判别模型进行定性分析,再使用定量分析模型进行预测,以免对纯甘薯淀粉粉丝产生误判。采用近红外光谱结合支持向量机构建快速检测模型,样品无需前处理,操作简便迅速,为甘薯粉丝掺假快速检测、品质控制提供了一种新的思路与方法。在相关法律法规允许范围内使用复合淀粉生产粉丝,以改善粉丝品质或降低生产成本是允许的,如WU等[34]将绿豆淀粉和大米淀粉按1∶20的比例混合,以提升粉丝的烹煮性能、质构品质及口感;PHOTINAM等[35]利用豇豆、绿豆淀粉1∶1混合生产高含量抗性淀粉粉丝。但是以复合淀粉原料生产的粉丝,应在食品标签中标注,并在商品销售环节应明确告知消费者,否则就是以廉价原料掺假隐瞒消费者的行为。
[1] 康维民, 肖念新. 甘薯淀粉掺假的快速检测研究[J]. 食品科技, 2003,29(1):78-79.
[2] 侯汉学, 董海洲,刘传富. 甘薯淀粉中掺有玉米淀粉的检测方法[J]. 食品与发酵工业, 2010,36(1):134-137.
[3] 杜连起. 甘薯粉条掺杂异种淀粉检验方法的研究[J]. 河北职业技术师范学院学报, 2000,26(2):24-26.
[4] 陈嘉, 刘嘉,马雅钦,等. 葛粉掺假的近红外漫反射光谱快速检测[J]. 食品科学, 2014,35(8):133-136.
[5] SANS S, FERRE J, BOQUE R, et al. Determination of chemical properties in 'calcot' (Allium cepa L.) by near infrared spectroscopy and multivariate calibration[J]. Food Chemistry, 2018,262:178-183.
[6] 贾柳君, 张海红,王健,等. 采用近红外光谱定量分析葡萄酒发酵液中总酸含量和pH值[J]. 食品与发酵工业, 2017,43(2):191-195.
[7] 何佳艳, 李亭,郭长凯,等. 近红外光谱法快速无损测定奶粉的脂肪含量[J]. 食品与发酵工业, 2017,43(10):228-233.
[8] TAHIR H E, ZOU X, SHEN T, et al. Near-Infrared (NIR) spectroscopy for rapid measurement of antioxidant properties and discrimination of sudanese honeys from different botanical origin[J]. Food Analytical Methods, 2016,9(9):2 631-2 641.
[9] RIOS-REINA R, LUIS GARCIA-GONZALEZ D, MARIA CALLEJON R, et al. NIR spectroscopy and chemometrics for the typification of Spanish wine vinegars with a protected designation of origin[J]. Food Control, 2018,89:108-116.
[10] BALLABIO D, ROBOTTI E, GRISONI F, et al. Chemical profiling and multivariate data fusion methods for the identification of the botanical origin of honey[J]. Food Chemistry, 2018,266:79-89.
[11] LIU N, PARRA H A, PUSTJENS A, et al. Evaluation of portable near-infrared spectroscopy for organic milk authentication[J]. Talanta, 2018,184:128-135.
[12] 潘冰燕, 鲁晓翔,张鹏,等. 近红外光谱对甜椒果实质地的无损检测[J]. 食品与发酵工业, 2015,41(11):143-147.
[13] GHOSH S, MISHRA P, MOHAMAD S N H, et al. Discrimination of peanuts from bulk cereals and nuts by near infrared reflectance spectroscopy[J]. Biosystems Engineering, 2016,151:178-186.
[14] MEES C, SOUARD F, DELPORTE C, et al. Identification of coffee leaves using FT-NIR spectroscopy and SIMCA[J]. Talanta, 2018,177:4-11.
[15] LIU J, WEN Y, DONG N, et al. Authentication of lotus root powder adulterated with potato starch and/or sweet potato starch using Fourier transform mid-infrared spectroscopy[J]. Food Chemistry, 2013,141(3):3 103-3 109.
[16] XU L, SHI P, YE Z, et al. Rapid analysis of adulterations in Chinese lotus root powder (LRP) by near-infrared (NIR) spectroscopy coupled with chemometric class modeling techniques[J]. Food Chemistry, 2013,141(3):2 434-2 439.
[17] MAHOOD F, JABEEN F, HUSSAIN J, et al. FT-NIRS coupled with chemometric methods as a rapid alternative tool for the detection & quantification of cow milk adulteration in camel milk samples[J]. Vibrational Spectroscopy, 2017,92:245-250.
[18] LU G, HUANG H, ZHANG D. Application of near-infrared spectroscopy to predict sweetpotato starch thermal properties and noodle quality[J]. Journal of Zhejiang University Science B, 2006,7(6):475-481.
[19] DING X, NI Y, KOKOT S. NIR spectroscopy and chemometrics for the discrimination of pure, powdered, purple sweet potatoes and their samples adulterated with the white sweet potato flour[J]. Chemometrics and Intelligent Laboratory Systems, 2015,144:17-23.
[20] CHUANG C C, SU S F, JENG J T, et al. Robust support vector regression networks for function approximation with outliers[J]. IEEE Transactions on Neural Networks, 2002,13(6):1 322-1 330.
[21] 张丽华, 郝莉花,李顺峰,等. 基于支持向量机的近红外光谱技术快速鉴别掺假羊肉[J]. 食品工业科技, 2015,36(23):289-293.
[22] PIERNA J, VOLERY P, BESSON R, et al. Classification of modified starches by Fourier transform infrared spectroscopy using support vector machines[J]. Journal of Agricultural and Food Chemistry, 2005,53(17):6 581-6 585.
[23] CHEN J, ZHU S, ZHAO G. Rapid determination of total protein and wet gluten in commercial wheat flour using siSVR-NIR[J]. Food Chemistry, 2017,221:1 939-1 946.
[24] 褚小立, 袁洪福,陆婉珍. 近红外分析中光谱预处理及波长选择方法进展与应用[J]. 化学进展, 2004,16(4):528-542.
[25] CHANG C, LIN C. LIBSVM: A library for support vector machines[J]. ACM Transactions on Intelligent Systems and Technology, 2011,2(3):1-27.
[26] FILGUEIRAS P R, ALVES J C L, POPPI R J. Quantification of animal fat biodiesel in soybean biodiesel and B20 diesel blends using near infrared spectroscopy and synergy interval support vector regression[J]. Talanta, 2014,119:582-589.
[27] 邹婷婷, 窦英,王莹,等. 近红外光谱法结合C-SVM及v-SVM方法快速无损鉴别淀粉种类[J]. 食品工业科技, 2013,34(17):317-319.
[28] CHEN J, YE F, ZHAO G. Rapid determination of farinograph parameters of wheat flour using data fusion and a forward interval variable selection algorithm[J]. Analytical Methods, 2017,9(45):6 341-6 348.
[29] RANZAN C, TRIERWEILER L F, HITZMANN B, et al. NIR pre-selection data using modified changeable size moving window partial least squares and pure spectral chemometrical modeling with ant colony optimization for wheat flour characterization[J]. Chemometrics and Intelligent Laboratory Systems, 2015,142:78-86.
[30] CHUANG C C, JENG J T, TAO C W. Two-Stages support vector regression for fuzzy neural networks with outliers[J]. International Journal of Fuzzy Systems, 2009,11(1):20-28.
[31] SHAO Y, CEN Y, HE Y, et al. Infrared spectroscopy and chemometrics for the starch and protein prediction in irradiated rice[J]. Food Chemistry, 2011,126(4):1 856-1 861.
[32] LIN C, CHEN X, JIAN L, et al. Determination of grain protein content by near-infrared spectrometry and multivariate calibration in barley[J]. Food Chemistry, 2014,162:10-15.
[33] AL-MBAIDEEN A, BENAISSA M. Frequency self deconvolution in the quantitative analysis of near infrared spectra[J]. Analytica Chimica Acta, 2011,705(1-2):135-147.
[34] WU F, MENG Y, YANG N, et al. Effects of mung bean starch on quality of rice noodles made by direct dry flour extrusion[J]. LWT-Food Science and Technology, 2015,63(2):1 199-1 205.
[35] PHOTINAM R, MOONGNGARM A, PASEEPHOL T. Process optimization to increase resistant starch in vermicelli prepared from mung bean and cowpea starch[J]. Emirates Journal of Food and Agriculture, 2016,28(7):449-458.