琥珀酸是一种二元羧酸,作为众多化工产品的合成前体,广泛应用于医药、农药、染料、香料、油漆、食品、塑料等行业[1]。研究发现,琥珀酸能够促进脂肪燃烧,这将为解决肥胖症提供新的研究思路[2]。目前琥珀酸的工业化生产主要依赖于化学合成法[3-4],而化学合成法生产的琥珀酸难以达到食用要求。为解决这一难题,越来越多的研究者致力于微生物发酵法生产琥珀酸[5-8]。酿酒酵母因其安全、无毒素分泌已经被广泛应用于食品发酵工业。同时酿酒酵母具备耐高渗[9]、耐低pH[10]的优点,因此更多的研究者致力于通过代谢工程手段促进酿酒酵母积累琥珀酸。但目前存在以下问题尚须解决[11]:(1)高效的琥珀酸合成途径的构建;(2)进入三羧环循环(tricarboxylic acid, TCA)的碳流量的加强。
菌体内的琥珀酸作为TCA循环的中间产物,在琥珀酸脱氢酶作用下转化为富马酸,而琥珀酸脱氢酶由基因SDH1、SDH2、SDH3和SDH4编码[12]。因此,弱化琥珀酸脱氢酶成为构建琥珀酸积累途径的首要条件。如OTERO等[13]敲除琥珀酸脱氢酶基因SDH3、3-磷酸甘油酸脱氢酶同工酶基因SER3和SER33,成功利用乙醛酸支路积累琥珀酸;RAAB等[14]则敲除基因SDH1、SDH2和异柠檬酸脱氢酶基因IDH1、IDP1,同时利用TCA还原途径和乙醛酸支路实现琥珀酸积累。丙酮酸羧化酶(pyruvate acid,PC)作为菌体内草酰乙酸回补途径关键酶,成为增强TCA循环碳流量的关键。如YAN等[15]过量表达酿酒酵母本身的丙酮酸羧化酶基因PYC2,促进了琥珀酸的积累。本课题组前期在探究代谢工程手段改造酿酒酵母积累富马酸时发现,来源于米根霉的丙酮酸羧化酶基因(RoPYC)较酿酒酵母本身的PYC2更能促进富马酸的积累[16]。
本实验以酿酒酵母为研究对象,敲除对琥珀酸脱氢酶酶活影响最大的基因SDH2,使琥珀酸通过TCA氧化途径得到积累。在此基础上考察了来源于米根霉的丙酮酸羧化酶基因RoPYC的表达对琥珀酸积累的影响,进而考察了微量元素Ca2+和生物素对PC酶表达及琥珀酸积累的作用。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒、及引物
S.cerevisiae CEN.PK 2-1C,由本实验室于-80 ℃保藏;敲除型质粒pUG72和Cre蛋白分泌质粒pSH63,由本实验室保藏;基因敲除时所用引物,由金唯智生物技术有限公司合成(表1)。
表1 本研究所用引物
Table 1 Primers used in this study

引物序列(5’-3’)SDH2-FATGTTGAACGTGCTATTGAGAAGGAAGGC-CTTTTGTTTGGTGACGCAGCTGAAGCTTCGTACGCSDH2-RTGAGAGGCTTCTGAGTTCT-TCGGGGCTAGCTGTTTTTCTGATAGTGCATAGGCCACTAGTGGATCTGA(SDH2)CGTATTATTGTAGCTCCGCACGB(SDH2)CGATGATCTCTGTAAGTAAGTAAGGTTGGC(SDH2)CCATTACCCCACATGTTTATTGTCD(SDH2)CTATGATGGACATATATATACAGACCGGBM(SDH2)CATAGCCTGCTTGAATGCAATACCM(SDH2)TACTATATTCCTTTTCGGTAGC
1.1.2 酶和试剂
质粒提取试剂盒、胶回收试剂盒、限制性内切酶、T4 DNA连接酶,均来自上海皓嘉生物技术有限公司;生物素,上海生物工程股份有限公司;琥珀酸脱氢酶酶活测定试剂盒、丙酮酸羧化酶酶活测定试剂盒,苏州科铭生物技术有限公司。
1.1.3 培养基
LB培养基(g/L):酵母粉5,NaCl 10,蛋白胨 10。YPD培养基(g/L):Peptone 20,酵母浸膏10,葡萄糖20,固体培养基加琼脂粉20。YPG培养基(g/L):Peptone 20,酵母浸膏10,半乳糖20,固体培养基加琼脂粉20。SD-Leu种子培养基:葡萄糖20 g/L,YNB 1.7 g/L,(NH4)2SO4 5 g/L,10×AA 100 mL,100×His、100×Ura、100×Arg、100×Trp各10 mL。SD种子培养基:在SD-Leu培养基的基础上添加100×Leu 10 mL/L。SD-Leu发酵培养基(g/L):葡萄糖 40,KH2PO4 3,K2SO4 6.6,MgSO4·7H2O 0.5,尿素 1,YNB 3.4,CaCO3 5, 10×AA 100 mL,100×Ura、 10×His、10×Arg、10×Trp各10 mL。SD发酵培养基(g/L):在SD-Leu培养基的基础上添加100×Leu 10 mL/L。
1.2 仪器与设备
凝胶成像仪、PCR仪,Bio-Rad公司;高效液相色谱,有机酸色谱柱,Waters公司。
1.3 方法
1.3.1 SDH2基因敲除
参照徐国强[17]的方法利用筛选标记,通过同源重组进行目的基因的敲除。
1.3.2 丙酮酸羧化酶异源表达
将本实验室保存的携带有米根霉丙酮酸羧化酶的质粒pY15TEF1-RoPYC利用醋酸锂转化法[18]转化至酿酒酵母琥珀酸生产菌株2-1CΔSDH2中。
1.3.3 培养方法
种子培养:从平板上挑取单菌落接种至装有4 mL SD或SD-Leu培养基的试管中,30 ℃、220 r/min过夜培养。
发酵培养:将过夜培养的种子液接入40 mL/250 mL锥形瓶SD或SD-Leu培养基中,30 ℃、220 r/min振荡培养。分别培养至12、24、36、48、60、72 h,取样进行发酵产物测定。
1.3.4 酶活测定
粗酶提取:菌株进行发酵培养分别至24、36、48 h时取样30 mL左右,在12 000 r/min条件下离心1 min,得到适量的菌体。向菌体中加入400 μL提取液,同时加入和菌体等体积的玻璃珠,在旋涡仪上进行振荡,每振荡30 s,立刻置于冰上1 min,重复操作10次;12 000 r/min离心10 min,取上清即为待测蛋白液。
酶活反应体系:50 μL样本,50 μL试剂四,900 μL工作液,在340 nm的波长下测定吸光度A1和2 min后的吸光度A2,两者差值即为ΔA。
琥珀酸脱氢酶酶活单位定义为:1 g组织1 min消耗1 nmol 2,6-二氯酚靛酚定义为1个酶活力单位。
琥珀酸脱氢酶酶活=
(1)
PC酶活=
(2)
1.3.5 发酵产物的分析方法
生物量测定:采用紫外可见分光光度计测定600 nm波长处的吸光值。
葡萄糖测定:采用生物传感器SBA-40D测定葡萄糖浓度。
琥珀酸及丙酮酸测定:采用高效液相色谱,色谱柱型号:Waters Atlantis T3,4.6 mm×250 mm,5 μm;检测条件:30 ℃柱温;20 mmol/L NaH2PO4流动相;0.7 mL/min流速;紫外210 nm波长下检测琥珀酸和丙酮酸产量。
2 结果与分析
2.1 SDH2缺失株的构建及SDH2基因缺失对琥珀酸积累的影响
酿酒酵母中琥珀酸进一步代谢所需的琥珀酸脱氢酶复合体由4个亚基组成,分别由基因SDH1、 SDH2、 SDH3、SDH4编码[19],其中基因SDH2的缺失使琥珀酸脱氢酶酶活下降57.8%,SDH2基因成为琥珀酸脱氢酶酶活的关键编码基因。为构建琥珀酸合成途径本研究敲除基因SDH2,分别以SDH2-A和SDH2-D引物进行PCR验证,获得SDH2缺失株如图1-a所示,若敲除成功,条带大小应为2 075 bp,否则为1 244 bp。对成功敲除菌株进行保藏和后续研究。图1-b为构建成功的SDH2基因缺失株和野生型菌株2-1C琥珀酸脱氢酶酶活测定结果。由图1-b可知,敲除SDH2基因后,琥珀酸脱氢酶的酶活在24、36、48 h较对照组分别下降30.32%、33.31%、57.13%,且48 h酶活最低。
图2对SDH2缺失株和对照菌株进行了发酵性能的对比。敲除SDH2基因后,菌株2-1C SDH2发酵至48 h生物量达到最大值,较对照菌株最大生物量下降8.4%。由此可知,敲除基因SDH2未造成菌株的停滞生长,故基因SDH2并非菌株生长的必需基因。而在敲除基因SDH2后,菌株2-1C SDH2较对照菌株实现了琥珀酸积累,由(0.011±0.002) g/L提高至(0.709±0.015)g/L。同时,敲除基因SDH2后,丙酮酸的产量有所提高,如图2-c,且发酵至36 h达到最大值(0.939±0.01) g/L,相较于原始菌株提高了47.64%。同时,由图2(b)可知,发酵至24 h后菌株2-1CΔSDH2及对照的葡萄糖剩余量均接近0。同时发酵84 h,菌株2-1CΔSDH2葡萄糖消耗能力较对照有所下降。说明敲除基因SDH2使菌株葡萄糖消耗能力减弱。
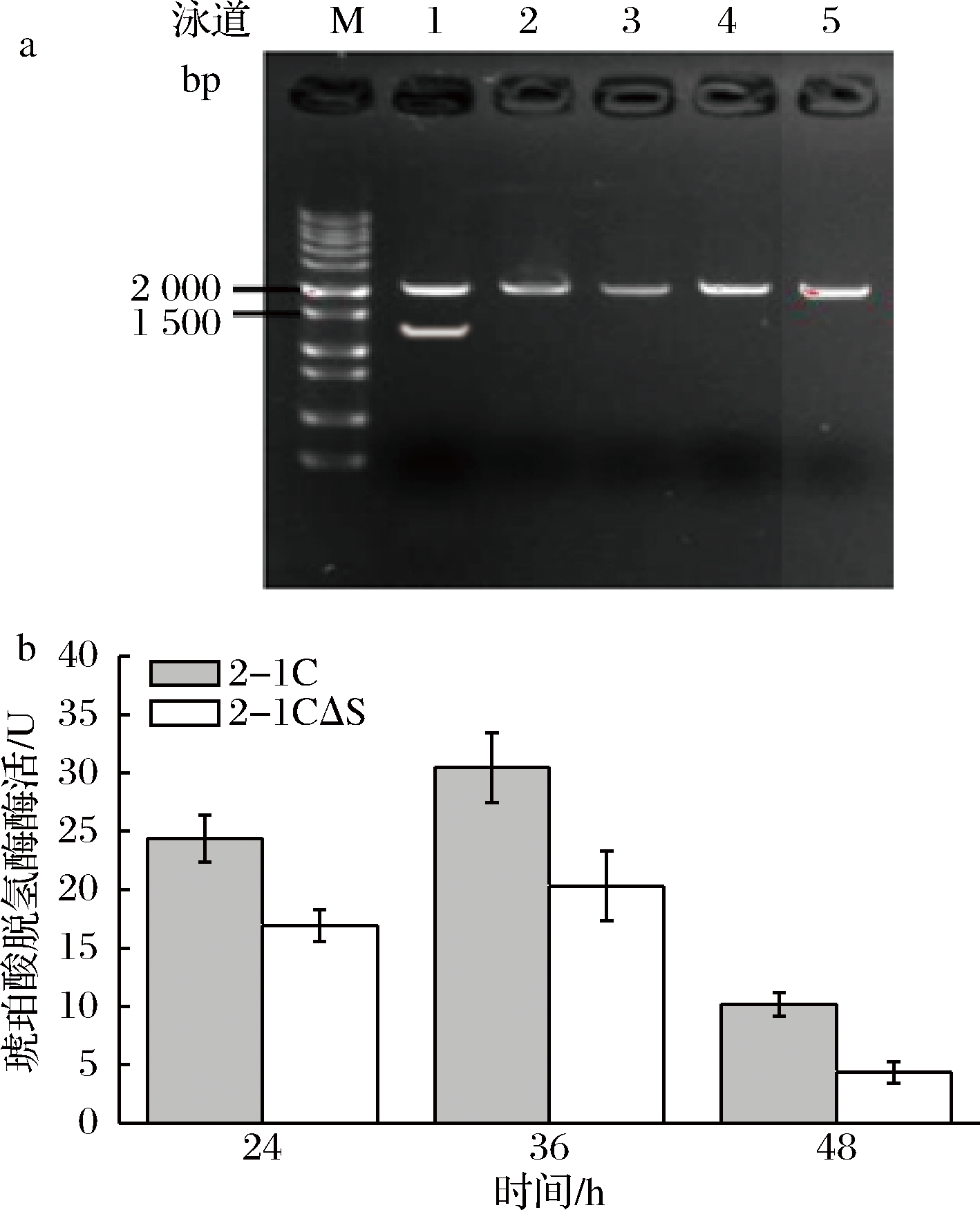
M-marker;1-空白对照;2~5-sdh2缺失菌株
图1 sdh2缺失株PCR验证结果(a)和菌株2-1C、2-1CΔsdh2琥珀酸脱氢酶酶活测定结果(b)
Fig.1 The PCR result of strain without gene sdh2(a) and the activity of succinate dehydrogenase in strain 2-1C and 2-1CΔsdh2(b)
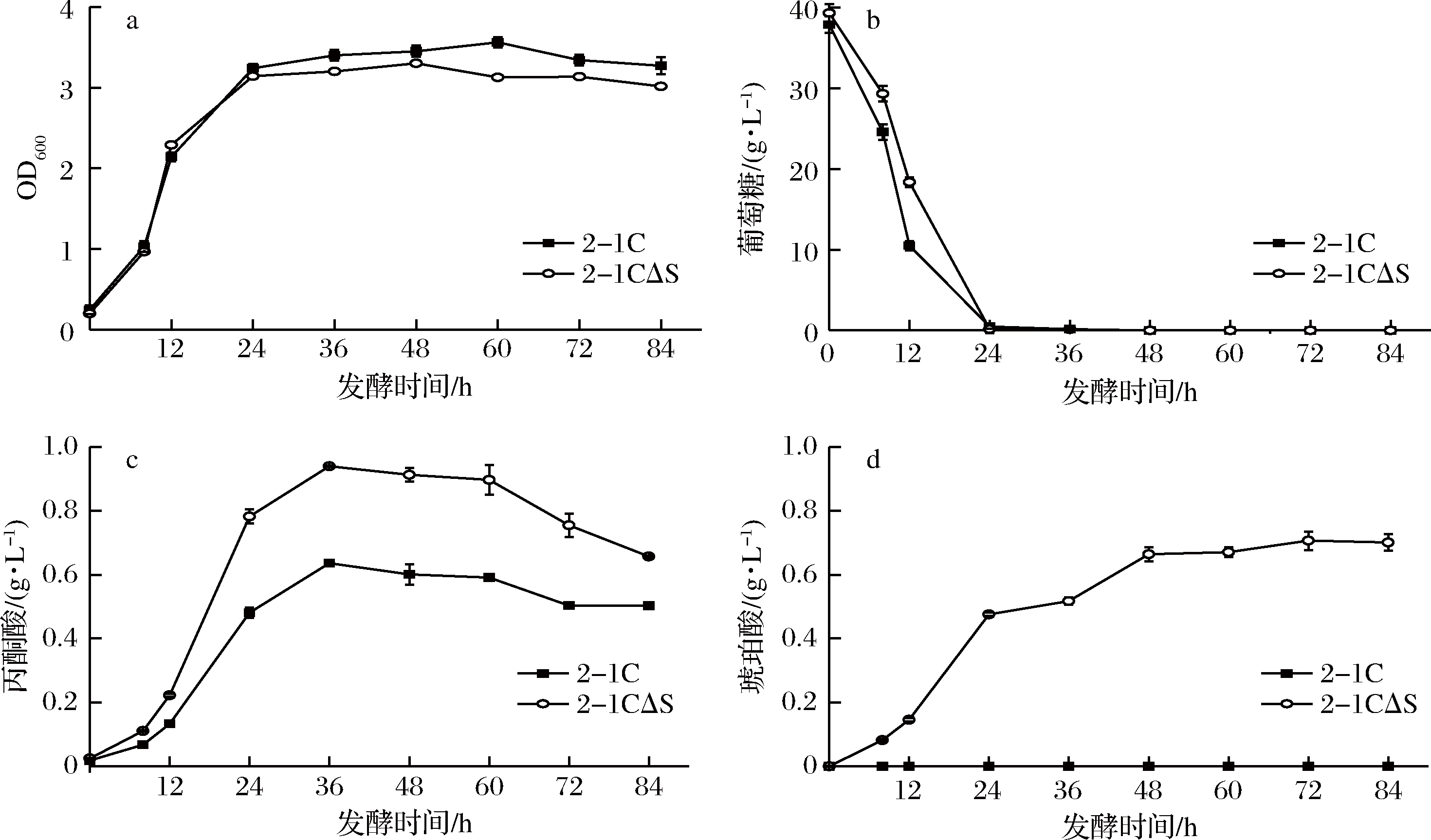
a-生物量;b-葡萄糖;c-丙酮酸;d-琥珀酸
图2 2-1C和2-CΔSDH2发酵曲线
Fig.2 Fermentation curve of 2-1C and 2-CΔSDH2
2.2 异源表达丙酮酸羧化酶对琥珀酸积累的影响
在敲除基因SDH2后,除琥珀酸得到积累外,丙酮酸含量也提高46.7%,而丙酮酸是连接糖酵解和三羧酸循环的关键点[5,20-21]。因此将丙酮酸处积累的碳流量“引入”琥珀酸合成途径成为急需解决的问题。而将丙酮酸转化为有机酸的关键酶是PC酶,KENEALY等研究发现,在R. oryzae发酵生产延胡索酸的过程中,PC酶具有较高的活性[22]。为此本研究在酿酒酵母中异源表达了来自米根霉的PC酶基因。
图3-a为异源表达丙酮酸羧化酶PYC基因获得的转化子菌落PCR产物图,图3-b比较了重组菌2-1CΔS-RoPYC和 2-1CΔSDH2丙酮酸羧化酶酶活,结果表明,发酵过程中丙酮酸羧化酶酶活呈现先增后减的趋势,且发酵至24 h,菌株2-1CΔS-RoPYC PC酶活提高最为明显,比菌株2-1C ΔSDH2高22.5%。
在异源表达PC酶基因后,进行发酵性能测定,探究其对琥珀酸积累的作用。如图4所示,异源表达丙酮酸羧化酶发酵至36 h,菌株2-1CΔS-RoPYC丙酮酸含量较2-1CΔS下降44.3%,同时,菌株2-1C、2-1CΔSDH2、2-1CΔSDH2-RoPYC发酵24 h后葡萄糖消耗殆尽,且葡萄糖消耗能力依次减弱。发酵至72 h琥珀酸产量达到最大值(0.841±0.020) g/L,较菌株2-1C ΔSDH2提高了28.6%。说明提高PC酶的酶活促进了琥珀酸的积累。
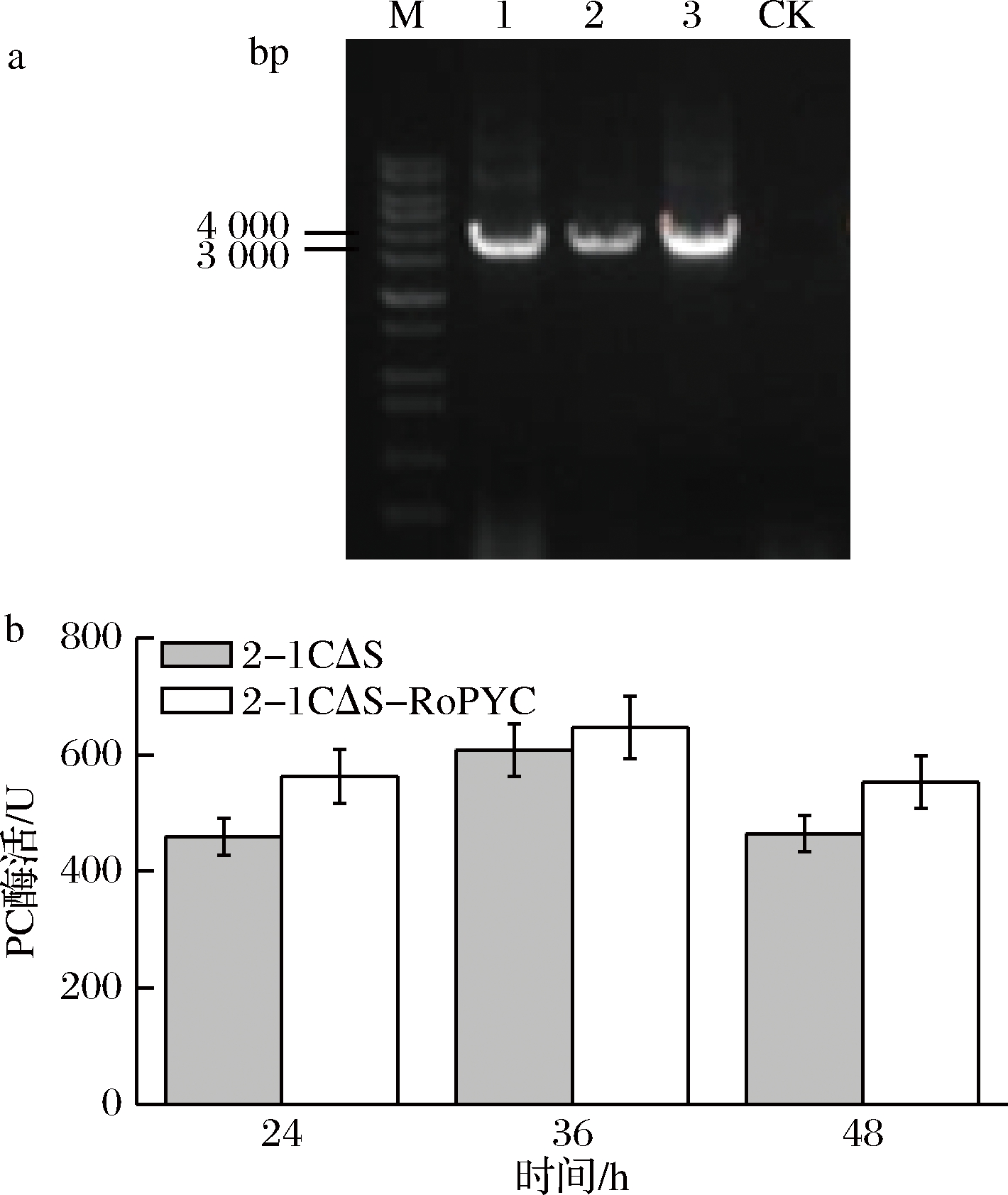
M-marker;CK-空白对照;1~3-转化成功的菌株
图3 异源表达RoPYC基因转化子菌落PCR验证凝胶电泳图(a)和菌株2-1CΔS-RoPYC与2-1CΔS的丙酮酸羧化酶酶活(b)
Fig.3 The PCR result of construct strain 2-1CΔS-RoPYC(a) and the activity of pyruvate carboxylase in strain 2-1CΔS-RoPYC and 2-1CΔSDH2(b)

a-生物量;b-葡萄糖;c-丙酮酸;d-琥珀酸
图4 菌株2-1C、2-1CΔSDH2、2-1CΔSDH2-RoPYC发酵曲线
Fig.4 Fermentation curve of 2-1C,2-1CΔSDH2,2-1CΔSDH2-RoPYC
2.3 Ca2+及生物素对丙酮酸羧化酶的作用探究
在琥珀酸初步积累的基础上通过辅因子水平探究来进一步提高丙酮酸羧化酶酶活。Ca2+作为乙酰CoA羧化酶的激活剂[23],在Ca2+存在时其酶活提高1倍。本实验室前期研究发现在过量表达PC酶的酿酒酵母发酵中,单独添加适量的Ca2+可以提高富马酸的含量,因此本实验首先考察了Ca2+的添加对PC酶活性及琥珀酸积累的作用。
在发酵36 h选择外源添加浓度为0、25、50、75、100、125 mmol/L CaCl2溶液,探究其对丙酮酸羧化酶的影响。从实验结果(图5、图6)可知,不同浓度的CaCl2溶液对PC酶的酶活作用不同。添加50 mmol/L CaCl2溶液和未添加CaCl2溶液PC酶活一致,而添加25、75、100、125 mmol/L CaCl2溶液后,PC酶活分别下降7%、30%、39%、13%。之后选择添加0、25、125 mmol/L CaCl2溶液探究其对琥珀酸积累的影响。结果发现,加入25、125 mmol/L的CaCl2溶液后,菌体浓度上升,相较于对照增加了29.7%和107.6%,但琥珀酸产量随Ca2+浓度增加逐渐下降,说明加入较多的Ca2+有利于菌体的生长,但却不利于丙酮酸和琥珀酸生产。图6(b)为添加不同浓度Ca2+后菌株2-1CΔSDH2-RoPYC葡萄糖消耗曲线。由图可知,添加不同浓度Ca2+后菌株2-1CΔSDH2-RoPYC葡萄糖剩余量无明显变化。说明Ca2+外源添加对菌株2-1CΔSDH2-RoPYC葡萄糖消耗能力无明显作用。
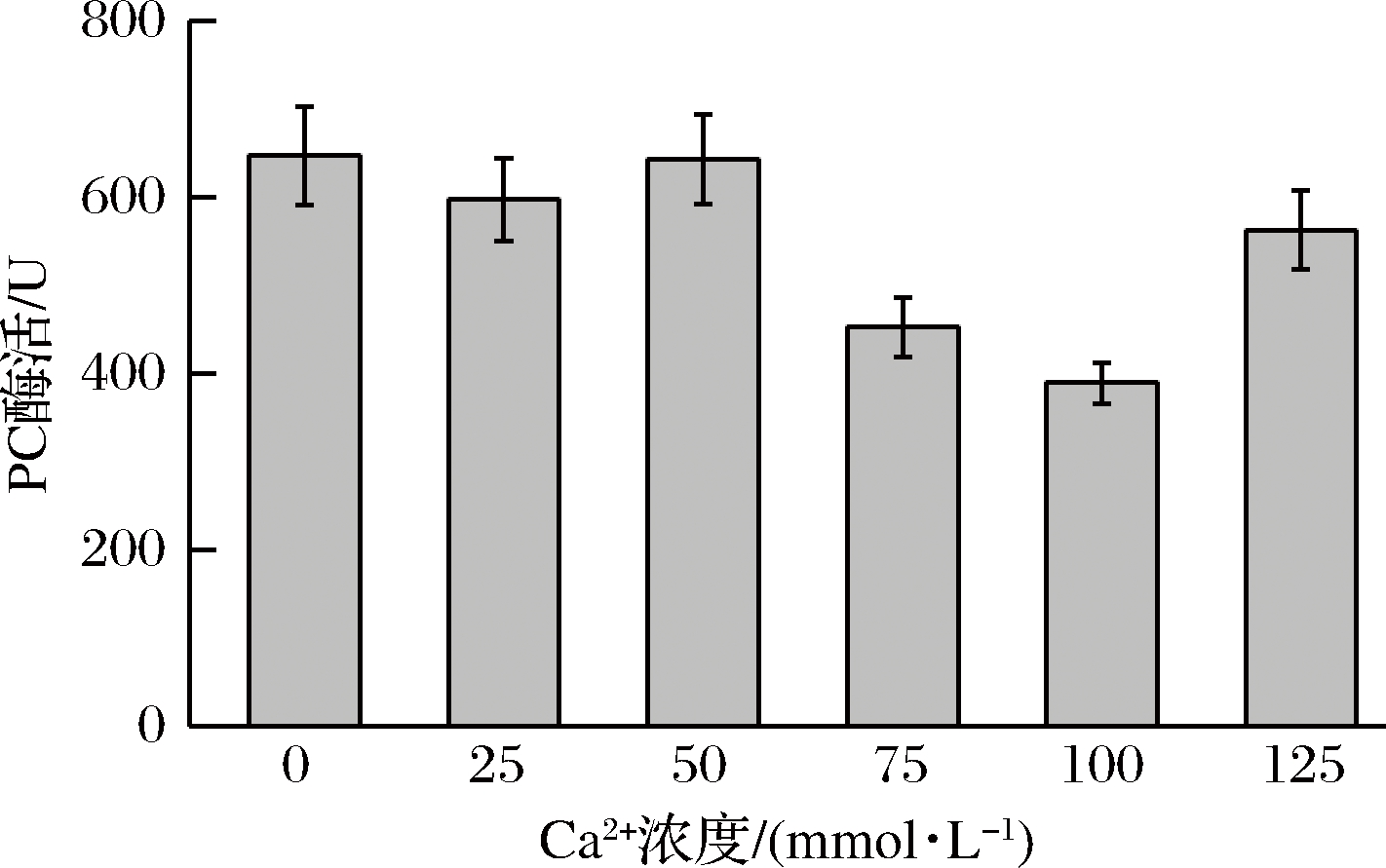
图5 36 h添加不同浓度Ca2+时丙酮酸羧化酶酶活
Fig.5 Activity of PC enzyme with different concentrations of Ca2+ at 36 h
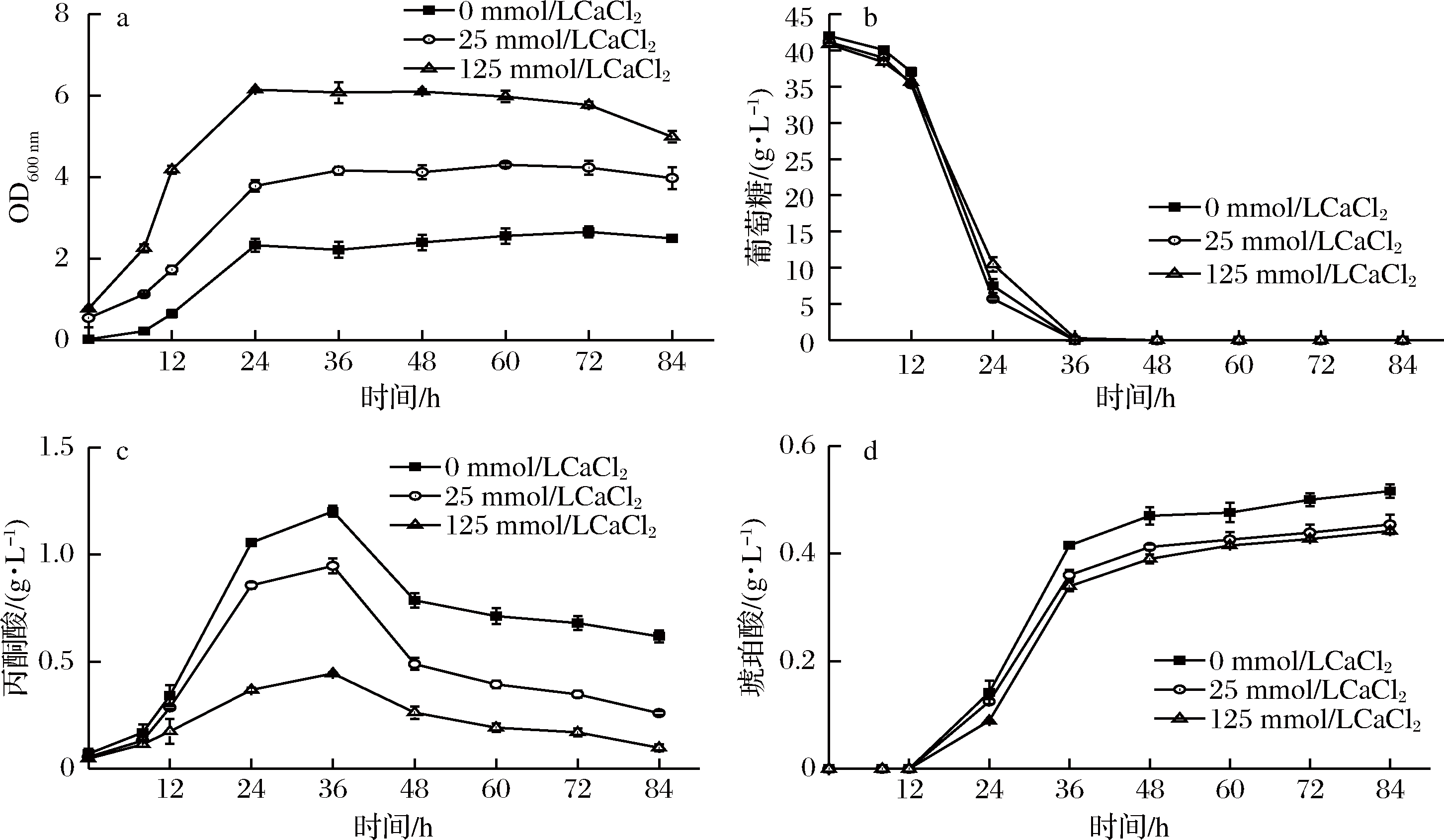
a-生物量;b-葡萄糖;c-丙酮酸;d-琥珀酸
图6 添加不同浓度Ca2+后菌株2-1CΔSDH2-RoPYC发酵曲线
Fig.6 Fermentation curve of 2-1CΔSDH2-RoPYC with diverse concentration of Ca2+
PC酶是生物素依赖型酶,在催化过程中,羧基生物素易位至羧基转移酶结构域后转移至受体底物丙酮酸,故缺乏生物素不利于酶促反应[24]。本实验考察了不同浓度生物素对丙酮酸羧化酶活性及琥珀酸积累的作用。添加质量浓度为16、32、64、96 μg/L生物素,PC酶酶活较未添加生物素分别提高5.87%、22.24%、15.12%、14.05%。且添加32 μg/L生物素时,PC酶酶活提高最明显。发酵实验表明,添加生物素琥珀酸产量提高,且生物素质量浓度为32 μg/L时,琥珀酸产量最高,为(0.964±0.02) g/L,相较于未添加生物素提高了87.5%,但添加适当浓度生物素,丙酮酸产量下降(图7、图8)。由图8(b)可知,外源添加生物素有利于菌株2-1CΔSDH2-RoPYC葡萄糖消耗能力的提高。
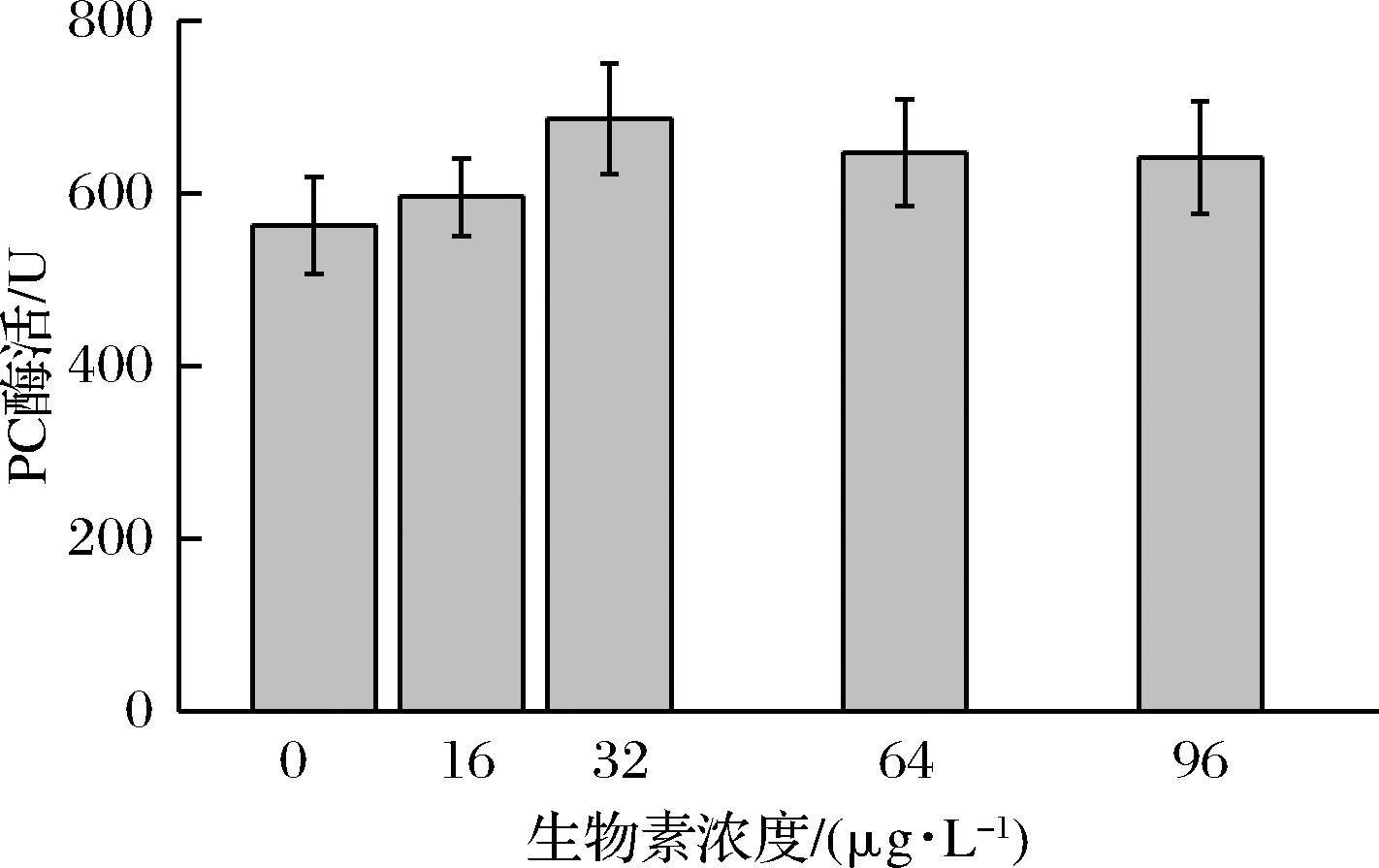
图7 36 h添加不同浓度生物素时PC酶活
Fig.7 Activity of PC enzyme with different concentrations of biotin at 36 h
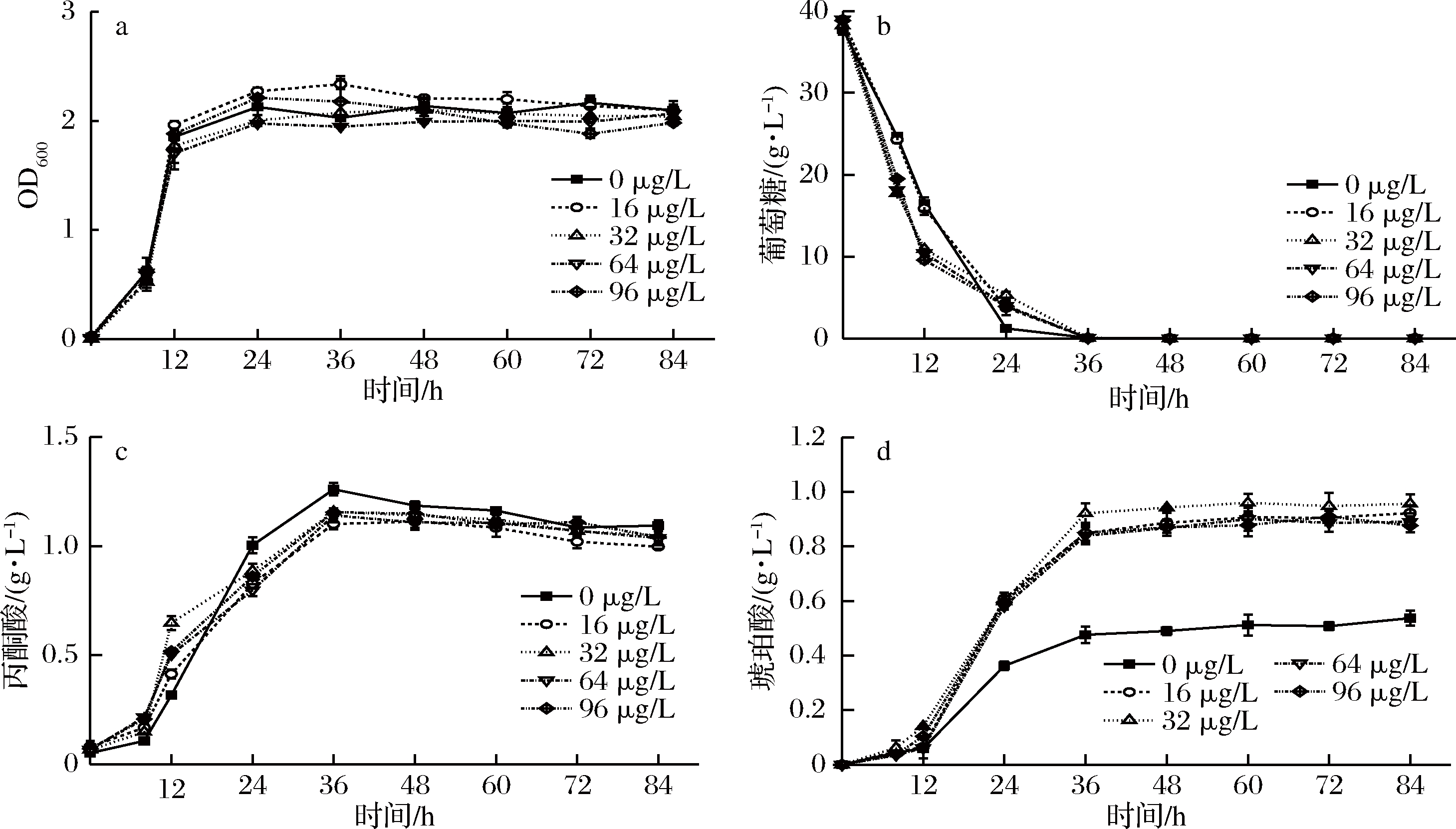
a-生物量;b-葡萄糖;c-丙酮酸;d-琥珀酸
图8 添加不同浓度生物素后菌株2-1CΔSDH2-RoPYC发酵曲线
Fig.8 Fermentation curve of 2-1CΔSDH2-RoPYC with diverse concentration of biotin
3 结论
早前已有研究人员在谷氨酸棒杆菌中共表达丙酮酸羧化酶和磷酸烯醇式丙酮酸羧化酶编码基因PYC和PPC促进了琥珀酸积累[25],且在缺乏丙酮酸羧化酶的大肠杆菌[21]中引入外源PC酶可以增强草酰乙酸的回补增加TCA循环的碳流量,从而提高琥珀酸产量。本研究敲除琥珀酸脱氢酶复合体基因SDH2,成功构建了琥珀酸合成途径,使琥珀酸产量由(0.011±0.002) g/L提高至(0.709±0.015) g/L。在此基础上过量表达米根霉丙酮酸羧化酶基因提高PC酶活,使琥珀酸产量进一步提高至(0.841±0.020) g/L。说明PC酶酶活提高有利于琥珀酸积累,进而探究其辅因子对琥珀酸积累作用,发现添加32 μg/L生物素,琥珀酸达到(0.964±0.02) g/L。综上,丙酮酸羧化酶作为连接糖酵解途径和TCA循环的节点,对琥珀酸积累起促进作用,同时也成为改造酿酒酵母积累琥珀酸的关键点,为代谢工程策略改造酿酒酵母积累琥珀酸提供新思路。在今后的研究中则要进一步考察PC酶不同表达量对酿酒酵母积累琥珀酸的作用。
[1] ZEIKUS J G, JAIN M K, ELANKOVAN P. Biotechnology of succinic acid production and markets for derived industrial products[J]. Applied Microbiology and Biotechnology, 1999, 51: 545-552.
[2] MILLS E L, PIERCE K A, JEDRYCHOWSKI M P,et al. Accumulation of succinate controls activation of adipose tissue thermogenesis[J]. Nature, 2018.
[3] LIU R, LIANG L, CAO W, et al. Succinate production by metabolically engineered Escherichia coli using sugarcane bagasse hydrolysate as the carbon source[J]. Bioresource Technology, 2013, 135(C): 574-577.
[4] LIANG L, LIU R, LI F, et al. Repetitive succinic acid production from lignocellulose hydrolysates by enhancement of ATP supply in metabolically engineered Escherichia coli[J]. Bioresource Technology, 2013, 143(C): 405-412.
[5] MAO Y, LI G, CHANG Z, et al. Metabolic engineering of Corynebacterium glutamicum for efficient production of succinate from lignocellulosic hydrolysate[J]. Biotechnology for Biofuels, 2018, 11(1):95.
[6] WANG J, WANG H, YANG L,et al. A novel riboregulator switch system of gene expression for enhanced microbial production of succinic acid[J]. Journal of Industrial Microbiology and Biotechnology, 2018, 45: 253-269.
[7] CUI Z, GAO C, LI J, et al. Engineering of unconventional yeast Yarrowia lipolytica for efficient succinic acid production from glycerol at low pH[J]. Metabolic Engineering, 2017, 42: 126-133.
[8] AHN J H, JANG Y S, LEE S Y. Production of succinic acid by metabolically engineered microorganisms[J]. Current Opinion in Biotechnology, 2016, 42: 54-66.
[9] 郑美娟,余华顺.酵母菌耐高渗机理研究[J].酿酒科技,2008(10):37-39.
[10] 徐国强,吴满珍.微生物发酵生产C4二元羧酸研究进展[J].中国生物工程杂志,2014,34(8):97-104
[11] ABBOTT D A, ZELLE R M, PRONK J T,et al. Metabolic engineering of Saccharomyces cerevisiae for production of carboxylic acids: Current status and challenges[J]. FEMS Yeast Research, 2009, 9(8): 1 123-1 136.
[12] KUBO Y, TAKAGI H, NAKAMORI S. Effect of gene disruption of succinate dehydrogenase on succinate production in a sake yeast strain[J]. Journal of Bioscience and Bioengineering, 2000, 90(6): 619-624.
[13] OTERO J M, CIMINI D, PATIL K R, et al. Industrial systems biology of Saccharomyces cerevisiae enables novel succinic acid cell factory[J]. PLoS One, 2013, 8(1):e54 144.
[14] RAAB A M, GEBHARDT G, BOLOTINA N, et al. Metabolic engineering of Saccharomyces cerevisiae for the biotechnological production of succinic acid[J]. Metabolic Engineering, 2010, 12(6): 518-525.
[15] YAN D, WANG C, ZHOU J, et al. Construction of reductive pathway in Saccharomyces cerevisiae for effective succinic acid fermentation at low pH value[J]. Bioresource Technology, 2014, 156: 232-239.
[16] XU G, WU M, JIANG L.Site-saturation engineering of proline 474 in pyruvate carboxylase from Rhizopus oryzae to elevate fumaric acid production in engineered Saccharomyces cerevisiae cells[J]. Biochemical Engineering Journal, 2017, 117(part_PB): 36-42.
[17] 徐国强.代谢工程改造酿酒酵母生产延胡索酸[D].无锡:江南大学,2012.
[18] XU G, ZOU W, CHEN X, et al. Fumaric acid production in Saccharomyces cerevisiae by in silico aided metabolic engineering[J]. PLoS One, 2012, 7(12): 1-10.
[19] KURO Y, TAKAG H, NAKAMORI S. Effect of gene disruption of succinate dehydrogenase on succinate production in a Sake Yeast Strain[J]. Journal of Bioscience and Bioengineering, 2000, 90(6): 619-624.
[20] KOFFAS M A G, JUNG G Y, AON J C, et al. Effect of pyruvate carboxylase overexpression on the physiology of Corynebacterium glutamicum [J]. Applied and Environmental Microbiology, 2002, 68(11): 5 422-5 428.
[21] LIN H, SAN K Y, BENNETT G N.Effect of Sorghum vulgare phosphoenolpyruvate carboxylase and Lactococcus lactis pyruvate carboxylase coexpression on succinate production in mutant strains of Escherichia coli[J]. Applied Microbiology and Biotechnology, 2005, 67(4): 515-523.
[22] KENEALY W, ZAADY E, DU PREEZ J C, et al. Biochemical aspects of fumaric acid accumulation by Rhizopus arrhizus[J]. Applied and Environmental Microbiology, 1986, 52(1): 128-33.
[23] MCNEILLIE E M, CLEGG R A, ZAMMIT V A. Regulation of acetyl-CoA carboxylase in rat mammary gland. effects of incubation with Ca2+, Mg2+ and ATP on enzyme activity in tissue extracts[J]. The Biochemical Journal, 1981, 200(3): 639-644.
[24] LIETZAN A D, LIN Y, MAURICE M S T. The role of biotin and oxamate in the carboxyltransferase reaction of pyruvate carboxylase[J]. Archives of Biochemistry and Biophysics, 2014, 562: 70-79.
[25] LITSANOV B, KABUS A, BROCKER M, et al. Efficient aerobic succinate production from glucose in minimal medium with Corynebacterium glutamicum[J]. Microb. Biotechnol., 2012, 5(1): 116-128.