大曲是中国白酒发酵所特有的糖化剂和发酵剂,它是在开放式环境中,通过自发的传统固态发酵生产而来,并且其生产原料通常是未经灭菌处理的小麦、大麦和/或豌豆混合物[1],因此大曲中包含多种类型的微生物,如细菌、酵母和丝状真菌[1-2]。其中丝状真菌对白酒的发酵至关重要,因为丝状真菌能够分泌大量的水解酶类,可以将原料中的大分子物质水解成小分子的肽类、单糖等,从而为后续的酒精发酵提供能量物质[3]。
从分类学上来看,丝状真菌是一类多样性非常复杂的真核微生物,并且许多丝状真菌的生长条件尚不明确,通过传统培养的方法很难全面了解大曲中丝状真菌的物种多样性,因此需要借助未培养的方法。近年来兴起的高通量测序技术可以对数百万个DNA分子进行快速、准确地测序,使得更加深入研究微生物的多样性成为可能[4],因此该技术也迅速在食品微生物领域得到广泛的应用。例如,乔晓梅等[5]利用高通量测序对清香大曲真菌群落结构研究发现,在微生物物种水平上,中温大曲包含米根霉(Rhizopus oryzae)、淀粉丝菌(Amylomyces rouxii)、微小根毛霉(Rhizomucor pusillus)、横梗霉(Lichtheimia)和伞枝犁头霉(Lichtheimia corymbifera)、伞形卷霉等多种丝状真菌[5]。夏玙等[6]同样利用高通量测序发现,中高温大曲中的优势丝状真菌属主要为嗜热子囊菌属(Thermoascus)、根毛霉属(Rhizomucor)和曲霉菌属(Aspergillus)3种。除此以外,还观察到了一些低丰度的Byssochlamys、Rhizopus、Monascus、Penicillium、Lichtheimia、Rasamsonia等丝状真菌属。最近,笔者通过高通量测序技术结合微生物溯源进行研究还发现,大曲中的部分丝状真菌,如A. flavus和A. oryzae等,还能进入到白酒的酒精发酵过程[7-8],说明丝状真菌除了产生各种水解酶类,可能还具有其他功能。
最近的研究表明,丝状真菌具有强大的次级代谢产物合成潜力,并且许多次级代谢产物具有重要的药理学活性[9],因而近年来许多研究者试图从一些丝状真菌丰富的栖息地(深海[10]、植物内生真菌[11]和海绵等[12])中研究这类微生物的物种多样性,并挖掘具有生物活性的天然产物。且大曲中含有丰富的丝状真菌资源,这些丝状真菌可能也具有合成多种天然产物的潜力。但是,目前系统性地研究大曲中丝状真菌的物种多样性及其次级代谢产物的合成潜力较少。
本研究的目的有2个:(1)利用高通量测序的方法比较中温、中高温和高温3种大曲中丝状真菌的菌群结构差异;(2)结合纯培养的方法研究大曲中丝状真菌的物种多样性及其次级代谢产物合成潜力。本研究为大曲来源的丝状真菌次级代谢产物合成潜力研究奠定理论基础,为与大曲相关的研究提供新的思路。
1 材料与方法
1.1 材料与仪器
1.1.1 大曲样本采集
本研究使用的大曲样本均为成品曲粉(表1),取样时,每种样本均使用无菌自封袋取500 g,并立即存放于-80 ℃,保存备用。
表1 本研究所使用的大曲样本
Table 1 Daqu samples used in this study

样本名称大曲类型发酵最高温度/℃样品来源ZW中温大曲40~50某清香型酒厂ZGW中高温大曲50~60某浓香型酒厂GW高温大曲60~70某酱香型酒厂
1.1.2 试剂与设备
麦芽提取物琼脂(malt extract agar,MEA)、马铃薯葡萄糖琼脂(potato dextrose agar,PDA)、玉米粉琼脂(corn meal agar,CMA)、沙氏葡萄糖琼脂(sabouraud dextrose agar,SDA)和察氏琼脂(czapek dox agar,CDA),青岛海博生物技术有限公司;基因组DNA提取试剂盒(DP305)、1×Taq PCR Mastermix、DNA纯化试剂盒(DP204)、琼脂糖凝胶DNA回收试剂盒(DP209),天根生化科技(北京)有限公司;QuantiNova SYBR Green PCR试剂盒,德国QIAGEN公司。
Illumina Miseq PE300二代测序仪,美国Illumina公司;HWY-2112型恒温培养摇床,上海智城分析仪器制造有限公司;LDZX-50KBS立式压力灭菌锅、SPX-250B型生化培养箱,上海博迅实业有限公司;SW-CJ-1D超净工作台,上海沪净医疗器械有限公司;Nanodrop 2000/2000C 分光光度计,美国Thermo Fisher Scientific公司;PowerPac通用电泳仪电源、C1000 Touch PCR仪、CFX96 Touch实时定量PCR仪、ChemiDoc成像系统,美国Bio-Rad公司。
1.2 DNA提取
大曲微生物宏基因组的提取参考本研究室的前期研究[13-14]。
1.3 PCR扩增、Illumina Miseq测序和高通量数据分析
通过真菌内转录间隔区(ITS)1区研究真菌种群结构,利用ITS1F (5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2(5′-TGCGTTCTTCATCGATGC-3′)为引物对ITS1区序列进行扩增。PCR扩增程序:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,共25个循环;最后72 ℃ 10 min。扩增产物通过琼脂糖凝胶回收、定量后,依据所需数据深度进行文库制备,然后在Illumina Miseq PE300平台上进行2×150 bp双端测序。原始数据经Qiime (v1.8.0)处理后,再对序列进行拼接,并根据接头序列将不同样本进行分类。随后依次去除序列上的接头序列、标签和引物序列,并去除低质量序列和嵌合体序列,得到优质数据(clean data)。再利用UCLUST将全部优质数据按照97%序列相似度聚类,进行操作分类单元(operational taxonomic units,OTU)划分,提取代表序列,并得到OTU表[15]。将OTU代表序列与NCBI nt数据库和UNITE数据库(v 6.0)进行比对[16],获得OTU物种分类信息。
1.4 荧光定量PCR
真菌18S rRNA基因的相对丰度定量参考ROUSK等[17]的研究。引物Fung(5′-ATTCCCCGTTACCCGTTG-3′)和NS1(5′-GTAGTCATATGCTTGTCTC-3′)用于扩增真菌18S rRNA基因部分序列[18]。通过扩增一系列浓度梯度的含有酿酒酵母18S rRNA基因全长拷贝的质粒建立标准曲线。PCR反应体系参考QuantiNova SYBR Green PCR试剂盒说明书。使用CFX96 Touch实时定量PCR仪进行所有qPCR反应,每个样本重复3次。PCR扩增程序: 94 ℃、4 min,94 ℃、30 s,55 ℃、30 s,72 ℃、30 s,共40个循环;最后72 ℃、2 min。
1.5 丝状真菌的纯培养
取10 g曲粉加入到盛有90 mL无菌生理盐水的250 mL锥形瓶中,并在摇床中以200 r/min振荡1 h。然后将大曲生理盐水混合物稀释至终浓度为1 g/L,取200 μL均匀涂布于5种不同的真菌培养基上,并放置于28、37 和50 ℃培养。每种大曲在不同培养基和温度条件下重复3次。
1.6 丝状真菌鉴定与系统发育分析
使用引物ITS1(5′-TCCGTAGGTGAACCTGCGG-3′)和ITS4(5′-TCCTCCGCTTATTGATATGC-3′)对纯培养获得的丝状真菌的ITS全长序列进行扩增[19]。50 μL的PCR反应体系包括:1×Taq PCR Mastermix、0.5 μmol/L引物、50 ng基因组DNA。PCR扩增程序:94 ℃、5 min;94 ℃、30 s,55 ℃、30 s,72 ℃、1 min,共35个循环;最后72 ℃、10 min。PCR产物使用通用DNA纯化试剂盒纯化后进行测序分析。利用NCBI BLAST工具(http://www.ncbi.nlm.nih.gov/blast)将获得核酸序列进行手动比对,并使用MEGA (version 6.0)软件对获得的序列进行系统发育分析[20]。最后将获得的系统发育树通过iTOL进行可视化[21]。
1.7 次级代谢产物合成酶基因扩增
通过对次级代谢产物合成酶表达基因进行扩增,研究大曲丝状真菌次级代谢产物合成能力。使用简并引物(表2)扩增的次级代谢物合成基因包括非核糖体多肽类合成酶(non-ribosomal peptides synthase,NRPSs)、I型聚酮类合成酶(type I polyketide synthase,PKS I)、III型聚酮类合成酶(type III polyketide synthase,PKS III)、PKS-NRPS杂合类合成酶和萜烯类合成酶(terpene synthase,TPS)5种。25 μL的PCR反应体系包括:1×Taq PCR Mastermix、0.5 μmol/L引物、100 ng基因组DNA。每种基因的扩增包含1组阳性对照和2组阴性对照。阳性对照使用包含上述所有5种基因的A. parasiticus (CGMCC 3.6155)基因组;阴性对照除了使用无菌水作为扩增模板,还使用Saccharomyces cerevisiae基因组作为扩增模板,因为该物种基因组不含任何上述5种次级代谢物合成基因[22]。PCR产物使用1%琼脂糖凝胶电泳检测,并使用Bio-Rad凝胶成像系统进行拍照。
表2 次级代谢产物合成基因及其简并引物
Table 2 Target genes and degenerate primer pairs

目的基因引物序列(5’-3’)PKSIKAF1GARKSiCAYGGiACiGGiACKAR1CCAYTGiGCiCCRTGiCCiGARAAPKSIIICHS1GAYTGGGCiVTNCAYCCBGGiGGDCHS2YTCNAYNKTRAKVCCiGGVCCRAAPKS-NRPS杂合类XKS1TTYGAYGCiBCiTTYTTYRAXKS2CRTTiGYiCCiCYDAAiCCAAANRPSAUG003CCGGCACCACCGGNAARCCHAAAUG007GCTGCATGGCGGTGATGSWRTSNCCBCCTPSTPS1GCiTAYGAYACiGCiTGGGTTPS2RAAiGCATiGCiGTRTCRTC
注:简并碱基符号:B=C/G/T;D=A/G/T;i=肌酐(inosine);K=G/T;M=A/C;R=A/G;S=C/G;Y=C/T。
2 结果与分析
2.1 高通量测序比较不同大曲中丝状真菌的群落结构
通过利用高通量测序技术,在ZW、AGW和GW 3种大曲样品中分别检测到了217、235和207个真菌OTU。其中,丝状真菌在3种大曲样本中所占的比例稳定在25.6%~26.4%(表3),说明丝状真菌的物种比例在不同类型的大曲中非常稳定,基本不受大曲类型的影响。
表3 大曲真菌及丝状真菌多样性分析
Table 3 Diversity of fungal and filamentous fungal community in Daqu
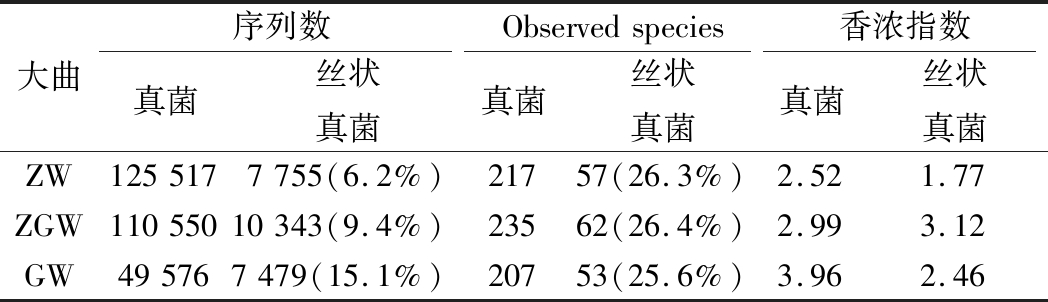
大曲序列数Observedspecies香浓指数真菌丝状真菌真菌丝状真菌真菌丝状真菌ZW1255177755(6.2%)21757(26.3%)2.521.77ZGW11055010343(9.4%)23562(26.4%)2.993.12GW495767479(15.1%)20753(25.6%)3.962.46
进一步将丰度高于1%的OTU序列使用NCBI BLAST进行手动比对,获得OTU在种水平的物种分类信息。如图1所示,不同样本之间丝状真菌的组成差异非常明显。Aspergillus属是丝状真菌中主要的微生物属,该属在ZW、ZGW和GW样本中比例分别为85%、58%和31%。Aspergillus属是环境中广泛存在的1种丝状真菌,文献报道该属能高效产生葡糖淀粉酶(2 463±343 U/g)和α-淀粉酶(1 491±324U/g)[23]。
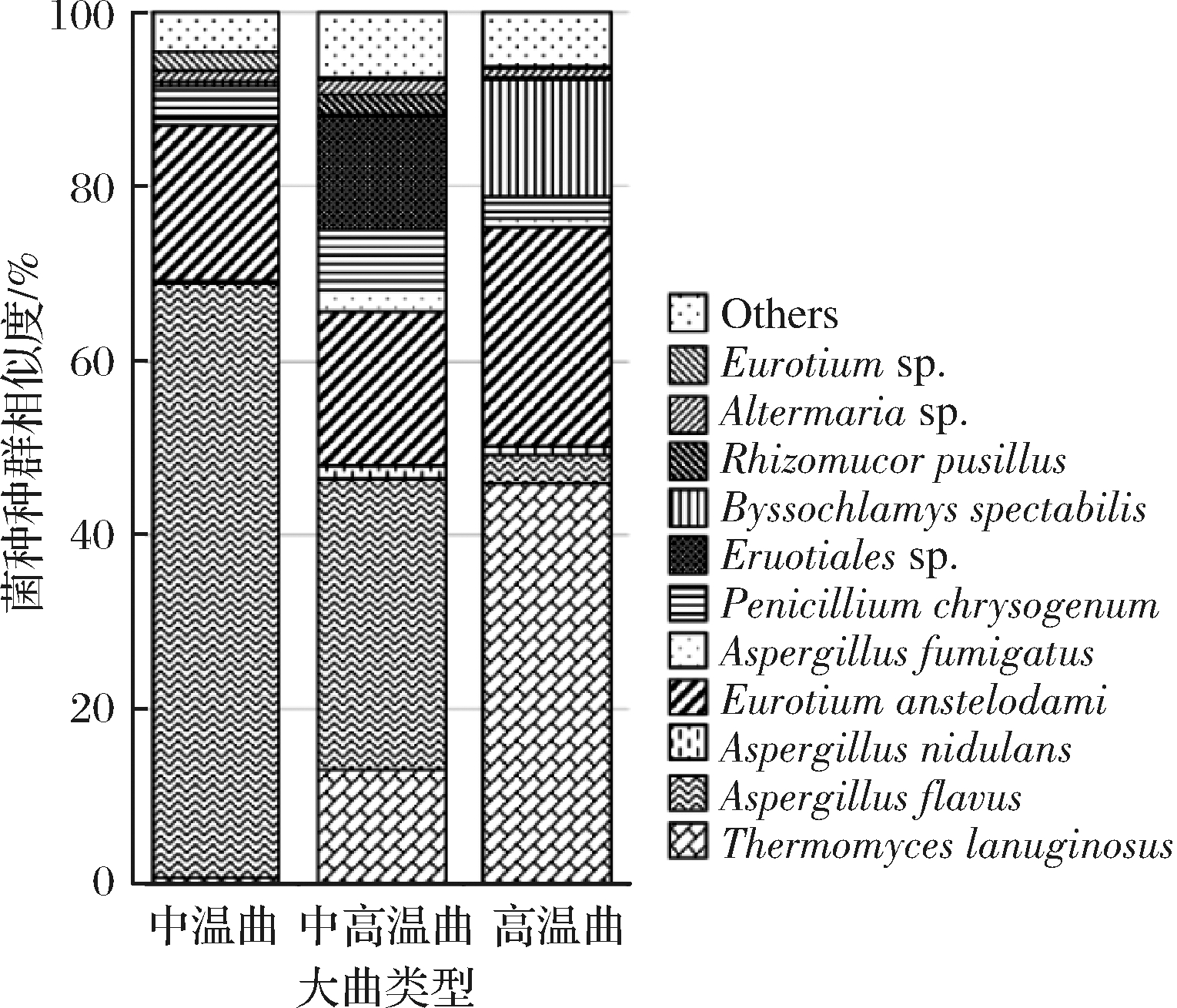
图1 大曲中丝状真菌种群结构(种水平)
Fig.1 Taxonomic classification of filamentous fungal reads at species level based on ITS1 region
高通量测序结果表明,该属在大曲中主要包括A. flavus、A. nidulans、A. fumigatus和Eurotium amstelodami等微生物种。与ZW样本中Aspergillus属占绝对优势不同,GW样本中Thermomyces属丰度最高。并且该属的比例随大曲发酵温度的升高而增加,其在ZW、ZGW和GW样本中比例分别为1%、13%和46%。由此可见,Thermomyces和Aspergillus的比例可用于对大曲进行区分。文献报道Thermomyces属是一种嗜热的丝状真菌,其最适生长温度为50 ℃,甚至能在60 ℃条件下生长[24]。高温曲在发酵过程中的最高温度可以达到60~70 ℃,因此有利于嗜热的Thermomyces属生长繁殖。Thermomyces属可以高效表达半纤维素酶,半纤维素酶可以将植物细胞壁的主要成分半纤维素水解成可发酵性的寡糖[24],在酒精发酵过程中为酵母和细菌提供能量来源。
2.2 大曲中丝状真菌定量分析
大曲中丝状真菌的数量通过实时荧光定量PCR(RT-PCR)技术结合高通量测序数据确定。首先利用一系列浓度梯度的含有酿酒酵母18S rRNA基因全长拷贝的质粒建立RT-PCR标准曲线。如图2-A所示,良好的扩增效率(97%,通过E =10-1/斜率-1计算而来)和线性关系(R2=0.994 9)表明了标准曲线的准确性。然后根据丝状真菌的序列数占真菌总序列数的比例,计算出丝状真菌在不同大曲中的数量。从图2-B可以看出,ZW样本有最多数量的真菌(1.73×107个)和丝状真菌(1.07×106个),GW样本真菌(1.38×106个)和丝状真菌(2.08×105个)最少,说明大曲中真菌和丝状真菌的数量随大曲发酵最高温度的升高而减少,原因可能是因为较少的微生物可以在高温环境下存活。
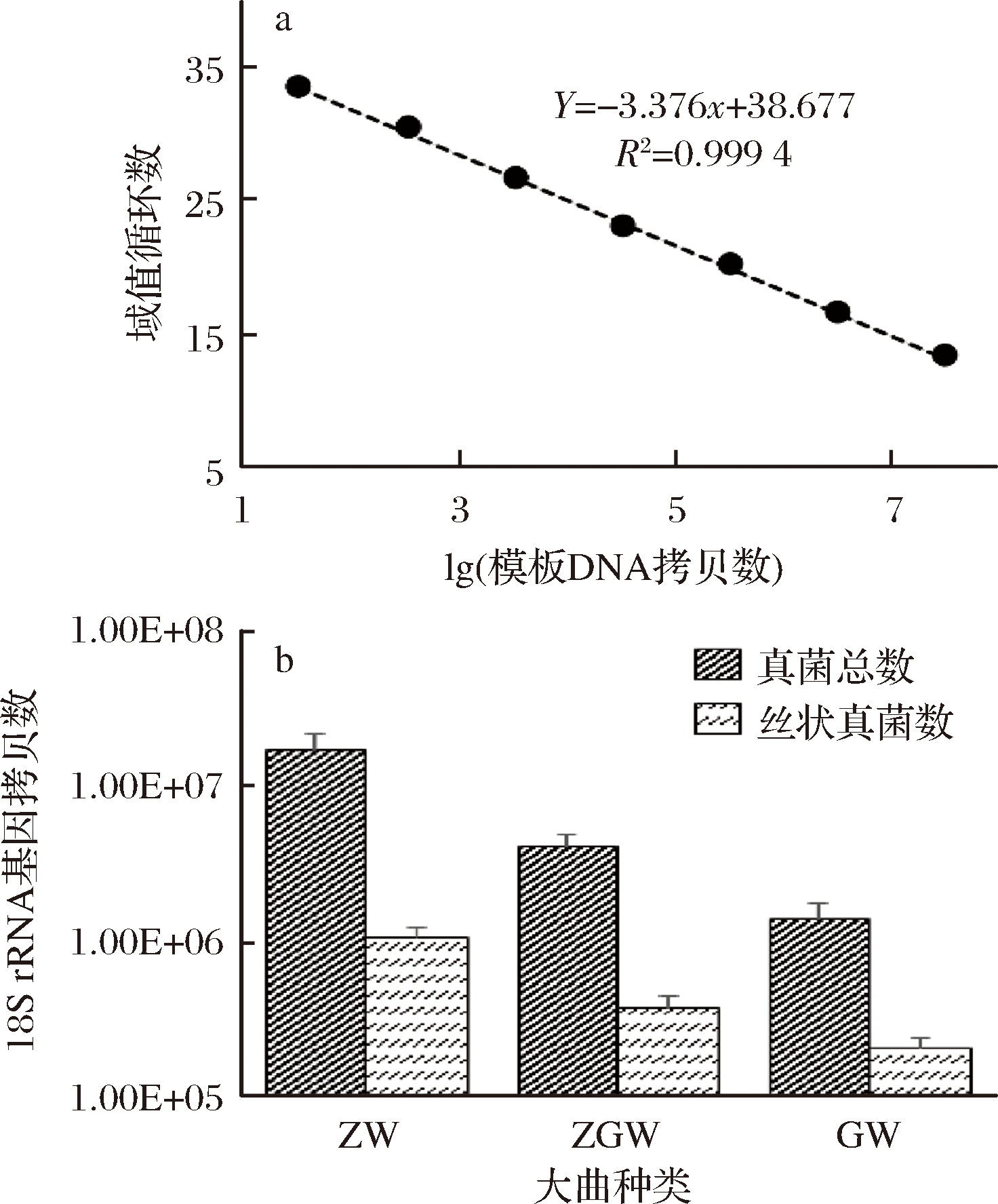
a-RT-PCR定量曲线;b-真菌与丝状真菌18S rRNA基因拷贝数
图2 RT-PCR结合高通量测序数据确定大曲中丝状 真菌数量
Fig.2 Quantification of the filamentous fungi by using real- time PCR coupled with pyrosequencing
2.3 基于可培养的方法研究大曲中丝状真菌的物种多样性
本研究选择GW样本来研究丝状真菌的物种多样性,一方面,由于GW样本中丝状真菌序列数占真菌序列总数的比例最大,因而筛选到丝状真菌的可能性更高;另一方面,来自极端环境的微生物产生天然产物的可能性更高。因为这些微生物可能是系统发育上全新的微生物,并且为了适应独特的环境,还可能存在全新的天然产物合成途径[25]。
由于不同类型的丝状真菌对营养物质的需求可能不同,因此为了获得尽可能多的丝状真菌物种,使用5种不同类型的培养基进行筛选。在筛选过程中,对于具有相似形态和生长特征的丝状真菌,仅保留1株。最终共筛选获得50株丝状真菌(表4),其中有45株是在28 ℃培养条件下获得(PDA培养基24株,MEA培养基7株,CMA培养基3株,SDA培养基5株,CDA培养基6株),在37 ℃培养条件下并未获得丝状真菌单菌落,剩下5株在50 ℃培养条件下获得(PDA培养基3株,MEA培养基2株)。
进一步对丝状真菌的rDNA-ITS序列进行BLAST比对分析发现,50株分离株大部分与已报道的菌株具有99%以上的相似度,除了PDA-7和PDA-16分别只有98%和97%的相似度,说明PDA-7和PDA-16可能是分类上新的微生物种。这50株菌株由散囊菌目(Eurotiales)、毛霉目(Mucorales)、炭角菌目(Xylariales)和格孢菌目(Pleosporales)4个微生物目组成,共分为曲霉属(Aspergillus)、红曲霉属(Monascus)、丝衣霉属(Byssochlamys)、横梗霉属(Lichtheimia)、根毛霉属(Rhizomucor)、毛霉属(Mucor)、节菱孢属(Arthrinium)、链格孢属(Alternaria)、嗜热真菌属(Thermomyces)和Rasamsonia十个属,其中Aspergillus属丝状真菌的种类最为丰富,共30株分离株属于该微生物属,隶属于19个不同的种。
过去的研究曾采用单一培养基对高温大曲中的丝状真菌进行过筛选,但是仅能获得有限种类的丝状真菌(P. variotii、 A. oryzae、P. namyslowskii、R. microsporus、Microascus cirrosus、Monascus purpureus、P. chrysogenum和A. terreus [23])。而本研究中,同样在高温大曲中,筛选鉴定出至少28个不同的丝状真菌种,说明大曲中丝状真菌的物种丰富度可能被严重低估。
另外还发现,虽然大部分丝状真菌分离株能在高通量测序结果中找到对应的微生物属,但仍然有少数丝状真菌仅能通过纯培养获得,例如Mucor和Arthrinium属的微生物,其原因可能是用于高通量测序的通用引物的选择性和覆盖范围限制了对所有物种的完整表征[26]。
表4 基于纯培养获得的丝状真菌的物种多样性和分类群信息
Table 4 Species diversity and taxon information of filamentous fungi by culture-dependent approach

培养温度菌株编号物种分类目水平属水平最近亲缘关系(检索号)相似度/%文献报道来源28℃PDA-1EurotialesAspergillusAspergillustamarii(KP784375)100咖啡豆,巴西CDA-3Aspergillussydowii(KP269026)100海藻,中国CDA-4Aspergillussydowii(AY373868)100室内环境,美国PDA-17Aspergillussydowii(AY373869)99室内环境,美国SDA-43Aspergillussydowii(KP994297)99未给出MEA-49Aspergillussydowii(KP942928)100香蕉,埃塞俄比亚PDA-6Aspergillusoryzae(KR265109)Aspergillusflavus(KJ482648)100牛,埃及没有给甘蔗渣,泰国PDA-15Aspergillusflavus(KR296888)99土壤,新加坡CDA-28Aspergillusoryzae(KR871216)Aspergillusflavus(KT004403)100马铃薯皮,尼日利亚未给出PDA-9,CMA-13Aspergillusfumigatus(KR023997)99亚马逊土壤,巴西PDA-20Aspergillusfumigatus(KP881424)99堆积有机残留物,巴西SDA-44Aspergillussp.(KT164776)100海绵,中国SDA-46Aspergillussp.(GQ169462)100窝奶酪,意大利CDA-53Aspergillusnidulans(KP278174)99加拉帕戈斯雀PDA-40Aspergillusnidulans(AY373888)99室内环境,美国MEA-18Aspergillusamstelodami(AY373885)100室内环境,美国MEA-24,MEA-25,MEA-26Aspergillusamstelodami(AY373885)99室内环境,美国PDA-63Aspergillusamstelodami(KR265111)99病牛的鼻子,埃及PDA-60Aspergillusparasiticus(JF412785)99杏仁和玉米,葡萄牙SDA-36Aspergillussp.SCSIOXWS02F40(KT164776)99海绵,中国PDA-64Aspergillusniger(KP940600)99重金属污染土壤,中国SDA-45,PDA-51Eurotiumamstelodami(JQ867376)99海藻,印度MEA-48Eurotiumsp.(GQ169454)100窝奶酪,意大利PDA-65Eurotiumcristatum(GU784865)100大米,马来西亚PDA-2Emericellasp.(JQ425379)99柑橘叶,埃及CDA-52Emericellanidulans(HQ285615)99米酒,韩国PDA-35MonascusMonascusruber(JN942658)100室内环境,美国PDA-5,PDA-14ByssochlamysPaecilomycesvariotii(KC411915)99石榴,印度PDA-8Byssochlamysspectabilis(KR812240)100蚁穴,巴西PDA-7MucoralesLichtheimiaLichtheimiaramosa(HQ285700)98米酒,韩国PDA-10Lichtheimiaramosa(JN315007)99豆饼,韩国CMA-21Lichtheimiaramosa(KF417720)99河口泥,英国CDA-22Lichtheimiaramosa(HQ285692)100米酒,韩国MEA-12,PDA-42Lichtheimiaramosa(HQ285650)100米酒,韩国PDA-34RhizomucorRhizomucorvariabilis(HQ285715)99米酒,韩国PDA-11MucorMucorcircinelloides(KT192269)100槐花,中国PDA-16Mucorhiemalis(LN714573)97森林土壤,捷克CMA-23XylarialesArthriniumArthriniumsacchari(HQ914941)99海洋巨藻,中国PDA-61PleosporalesAlternariaAlternariatenuissima(KT218501)100植物叶子,中国50℃PDA-31EurotialesThermomycesThermomyceslanuginosus(AB085929)99土壤,日本PDA-32,PDA-33,MEA-37,MEA-38RasamsoniaRasamsoniacomposticola(JF417481)99未给出
基于以上结果可以说明,大曲中丝状真菌的物种丰富度可能被显著低估,同时基于纯培养的方法在目前微生物群落研究中仍然发挥着不可替代的作用。
2.4 大曲丝状真菌来源的次级代谢产物合成基因多样性分析
为了进一步探究大曲来源的丝状真菌合成天然生物活性物质的遗传潜力,利用简并引物对筛选得到的50株丝状真菌的NRPSs、TPS、PKS-NRPS、I型和III型PKSs 5种次级代谢产物合成基因进行了扩增。从图3可以看出,50株分离株全部含有至少1个次级代谢产物合成基因。其中48%的丝状真菌(24株)含有所有靶基因;32%(16株)含有4个靶基因;16%(8株)含有3个靶基因;2%(1株)含有2个靶基因;2%(1株)仅含有1个靶基因。结果表明,大曲来源的丝状真菌具有合成多种次级代谢产物的遗传潜力。
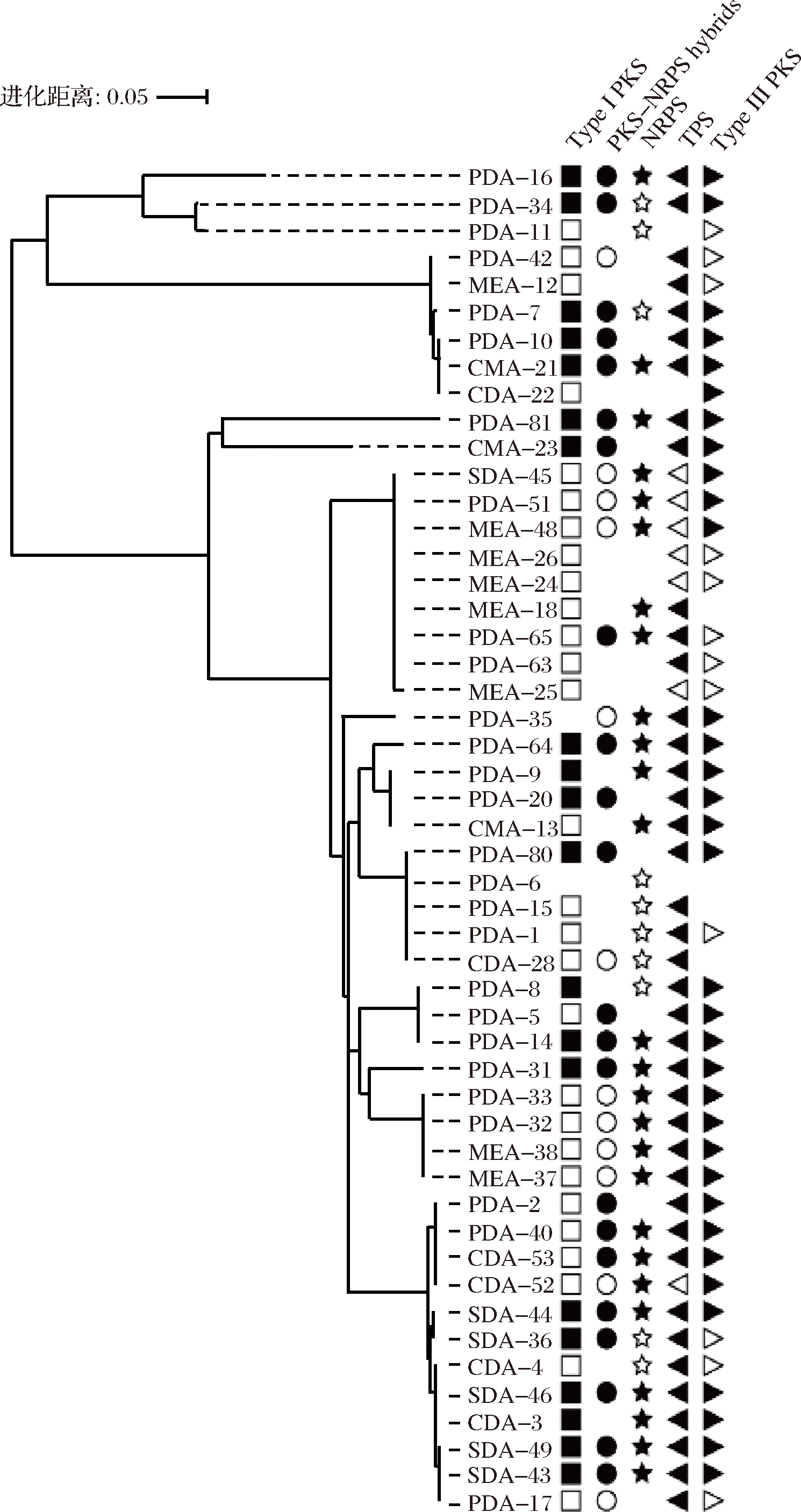
图3 GW来源的丝状真菌的系统发育树及其次级代谢产物合成基因分布
Fig.3 Phylogenetic tree of filamentous fungi derived from GW coupled with presence of genes involved in synthesis of bioactive compounds
注:不同的形状代表不同的次级代谢物合成基因;填充的形状表示含有2个以上的SM合成基因,无填充的形状表示仅含有1个SM合成基因,空缺代表没有相应的SM合成基因。
另外,与前人研究发现真菌含有多种PKS基因的结果相一致[27],本研究几乎在所有分离株中都观察到至少1个I型和III型PKSs基因。文献报道,PKS基因参与多种天然产物的合成,例如,I型PKS基因能够合成大量具有药理学活性的次级代谢产物,包括红霉素、免疫抑制剂FK506以及抗寄生虫的阿维菌素类化合物[28],因此近年来PKS基因受到了广泛的关注[25, 27]。
相同微生物种的丝状真菌也有可能含有不同的次级代谢产物合成基因,如Lichtheimia ramosa(PDA-10、CMA-21和CDA-22)和A. sydowii(CDA-3、CDA-4、PDA-17、SDA-43和MEA-49)。YU等[12]从海绵中筛到的8株Aspergillus terreus,也显示出能够产生不同的次级代谢产物的现象。造成这一现象的原因可能是真菌的次级代谢基因簇通过多次重排、重复或丢失而迅速进化[29]。大曲中的丝状真菌在适应环境的过程中,可能丢失或者获得了一些次级代谢基因。
为验证真菌天然产物的合成,仍需通过模拟大曲发酵环境对分离株进行培养,并对产物进行化学鉴定和细胞毒性测试,以证实大曲来源的丝状真菌是否能够合成天然生物活性物质。
3 结论
通过高通量测序手段对3种不同类型的成品曲中的丝状真菌的群落结构进行了分析,观察到Thermomyces和Aspergillus属的丰度在不同类型的大曲中差异非常明显,能用于区分不同类型的大曲;另外利用纯培养的手段对高温大曲中丝状真菌进行了筛选,共获得了来自10个不同属的50株丝状真菌,表明大曲中丝状真菌的物种丰富度被低估;进一步对这50株丝状真菌基因组中参与次级代谢产物合成的基因进行了扩增,结果发现高温大曲来源的丝状真菌具有强大的次级代谢产物合成潜力。但是由于在大多数情况下,次级代谢产物合成基因是沉默的,因此仍需有针对性的检测大曲以及白酒中的生物活性成分,并分析这些物质的微生物来源。综合以上结论可以看出,大曲是一种优良的微生物库和基因库,包含丰富的微生物资源和功能基因资源。
[1] ZHENG X, TABRIZI M R, NOUT M J R, et al. Daqu-a traditional Chinese liquor fermentation starter [J]. J Inst Brew, 2011, 117(1):82-90.
[2] DU H, WANG X S, ZHANG Y H, et al. Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter [J]. Int J Food Microbiol, 2019, 297:32-40.
[3] CHEN B, WU Q, XU Y. Filamentous fungal diversity and community structure associated with the solid state fermentation of Chinese Maotai-flavor liquor [J]. Int J Food Microbiol,2014, 179:80-84.
[4] KNIGHT R, VRBANAC A, TAYLOR B C, et al. Best practices for analysing microbiomes [J]. Nature Reviews Microbiology,2018, 16(7):410-422.
[5] 乔晓梅, 赵景龙,杜小威,等. 高通量测序法对清香大曲真菌群落结构的分析[J]. 酿酒科技,2015(4):28-31.
[6] 夏玙, 罗惠波,周平,等. 不同处理方式的大曲真菌群落差异分析[J]. 食品科学,2018, 39(22):173-179.
[7] WANG X S, DU H, ZHANG Y, et al. Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation [J]. Appl Environ Microbiol,2018, 84(4):13.
[8] WANG X S, DU H, XU Y. Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor [J]. Int J Food Microbiol, 2017, 244: 27-35.
[9] ANDERSEN M R, NIELSEN J B, KLITGAARD A, et al. Accurate prediction of secondary metabolite gene clusters in filamentous fungi [J]. Proceedings of the National Academy of Sciences of the United States of America,2013, 110(1):E99-E107.
[10] IMHOFF J F. Natural products from marine fungi—Still an underrepresented resource [J]. Mar Drugs, 2016, 14(1):19.
[11] JALGAONWALA R E, MOHITE B V, MAHAJAN R T. A review: Natural products from plant associated endophytic fungi [J]. Journal of Microbiology and Biotechnology Research,2017, 1(2):21-32.
[12] YU Z, ZHANG B, SUN W, et al. Phylogenetically diverse endozoic fungi in the South China Sea sponges and their potential in synthesizing bioactive natural products suggested by PKS gene and cytotoxic activity analysis [J]. Fungal Diversity, 2013, 58(1):127-141.
[13] 杜海, 杜小威,赵景龙,等. 贮存过程中大曲原核微生物多样性及土味素含量变化规律[J]. 食品与发酵工业,2017, 43(4):1-6.
[14] 王雪山, 杜海,徐岩. 清香型白酒发酵过程中微生物种群空间分布[J]. 食品与发酵工业,2018, 44(9):1-8.
[15] EDGAR RC. Search and clustering orders of magnitude faster than BLAST [J]. Bioinformatics,2010, 26(19):2 460-2 461.
[16] K LJALG U, LARSSON K H, ABARENKOV K, et al. Unite: A database providing web-based methods for the molecular identification of ectomycorrhizal fungi [J]. New Phytol,2005, 166(3):1 063-1 068.
LJALG U, LARSSON K H, ABARENKOV K, et al. Unite: A database providing web-based methods for the molecular identification of ectomycorrhizal fungi [J]. New Phytol,2005, 166(3):1 063-1 068.
[17] ROUSK J, BÅÅTH E, BROOKES P C,et al. Soil bacterial and fungal communities across a pH gradient in an arable soil [J]. The ISME Journal,2010, 4(10):1 340-1 351.
[18] KRISHNAN Y, BONG C P C, AZMAN N F,et al. Co-composting of palm empty fruit bunch and palm oil mill effluent: microbial diversity and potential mitigation of greenhouse gas emission [J]. Journal of Cleaner Production,2017, 146:94-100.
[19] WHITE T J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics [J]. PCR Protocols: a guide to methods and applications,1990, 18:315-322.
[20] TAMURA K, STECHER G, PETERSON D,et al. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]. Mol Biol Evol,2013, 30(12):2 725-2 729.
[21] LETUNIC I, BORK P. Interactive tree of life v2: Online annotation and display of phylogenetic trees made easy [J]. Nucleic Acids Res,2011,39(2):W475-W478.
[22] KHALDI N, SEIFUDDIN F T, TURNER G,et al. SMURF: Genomic mapping of fungal secondary metabolite clusters [J]. Fungal Genet Biol,2010, 47(9):736-741.
[23] CHEN B, WU Q, XU Y. Filamentous fungal diversity and community structure associated with the solid state fermentation of Chinese Maotai-flavor liquor [J]. Int J Food Microbiol,2014, 179:80-84.
[24] SINGH S, MADLALA A M, PRIOR B A. Thermomyces lanuginosus: properties of strains and their hemicellulases [J]. FEMS Microbiol Rev,2003, 27(1):3-16.
[25] ZHAO J, YANG N, ZENG R Y. Phylogenetic analysis of type I polyketide synthase and nonribosomal peptide synthetase genes in Antarctic sediment [J]. Extremophiles,2008, 12(1):97-105.
[26] SCANLAN P D, MARCHESI J R. Micro-eukaryotic diversity of the human distal gut microbiota: Qualitative assessment using culture-dependent and-independent analysis of faeces [J]. The ISME Journal,2008, 2(12):1 183-1 193.
[27] LIN X, HUANG Y J, ZHENG Z H, et al. Endophytes from the pharmaceutical plant, Annona squamosa: isolation, bioactivity, identification and diversity of its polyketide synthase gene [J]. Fungal Diversity,2010, 41(1):41-51.
[28] PIEL J, HUI D, FUSETANI N, et al. Targeting modular polyketide synthases with iteratively acting acyltransferases from metagenomes of uncultured bacterial consortia [J]. Environ Microbiol,2004, 6(9):921-927.
[29] KHALDI N, COLLEMARE J, LEBRUN M H, et al. Evidence for horizontal transfer of a secondary metabolite gene cluster between fungi [J]. Genome biology,2008, 9(1):R18.