芝麻香型白酒是我国的创新型白酒,具有浓香型、清香型、酱香型白酒的综合风格,在风味上又别具一格,有着炒芝麻的特有香气[1-2]。大曲是我国固态白酒酿造特有的糖化发酵剂,它不仅含有重要的粗酶和复杂的微生物区系,而且是决定其产品风味特征的香气前体物质等重要的生物活性物质来源,在传统白酒酿造过程中发挥重要的作用[3-5]。“好曲产好酒”,芝麻香型白酒特殊风味的形成亦离不开好曲,高温大曲是芝麻香型白酒酿造的关键原料[6],其为酿酒发酵富集了微生物[7],这些微生物大多具有分泌淀粉酶、蛋白酶的能力,为芝麻香型白酒酿造提供了酶系和前体物质,对芝麻香型白酒特殊风味的形成发挥至关重要的作用[8-9]。
16S rRNA基因序列广泛应用于构建细菌的系统发育树,但在亲缘关系很近的分类类群间,由于序列间的相似度太高而不能有效区分。其中,16S rRNA基因序列不能有效区分枯草芽孢杆菌属的菌种[10]。但近年来研究者发现以编码蛋白的DNA促旋酶A亚基基因(gyrA gene)[11]作为系统发育鉴定标记可以弥补16S rRNA基因的不足。随机扩增多态性DNA标记(random amplifiea polmorphic,RAPD)是1990年WILLIAMS根据限制性内切酶片段长度多态性(RFLP)法研究分子标记时提出的[12]。RAPD是建立在PCR(polymerase chain reaction)基础之上的一种可对整个未知序列的基因组进行多态性分析的分子技术。其以基因组DNA为模板,以单个人工合成的随机多态核苷酸序列为引物,进行PCR扩增。对于不同模板DNA,用同一引物扩增总是有差异的,所以用RAPD就可以进行分子标记研究。理论上,在一定的扩增条件下,扩增的条带数取决于基因组的复杂性。对特定的引物,复杂性越高的基因组所产生的扩增条带数也越多。
本文利用传统筛选方法从本实验室提供的芝麻香型白酒制曲3个过程的细菌资源中筛选出产淀粉酶、蛋白酶的菌株,利用16S rRNA和gyrA基因序列分析鉴定技术对其鉴定到种,并用RAPD分子分型技术对其在制曲过程中的动态演替进行追踪。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
供试菌株为本实验室前期在芝麻香型白酒大曲制曲3个关键阶段分离获得的细菌资源(表1)。
1.1.2 主要试剂
LB成品培养基,北京陆桥技术有限公司;引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’)、1492R(5’-GGTTACCTTGTTACGACTT-3’)、BS-gyrA-F(5’- CAGTCAGGAAATGCGTACGTCTT-3’)、BS-gyrA-R(5’- CAAGGTAATGCTCCAGGCATTGCT-3’)、PCR引物,北京诺赛生物公司;可溶性淀粉,西陇化工股份有限公司;分子生物学试剂,TIANGEN公司;脱脂奶粉、碘液,市售。
表1 大曲发酵过程中的理化指标和菌种数
Table 1 Physicochemical index and number of strains during the process of Daqu fermentation

时期温度/℃湿度/%菌株数编号第1次翻曲阶段(Q1期)6531.027Q1B1-Q1B27第2次翻曲阶段(Q2期)5925.66Q2B1、Q2B2、Q2B4、Q2B5、Q2B7、Q2B8出房期阶段(Q3期)5812.64Q3B1-Q3B4
1.1.3 主要仪器与设备
精密恒温培养箱,上海一恒科学仪器有限公司;FTC-3000P实时荧光定量PCR仪,Canada Funglyn Biotech Inc;Bio-Rad GelDoc EZ凝胶成像系统,伯乐生命医学产品(上海)有限公司;DYY-8C型电泳仪,北京市六一仪器厂。
1.2 方法
1.2.1 产淀粉酶、蛋白酶微生物的筛选
1.2.1.1 产淀粉酶微生物的筛选
将菌种接种在含1.0%淀粉LB琼脂培养基平板上,于30 ℃培养48 h后,在菌落周围滴加碘液,如菌落周围呈现透明圈时,表示菌种产生淀粉酶。若菌落周围染成蓝色,不出现透明圈者,说明该菌株不产生淀粉酶。
1.2.1.2 产蛋白酶微生物的筛选
将菌株分别划线接种于含1.0%的脱脂奶粉的LB琼脂培养基平板上,30 ℃培养48 h后,观察菌落周围是否出现透明圈。
1.2.2 16S rRNA与DNA促旋酶A亚基基因(gyrA gene)序列分析
1.2.2.1 细菌基因组DNA提取
从平板上挑取细菌溶于30 μL无菌水中,先用沸水煮3 min,再放入-20 ℃冰箱中3 min,重复3次,离心,上清液中即含有细菌DNA。
1.2.2.2 16S rRNA与DNA促旋酶A亚基基因PCR扩增和序列测定
16S rRNA基因序列扩增引物:上游引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’),下游引物1492R(5’-GGTTACCTTGTTACGACTT-3’);gyrA基因序列PCR扩增引物:上游引物BS-gyrA-F(5’- CAGTCAGGAAATGCGTACGTCTT -3’),下游引物BS-gyrA-R(5’- CAAGGTAATGCTCCAGGCATTGCT -3’)。16S rRNA基因和gyrA基因的扩增条件均为94 ℃,5min;(94 ℃,1 min,55 ℃,1 min,72 ℃,1.5 min,35个循环);72 ℃,10 min。扩增体系见表2。扩增完成后,取10 μL PCR产物点样1.0 %琼脂糖凝胶电泳来确认PCR产物质量。扩增产物由北京诺赛基因组研究中心有限公司测序。
表2 16S rRNA和gyrA基因序列PCR扩增体系
Table 2 PCR amplification system of 16S rRNA and gyrA gene sequence

试剂50μL反应体系中加样体积/μL基因组模板DNA12×EasyTaqTMPCRSuperMix25引物27F或BS-gyrA-F2引物1492R或BS-gyrA-R2ddH2O补足至20
1.2.2.3 系统发育树的构建
将测序序列提交到GenBank,获得接收号,并在EzTaxon server 2.1中进行比对分析[13],以确定其与模式菌种的同源关系,并下载各模式菌种的有效序列。利用CLUSTAL W软件进行多重序列比对后[14],利用MEGA 7.0软件使用邻接法(neighbor-joining)进行系统发育树构建[15]。
1.2.3 RAPD分子分型
使用1.2.2中提取的细菌基因组DNA为模板,利用RAPD分子分型技术对Q1B16、Q1B18、Q2B2、Q2B7、Q2B8、Q3B2、Q3B3、Q3B4八株贝莱斯芽孢杆菌(Bacillus velezensis)进行菌株分型与动态演替的追踪研究。使用的引物为OPA-02(5’-TGCCGAGCTG-3’)、OPA-18 (5’-AGGTGACCGT-3’)、OPL-07 (5’-AGGCGGGAAC-3’)、OPL-16(5’-AGGTTGCAGG-3’)、OPM-05(5’-GGGAACGTGT-3’)。扩增条件为95 ℃,5 min;(94 ℃,1 min,55 ℃,1 min,72 ℃,2 min,45个循环);72 ℃,10 min。扩增体系见表3。扩增完成后,取10 μL PCR产物点样1.0%琼脂糖凝胶进行电泳分析[16]。
表3 RAPD PCR扩增体系
Table 3 PCR amplification system of RAPD
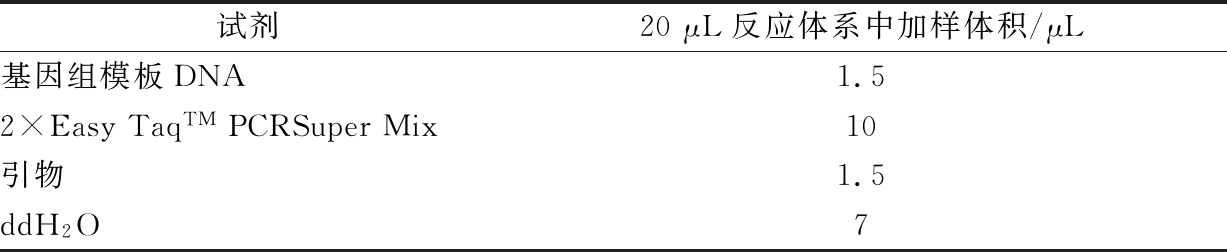
试剂20μL反应体系中加样体积/μL基因组模板DNA1.52×EasyTaqTMPCRSuperMix10引物1.5ddH2O7
2 结果与分析
2.1 产淀粉酶、蛋白酶微生物的筛选结果
根据含1%的淀粉或脱脂奶粉的LB琼脂培养基平板是否有透明圈,筛选出产淀粉酶的细菌5株,Q1B18、Q2B2、Q2B7、Q3B2和Q3B4;产蛋白酶的细菌9株,Q1B16、Q1B25、Q2B2、Q2B7、Q2B8、Q3B1、Q3B2、Q3B3和Q3B4;既产淀粉酶又产蛋白酶的细菌4株,Q2B2、Q2B7、Q3B2和Q3B4(表4,图1和图2)。
表4 产酶微生物的筛选结果
Table 4 Screening results of enzyme-producing microorganisms

菌株编号淀粉酶蛋白酶Q1B16-+Q1B18+-Q1B25-+Q2B2++Q2B7++Q2B8-+Q3B1-+Q3B2++Q3B3-+Q3B4++
注:“+”,阳性;“-”,阴性。
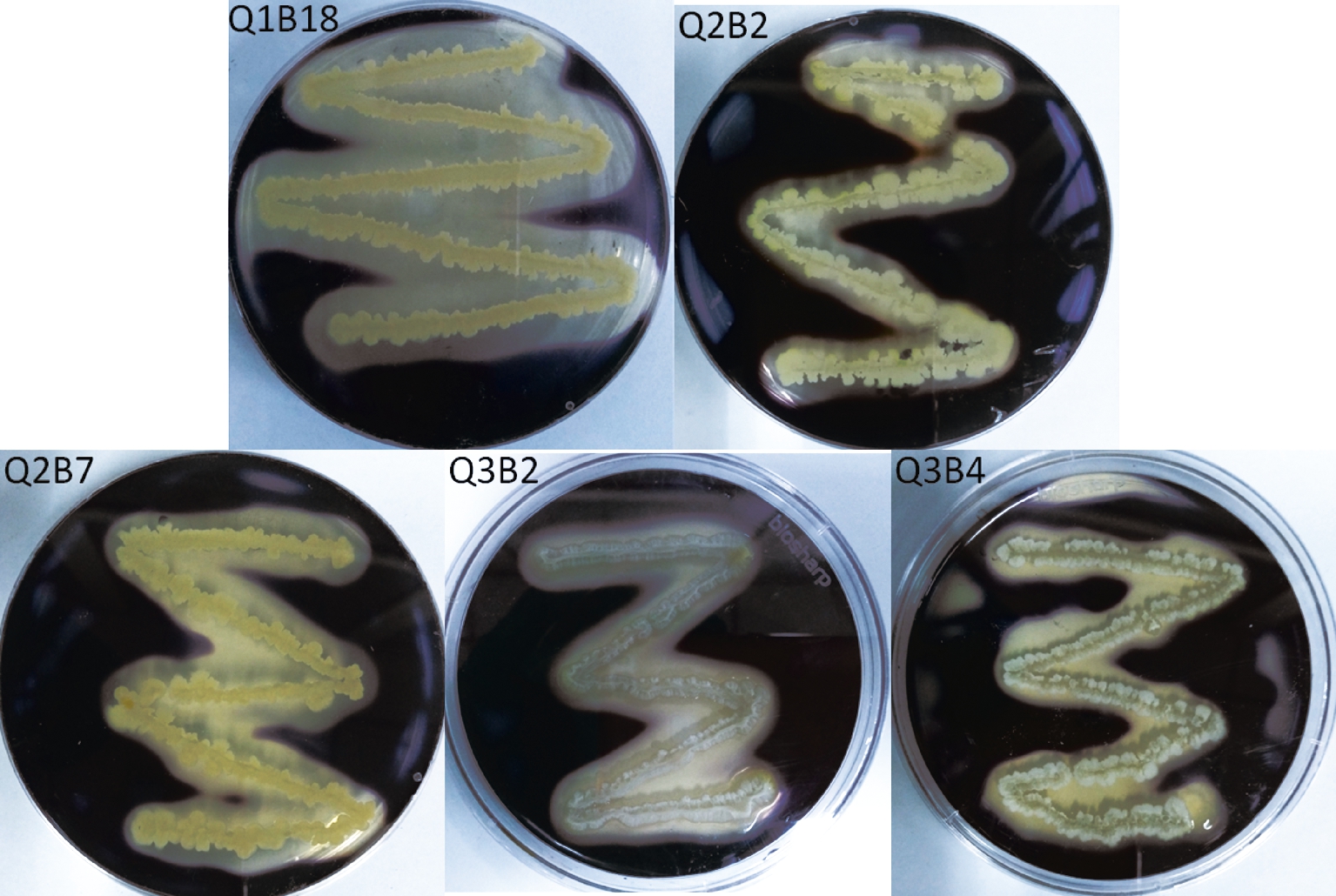
图1 产淀粉酶微生物在淀粉平板上形成透明圈 (加碘液显色)
Fig.1 Transparent circle of amylase-producing microorg- anisms on starch plate
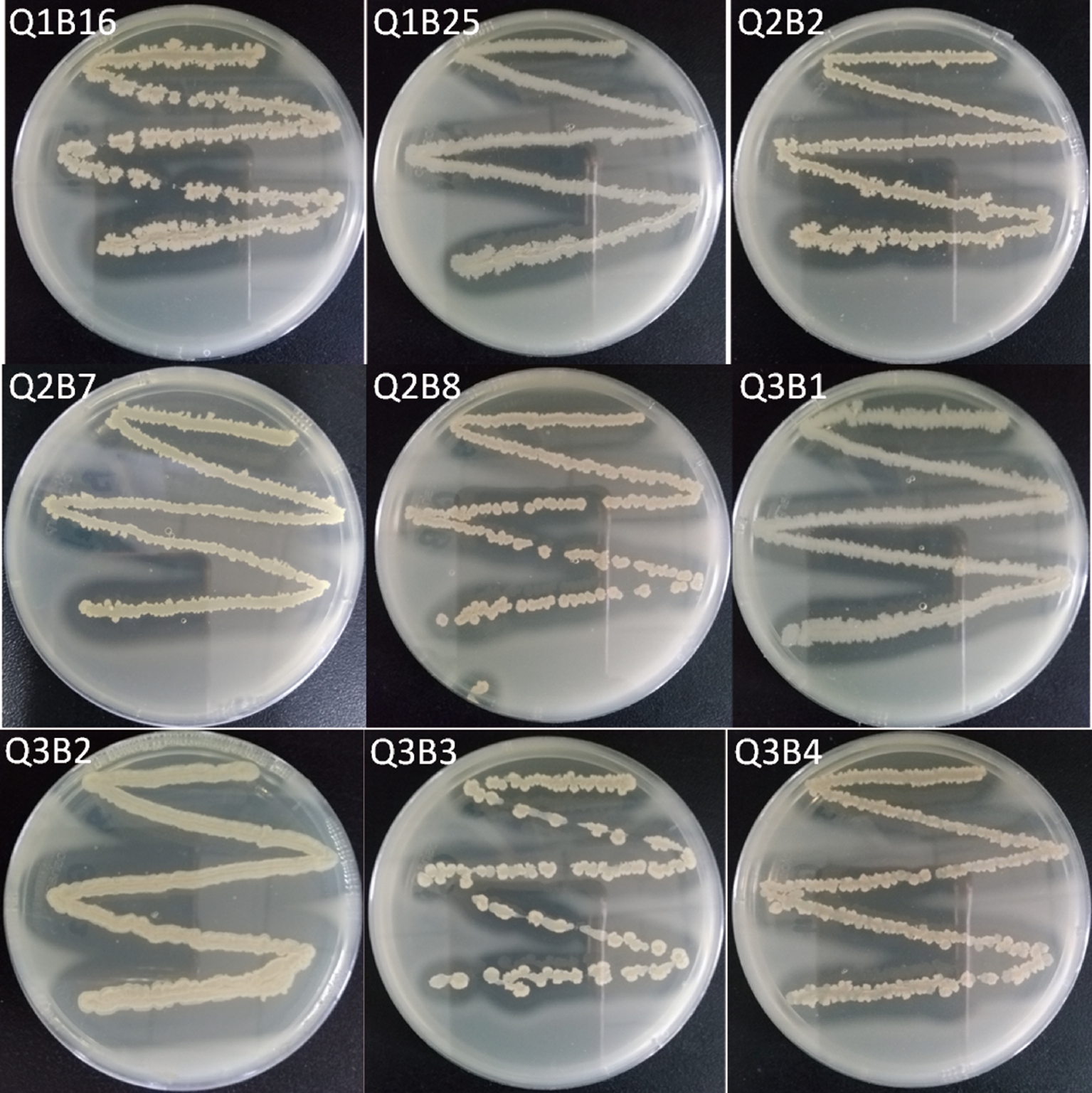
图2 产蛋白酶微生物在脱脂奶粉平板上形成透明圈
Fig.2 Transparent circle of Protease-producing microorganisms on plate of skimmed milk powder
2.2 16S rRNA与DNA促旋酶A亚基基因序列分析结果
利用MEGA 7.0软件使用邻接法(neighbor-joining)对16S rRNA与DNA促旋酶A亚基基因进行系统发育分析。16S rRNA基因构建系统发育树结果表明,所有可产酶菌株归1个属,即芽孢杆菌属(Bacil-lus)(图3)。gyrA基因构建系统发育树结果表明,所有可产酶菌株归属2个种,即枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp.subtilis)和贝莱斯芽孢杆菌(Bacillus velezensis),其中Q1B25、Q3B1归属为枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp.subtilis);Q1B16、Q1B18、Q2B2、Q2B7、Q2B8、Q3B2、Q3B3、Q3B4归属为贝莱斯芽孢杆菌(Bacillus velezensis)(图4)。
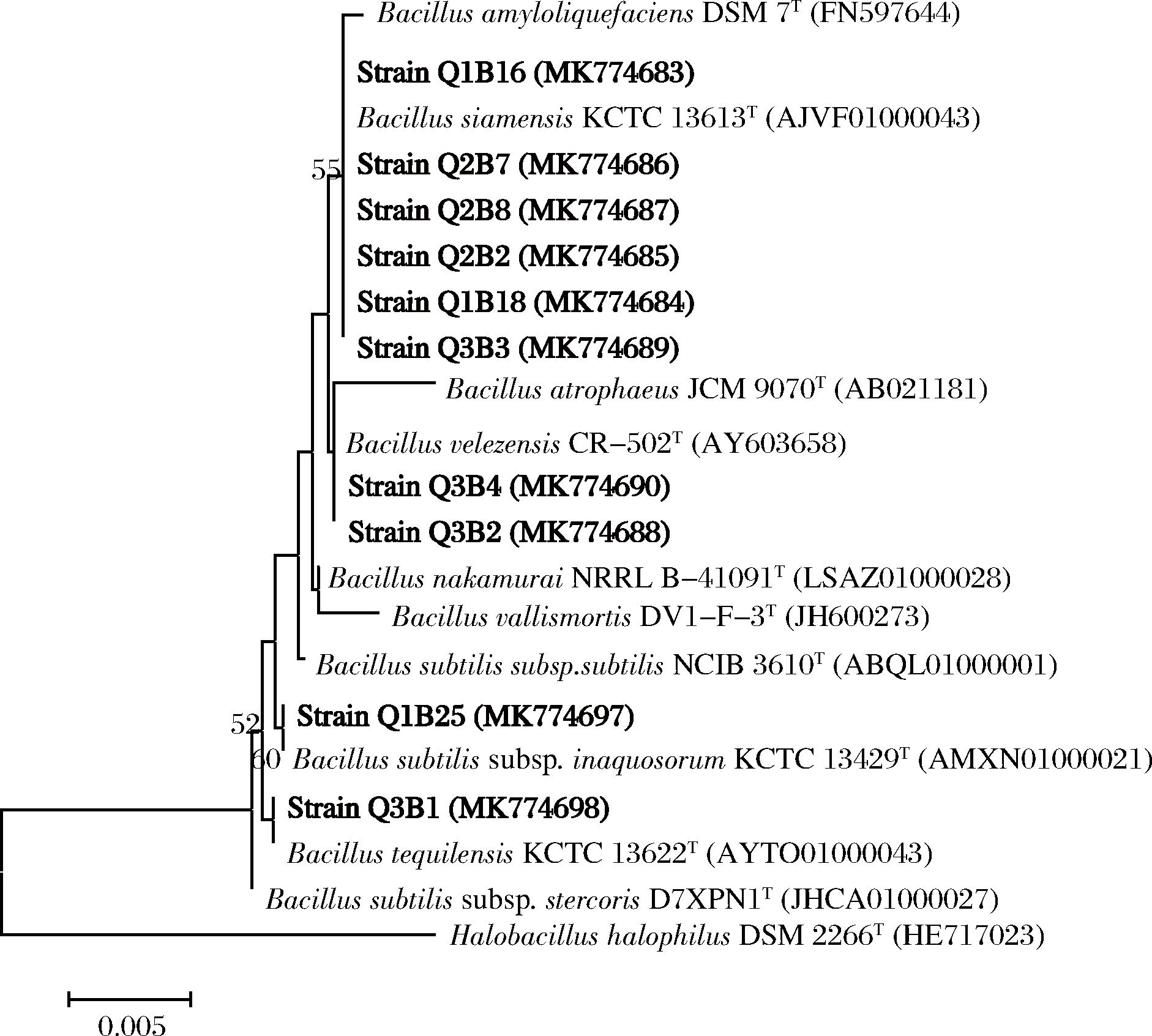
图3 大曲菌株的16S rRNA基因系统发育分析
Fig.3 Phylogenetic analysis of the 16S rRNA gene of strain isolated from Daqu
注:标尺代表0.5%的序列差异度
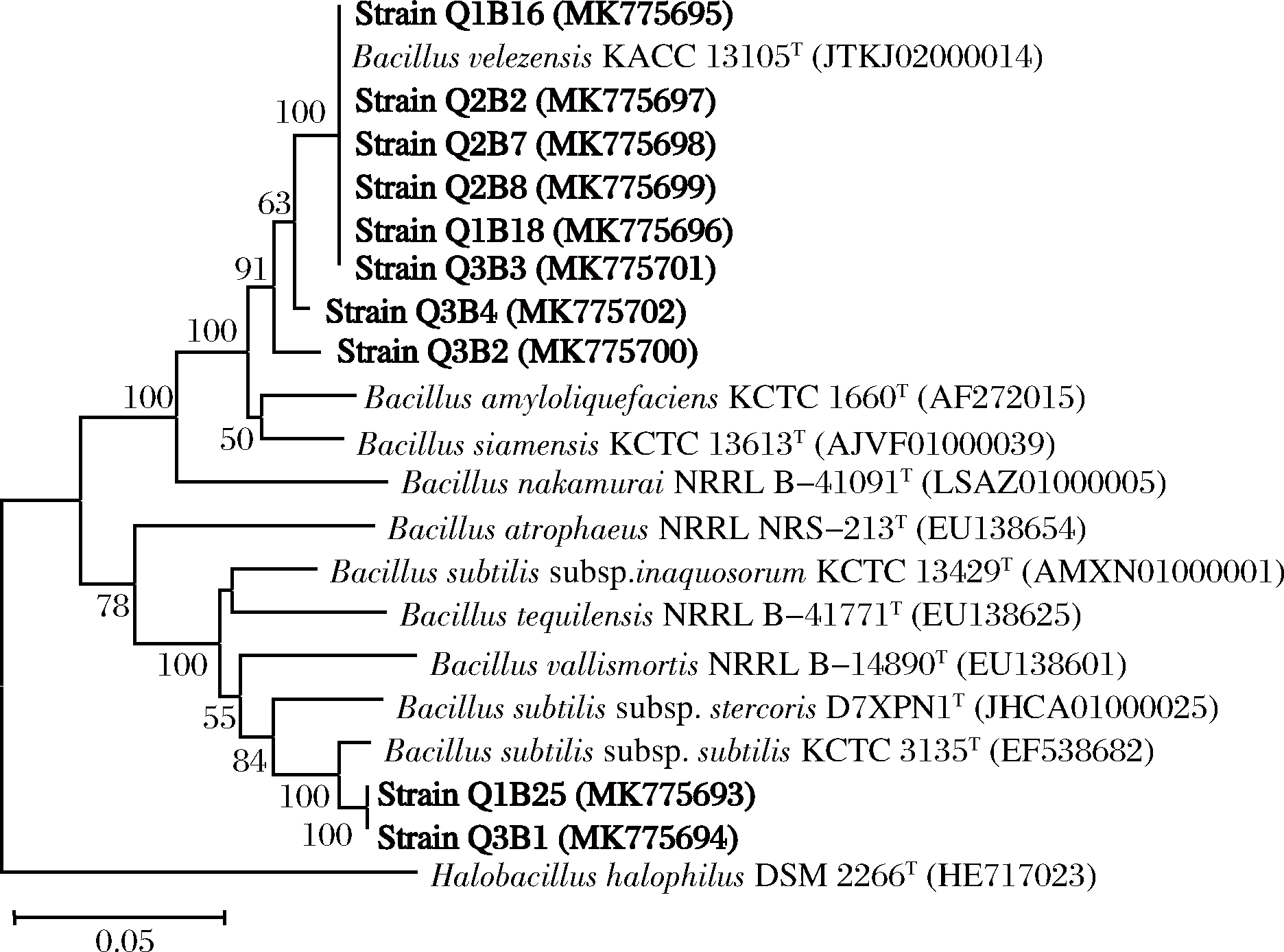
图4 大曲菌株的gyrA基因系统发育分析
Fig.4 Phylogenetic analysis of the 16S rRNA gene of strain isolated from Daqu
注:标尺代表5%的序列差异度
2.3 RAPD分型结果
对Q1B16、Q1B18、Q2B2、Q2B7、Q2B8、Q3B2、Q3B3、Q3B4八株贝莱斯芽孢杆菌(Bacillus velezensis)利用5种RAPD菌株分型引物OPA-02、OPA-18、OPL-07、OPL-16、OPM-05扩增结果的综合分析,表明Q1B16、Q2B2、Q2B7、Q2B8、Q3B3为同一株型,Q1B18、Q3B2、Q3B4分属于不同的株型(图5)。Q1B16和Q1B18同分离于Q1期,但属于同一株型;Q3B2和Q3B4同分离于Q3期,但不属于同一株型,表明芝麻香型白酒高温制曲过程中贝莱斯芽孢杆菌菌群发生了动态演替;Q1B16、Q2B2、Q2B7、Q2B8、Q3B3分别分离于制曲过程中的不同时期,但属于同一株型,表明该株型的细菌具有纵向传递性,在制曲过程中发生了积累与传递。
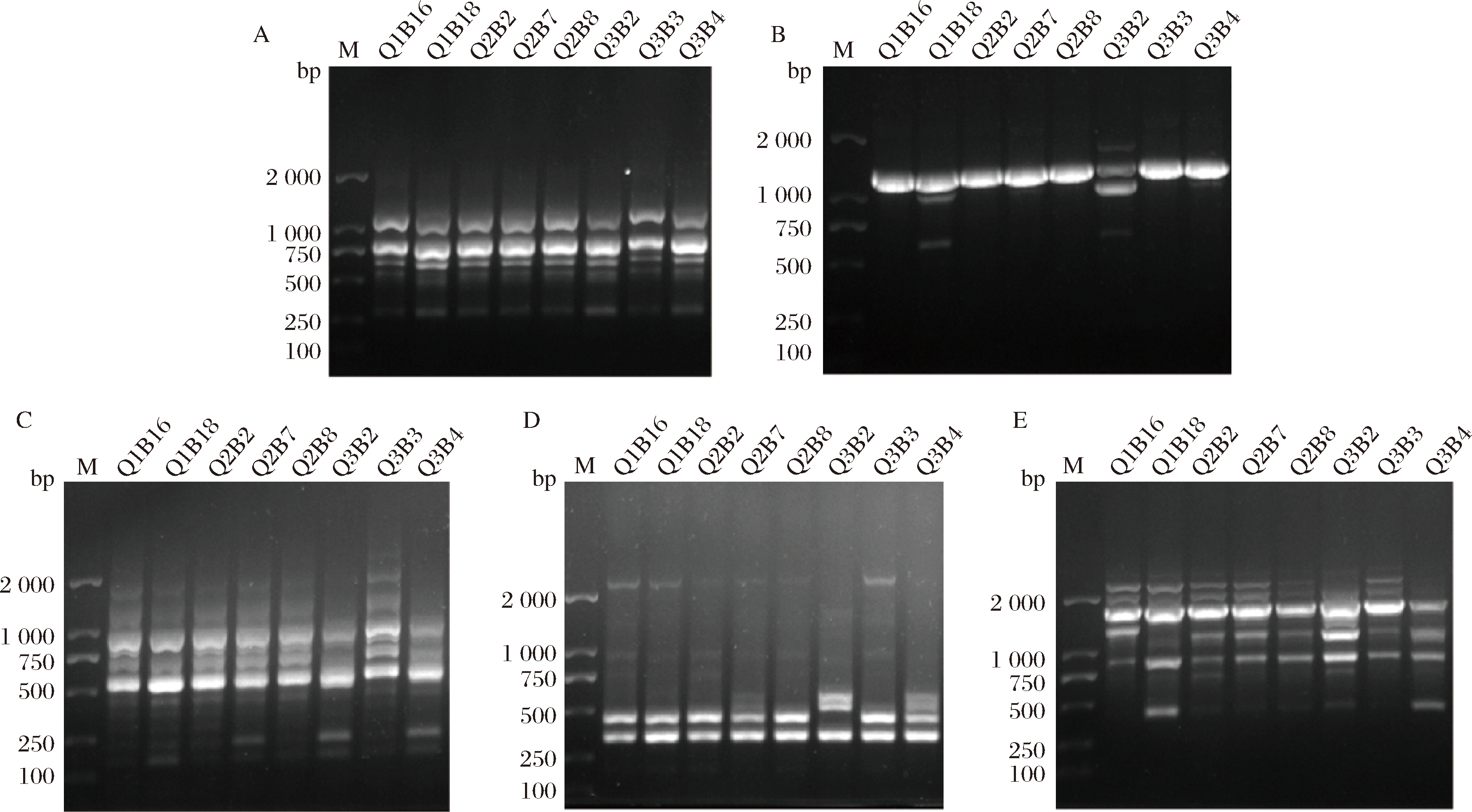
DNA Marker;A-OPA-02;B-OPA-18;C-OPL-07;D-OPL-16;E-OPM-05
图5 RAPD产物电泳图谱
Fig.5 Electropherogram of RAPD product
3 结论与讨论
大曲培养过程是酿酒过程中从自然环境中富集发酵菌株的主要途径,里面不仅有酵母菌,还有细菌、霉菌[17],但是随着制曲温度的逐步提高,酒曲中细菌发挥的作用却越来越重要,其所进行的生理生化代谢活动产生的物质和能量是制曲发酵的主要动力源泉,是形成大曲香味物质的关键[18-20]。
细菌作为酿酒过程中的一类重要微生物,对提高酿酒产量,增强风味具有重要作用,一直是科研工作者的研究热点。李永博等[21]以浓香型大曲为菌源,分离纯化出一株高产β-葡萄糖苷酶的菌株,为进一步开发该菌株奠定了一定的基础。TANG等[22]使用16S rRNA基因的V4高变区的高通量测序研究了茅台和国泰大曲的细菌多样性,发现虽然有些细菌的丰度不同,但这2种酱香型白酒大曲的细菌群落组成是一样的。罗小叶等[23]从茅台大曲中分离出了3株放线菌,对其进行形态学特征、生理生化实验及分子生物学鉴定,并对菌株的产酶、产香特性进行了研究,发现3株菌产果胶酶能力较为突出,其固态发酵挥发性成分以酯类物质为主,且均能产生吡嗪类物质。HE等[24]为研究强化大曲对浓香型白酒生产过程的影响利用Illumina MiSeq平台分析了3种酒醅(FG)接种模式中微生物群落的差异,发现在FG50(传统∶强化大曲=1∶1,w/w,50%强化大曲)中芽孢杆菌、乳球菌、曲霉和念珠菌的相对丰度较高,乳酸菌相对丰度较低,对制定有效的策略来调节酿造过程中的微生物并进一步改善中国白酒的风味有一定作用。
而对芝麻香型白酒的研究目前集中在菌落多样性及分离鉴定的研究。曹宇等采用磷脂脂肪酸分析方法对芝麻香型白酒高温大曲生产过程中微生物生物量和群落结构的动态变化进行了研究,结果表明,真菌在整个制曲过程中占主导地位,细菌的含量和种类在第1次和第2次翻曲的过程中出现规律性变化,推测这2次翻曲在制曲过程中起着关键作用[25]。YAO等[26]从芝麻型白酒高温大曲中分离出一种新型嗜热菌种Thermoactinomyces daqus H-18,并完成基因组测序。李斌等[27]基于Illumina Miseq测序方法研究了浓香型白酒中高温酒曲和芝麻香型白酒高温酒曲的微生物菌群结构,并对酒曲中的细菌种类和丰度进行分析,结果表明,芝麻香型白酒高温酒曲的优势类群主要有芽孢杆菌属(Bacillus)和别样芽胞杆菌属(Allobacillus),其中别样芽胞杆菌属(Allobacillus)是首次在酒曲中发现且丰度较高的属,其特性和功能尚不清晰,有待更加深入地研究。
本研究从本实验室提供的从芝麻香型白酒高温大曲中分离的细菌资源中筛选出产淀粉酶细菌5株;产蛋白酶细菌9株;既产淀粉酶又产蛋白酶细菌4株。采用分子生物学的鉴定技术确定了这10株菌归属于1个属,即芽孢杆菌属(Bacillus);2个种,即枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp.subtilis)和贝莱斯芽孢杆菌(Bacillus velezensis),为增强白酒风味、提高白酒产量提供了菌种资源。在此之前,ZHI等[28]曾从多种白酒大曲中分离得到枯草芽孢杆菌枯草亚种,并用于白酒生产过程中产生土臭味素的链霉菌属的生物防治研究;WANG等[29]曾在研究高温大曲是否对耐热微生物有选择性时也从高温大曲中分离出贝莱斯芽孢杆菌。此外,本研究对8株贝莱斯芽孢杆菌的分子分型结果表明其属于4个不同株型,表明芝麻香型白酒特殊风味的形成需要不同株系的贝莱斯芽孢杆菌共同起作用;且发现Q1B16、Q2B2、Q2B7、Q2B8、Q3B3为同一株型,表明其在制曲过程中具有纵向传递性,在芝麻香型白酒风味形成过程中可能起重要作用,本研究对芝麻香型白酒高温大曲产酶细菌进行分离与鉴定,为下一步研究对芝麻香型白酒高温大曲制曲过程中细菌群落的控制提供了理论基础。
[1] 高传强,阳飞,张华山.芝麻香型白酒微生物菌群及风味物质研究进展[J].微生物学通报,2017,44(4):940-948.
[2] 杜海,邢敏钰,徐岩.芝麻香型白酒酿造过程中乳酸菌分离及其碳源利用特征[J].食品与发酵工业,2018,44(1):13-18.
[3] 李登勇,黄钧,丁晓菲,等.酱香大曲间微生物群落结构时空特征的表征[J].食品工业科技,2018,39(23):139-145.
[4] LIU J,CHEN J,FAN Y,et al.Biochemical characterization and dominance of different hydrolases in different types of Daqu-a Chinese industrial fermentation starter[J].Journal of the Science of Food &Agriculture,2018,98(1):113-121.
[5] FAN G,FU Z,SUN B,et al.Roles of aging in the production of light-flavored Daqu[J].Journal of Bioscience and Bioengineering,2019,127(3):309-317.
[6] 刘洋,赵婷,姚粟,等.一株芝麻香型白酒高温大曲嗜热放线菌的分离与鉴定[J].生物技术通报,2012(10):210-216.
[7] 姚粟,葛媛媛,李辉,等.利用非培养技术研究芝麻香型白酒高温大曲的细菌群落多样性[J].食品与发酵工业,2012,38(6):1-6.
[8] 于学健,冯慧军,翟磊,等.芝麻香型白酒高温大曲中高温放线菌及其功能基因的动态变化规律[J/OL].食品与发酵工业,2019,DOI:10.13995/j.cnki.11-1802/ts.019959.
[9] YAO S,LIU Y,LI H,et al.Bacterial communities during the process of high-temperature Daqu production of roasted sesame-like flavour liquor[J].Journal of the Institute of Brewing,2015,121(3):440-448.
[10] WANG L T,LEE F L,TAI C J,et al.Comparison of gyrB gene sequences,16S rRNA gene sequences and DNA-DNA hybridization in the Bacillus subtilis group[J].Int J Syst Evol Microbiol,2007,57(8):1 846-1 850.
[11] CHUN J,BAE KS.Phylogenetic analysis of Bacillus subtilis and related taxa based on partial gyrA gene sequences[J].Antonie Van Leeuwenhoek,2000,78(2):123-127.
[12] WILLIAMS J G K,KUBELIK A R,LIVAK K J,et al.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J].Nucleic Acids Research,1990,18(22):6 531-6 535.
[13] CHUN J,LEE J H,JUNG Y,et al.EzTaxon:a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J].Int J Syst Evol Microbiol,2007,57(10):2 259-2 261.
[14] THOMPSON J D,HIGGINS D G,GIBSON T J CLUSTAL W.Improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Res,1994,22(22):4 673-4 680.
[15] KUMAR S,STECHER G,TAMURA K MEGA.Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1 870-1 874.
[16] 刘毅,姚粟,李辉,等.三种DNA指纹图谱技术在菌株分型中的应用[J].生物技术通报,2015,31(6):81-86.
[17] ZOU W,ZHAO C,LUO H.Diversity and function of microbial community in Chinese strong-flavor baijiu ecosystem:A Review[J].Front Microbiol,2018,9:671.doi:10.3389/fmicb.2018.00671.
[18] ZHENG X W,YAN Z,NOUTT M J,et al.Microbiota dynamics related to environmental conditions during the fermentative production of Fen-Daqu,a Chinese industrial fermentation starter[J].Int J Food Microbiol,2014,182-183:57-62.
[19] DU H,WANG X,ZHANG Y,XU Y.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].Int J Food Microbiol,2019,297:32-40.
[20] XIAO C,LU ZM,ZhANG XJ,et al.Bio-Heat is a key environmental driver shaping the microbial community of medium-temperature Daqu[J].Applied and Environmental Microbiology,2017,83(23):e01 550-e01 517.
[21] 李永博,李星,唐敏,等.浓香型大曲中产β-葡萄糖苷酶微生物的筛选鉴定及其产酶条件优化[J].食品科技,2017,42(12):17-22.
[22] TANG J,TANG X,TANG M,et al.Analysis of the bacterial communities in two liquors of soy sauce aroma as revealed by high-throughput sequencing of the 16S rRNA V4 hypervariable region[J].Biomed Res Int,2017(2):1-9.
[23] 罗小叶,王晓丹,邱树毅.酱香大曲中3株放线菌的分离筛选及挥发性成分分析[J].中国酿造,2018,37(8):62-67.
[24] HE G,HUANG J,ZHOU R,et al.Effect of fortified Daqu on the microbial community and flavor in Chinese strong-flavor liquor brewing process[J].Front Microbiol,2019,10:56.
[25] 曹宇,翟磊,信春晖,等.芝麻香型白酒高温大曲制曲过程中微生物群落结构特征的磷脂脂肪酸(PLFA)分析[J].酿酒科技,2016(3):33-36;41.
[26] YAO S,XU Y,XIN C,et al.Genome sequence of Thermoactinomyces daqus H-18,a novel thermophilic species isolated from high-temperature Daqu[J].Genome Announc,2015,3(1):e01 394-e01 414.
[27] 李斌,闫志鹏,李慧星,等.基于高通量测序技术的浓香型和芝麻香型白酒酒曲细菌群落结构分析[J].中国酿造,2018,37(8):148-152.
[28] ZHI Y,WU Q,DU H,et al.Biocontrol of geosmin-producing Streptomyces spp.by two Bacillus strains from Chinese liquor[J].International Journal of Food Microbiology,2016,231(9):1-9.
[29] WANG Z,LI P,LUO L,et al.Daqu fermentation selects for heat-resistant Enterobacteriaceae and Bacilli[J].Applied and Environmental Microbiology,2018,84(21):e01 483-e01 518.