氯霉素(chloramphenicol,CAP)是一种广谱抗生素,又名左旋霉素,是一种由链霉菌产生并对多种有氧和厌氧微生物具有活性抑制作用的抗生素,分子式为C11H12Cl2Na2O3,其化学结构如图1所示。自1949年被引入临床实践,CAP就由于其廉价和高效的特点,被广泛用作治疗各种传染病的兽药[1]。然而,随着对CAP研究的不断深入,科学家们发现其苯环上含有的硝基半衰期时间较长,在动物性食品中的残留可通过食物链进行富集,对人体有较强的毒性,长期积累会使细菌产生强的耐药性,甚至导致白血病,再生障碍性贫血和灰色婴儿综合症等疾病发生[2]。由于其具有种种危害人类健康的毒副作用,美国、澳大利亚、日本以及中国等许多国家都禁止在动物食品中使用CAP。欧盟(European Union,EU)规定在海鲜、牛奶、肉等动物性食品中CAP的最高残留限量(maximum residue limits,MRL)为0.3 μg/kg[3],我国农业农村部在2002年第235号公告中也明文规定将氯霉素及其盐、酯类列入禁止使用药物,并在所有动物性食品的靶组织中不得检出[4]。为满足市场监管需要,相关行业不断研究发展快速、准确和便捷检测食品中CAP残留的高新技术,为规范市场行为和保障食品安全建立了一道重要防线。
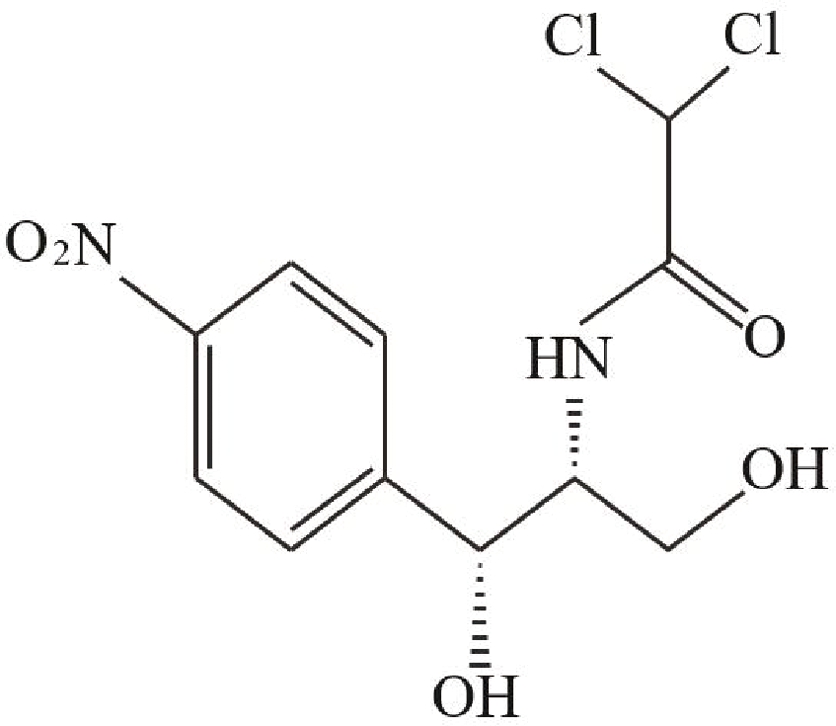
图1 氯霉素分子结构式
Fig.1 Molecule structure of chloramphenicol
近年来对CAP残留检测主要包括仪器分析法和免疫分析法两大类主流检测方法。仪器检测方法拥有高通量、灵敏、准确的分析检测能力,在大型分析检测机构有着较为广泛的应用,如气相色谱法(GC)[5]、高效液相色谱法(HPLC)[6]、气相色谱-质谱联用法(GC-MS)[7]、液质联用法(HPLC-MS)[8]等,但仪器操作专业度高,前处理过程复杂耗时,且后期设备维护成本较高,难以推广到市场中进行现场检测,因而对于大批量样品的快速高通量检测具有一定的局限性[9]。免疫分析方法,如酶联免疫吸附测定法(enzyme linked immunosorbent assay, ELISA)[10]、化学发光免疫分析法(chemiluminescence immunoassay, CLIA)[11]、放射免疫测定法(radio immunoassay, RIA)[12]、荧光免疫分析法(fluorescent immunoassay, FIA)[13]和胶体金免疫层析法(gold immunochromatography assay, GICA)[14]等,操作简便、反应快速,灵敏度高、准确度高,能满足大多数检测需求,因此发展快速,应用广泛。然而传统的免疫分析方法均是基于抗原-抗体特异性识别反应建立起来的,其原料抗体生产成本高、周期长,免疫活性易受离子强度、pH等环境因素影响,标记修饰复杂且难以定向标记。核酸适配体作为一种新型识别元件,合成简单快速、成本低、亲和力高、性质稳定且易于修饰标记[16-17],是一种优良的抗体替代识别元件。因此,基于核酸适配体的分析检测研究在近些年得到了不断发展。本文对核酸适配体进行了简要介绍,并综述了近5年基于该生物识别元件检测动物性食品中氯霉素残留的分析方法,以期为动物性食品中氯霉素残留的有效监管和检测技术的深入研究提供有价值的参考。
1 核酸适配体
核酸适配体(aptamer,Apt)是通过体外配体指数级富集系统进化技术(systematic evolution of ligands by exponential enrichment, SELEX)从人工构建的寡核苷酸文库中筛选出来的一种对目标靶标物质具有高度亲和力的单链寡核苷酸序列[17],一般由25~80个核苷酸碱基组成,既可以是RNA也可以是单链DNA,其功能与抗体相似,不但能与多种目标物进行高特异性、高灵敏度地结合,并且还具有许多优于抗体的优点,例如核酸适配体是通过体外合成,不需要依赖动物产生受体,批次差异小,生产周期短,并且核酸稳定性好,适用范围更广,能够特异性结合蛋白质、细胞、药物以及金属离子等,因此常常被视为更有前途的“化学抗体”[18]。凭借优越的性能,适配体已作为一种新的生物识别元件被广泛应用于分析化学、食品安全、分子识别和药物残留检测等领域的研究[19-21]。我们发现在近5年结合核酸适配体检测CAP的相关报道中,不同的实验所设计的适配体序列虽不尽相同,但通过仔细比对发现大部分核酸适配体的完整序列中都包含“5′-ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTA G-3′”这一共同序列,所以推测这就是高特异性识别目标分子CAP的适配体核心序列,并且亲和性能被大家所普遍认同。
1.1 核酸适配体与靶标物结合模式
核酸适配体对靶标物的亲和性类似于抗体抗原的特异性免疫识别,其结合模式可划分为两大类,即单站点结合和双站点结合模式[23]。如图2所示,单站点结合主要是针对小分子靶标物,当核酸适配体与小分子物质结合后自身构象会发生一定的改变,核磁共振(nuclear magnetic resonance, NMR)研究证实了小分子通常被结合于适配体自身的折叠区域[24],这种结合模式也是被运用最多的。双站点的结合模式则常常被设计用于结合蛋白质等大分子物质,与小分子相比,类似于蛋白质等大分子靶标物在结构上往往更加复杂,但是这些复杂结构为堆叠、结构互补、氢键结合等结合方式提供了更多的可能,使有多个结合位点大分子常被设计成“三明治”夹心法检测模式。这两种结合模式的探明为后续研究的实验方案设计提供了强有力的理论支撑。

图2 核酸适配体结合模式[23]
Fig.2 Aptamer-based assay formats
2 检测方法
近年来应用核酸适配体检测氯霉素残留的研究有大量报道,主要有比色检测法、荧光检测法、化学发光检测法、表面增强拉曼散射检测法和生物传感器检测法等,表1为相关检测方法的文献简要概括对比。研究者们通过对核酸适配体的巧妙设计,同时结合纳米金颗粒(AuNPs)、磁性纳米粒子、荧光素和量子点(quantum dots, QDs)等各种类型的新型纳米标记材料,使动物性食品中氯霉素残留的检测方式多样化,并且朝着快速简便、高灵敏度、高准确度和高通量的方向不断发展,表2为相关新型纳米标记材料特性的简要概括总结。
2.1 比色分析法
比色法是指基于有色待测物质颜色的深浅与组分含量在一定范围内成比例的原理,通过比较或测量物质溶液的颜色深浅来确定含量,早在20世纪40年代就被应用于定量分析,是一种常见的分析方法[25-26]。基于核酸适配体检测氯霉素残留的比色法成本低,响应快速,灵敏度及精密度高[27],有一定的发展前景。
在传统比色法检测中,ELISA方法利用辣根过氧化物酶(HRP)催化H2O2和3,3′,5,5′-四甲基联苯胺(TMB)反应产生颜色信号,且待测物浓度与颜色深浅相关。该方法所需的仪器设备简单,适合批量检测,然而这类非均相反应需要多次洗涤孵育,操作繁琐耗时。类似于ELISA反应,YAN等[28]开发了一种基于核酸适配体的比色法用于检测动物性食品中CAP残留,运用经典的生物素-链霉亲和素信号级联放大系统,既可通过目测颜色变化定性确定样品中是否存在CAP超标残留,也可进一步使用酶标仪测定吸光度达到定量测定的目的,在蜂蜜及鱼肉中的检测限达到了0.003 1 μg/kg,添加回收率为96.2%~109.0%,该方法灵敏度高且准确度高,表现出了适配体对CAP优异的识别能力。此外,新型纳米材料的开发为构建比色法检测技术提供了全新的思路和平台,同样是催化TMB显色,HUANG等[29]将核酸适配体特异性识别结合的特性与DNAzyme功能化纳米探针催化显色的原理相结合,建立了一种新型比色生物传感器用于快速检测CAP残留,将过氧化氢酶模拟物及适配体互补探针cDNA与纳米金结合,构建新的纳米金探针,过氧化氢酶模拟物能催化H2O2使TMB的氧化并产生比色信号,该方法中新型纳米材料的引入使检测信号大大增强,奶粉样品中添加回收率在92.0%~104.0%,相对标准偏差(RSD)<2.7%,表明了基于核酸适配体的比色法对氯霉素的检测具有较强的检测能力,但不足之处是这种四联体DNAzyme制备纳米探针过程复杂,在大批量检测效率上应用相对受限。
表1 核酸适配体检测氯霉素残留的检测方法
Table 1 The methods of chloramphenicol residue detection via aptamers

检测方法检测样本适配体序列5’-3’检测策略检测限线性范围参考文献比色法蜂蜜、鱼5'- CAT TCC TTT TAC ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCGGTG GTA G-Biotin-3'HRP-H2O2-TMB0.003 1 μg/L0.001-1000 μg/L[28]比色法奶粉5'-Biotin-ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTA G-3'DNAzyme催化0.13 ng/L0.001-100 μg/L[29]比色法牛奶5'-ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTA G-3'盐诱导AuNPs聚集0.032 nmol/L0.1-10 nM[31]荧光法牛奶5'-CAATAAGCGATGCGCCCTCGCCTGGGGGCCTAGTCCTCTCCTATGCGTGCTACCGTGAA-3'PCR0.10 μg/L0.1-20 μg/L[2]荧光法牛奶5'-NH2-C6-AGCAGCACAGAGGTCAGATGCACTCGGAC-CCCATTCTCCTTCCATCCCTCATCCGTCCACCCTATGCGTGCTACCGTGAA-3'FRET0.03 μg/L0.1-10 nM[35]荧光法鱼5'-(CH 2)6 -ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTAG-3'FRET0.002 μg/L0.001-10 μg/L[36]荧光法牛奶5'-(CH 2)6 -ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTAG-3'FRET0.0003 μg/L0.001-10 μg/L[37]荧光法牛奶5'-Biotin-AGCAGCACAGAGGTCAGATGACTTCAGT-GAGTTGTCCCACGGTCGGCGAGTCGGTGGTAGCCTATGCGTGCTACCGTGAA-3'UCNPs0.01 μg/L0.01-1.0 μg/L[38]化学发光法牛奶5'-Biotin-TTTTTAGCAGCACAGAGGTCAGATGACT-TCAGTGAGTTGTCCCACGGTCGGCGAGTCGGTAGCCTATGCGTGCTACCGTGAA-3'ABEI-H2O2-PIP0.01 μg/L0.01-0.20 μg/L[39]拉曼光谱法乳粉5'-ACTTCAGTGAGTTGTCCCACGGTCGGCGAG TCGGTGGTAG-Cy5 3'AuNPs0.19 ng/L0.001-1.0 μg/L[41]电化学法牛奶5'-NH2-(CH2)6-ACTTCAGTGAGTTGTCCCACGGTCGGCGAGTCGGTGGTAG-3'NMOF0.19 pmol/L 0.002-100 nM[43]电化学法鱼肉5'-ACTTCAGTGAGTTGTCCCACGGTCGGCGAGTCGGTGGTAG-COOH-3'cDNA竞争0.07 nmol/L0.2-150 nM[44]微流控电泳法牛奶、鱼5'-ACT TCA GTG AGT TGT CCC ACG GTC GGC GAG TCG GTG GTAG-3'cDNA竞争0.003 μg/L0.008-1.0 ng/mL[45]微流控电泳法牛奶、鱼5'- CATCTCTTCTCCGAGCCATCTCTTCTCCGAGCCGGTCGAAATAGCC ATAGTGGGTG GCCTAACTACCACCGA -3'Microchip阵列0.006 nmol/L0.006-10 nM[46]
近年来关于纳米金的比色研究在分析检测领域十分热门,金纳米粒子高消光系数和良好的光学效应使得适配体和金纳米粒子的组合成为比色生物测定中成为最强大的生物传感之一[30]。核酸适配体的单链DNA(ssDNA)上的N原子与金原子的静电吸附作用可以使其结合在纳米金上,AuNPs在分散状态下溶液为酒红色,当加入盐离子后会引起AuNPs聚集从而呈现蓝紫色。根据AuNPs在不同状态下显色不同的原理,JAVIDI等[31]设计了一个基于Watson-Crick碱基配对的适配体终端锁(ATL)比色法来检测CAP,具有环状结构的ATL由完整的适配体序列和用作锁定探针(LP)的短序列寡核苷酸共同组成,当待测物中存在CAP时,特异性适配体识别CAP形成复合物,LP片段从ATL中解离吸附在纳米金颗粒上,避免了加入盐离子而引起的聚集,溶液依然保持透亮的酒红色,反之盐离子诱导纳米金聚集而呈现蓝紫色,奶粉样品中在0.1~10.0 nmol/LCAP与相对响应信号有良好的线性关系,最低检测限为0.03 nmol/L。该方法的最大优势在于无需对适配体以及其他分子进行任何修饰,检测所需设备少,步骤简单,通过紫外可见光光谱即可定量检测,弊端是样品的前处理要求较高,否则基质会影响AuNPs的聚集从而造成检测结果不准确。
2.2 荧光分析法
荧光分析法是基于一些荧光物质能够在一定的激发波长下发出荧光的原理,通过检测荧光的强弱来进行定性定量分析的一种方法,具有高灵敏度、低背景值和选择性好等明显的优点,在生命医学,化学等各种检测领域应用广泛[32-33]。核酸适配体新技术与荧光分析法的结合为氯霉素等抗生素残留检测提供了新思路,同时新型纳米荧光材料的应用也大大提升了荧光分析法的检测能力。核酸适配体具有易被化学键修饰的特点,因此常常对核酸适配体或其互补链cDNA末端进行荧光标记,对CAP的检测根据核酸适配体的标记与否可以划分为荧光标记型检测法和荧光非标记型检测法[34]。
表2 新型纳米标记材料主要特性
Table 2 Main features of new marking nanomaterials

新型标记物材料荧光寿命/ns优点缺点纳米金胶体金颗粒比表面积大,易修饰,易制备,高消光系数[30-31]磁性纳米颗粒金属氧化物,介孔Fe3O4材料等稳定性好,形状可控,生物兼容性好,易修饰[37-38]磁性粒子提取过程复杂荧光素荧光前提物质,荧光化合物1~10种类多,价格低廉,工艺成熟[41]荧光稳定性差,荧光寿命短,Stokes位移小,易光漂白,易受背景荧光物质的干扰量子点半导体纳米材料,Ⅱ-Ⅵ、Ⅲ-Ⅴ、Ⅳ-Ⅵ族元素,CdTe等10~103激发波长宽,可实现单激发、多波长辐射,通过控制尺寸可控制发射波长,用于多组分检测[35-37]易发生荧光猝灭[36]上转换纳米粒子掺杂稀土离子的化合物,NaYF4等104~105逆Stokes发光,检测的背景值低;信噪比大[38]制备成本高;应用少
2.2.1 荧光标记型检测法
荧光标记的捕获探针与目标物结合能够显著性地改变体系中的荧光信号强度,且待测物浓度与信号强度相关,通过检测反应前后体系荧光信号的变化即可测算出待测物浓度。目前常应用于标记核酸适配体的荧光物质有量子点、荧光染料、上转换材料以及贵金属纳米簇等。
量子点(QDs)又称半导体纳米粒子,是一种准零维纳米材料,其作为新型的纳米材料凭借量子产率高,发射光谱窄、激发光谱宽,抗光漂白以及可测量的荧光强度等优点,被广泛应用作荧光标记材料。ALIBOLANDI等[35]以量子点作为能量供体,氧化石墨烯(GO)作为能量受体,构建了一个基于适配体的QDs-GO荧光共振能量转移(fluorescence resonance energy transfer, FRET)平台用于检测牛奶样品中的氯霉素,反应中荧光猝灭效率超过90%,检测限为0.03 μg/L,检测范围为0.1~10 nmol/L,该方法与CAP类似物无交叉反应,准确度高,达到了良好的检测效果,最大缺点在于GO的制备及纯化耗时约1周,实验周期长。由于FRET检测模式具有高灵敏度、高效率的优势,因此近几年在荧光检测法中的研究十分热门,MIAO等[36]将CdSe量子点作为能量供体,AuNPs探针作为受体,建立了一种基于FRET的适配体检测方法用于检测氯霉素,在鱼肉的实际样品中检测限为0.002 μg/L,线性范围为0.001~10 μg/L,与传统的ELISA相比,该方法不但检测步骤少,而且更加省时,从添加样品到测出荧光时间不足10 min,不足之处是探针的制备技术难度大,成本较高。在后面的进一步研究中,MIAO等[37]又创新性地开发出了一种脂质体囊泡探针用于氯霉素FRET检测。如图3所示,脂质体囊泡由双磷脂分子层结构的两端亲水基团和中间疏水基团构成,包裹的大量QDs可以放大探针信号,在牛奶样品中检测限为0.000 3 μg/L,线性范围为0.001~10 μg/L,添加回收率均在90%~103%。这种均相检测荧光“开关”模式操作简便,结合荧光分析仪即可测定,并且较未改进的FRET模式检测灵敏度提高了近7倍,是目前基于适配体荧光法检测CAP残留的所有报道中检测限最低的方法。
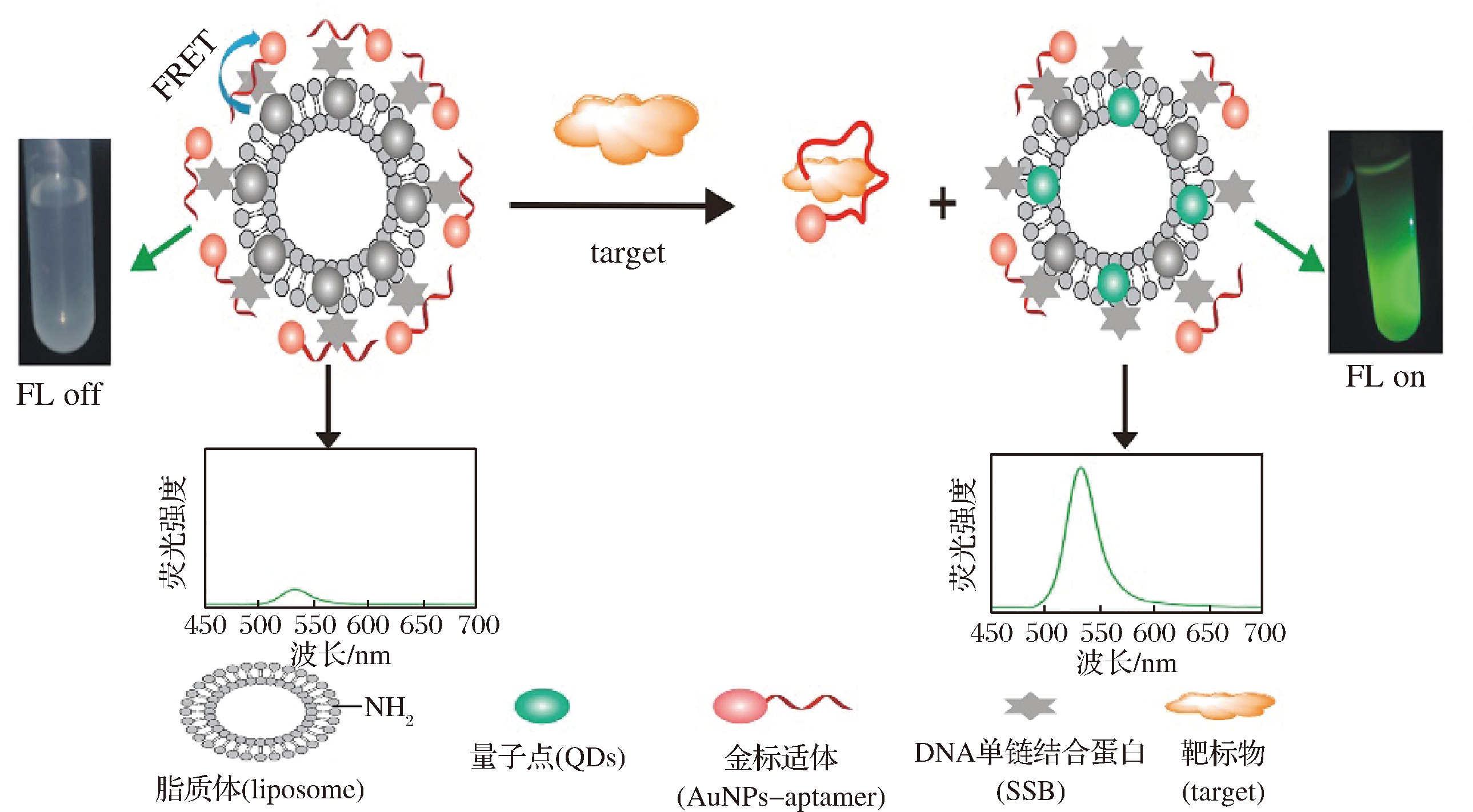
图3 基于核酸适配体的荧光共振能量转移检测氯霉素[37]
Fig.3 FRET detection based on aptamer for chloramphenicol
上转换纳米粒子(upconversion nanoparticles, UCNPs)是一种优秀的生物材料,具有检测背景值低,抗光漂白,低毒性,寿命长,大的逆斯托克斯(Stokes)位移,改变组分比例还可实现多重检测等特点,常被用作荧光标记材料。WU等[38]建立了一个上转换纳米粒子的适配体检测平台,将连接有CAP适配体的磁性纳米粒子(magnetic nanoparticles,MNPs)作为捕获探针,连接有cDNA上转换纳米粒子作为信号探针,捕获探针与靶标物结合后引起cDNA解离并释放上转换纳米粒子,导致MNPs表面荧光信号减弱,在牛奶样品中检测限达到0.01 ng/mL,添加回收率为93.67%~101.41%。该方法原理简单,灵敏度高,由于使用980 nm激光激发,可以完全避免源自溶液当中生物分子的自发荧光,同时这种检测方法具有一定的通用性,基于该原理通过替换合适的适配体,可以尝试用于检测其他抗生素。但样品的制备涉及2个标记过程,需要人工设计,具有一定的难度,并且标记的批间差异性也难以保证。
2.2.2 荧光非标记型检测法
同荧光非标记型检测法相比,标记型的检测方法的检测灵敏度更高,但是需要对探针进行化学合成标记,过程繁琐耗时,非标记型检测模式则相对更加简便,无需使用化学试剂进行复杂的标记及最后的分离纯化。DUAN等[2]首次报道了使用实时荧光定量PCR高灵敏度检测食品中的氯霉素残留,设计的方法中CAP适配体首先与生物素化的cDNA进行互补配对,cDNA通过生物素-链霉亲和素结合系统固定在缀合有亲和素蛋白的MNPs上,当存在CAP时,适配体与CAP特异性结合形成发夹结构,并从MNPs上释放用于实时荧光定量PCR(qRT-PCR)检测。该方法优化后能对真实牛奶样品中CAP残留的进行高灵敏度检测,检出限为0.1 ng/mL,线性范围为0.1~20.0 ng/mL,添加回收率为94.0%~102.0%,并且对氯霉素类似物甲砜霉素(TAP)和氟苯尼考(FF)显示出了较高的选择性。这种非标记荧光检测方法中PCR技术灵敏度高,但是操作过程复杂,对实验条件有一定的要求,因此在现场的快速检测场景中有一定的局限性,但依然提供了一个新的检测思路与方法。
2.3 化学发光检测法
化学发光(chemiluminescence,CL),指由发光物质自身电子从激发态跃迁到基态所产生的发光,是不需要使用外部光源或光学系统的化学反应,与荧光分析法相比可以有效避免光漂白效应,其检测信号是由光子数决定的,具有灵敏度高,仪器简单,校准范围宽,操作简便,检测时间短等优点,因此在分析检测领域中占有重要地位。HAO等[39]开发了一种基于化学发光的适配体检测方法,将连有生物素化CAP适配体的MNPs作为功能化的捕获探针,连有乙基异鲁米诺(ABEI)的花状金纳米结构(AuNF)作为信号探针,结合对碘苯酚(PIP)共同构建了一个稳定的ABEI-H2O2-PIP化学发光系统;该方法基于竞争的形式实现了对CAP的高灵敏度检测,牛奶样品中检测限为0.01 ng/mL,线性范围为0.01~0.20 μg/L。与于秀霞[40]使用HRP催化鲁米诺的化学发光的方法相比,基于核酸适配体的化学发光分析法中AuNF灵敏度高,适配体和磁分离稳定性较好,测定重现性好,是更具有前景的小分子药物残留的检测方法,但制备修饰的ABEI-AuNF信号探针相当耗时,相比之下总体优势不够突出,同时该方法也是近5年基于化学发光检测仅有的一篇报道。
2.4 表面增强拉曼散射检测法
表面增强拉曼散射(surface-enhanced Raman scattering, SERS)是一种表面敏感技术,具有快速响应,超高灵敏度,操作简单,能同时进行多重检测等特点,是超灵敏检测复杂基质中特定分析物的最有效的技术之一,已广泛应用于食品安全、生物诊断、医学和化学等领域。YAN等[41]通过使用银材料封装的内层金,制成了一种金核银壳纳米结构材料用于SERS检测CAP,将荧光染料Cy5修饰的适配体与纳米金粒子通过共价键连接作为分子识别探针,当存在CAP时,适配体结合CAP形成茎环结构并引起解离,随着Cy5-适配体与金核表面分离,SERS信号强度显著降低。与在纳米颗粒外层附着染料的其他SERS传感器相比,该方法表现出的灵敏度更高,信号稳定性也更好,实际的加标乳样品中,检测限为0.19 ng/L,回收率为96.6%~110.2%,变异系数为1.8%~4.9%,具有对CAP残留的高灵敏度和检测的能力。与免疫分析方法相比,YANG等[42]开发出了1种竞争性免疫分析和磁分离的SERS生物传感器用于精确和灵敏地检测氯霉素,这种SERS生物传感器由于磁分离技术的富集作用对氯霉素检测信号具有很好放大作用,检测限达到10 ng/L,线性范围0.001~10 μg/L,但结果表明,免疫分析方法检测限低于适配体检测方法。总而言之,结合适配体的SERS平台适用性广泛,检测效果好,通过适配体稍作变更即可以实现对其他类型的抗生素残留的检测,但遗憾的是,探针制备复杂以及昂贵的检测成本限制了其应用推广。
2.5 电化学适配体生物传感器检测法
电化学适配体生物传感器是基于核酸适配体材料建立并利用电信号作为分析和检测信号的新型生物传感检测装置,具有灵敏度高,响应速度快等优点。CHEN等[43]首次基于纳米金属有机骨架(NMOF)建立了一种新型电化学适配体传感器同时检测包括氯霉素在内的多种抗生素;使用NMOF包封金属离子并固定cDNA作为信号标签,CAP与cDNA竞争性地结合适配体,CAP靶浓度与测定的电流信号成比例,该方法的检测限低至0.19 pmol/L,同时线性范围为0.002~100 nmol/L,达到了5个数量级,对CAP的检测性能非常优秀,并且通过改变相应的适配体还能检测其他靶标物,但电化学生物传感器需要对电极进行改性设计,可能会影响生物活性分子的构型从而降低识别能力,检测性能的稳定性也难以保证。电化学发光(electro chemiluminescence,ECL)同时具有电化学和化学发光2种方法的优点,与化学发光方法相比可以实现更精确的分析物测定。FENG等[44]建立的一种“双电势”的新型电化学发光适配体传感器可同时检测孔雀石绿(MG)和氯霉素,将与适配体互补的cDNA同鲁米诺-纳米金粒子连接并修饰到阳极上作为发射体,使电化学发光信号的增强取决于目标物MG和CAP的浓度,在实际鱼类样品的检测中,检测限分别为0.03 nmol/L和0.07 nmol/L。该方法反应迅速,从添加样品到读出结果仅需要1 min左右即可完成,精确度和重现性均表现良好,并且具有多重检测的能力,但实验中的电极修饰难度较大,对实验的条件要求苛刻,限制了大规模的推广应用。
2.6 微流控电泳法
微流控电泳法作为一种快速筛选和高通量检测的方法,已成功作为高通量DNA或ssRNA的分离系统,可根据不同序列的碱基对数不同的原理来检测ssDNA和dsDNA。核酸适配体本质也是ssDNA或RNA,基于此原理,ZHOU等[45]开发了一种新型无标记的适配体微流控电泳平台用于自动检测抗生素残留,利用靶标物CAP和cDNA同时竞争结合适配体,电泳平台根据不同比例的dsDNA和Apt-CAP产生不同的信号比率,实现了对CAP残留的精确检测,在实际的鱼肉以及牛奶的样品中,检出限达到0.003 μg/mL,并且回收率良好。ZHOU等[46]后来又进一步在此模型上采用PCR信号放大原理进行同时测定卡那霉素和CAP,将CAP检测的灵敏度再次提高了将近1倍。该方法在抗生素残留的检测方面具有非常大的优势,首先作为电泳方法,适配体探针不需要信号源的标记,其次整个检测过程可以在3 min之内完成,具备高通量检测的能力,并且需要的样品体积小,也适宜于痕量检测,但样品在检测前需离心、真空干燥以及氮吹处理,因此基于核酸适配体的微流控电泳法在实验室层面的CAP残留检测乃至其他种类的抗生素筛选均具有较大的应用潜力,但不适合现场的快速检测。
2.7 侧流层析试纸条法
可视化的层析试纸条法是一种比较常见的低成本并且无需专业技能即可上手操作的快捷检测方法。在近些年来得到了飞速的发展,已经成为食品检测乃至医药研究领域的热点方向[47]。传统的基于抗原抗体免疫分析试纸条技术已经在很多方面进行了商业化的生产,但抗体本身具有一些局限性,比如批次的不均一性,无免疫活性或具有毒性的物质难以去制备抗体等,使得利用核酸适配体代替抗体去设计测流层析试纸条成为了一个新的突破口。赵帅[47]建立了一种基于核酸适配体的侧流层析试纸条法用于快速检测氯霉素的残留,试纸条的样品垫上附着有适配体标记的纳米金,T线和C线分别通过生物素-链霉亲和素系统固定有不同序列的cDNA,连接有纳米金的适配体会先与靶标物CAP结合,此方法选择性良好,当存在与CAP结构类似物氟苯尼考和甲砜霉素时,也不会产生信号的变化,借助荧光检测读数仪能够定量准确检测出CAP含量,在加标样品的检测中检测限为0.06 μg/mL,然而BERLINA等[48]基于量子点并结合传统的抗原抗体免疫层析法对牛奶样品中的CAP检测限达到了0.2 ng/mL,相比之下提高灵敏度则是目前亟待解决的问题。基于核酸适配体的侧流层析法是一个快速定性定量检测CAP残留的好途径,但试纸条本身不稳定,不能够永远维持像实验室这样一个良好的检测环境,容易受到外界环境如温度、湿度等因素的影响,因此具有许多不可控的因素而最终影响检测的灵敏度和准确性,这是在以后开发类似方法中需要注意并加以解决的地方。
3 展望
氯霉素等兽药超标残留严重威胁着人们的生命健康,同时也影响我国动物源性食品对国际市场的外贸出口,因此发展简单快速并且高灵敏度的检测方法对保障食品安全和相关产业体系的良性发展具有重大的战略意义。
核酸适配体作为新型识别元件具有许多独特的优势,它可以通过化学合成进行大量低成本生产,性质稳定,并易于进行官能团修饰以用作标记或固定;可以与新型功能化的纳米材料如金属纳米粒子、磁性纳米材料、上转换纳米材料、荧光染料以及量子点等结合使用提高检测能力;与新的检测策略结合应用如FRET等在近几年的分析检测领域中更是炙手可热。这些优势使得适配体的研究应用极富价值,然而抗体所构建的检测体系仍是商业化应用的主流,核酸适配体的实际应用依然存在许多的不足与挑战,例如临床或环境样品基质中通常含有多种干扰物质,其中有些会强烈地阻碍靶标与适配体之间的特异性识别,生物样品中的某些成分也会导致适配体自身折叠从而无法进行准确检测[49];因此在核酸适配体检测真正商业化应用之前,这些问题都需要去一一解决。未来关于核酸适配体检测氯霉素的创新首先可能集中在提高灵敏度、选择性以及样品通量和多重检测系统的设计上;其次适配体与检测能力优异的电化学生物传感器相结合并解决电极改性问题,将会使其在实际应用中更进一步[50];最后,目前大多数报道的适配体都是对目标物进行体外检测,设计适当的传感器能在复杂基质中或体内定性定量检测也将是一个重点的突破方向。
更好的检测方法往往是基于现有的基础上去精进并不断探索出来的。随着科学技术的进步,新的检测策略、新型标记材料的研究不断深入,简便化、高通量、多残留和高精度的检测体系正日益完善,尽管仍有一些技术瓶颈需要去突破,但作为适用性更强的分子探针,相信核酸适配体在氯霉素等兽药残留检测领域的研究应用将会更加深入,未来在健康、环境和食品质量方面的应用也将潜力巨大。
[1] EFFIONG G S, EBONG P E, EYONG E U, et al. Amelioration of chloramphenicol induced toxicity in rats by coconut water[J]. Journal of Applied Sciences Research, 2010,6(4):331-335.
[2] DUAN Y, WANG L, GAO Z, et al. An aptamer-based effective method for highly sensitive detection of chloramphenicol residues in animal-sourced food using real-time fluorescent quantitative PCR[J]. Talanta, 2017, 165:671-676.
[3] 陶晓奇. 动物性食品中酰胺醇类残留化学发光检测技术研究[D]. 北京:中国农业大学, 2014.
[4] 中华人民共和国农业部. 农业部第235 号公告 动物性食品中兽药最高残留限量[S]. 北京:中国标准出版社,2002.
[5] 符靖雯, 黄子敬,陈孟君,等. 气相色谱-电子捕获检测器快速测定水产品中多种农药及兽药残留[J]. 理化检验(化学分册), 2018, 54(9):29-33.
[6] 肖婉娜, 何敏,陈怡佳. 高效液相色谱法检测蜂蜜中的氯霉素[J]. 农产品加工, 2018(6):56-58.
[7] LIU T, XIE J, ZHAO J, et al. Magnetic chitosan nanocomposite used as cleanup material to detect chloramphenicol in milk by GC-MS[J]. Food Analytical Methods, 2014, 7(4):814-819.
[8] 张燕, 徐幸,舒平, 等. 高效液相色谱-质谱联用法测定乳及乳制品中氯霉素类药物残留量[J]. 食品安全质量检测学报, 2015,6(2):710-717.
[9] 王安伟, 刘天密,覃锐, 等. 水产品中氯霉素残留检测方法研究进展[J]. 食品安全质量检测学报, 2017,8(11):179-184.
[10] SARWER UG, RONY M M H,SHARMIN M S S, et.al. ELISA validation and determination of cut-off level for chloramphenicol (CAP) residues in shrimp and fish[J]. Our Nature,2017,15(1-2):13-18.
[11] TAO X, JIANG H, ZHU J, et al. Detection of ultratrace chloramphenicol residues in milk and chicken muscle samples using a chemiluminescent ELISA[J]. Analytical Letters, 2012, 45(10):1 254-1 263.
[12] 吴丽媛. 动物源性食品氯霉素类残留检测方法概述[J]. 中国畜禽种业, 2014, 10(4):43-44.
[13] ZHANG Z, LIU J F, YAO Y, et al. A competitive dual-label time-resolved fluoroimmunoassay for the simultaneous determination of chloramphenicol and ractopamine in swine tissue[J]. Chinese Science Bulletin, 2011, 56(15): 1 543-1 547.
[14] 刘文珍, 王振国,田飞焱, 等. 胶体金免疫层析法快速检测水产品中氯霉素的应用与验证[J]. 江西水产科技, 2016(5):23-27.
[15] 段烨. 氯霉素核酸适体的筛选及基于核酸适体生物传感器的建立[D]. 北京:北京化工大学, 2016.
[16] DUAN Y, GAO Z, WANG L, et al. Selection and identification of chloramphenicol-specific DNA aptamers by Mag-SELEX[J]. Applied Biochemistry and Biotechnology, 2016, 180(8):1 644-1 656.
[17] OHON Y, MAEHASHI K, MARSUMOTO K. Label-free biosensors based on aptamer-modified graphene field-effect transistors[J]. Journal of the American Chemical Society, 2010, 132(51): 18 012-18 013.
[18] ZHANG S, MA L, MA K, et al. Label-free aptamer-based biosensor for specific detection of chloramphenicol using AIE probe and graphene oxide[J]. ACS omega, 2018, 3(10): 12 886-12 892.
[19] GAO H, GAN N, PAN D, et al. A sensitive colorimetric aptasensor for chloramphenicol detection in fish and pork based on the amplification of a nano-peroxidase-polymer[J]. Analytical Methods, 2015, 7(16): 6 528-6 536.
[20] LU Z, CHEN X, HU W. A fluorescence aptasensor based on semiconductor quantum dots and MoS2 nanosheets for ochratoxin A detection[J]. Sensors and Actuators B: Chemical, 2017, 246: 61-67.
[21] KIM S, LEE H J.Gold nanostar enhanced surface plasmon resonance detection of an antibiotic at attomolar concentrations via an aptamer-antibody sandwich assay[J]. Analytical chemistry, 2017, 89(12): 6 624-6 630.
[22] JAYASENA S D. Aptamers: An emerging class of molecules that rival antibodies in diagnostics[J]. Clin Chem,1999, 45(9):1 628-1 650.
[23] SONG S, WANG L, LI J, et al. Aptamer-based biosensors[J]. TrAC Trends in Analytical Chemistry, 2008, 27(2):108-117.
[24] HERMANN T. Adaptive recognition by nucleic acid aptamers[J]. Science, 2000, 287(5 454):820-825.
[25] MONTAZER M, PARVNZADEH M, KIUMARSI A. Colorimetric properties of wool dyed with natural dyes after treatment with ammonia[J]. Coloration Technology, 2006, 120(4):161-166.
[26] YE BF, ZHAO Y J, CHENG Y, et al. Colorimetric photonic hydrogel aptasensor for the screening of heavy metal ions[J]. Nanoscale, 2012, 4(19):5 998-6 003.
[27] XIE Y, HUANG Y, TANG D, et al. A competitive colorimetric chloramphenicol assay based on the non-cross-linking deaggregation of gold nanoparticles coated with a polyadenine-modified aptamer[J]. Microchimica Acta, 2018, 185(12):534.
[28] YAN C, ZHANG J, YAO L, et al. Aptamer-mediated colorimetric method for rapid and sensitive detection of chloramphenicol in food[J]. Food Chemistry, 2018,260: 208-212.
[29] HUANG W, ZHANG H, LAI G, et al. Sensitive and rapid aptasensing of chloramphenicol by colorimetric signal transduction with a DNAzyme-functionalized gold nanoprobe. [J] Food Chemistry, 2019, 270:287-292.
[30] ZHANG X, HUANG P J J, SERVOS M R, et al. Effects of polyethylene glycol on DNA adsorption andHybridization on gold nanoparticles and graphene oxide[J]. Langmuir, 2012, 28(40):14 330-14 337.
[31] JAVIDI M, HOUSAINDOKHT, MR, et al. Detection of chloramphenicol using a novel apta-sensing platform based on aptamer terminal-lock in milk samples[J]. Analytical Chimica Acta, 2018, 1039:116-123.
[32] ROSSINI E L, MILANI M I, PEZZA H R. Green synthesis of fluorescent carbon dots for determination of glucose in biofluids using a paper platform[J]. Talanta, 2019, 201: 503-510.
[33] 谷巧荣. 荧光分析法检测磷脂酶C及DnaseⅠ活性的研究[D].西安:陕西师范大学,2017.
[34] LEE J B, KURODA S, SHICHINOHE H, et al. Migration and differentiation of nuclear fluorescence-labeled bone marrow stromal cells after transplantation into cerebral infarct and spinal cord injury in mice[J]. Neuropathology, 2003, 23(3): 169-180.
[35] ALIBOLANDI M, HADIZADEH F, VAJHEDIN F, et al. Design and fabrication of an aptasensor for chloramphenicol based on energy transfer of CdTe quantum dots to graphene oxide sheet[J]. Materials Science and Engineering: C, 2015, 48:611-619.
[36] MIAO Y, GAN N, LI T, et al. An ultrasensitive fluorescence aptasensor for chloramphenicol based on FRET between quantum dots as donor and the magnetic SiO2@ Au NPs probe as acceptor with exonuclease-assisted target recycling[J]. Sensors and Actuators B: Chemical, 2016, 222: 1 066-1 072.
[37] MIAO Y,REN H X,GAN N, et al. A homogeneous and “off-on” fluorescence aptamer-based assay for chloramphenicol using vesicle quantum dot-gold colloid composite probes[J]. Analytica Chimica Acta, 2016,929:49-55.
[38] WU S, ZHANG H, SHI Z, et al. Aptamer-based fluorescence biosensor for chloramphenicol determination using upconversion nanoparticles[J]. Food Control, 2015, 50:597-604.
[39] HAO L DUAN N, WU S, et al. Chemiluminescent aptasensor for chloramphenicol based on N-(4-aminobutyl)-N-ethylisoluminol-functionalized flower-like gold nanostructures and magnetic nanoparticles[J]. Analytical and Bioanalytical Chemistry, 2015, 407(26):7 907-7 915.
[40] 于秀霞. 化学发光功能化纳米材料在新型生物传感器中的应用[D]. 合肥:中国科学技术大学,2014.
[41] YAN W, YANG L, ZHUANG H, et al. Engineered “hot” core-shell nanostructures for patterned detection of chloramphenicol[J]. Biosensors & Bioelectronics, 2015,78:67-72.
[42] YANG K, HU Y, DONG N. A novel biosensor based on competitive SERS immunoassay and magnetic separation for accurate and sensitive detection of chloramphenicol[J]. Biosensors and Bioelectronics, 2016, 80: 373-377.
[43] CHEN M, GAN N,ZHOU Y, et al. A novel aptamer- metal ions- nanoscale MOF based electrochemical biocodes for multiple antibiotics detection and signal amplification[J]. Sensors and Actuators, B: Chemical, 2017, 242:1 201-1 209.
[44] FENG X, GAN N, ZHANG H, et al. A novel “dual-potential” electrochemiluminescence aptasensor array using CdS quantum dots and luminol-gold nanoparticles as labels for simultaneous detection of malachite green and chloramphenicol[J]. Biosensors and Bioelectronics, 2015, 74:587-593.
[45] ZHOU L, GAN N, ZHOU Y, et al. A label-free and universal platform for antibiotics detection based on microchip electrophoresis using aptamer probes[J]. Talanta, 2017, 167(Complete):544-549.
[46] ZHOU L, GAN N, HU F, et al. Microchip electrophoresis array-based aptasensor for multiplex antibiotic detection using functionalized magnetic beads and polymerase chain reaction amplification[J]. Sensors and Actuators B: Chemical, 2018,263:568-574.
[47] 赵帅. 基于核酸适配体的侧流层析试纸条法快速检测氯霉素残留[D]. 北京:北京化工大学,2018.
[48] BERLINA A N, NADEZHDA A, TERANOVA N A,et al. Quantum dot-based lateral flow immunoassay for detection of chloramphenicol in milk[J]. Analytical and Bioanalytical Chemistry, 2013, 405(14): 4 997-5 000.
[49] SUN Y, LU J. Chemiluminescence-based aptasensors for various target analytes[J]. Luminescence, 2018,33(18):1 298-1 305.
[50] MEHLHORN A, RAHIMI P,JOSEPH Y. Aptamer-based biosensors for antibiotic detection: A review[J]. Biosensors, 2018, 8(2):54-62.