长双歧杆菌是在人体肠道微生物群中最广泛存在的一种双歧杆菌[1],具有防治便秘[2]、抑制肠道致病菌[3]、调节肠道平衡、降低胆固醇[4]、促进营养物质的消化吸收、延缓衰老[5]和增强机体免疫活性[6-7]等生理功能。长双歧杆菌可分为3个亚种,即长双歧杆菌长亚种、长双歧杆菌婴儿亚种和长双歧杆菌猪亚种[8],这3个亚种在生理生化特性以及遗传进化方面十分相近,常用的16S rRNA菌种鉴定的方法无法实现这3个亚种的区分[9-10]。目前多使用全基因组测序及系统发育树[11-13]、PCR-RFLP[14]、PCR-DGGE[15]、多位点序列分析(MLSA)、扩增片段长度多态性分析(AFLP)[16]、多位点测序分型(MLST)[17]等方法来区分不同亚种,但这些方法过程复杂,耗时长,成本高。《伯杰氏系统细菌学手册》中记录了长双歧杆菌的不同亚种对碳水化合物利用能力存在显著差异,这或许可作为区分不同亚种的依据。此外,利用基因型差异来区分亚种的方法也更加可靠,已有研究者尝试采用特异性基因扩增的方法进行一些细菌的亚种区分,该方法过程简单、快速,但需要找到能够可靠区分不同亚种的特异性基因[18-21]。
因此,本研究拟利用碳水化合物利用谱测定、特异性基因扩增等手段,确认一种有效、快速、低成本的区分方法,以实现长双歧杆菌的亚种区分。
1 材料与方法
1.1 材料与试剂
1.1.1 实验菌株
本文所用54株长双歧杆菌,均保存于江南大学食品生物技术研究中心菌种库(表1)。其中ATCC15697为长双歧杆菌婴儿亚种,13株已测定了基因组但亚种未知,其余40株亚种未知且无基因组序列。
表1 本文所用长双歧杆菌
Table 1Bifidobacterium longum in this study
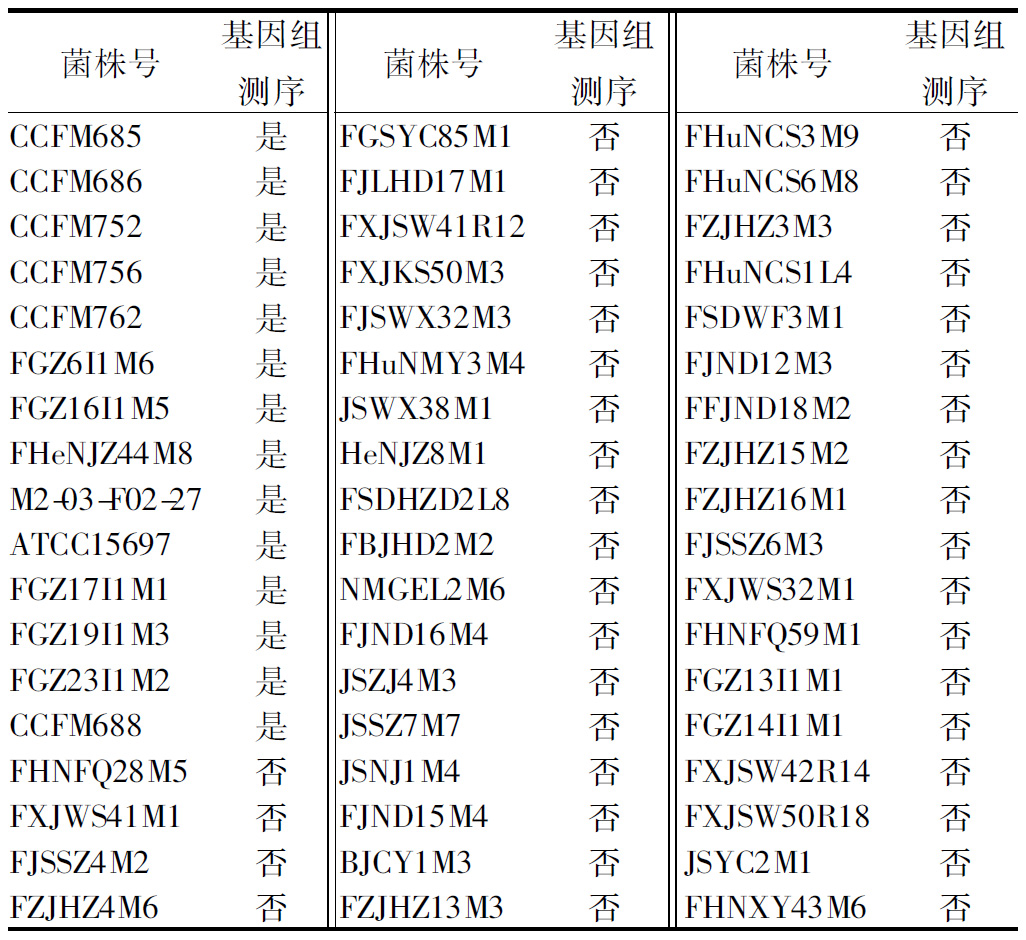
菌株号基因组测序菌株号基因组测序菌株号基因组测序CCFM685是FGSYC85M1否FHuNCS3M9否CCFM686是FJLHD17M1否FHuNCS6M8否CCFM752是FXJSW41R12否FZJHZ3M3否CCFM756是FXJKS50M3否FHuNCS1L4否CCFM762是FJSWX32M3否FSDWF3M1否FGZ6I1M6是FHuNMY3M4否FJND12M3否FGZ16I1M5是JSWX38M1否FFJND18M2否FHeNJZ44M8是HeNJZ8M1否FZJHZ15M2否M2-03-F02-27是FSDHZD2L8否FZJHZ16M1否ATCC15697是FBJHD2M2否FJSSZ6M3否FGZ17I1M1是NMGEL2M6否FXJWS32M1否FGZ19I1M3是FJND16M4否FHNFQ59M1否FGZ23I1M2是JSZJ4M3否FGZ13I1M1否CCFM688是JSSZ7M7否FGZ14I1M1否FHNFQ28M5否JSNJ1M4否FXJSW42R14否FXJWS41M1否FJND15M4否FXJSW50R18否FJSSZ4M2否BJCY1M3否JSYC2M1否FZJHZ4M6否FZJHZ13M3否FHNXY43M6否
1.1.2 培养基与试剂
改良MRS(modified MRS,mMRS)培养基:蛋白胨10 g;牛肉膏10 g;酵母粉5 g;葡萄糖20 g;乙酸钠5 g;吐温80 1mL;K2HPO42.0 g;柠檬酸氢二铵2.0 g;MgSO4·7H2O 0.1 g;MnSO4·H2O 0.05 g;L-半胱氨酸盐酸盐0.5 g;蒸馏水1 L;pH 6.2~6.4;115 ℃灭菌20 min。
不同碳水化合物替代葡萄糖的培养基:用不同的碳水化合物(终质量浓度为10 g/L)替代mMRS培养基中的葡萄糖,并添加0.5%溴甲酚紫母液15 mL/L;
作为阴性对照培养基:mMRS培养基添加0.5%溴甲酚紫母液15 mL/L;
2×Taq Plus MasterMix,康为世纪生物科技有限公司;GeneRuler DNA Ladder Mix,英潍捷基;HGV-Ⅱ核酸染液,北京赛百盛基因技术有限公司;琼脂糖,生工生物工程股份有限公司。
1.2 仪器与设备
AW500SG型Electrotek厌氧工作站,英国依莱泰科;GR60DA立式自动压力蒸汽灭菌器,致微(厦门)仪器有限公司;SW-CJ-1FD洁净工作台,苏州安泰空气技术有限公司;C1000 TouchTM型基因扩增仪,美国Bio-Rad公司;DYCP-31DN/DYCP-32B水平电泳仪,北京六一生物科技有限公司;Gel DocTM EZ imager凝胶成像系统,美国Bio-Rad公司。
1.3 方法
1.3.1 系统发育树的构建
采用orthomclV2.0.9软件对不同菌株的直系同源基因进行分析[22-23],根据同源基因分析结果,提取所有菌株的直系同源基因序列,利用mafft-7.313软件对不同菌株的直系同源基因进行序列比对[22,24],基于序列比对结果,进行进化分析构建系统发育树。
1.3.2 菌株的活化
将保藏于甘油中的菌株划线至mMRS固体培养基37 ℃厌氧培养48 h,后挑取单菌落接种至5 mL mMRS液体培养基中,37 ℃厌氧培养48 h,液体连续活化2代。
1.3.3 碳水化合物利用能力比较
将200 μL不同碳水化合物替代葡萄糖的培养基和阴性对照的培养基分装于无菌96孔板中,分别接种2次活化后的2 μL菌液至96孔中,每个菌每种碳水化合物测2个平行孔,培养48 h后观察孔板中培养基颜色变化及菌株生长情况。碳水化合物利用能力比较实验重复3遍测定。
1.3.4 不同亚种的特异性基因分析
本文所有引物信息如表2所示。PCR反应体系(20 μL):模板1 μL,上、下游引物各0.5 μL,2×Taq MasterMix 10 μL,ddH2O 8 μL。PCR条件:95 ℃预变性5 min,95 ℃变性35 s,60 ℃退火35 s,72 ℃延伸40 s,从变性到延伸需要重复30个循环,最后72 ℃补充延伸10 min。PCR产物进行1.5%的琼脂糖凝胶电泳,于凝胶成像分析仪中观察条带情况。实验重复3次确保引物具有良好的重复性。
表2 PCR引物
Table 2 PCR primers

亚种名称特异性基因引物名称序列(5'-3')片段长度/bp参考文献长双歧杆菌长亚种MarR family transcriptional regula-torMarR-FMarR-RATCCTCAACCAATGCGT-TCCGGCTTGTTCCAGCAGTTT-GA168[20]Tellurite resistance protein TerBTerB-FTerB-RTGAGGGTGTTGATG-CAGTCTATCGTCCGGGCAATAT-GTCT156[20]Sugar kinase genelon_Flon_RGAGGCGATGGTCTG-GAAGTTCCACATCGCCGAGAAGAT-TC181[18]Tuf geneBloFBloRGTATCCGTCCGACCCAG-CAGGGTGACGGAGCCCGGCTTG161[21]长双歧杆菌婴儿亚种GlyoxalaseGlyoxalase-FGlyoxalase-RGCTTTGCAGACAGTT-GAAACCGCTGTCCCCATGAATGT-TC176[20]ABC-2 type transporterABC-FABC-2AGCGGACAGGTACATTG-GAACCAATATGAGTTGC-CACGGG177[20]Sialidase geneinf_Finf_RATACAGCAGAACCTTGGC-CTGCGATCACATG-GACGAGAAC234[18]Tuf geneBinf-tFBinf-tRATCCGTCCGACCCAGACCCTCGACATCCTCACGGCC123[21]长双歧杆菌猪亚种Serine/threonine protein kinaseSerine-FSerine-RGCAACTCGAGGATTCCGACAACGGATTGGTGGTTT-GCTC166[20]
1.3.5 长双歧杆菌菌株的亚种确认
利用亚种特异性引物对40株亚种未知的长双歧杆菌进行PCR扩增,根据扩增产物电泳后条带情况区分长双歧杆菌的不同亚种。并选择扩增结果为婴儿亚种的菌株送至上海美吉生物医药科技有限公司进行基因组草图扫描,并构建系统进化树,以验证其为长双歧杆菌婴儿亚种。
2 结果与分析
2.1 13株已测基因组菌株的亚种确认
从NCBI数据库(https://www.ncbi.nlm.nih.gov/)中获取已确认亚种的长双歧杆菌长亚种、长双歧杆菌婴儿亚种、长双歧杆菌猪亚种菌株的基因组序列(各10个),与本团队前期已测全基因组但尚未确认亚种的13株长双歧杆菌进行直系同源基因分析,共获得核心基因941个,基于这些核心基因,构建了43株菌系统发育树(图1)。
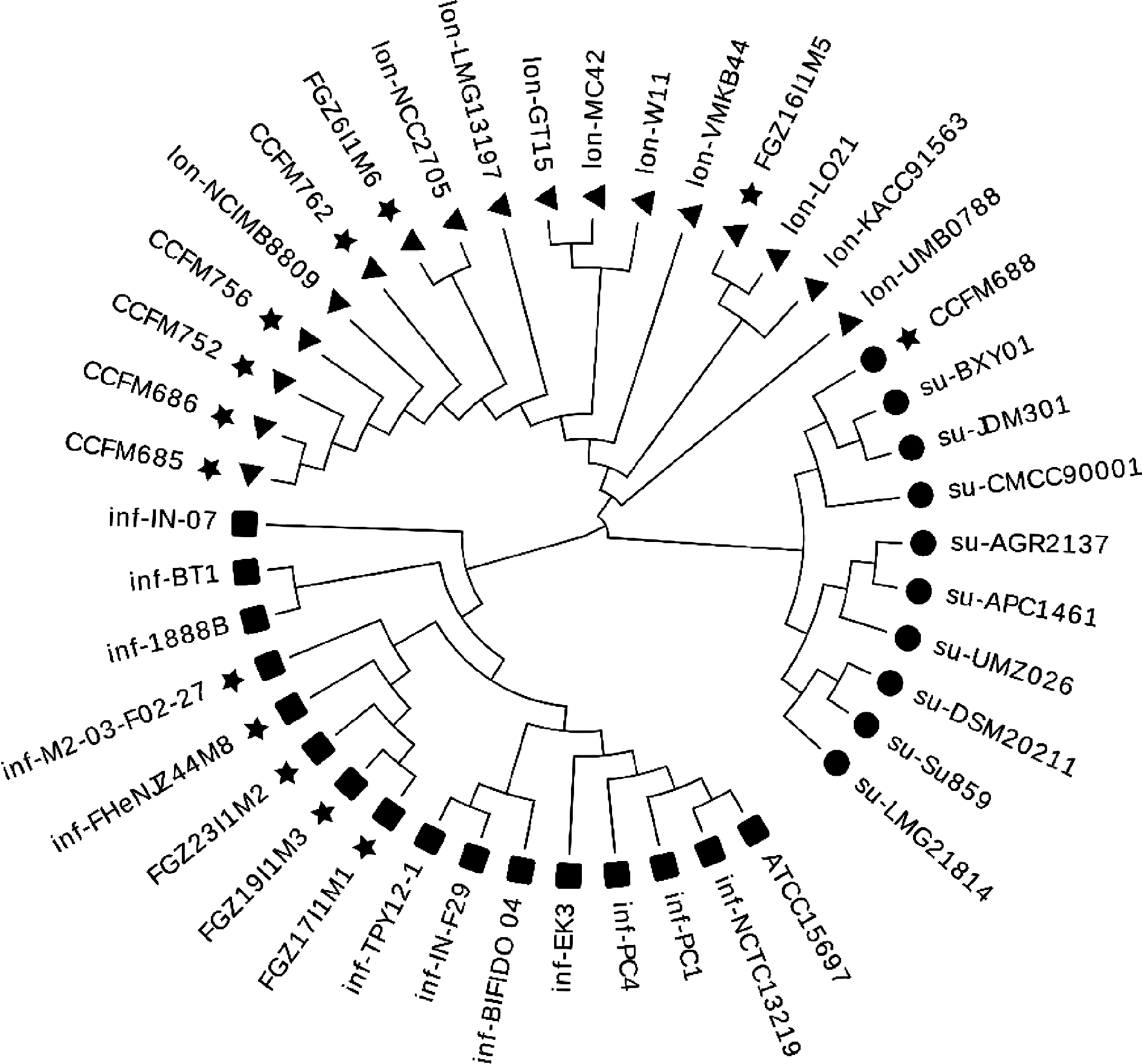
图1 基于43株长双歧杆菌核心基因构建系统发育树
Fig.1 Phylogenetic tree of 43 Bifidobacterium longum
based on core genes
注:“■”为长双歧杆菌婴儿亚种;“▲”为长双歧杆菌长亚种;“●”为长双歧杆菌猪亚种;“★”为已测定全基因组但尚未确认亚种的长双歧杆菌。
结果显示,30株长双歧杆菌不同亚种分别位于不同分支上,而本团队前期已测基因组的13株菌分别属于3个不同亚种,其中7株是长双歧杆菌长亚种、5株为长双歧杆菌婴儿亚种、1株为长双歧杆菌猪亚种。
2.2 不同长双歧杆菌碳水化合物利用能力比较
根据《伯杰氏系统细菌学手册》及相关文献报道[25-26]选择了长双歧杆菌不同亚种利用能力存在显著差异的6种碳水化合物进行实验,包括阿拉伯糖、木糖、核糖、葡萄糖醛酸钠、甘露糖和岩藻糖,以进一步确认长双歧杆菌不同亚种碳水化合物利用谱差异。
结果显示,所测定的长双歧杆菌长亚种、长双歧杆菌婴儿亚种、长双歧杆菌猪亚种共14株菌利用不同碳水化合物产酸能力存在差异(表3)。3个亚种均可利用核糖和岩藻糖;长双歧杆菌长亚种和长双歧杆菌猪亚种不能利用葡萄糖醛酸钠,而长双歧杆菌婴儿亚种FHeNJZ44M8、M2-03-F02-27和ATCC15697三株菌可利用葡萄糖醛酸钠,其余3株却不能利用;长双歧杆菌长亚种中除FGZ16I1M5外,均可利用阿拉伯糖和木糖产酸。同一亚种的菌株利用同一种碳水化合物的能力存在差异,故根据不同碳水化合物利用能力的差异难以实现长双歧杆菌不同亚种的区分。
表3 长双歧杆菌不同亚种的碳水化合物利用比较
Table 3 Carbohydrate utilization capacity of differentBifidobacterium longum strains
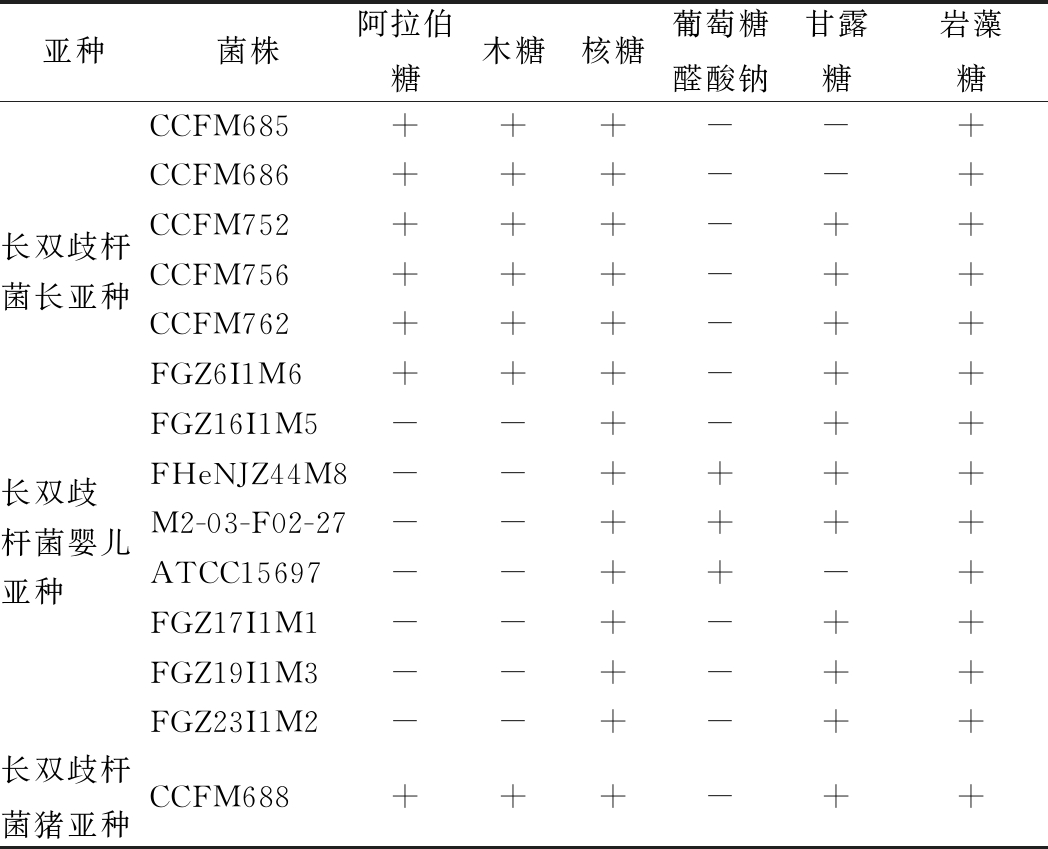
亚种菌株阿拉伯糖木糖核糖葡萄糖醛酸钠甘露糖岩藻糖长双歧杆CCFM685+++--+菌长亚种CCFM686+++--+CCFM752+++-++CCFM756+++-++CCFM762+++-++FGZ6I1M6+++-++FGZ16I1M5--+-++长双歧FHeNJZ44M8--++++杆菌婴儿M2-03-F02-27--++++亚种ATCC15697--++-+FGZ17I1M1--+-++FGZ19I1M3--+-++FGZ23I1M2--+-++长双歧杆菌猪亚种CCFM688+++-++
注:“+”代表菌株利用该碳水化合物产酸;“-”代表菌株未利用该碳水化合物产酸。
2.3 不同亚种的特异性基因分析
根据相关文献报道选择了9个亚种特异性基因(表2),对14株亚种确认的长双歧杆菌基因组DNA进行扩增,以评价这个特异性基因的有效性。MarR基因是长双歧杆菌长亚种的特异性基因,理论扩增条带为168 bp,但7株长双歧杆菌长亚种的扩增结果显示,仅CCFM762(图2-b)、CCFM756(图2-g)可成功扩增出168 bp的条带,其余长亚种菌株无扩增产物;TerB基因的扩增结果也类似,仅CCFM685(图2-c)、CCFM686(图2-d)和FGZ16I1M5(图2-f)可扩增出正确大小的条带(156 bp),其余长亚种菌株均没有扩增产物,因此,这2个基因无法用于长双歧杆菌长亚种的区分。尽管所有长亚种菌株可扩增出Sugar kinase基因(图2),婴儿亚种菌株也不能扩增出该基因(图3),但猪亚种同样扩增出该基因(图4),因此,该基因也无法区分长亚种和猪亚种,但可用于长双歧杆菌婴儿亚种的确认。所有长亚种菌株(图2)和猪亚种菌株(图4)可扩增出长亚种Tuf基因,而婴儿亚种菌株(图3)也可扩增出条带,该基因无法快速区分不同亚种。
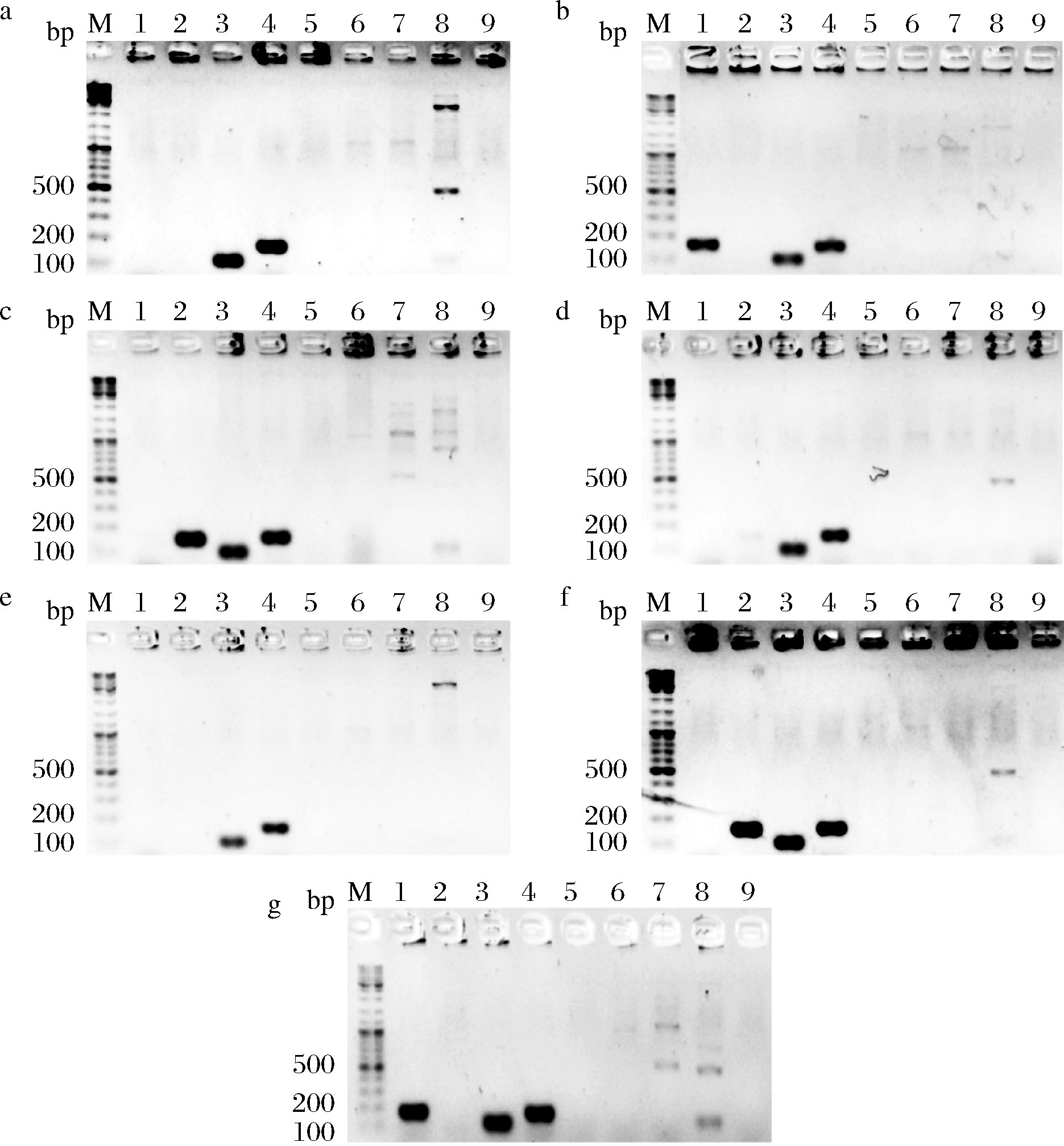
a~g- FGZ6I1M6、CCFM762、CCFM685、CCFM686、CCFM752、FGZ16I1M5、CCFM756
图2 长双歧杆菌长亚种菌株PCR电泳结果
Fig.2 PCR electrophoresis results of B. longum
subsp. longum
注:M- DNA Marker;1~9表示分别用MarR、TerB、lon、Blo、Glyoxalase、ABC、inf、Binf、Serine引物扩增。下同。
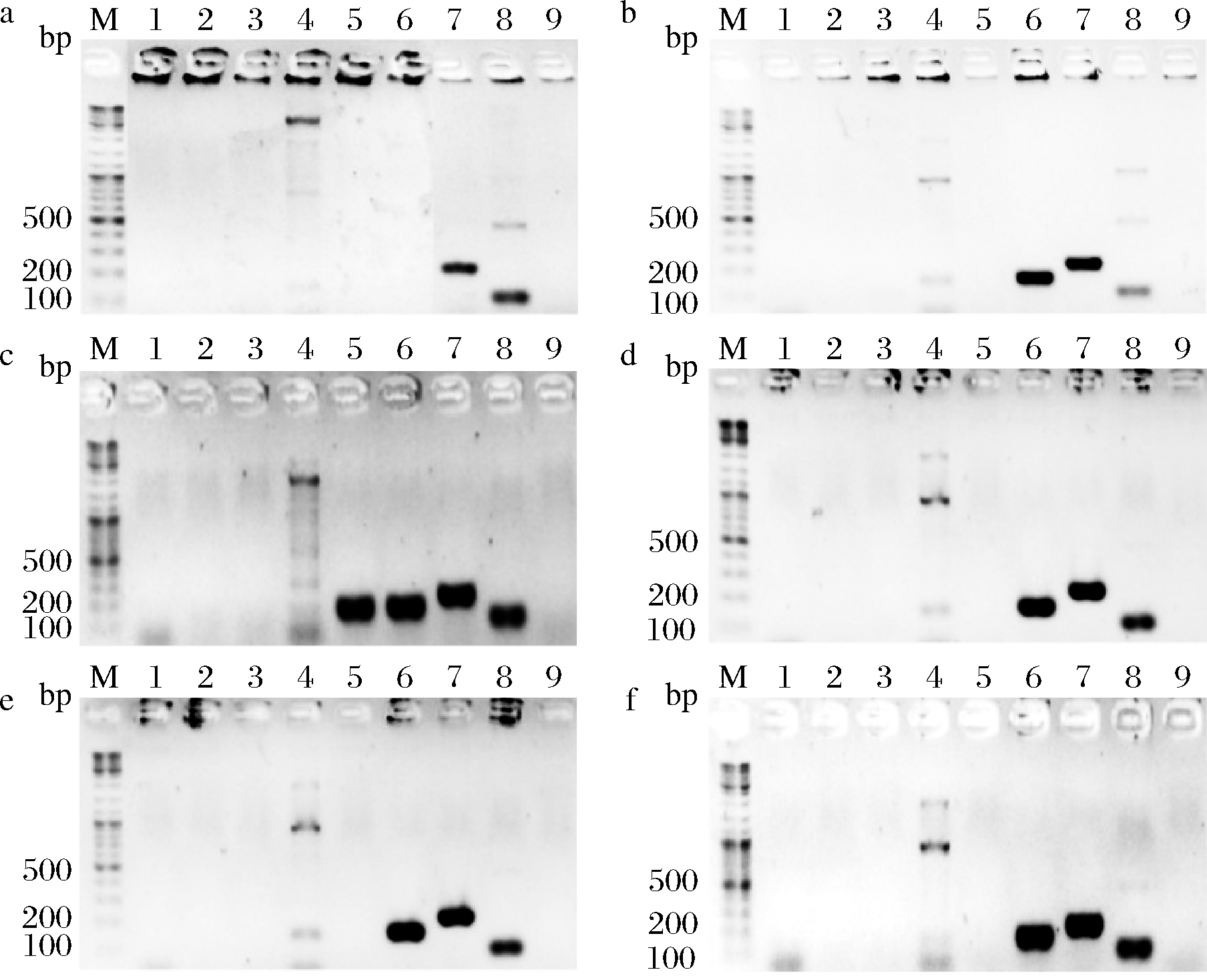
a~f- M2-03-F02-27、FHeNJZ44M8、ATCC15697、GZ17I1M1、GZ19I1M3、GZ23I1M2
图3 长双歧杆菌婴儿亚种菌株PCR电泳结果
Fig.3 PCR electrophoresis results of B. longum
subsp. infantis
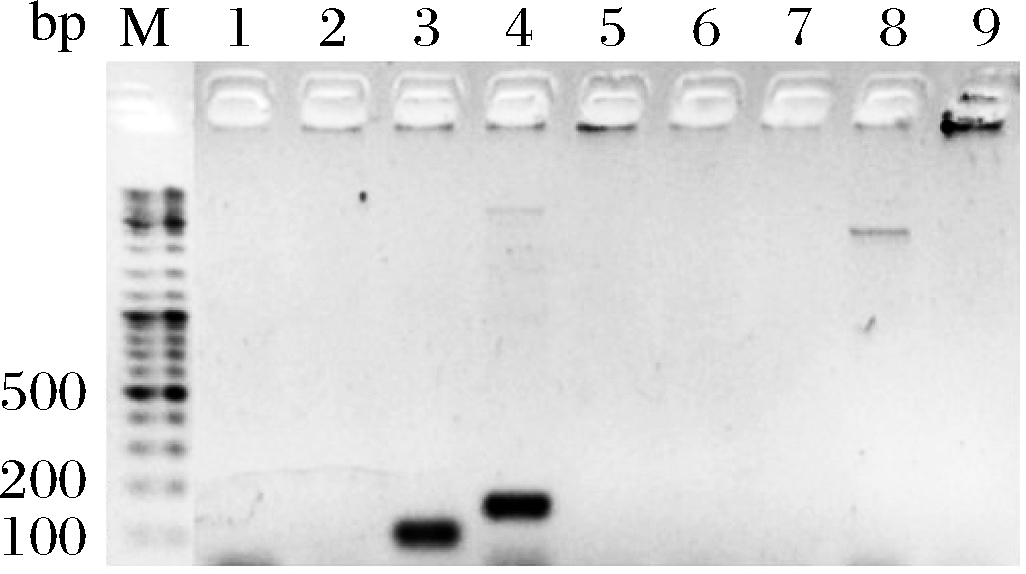
图4 长双歧杆菌猪亚种菌株PCR电泳结果
Fig.4 PCR electrophoresis results of B. longum subsp. suis
Glyoxalase基因是长双歧杆菌婴儿亚种的特异性基因,理论扩增条带为168 bp,但6株长双歧杆菌婴儿亚种的扩增结果显示,只有ATCC15697(图3-c)可扩增出168 bp的条带,其余婴儿亚种菌株没有扩增产物;ABC基因的结果也类似,M2-03-F02-27(图3-a)没有扩增产物,因此,这2个基因无法用于长双歧杆菌婴儿亚种的区分。Sialidase基因的扩增结果显示,仅长双歧杆菌婴儿亚种菌株可成功扩增得到正确大小的条带(图3),而长双歧杆菌长亚种(图2)和长双歧杆菌猪亚种(图4)均无扩增产物,因此,Sialidase基因可用于长双歧杆菌婴儿亚种的快速区分。婴儿亚种Tuf基因是长双歧杆菌婴儿亚种的特异性基因,该基因理论扩增条带为123 bp,但所用14株菌的扩增结果显示,长双歧杆菌长亚种FGZ6I1M6(图2-a)、CCFM685(图2-c)、CCFM756(图2-g)和长双歧杆菌婴儿亚种菌株(图3)均可以扩增出100 bp左右的条带,因此,该基因的区分效果同样不佳。而猪亚种的特异性基因Serine,理论扩增条带为166 bp,但扩增结果显示,CCFM688(图4)没有扩增产物,鉴于菌株数量太少,还需进一步确认。
为了解析不同基因扩增效果的差异,进一步利用Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)来分析引物的特异性(表4)。结果显示,MarR和TerB的引物只能扩增出长双歧杆菌长亚种菌株,但扩增出的菌株数较少,说明基因的特异性过强,可能不能广泛区分长亚种;Sugar kinase和长亚种Tuf基因的引物可扩增出长双歧杆菌长亚种和猪亚种,也就解释了这两个基因均无法实现长亚种和猪亚种的区分的原因,且长亚种Tuf基因可扩增出多个不同大小的片段;基因Glyoxalase和基因ABC Primer-BLAST只能扩增出部分长双歧杆菌婴儿亚种,基因的特异性较强,不能广泛用于区分婴儿亚种;Sialidase基因可扩增出全部长双歧杆菌婴儿亚种,基因的特异性较好;婴儿亚种Tuf基因可扩增出全部长双歧杆菌婴儿亚种,但可扩增出多个不同大小的片段;基因Serine Primer-BLAST只能扩增出部分长双歧杆菌猪亚种,无法实现猪亚种的区分。
表4 特异性基因Primer-BLAST结果
Table 4 The Primer-BLAST results of specific genes
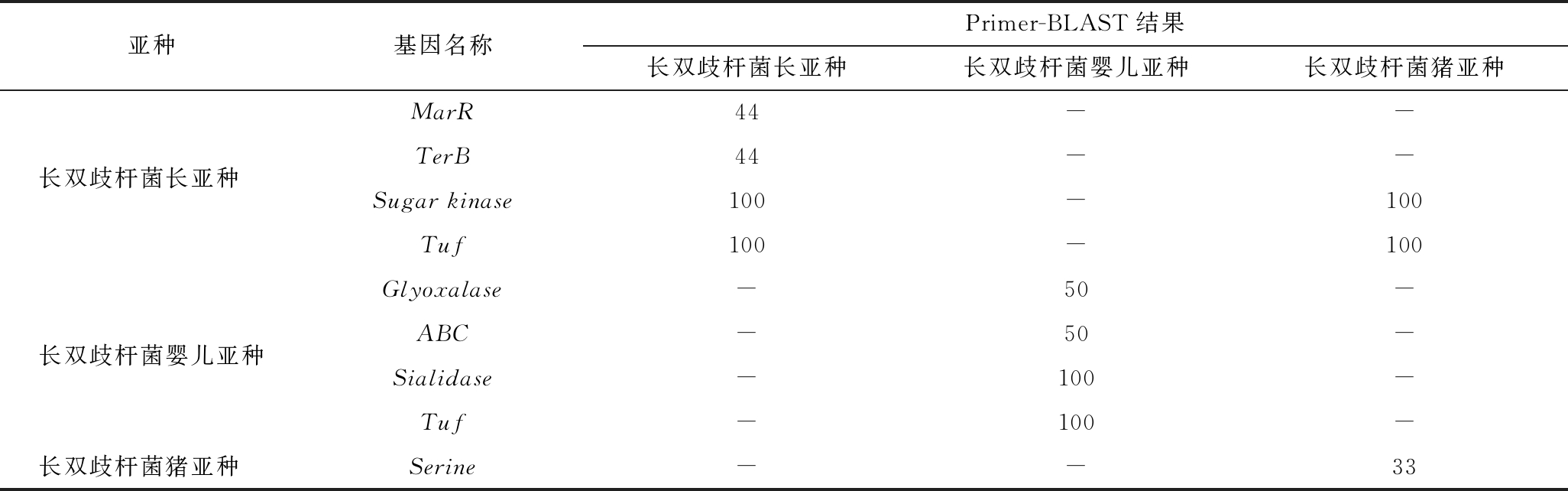
亚种基因名称Primer-BLAST结果长双歧杆菌长亚种长双歧杆菌婴儿亚种长双歧杆菌猪亚种长双歧杆菌长亚种 MarR44--TerB44--Sugar kinase100-100Tuf100-100长双歧杆菌婴儿亚种Glyoxalase-50-ABC-50-Sialidase-100-Tuf-100-长双歧杆菌猪亚种 Serine--33
注:表中数字为引物扩增出的菌株数占该亚种菌株总数的比例;“-”表示引物未扩增出该亚种菌株。
综上可知,长双歧杆菌长亚种糖激酶基因和长双歧杆菌婴儿亚种唾液酸酶基因可有效实现长双歧杆菌婴儿亚种与其他两亚种的区分,根据扩增产物电泳后条带的有无及大小初步实现长双歧杆菌婴儿亚种的区分。若婴儿亚种唾液酸酶基因扩增产物电泳后产生234 bp左右的条带且长亚种糖激酶基因扩增产物电泳后无条带产生,则该菌株可初步确认为长双歧杆菌婴儿亚种。
2.4 长双歧杆菌婴儿亚种的确认
采用上述区分效果较好的长双歧杆菌长亚种糖激酶基因和长双歧杆菌婴儿亚种唾液酸酶基因进行PCR扩增,对40株亚种未知的长双歧杆菌进行了PCR扩增,电泳后的条带结果显示,HeNJZ8M1、FJND16M4、JSSZ7M7、FHuNCS6M8和JSYC2M1共5株菌为长双歧杆菌婴儿亚种,其余35株菌均为长双歧杆菌长亚种或猪亚种(图5)。

图5 40株长双歧杆菌PCR电泳结果
Fig.5 PCR electrophoresis results of
40 Bifidobacterium longum
为进一步验证目前所确认亚种的准确性,从5株新确认的长双歧杆菌婴儿亚种中随机选择了4株,即HeNJZ8M1、FHuNCS6M8、FJND16M4、JSSZ7M7寄送上海美吉生物医药科技有限公司进行基因组草图测序。并将上述4株菌与图1中43株菌合并进行同源基因分析,共获得938个核心基因。基于这些核心基因,构建了该47株菌系统发育树(图6)。结果显示,经特异性基因扩增确认为长双歧杆菌婴儿亚种的4株菌(HeNJZ8M1、FHuNCS6M8、FJND16M4和JSSZ7M7)确为长双歧杆菌婴儿亚种,进一步证实了基于长双歧杆菌长亚种糖激酶基因和长双歧杆菌婴儿亚种唾液酸酶基因的特异性扩增可有效实现长双歧杆菌婴儿亚种的快速区分。

图6 基于47株长双歧杆菌核心基因构建系统发育树
Fig.6 Phylogenetic tree of 47 Bifidobacterium
longum based on core genes
注:“■”长双歧杆菌婴儿亚种;“▲”长双歧杆菌长亚种;“●”长双
歧杆菌猪亚种;“★”新确认的长双歧杆菌婴儿亚种。
3 结论
本研究对13株已测定基因组的长双歧杆菌经生物信息学分析确认了亚种,其中7株为长双歧杆菌长亚种,5株为长双歧杆菌婴儿亚种,1株为长双歧杆菌猪亚种。对亚种确认的长双歧杆菌进行了碳水化合物利用谱比较,但碳水化合物利用谱难以实现不同亚种的区分。后采用特异性基因扩增的方法确认了长双歧杆菌长亚种糖激酶基因和长双歧杆菌婴儿亚种唾液酸酶基因可实现长双歧杆菌婴儿亚种的快速区分,并采用这对特异性基因从40株长双歧杆菌中成功确认了5株长双歧杆菌婴儿亚种,其余35株菌均为长双歧杆菌长亚种或猪亚种,随机选择4株进行全基因组草图测序,进一步分析证实确为长双歧杆菌婴儿亚种。遗憾的是,因长双歧杆菌猪亚种可用菌株数量极少,长双歧杆菌长亚种和长双歧杆菌猪亚种的快速区分仍有待进一步完善。
[1] CHAPLIN A V, EFIMOV B A, SMEIANOV V V, et al. Intraspecies genomic diversity and long-term persistence of bifidobacterium iongum[J]. PloS One, 2015,10(8):e01 356 588.
[2] 丁圣, 蒋菁莉,刘松玲,等. 长双歧杆菌BBMN68对便秘模型小鼠的通便作用[J]. 食品科学, 2011,32(3):195-198.
[3] YUN B, SONG M, PARK D, et al. Beneficial effect of Bifidobacterium longum ATCC 15707 on survival rate of Clostridium difficile infection in mice[J]. Korean Journal for Food Science of Animal Resources, 2017,37(3):368-375.
[4] 韦云路, 刘义,王瑶,等. 3株益生菌体外降胆固醇能力及体内降血脂效果评价[J]. 食品科学, 2017,38(23):129-134.
[5] 吕锡斌, 何腊平,张汝娇,等. 双歧杆菌生理功能研究进展[J]. 食品工业科技, 2013,34(16):353-358.
[6] 李平兰, 马长伟,江志杰,等. 长双歧杆菌TTF菌株增强机体免疫活性研究[J]. 微生物学通报, 2006,33(2):1-5.
[7] 杨景, 罗绪刚,任发政. 长双歧杆菌BBMN68诱导的树突状细胞对小鼠牛乳β-乳球蛋白过敏的缓解作用[J]. 现代食品科技, 2016,32(7):6-11.
[8] MATTARELLI P, BONAPARTE C, POT B, et al. Proposal to reclassify the three biotypes of Bifidobacterium longum as three subspecies: Bifidobacterium longum subsp. longum subsp nov., Bifidobacterium longum subsp. infantis comb. nov and Bifidobacterium longum subsp. suis comb. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2008,58(4):767-772.
[9] YOUN S Y, SEO J M, JI G E. Evaluation of the PCR method for identification of Bifidobacterium species[J]. Letters in Applied Microbiology, 2008,46(1):7-13.
[10] TANNOCK G W, LAWLEY B, MUNRO K, et al. Comparison of the compositions of the stool microbiotas of infants fed goat milk formula, cow milk-based formula, or breast milk[J]. Applied and Environmental Microbiology, 2013,79(9):3 040-3 048.
[11] ARBOLEYA S, BOTTACINI F, O’CONNELL-MOTHERWAY M, et al. Gene-trait matching across the Bifidobacterium longum pan-genome reveals considerable diversity in carbohydrate catabolism among human infant strains[J]. BMC GENOMICS, 2018,19(1):33.
[12] O’CALLAGHAN A, BOTTACINI F, MOTHERWAY M O C, et al. Pangenome analysis of Bifidobacterium longum and site-directed mutagenesis through by-pass of restriction-modification systems[J]. BMC GENOMICS, 2015,16(1):832.
[13] ALTMANN F, KOSMA P, O’CALLAGHAN A, et al. Genome analysis and characterisation of the exopolysaccharide produced by Bifidobacterium longum subsp. longum 35624TM[J]. PloS One, 2016,11(9):e01629839.
[14] JENA R, CHOUDHURY P K, PUNIYA A K, et al. Isolation and species delineation of genus Bifidobacterium using PCR-RFLP of partial hsp60 gene fragment[J]. LWT-Food Science and Technology, 2017,80:286-293.
[15] REQUENA T, BURTON J, MATSUKI T, et al. Identification, detection, and enumeration of human Bifidobacterium species by PCR targeting the transaldolase gene[J]. Applied and Environmental Microbiology, 2002,68(5):2 420-2 427.
[16] YANOKURA E, OKI K, MAKINO H, et al. Subspeciation of Bifidobacterium longum by multilocus approaches and amplified fragment length polymorphism: Description of B.longum subsp. suillum subsp. nov., isolated from the faeces of piglets[J]. Systematic and Applied Microbiology, 2015,38(5):305-314.
[17] 张秋雪, 刘晓婵,朱宗涛,等. 婴儿粪便长双歧杆菌的分离与多样性分析[J]. 食品科学, 2017,38(24):14-20.
[18] LAWLEY B, MUNRO K, HUGHES A, et al. Differentiation of Bifidobacterium longum subspecies longum and infantis by quantitative PCR using functional gene targets[J]. Peerj, 2017,5:e3375.
[19] LAWLEY B, CENTANNI M, WATANABE J, et al. Tuf gene sequence variation in Bifidobacterium longum subsp. infantis detected in the fecal microbiota of Chinese infants[J]. Applied and Environmental Microbiology, 2018,84(13):e00336-18.
[20] FERRARIO C, MILANI C, MANCABELLI L, et al. A genome-based identification approach for members of the genus Bifidobacterium[J]. FEMS Microbiology Ecology, 2015,91(3):fiv009.
[21] SHEU S, HWANG W, CHIANG Y, et al. Use of Tuf gene-based primers for the PCR detection of probiotic Bifidobacterium species and enumeration of Bifidobacteria in fermented milk by cultural and quantitative real-time PCR methods[J]. Journal of Food Science, 2010,75(8):M521-M527.
[22] 闫爽. 长双歧杆菌遗传与表型多样性及其与免疫调节功能的相关性研究[D]. 无锡:江南大学, 2019.
[23] CHEN F, MACKEY A J, STOECKERTJR C J, et al. OrthoMCL-DB: Querying a comprehensive multi-species collection of ortholog groups[J]. Nucleic Acids Research, 2006,34(S1):D363-D368.
[24] KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability[J]. Molecular Biology and Evolution, 2013,30(4):772-780.
[25] 冯仕云, 龚萍,蒋德意,等. 婴儿双歧杆菌BI-G201的分离与鉴定[J]. 生物化工, 2016,2(6):41-43;46.
[26] SAKATA S, KITAHARA M, SAKAMOTO M, et al. Unification of Bifidobacterium infantis and Bifidobacterium suis as Bifidobacterium longum[J]. International Journal of Systematic and Evolutionary Microbiology, 2002,52(6):1 945-1 951.