涪陵榨菜采用茎瘤芥(Brassica juncea)(俗称青菜头)腌制加工,是世界三大名腌菜之一[1-2]。榨菜加工工艺包括3次腌制和3次压榨:第1次腌制采用菜块质量2%~4%的食盐腌制约7 d,第2次加入4%~8%质量分数的食盐腌制20~30 d,第3次加入质量分数为10%左右的食盐腌制4~6个月,每次腌制结束时都将榨菜起池、压榨去水[3]。
青菜头属于膨大茎,粗壮肥硕,生食有苦辣味,口感不佳,经过腌制成为异常鲜美脆嫩的榨菜。榨菜腌制过程中有明显的产酸产气等发酵现象,因此学者们推测微生物对于榨菜风味品质的形成发挥了关键作用[3-6]。杨珺等[7]、贾巍[8]采用了培养法,鉴定出数十种微生物,细菌主要有:明串珠菌、乳杆菌、片球菌、魏斯氏菌等,酵母菌主要有鲁氏酵母、汉逊氏酵母等,霉菌主要有青霉和曲霉。榨菜微生物数量相关的研究主要集中于第3次腌制时期[7-8],即后熟阶段,因为这一阶段是风味品质形成的关键时期,例如根据贾巍[8]的研究,细菌、酵母初始(第1天)数量分别为2.7×103、2.1×102个/g,第60天均增加到105个/g,第120天分别降到104、103个/g,第180天均降至103个/g;霉菌初始数量为4.25×103 个/g,在整个后熟阶段保持于103个/g但数量一直下降。目前很少有文献分析第1、2次腌制时期微生物数量,缺少对整个腌制过程中微生物数量变化完整的描述。另一方面,培养法存在一定的局限性,不能检出不可培养的微生物,榨菜腌制3个阶段NaCl的质量浓度不同,而培养法中采用相同的培养基和培养条件,不一定适合所有微生物的生长[9-10]。更加深入地研究榨菜整个腌制过程中微生物数量变化过程,可以为榨菜腌制工艺改进、提高过程控制水平提供重要的参考数据。
近年来,实时荧光定量技术(quantitative real-time PCR,qPCR)已广泛应用于食品、环境样品中微生物的数量分析[11],提取样品的宏基因组DNA,采用细菌或者真菌的特异性引物做qPCR扩增,根据标准曲线定量即可检测出样品中细菌、真菌的数量[12]。qPCR为免培养方法,反映样品中可培养和不可培养的所有细菌或者真菌的数量。qPCR技术主要采用2种方法构建标准曲线:第1种方法是将标准菌株液体培养物做10倍梯度稀释,提取基因组DNA作为模板进行qPCR扩增,用Ct值和菌液浓度构建标准曲线[13],这种方法容易受到标准菌株培养性质、DNA提取操作的影响。第2种方法是将标准菌株的16S rRNA/ITS/18S rRNA基因与质粒载体连接,将重组质粒做10倍梯度稀释后作为模板进行qPCR扩增,用Ct值和重组质粒拷贝数构建标准曲线,此法更容易获得线性关系良好的标准曲线,目前在食品病源微生物检测中应用较多,也应用于四川泡菜[14]、西藏开菲尔粒[15]、镇江香醋[16]等传统发酵食品微生物数量变化过程分析,具有快速准确的优点。
本研究将标准菌株的16S rRNA/ITS基因与质粒载体连接作为标准参照物构建标准曲线,建立qPCR检测方法,检测榨菜不同腌制时期腌制汁液中的细菌、真菌数量,同时检测基本理化数据,探讨整个腌制过程中细菌、真菌数量变化过程,为榨菜腌制工艺改进、提高过程控制水平提供重要的参考数据。
1 材料与方法
1.1 材料
榨菜经过3次腌制,分别在第1次腌制的第4、7天,第2次腌制的第5、12、20天,第3次腌制的第9、30、90、180(成品)天采集腌制汁液样品。
1.2 标准菌株
植物乳杆菌(Lactobacillus plantarum, L. plantarum) ATCC 8014、酿酒酵母(Saccharomyces cerevisiae, S. cerevisiae) ATCC 9763:广东省微生物研究所菌种保藏中心。
1.3 试剂与仪器
1.3.1 试剂、培养基和引物
AgNO3标准溶液(0.100 0 mol/L),天津渤化化学试剂有限公司;NaOH、K2CrO4、酚酞,成都市科龙化工试剂厂;Premix Taq、TB Green Premix Ex Taq Ⅱ、RNase-free Water、pMD19-T Vector Cloning Kit、DL 2 000 DNA Ladder Marker、E.coli Competent Cells JM109,宝生物工程(大连)有限公司;IPTG溶液、X-Gal溶液,北京酷来搏科技有限公司;氨苄青霉素钠、细菌基因组DNA提取试剂盒(DP302)、酵母基因组DNA提取试剂盒(DP307)、深加工食品DNA提取试剂盒(DP326)、普通琼脂糖凝胶DNA回收试剂盒(DP209)、质粒小提试剂盒(DP103),天根生化科技(北京)有限公司。
MRS肉汤培养基(g/L,乳酸菌增殖培养用):蛋白胨 10.0、牛肉粉 8.0、酵母粉 4.0、葡萄糖 20.0、K2HPO4 2.0、柠檬酸氢二铵 2.0、乙酸钠 5.0、MgSO4·7H2O 0.2、MnSO4·4H2O 0.04、吐温80 1.0,pH 5.5~5.9,121 ℃高压灭菌15 min。
YM培养基培养基(g/L,酵母菌增殖培养用):胰蛋白胨 5.0、麦芽浸粉 3.0、葡萄糖 10.0、酵母浸粉 3.0,pH 6.0~6.4,121 ℃高压灭菌15 min。
LB肉汤培养基(g/L,大肠杆菌增殖培养用):胰蛋白胨 10.0、酵母浸粉 5.0、NaCl 10.0,pH 6.9~7.1,121 ℃高压灭菌15 min。以上培养基均从青岛高科技工业园海博生物技术有限公司购入。
含抗生素的LB固体平板:100 mL LB培养基(加入1.5 g琼脂粉),经过121 ℃、15 min灭菌后将温度降到55 ℃,以无菌操作加入抗生素,制作LB固体平板[17]。
SOC培养基(g/L,促进感受态细胞转化用):胰蛋白胨 2.0、酵母浸粉 0.5、NaCl 0.05、KCl 0.2、MgCl2 1.0、葡萄糖3.6,购于宝生物工程(大连)有限公司。
引物:引物序列如表1所示,委托生工生物工程(上海)股份有限公司合成。
表1 引物序列
Table 1 Sequences of primers

引物引物序列(5’-3’)扩增物长度/bpY1GCGGTAATTCCAGCTCCAAT-AGY2GCCACAAGGACTCAAGGT-TAG1518FAGAGTTTGATCCTGGCTCAG798RCCAGGGTATCTAATCCTGTT790338FACTCCTACGGGAGGCAGCAG518RATTACCGCGGCTGG18027FAGAGTTTGATCCTGGCTCAG1492RGGYTTACCTTGTTACGACTT1 465M13PrimerM4GTTTTCCCAGTCACGACM13PrimerRVCAGGAAACAGCTATGAC
1.3.2 仪器
CFX96 Real-time PCR仪、T100 PCR仪、power pacbasic电泳仪,美国Bio-Rad公司;5810R高速冷冻离心机,德国Eppendorf股份公司;SW-CJ-2F洁净工作台,苏州安泰空气技术有限公司;SIM-F1制冰机,日本Sanyo有限公司;303系列电热恒温培养箱,上海叶拓仪器仪表有限公司;YCX-2数显恒温水浴锅,金坛市城西峥嵘实验仪器厂;GR60DA立式自动压力蒸汽灭菌锅,美国Zealway有限公司;G:BOXEF凝胶成像系统,美国Gene公司;P100/P100+超微量分光光度计,美国Pultton公司;Mini-Q超级纯水仪,法国Synergy公司;ZWY-2102C双层恒温培养振荡器,上海智城分析仪器制造有限公司;pH计,上海仪电科学仪器股份有限公司。
1.4 实验方法
1.4.1 样品理化指标测定
pH值测定:每个样品重复3次,结果取算术平均值[18]。
可滴定酸度测定:参照李娇洋等[19]方法用NaOH标准溶液滴定,酚酞指示滴定终点,通过消耗的NaOH体积计算出可滴定酸含量。以乳酸计(TA,g/L),如公式(1)所示:
(1)
式中:C1,NaOH标准溶液的浓度,mol/L;Vd1,用于滴定的样品体积,mL;V1,滴定消耗的NaOH溶液体积,mL;V0,空白试验消耗的NaOH溶液体积,mL;0.090:乳酸的换算系数。
NaCl的质量浓度测定:用0.1 mol/L NaOH溶液滴定至出现浅红色,加入1 mL 10% K2CrO4溶液,边摇边滴加0.1 mol/L AgNO3标准溶液,溶液变为橙黄色保持1 min不褪色即为终点[17]。结果用氯化物的质量浓度(S,g/L)表示,计算公式(2)如下:
(2)
式中:0.035 5,与1.00 mL AgNO3标准溶液[c(AgNO3)=1.000 mol/L]相当的氯的质量,g;C2,AgNO3标准溶液的浓度,mol/L;Vd2:用于滴定的样品体积,mL;V2,滴定样品时消耗的AgNO3标准溶液体积,![]() 空白试验消耗的AgNO3标准溶液体积,mL。
空白试验消耗的AgNO3标准溶液体积,mL。
1.4.2 榨菜腌制汁液样品基因组DNA的提取
取榨菜腌制水10 mL于离心管中,7 000 r/min离心10 min去上清,沉淀菌体用深加工食品DNA提取试剂盒提取宏基因组DNA,用超微量分光光度计测定DNA浓度,并且贮存于-20 ℃备用。
1.4.3 标准质粒构建过程
L.plantarum ATCC 8014用MRS肉汤培养,用细菌基因组DNA抽提试剂盒提取基因组DNA。S. cerevisiae ATCC 9763用YM肉汤培养,用酵母基因组DNA提取试剂盒提取基因组DNA。
乳杆菌基因组DNA用引物8F和798R进行PCR扩增,用普通琼脂糖凝胶DNA回收试剂盒纯化PCR产物,与载体pMD19-T连接,转化到E.coli JM109感受态细胞中,于SOC液体培养基中培养,取适量菌液涂布于含氨苄青霉素(100 g/L)、IPTG(20 g/L)、X-gal(50 g/L)的LB平板上,37 ℃培养24 h,用蓝白斑法筛选阳性克隆[19-20]。酵母菌基因组DNA用引物Y1和Y2进行ITS的PCR扩增,同法构建重组质粒。
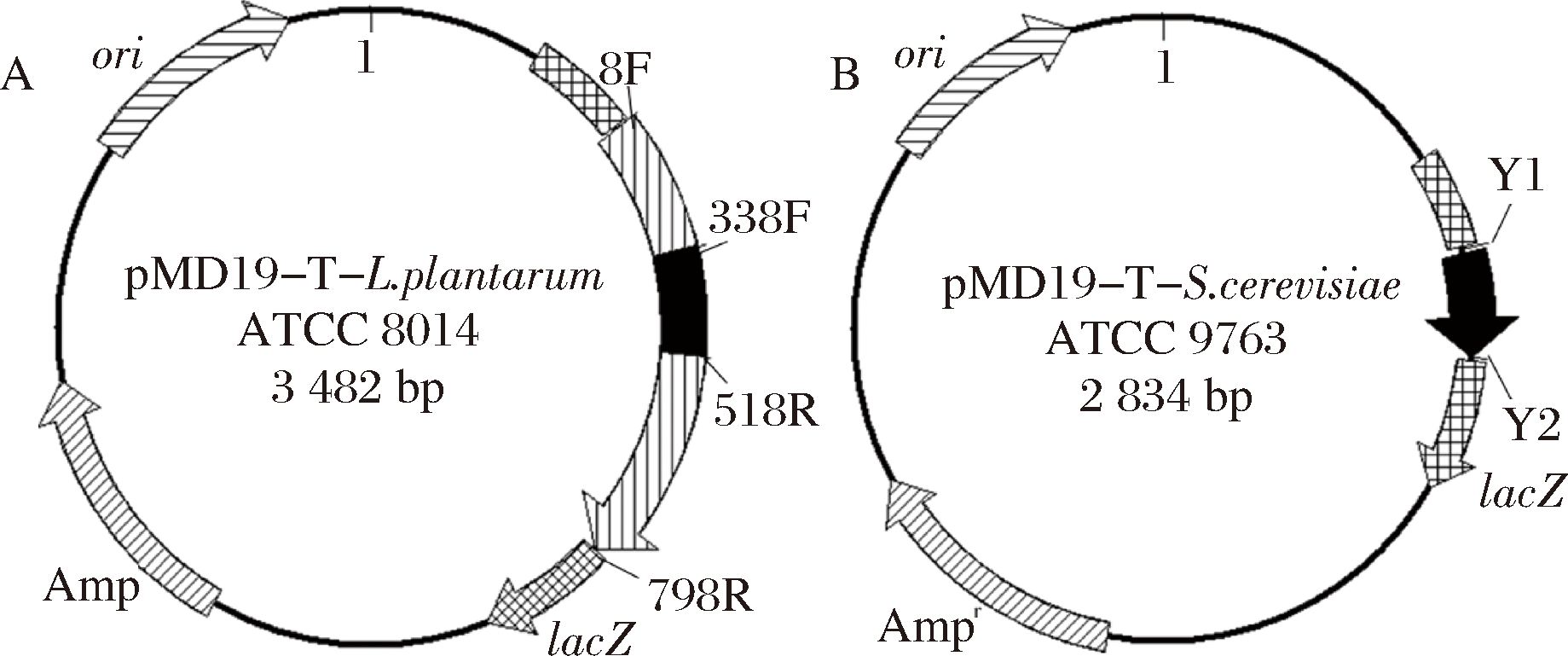
A-pMD19-T-L. plantarum重组质粒;B-pMD19-T-S. cerevisiae重组质粒
图1 重组质粒构建模拟模型
Fig.1 Construction simulation of recombinant plasmid
挑取白色阳性克隆菌落,用LB液体培养基培养,提取质粒,分别用338F、518R和Y1、Y2引物做PCR扩增验证乳杆菌、酵母标准菌株构建的质粒,PCR反应体系25 μL:2×PCR buffer 12.5 μL,质粒模板2 μL,10 μmol/L引物各0.8 μL,ddH2O 8.9 μL。以ddH2O代替质粒作为阴性对照。反应条件:94 ℃预变性3 min;94 ℃、30 s,56 ℃、30 s,72 ℃、30 s,扩增40个循环;最后72 ℃延伸10 min。将阴性对照、质粒扩增产物、pMD19-T质粒(空载)、重组质粒一起点样电泳,通过条带的相对位置验证插入序列。
经检验含有适当插入序列的质粒送生工生物工程(上海)股份有限公司用M4和RV引物测序验证。将测出的序列与插入序列输入ClustalW网页(https://www.genome.jp/tools-bin/clustalw)做序列比对,比对文件输入ENDscript/ESPript网页(http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)生成序列比对图。
1.4.4 qPCR标准曲线的构建
用超微量分光光度计测重组质粒的浓度,按公式(3)计算拷贝数(copies/μL):
拷贝数![]()
(3)
式中:DNA浓度单位为ng/μL;DNA长度单位为bp(乳杆菌重组质粒为3 842 bp,酵母重组质粒为2 834 bp)。
将重组质粒作10倍梯度稀释,稀释液作为DNA模板做qPCR扩增,细菌和真菌的反应体系(20 μL)均为:TB Green Premix Ex Taq Ⅱ 10 μL,DNA模板2 μL,10 μmol/L上下游引物各0.5 μL,ddH2O 7 μL。细菌的引物为338F和518R,扩增程序为:94 ℃预变性45 s;94 ℃、20 s,60 ℃、30 s,72 ℃、30 s,扩增45个循环;最后72 ℃延伸10 min。真菌的引物为Y1和Y2,扩增程序为:94 ℃预变性45 s;94 ℃、15 s,58 ℃、 15 s,72 ℃、15 s,扩增45个循环;最后72 ℃延伸5 min。在扩增结束时设置熔解曲线步骤以确定产物的特异性。每个实验重复5次。
根据Ct值与质粒拷贝数构建标准曲线,评价标准曲线的线性系数、线性范围和扩增效率[21]。扩增效率(E)与标准曲线的斜率(K)相关,如公式(4)所示:
(4)
1.4.5 榨菜腌制汁液中细菌和真菌数量的检测
采用建立的qPCR反应方法检测腌制汁液样品中的细菌和真菌的数量,3次重复。
1.5 数据处理
采用Excel 2016软件进行数据的处理和分析,结果采取均值±标准差形式。用Origin 2017软件进行数据作图。
2 结果与讨论
2.1 榨菜腌制汁液理化指标的变化过程分析
榨菜的3次腌制采用不同的NaCl的质量浓度。第1次腌制时,青菜头拌盐后,随着菜块中的汁液在食盐的高渗作用下流出和盐的溶解过程,腌制汁液的NaCl的质量浓度从8.99 g/L上升到17.13 g/L,pH值由6.72快速下降到5.08,酸度从0.06 g/L增加到1.82 g/L,表明这一时期微生物快速产酸。第2次腌制时,腌制水的NaCl的质量浓度从41.42 g/L逐渐降低到30.03 g/L,可能因为相对于第1次腌制时NaCl的质量浓度有所提高,菜块中大量水渗出使盐浓度稀释,因此呈现一个下降的过程,pH值从4.65缓慢增加到了5.24,可滴定酸从5.14 g/L增加到5.78 g/L,末期下降到3.44 g/L,表明这一时期微生物也发酵产酸,可能也因为菜块水渗出的稀释作用而使酸浓度略有下降。第3次腌制时,腌制水中NaCl的质量浓度从71.36 g/L下降到62.48 g/L,再增加到67.31 g/L,pH值持续下降,从5.04降到了4.43,可滴定酸度从6.15 g/L增加到7.00 g/L,再下降到3.82 g/L,表明尽管受到高盐浓度的抑制作用,微生物仍然继续发酵产酸。最后,3Y180d(成品)样品经过一定的脱盐步骤后,NaCl的质量浓度为36.23 g/L,pH值为4.24,可滴定酸度为3.91 g/L。
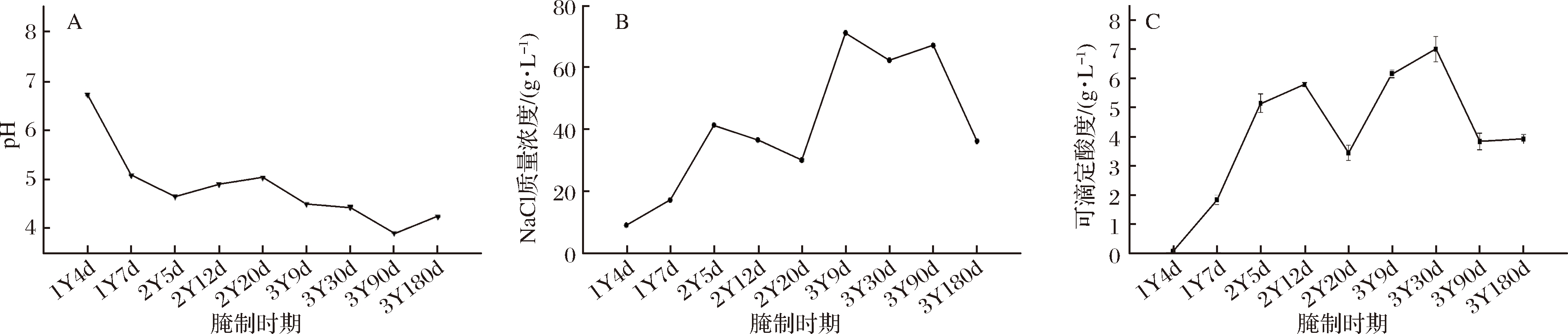
A-pH值;B- NaCl质量浓度;C-可滴定酸度
图2 榨菜腌制过程中理化指标的变化
Fig.2 Physical and chemical indicators in the curing process of Zhacai
2.2 标准质粒的构建和验证
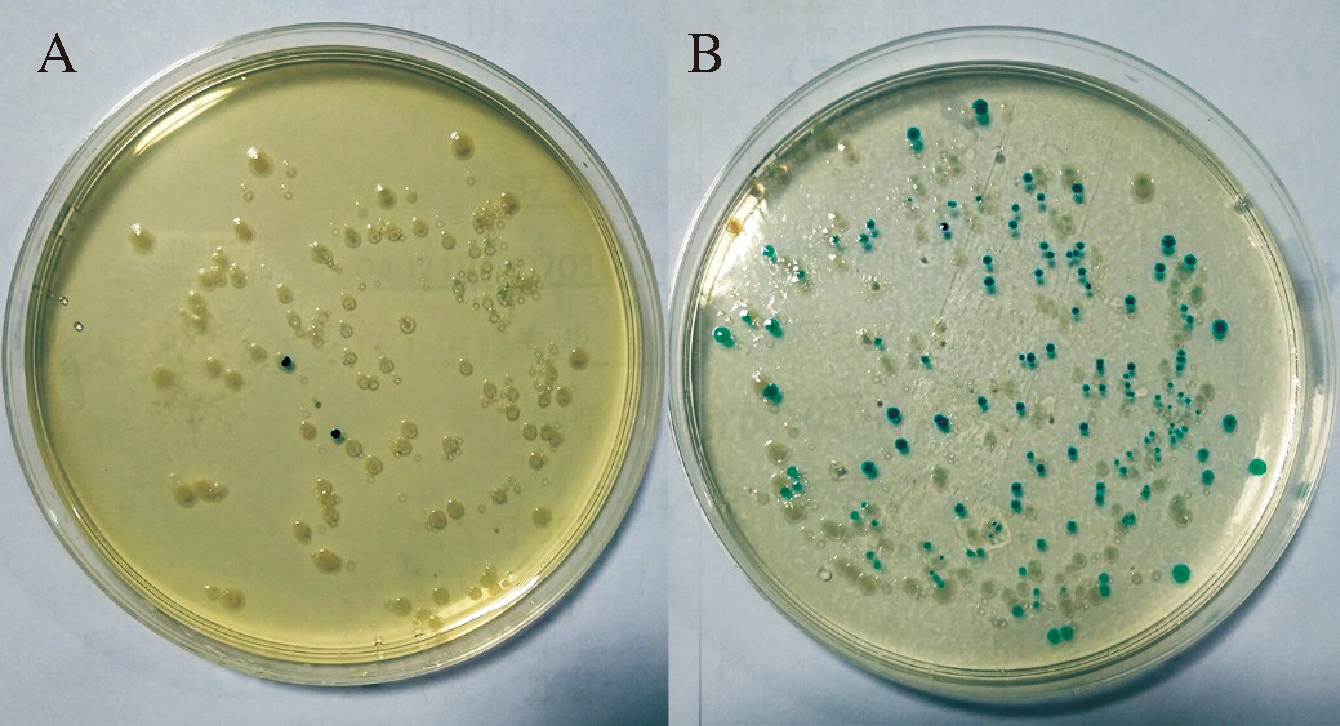
分别用标准菌株L. plantarum ATCC 8014的16S rRNA基因和S. cerevisiae ATCC 9763的ITS基因与质粒pMD19-T连接,转化大肠杆菌,用蓝白斑法筛选阳性克隆(图3)。图3-A中菌落几乎都是白色,而图3-B中蓝色菌落较多,表明乳杆菌重组质粒的连接转化效率较高。
重组质粒的PCR扩增和电泳验证实验结果如图4所示。阴性PCR扩增没有条带,随机挑选的3个乳杆菌重组质粒扩增均在180 bp处显示条带,重组质粒(第6泳道)相对于空载质粒(第5泳道)处于凝胶中分子质量稍低的位置,这是由于pMD19-T空载质粒是开环线性的,在凝胶中泳动受到的空间位阻较大,而重组质粒是闭合环状的,在凝胶中的空间位阻较小。酵母菌重组质粒的电泳图与乳杆菌相似,其阳性扩增条带位于150 bp。
A-pMD19-T-L. plantarum重组质粒转化的JM109;B-pMD19-T-S. cerevisiae重组质粒转化的JM109
图3 蓝白斑筛选重组质粒
Fig.3 The recombinant clones were screened by white-blue plaque selection method

A-pMD19-T-L. plantarum重组质粒验证;B-pMD19-T-S. cerevisiae重组质粒验证
图4 PCR和电泳验证重组质粒
Fig.4 PCR and electrophoretic profile of recombinant plasmid
注:M-DL2 000 DNA Ladder Marker;1-阴性对照;2、3、4-重组质粒PCR产物;5-pMD19-T质粒(空载);6-pMD19-T重组质粒。
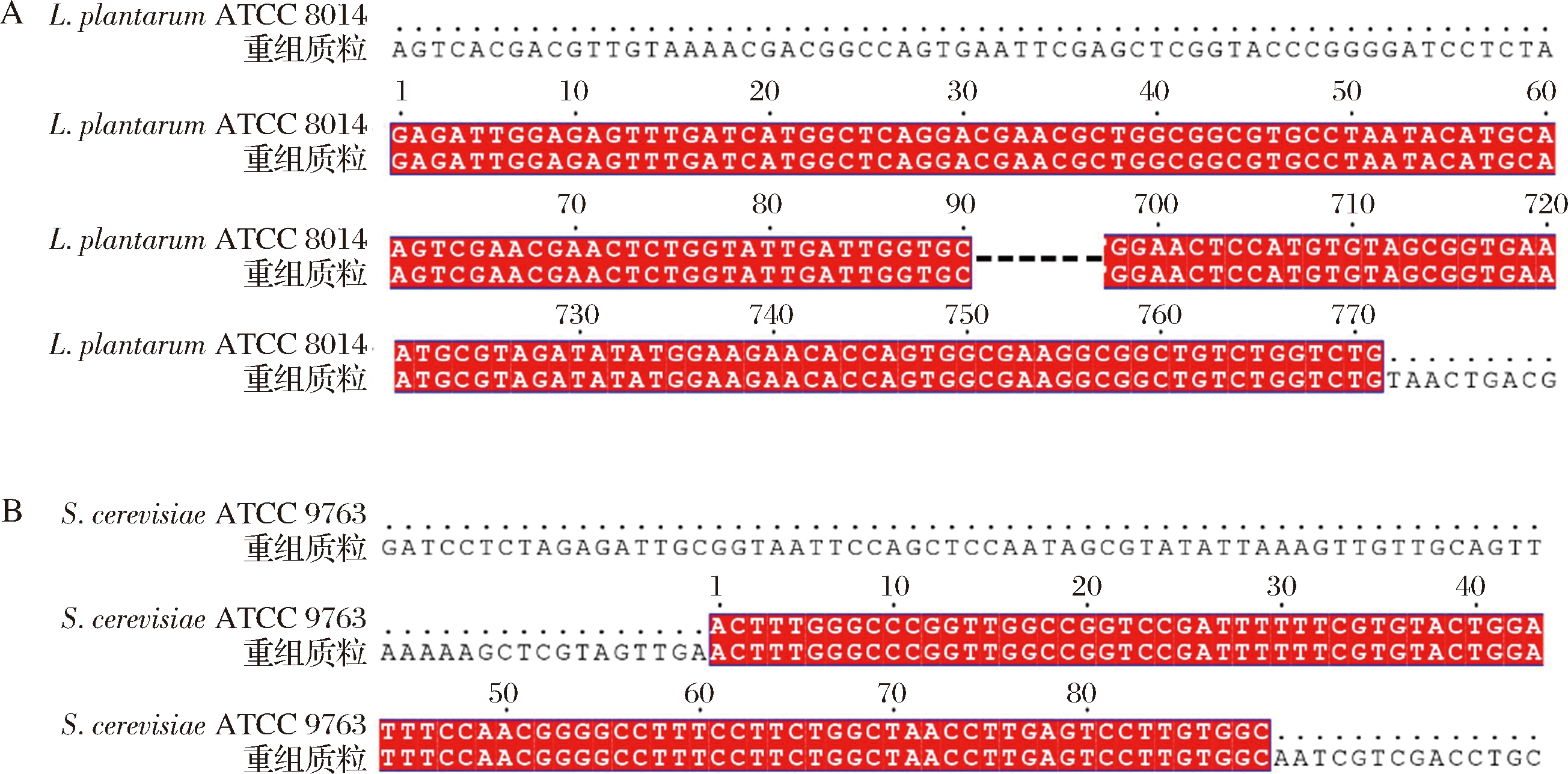
乳杆菌重组质粒与L. plantarum ATCC 8014的16S rRNA基因、酵母菌重组质粒与S. cerevisiae ATCC 9763的ITS基因序列比对的相似性都为100%,如图5所示,进一步证明目标序列正确插入质粒载体中。
2.3 qPCR定量检测方法的建立
将乳杆菌重组质粒做10倍梯度稀释后用于qPCR扩增,基线平整,扩增曲线呈S型,熔解曲线为单一熔解峰(图6-A、图6-B),说明无引物二聚体及非特异性产物产生,标准曲线为Ct=-3.408 7lgC+35.793,相关系数为R2=0.999 9,扩增效率E=96.5%,线性范围是3.64×104~3.62×109 copies/μL。酵母菌重组质粒的qPCR扩增曲线也呈S型,熔解曲线为单峰(图6-C、图6-D),标准曲线为Ct=-3.227 8lgC+35.294,相关系数为R2=0.998 9,扩增效率E=104.1%,线性范围是7.07×102~7.07×109 copies/μL。FREDLUND等[22]和SUANTHIE等[23]报道,可以根据如下标准判断qPCR标准曲线是否构建成功:斜率在-3.1~-3.6,PCR扩增效率在80%~110%,R2≥0.98。用乳杆菌、酵母菌重组质粒构建的标准曲线均符合上述标准,可分别应用于样品中细菌、真菌数量的检测。
A-pMD19-T-L. plantarum重组质粒验证;B-pMD19-T-S. cerevisiae重组质粒验证
图5 重组质粒基因序列比对验证
Fig.5 Comparison and validation of recombinant plasmid gene sequence
2.4 榨菜腌制汁液中细菌和真菌数量的检测
采用建立的qPCR方法检测腌制汁液样品中细菌和真菌的数量,结果如图7所示。
第1次腌制时,细菌数量从109.28 copies/μL上升到1010.43 copies/μL,这一时期细菌快速增殖。真菌的数量一直保持于106 copies/μL。第2次腌制时,细菌的初始数量为108.10 copies/μL,细菌数量的急剧降低可能是由于菜块转池并且用量盐提高,此后细菌数量逐渐增加至109.11 copies/μL,表明在较高NaCl的质量浓度下细菌以比较缓慢的速度增殖。真菌数量最初保持于106 copies/μL,第20天时急剧下降至105.29 copies/μL,可能部分真菌对盐耐受性较低而被抑制。第3次腌制时,细菌的初始数量为109.49 copies/μL,先升高至1010.13 copies/μL,然后略有下降,最终稳定至109.92 copies/μL,在较高NaCl的质量浓度下细菌仍然缓慢增殖并且数量保持在较高的水平。真菌数量从105.97 copies/μL稳步增加至107.75 copies/μL,最终3Y180d(成品)为107.26copies/μL。
在整个腌制过程中,细菌数量高于真菌数量,3个腌制时期都有明显的数量增殖过程,表明细菌对于发酵过程是最主要的贡献者。第3次腌制时真菌才有明显的数量增殖过程,表明真菌在后熟阶段也发挥了重要的作用。细菌数量在1Y7d、2Y20d达到较高水平时,真菌数量均降低,细菌数量在2Y5d、3Y90d较低时,真菌数量略有上升,由此可推测细菌增殖可能对真菌有一定的抑制作用。
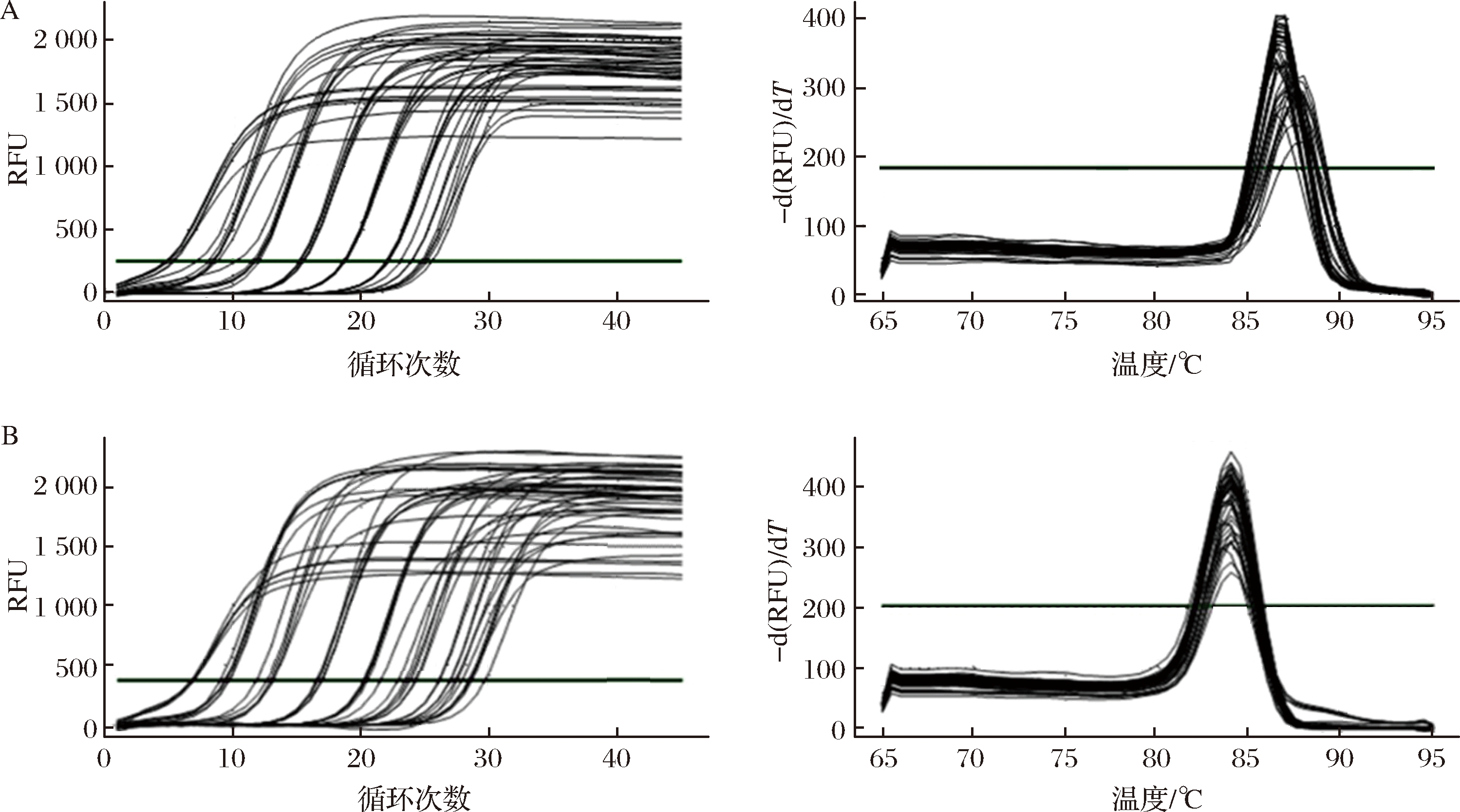
A、B-L. plantarum重组质粒的扩增曲线和熔解曲线;C、D-S. cerevisiae重组质粒的扩增曲线和熔解曲线
图6 重组质粒的qPCR扩增曲线和熔解曲线
Fig.6 Amplification curve and melting curve analysis of PCR products of bacterial and fungi

图7 榨菜腌制过程中细菌和真菌数量变化过程
Fig.7 The change of the quantity of bacteria and fungi during the curing process
本研究用qPCR法检测出第3次腌制期间细菌、真菌的数量都比较高,在腌制末期(3Y90d、3Y180d)仅仅略微降低,而现有的文献报道,后熟末期(60~180 d)细菌从106 个/g下降至102个/g[5],或者从105个/g下降至103个/g[6]。结果的差异可能是因为腌制末期盐浓度很高,检测培养基与腌制汁液环境差异较大,不适宜部分微生物的生长[24],qPCR技术是一种免培养技术,不受微生物可培养性的影响。
3 结论
榨菜是我国著名的传统酱腌制品,其独特的风味和品质深受人们的喜爱。微生物发酵作用对其风味品质的形成有着重要的影响。本研究构建了qPCR检测方法,检测榨菜不同腌制时期腌制汁液中的细菌、真菌数量,探讨了3个腌制阶段细菌、真菌数量变化过程。细菌的数量变化范围是108.10~1010.43 copies/μL,在3个腌制阶段中细菌都有明显的数量增殖过程;真菌的数量变化范围是105.29~107.75 copies/μL,仅在第3次腌制期间有明显的数量增殖过程。同时分析了腌制过程中NaCl的质量浓度、pH值、酸度的变化过程。本研究采用qPCR方法,反映榨菜腌制汁液中所有可培养和不可培养微生物的数量,为进一步研究不同菌属在榨菜发酵中所起到的作用提供一定的理论基础,同时也对榨菜腌制工艺改进、提高过程控制水平具有重要的参考意义。
[1] 庞杰,向珣.涪陵榨菜的加工及生产现状[J].长江蔬菜, 1999(2): 42-44.
[2] 苏扬,张聪,王朝辉.榨菜加工工业及其发展战略的研究[J].食品科技,2010,35(4):114-119.
[3] 贺云川,周斌全,刘德君.涪陵榨菜传统工艺概述[J].食品与发酵科技, 2013,49(4):57-60.
[4] 王中凤,吴永娴,曾凡坤.榨菜风味形成机理及其影响因素[J].中国酿造, 1995, 14(1): 10-11.
[5] 袁美,韩彦慧,徐柯,等.机械脱水一次腌制榨菜在腌制过程中香气成分的变化[J].食品工业科技,2018,39(23):234-240.
[6] 阮祥林.涪陵榨菜腌制机理与味感[J].中国调味品, 1983(2): 9-12.
[7] 杨珺,吴永娴,曾凡坤.四川榨菜后熟时期微生物区系的研究[J].中国调味品, 2001(10): 18-20.
[8] 贾巍.榨菜后熟优势微生物分离鉴定及发酵剂研制[D].重庆:西南大学, 2008.
[9] 张先琴.PCR-DGGE技术分析传统发酵蔬菜中微生物群落结构[D].雅安:四川农业大学,2012.
[10] 李正国,付晓红,邓伟,等.传统分离培养结合DGGE法检测榨菜腌制过程的细菌多样性[J].微生物学通报,2009,36(3):371-376.
[11] 程海星,郭月英,任霆,等.实时荧光定量PCR技术原理及在食品检测中的应用[J].食品与发酵工业,2015,41(3):243-247.
[12] POSTOLLEC F, FALENTION H, PAVAN S, et al. Recent advances in quantitative PCR (qPCR) applications in food microbiology[J]. Food Microbiology, 2011, 28(5): 848-861.
[13] 张倩,叶可萍,关正萍,等.运用Real-time PCR方法建立大肠杆菌O157∶H7生长预测模型[J].中国食品学报,2017,17(9):203-212.
[14] 张亚豪,梁会朋,常聪,等.基于实时荧光定量PCR技术监测四川工业泡菜发酵过程中主要细菌的变化[J].中国调味品,2018,43(1):35-38;47.
[15] 王兴兴,丁梦帆,潘迎捷,等. 基于定量PCR方法初步分析西藏开菲尔粒中细菌与酵母菌数量变化[J]. 上海海洋大学学报, 2015, 24(4):625-631.
[16] 陶京兰,陆震鸣,王宗敏,等.实时荧光定量PCR监测镇江香醋醋酸发酵过程中微生物变化[J].食品与发酵工业,2013,39(2):156-160.
[17] SAMBROOK J, RUSSELL D W.分子克隆实验指南[M].北京:科学出版社,2002.
[18] YANG Jixia, CAO Jialu, XU Haiyan, et al. Bacterial diversity and community structure in Chongqing radish paocai brines revealed using PacBio single-molecule real-time sequencing technology [J]. Journal of the Science of Food and Agriculture, 2018, 98(9):3 234 - 3 245.
[19] 李娇洋,杨帆,包斌.叶类蔬菜贮藏中品质变化评价指标及其分析方法的研究进展[J].食品工业科技,2018,39(15):334-340.
[20] 许艺珊,何晶晶,陈婉娟,等.TA克隆技术的原理与应用[J].科技展望,2015,25(6):89-90.
[21] 王小花,李建祥,王国卿,等.SYBR Green实时荧光定量PCR检测大豆转基因成分[J].食品科学,2009,30(8):171-176.
[22] FREDLUND E, GIDLUND A, OLSEN M, et al. Method evaluation of Fusarium DNA extraction from mycelia and wheat for down-stream real-time PCR quantification and correlation to mycotoxin levels[J]. Journal of Microbiological Methods, 2008, 73(1):33-40.
[23] SUANTHIE Y, COUSIN M A, WOLOSHUK C P. Multiplex real-time PCR for detection and quantification of mycotoxigenic Aspergillus, Penicillium and Fusarium[J]. Journal of Stored Products Research, 2009, 45(2):139-145.
[24] 王香君,李梦茹,段杉.实时荧光定量PCR法定量微生物的条件及在鱼露和虾油微生物检测中的应用研究[J].食品与发酵工业,2018,44(2):194-201.