人体内某些蛋白质在特定病理条件下会发生错误折叠和聚集,最终在组织或器官内形成不溶性淀粉样蛋白沉积,进而引发如阿尔兹海默病(Alzheimer’s disease,AD),帕金森综合症和II型糖尿病等疾病[1]。AD是以进行性认知功能障碍和行为损害为特征的神经退行性疾病,多发生在老年及老年前期。目前,AD严重影响着世界上数以千万计的患者,对家庭和社会造成了极大的负担[2]。AD的病因复杂,其中淀粉样级联假说是目前AD发病机制的主流学说,即淀粉样β蛋白(Aβ)在脑内的错误折叠和聚集是AD发生发展的关键因素之一[3-4]。Aβ是由β-分泌酶和γ-分泌酶依次水解淀粉样前体蛋白APP产生的多肽片段,一般含有39~43个氨基酸。大量证据表明Aβ42的聚集性和神经毒性最强,在AD的发病过程中扮演着更关键的角色[5-10]。有研究报道指出,患者脑脊液内Aβ42的浓度在纳摩尔每升水平[11-12]。纳摩尔到微摩尔每升的低浓度水平上可溶性Aβ寡聚体对细胞轴突和突触已存在明显毒性作用[13],表明Aβ42在较低浓度时便能在体内发生聚集。因此,筛选Aβ42聚集抑制剂成为开发抗AD药物的研究热点。
开发快速、简便和准确的Aβ42聚集抑制剂筛选方法,对于开发抗聚集药物至关重要。硫磺素T(thioflavin T,ThT)荧光染色法是目前鉴定淀粉样蛋白聚集抑制剂的“黄金方法”[14],其基本原理是,当ThT分子结合到富含β-折叠的淀粉样纤维上时,在特定激发波长下会发出较强荧光。根据特定波长下样品的荧光强度来判断淀粉样蛋白纤维的含量,进一步分析受试化合物的聚集抑制效果[8, 15-16]。然而该方法存在工作量大、效率低、假阳性高等缺陷。另外,由于体内外生理环境的巨大差异,体外检测获得的抑制剂分子在体内可能难以发挥有效的聚集抑制作用[17-19]。
前期研究表明,将易于错误折叠和聚集的蛋白质基因插入到报告蛋白的合适位置,报告蛋白的活性将依赖于插入蛋白的错误折叠或聚集状态[20]。TEM1-β-内酰胺酶(TEM1-β-lactamase, βlac)是一种常用的报告蛋白,βlac位于革兰氏阴性菌的细胞周质中,分为αβ和α两个结构域,这2种结构域相互作用,在表面形成一个底物结合裂口[21-23]。研究表明,βlac的196位氨基酸残基位于αβ和α结构域之间,βlac对该位置附近发生的插入或缺失特定片段具有耐受性[21, 23-26]。基于上述特点,将受试蛋白插入到βlac的196和197残基之间,构建βlac-受试蛋白三联体融合体系,可将蛋白质稳定性与宿主菌对抗生素的抗性有效联系起来[27]。目前,基于βlac的三联体融合体系目前已广泛应用于优化蛋白质的体内折叠和体内识别小分子聚集抑制剂等[20-21, 28-29]。本文将Aβ42多肽插入到βlac的196和197残基之间,构建大肠杆菌体内βlac-Aβ42筛选体系(图1),并利用已知聚集抑制剂,结合3种检测方法对该系统进行验证(图2)。

A-βlac;B-βlac-Aβ42
图1 βlac和βlac-Aβ42融合体系示意图
Fig.1 Schematic diagram of βlac and βlac-Aβ42 fusion systems
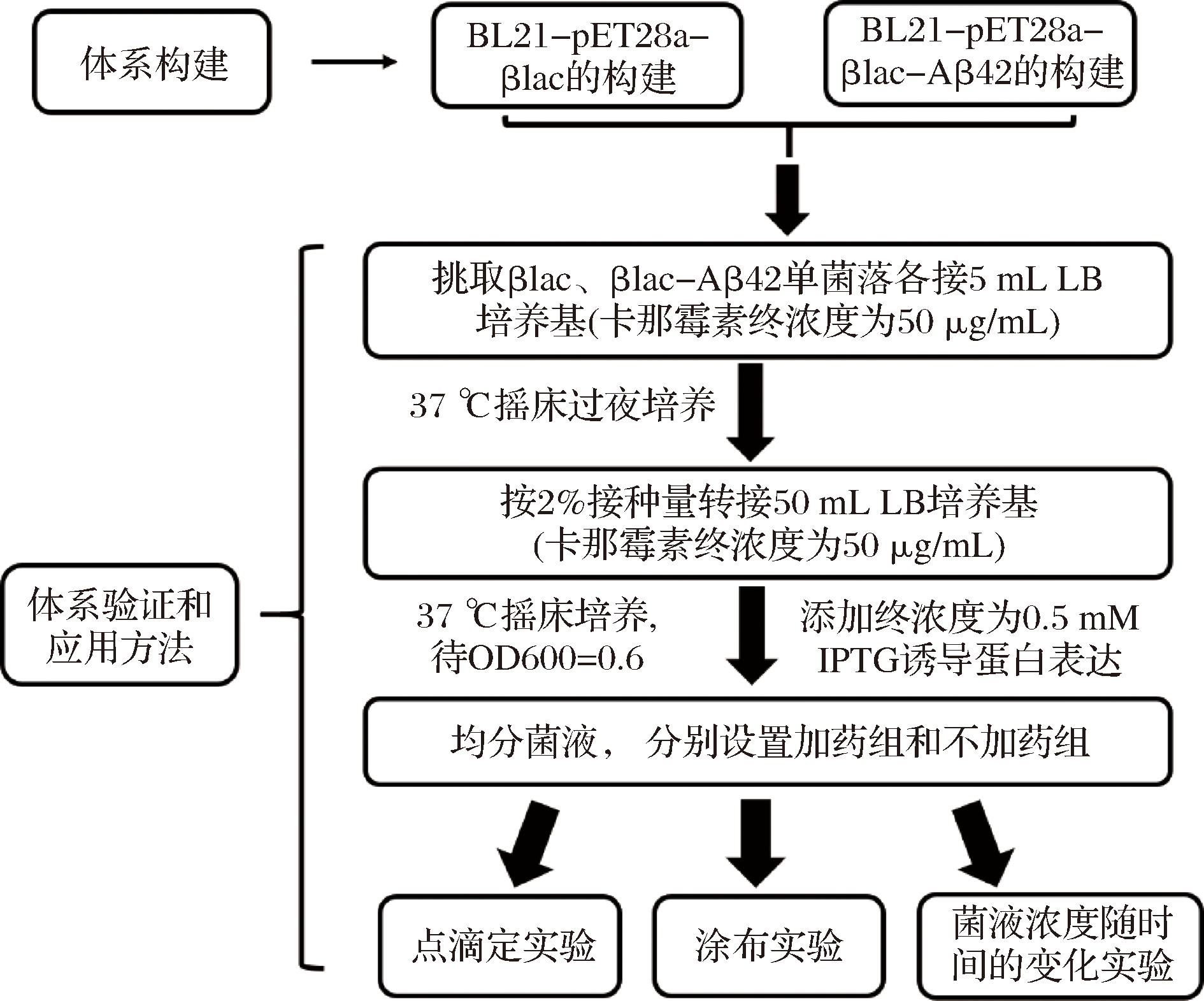
图2 筛选体系构建流程图
Fig.2 Flow chart of the construction of the inhibitor screening system
1 材料与方法
1.1 材料与试剂
大肠杆菌JM109、BL21(DE3),pMD19T(Simple)克隆载体和pET28a表达载体质粒保存于本实验室;高保真DNA聚合酶、T4 DNA连接酶及限制性内切酶Nde I、EcoR I、Xho I、BamH I,大连宝生物公司;质粒小量提取试剂盒,北京索来宝科技有限公司;姜黄素(curcumin)、EGCG、固绿(fast green),上海源叶生物科技有限公司;其他未注明试剂均为分析纯,上海生工生物工程股份有限公司。
1.2 仪器与设备
Mastercycler nexus PCR仪,德国eppendorf公司;ZXGP-B2080隔水恒温箱、ZHJH-C1106C超净工作台,上海智城分析仪器制造有限公司;Multifuge X1R高速冷冻离心机、NanoDrop 2000蛋白核酸定量仪,美国ThermoFisher Scientific公司;Infinite 200PRO多功能酶标仪,奥地利TECAN公司。
1.3 实验方法
1.3.1 DNA片段、引物合成,pET28a-βlac、pET28a-βlac-Aβ42表达载体的构建
根据βlac蛋白氨基酸序列,密码子优化为偏好大肠杆菌表达的DNA序列。DNA合成及测序由苏州金唯智生物科技有限公司完成,引物由深圳华大基因公司合成,DNA及引物序列见表1。
βlac DNA序列以pUC57-βlac形式合成,Nde I和EcoR I双酶切pUC57-βlac质粒获得βlac片段,与具有相同黏性末端的pET28a载体框架于16 ℃连接4 h,转化JM109大肠杆菌感受态细胞,37 ℃培养12 h后,挑取阳性转化子进行菌落PCR验证,并提取质粒送至金唯智基因公司进行测序鉴定,将鉴定正确的质粒命名为pET28a-βlac。
以Nde I-βlac-F,EcoR I-βlac-R为引物,通过PCR技术,获得含有酶切位点Nde I/EcoR I的目的片段βlac。参考上述分子生物学方法,将βlac与T载体片段连接,构建T-βlac。用Xho I和BamH I同时酶切质粒T-βlac和Aβ42基因,以相同方法构建T-βlac-Aβ42质粒。Nde I和EcoR I酶切质粒T-βlac-Aβ42,回收目的片段与含有相同黏性末端的pET28a载体连接并转化大肠杆菌JM109,最终获得表达载体pET28a-βlac-Aβ42。
1.3.2 斑点滴定实验[21, 23]
将pET28a-βlac、pET28a-βlac-Aβ42转化大肠杆菌BL21(DE3),获得重组工程菌BL21-βlac,BL21-βlac-Aβ42。挑取单菌落至5 mL LB培养基中,37 ℃过夜培养,按体积分数2%的接种量转接至5 mL新鲜LB培养基中,37 ℃培养至OD600为0.6,添加终浓度0.5 mmol/L异丙基-β-D-硫代半乳糖苷(IPTG)进行诱导,37 ℃继续孵育4 h,用无菌LB培养基将BL21-βlac,BL21-βlac-Aβ42菌液调至OD600=0.7,然后用170 mmol/L NaCl 10倍递增连续稀释。取3 μL稀释液点滴定至LB固体培养基(卡那霉素50 μg/mL,IPTG 0.5 mmol/L),其中含有终浓度100 μmol/L小分子药物以及不同浓度的氨苄青霉素(ampicillin, Amp)。将平板置于37 ℃孵育18 h,观察各Amp浓度下允许菌体生长的最大细胞稀释度。
表1 βlac、βlac-Aβ42基因序列及PCR反应所需引物序列
Table 1 Sequence of βlac and βlac-Aβ42, and the primers used in the PCR reaction

name5′-3′βlacATGAGTATTCAACATTTCCGTGTCGCCCTTATTCCCTTTTTTGCGGCATTTTGCCTTCCTGTTTTTGCTCACCCAGAAACGCTGGTGAAAGTAAAAGATGCTGAAGATCAGTTGGGTGCACGAGTGGGTTACATCGAACTGGATCTCAACAGCGGTAAGATCCTTGAGAGTTTTCGCCCCGAAGAACGTTTTCCAATGATGAGCACTTTTAAAGTTCTGCTATGTGGCGCGGTATTATCCCGTGTTGACGCCGGGCAAGAGCAACTCGGTCGCCGCATACACTATTCTCAGAATGACTTGGTTGAGTACTCACCAGTCACAGAAAAGCATCTTACGGATGGCATGACAGTAAGAGAATTATGCAGTGCTGCCATAACCATGAGTGATAACACTGCGGCCAACTTACTTCTGACAACGATCGGAGGACCGAAGGAGCTAACCGCTTTTTTGCACAACATGGGGGATCATGTAACTCGCCTTGATCGTTGGGAACCGGAGCTGAATGAAGCCATACCAAACGACGAGCGTGACACCACGATGCCTGCAGCAATGGCAACAACGTTGCGCAAACTATTAACTGGCGAACTAGGTGGTGGTGGTTCTGGTGGTGGTGGCTCGAGCTCAGGATCCGGGAGCGGTTCCGGAAGCGGAGGAGGTGGTTCAGGCGGAGGTGGAAGCTTGACTCTAGCTAGCCGGCAGCAGCTCATAGACTGGATGGAGGCGGATAAAGTTGCAGGACCACTTCTGCGCTCGGCCCTTCCGGCTGGCTGGTTTATTGCTGATAAATCTGGAGCCGGTGAGCGTGGGTCTCGCGGTATCATTGCAGCACTGGGGCCAGATGGTAAGCCCTCCCGTATCGTAGTTATCTACACGACGGGGAGTCAGGCAACTATGGATGAACGAAATAGACAGATCGCTGAGATAGGTGCCTCACTGATTAAGCATTGGTAAβlac-Aβ42ATGAGTATTCAACATTTCCGTGTCGCCCTTATTCCCTTTTTTGCGGCATTTTGCCTTCCTGTTTTTGCTCACCCAGAAACGCTGGTGAAAGTAAAAGATGCTGAAGATCAGTTGGGTGCACGAGTGGGTTACATCGAACTGGATCTCAACAGCGGTAAGATCCTTGAGAGTTTTCGCCCCGAAGAACGTTTTCCAATGATGAGCACTTTTAAAGTTCTGCTATGTGGCGCGGTATTATCCCGTGTTGACGCCGGGCAAGAGCAACTCGGTCGCCGCATACACTATTCTCAGAATGACTTGGTTGAGTACTCACCAGTCACAGAAAAGCATCTTACGGATGGCATGACAGTAAGAGAATTATGCAGTGCTGCCATAACCATGAGTGATAACACTGCGGCCAACTTACTTCTGACAACGATCGGAGGACCGAAGGAGCTAACCGCTTTTTTGCACAACATGGGGGATCATGTAACTCGCCTTGATCGTTGGGAACCGGAGCTGAATGAAGCCATACCAAACGACGAGCGTGACACCACGATGCCTGCAGCAATGGCAACAACGTTGCGCAAACTATTAACTGGCGAACTAGGTGGTGGTGGTTCTGGTGGTGGTGGCTCGAGCGATGCCGAGTTTCGCCATGATAGCGGCTATGAGGTGCACCACCAGAAACTGGTGTTCTTTGCCGAGGATGTGGGCAGCAACAAAGGCGCCATTATTGGCCTGATGGTGGGTGGCGTGGTGATTGCCGGATCCGGGAGCGGTTCCGGAAGCGGAGGAGGTGGTTCAGGCGGAGGTGGAAGCTTGACTCTAGCTAGCCGGCAGCAGCTCATAGACTGGATGGAGGCGGATAAAGTTGCAGGACCACTTCTGCGCTCGGCCCTTCCGGCTGGCTGGTTTATTGCTGATAAATCTGGAGCCGGTGAGCGTGGGTCTCGCGGTATCATTGCAGCACTGGGGCCAGATGGTAAGCCCTCCCGTATCGTAGTTATCTACACGACGGGGAGTCAGGCAACTATGGATGAACGAAATAGACAGATCGCTGAGATAGGTGCCTCACTGATTAAGCATTGGTAANdeI-βlac-FGGAATTATATGAGTATTAAATTTEcoRI-βlac-RGGAATTTTAAATGTTAATAGTXhoI-Aβ42-FCCGCTCGAG CGATGCCGAGTTTCGCCATBamHI-Aβ42-RCGGGATCC GGCAATCACCACGCCACC
1.3.3 平板涂布实验
融合蛋白的诱导表达方法同1.3.2,37 ℃诱导4 h后,用无菌LB培养基将OD600调至0.7,并用170 mmol/L NaCl 10倍递增连续稀释,吸取50 μL各稀释液均匀涂布于含有卡那霉素、IPTG、100 μmol/L小分子药物和不同浓度Amp的6孔板固体培养基中,37 ℃培养箱恒温孵育18 h。
1.3.4 菌体浓度检测
融合蛋白的诱导表达方法同1.3.2,37 ℃诱导4 h后,用无菌LB培养基将OD600调至0.7。于96孔板中分别加入200 μL的目的菌液,同时加入卡那霉素(终浓度50 μg/mL),IPTG (终浓度0.5 mmol/L),小分子药物(终浓度100 μmol/L)以及不同浓度的Amp,37 ℃恒温振荡培养,于不同的时间点检测菌液浓度变化。
2 结果与分析
2.1 BL21-pET28a-βlac、BL21-pET28a-βlac-Aβ42表达载体的构建
根据βlac氨基酸序列,对其基因序列进行大肠杆菌密码子偏好性优化,获得完整的βlac DNA序列(表1)。将βlac与pET28a表达载体框架连接,构建pET28a-βlac表达载体质粒(图3-A)。转化大肠杆菌JM109,转化子经菌落PCR验证。结果如图3-C所示,4个转化子PCR产物与βlac基因理论大小一致(951 bp)。同时,采用Nde I和EcoR I双酶切验证pET28a-βlac是否构建成功,结果如图3-E (泳道1和泳道2)所示,2个转化子酶切产物与pET28a和βlac基因理论大小一致(5 369 bp和951 bp)。基因测序结果表明目标基因序列未发生突变,证明pET28a-βlac表达载体质粒构建成功。

A-pET28a-βlac载体示意图;B-pET28a-βlac-Aβ42载体示意图;C-菌落PCR验证pET28a-βlac转化Jm109转化子: M-1 kb marker; 1~4-4个pET28a-βlac转化Jm109转化子;D-菌落PCR验证pET28a-βlac-Aβ42转化Jm109转化子: M-1 kb marker; 1~2-2个pET28a-βlac-Aβ42转化Jm109转化子; 3-含有pET28a-βlac的对照菌;E-双酶切验证pET28a-βlac和pET28a-βlac-Aβ42转化Jm109转化子: M-1 kb marker; 1~2-2个pET28a-βlac转化Jm109转化子; 3~5-3个pET28a-βlac-Aβ42转化Jm109转化子
图3 表达载体pET28a-βlac/βlac-Aβ42的构建和验证
Fig.3 Construction and identification of pET28a-βlac/βlac-Aβ42
pET28a-βlac-Aβ42表达载体如图3-B所示。菌落PCR验证结果如图3-D所示,其中2个转化子(泳道1和泳道2) PCR产物大小与βlac-Aβ42基因理论大小一致(1 074 bp)。进一步双酶切验证,可观察到与理论大小一致的pET28a (5 369 bp)片段和βlac-Aβ42 (1 074 bp)片段,如图3-E中泳道3、4和5所示。结合测序验证结果证明表达载体pET28a-βlac-Aβ42构建成功。
2.2 βlac和βlac-Aβ42蛋白的表达
重组质粒pET28a-βlac、pET28a-βlac-Aβ42转化大肠杆菌BL21(DE3)感受态细胞中,挑取单克隆菌株并用IPTG诱导表达。SDS-PAGE分析蛋白表达情况,由图4-A可以看出,与BL21-pET28a空白对照相比,BL21-βlac和BL21-βlac-Aβ42中均观察到明显的蛋白条带(泳道2、3),片段大小与βlac和βlac-Aβ42理论值一致(33.5和38.0 kDa),证明2种融合蛋白成功表达。
同时,分别检测了IPTG诱导后BL21-βlac和BL21-βlac-Aβ42的菌体浓度,结果如图4-B所示,2种重组菌株OD600值分别为1.297 5和0.736 5,表明BL21-βlac-Aβ42生长状况较差。造成该现象的原因可能是在βlac-Aβ42融合蛋白表达过程中,Aβ42的快速聚集导致βlac-Aβ42折叠发生改变,对Amp抗性降低,导致部分细胞死亡,菌体浓度下降。

A-pET28a-βlac和pET28a-βlac-Aβ42表达产物SDS-PAGE分析;M-蛋白Marker; 1-BL21-pET28a; 2-BL21-βlac; 3-BL21-βlac-Aβ42;B-BL21-βlac与BL21-βlac-Aβ42诱导结束后的菌液浓度
图4 βlac和βlac-Aβ42蛋白表达
Fig.4 Expression of the βlac and βlac-Aβ42
2.3 重组大肠杆菌菌株对氨苄青霉素耐受性分析
通过斑点滴定实验检测重组融合蛋白菌株对Amp的耐受能力,具体实验方法见1.3.2。为确定最佳Amp浓度,将Amp质量浓度设置为20,50和100 μg/mL。结果如图5-A所示,上述3种Amp浓度下,对照组BL21-βlac均有菌落生长,说明βlac蛋白诱导表达,使重组菌株BL21-βlac具有Amp抗性。与之相反,BL21-βlac-Aβ42未出现明显的菌落说明由于Aβ42的聚集阻碍了βlac的2个结构域的正常结合,导致重组BL21-βlac-Aβ42对Amp的敏感性增加,因此在同浓度抗性平板上无法正常生长。
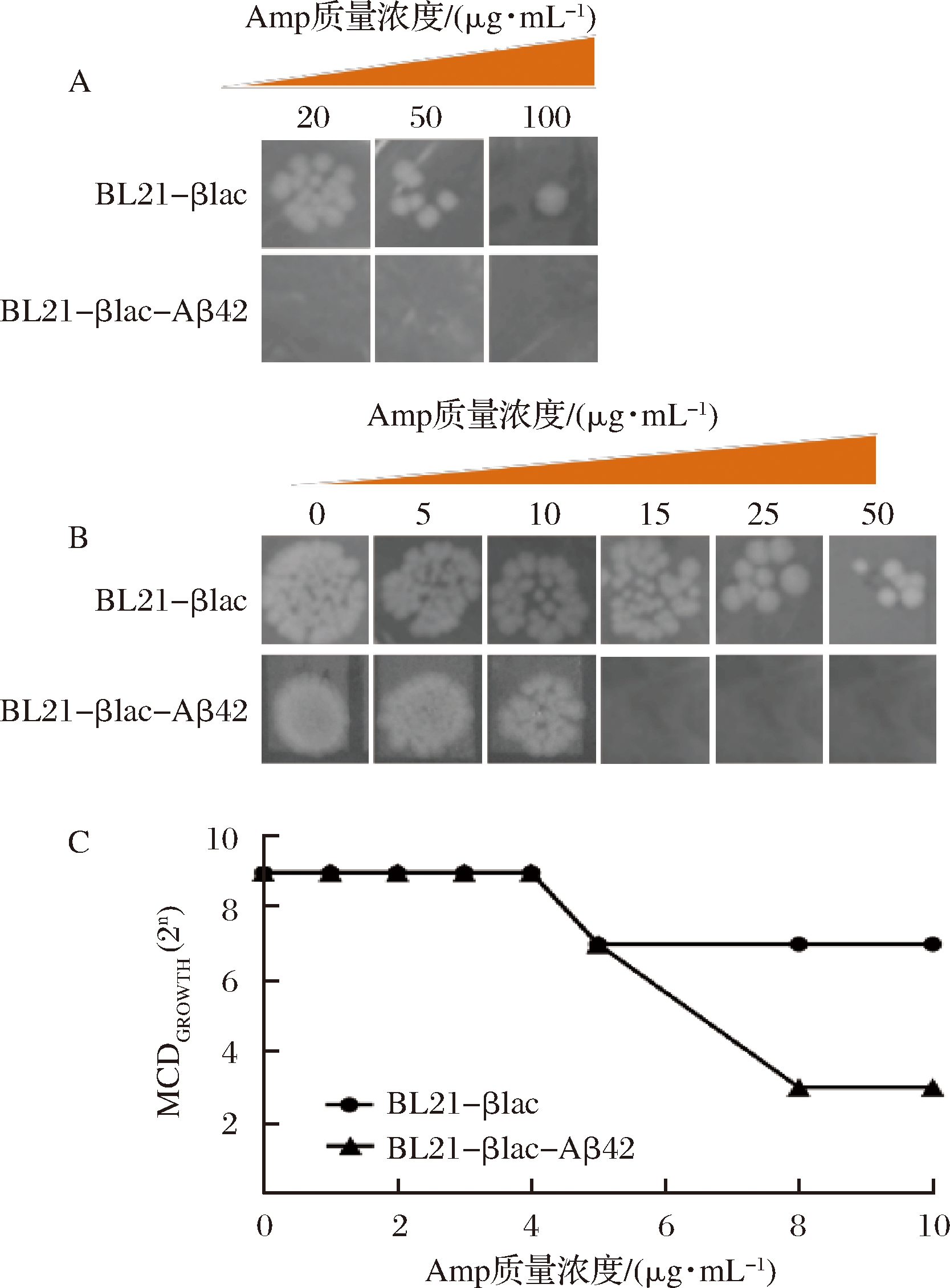
A-Amp质量浓度为20、50、100 μg/mL时,BL21-βlac和BL21-βlac-Aβ42菌落生长状况;B-Amp质量浓度为0、5、10、15、25、50 μg/mL时,BL21-βlac和BL21-βlac-Aβ42菌落生长状况;C-抗生素浓度与允许菌体生长的最大细胞稀释倍数(MCDGROWTH)之间的关系
图5 不同浓度Amp对BL21-βlac和BL21-βlac-Aβ42生长的影响
Fig.5 Effect of different concentrations of Amp on the growth of BL21-βlac and BL21-βlac-Aβ42
为确定BL21-βlac和BL21-βlac-Aβ42允许菌体生长的最大Amp浓度。将不同Amp浓度梯度0,5,10,15,25,50 μg/mL来进行斑点滴定实验。结果如图5-B所示,BL21-βlac在Amp质量浓度为50 μg/mL时仍能正常生长,而BL21-βlac-Aβ42在Amp质量浓度为15 μg/mL时菌落已不再生长。因此,选择在低Amp浓度范围(0~10 μg/mL)内,以允许菌体生长的最大细胞稀释倍数(MCDGrowth)来衡量2种重组菌株的生长情况(图5-C)。结果表明,当Amp质量浓度低于4 μg/mL时,2种菌的MCDGROWTH值为29,随着Amp浓度的继续增大,BL21-βlac MCDGROWTH值明显高于BL21-βlac-Aβ42。
综上所述,当Amp质量浓度为15 μg/mL时,BL21-βlac-Aβ42的菌落不能生长,质量浓度降为10 μg/mL时,2种重组菌的原液不仅可以正常生长,且随着稀释倍数的增加呈现明显的梯度效果(图6)。
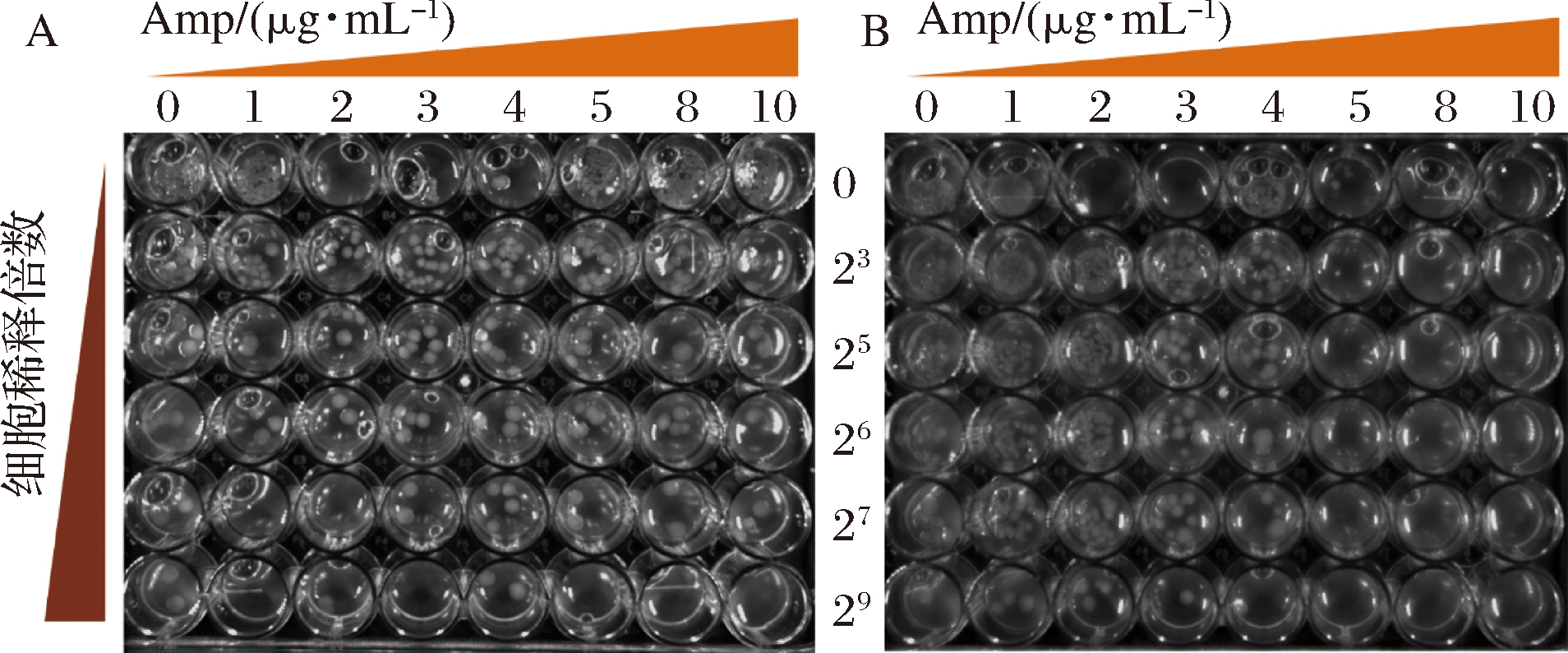
A-BL21-βlac;B-BL21-βlac-Aβ42
图6 0~10 μg/mL下Amp重组菌株的生长状况
Fig.6 The growth of recombinant strains in the presence of Amp (0~10 μg/mL)
2.4 已知Aβ42聚集抑制剂对体内筛选系统的验证
根据上文确定的Amp浓度范围,利用已知Aβ42聚集抑制剂,分别通过斑点滴定实验、涂布实验以及检测重组菌株OD 3种方法来验证BL21-βlac-Aβ42体内聚集抑制剂筛选体系的有效性。
2.4.1 斑点滴定实验验证
姜黄素是从姜科、天南星科中一些植物根茎中提取的化学成分,具有抗炎、抗氧化、抗肿瘤等作用,并且是一种典型的Aβ42聚集抑制剂[30-31]。分析姜黄素对BL21-βlac在不同浓度Amp条件下菌体细胞生长的影响,结果如图7-A所示,Amp在质量浓度0~10 μg/mL范围内,加药组与未加药组BL21-βlac在各稀释倍数下均有菌落生长,且随稀释倍数增加,菌落数目逐渐减少,表明Amp质量浓度在0~10 μg/mL范围内,姜黄素的添加不会显著影响菌体的生长。
分别在加与不加姜黄素的条件下诱导表达βlac-Aβ42,并进行斑点滴定实验检测,观察菌落生长状态和允许菌体生长的最大细胞稀释倍数。图7-B结果显示,未加药组BL21-βlac-Aβ42允许菌体生长的最大稀释倍数为26。添加姜黄素后,允许菌体生长的最大稀释倍数变为210(图5)。由此可见,由于姜黄素抑制了Aβ42的聚集,使得βlac的2个结构域正常结合,恢复BL21-βlac-Aβ42菌株对Amp的抗性。以上结果表明该体系可应用于Aβ42聚集抑制剂的筛选。

图7 (A)0~10 μg/mL Amp下姜黄素对BL21-βlac生长状况的影响;(B)不同稀释倍数BL21-βlac-Aβ42在加与不加100 μmol/L姜黄素条件下菌落生长状况
Fig.7 (A)Effect of curcumin on the growth of BL21-βlac in the presence of 0-10 μg/mL Amp;(B) The growth of BL21-βlac-Aβ42 in different dilution ratios with or without 100 μmol/L curcumin
2.4.2 BL21-βlac-Aβ42发酵过程中菌液浓度变化
根据βlac-Aβ42蛋白表达过程中宿主菌OD600的变化来验证筛选体系。将初始OD600一致的BL21-βlac-Aβ42菌液添加于96孔板中,并分别加入不同质量浓度Amp(0,2,4,6,8,10 μg/mL)和终浓度为100 μmol/L的姜黄素。不加姜黄素菌液为对照组,观察各组菌液OD600值随时间的变化。结果如图8所示,在不同Amp浓度下,随着时间的延长,加药组与未加药组菌液OD600值均逐渐增大。当培养时间超过5 h后,加药组OD600值明显高于未加药组。综上所述,当添加姜黄素后,Aβ42的聚集受到一定程度的抑制,促使βlac的2个结构域正常结合,并发挥对Amp的抗性作用,改善重组菌株的生长状况。
2.4.3 平板涂布实验
本研究利用3种已知Aβ42聚集抑制剂(Curcumin、EGCG、Fast Green),通过菌液涂布实验进一步验证BL21-βlac-Aβ42筛选体系。将初始OD600值相同的菌液进行相同倍数的梯度稀释,取稀释倍数为0、10、102、103、104和105的菌液进行涂板(Amp质量浓度为10 μg/mL)。结果如图9-A所示,随着菌体稀释倍数的增加,BL21-βlac、BL21-βlac-Aβ42和添加抑制剂后的BL21-βlac-Aβ42菌落数目均逐渐减少。当稀释倍数为102时,菌落数目明显减少,但加药组菌落数目多于未加药组。稀释倍数为103时,BL21-βlac-Aβ42无菌落生长,添加Curcumin、EGCG和Fast Green后,BL21-βlac-Aβ42依然有菌落生长,且菌落状态正常。基于上述实验结果,在104范围内将菌液密集稀释,取稀释倍数为0、23、25、26、27、29、210、104的菌液涂板,并选取Curcumin为代表药物,结果如图9-B所示。添加姜黄素的BL21-βlac-Aβ42菌液允许细胞生长的最大细胞稀释倍数为104,明显高于未加药组(29),证明姜黄素抑制了Aβ42的聚集,恢复BL21-βlac-Aβ42对Amp的抗性,改善菌体生长状态。上述结果进一步证明该体内系统可以用来筛选Aβ42聚集抑制剂。

图8 BL21-βlac-Aβ42菌体质量浓度随诱导时间的变化
Fig.8 Concentrations changes of BL21-βlac-Aβ42 along with the induction time

A-姜黄素、EGCG、固绿作用下BL21-βlac-Aβ42菌落生长状况;B-密集稀释后,姜黄素作用下菌落生长状况
图9 不同Aβ42聚集抑制剂作用下BL21-βlac-Aβ42生长状况
Fig.9 The growth of BL21-βlac-Aβ42 in the presence of different Aβ42 inhibitors
3 结论
本研究基于βlac结构特征及Aβ42聚集特性,成功构建了BL21-βlac-Aβ42重组菌株,并将该重组菌株应用于体内筛选Aβ42聚集抑制剂。利用已知Aβ42聚集抑制剂,通过斑点滴定实验、涂布平板实验和检测菌液浓度变化实验对该体内筛选系统进行了验证。结果表明添加聚集抑制剂后,BL21-βlac-Aβ42菌体在特定Amp浓度条件下生长状况得到改善,允许菌体生长的最大细胞稀释倍数增大,证明了该体系可以应用于大肠杆菌体内筛选Aβ42聚集抑制剂。本文构建的大肠杆菌体内筛选体系拓宽了淀粉样蛋白聚集抑制剂筛选的思路,提供了抑制剂开发的新方法。
[1] CHITI F, DOBSON C M. Protein misfolding, functional amyloid, and human disease [J]. Annual Review of Biochemistry, 2006, 75(1): 333-366.
[2] CANTER R G, PENNEY J, TSAI L H. The road to restoring neural circuits for the treatment of Alzheimer's disease [J]. Nature, 2016, 539(7 628): 187-196.
[3] HARDY J A, HIGGINS G A. Alzheimer's disease: the amyloid cascade hypothesis [J]. Science, 1992, 256(5 054): 184-185.
[4] 李丽, 刘夫锋. 淀粉样β蛋白质构象转换及其抑制的分子动力学模拟[J]. 生物加工过程, 2019, 17(1): 50-58.
[5] GEERT V G, WIM A. Amyloid, presenilins, and Alzheimer's disease [J]. Neuroscientist A Review Journal Bringing Neurobiology Neurology & Psychiatry, 2003, 9(2): 117-126.
[6] GUO J, SUN W, LIU F F. Brazilin inhibits the Zn2+-mediated aggregation of amyloid β-protein and alleviates cytotoxicity [J]. Journal of Inorganic Biochemistry, 2017, 177: 183-189.
[7] JIA L G, WANG W J, SHANG J Z, et al. Highly efficient soluble expression, purification and characterization of recombinant Ab42 from Escherichia coli [J]. RSC Advances, 2018, 8(33): 18 434-18 441.
[8] LIU F F, WANG W J, SANG J C, et al. Hydroxylated single-walled carbon nanotubes inhibit a beta(42) fibrillogenesis, disaggregate mature fibrils, and protect against a beta(42)-induced cytotoxicity [J]. ACS Chemical Neuroscience, 2019, 10(1): 588-598.
[9] 郑嵋戈, 阮志刚,刘靖,等. β-淀粉样蛋白的代谢机制[J]. 神经解剖学杂志, 2011, 27(3): 344-348.
[10] HARDY J, SELKOE D J. The amyloid hypothesis of Alzheimer's disease: Progress and problems on the road to therapeutics [J]. Science, 2002, 297(5 580): 353-356.
[11] TAPIOLA T, ALAFUZOFF I, HERUKKA S K, et al. Cerebrospinal fluid β-Amyloid 42 and tau proteins as biomarkers of Alzheimer-Type pathologic changes in the brain [J]. Alzheimers & Dementia the Journal of the Alzheimers Association, 2009, 6(4): S170-S170.
[12] COHEN S I, LINSE S, LUHESHI L M, et al. Proliferation of amyloid-β42 aggregates occurs through a secondary nucleation mechanism [J]. Proc Natl Acad Sci USA, 2013, 110(24): 9 758-9 763.
[13] SHANKAR G M, BLOODGOOD B L, TOWNSEND M, et al. Natural oligomers of the alzheimer amyloid-β protein induce reversible synapse loss by modulating an NMDA-Type glutamate receptor-dependent signaling pathway [J]. Journal of Neuroscience, 2007, 27(11): 2 866-2 875.
[14] BIANCALANA M, KOIDE S. Molecular mechanism of Thioflavin-T binding to amyloid fibrils [J]. Biochimica et Biophysica Acta-Proteins and Proteomics, 2010, 1 804(7): 1 405-1 412.
[15] GROENNING M. Binding mode of Thioflavin T and other molecular probes in the context of amyloid fibrils-current status [J]. Journal of Chemical Biology, 2010, 3(1): 1-18.
[16] ZHAO D S, CHEN Y X, LIU Q, et al. Exploring the binding mechanism of thioflavin-T to the β-amyloid peptide by blind docking method [J]. Science China:Chemistry, 2012, 55(1): 112-117.
[17] LI S, LIU F F, YU L L, et al. Dual effect of thioflavin T on the nucleation kinetics of amyloid beta-protein 40 [J]. Acta Physico-Chimica Sinica, 2016, 32(6): 1 391-1 396.
[18] BIANCALANA M, MAKABE K, KOIDE A, et al. Molecular mechanism of thioflavin-T binding to the surface of beta-rich peptide self-assemblies [J]. Journal of Molecular Biology, 2009, 385(4): 1 052-1 063.
[19] MENG F, MAREK P, POTTER K J, et al. Rifampicin does not prevent amyloid fibril formation by human islet amyloid polypeptide but does inhibit fibril thioflavin-T interactions: Implications for mechanistic studies of β-cell death [J]. Biochemistry, 2008, 47(22): 6 016-6 024.
[20] OSTERMEIER M. Designing switchable enzymes [J]. Current Opinion in Structural Biology, 2009, 19(4): 442-448.
[21] SAUNDERS J C, YOUNG, L M, MAHOOD R A, et al. An in vivo platform for identifying inhibitors of protein aggregation [J]. Nature Chemical Biology, 2016, 12(2): 94-101.
[22] DELAIRE M, LENFANT F, LABIA R, et al. Site-directed mutagenesis on TEM-1 beta-lactamase: role of Glu166 in catalysis and substrate binding [J]. Protein Engineering, 1991, 4(7): 805-810.
[23] FOIT L, BARDWELL J C. A tripartite fusion system for the selection of protein variants with increased stability in vivo [J]. Methods in Molecular Biology, 2013, 978: 1-20.
[24] ZEBALA J, BARANY F. Mapping catalytically important regions of an enzyme using two-codon insertion mutagenesis: a case study correlating β-lactamase mutants with the three-dimensional structure [J]. Gene, 1991, 100: 51-57.
[25] FOIT L, MORGAN G J, KERN M J, et al. Optimizing protein stability in vivo [J]. Molecular Cell, 2009, 36(5): 861-871.
[26] HALLET B, SHERRATT D J, HAYES F. Pentapeptide scanning mutagenesis: random insertion of a variable five amino acid cassette in a target protein [J]. Nucleic Acids Research, 1997, 25(9): 1 866-1 867.
[27] GALARNEAU A, PRIMEAU M, TRUDEAU L E, et al. Beta-Lactamase protein fragment complementation assays as in vivo and in vitro sensors of protein-protein interactions [J]. Nature Biotechnology, 2002, 20(6): 619-622.
[28] EDWARDS W R, BUSSE K, ALLEMANN R K, et al. Linking the functions of unrelated proteins using a novel directed evolution domain insertion method [J]. Nucleic Acids Research, 2008, 36(13): e78-e78.
[29] GUNTAS G, MANSELL T J, KIM J R, et al. Directed evolution of protein switches and their application to the creation of ligand-binding proteins [J]. Proceedings of the National Academy of Sciences, 2005, 102(32): 11 224-11 229.
[30] YANG F, LIM G P, BEGUM A N, et al. Curcumin inhibits formation of amyloid beta oligomers and fibrils, binds plaques, and reduces amyloid in vivo [J]. Journal of Biological Chemistry, 2005, 280(7): 5 892-5 901.
[31] 刘彬, 赵莹,于思礼, 等. 微生物代谢工程发酵姜黄素研究进展[J]. 食品与发酵工业, 2016, 42(7): 254-260.