脂肪酶,三酰基甘油酰基水解酶(EC.3.1.1.3),可催化甘油三酯,使其水解为甘油和脂肪酸[1-2]。1834年,EBERLE首次在胰腺中发现脂肪酶,随后,EIJKMANN首次分别从Serratia marcescens、Pseudomonas aeruginosa和Pseudomonas fluorescens中分离到脂肪酶[3]。脂肪酶是继蛋白酶和淀粉酶之后第三大工业化应用酶制剂。脂肪酶不仅可催化水解反应,还可催化无水混合物的合成反应,包括酯化和酯交换(酸解、醇解)等[4-5]。因此,脂肪酶可应用于食品、油脂加工、生物柴油、洗涤剂、生物医药、化妆品和皮革加工等工业领域[6-7]。
脂肪酶广泛存在于动物、植物和微生物细胞中[1-2]。微生物来源的脂肪酶因其催化特异性强、种类丰富、胞外分泌表达和易于回收等特征,成为脂肪酶最主要的来源[3]。2007年,黑曲霉CBS513.88基因组解析完成并公布[8],通过生物信息学预测方法预测到黑曲霉具有丰富的脂肪酶,但功能未知,绝大部分仍停留在生物信息学推断水平与组学分析阶段。我国学者于2007年进行了黑曲霉F044脂肪酶的基因克隆和其在大肠杆菌和毕赤酵母中的异源表达[9];通过两步基因合成优化密码子,实现了黑曲霉lip2基因在毕赤酵母中的高效表达[10];通过同源比对筛选黑曲霉中新型活性脂肪酶基因an1-1,并在Escherichia coli BL21中实现表达[11]。2012年,NAKAJIMA-KAMBE等[12]克隆了A. niger MTCC 2594中lipA基因,在毕赤酵母中表达,并研究了其在洗涤剂行业中的应用潜力。
本研究在黑曲霉基因组分析的基础上,对其中一个编码功能未知的脂肪酶编码基因tglE进行了基因克隆和表达,并对重组酶的酶学性质和酯化功能进行了解析。
1 材料与方法
1.1 菌种
黑曲霉(A. niger)CICIM F0215由江南大学中国高校工业微生物资源与信息中心(http://cicim-cu.jiangnan.edu.cn)提供。大肠杆菌(E. coli)JM109、毕赤酵母(Pichia pastoris)GS115和质粒pPIC9k保藏于天津科技大学生物催化与生物转化研究室。
1.2 主要试剂
限制性内切酶和LA Taq DNA多聚酶,大连宝生物工程有限公司;T4 DNA连接酶、质粒小量提取试剂盒、小量DNA产物纯化回收试剂盒和cDNA合成试剂盒(1st strand cDNA synthesis kit),ThermoFisher公司;G418,Invitrogen公司;氨苄青霉素钠,Sigma公司;C4~C16系列对硝基苯酚羧酸酯,Sigma公司;其他生化试剂为分析纯,国药化学试剂有限公司。
1.3 脂肪酶基因的生物信息分析
利用NCBI(https://www.ncbi.nlm.nih.gov)查询脂肪酶的氨基酸序列。序列比对采用Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)程序。信号肽分析采用SignalP 4.1软件(http://www.cbs.dtu.dk/services/SignalP/)。
1.4 黑曲霉脂肪酶基因克隆与表达
1.4.1 基因克隆与重组菌构建
黑曲霉总RNA提取、cDNA的制备与纯化按照试剂盒说明书进行,质粒和PCR产物酶切、连接、电转及转化子筛选等均采用实验室常规方法进行[13]。根据A. niger CBS 513.88基因组注释信息设计引物,上游引物(5’→3’)为gtaGTGCCAATGCTCCAGTCTCGAG,下游引物(5’→3’)为![]() (下划线部分为限制性酶切位点)。
(下划线部分为限制性酶切位点)。
1.4.2 重组酶的制备与纯化
根据毕赤酵母表达手册(Invitrogen EasyselectTM Pichia Expression Kit)制备发酵液。在4 ℃下,10 000 g离心5 min,获得粗酶液。进一步利用硫酸铵沉淀、GE系列PD-10脱盐柱除盐和G75凝胶色谱法对酶液进行纯化,纯化后经冷冻干燥,留酶样备用。通过SDS-PAGE分析蛋白纯化情况,按照文献方法[14]进行。
1.5 重组酶酶活测定与酶学性质分析
1.5.1 脂肪酶酶活测定方法
脂肪酶酶活测定参照GUPTA等方法,并作改进[15]。在一定温度和pH下,1 h生成1 μmol的对硝基苯酚为1个酶活力单位。
1.5.2 最适温度
在不同温度下(25~70 ℃),测定重组脂肪酶tglE的酶活力。以最高酶活为100%计算相对酶活。
1.5.3 最适pH
在不同反应pH下(pH 4.0~10.0),测定重组脂肪酶tglE的酶活力。以最高酶活为100%计算相对酶活。
1.5.4 温度稳定性
将酶液分别放在不同温度下(20~60 ℃)保温,分别在不同时间取样,测定残余酶活。以最高酶活力为100%计算相对酶活。
1.5.5 pH稳定性
称取一定量纯化后的酶样,用不同pH(pH 5.0~9.0)的缓冲液溶解(终浓度一致),于室温下放置,在不同时间取样,测定残余酶活。以最高酶活力为100%计算相对酶活。
1.5.6 金属离子及螯合剂对酶活力的影响
将酶液分别与不同的金属离子、螯合剂混合,室温放置2 h。测定不同金属离子对酶活力的影响。金属离子在反应体系中的终浓度为1 mmol/L。以未加金属离子的反应体系的酶活力为100%计算相对酶活。
1.5.7 有机溶剂对酶活力的影响
按照文献[16],在500 μL反应体系中,加入40 μL酶液和10 μL有机溶剂,于150 r/min,30 ℃下放置2 h。测定残余酶活力。以未加有机溶剂的反应体系的酶活力为100%计算相对酶活。
1.5.8 底物特异性和动力学参数
以5种不同碳链长度的对硝基苯酚羧酸酯(C2、C4、C8、C12、C16)为底物,测定该重组酶的酶活力。以最高酶活力为100%计算相对酶活。采用Lineweave-Burk双倒数作图法测定脂肪酶的动力学参数。以C4为底物,用异丙醇配制一系列不同浓度的底物,测定在不同底物浓度下的酶活力,以底物浓度的倒数1/[S]为横坐标,以反应速率的倒数1/V为纵坐标,作图,并利用公式1/V=1/Vmax+(Km/Vmax)·(1/[S])得出Km和![]()
1.6 重组酶酯化活性鉴定
1.6.1 重组酶合成辛酸乙酯和乳酸乙酯
1.6.1.1 辛酸乙酯反应体系
按照文献[18]进行。配制辛酸和无水乙醇摩尔浓度分别为1.2 mol/L和1.44 mol/L(溶剂为正庚烷),分别加入2.5 mL制备好的辛酸和乙醇和10 mg酶粉,于40 ℃,150 r/min下振荡反应12 h,以高温灭活的酶为对照。并离心,取上清液,留作气相分析。
1.6.1.2 乳酸乙酯反应体系
按照文献[19]。配制乳酸和乙醇摩尔浓度分别为1.17 mol/L和4.68 mol/L,溶剂为叔丁醇,分别加入5 mL制备好的乳酸和乙醇和10 mg酶,于40 ℃,150 r/min反应12 h。以高温灭活的酶为对照。并离心,取上清液。留作气相分析。
1.6.2 辛酸乙酯和乳酸乙酯的检测
采用气相色谱法进行。检测条件是:色谱柱为HP-INNOWax,30 m×0.32 mm×0.5 μm毛细血管柱,检测器为FID。进样口的温度为200 ℃,检测器温度为250 ℃,H2流量为40 mL/min,空气流量为300 mL/min,尾吹流量为25 mL/min。进样量条件:进样量1 μL,分流比10∶1。载气为N2,色谱柱流速为0.8 mL/min。升温程序:起始柱温50 ℃,保持8 min,以5 ℃/min的速度上升至200 ℃,保持5 min[20]。
2 结果与分析
2.1 黑曲霉脂肪酶基因克隆及重组菌的获得
依据黑曲霉CBS 513.88基因组序列信息(EMBL AM270980-AM270998)并采用Blast等分析方法进行分析,发现一个疑似编码脂肪酶的基因,但功能未知。为此以黑曲霉F0215的cDNA为模板,对该基因进行PCR扩增(如图1-A),并命名为tglE,将其连接到载体pPIC9K上,获得重组质粒pPIC-tglE,使用限制性内切酶Sal I进行酶切验证,获得2条大小约为7.6 kb和2.5 kb的条带(如图1-B)。经进一步序列测定与分析发现,tglE编码的氨基酸序列与黑曲霉CBS 513.88基因组中相应序列仅有1个氨基酸残基存在差异(在275位上,CBS 513.88基因序列碱基为C, F0215基因序列碱基为G),该差异导致了1个氨基酸序列不同(在92位上,CBS 513.88的氨基酸残基为S,F0215的氨基酸残基为C),但其对酶催化活性中心(S139、D195、H248)、GXSXG保守序列和“盖子”结构无影响(数据未呈现)。重组质粒pPIC -tglE使用限制性内切酶Sac I进行线性化,转化在毕赤酵母GS115中,获得了tglE重组菌,命名为ANL-TglE。
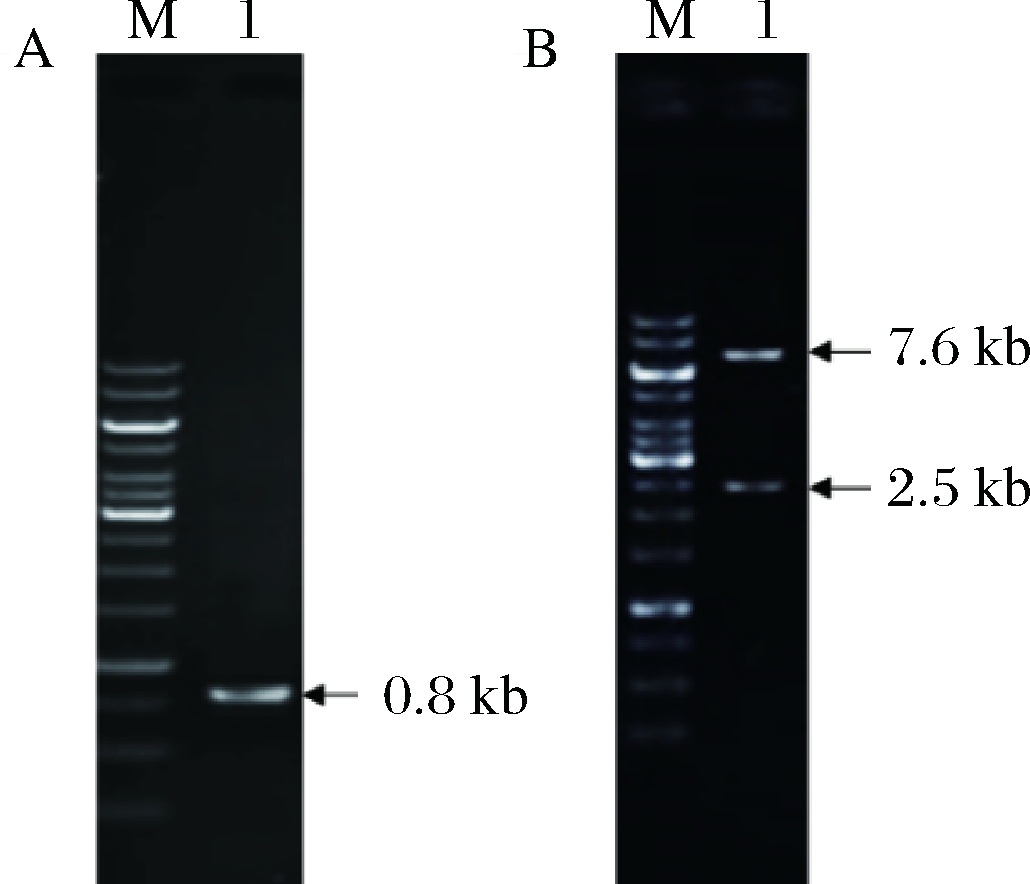
M-DNA Marker;A1-tglE;B1-pPIC-tglE/Sal I
图1 目的基因及重组质粒验证电泳图谱
Fig.1 Agarose gel electrophoresis of target gene and
recombination vectors
2.2 重组酶的制备与纯化
根据毕赤酵母表达手册,制备发酵酶液,酶液经硫酸铵沉淀、PD-10脱盐柱除盐和G75凝胶色谱纯化后,在SDS-PAGE上呈现单一条带(图2)。重组酶tglE的分子质量约为32 kDa,略大于预测的28 kDa,可能与重组酶tglE表达过程中被糖基化修饰有关。纯化后的tglE,其比酶活为136.42 U/mg,纯化倍数和得率分别为13.95倍和13.36%(表1)。

M-蛋白分子量标准;1-GS115发酵液;2-ANL-tglE发酵液;
3-纯化后的脂肪酶tglE
图2 脂肪酶tglE SDS-PAGE
Fig.2 SDS-PAGE analysis of tglE
表1 脂肪酶tglE的纯化结果
Table 1 Purification on tglE
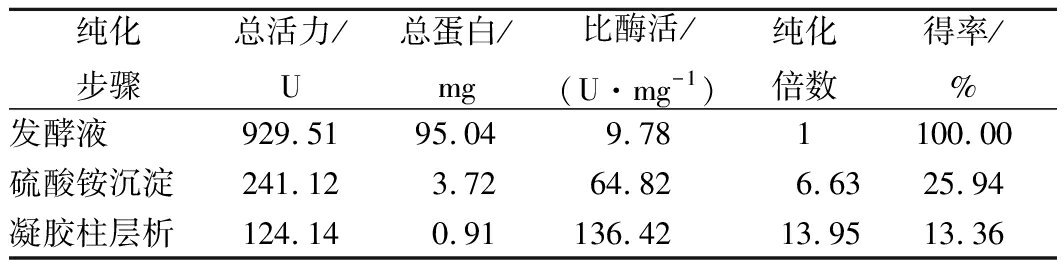
纯化步骤总活力/U总蛋白/mg比酶活/(U·mg-1)纯化倍数得率/%发酵液929.5195.049.781100.00硫酸铵沉淀241.123.7264.826.6325.94凝胶柱层析124.140.91136.4213.9513.36
2.3 黑曲霉脂肪酶酶学性质解析
2.3.1 最适温度和最适pH
在不同温度或pH下测定tglE的酶活。当温度高于25 ℃时,酶活力开始急剧上升,在40 ℃时达到最大酶活力,超过40 ℃后,酶活力开始下降(图3-A),表明40 ℃为该酶的最适温度。在30~60 ℃之间,该酶的酶活力保持在60%以上。当温度在25 ℃或者高于65 ℃,酶活力低于50%。当pH高于5.5,酶活力急剧上升,在pH为7.0时,达到最大酶活力,在pH 6.5~9.0之间,酶活力保持在60%以上(图3-B)。可见,该酶的最适温度和pH分别为40 ℃和7.0。
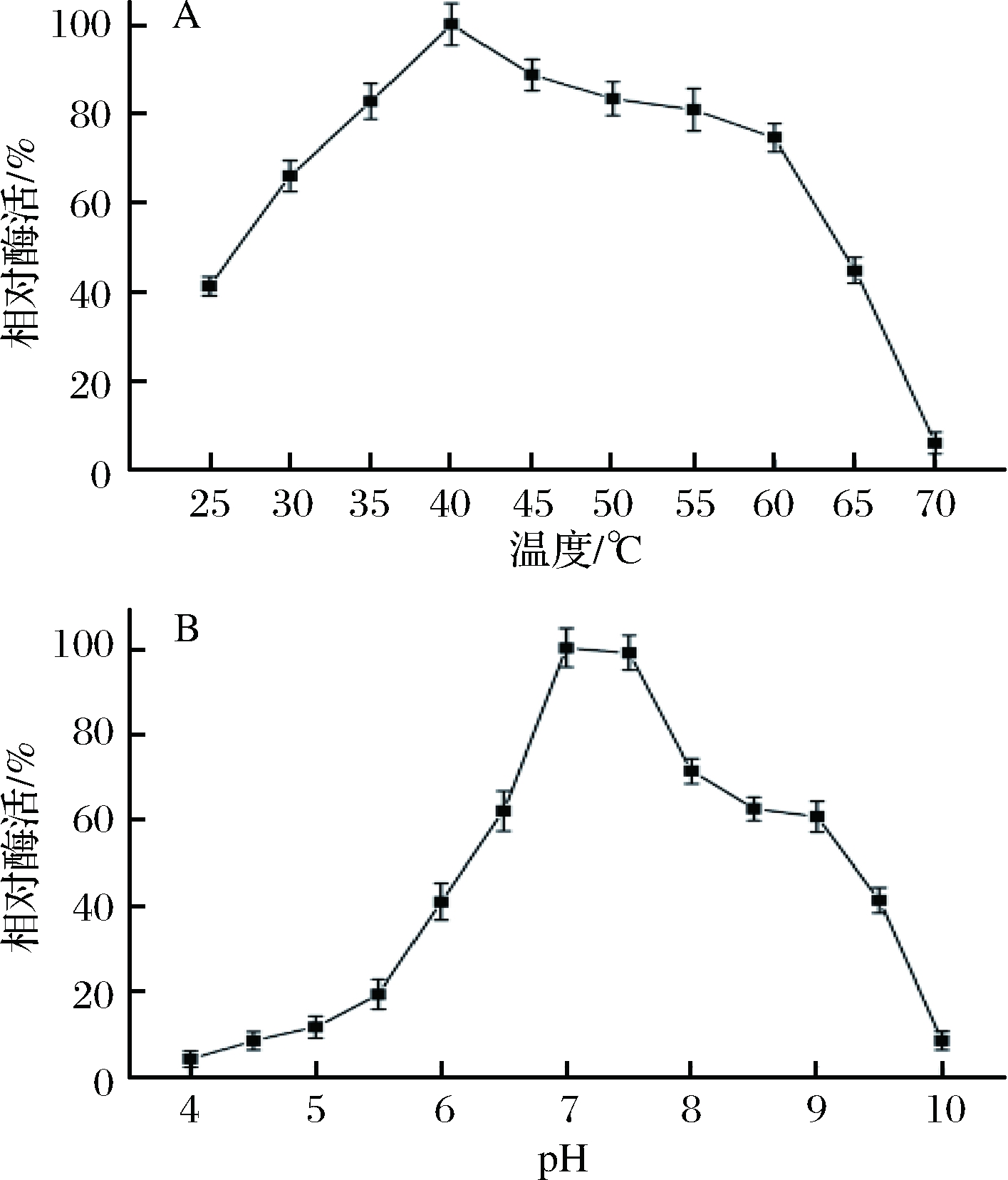
图3 tglE的最适作用温度和最适作用pH
Fig.3 The optimum temperature and pH of tglE
2.3.2 热稳定性和pH稳定性
tglE在不同温度或pH下孵育,定时取样检测残余酶活。tglE在20 ℃和30 ℃时,4 h内残余酶活均在80%以上;随着温度的上升,酶活力下降速率明显上升;在30 ℃以下保温3 h, tglE的残余酶活力高于60%(图4-A)。tglE在pH为7.0的缓冲体系下最稳定,4 h内残余酶活在80%以上;在3 h内,该酶在pH为6.0~8.0的缓冲体系中,残余酶活均大于60%(图4-B)。可见,该酶在20~30 ℃和pH 6.0~8.0稳定性良好。

图4 tglE温度稳定性和pH稳定性
Fig.4 Effect of temperature and pH on enzyme
stability of tglE
2.3.3 金属离子及螯合剂对重组脂肪酶活力的影响
将tglE与金属离子或螯合剂混匀,反应的终浓度为1 mmol/L,保温2 h。Ca2+、Sn2+、K+、Cu2+、Mg2+、Mn2+对TglE的水解活性有促进作用,其中Ca2+的促进作用最明显;Fe3+、Zn2+和EDTA对酶的水解活性有抑制作用,其中EDTA最强(表2)。EDTA为金属离子螯合剂,可吸附体系中的金属离子,严重抑制酶的水解活性,表明脂肪酶tglE属于金属酶。
表2 金属离子及EDTA对tglE的影响
Table 2 Effects of metal ions and chelate agent on the
activities of tglE
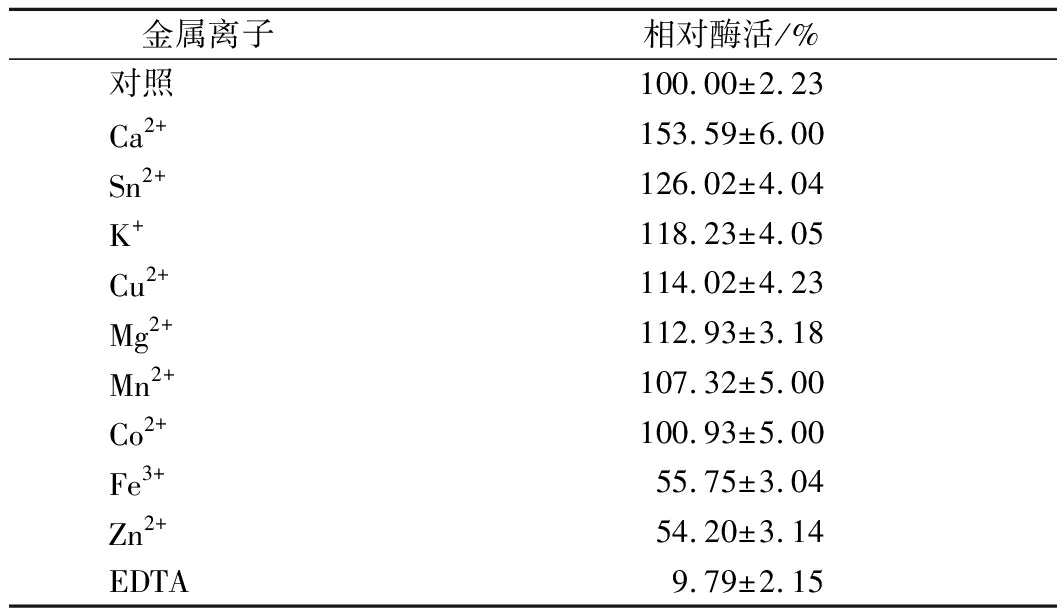
金属离子相对酶活/%对照100.00±2.23Ca2+153.59±6.00Sn2+126.02±4.04K+118.23±4.05Cu2+114.02±4.23Mg2+112.93±3.18Mn2+107.32±5.00Co2+100.93±5.00Fe3+55.75±3.04Zn2+54.20±3.14EDTA9.79±2.15
2.3.4 有机溶剂耐受性分析
将tglE与有机溶剂混合(有机溶剂占总体积的20%)。庚烷、甲醇和二甲基亚砜对tglE的水解活性有一定的促进作用;乙酸甲酯、十二烷则对该酶有抑制作用。甲苯、乙腈、乙醇、丙酮、四氯化碳、正己烷和氯仿对酶有轻微抑制作用(图5)。

图5 有机溶剂对tglE酶活的影响
Fig.5 Effects of organic solvent on the activities of tglE
2.3.5 底物特异性分析
tglE水解不同碳链长度合成底物发现,其最适反应底物为C4,其次依次为C8、C12、C2和C16(图6)。
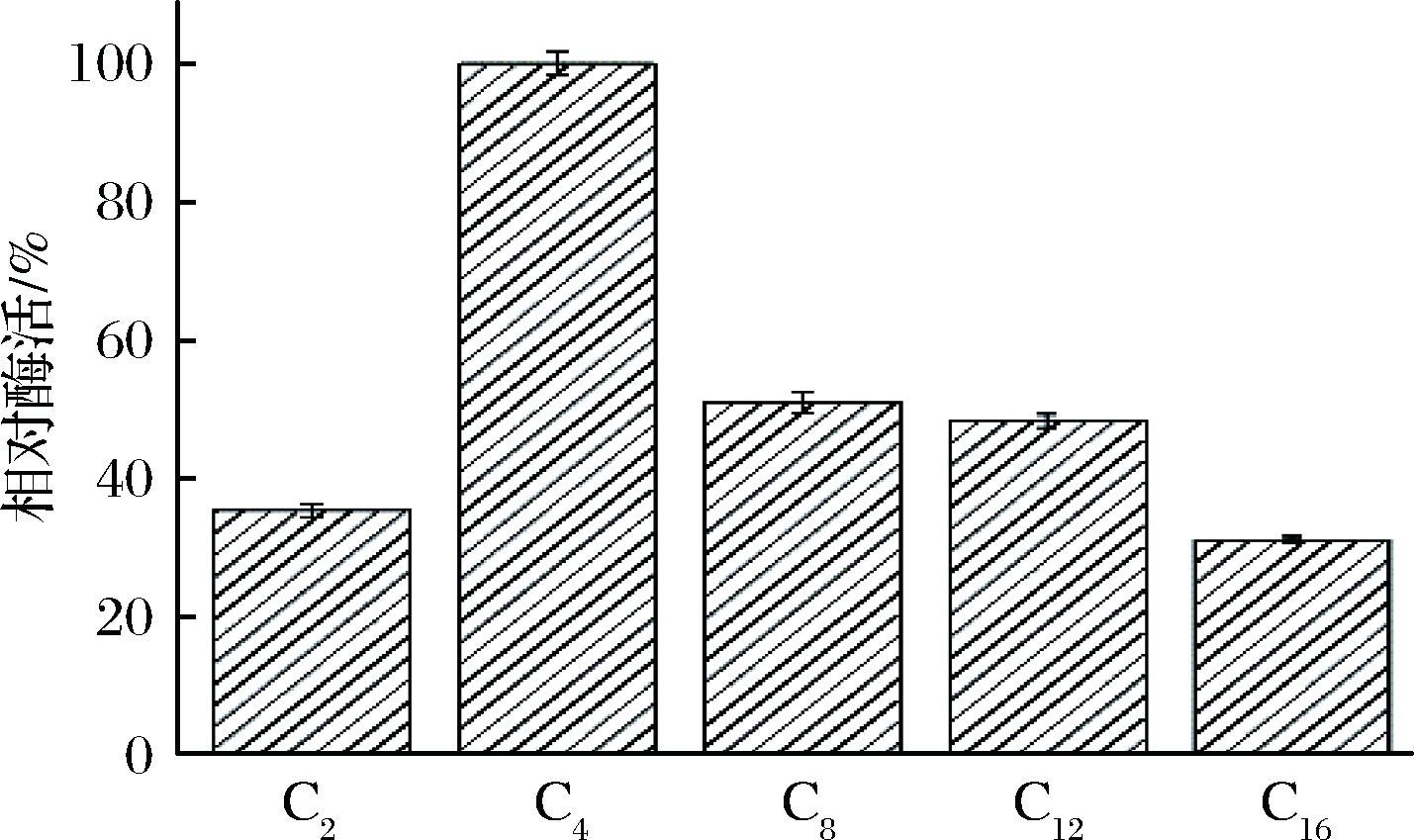
图6 tglE脂肪酸特异性
Fig.6 Substrate specificity of tglE
酶对C2-C16底物均有不同程度的水解作用,说明该酶具有宽底物谱。以不同浓度的对硝基苯酚丁酸酯为底物,采用双倒数法,绘制tglE的动力学曲线,求得tglE的Km值为14.40 mmoL/L,Vmax为46.72 mmol/(mL·h)。
tglE对于不同的植物油,酶活力不同(图7)。其中以橄榄油为底物时,酶活最高,表明tglE对橄榄油的水解程度最高;以棕果油和菜籽油为底物时,酶活较低,相对于橄榄油为底物时,酶活分别为45%和47%,表明该酶对菜籽油和棕果油的水解程度较低;对其他植物油的水程度略有不同,相对于以橄榄油为底物,其水解活性在65%~85%之间。
2.4 酯化活性鉴定
以辛酸、乳酸和乙醇为底物,利用tglE催化合成乳酸乙酯和辛酸乙酯,经气相色谱检测可知,酶可合成2种酯,但合成辛酸乙酯的效率要明显高于乳酸乙酯(图8)。其中,合成乳酸乙酯和辛酸乙酯的转化率,分别为0.45%和12.40%。

图7 tglE天然底物特异性
Fig.7 Natural substrate specificity of tglE

图8 tglE催化合成乳酸乙酯和辛酸乙酯气相图
Fig.8 GC profiles of synthesis of ethyl lactate and ethyl caprylate by tglE
3 结论
本研究对黑曲霉F0215来源的但功能未知的一种脂肪酶进行分子克隆、异源表达,首次系统地解析了该脂肪酶的酶学性质并研究其酯化活性。经克隆表达与纯化后,该酶的比酶活为137.08 U/mg,纯化倍数为13.95倍,得率为13.36%,与前人在其它脂肪酶研究的结果类似[21]。研究中发现,EDTA明显抑制tglE的水解活性,提示该酶是一种金属酶,后续将进一步就这一属性进行深入分析。本研究获得的tglE具有较好的有机溶剂的耐受性,与来源于Proteus sp. SW1[22]、Acinetobacter sp. XMZ-26[23]和Staphylococcus epidermidis AT2[24]脂肪酶相似。这一属性使其在有机相催化反应中,具有重要的潜在应用价值。tglE对C4底物表现出最高的水解活性,以C4为底物时的Km为14.40 mmol/L,Vmax为46.72 mmol/(mL·h);对不同的天然植物油也表现出不同的水解活性。这一属性与来源于Yersinia enterocolitica[25]、Psychrobacter pacificensis[26]、Candida rugosa[27]和Geotrichum candidum[27]的脂肪酶相似。且本文鉴定出的tglE,具有较好的低温合成辛酸乙酯的酯化活性,预示着该酶在相关食品加工中也具有潜在的应用价值。
[1] SARMAH N, REVATHI D, SHEELU G, et al. Recent advances on sources and industrial applications of lipases[J]. Biotechnology Progress, 2018, 34(1):5-28.
[2] ANGAJALA G, PAVAN P, SUBASHINI R. Lipases: an overview of its current challenges and prospectives in the revolution of biocatalysis[J]. Biocatalysis and Agricultural Biotechnology, 2016, 7:257-270.
[3] CASAS- GODOY L, DUQUESNE S, BORDES F, et al. Lipase: an overview[J]. Lipases and Phospholipases: Methods and Protocols, 2012: 3-29.
[4] QUILLES J C J, BRITO R R, BORGES J P, et al. Modulation of the activity and selectivity of the immobilized lipases by surfactants and solvents[J]. Biochemical Engineering Journal, 2015, 93:275-280.
[5] STERGIOU P Y, FOUKIS A, FILIPPOU M, et al. Advances in lipase-catalyzed esterification reactions[J]. Biotechnology Advances, 2013, 31:1 846-1 859.
[6] VERMA N, THAKUR S, BHATT A K. Microbial lipases: industrial applications and properties[J]. International Research Journal of Biological Sciences, 2012, 1:88-92.
[7] PRIJI P, SAJITH S, FAISAL PA, et al. Microbial lipases-properties and applications[J]. Journal of Microbiology Biotechnology & Food Sciences, 2016, 6(2): 799-807.
[8] PEL H J, de WINDE J H, ARCHER D B, et al. Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88 [J]. Nature Biotechnology, 2007, 25(2): 221-231.
[9] 舒正玉, 杨江科,徐莉,等. 黑曲霉脂肪酶基因的克隆及其在毕赤酵母中的表达[J]. 武汉大学学报(理学版), 2007, 53(2): 204-208.
[10] YANG Jiangke, LIU Liying. Codon optimization through a two-step gene synthesis leads to a high-level expression of Aspergillus niger lip2 gene in Pichia pastoris[J]. Journal of Molecular Catalysis B: Enzymatic, 2010, 63(3-4): 164-169.
[11] 方珊, 刘浩浩,李华林,等. 新型脂肪酶基因的克隆与表达[J]. 食品与药品, 2013, 15(1): 15-18.
[12] NAKAJIMA K T, EDWINOLIVER N G, MAEDA H, et al. Purification, cloning and expression of an Aspergillus niger lipase for degradation of poly (lactic acid) and poly(ε-caprolactone) [J]. Polymer Degradation and Stability, 2012, 97(2): 139-144.
[13] 诸葛健, 王正祥. 工业微生物实验技术手册[M]. 北京:中国轻工业出版社, 1994: 94-302.
[14] SOLIMAN E A M, ALY N A H, MOHARAM M E. Genetic modification of alkaline protease, lipase activities, SDS-PAGE proteins and other characters in some bacterial strains[J]. Asian Journal of Biotechnology, 2001, 1(4): 129-141.
[15] GUPTA N, RATHI P, GUPTA R. Simplified para-nitrophenyl palmitate assay for lipases and esterases[J]. Analytical Biochemistry, 2002, 311(1): 98-99.
[16] SHARMA P, SHARMA N, PATHANIA S, et al. Purification and characterization of lipase by Bacillus methylotrophicus PS3 under submerged fermentation and its application in detergent industry[J]. Journal of Genetic Engineering and Biotechnology, 2017, 15: 369-377.
[17] SINGH A K, MUKHOPADHYAY M. Immobilization of lipase on carboxylic acid-modified silica nanoparticles for olive oil glycerolysis[J]. Bioprocess & Biosystems Engineering, 2018, 41(1): 115-127.
[18] 滕云. 酯合成脂肪酶高产菌的选育及其产酶发酵调控的研究[D]. 无锡:江南大学, 2008.
[19] 赵天涛. 有机相脂肪酶催化合成乳酸乙酯的研究[D]. 天津:河北工业大学, 2005.
[20] 王沙木. 气相色谱法测定白酒中乙酸乙酯含量的探讨[J]. 黑龙江科技信息, 2007(2): 30-212.
[21] JAVED S, AZEEM F, HUSSAIN S, et al. Bacterial lipases: a review on purification and characterization[J]. Progress in Biophysics and Molecular Biology, 2017, 132:23-34.
[22] WANG Yu, LUO Dan, ZHAO Yunshi, et al. High-level expression and characterization of solvent-tolerant lipase[J]. Journal of Bioscience & Bioengineering, 2017,125(1): 23-29.
[23] ZHENG Xiaomei, CHU Xiaoyu, ZHANG Wei, et al, A novel cold-adapted lipase from Acinetobacter sp. XMZ-26: gene cloning and characterisation[J]. Applied Microbiology and Biotechnology, 2011, 90:971-980.
[24] KAMARUDIN N H A, RAHMAN R N Z R A, ALI M S M, et al. Unscrambling the effect of C-terminal tail deletion on the stability of a coldadapted, organic solvent stable lipase from Staphylococcus epidermidis AT2[J]. Molecular Biotechnology, 2014, 56 (8): 747-757.
[25] JI Xiuling, CHEN Guiyuan, ZHANG Qi, et al. Purification and characterization of an extracellular cold-adapted alkaline lipase produced by psychrotrophic bacterium Yersinia enterocolitica strain KM1[J]. Journal of Basic Microbiology, 2015, 55(6): 718-728.
[26] WU Gaobing, ZHANG Xiangnan, WEI Lu, et al. A cold-adapted, solvent and salt tolerant esterase from marine bacterium Psychrobacter pacificensis[J]. International Journal of Biological Macromolecules, 2015, 81:180-187.
[27] DE MORAIS W G, KAMIMURA E S, RIBEIRO E J, et al. Optimization of the production and characterization of lipase from Candida rugosa and Geotrichum candidum in soybean molasses by submerged fermentation[J]. Protein Expression and Purification, 2016, 123:26-34.