谷氨酸脱羧酶(glutamate decarboxylase,GAD,EC 4.1.1.15)可专一地催化L-谷氨酸(L-glutamic acid, L-Glu)α-羧基脱羧作用,在γ-氨基丁酸(γ-aminobutyric acid, GABA)生物合成和手性物质DL-谷氨酸(DL-glutamic acid,DL-Glu)拆分方面发挥着重要作用[1-3]。由于乳酸菌的安全性较好,便于在发酵食品中应用,乳酸菌GAD受到了极大关注,已发现Lactobacillus brevis [4]、Lactobacillus futsaii [5]、Lactobacillus rhamnosus [6]、Lactobacillus sakei [7]、Streptococcus salivarius ssp. thermophilus [8]、Pediococcus pentosaceus [9]、 Enterococcus faecium [10]和Lactobacillus plantarum [11]等微生物均具有GAD。然而,由于乳酸菌生长易受其代谢产生的乳酸抑制,生长缓慢,GAD产量低,难以满足生产需要。为了降低GABA合成和DL-Glu拆分成本,一些学者对GAD进行了固定化研究,以期重复利用GAD。已有文献报道利用多孔硅球[11]、壳聚糖、戊二醛[12]、羧基化磁性微球[13]、细菌纤维素[14]和金属亲和树脂[15-16]对不同来源GAD进行了固定化,然而,从固定化成本、固定化酶强度、固定化载体安全性、固定化酶活力等工业应用角度综合考量,现有方法尚有待完善,所以研发工艺简单、成本低和环境友好的固定化方法仍是酶工程领域的重要课题。
纤维素来源丰富,价格低廉,化学惰性,对大多数蛋白质非特异性亲和力低,是一种优质的载体。大多数纤维素降解酶包含3个结构域:纤维素结合结构域(cellulose-binding domain,CBD)、柔性连接区域和催化结构域[17-19],因此可利用CBD能与纤维素特异和不可逆结合的特性,将CBD开发为亲和标签,构建融合蛋白,实现与纤维素亲和吸附的目的[19-23]。要实现这一目标,CBD融合酶的构建、高效表达以及有效维持或改善原天然酶的酶学特性是该技术的关键。
不同种类的酶结构存在差异,甚至是同一种酶,其结构也可能因来源不同而差异显著。例如,Lactococcus lactis subsp. lactis GAD只有一个亚基,亚基分子量约为54 kDa[24];Streptococcus salivarius ssp. thermophilus GAD具有2个亚基,亚基分子量约为46.9 kDa[25];而Escherichia coli GAD含有6个相同的亚基,分子量约为53 kDa[26]。由此可见,虽然通过构建CBD融合酶实现酶的固定化已有报道,但是由于酶的结构差异性,CBD融合酶能否构建成功、融合酶的活性以及酶学性质是否会发生改变等方面尚存在极大不确定性。由此可见,该项技术尚未成熟,亟需更多研究进行补充和完善。
前期研究已表明,屎肠球菌(E. faecium)具有较高GAD活性,在GABA生物合成方面具有较好的开发利用前景[10]。本实验主要对E. faecium纤维素结合域谷氨酸脱羧酶(CBD-GAD)的构建、纯化及其酶学性质进行探讨,以期为纤维素亲和固定化E. faecium GAD提供理论和应用依据。
1 材料与方法
1.1 材料与试剂
E. faecium GDMCC 60203作为专利菌种保藏于广东省微生物菌种保藏中心,是岭南师范学院绿色生物制造研究室从泡菜中分离获得,是本实验GAD基因gadB来源菌株。大肠杆菌(Escherichia coli)DH5ɑ由玉林师范学院生物与制药学院林谦博士提供,用于CBD-GAD融合酶的表达。E. coli GDMCC 60445作为专利菌种保藏于广东省微生物菌种保藏中心,为本实验自行构建的可表达CBD-GAD融合酶的重组菌株。
层析硅胶G薄板,浙江省台州市路桥四甲生化塑料厂;γ-氨基丁酸(质量分数≥99%)、5′-磷酸吡哆醛(pyridoxal 5′-phosphate, PLP)、异硫氰酸苯酯(phenylisothiocyanate,PITC),美国Sigma公司;乙腈、乙酸、三乙胺(均为色谱纯),美国TEDIA公司;氨苄青霉素(美国药典级),生工生物工程(上海)有限公司;细菌基因组提取试剂盒和DNA胶回收试剂盒康,世纪生物技术公司;TransStart Fast Pfu DNA聚合酶,全式金生物技术公司;Taq DNA聚合酶、pMD19 Simple T载体、In-fusion HD基因克隆试剂盒,大连宝生物工程公司。其他试剂为市售生化试剂或分析纯试剂。
载体pRPOCB为自行构建的E. coli质粒,主要在pUC57质粒中插入了胁迫诱导型启动子PrpoS、cbm3(来自Clostridium thermocellum的家族Ⅲ纤维素结合域)编码序列和T7终止子序列。
引物efa-1、efa-2、Infu-13、Infu-22、Infu-24和Infu-27均委托生工生物工程(上海)股份有限公司合成。
LB培养基参照文献[27]配制,略有改变。胰蛋白胨10 g/L、酵母粉 5 g/L、NaCl 10 g/L,pH 6.0。
PSB(peptone-sucrose-beef extract)培养基参照文献[10]配制,略有改变。蛋白胨15 g/L、牛肉膏10 g/L、蔗糖12.5 g/L、乙酸钠6 g/L、L-谷氨酸一钠(monosodium glutamate,MSG)10 g/L、吐温-80 1.0 g/L,pH 7.0。
上述培养基按照配方配制,分装于规格为250 mL三角瓶,每瓶120 mL,121 ℃灭菌20 min,临用前加入240 μL 50 mg/mL氨苄青霉素。
再生无定形纤维素(regenerated amorphous cellulose,RAC)参照文献[28]制备。
1.2 仪器与设备
1200 Series高效液相色谱仪(配置G1354A四元梯度泵、G1316A 柱温箱、G1314B可变波长紫外检测器、Chemstation工作站),美国安捷伦公司;HZQ-F100恒温振荡培养箱,金坛市精达仪器制造有限公司;TGL16M台式高速冷冻离心机,盐城市凯特实验仪器有限公司;SHA-2冷冻水浴恒温振荡器,江苏省金坛市三和仪器有限公司;AUW120电子分析天平,日本Shimadzu公司。
1.3 实验方法
1.3.1 种子液的制备
E. faecium GDMCC 60203种子液制备:从4 ℃保藏的菌种斜面挑取1环,接入PSB培养基中,37 ℃静置培养12 h,然后按体积分数1%的接种量再次接入PSB培养基,37 ℃静置培养12 h。
E. coli DH5ɑ和E. coli GDMCC 60445种子液制备:吸取-25 ℃甘油保藏的菌种1 mL,接入LB培养基,37 ℃,120 r/min振荡培养12 h。
1.3.2 E. faecium CBD-GAD重组大肠杆菌的构建
1.3.2.1 E. faecium的gadB基因克隆
采用细菌基因组提取试剂盒,按照其说明书提取E. faecium基因组DNA,具体方法为:取E. faecium GDMCC 60203种子液5 mL,4 ℃、8 500 r/min离心15 min,收集菌体,加入180 μL酶解缓冲液重悬菌体,37 ℃保温30 min,再依次加入20 μL蛋白酶K和200 μL缓冲液GL,旋涡振荡混匀, 56℃保温30 min,然后加入200 μL无水乙醇,旋涡振荡混匀,即为细胞酶解液。将细胞酶解液加入到已装入收集管的吸附柱,以12 000 r/min离心1min,弃收集管中的废液,按此相同的加液、离心、弃废液操作,依次分别用500 μL缓冲液GW1、500 μL缓冲液GW2洗涤吸附柱。将经离心脱液的吸附柱置于室温数分钟,以彻底晾干,然后将吸附柱置于一个新的1.5 mL离心管中,向吸附柱中间悬空部位加入100 μL缓冲液GE,室温放置2~5 min,12 000 r/min离心1 min,收集离心管中的DNA溶液即为E. faecium GDMCC 60203基因组DNA,于-20 ℃保存备用。
根据NCBI中的E. faecium基因组序列,设计和合成引物efa-1和efa-2,用于扩增谷氨酸脱羧酶基因gadB:
efa-1: 5′-ATGTTATACGGAAAAGATAATCAAGAA G-3′
efa-2: 5′-TTAGTGAGTAAAGCCGTACGTTTTCAC-3′
以基因组DNA为模版,以efa-1和efa-2为引物,使用TransStart Fast Pfu DNA聚合酶扩增,PCR程序为:95 ℃、2 min;95 ℃、20 s,50 ℃、30 s,72 ℃、55 s,循环33次;72 ℃、5 min。扩增结束后,在反应体系中加入1 μL Taq DNA聚合酶,72 ℃、20 min,使PCR产物3′端得到突出的碱基A。电泳切胶,用DNA胶回收试剂盒回收PCR产物,与pMD19 Simple T载体连接,转化E. coli DH5α感受态细胞,挑取单菌落,提取重组质粒pMD-EfagadB,委托生工生物工程(上海)有限公司测序。
1.3.2.2 CBD-GAD融合酶表达载体及重组大肠杆菌的构建
设计和合成引物Infu-13、Infu-24、Infu-22和Infu-27:
Infu-13: 5′-GTTTGGGGTAAAGAACCGGGATCC ATGTTATACGGAAAAGATAATCAAGAAG-3′
Infu-24: 5′-GCCCTCGAGGAATTCTTAGTGAGTAAAGCCGTACGTTTTCAC-3′
Infu-22: 5′-GAATTCCTCGAGGGCTCTTCCAG-3′
Infu-27: 5′-GGATCCCGGTTCTTTACCCCAAACC-3′
以Infu-13和Infu-24为引物,以pMD-EfagadB作为模板,通过PCR扩增gadB基因,电泳切胶,回收纯化gadB基因;以Infu-22和Infu-27为引物,以pRPOCB质粒作为模板,通过PCR扩增制备线性化的载体,电泳切胶,回收纯化目的片段。使用TransStart Fast Pfu DNA聚合酶扩增,PCR程序为95 ℃、2 min;95 ℃、20 s,54 ℃、30 s,72 ℃、105 s,循环33次;72 ℃、5 min。
将gadB基因与线性化载体pRPOCB用In-fusion HD基因克隆试剂盒进行拼接,构建重组质粒pRPOCB-EfagadB。反应体系为:2 μL In-fusion酶、1 μL gadB基因、2 μL 线性化载体、5 μL ddH2O,总计10 μL。离心30 s,50 ℃连接15 min。
将pRPOCB-EfagadB重组质粒转化E. coli DH5α感受态细胞。挑取单菌落,提取质粒后,用BamH I、EcoR I双酶切质粒,进行电泳检测。转化了重组质粒pRPOCB-EfagadB的E. coli DH5α菌株即为E. coli GDMCC 60445。
1.3.2.3 CBD-GAD融合酶的表达
将20 mL E. coli GDMCC 60445接于LB液体培养基,于37 ℃、120 r/min振荡培养9~10 h,再取1 mL培养液转接到120 mL LB培养基,37 ℃振荡培养24 h作为发酵液。
1.3.2.4 CBD-GAD融合酶的提取与固定化
将120 mL E. coli GDMCC 60445发酵液于4 ℃、8 500 r/min离心15 min,收集菌体,加入30 mL生理盐水,搅拌分散洗涤菌体,再次离心收集菌体,然后加入pH 8.0、50 mmol/L Tris-HCl缓冲液10 mL,在冰水浴中以400 W功率,超声波破碎细胞(工作5 s,冷却5 s,全程50 min)。将细胞破碎液于8 500 r/min、 4 ℃离心15 min,上清液即为CBD-GAD融合酶粗酶液。
称取0.8 g RAC湿质量与8 mL CBD-GAD粗酶液混合,30℃、50 r/min振荡15 min,滤出液体,然后采用40 mL pH 8.0、50 mmol/L Tris-HCl缓冲液分5次洗涤固形物,再采用40 mL pH 4.8、0.2 mol/L乙酸-乙酸钠缓冲液分5次洗涤固形物,滤除洗涤液,即可获得RAC固定化CBM-GAD(RAC-CBD-GAD)。
1.3.2.5 RAC-CBD-GAD活性测定
为了避免E. coli受自身基因组表达的E. coli GAD的影响,本实验通过测定RAC-CBD-GAD活性以评价CBD-GAD融合酶的表达情况。
将0.2 g RAC-CBD-GAD与10 mL pH 4.8、0.25 mol/L L-Glu溶液(溶于0.2 mol/L乙酸盐缓冲液,含0.2 mmol/L PLP)混合,40 ℃、80 r/min水浴振荡反应30 min,立即吸取0.5 mL反应液与0.5 mL无水乙醇混合以终止反应, 8 500 r/min离心15 min,取上清液定性和定量测定GABA。
GABA定性检测:将上清液用蒸馏水适当稀释后,采用薄层层析(thin-layer chromatography, TLC)进行鉴定;展开剂为正丁醇∶冰乙酸∶水(体积比6∶1∶1),且含3 g/L茚三酮;发色温度90 ℃。
GABA定量检测:参照文献[29]采用HPLC进行测定。取100 μL标准溶液或样品液注入1.5 mL的锥形离心管,加入50 μL乙腈-水-三乙胺-PITC(体积比7∶1∶1∶1),混匀,室温放置1 h,加入150 μL流动相A-流动相B(体积比80∶20),旋涡混合器振荡1 min,然后加入600 μL正己烷,旋涡混合器振荡1 min,最后静置10 min。用注射器吸取下层溶液,经0.22 μm滤膜过滤备测。HPLC色谱条件为:色谱柱ZORBAX Eclipse XDB C18(4.6 mm×250 mm,5 μm);检测波长254 nm;进样量20 μL,柱温25℃;采用流动相A与流动相B(体积比80∶20)以0.6 mL/min线性等梯度洗脱。流动相A:pH 5.6、0.1 mol/L乙酸盐缓冲液,1 L缓冲液中分别含三乙胺0.5 mL、乙酸0.7 mL和乙腈 5.0 mL。流动相B:体积分数为60%的乙腈溶液。
RAC-CBD-GAD活力单位定义为:测定条件下1 min生成1 μmol GABA所需要的酶量为1个酶活单位(U)。以单位质量RAC-CBD-GAD所具有的GAD活力表示(U/g)。
1.3.2.6 重组E. coli GDMCC 60445的稳定性
吸取-25 ℃甘油保藏的菌种1 mL,转接于不含氨苄青霉素的LB培养基,37 ℃、120 r/min振荡培养12 h,再吸取1 mL培养液,转接于不含氨苄青霉素的LB培养基,37 ℃、120 r/min振荡培养12 h,重复转接5次,然后吸取2 mL接入120 mL不含氨苄青霉素的LB培养基,37 ℃、120 r/min振荡培养24 h。收集细胞,制备RAC-CBD-GAD并测定其活性。以含0.1 mg/mL氨苄青霉素的LB培养基按照同样操作收集的细胞作为对照。
1.3.3 CBD-GAD融合酶的纯化
按RAC与CBD-GAD粗酶液的质量体积比为1∶20的比例按上述方法进行固定化,将固定化酶RAC-CBD-GAD用pH 8.0、50 mmol/L的Tris-HCl缓冲液充分洗涤,然后将每1 g RAC-CBD-GAD与20 mL乙二醇混合,静置脱吸附10 min,滤出酶液,将酶液用超滤管以3 000 r/min进行离心超滤,再通过多次加入生理盐水、离心超滤的循环操作去除乙二醇,即可获得基本不含乙二醇的CBD-GAD酶液。参照SDS-PAGE变性丙烯酰胺凝胶快速制备试剂盒说明书,采用SDS-PAGE电泳鉴定CBD-GAD,浓缩胶质量浓度5%,分离胶质量浓度8%,胶厚1.0 mm。蛋白质浓度参照Bradford法蛋白浓度测定试剂盒说明书进行测定。
1.3.4 CBD-GAD纯酶的酶学性质研究
1.3.4.1 CBD-GAD活性测定方法
除特别说明外,CBD-GAD活性测定条件为:取1.5 mL pH 4.8、0.25 mol/L L-Glu溶液(溶于0.2 mol/L乙酸盐缓冲液,含0.2 mmol/L PLP),加入1.5 mL CBD-GAD纯酶,40 ℃水浴反应30 min,加入4 mL无水乙醇,混匀、终止反应,8 500 r/min离心15 min,取上清液适当稀释后采用HPLC测定GABA含量。CBD-GAD活力单位定义为:测定条件下1 min生成1 μmol GABA所需要的酶量为1个酶活单位(U)。除对照组平均CBD-GAD活力为100%外,未设置对照组的实验以活力最高的实验组平均CBD-GAD活力为100%计算相对酶活(%)。
1.3.4.2 pH对CBD-GAD的影响
pH对CBD-GAD反应活性的影响:分别以pH 4.0、4.2、4.4、4.6、4.8、5.0、5.2和5.4的0.25 mol/L L-Glu溶液(溶于0.2 mol/L乙酸盐缓冲液,含0.2 mmol/L PLP)作为底物进行酶活测定。
pH对CBD-GAD稳定性的影响:将CBD-GAD酶液分别与pH 3.6、4.0、4.4、4.8、5.2、5.6 的0.05 mol/L乙酸盐缓冲液按体积比1∶1混合,于40 ℃保温5 h,然后对处理后的CBD-GAD酶液进行酶活测定。
1.3.4.3 温度对CBD-GAD的影响
温度对CBD-GAD反应活性的影响:分别在40、45、50、55、60、65和70 ℃下进行酶活测定。
温度对CBD-GAD稳定性的影响:将CBD-GAD酶液与pH 4.8、0.05 mol/L乙酸盐缓冲液按体积比1∶1混合,分别于-25、-20、-7、4、40、50、60和70 ℃保温3 h,然后室温下放置1 h,再取处理后的CBD-GAD酶液进行酶活测定。以4 ℃处理组为对照。
1.3.4.4 CBD-GAD底物特异性
分别取pH 4.8的0.25 mol/L L-Glu、D-Glu、L-Asp溶液1.5 mL,加入0.5 mL CBD-GAD酶液,快速混匀,于40 ℃水浴反应2 h,然后100 ℃保温10 min终止酶活,4 ℃、8500 r/min离心15 min,用HPLC测定L-Glu、D-Glu、L-Asp含量,测定方法参照上述HPLC测定GABA的方法。以121 ℃灭活10 min的CBD-GAD酶液进行同样操作作为对照。
以底物浓度的平均变化率[(实验组浓度-对照组浓度)/对照组浓度×100%]对反应活性进行比较。根据试验过程存在系统误差,将底物的平均变化率绝对值≤5%判定为阴性(-),变化率绝对值>5%判定为阳性(+)。
1.3.4.5 CBD-GAD动力学常数
分别以pH 4.8的0.4、0.8、1.2、2.4、3.6、4.8、6.4、9.6、12.8、19.2、25.6、51.2、76.8、89.6、102.4、128、153.6、204.8 mmol/L L-Glu溶液作为底物进行酶活测定,选择呈一级反应关系的底物浓度和反应速度,采用Lineweaver-Burk双倒数作图法计算GAD的Km和Vmax。
1.3.4.6 GABA对CBD-GAD活性的影响
以含0.1 mol/L GABA的0.25 mol/L L-Glu混合溶液为底物进行酶活测定,同时以不含GABA的0.25 mol/L L-Glu溶液作为对照。
1.4 数据分析
所有实验均为3个平行,结果以“平均值±标准偏差”表示。采用IBM SPSS Statistics 19.0软件以独立样本t检验进行统计分析。
2 结果与分析
2.1E.faecium CBD-GAD重组大肠杆菌的构建
2.1.1 gadB基因克隆
以E. faecium GDMCC 60203的基因组DNA为模板,扩增出的产物为大小1.4 kb(图1)。测序结果表明,该基因与Enterococcus sp. JK29的gadB基因(1 401 bp,GenBank登录号:KM649684)100%一致。
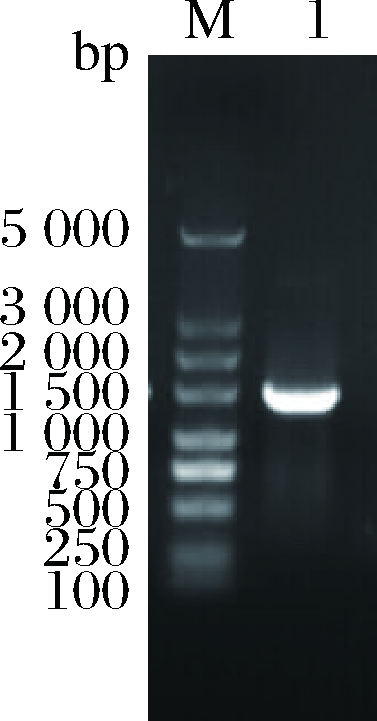
M-Marker;1-PCR产物
图1 PCR扩增E. faecium GDMCC 60203 gadB基因的电泳图
Fig.1 Electrophoretic map of gadB gene by PCR amplified from E. faecium GDMCC 60203
2.1.2 CBD-GAD融合酶表达载体及重组大肠杆菌的构建
将E. faecium GDMCC 60203的gadB基因与pRPOCB线性化载体拼接后,得到pRPOCB-EfagadB重组质粒(图2),该重组质粒包含了胁迫诱导型启动子PrpoS、E. coli中 5′-非翻译区和来自编码C. thermocellum家族3纤维素结合域的序列cbm3和E. faecium LNSF2 GAD的基因gadB。从序列推断,用BamH I、EcoR I双酶切重组质粒,应切出3 723 bp、1 407 bp两条带,对酶切产物进行电泳,电泳结果(图3)与理论推断一致,表明重组质粒pRPOCB-EfagadB构建成功。
将pRPOCB-EfagadB重组质粒转化E. coli DH5α,获得了携带CBD-GAD融合酶表达载体的重组E. coli GDMCC 60445。
2.1.3 CBD-GAD融合酶的表达
RAC对CBD具有特异性吸附性能,采用RAC对E. coli GDMCC 60445的CBD-GAD进行吸附固定化,避免了E. coli自身基因组所表达的GAD的影响。经采用TLC对RAC-CBD-GAD的转化产物进行检测,图4显示存在与GABA标准品迁移率一致的斑点,HPLC定量分析表明RAC-CBD-GAD活力为(349.54±33.47) U/g,结果表明E. coli GDMCC 60445可高效表达具有GAD催化活性的CBD-GAD。

图2 表达载体pRPOCB-EfagadB示意图
Fig.2 Map of the expression vector pRPOCB-EfagadB

M-Marker;1-pRPOCB-EfagadB的BamH I和EcoR I酶切产物
图3 pRPOCB-EfagadB酶切产物电泳图
Fig.3 Electrophoretic map of the digestion products of pRPOCB-EfagadB

1- GABA标准品;2-RAC-CBD-GAD转化反应液;3-L-Glu标准品
图4 RAC-CBD-GAD转化液薄层层析图谱
Fig.4 Thin-layer chromatography of the conversion liquid of RAC-CBD-GAD
2.1.4 重组E. coli GDMCC 60445的稳定性
对含氨苄青霉素抗性标记的重组菌,通常需要在培养基中添加氨苄青霉素以防止重组菌丢失质粒。然而,氨苄青霉素价格昂贵,安全性差,工业应用成本高、风险大。经对E. coli GDMCC 60445稳定性进行测试,实验组RAC-CBD-GAD活力为(347.93±27.63) U/g,与对照组(329.29±10.37) U/g无显著性差异(P>0.05)。结果表明,连续转接培养5批次,E. coli GDMCC60445性能稳定,未受氨苄青霉素影响。
2.2 CBD-GAD融合酶的纯化
由于RAC对CBD具有特异性吸附性能,因此只需对RAC-CBD-GAD充分洗涤以去除未吸附的杂蛋白,然后采用乙二醇进行脱吸附处理,即可实现CBD-GAD纯化。图5显示,纯化后的CBD-GAD酶液在SDS-PAGE电泳图上呈现单一蛋白质条带,说明CBD-GAD已经达到了电泳纯。CBD-GAD纯酶的比酶活为(50.57±4.44) U/mg。

1-蛋白质Marker;2-CBD-GAD粗酶液;3,4-脱吸附CBD-GAD
图5 CBD-GAD融合酶的SDS-PAGE电泳图
Fig.5 SDS-PAGE electrophorogram of fusion enzyme CBD-GAD
根据电泳分离胶的浓度和蛋白质Marker分子量分布情况,选择SDS-PAGE电泳分子量分布较均匀的条带(15~100 kDa)的相对迁移距离与蛋白质分子量标准的对数值绘制标准曲线(图6),由曲线计算可知CBD-GAD的相对分子量为74.02 kDa,与CBD-GAD理论分子量71.52 kDa基本相符。电泳结果也表明CBD-GAD融合酶在E. coli GDMCC 60445中获得成功表达。
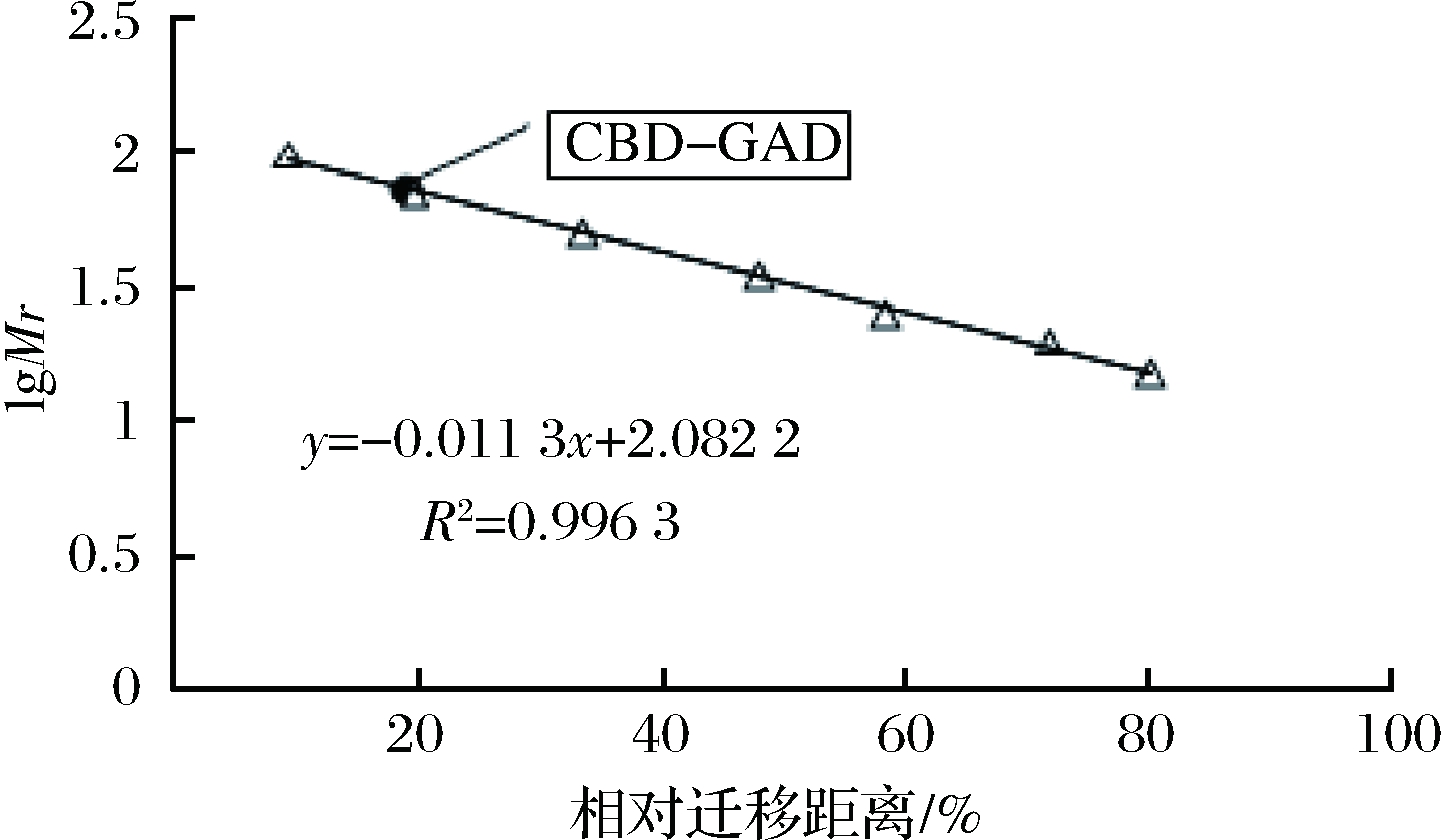
图6 SDS-PAGE测定GAD分子量
Fig.6 Molecular weight of GAD on SDS-PAGE
2.3 CBD-GAD纯酶的酶学性质
2.3.1 pH对CBD-GAD的影响
图7-a显示,当反应pH小于4.6,CBD-GAD活力随pH增加而增强;当反应pH为4.6,CBD-GAD活力最强;当反应pH大于4.6, CBD-GAD活力随反应pH增加而逐渐下降,但在pH 4.6~5.2范围内下降较为平缓,CBD-GAD活力差异不显著(P>0.05)。结果表明,CBD-GAD在pH 4.6~5.2可保持90%以上的活性,适宜反应pH范围较宽,最适反应pH值为4.6。从图7-b可见,pH值小于4.8,CBD-GAD随pH降低失活越严重,在pH 4.8~5.6相对较稳定,40 ℃保温5 h仍可保持90%以上活力,在pH 4.8最稳定。

a-pH对CBD-GAD催化反应活性的影响;b-pH对CBD-GAD稳定性的影响
图7 pH对CBD-GAD的影响
Fig.7 Effect of pH on CBD-GAD
杨胜远等[30]对E. faecium GAD天然酶纯酶的酶学性质研究表明,GAD最适反应pH为5.0,当pH偏离最适反应pH时,GAD活力快速下降,维持75%以上活力的pH范围仅为4.8~5.2。由此可见,融合CBD后,在pH对GAD催化活性影响方面发生了较大变化。
2.3.2 温度对CBD-GAD的影响
如图8-a所示,CBD-GAD的最适反应温度为60 ℃,在55~60 ℃范围可保持90%以上的CBD-GAD酶活;当温度高于65 ℃,酶活快速下降。图8-b显示,CBD-GAD在4~40 ℃相对较稳定,经pH 4.8、0.05 mol/L乙酸盐缓冲液处理3 h后,仍可保持95%以上的酶活;冰冻处理(低于-7 ℃)和高于50 ℃处理对CBD-GAD稳定性影响较大,酶活损失较大,当温度达到70 ℃,CBD-GAD基本完全失活。
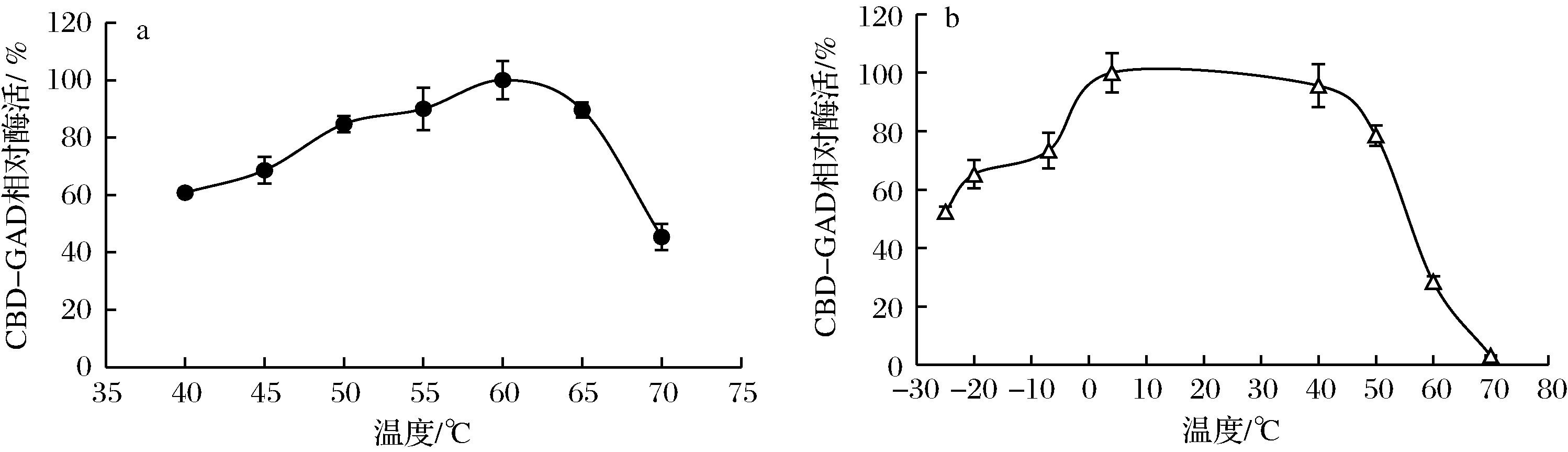
a-温度对CBD-GAD催化反应活性的影响;b-温度对CBD-GAD稳定性的影响
图8 温度对CBD-GAD的影响
Fig.8 Effect of temperature on CBD-GAD
2.3.3 CBD-GAD底物特异性
由表1可见,CBD-GAD仅对L-Glu具有催化活性,对仅有一个-CH2差异的L-Asp和仅具有立体结构差异的D-Glu均无催化活性。结果表明CBD-GAD具有很强的底物专一性。
表1 CBD-GAD对不同氨基酸的催化反应活性
Table 1 Catalytic activity of CBD-GAD for different amino acids

氨基酸对照组/(mmol·L-1)实验组/(mmol·L-1)浓度差绝对值/(mmol·L-1)变化率/%反应活性L-Glu162.64±2.30133.84±0.7028.8016.68+D-Glu166.69±4.68164.15±6.922.541.36-L-Asp169.61±5.89165.90±4.893.711.77-
注:“+”为具有催化活性;“-”为不具有催化活性。
2.3.4 CBD-GAD动力学常数
从图9可知,L-Glu浓度低于12.8 mmol/L时,反应呈一级反应; L-Glu浓度在12.8~76.8 mmol/L范围内,反应呈混合级反应;L-Glu浓度高于76.8 mmol/L,反应呈零级反应;CBD-GAD催化反应没有底物抑制现象。结果表明,CBD-GAD催化反应符合Michaelis-Menten的快速平衡学说。根据图10计算CBD-GAD的Km和Vmax分别为10.58 mmol/L和3.83 μmol/(mL·min)。
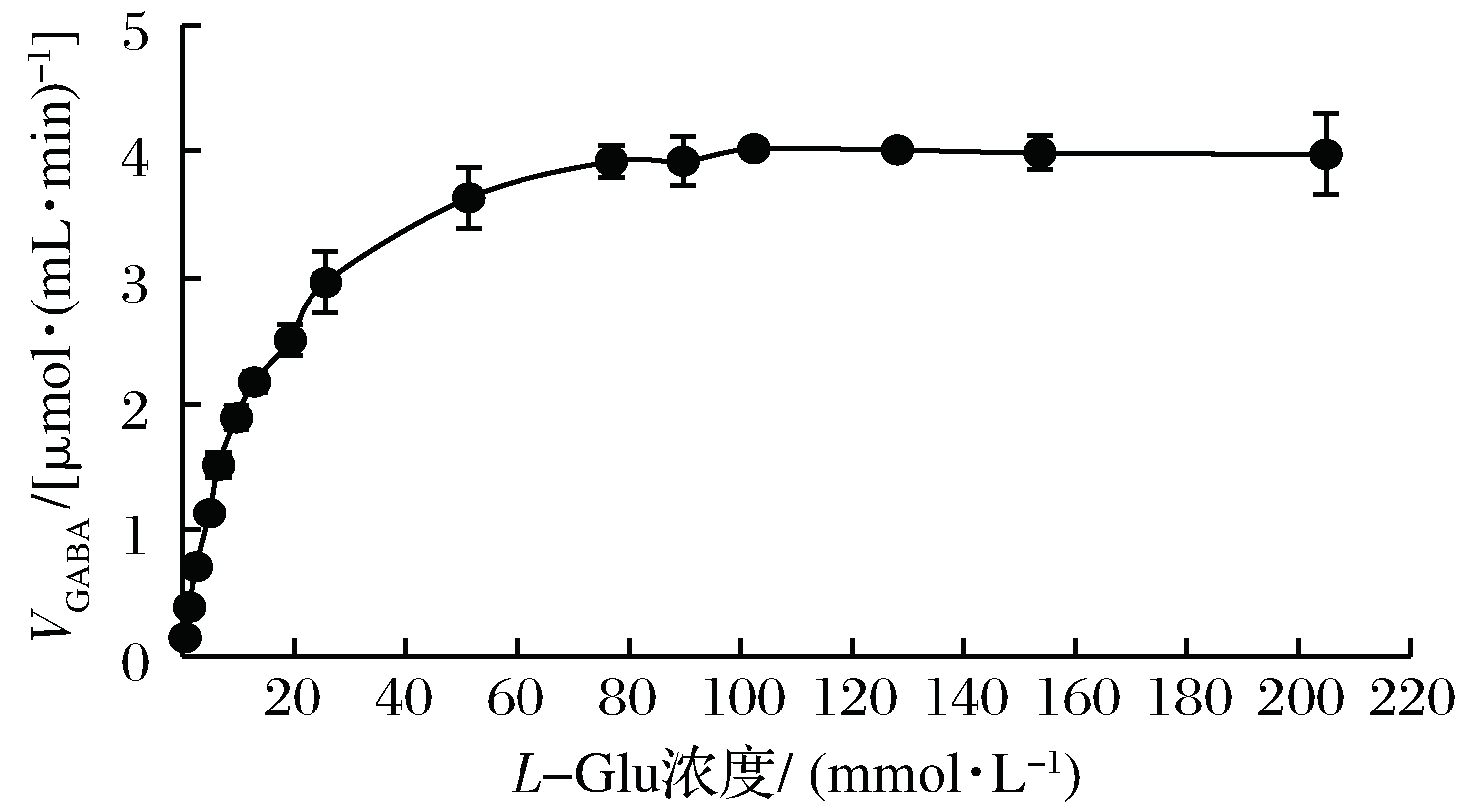
图9 L-Glu浓度对CBD-GAD催化反应速度的影响
Fig.9 Effect of L-Glu concentration on the catalytic reaction rate of CBD-GAD

图10 CBD-GAD动力学常数Lineweaver-Burk图
Fig.10 Lineweaver-Burk plot of the kinetic constants of CBD-GAD
2.3.5 GABA对CBD-GAD活性的影响
由图11可知,初始L-Glu底物溶液中添加 0.1 mol/L GABA的实验组与不含GABA的对照组的CBD-GAD活性无显著性差异(P>0.05),说明CBD-GAD催化活性无产物抑制现象。
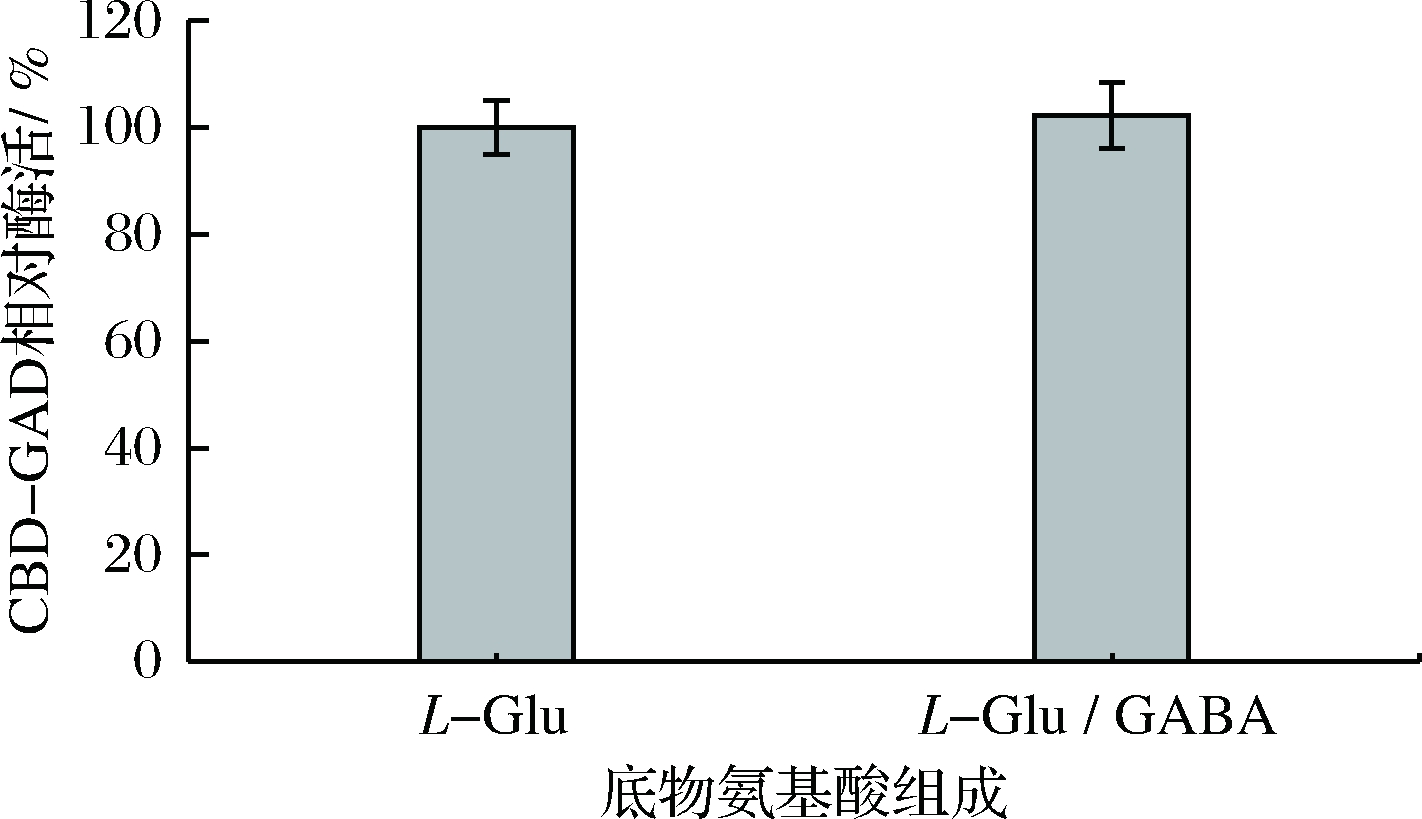
图11 GABA对CBD-GAD活性的影响
Fig.11 Effects of GABA on CBD-GAD activity
3 讨论
本实验通过以PrpoS作为启动子,成功构建了可高效表达CBD-GAD的E. coli GDMCC 60445,该重组菌株在无氨苄青霉素LB培养基中性能稳定。构建的CBD-GAD对RAC亲和性强,只经RAC一步纯化即可获得电泳纯化CBD-GAD。与GAD天然酶相比, CBD-GAD对pH的依赖性得到了较大改善,酶的催化专一性未受影响,催化反应不受底物和产物抑制,酶学性质优良。通过构建CBD-GAD融合酶,利用纤维素可快速实现GAD固定化和纯化,方法简便、成本低,具有很好的推广价值。
[1] ZHU Hui, SADIQ F A, LI Yun, et al. Application of ion-exchange resin as solid acid for buffer-free production of γ-aminobutyric acid using Enterococcus faecium cells[J]. LWT-Food Science and Technology, 2018, 98: 341-348.
[2] JORGE J M P, LEGGEWIE C, WENDISCH V F. A new metabolic route for the production of gamma-aminobutyric acid by Corynebacterium glutamicum from glucose[J]. Amino Acids, 2016, 48(11): 2 519-2 531.
[3] 杨成丽, 马子玉,胡晓丽,等. 化学-酶催化法制备D-谷氨酸与γ-氨基丁酸[J]. 化学与生物工程, 2013, 30(11): 55-56.
[4] VILLEGAS J M, BROWN L, DE GIORI G S, et al. Optimization of batch culture conditions for GABA production by Lactobacillus brevis CRL 1942, isolated from quinoa sourdough[J]. LWT-Food Science and Technology, 2016, 67(22): 22-26.
[5] SANCHART C,RATTANAPON O,HALTRICH D, et al. Lactobacillus futsaii CS3, a new GABA-producing strain isolated from Thai fermented shrimp (Kung-Som)[J]. Indian Journal of Microbiology, 2017, 57(2): 211-217.
[6] SONG H Y, YU R C. Optimization of culture conditions for gamma-aminobutyric acid production in fermented adzuki bean milk [J]. Journal of Food and Drug Analysis, 2018, 26(1): 74-81.
[7] SA H D, PARK J Y, JEONG S J, et al. Characterization of glutamate decarboxylase (GAD) from Lactobacillus sakei A156 isolated from Jeot-gal[J]. Journal of Microbiology and Biotechnology, 2015, 25(5): 696-703.
[8] 杨胜远, 陆兆新,吕凤霞,等. 唾液链球菌嗜热亚种Y-2细胞转化法制备γ-氨基丁酸[J]. 食品科学, 2011, 32(1): 162-167.
[9] AGUNG Y I B, KUSUMAWATI I G A W, SUMADEWI N L U, et al. Isolation and identification of lactic acid bacteria from Indonesian fermented foods as γ-aminobutyric acid-producing bacteria[J]. International Food Research Journal, 2018, 25(4): 1 753-1 757.
[10] 杨胜远, 李云. 双向单因素与田口法优化屎肠球菌产谷氨酸脱羧酶培养基[J]. 食品科学, 2018, 39(4): 90-98.
[11] LEE S J, LEE H S, LEE D W. Production of γ-amminobutyric acid using immobilized glutamate decarboxylase from Lactobacillus plantarum[J]. Microbiology and Biotechnology Letters, 2015, 43(3):300-305.
[12] 姜启兴, 何莎,张可,等. 影响壳聚糖固定化谷氨酸脱羧酶的因素[J]. 食品与机械, 2010, 26(5): 15-18.
[13] 李佳男, 谢湉,胡升,等. 羧基化磁性微球固定化谷氨酸脱羧酶[J]. 化工学报, 2017, 68(4): 1 550-1 557.
[14] YAO Wanying, WU Xiao, ZHU Jun, et al. In vitro enzymatic conversion of γ-amminobutyric acid immobilization of glutamate decarboxylase with bacterial cellulose membrane (BCM) and non-linear model establishment[J]. Enzyme and Microbial Technology, 2013, 52(4-5): 258-264.
[15] LEE J Y, JEON S J. Characterization and immobilization on nickel-chelated sepharose of a glutamate decarboxylase A from Lactobacillus brevis BH2 and its application for production of GABA[J]. Bioscience, Biotechnology, and Biochemistry, 2014, 78(10): 1 656-1 661.
[16] LEE S, AHN J, KIM Y G, et al. Gamma-aminobutyric acid production using immobilized glutamate decarboxylase followed by downstream processing with cation exchange chromatography[J]. International Journal of Molecular Sciences, 2013, 14(1): 1 728-1 739.
[17] MÉNDEZ-L TER J A, GIL-MU
TER J A, GIL-MU OZ J, NIETO-DOM
OZ J, NIETO-DOM NGUEZ M, et al. A novel, highly efficient β-glucosidase with a cellulose-binding domain: characterization and properties of native and recombinant proteins[J]. Biotechnology for Biofuels, 2017, 10: 256-271.
NGUEZ M, et al. A novel, highly efficient β-glucosidase with a cellulose-binding domain: characterization and properties of native and recombinant proteins[J]. Biotechnology for Biofuels, 2017, 10: 256-271.
[18] JOHNS M A, BERNARDES A, de AZEV DO E R, et al. On the subtle tuneability of cellulose hydrogels: implications for binding of biomolecules demonstrated for CBM 1[J]. Journal of Materials Chemistry B, 2017, 5: 3 879-3 887.
DO E R, et al. On the subtle tuneability of cellulose hydrogels: implications for binding of biomolecules demonstrated for CBM 1[J]. Journal of Materials Chemistry B, 2017, 5: 3 879-3 887.
[19] GOLDSTEIN M A, TAKAGI M, HASHIDA S, et al. Characterization of the cellulose-binding domain of the Clostridium cellulovorans cellulose-binding protein A[J]. Journal of Bacteriology, 1993, 175(13): 5 762-5 768.
[20] BULAKHOV A G, GUSAKOV A V, ROZHKOVA A M, et al. Properties of chimeric polysaccharide monooxygenase with an attached cellulose binding module and its use in the hydrolysis of cellulose-containing materials in the composition of cellulase complexes [J]. Catalysis in Industry, 2018, 10(2): 152-158.
[21] ITO S, KUNO A, SUZUKI R, et al. Rational affinity purification of native Streptomyces family 10 xylanase[J]. Journal of Biotechnology, 2004, 110(2): 137-142.
[22] RICHINS R D, MULCHANDANI A, CHEN W. Expression, immobilization, and enzymatic characterization of cellulose-binding domain-organophosphorus hydrolase fusion enzymes[J]. Biotechnology and Bioengineering, 2015, 69(6): 591-596.
[23] PARK H, AHN J, LEE J, et al. Expression, immobilization and enzymatic properties of glutamate decarboxylase fused to a cellulose-binding domain[J]. International Journal of Molecular Sciences, 2012, 13(1): 358-368.
[24] NOMURA M, NAKAJIMA I, FUJITA Y, et al. Lactococcus lactis contains only one glutamate decarboxylase gene[J]. Microbiology, 1999, 145(6):1 375-1 380.
[25] YANG Shengyuan, Lin Qian, LU Zhaoxin, et al. Characterization of a novel glutamate decarboxylase from Streptococcus salivarius ssp. thermophilus Y2[J]. Journal of Chemical Technology and Biotechnology, 2008, 83(6):855-861.
[26] STRAUSBAUCH P H, FISCHER E H. Chemical and physical properties of Escherichia coli glutamate decarboxylase[J]. Biochemistry, 1970, 9(2): 226-232.
[27] KANG Zhen, WANG Qian, ZHANG Hao-jun, et al. Construction of a stress-induced system in Escherichia coli for efficient polyhydroxyalkanoates production[J]. Applied Microbiology and Biotechnology, 2008, 79: 203-208.
[28] HONG Jiong, YE Xinhao, WANG Yiran, et al. Bioseparation of recombinant cellulose-binding module-proteins by affinity adsorption on an ultra-high-capacity cellulosic adsorbent[J]. Analytica Chimica Acta, 2008, 621(2): 193-199.
[29] 杨胜远, 江敏,胡小军,等. 一种富含天然γ-氨基丁酸的新食材[J]. 食品研究与开发, 2017, 38(22): 153-157.
[30] 杨胜远, 郭文怡,李紫君,等. 732阳离子交换树脂对全细胞谷氨酸脱羧酶活性的促进机制[J]. 食品工业科技,2018,39(20): 105-111.