自然发酵辣椒酱营养丰富,色泽鲜艳,其风味不仅取决于辣椒原料和生产工艺,还与发酵过程中的微生物代谢密切相关[1]。不同的微生物会利用辣椒原料进行发酵,可能产生不同的有机酸和氨基酸等风味物质,从而对辣椒酱的品质造成一定的影响[2]。新疆地区的辣椒酱多以农户家庭自制为主,众所周知,农家自制辣椒酱的环境相对开放,基质中存在大量微生物,可能蕴藏一定数量对辣椒酱品质形成有积极意义或有潜在益生特性的菌株,也可能含有一些致病菌及条件致病菌[3-4]。传统微生物检测主要通过观察形态、革兰氏染色和生化反应等进行比对分析,虽然操作简单且可直接得到结果,但不适用于菌群大批量分析鉴定[5]。随着现代生物技术的发展和测序成本的降低,宏基因组测序、扩增子测序和16S rDNA测序等手段越来越受到国内外研究学者的关注[6]。如沈馨等[7]利用Miseq高通量测序技术研究了辣椒酱核心细菌类群,发现湖北当阳地区辣椒酱中的优势菌是芽孢杆菌(Bacillus)和葡萄球菌(Staphylococcus);赵玲艳等[8]研究了自然发酵辣椒微生物多样性及其宏转录组,发现发酵线椒中的优势细菌为Weissella、Lactobacillus、Aureimonas和Rhizobium,优势真菌为Hanseniaspora、Debaryomyces 、Rhodotorula和Trichosporon等;钟燕青[9]研究湖南地区自然发酵剁辣椒,发现主要优势细菌是植物乳杆菌(Lacto-bacillus plantarum)、戊糖片球菌(Pediococcus pentosaceus)和短乳杆菌(Lactobacillus breris);韩俊燕等[10]研究发酵辣椒多样性,发现剁辣椒中的优势菌是魏斯氏菌、明串珠菌和乳杆菌。
新疆地域辽阔,辣椒产业是当地红色产业,辣椒适栽区具有复杂多样的生态地理环境,蕴含着丰富的微生物菌株资源[11]。但遗憾的是目前关于新疆地区辣椒酱细菌微生物多样性的研究一直未得到应有的重视,其多样性研究在新疆尚属空白。沙漠等[12]围绕新疆辣椒酱在加工中的问题,筛选了适用于发酵辣椒制品发酵性能稳定的乳酸菌;但是未对辣椒酱发酵过程中的真菌进行深入研究,尤其是酵母菌在发酵过程中的作用。
本研究利用MiSeq技术对新疆哈密(A)、伊犁(B)、昌吉(C)、阿克苏(D)和乌鲁木齐(E)地区自然发酵辣椒酱中的微生物组成进行解析,以期揭示自然发酵辣椒酱中细菌和真菌的多样性,为发酵辣椒的安全生产提供理论指导,同时为适合辣椒酱发酵微生物的筛选提供基础。
1 材料与方法
1.1 样品采集
利用无菌袋,对5个地区的自然发酵辣椒酱进行取样,每个地区取5份样品,具体如表1所示。
表1 试验样品
Table 1 Experimental samples

样品来源样品编号哈密地区AY5、Y62、Y61、Y65、Y9伊犁地区BY12、Y18、Y72、Y73、Y75昌吉自治州奇台地区CY13、Y15、Y80、Y79、Y66阿克苏地区DY23、Y28、Y30、Y44、Y78乌鲁木齐地区EY57、Y58、Y59、Y60、Y22
1.2 试剂与仪器
琼脂糖,西班牙Biowest Agarose公司;AxyPrep DNA凝胶回收试剂盒、QIAamp Fast DNA Stool Mini Kit,AXYGEN公司;Gold View I核酸染料,北京中生瑞泰科技有限公司;所有无机、有机溶剂均为国产分析纯。
5430R型高速冷冻离心机,德国Eppendorf公司;My Cycler型聚合酶链式反应(polymerase chain reaction,PCR)仪,美国Bio-rad公司; Bio-Best 200E 型凝胶成像分析系统,赛多利斯公司; Miseq测序仪,Illumina公司。
1.3 实验方法
1.3.1 DNA提取和PCR扩增
称取10 g辣椒酱样品,DNA提取严格按照QIAamp Fast DNA Stool Mini Kit说明书进行。用Primer 6.0软件设计引物,细菌16S rDNA V3-V4扩增通用引物:341F(5′-CCTACGGGRSGCAGCAG-3′)和806R(5′-GGACTACVVGGGTATCTAATC-3′);真菌I T S 2区域扩增通用引物:2045F(5′-GCATCGATGAAGAACGCAGC-3′)和2930R(5′-TCCTCCGCTTATTGATATGC-3′)。PCR反应体系为:2×KAPA Library Amplification ReadyMix 15 μL,引物(10 μM)F和R各为1 μL,模板DNA为50 ng,最后加ddH2O 至30 μL。细菌PCR反应条件:95 ℃预变性180 s;95 ℃变性45 s,50 ℃退火30 s,72 ℃延伸60 s,反应35 个循环;72 ℃延伸48 s,-20 ℃保存。真菌PCR反应条件为:98 ℃预变性180 s;98 ℃变性20 s,58℃退火15 s,72℃延伸20 s,执行35 个循环;最后72 ℃维持300 s,-20 ℃保存。利用Thermo NanoDrop 2 000紫外微量分光光度计和2%琼脂糖凝胶电泳检测PCR产物,而后根据AxyPrep DNA凝胶提取试剂盒说明书切胶回收PCR产物。
1.3.2 Illumina MiSeq HiSeq PE250 测序
文库质检合格后,使用Qubit 2.0进行文库定量,并根据每个样品的数据量要求,进行相应比例的混合。采用MiSeq平台PE250策略进行双末端测序,通过重叠关系进行拼接,获得高变区的长reads。
1.4 数据处理
16S序列长为220~500 bp,保证每条reads含N数不超过3个,且平均质量值不低于20。对序列完全相同的Clean Reads,过滤掉其中的Singletons,根据其丰度大小进行排序。使用UPARSE(http://drive5.com/uparse/)将97%相似度进行OTU聚类,利用Userach 7.0鉴定并移除嵌合体序列。使用RDP数据库(http://rdp.cme.msu.edu/),将OTU置信度阈值设置为0.8,利用RDPC lassifer(http://rdp.cme.msu.edu/)将具有代表性的序列进行物种注释。OTU proflingtable和alpha多样性分析通过Qiime的python脚本实现[13-14]。使用R语言和Network工具分析和作图;使用主坐标分析(principal coordinates analysis,PCoA)的方法展示各个样品间的差异大小;使用单因素方差分析比较样本间是否存在显著性差异,采用fdr方法对p值进行多重检验校正。
2 结果与分析
2.1 OTU分析
由图1-a看出,5个地区的25个样品共含有1 562个细菌OTU,5个地区共有的细菌种类有440个,在不同组样品间有显著差异的OTU(P<0.05)共57个。其中D地区OTU数量最多,有1 182个,A地区最少,有724个;B、C和E 3个地区OUT差异不显著(P>0.05),说明细菌种类比较相似。由图1-b看出,25个样品中含有396个真菌OTU,5个地区共有的真菌种类有88个,在不同组样品间有显著差异的OTU(P<0.05)共14个。其中A地区OTU数量最多,有249个;C地区最少,有198个;B、D和E 3个地区OTU差异不显著(P>0.05),真核微生物种类相似度较高。综合分析,在新疆自然发酵辣椒酱中,不同地区真菌种类明显少于细菌种类,伊犁地区和乌鲁木齐地区微生物多样性比较相似。

a-细菌;b-真菌
图1 微生物群落维恩图
Fig.1 Venn diagram of microbial community
2.2 α-多样性分析
Coverage指数代表测序对物种的覆盖度,Shannon指数能够对样品中微生物多样性进行衡量,Shannon数值越大说明微生物的多样性越高。Chao指数可以估算样品中物种的数目。
表2 α-多样性指数
Table 2 α-diversity index of samples

地区样品编号真菌细菌CoverageChaoindexShannonindexCoverageChaoindexShannonindexY9152.501.421256.061.29Y51114.691.871322.951.86AY61196.52.291660.354.69Y621119.271.411550.521.73Y651153.071.561358.571.62Y121161.431.761656.823.76Y18165.502.471748.736.13BY72192.231.331351.752.74Y73198.001.711531.876.78Y75166.772.121160.052.79Y13189.501.291333.802.32Y15189.003.601392.563.50CY66190.750.571461.701.93Y791143.551.931412.531.46Y80121.200.051515.085.06Y23194.601.311819.975.40Y28155.001.301730.686.34DY30121.000.091754.226.27Y441113.050.631560.132.38Y781132.643.191484.522.26Y221119.621.481176.271.45Y571114.082.081835.584.43EY581124.751.390.99858.363.30Y591119.091.831936.256.65Y601146.831.681293.002.87
由表2可知,辣椒酱中微生物Coverage指数都大于0.99,说明样本测序结果可以反应样品的真实情况。根据Chao指数和Shannon指数可知,不同地区辣椒酱样品中细菌平均Chao指数差异较明显。E地区的平均Chao指数最大,为124.87;D地区的平均Chao指数最小,为83.26,其中Y78和Y44样品的Chao指数较大,属于异常值;B地区样品的平均细菌Shannon指数为1.88,显著高于其他地区。综上说明,乌鲁木齐地区的细菌物种数目多于其他地区,伊犁地区细菌群落多样性优于其他地区。
就真菌而言,D地区的平均Chao指数和平均Shannon指数最大,分别为669.904和4.53,说明阿克苏地区真菌群落丰度和多样性都高于其他地区。从α-多样性指数可以得出,辣椒酱样品中细菌物种丰度显著高于真菌,说明在辣椒自然发酵过程中主要优势菌群是细菌,由于自然发酵的辣椒酱多在室温厌氧环境中进行,该环境更有利于细菌生长。
2.3 新疆不同地区辣椒酱样品间细菌群落结构差异性分析
从微生物分类门的水平上来看,A、B、C、D、E地区的样品中分别含有15、16、16、17、16个门水平的细菌,主要包括厚壁菌门(Firmicutes)、蓝藻菌门(Cyanobacteria)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、梭杆菌门(Fusobacteria)、疣微菌门(Verrucomicrobia)、螺旋体门(Spirochaetes)和异常球菌-栖热菌门(Deinococcus-Thermus)等。
由图2可知,在门水平下,不同地区辣椒酱样品中细菌群落结构组成基本相同,差异较大的是相对丰度。厚壁菌门是5个地区辣椒酱样品中的优势细菌,E地区样品的平均丰度最大,为57.93%;D地区样品丰度最小,为37.86%;A、B和C地区的丰度分别为48.73%、43.21%和47.64%。蓝藻菌门是5个地区样品中相对丰度较大的细菌,平均丰度达到41.46%;在B、C、D和E地区中的平均相对丰度分别为26.39%、25.84%、27.44%和22.39%。厚壁菌门和蓝藻菌门丰度之和均大于60%,这可能是由于厚壁菌门中的乳杆菌和乳球菌大多数为厌氧细菌,能够产酸且在强酸环境下生长。蓝藻菌门可能来自原料辣椒中的叶绿体。从各样品在门水平的分类情况来看,5个地区样品中都出现了大量的蓝藻菌门,但在接下来的分类水平中没有具体分类。据报道线粒体、叶绿体分别起源于原始的好氧细菌和蓝藻类原核细胞,它们长期与宿主进行共生而逐渐演化为细胞器[15]。因为辣椒的叶绿体和线粒体DNA与原核生物的DNA具有同源性,故推测蓝藻菌门可能是来自辣椒原料中的叶绿体和线粒体。

图2 不同地区辣椒酱样品中门水平细菌群落结构分布图
Fig.2 Distribution of bacterial communities’ structure in
chili sauce samples from different areas at phylum level
根据物种分类结果,由于样品中所检测出的微生物种类繁多,其中许多在物种中含量相对较少,因此筛选出优势物种,分别对丰度前20的细菌和真菌进行物种分类统计。由图3可知,在属水平下,不同地区辣椒酱样品中细菌群落结构组成基本相同,差异较大的是相对丰度。A、B、C和E地区样品中的优势细菌是链形植物属(Streptophyta)和乳杆菌属(Lactobacillus),其中A地区两者丰度之和最大,为62.97%,B地区两者丰度之和最小,为46.09%;D地区样品中的优势细菌是链形植物和肠球菌属(Enterococcus),丰度之和为34.25%。在属水平下,链形植物、乳杆菌属和明膜串珠菌属是新疆地区辣椒酱中的优势菌株。存在大量链形植物属是由于采集的样品多为秋季农家自制的辣椒酱,在大气颗粒物中大量存在[16]。乳杆菌属和明膜串珠菌属均隶属乳酸菌,辣椒发酵过程中乳酸菌利用糖类发酵产生乳酸,同时产生醇类、醛类、酮类等多种风味物质,各个地区间丰度的差异也致使不同地区辣椒酱风味物质各有千秋。肠球菌属通常为非条件致病菌,部分为人体肠道内正常微生物,能利用糖酵解和戊糖磷酸途径对糖进行降解,产生有机酸[17]。该属细菌在不同地区样品中丰度差异较大,其中在E地区中丰度最小,仅占2.43%,与样品有机酸含量相对较低,酸味口感不突出的结果一致。
在A、B、C、D和E地区自然发酵辣椒酱样品中分别存在 0.011%、0.017%、0023%、4.471%、0.021%的芽孢杆菌,分析原因可能与辣椒酱中食盐含量不同有关,D地区辣椒酱样品的食盐含量较低,芽孢杆菌含量相对较高。值得关注的是,芽孢杆菌是自然发酵豆豉中的优势菌,该菌株的存在可能会赋予辣椒酱特殊的风味[18]。
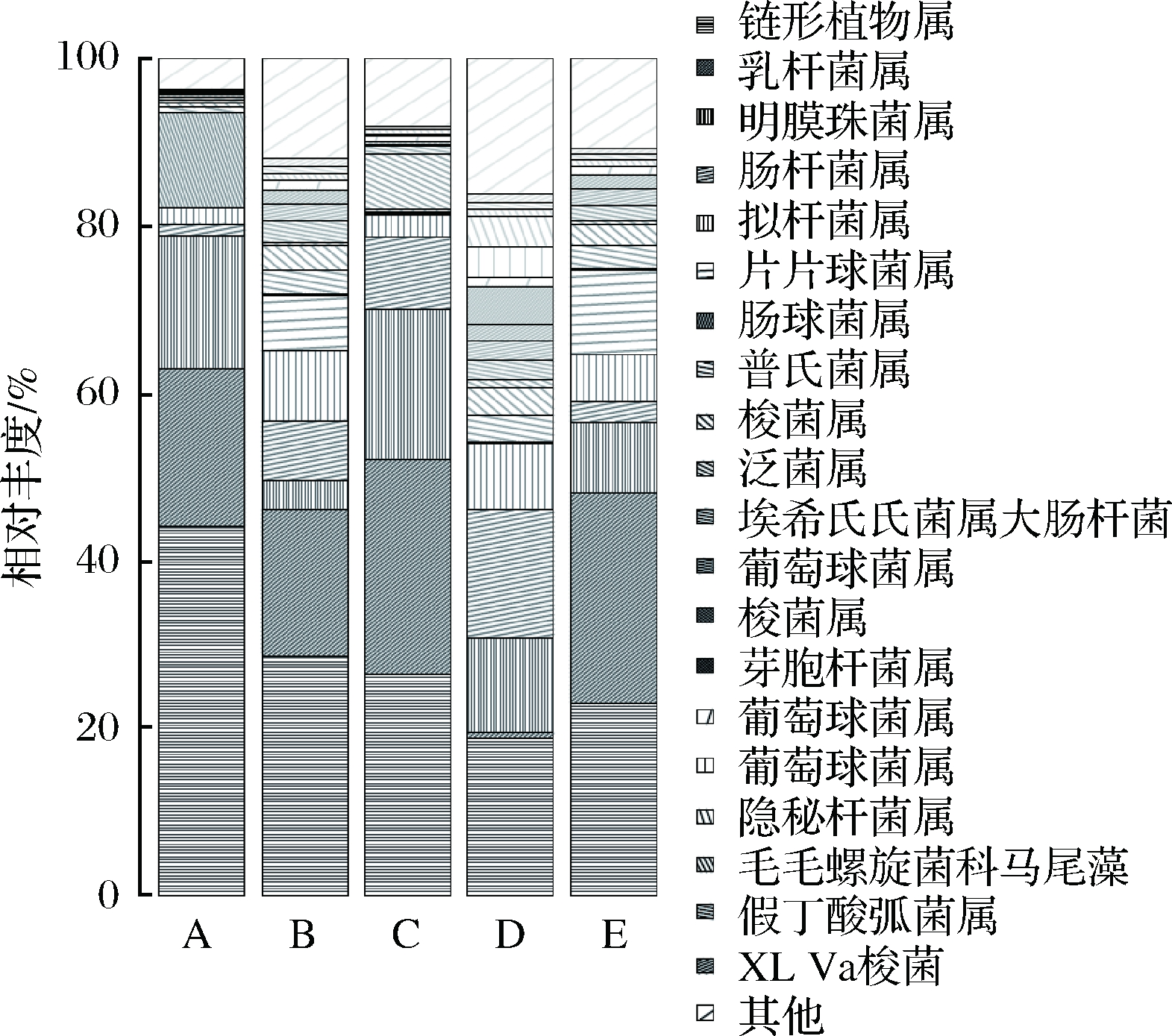
图3 不同地区辣椒酱样品中属水平细菌群落结构分布图
Fig.3 Distribution of bacterial communities’ structure in chili sauce samples from different areas at genus level
2.4 新疆不同地区辣椒酱样品间真菌群落结构差异性分析
由图4可知,在门水平下,从A、B、C、D和E地区中一共鉴定出子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、接合菌门(Zygomycota)和鞭毛菌亚门壶菌纲(Mastigomycotina Chytridiomycota)4个真菌物种,优势真菌都是子囊菌门(Ascomycota),其丰度分别为31.45%、29.15%、33.80%、36.89%和18.81%。说明子囊菌门在辣椒酱发酵中起重要作用。担子菌门相对丰度在5个地区之间的差异较明显,A、C、E地区辣椒酱样品中担子菌门的含量显著低于B、D 2个地区。值得注意的是,在E地区还检测到鞭毛菌亚门壶菌纲,有研究发现隶属鞭毛菌亚门壶菌纲的马铃薯癌肿病菌是一种专性寄生的低等真菌,目前尚不能人工培养[19];发现5个地区真菌的优势菌群差异较大,未知酵母的相对丰度都超过60%,E地区样品中未知酵母高达80.55%,说明新疆不同地区辣椒酱中真菌多样性有待进一步挖掘和发现。
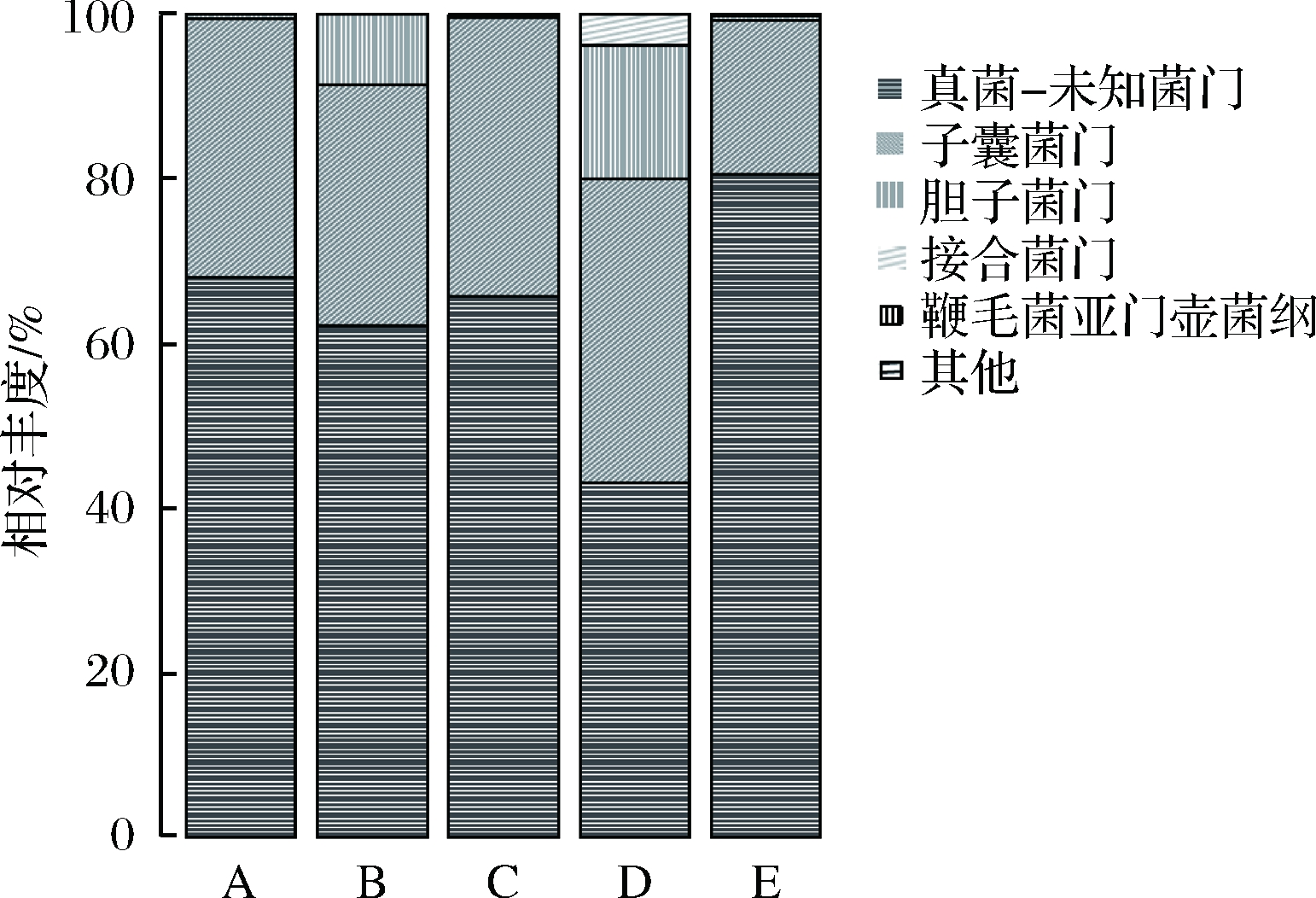
图4 不同地区辣椒酱样品门水平真菌群落结构分布图
Fig.4 Distribution of fungal communities’ structure in chili sauce samples from different areas at phylum level
由图5可看出,在属水平下,A地区样品中优势真菌菌属是毕赤酵母属(Pichia),相对丰度为9.31%,丝孢毕赤酵母属是发酵饮料中常见的产香酵母,对发酵产品有增香作用[20];B地区样品中优势菌属是孢汉逊酵母属(Hanseniaspora),相对丰度为17.44%,Hanseniaspora 主要存在于土壤、水果、腐败的食品和饮料中,可发酵葡萄糖,生长需要肌糖[21];C地区样品中优势菌属是威克汉姆酵母(Wickerhamomyces),相对丰度为19.99%,据报道异常威克汉姆酵母菌株具有低产尿素、产风味,耐酒精、耐酸的特征,是一种产香酵母,具有较高的产 3-甲基-1-丁醇能力,在发酵过程中能产生乙酸乙酯、2-苯基乙酸乙酯、乙酸异戊酯等对发酵辣椒风味具有促进作用的酯类物质,对发酵制品风味有很好的贡献作用[22];D和E地区样品中优势菌属是曲霉属(Aspergillus),相对丰度分别为27.17%和2.28%,LV等[23]从10份红曲米酒酒曲中分离鉴定米曲霉和黄曲霉很强的α-淀粉酶或糖化酶活性,这说明曲霉属在米酒发酵过程中能够有效地分解淀粉,增加小分子糖的含量,因而使米酒含糖量升高,导致口感偏甜。这与D、E 2地区辣椒酱样品口味偏甜,酸味不突出结果一致。
本研究采集的样品为发酵中后期的辣椒酱,由于酸度的增加,真菌的种类和相对丰度都大大降低。A、C地区中包含德巴利(氏)酵母属(Debaryomyces),其丰度分别为6.60%、1.16%。B、C、D和E地区中都含有假丝酵母(Candida),丰度分别为1.44%、2.98%、1.85%和0.58%。B、D地区中含有未知伞菌属(Agaricomycetes_unidentified_1),丰度分别为8.07%、16.29%。D地区中含有丰度较高的毛霉菌属(Mucor)和青霉属(Penicillium),丰度分别为1.25%和0.53%。张仁凤等[24]指出霉菌可产生物胺,不同菌株产生物胺的能力各不相同,但该地区样品中生物胺含量还需要通过进一步验证说明。A地区样品中特有的菌种是孢圆酵母(Torulaspora),丰度为3.98%。李华敏等[25]指出孢圆酵母能显著提高樱花酒中β-苯乙醇、丁酸乙酯、异戊酸乙酯等挥发性组分的含量,能够增加发酵香气。C地区特有的菌种是螺旋聚孢霉属(Clonostachys),丰度为5.44%。据报道,粉红螺旋聚孢霉(Clonostachys rosea)可以通过分泌碱性泛解酸内酯水解酶,将毒素玉米赤霉烯酮转化成无雌激素性质的产物[26]。E地区特有的菌是链格孢属(Alternaria),其丰度为0.63%。链格孢属是重要的植物病原真菌,种类繁多,寄主广泛,近年来其在葡萄上引起的病害引起了国内外学者广泛关注[27]。5个地区包含了绝大多数的未知真菌物种,相对丰度都超过60%, E地区的样品中未知真菌物种高达80.62%,优势菌群比较少。发现不同地区辣椒酱发酵过程中特有的真菌,其功能需要进一步探索,以便于更好地为辣椒酱产业的规模化和现代化服务。

图5 不同地区辣椒酱样品中属水平真菌群落结构分布图
Fig.5 Distribution of fungal communities’ structure in chili sauce samples from different areas at genus level
2.5 不同地区样品间物种组成分析
如图6-a所示, PCoA分析可以直观地将样品间相似或差异程度体现在二维坐标图上,距离越近则表示越相似。统计分析的可信度用P表示,P小于0.05表示统计具有显著性。使用秩和检验的方法对不同分组之间进行显著性差异分析,以找出对组间划分产生显著性差异影响的物种,对于不同组样品细菌群落,有19个属、7个科和3个目,存在显著性差异(P<0.05)。R为0.116,介于(-1,1)之间且大于0,说明组间差异显著。PCoA1和PCoA2分别解释了不同地区样品中细菌群落 39.54%和 33.96%的信息,2个主成分之和大于70%,表明2个成分较好地代表了样品中的细菌群落信息。不同地区可明显地划分为2组,C、E和A、B、D,E地区样品中2个点虽然偏离较远,但根据主成分一(39.54%) 可与其余样品聚为一类。发现A、B和D组样品较为离散,各自独立,说明这3个地区样品间细菌群落相似性不高;C、E组样品间的距离较近,说明这2个地区样品间的细菌群落相似性高,该结果与这2个地区距离较近有密切关系。
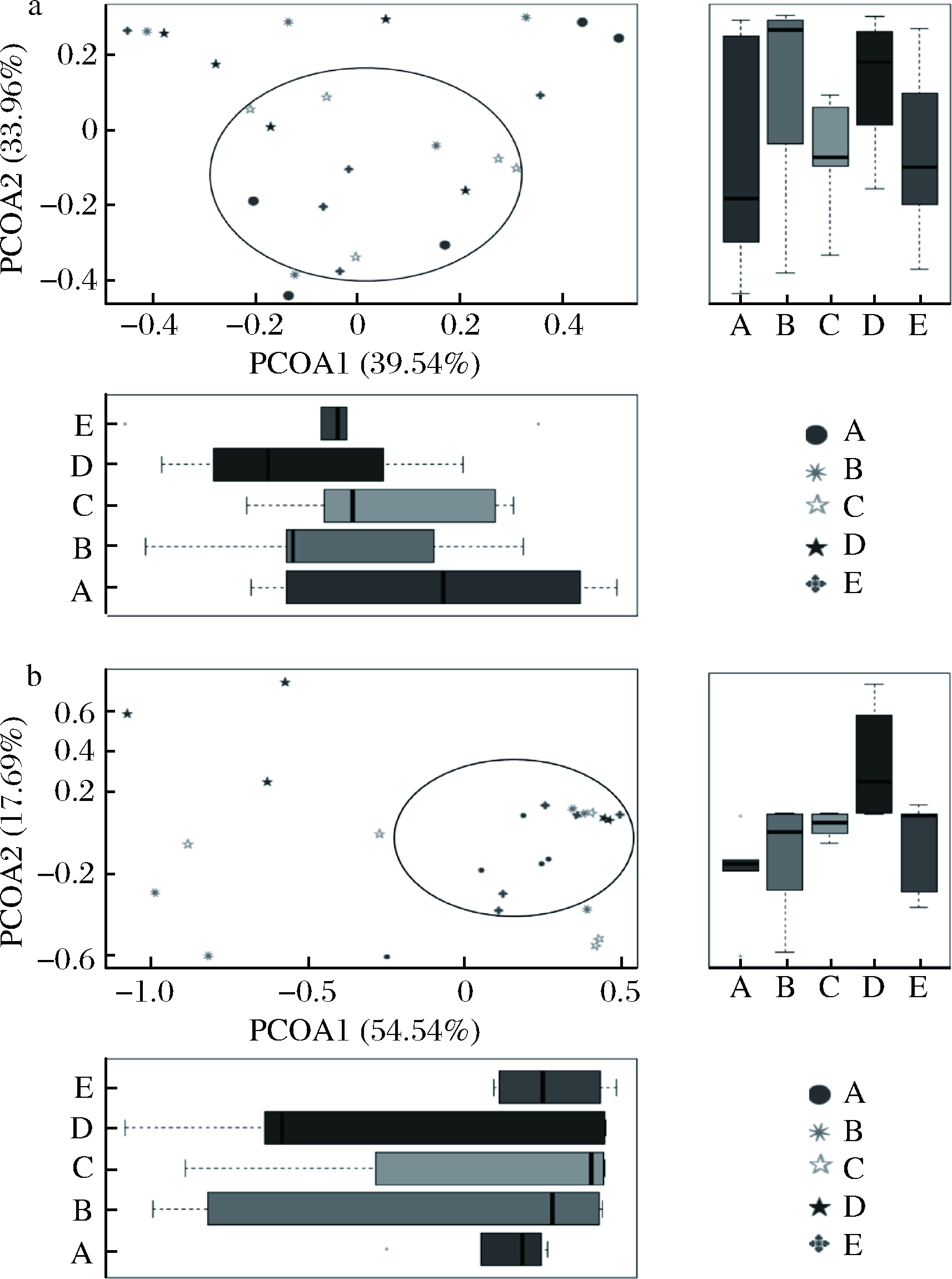
a-细菌;b-真菌
图6 不同地区辣椒酱中微生物群落主成分分析图
Fig.6 The principal coordinate analysis of microbial community structure in chili sauce samples from different reas
注:横坐标表示第1主坐标,纵坐标表示第2主坐标,括号中的百分比则表示对样品差异的贡献率;图中各点分别表示各个样品,不同形状代表样品属于不同的分组。盒形图可以显示最小值,第1个四分位数,中位数,第3个中位数和最大值,及由下到上的5条线,异常值以“o”标出。
由图6-b可知,对不同组微生物群落之间的物种进行假设检验,属、科和目各4个,在不同组样品真菌间有显著差异(P<0.05)。R为0.193,介于(-1,1)之间且大于0,说明组间差异显著。PCoA1和PCoA2分别解释了不同样品真菌群落 54.54%和 17.69%的信息,2个主成分之和大于 70%,表明2个成分较好地代表了样品中真菌群落信息。不同地区可明显地划分为2组,B、C、D和A、E,A地区样品中的一点虽然偏离较远,但根据主成分一(54.54%) 可与其余样品聚为一类。分析2组发现B、C和D组样品较为离散,各自独立,说明这3个地区样品间真菌群落相似性不高;A和E组的样品距离较近,说明这2个地区样品间真菌群落相似性高。
3 讨论
本研究检测辣椒酱样品微生物多样性,发现A、B、C、D、E地区样品中都含有肠球菌属、葡萄球菌和梭菌,其累计平均含量分别为2.476%、10.778%、9.445%、25.366%、7.242%。隶属于上述3个属的部分细菌是条件致病菌,例如隶属于肠杆菌属的阪崎肠杆菌[28]是引起新生儿脑膜炎、小肠结肠炎和菌血症的主要微生物;葡萄球菌中的金黄色葡萄球菌[29]可引起局部化脓感染,也可引起肺炎、心包炎和伪膜性肠炎;梭菌属中的破伤风菌[30]属是人畜破伤风的病原体,产气荚膜梭菌是人类气性坏疽的主要病原体。综上所述,农户家自制的辣椒酱样品中可能存在一定质量安全隐患,只是不同地区样品间菌株含量存在差异。阿克苏地区含量明显高于其他地区,其中肠球菌属的含量明显高于其他地区,分析原因可能与其样品发酵周期较长有关,也可能和阿克苏地区的原料、水、土壤和空气有关。哈密、昌吉和乌鲁木齐地区样品中特有的真菌菌种分别是孢圆酵母、螺旋聚孢霉属和链格孢属,因此后续试验研究孢圆酵母与发酵香气的关系具有重要意义。值得关注的是,链格孢属是否对乌鲁木齐地区辣椒生长有影响,因而后续研究原料辣椒中的微生物多样性以及新疆不同地区水、土壤和空气的微生物多样性,无论是对了解辣椒酱的微生物多样性,还是减少食品安全隐患均具有积极的意义。
沈馨等[7]以湖北当阳地区辣椒酱为研究对象,发现酸味是辣椒酱样品间差异最大的滋味指标,微生物构成对辣椒酱滋味品质的形成具有较大的影响。韩俊燕等[10]研究安徽省黄山、湖南省永川、山西省平遥、吉林省延边地区的发酵辣椒制品,发现菌群丰度在不同样品中差异较大。门水平上,主要为厚壁菌门、变形菌门和蓝藻菌门;属水平上,主要微生物为乳杆菌属、魏斯氏菌属、乳球菌属、片球菌属和明串珠菌属。尚雪娇等[31]研究恩施市3种泡辣椒样品,发现优势细菌门为硬壁菌门(Fir-micutes)和变形菌门(Proteobacteria);优势细菌属为乳酸杆菌属(Lactobacil-lus)。对比全国其他地区辣椒酱微生物多样性研究,本研究结果中微生物多样性特征与其他学者研究结果大体相同,但微生物种类及其数量上具有差异。发现新疆不同地区辣椒酱样品中细菌群落结构组成基本相同,差异较大的是相对丰度,发酵辣椒酱中细菌的构成比单纯的发酵辣椒更为复杂,菌群的种类和数量也不尽相同,与武俊瑞[32]的研究结果一致。5个地区的辣椒酱样品中微生物在数量、种类等方面都具有一定的差异,分析其原因可能是:采样地区的气候、地理位置、光照时间、制作方法(如食盐添加量、发酵时间、发酵温度、使用容器等)、饮食习惯等客观因素的不同引起发酵辣椒酱成分的不同,因此不同地区辣椒酱中的微生物多样性具有一定的差异。
4 结论
本研究通过高通量测序技术对不同地区自然发酵辣椒酱样品进行分析,主要优势细菌菌门为厚壁菌门、蓝藻菌门、变形菌门、拟杆菌门。厚壁菌门是5个地区辣椒酱样品中的优势细菌,蓝藻菌门是5个地区样品中相对丰度较大的细菌。链形植物属和乳杆菌属是哈密、伊犁、昌吉和乌鲁木齐地区样品中的优势细菌;链形植物和肠球菌属是阿克苏地区样品中的优势细菌。从5个地区中一共鉴定出 4个真菌物种,优势真菌都是子囊菌门。毕赤酵母属、孢汉逊酵母属、威克汉姆酵母、曲霉属和Davidiella分别是A、B、C、D和E地区样品中优势真菌。毕赤酵母属、孢汉逊酵母属和威克汉姆酵母是哈密、伊犁和昌吉地区样品中优势真菌,曲霉属是阿克苏和乌鲁木齐地区的优势真菌。孢圆酵母是哈密地区样品中特有的菌种;粘帚霉属是昌吉地区特有的菌种;链格孢属是乌鲁木齐地区特有的菌种。
本研究选择新疆不同地区自然发酵辣椒酱为研究对象,获得的微生物信息涵盖所有微生物包括死亡的微生物,也包括取样时样品体系中的微生物和取样过程中增加的微生物,但研究结果并不能区分时间段,具有一定局限性。细菌和真菌无法同时进行定量,也不能将微生物与其他相关指标建立联系,未来研究中还需进行宏转录组测序。本试验中存在大量未知菌属,可能是因为测序数据中包含着较多未知数量菌种,很难通过一段DNA序列来确定来源。虽然有一定的局限性,但是也能在一定程度上反映出5个地区自然发酵辣椒酱中微生物群落。本研究首次大量采集新疆不同地区自然发酵辣椒酱,利用高通量测序的方法研究了新疆不同地区自然发酵辣椒酱中细菌的群落结构和多样性,为后续建立新疆辣椒酱中微生物体系指标提供了理论支持,同时为适合辣椒酱发酵微生物的筛选提供基础。
[1] 郭琳, 孟梦,郭淼,等. 接种乳酸菌改善辣椒酱风味的研究[J]. 中国调味品, 2016, 41(5): 1-7.
[2] 白露露, 胡文忠,姜爱丽,等. 辣椒加工工艺及其设备的应用现状[J]. 食品工业科技, 2014, 35(15): 369-372;376.
[3] SIRIPATTANAKULRATPUKDI S, VANGNAI A S, SANGTHEAN P, et al. Profenofos insecticide degradation by novel microbial consortium and isolates enriched from contaminated chili farm soil[J]. Environmental Science & Pollution Research, 2015, 22(1): 320-328.
[4] 叶陵, 李勇,王蓉蓉,等. 剁辣椒中优良乳酸菌的分离鉴定及其生物学特性分析[J]. 食品科学, 2018, 39(10): 112-117.
[5] COCOLIN L, ALESSANDRIA V, DOLCI P, et al. Culture independent methods to assess the diversity and dynamics of microbiota during food fermentation[J]. International Journal of Food Microbiology, 2013, 167(1): 29-43.
[6] TURCATTI G, ROMIEU A, FEDURCO M, et al. A new class of cleavable fluorescent nucleotides synthesis and optimization as reversible terminators for DNA sequencing by synthesis[J]. Nucleic Acid Research Joumal, 2008, 36(4): 25.
[7] 沈馨, 王艳,代凯文,等. 基于Miseq高通量测序技术的辣椒酱核心细菌类群研究[J]. 食品研究与开发, 2018, 39(10): 151-157.
[8] 赵玲艳. 自然发酵辣椒微生物多样性及其宏转录组研究[D]. 长沙:湖南农业大学, 2017.
[9] 钟燕青. 自然发酵辣椒中乳酸菌分离筛选及香气成分分析[D]. 长沙:湖南农业大学, 2012.
[10] 韩俊燕, 赵国忠,赵建新,等. 发酵辣椒细菌多样性的16S rDNA测序分析[J]. 中国食品学报, 2018, 18(5): 246-251.
[11] 武亚婷, 武运,杜木英,等. 新疆辣椒酱产业研究现状和发展趋势[J]. 中国酿造, 2019, 38(3): 1-4.
[12] 沙漠, 逄焕明,古丽娜孜,等. 自然发酵辣椒酱中乳酸菌的分离与鉴定[J]. 食品与机械, 2012, 28(1): 35-37.
[13] COLE J R, CHAI B, FARRIS R J, et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis[J]. Nucleic Acids Research, 2005, 33(Database issue): 294-296.
[14] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive Bayesian Classifier for Rapid Assignment of rRNA Sequences into the New Bacterial Taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5 261-5 267.
[15] 李峰, 管敏鑫. 线粒体起源和蛋白含量进化[J]. 科技通报, 2015, 31(1): 61-66.
[16] 苟欢歌. 北疆重要城市大气颗粒物中微生物群落结构研究[D]. 石河子:石河子大学, 2016.
[17] 苏悟, 郑小芬,徐睿烜,等. 1种细菌型豆豉自然发酵过程中生物胺的变化[J]. 食品与发酵工业, 2014, 40(7): 40-45.
[18] PRICE M N, DEHAL P S, ARKIN A P. Fasttree: computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular biology and evolution, 2009, 26(7): 1 641-1 650.
[19] 王云月, 马俊红,何霞红,等. 马铃薯癌肿病菌分离及基因组DNA提取[J]. 云南农业大学学报, 2002(4): 432-433.
[20] CHEN B, WU Q, XU Y. Filamentous fungal diversity and community structure associated with the solid state fermentation of Chinese Maotai-flavor liquor[J]. International Journal of Food Microbiology, 2014, 179(6): 80-84.
[21] 王泽举. 新疆、甘肃地区葡萄酒相关酵母菌的鉴定及多样性研究[D]. 杨凌:西北农林科技大学, 2008.
[22] 李凯敏, 付桂明,吴酬飞,等. 特香型白酒酿造过程中真核微生物菌群演替[J]. 食品科学, 2017, 38(22): 131-136.
[23] LV X C, WENG X, ZHANG W, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2): 426-434.
[24] 张仁凤, 陈光静,杨万明,等. 豆豉发酵常用毛霉和米曲霉菌株产生物胺能力的评价[J]. 食品与发酵工业, 2017, 43(9): 15-21.
[25] 李华敏, 孙舒扬,黄萍萍,等. 戴尔有孢圆酵母WA19与酿酒酵母F33混合发酵在樱桃酒酿造中的应用[J]. 食品科学, 2018, 39(24): 123-130.
[26] KAKEYA H, TAKAHASHI-ANDO N, KIMURA M, et al. Biotransformation of the mycotoxin, zearalenone, to a non-estrogenic compound by a fungal strain of Clonostachys sp[J]. Bioscience Biotechnology & Biochemistry, 2002, 66(12):2 723-2 726.
[27] 王彩霞, 李兴红,魏艳敏,等. 引起葡萄叶斑病的链格孢种类的初步鉴定[J]. 植物保护学报, 2019, 46(1): 175-183.
[28] 裴晓燕, 刘秀梅. 阪崎肠杆菌的生物学性状与健康危害[J]. 中国食品卫生杂志, 2004, 16(6): 550-555.
[29] 李晓梅, 耿艺介. 金黄色葡萄球菌及其肠毒素B的生物学特性[J]. 中国热带医学, 2007, 7(4): 617-619.
[30] 周笑娉, 陈忠良,刘长秀. 海南省新生儿破伤风高发区外环境破伤风杆菌检测[J]. 中华疾病控制杂志, 2001, 5(4): 367-368.
[31] 尚雪娇, 折米娜,邓长阳,等. 恩施市3种泡辣椒样品中细菌多样性研究[J]. 中国酿造, 2019, 38(2): 53-57.
[32] 武俊瑞. 东北传统发酵特色食品中主要微生物多样性研究[D]. 沈阳:沈阳农业大学, 2013.