啤酒是世界上最受欢迎的酒精饮料之一。尽管工业生产中对微生物污染高度重视,但由于啤酒微生物的高度适应性,微生物污染的情况仍时有发生,这给啤酒生产企业带来了极大的困扰[1]。
啤酒中的腐败菌种类较多,以乳酸菌为主。近年,啤酒中发现的1个新啤酒腐败菌菌种——耐乙酸乳杆菌(Lactobacillus acetotolerans),因其特殊的生理特性引起了人们的关注[2]。啤酒中的耐乙酸乳杆菌保持了乳杆菌的菌种特性,以乳酸、醋酸和双乙酰为糖酵解终产物,大量的双乙酰使啤酒产生了令消费者不愉快的黄油味和油腻的口感,严重影响了啤酒的品质;同时,该菌具有在一般营养条件下不易培养的生理特性,国标等常规方法难以检测到此菌,出现假阴性结果[3]。这使得啤酒在贮存或销售环节易出现质量问题,严重影响产品形象和企业声誉。
基于微生物检测技术的快速发展,除常规的检测方法外,蛋白质和核酸等分子检测手段也得到广泛应用。其中聚合酶链式反应(polmerase chain reaction, PCR)技术以其较高的灵敏性和准确性在啤酒腐败菌检测中得到普遍认可[4]。PCR检测多以啤酒腐败菌某些特定的靶标进行检测,包括16SrDNA通用引物和四联球菌特异性引物等[5-7];或利用某些特殊抗性基因,如酒花抗性基因horA[8]、horB[9]或horC[10-11]进行跨种属检测,以实现对啤酒腐败菌的快速检测。但对于新出现的啤酒污染菌仍然需要建立对应的检测方法,以实现对其进行有效的监控[12]。因此本研究以建立啤酒中耐乙酸乳杆菌种特异性检测体系为切入点,建立特异性的检测体系,以实现对啤酒生产过程质量检测和对出厂啤酒提供必要的技术保障。
1 材料与方法
1.1 仪器与材料
2-16N高速微量离心机,湖南恒诺仪器设备有限公司;NanoDrop One,赛默飞世尔仪器有限公司;ABI7500 real time PCR system,美国ABI公司;Multiskan FC酶标仪,赛默飞世尔仪器有限公司;SW-CJ-2FD超净工作台,苏州净化设备有限公司;BX51显微镜,奥林巴斯(中国)有限公司;LRH-250培养箱,上海一恒科学仪器有限公司;BC-C57PCR仪,北京天林恒泰科技有限公司;BG-sub MIDI多用途水平电泳仪、Tanon 1600凝胶成像系统,上海天能科技有限公司。
NBB-P培养基,德乐公司;KK4601 KAPA SYBR FAST试剂盒,KAPA BIOSYSTEMS;细菌基因组DNA提取试剂盒,北京天根生化科技有限公司。
1.2 本研究中使用的菌株
本研究中使用的菌株均分离自出现混浊、异味或产生沉淀的啤酒样品,共分离鉴定了12株啤酒腐败菌,菌株信息见表1。所有菌株均采用平板分离后再进行厌氧培养[13-14]。
表1 本研究中分离和使用的啤酒腐败菌菌株
Table 1 Beer spoilage strains isolated and used in the study

序号菌株编号鉴定结果1L2Lactobacillus lindneri2L150Lactobacillus brevis3L238Lactobacillus casei4L758Lactobacillus plantarum5L1104Lactobacillus backii6CN004Lactobacillus paracasei7CN009Lactobacillus harbinensis8CN124Lactobacillus parabuchneri9CN134Lactobacillus brevis10CN247Lactobacillus acetotolerans11SaTPediococcus damnosus12250Lactobacillus hilgardii
1.3 引物设计与验证
1.3.1 耐乙酸乳杆菌特异性基因的筛选与引物设计
目前开放的GenBank数据库中仅有一株较为完整的耐乙酸乳杆菌的基因组数据,其余均为基因组草图[15-16]。研究中根据以上数据和预测的蛋白质信息,利用Mauve软件比对GenBank中23株耐乙酸乳杆菌基因组,从中筛选出10个耐乙酸乳杆菌较为特异的基因编码区片段,然后将基因编码序列与数据库中的微生物序列进行BLAST比对,在比对结果中排除具有较多相似菌株且相似度较高的基因序列,保留比对结果中只有耐乙酸乳杆菌的特异性基因序列,以此筛选出耐乙酸乳杆菌特有的基因作为检测的备选基因用于后续研究。
1.3.2 引物设计
下载筛选到的目的基因序列,使用Primer premier 5软件进行引物设计。引物长度18~24 bp,GC含量40%~60%,目标产物在130 bp~300 bp,Tm值在55~60℃,设计完成后登录在线引物设计工具[17-18],在模板输入框中分别输入引物上下游序列,在线检测合成产物。
1.4 扩增体系与反应条件
将设计好的引物送上海生工生物有限公司合成。扩增反应总体系为20 μL,包括上下游引物各0.4 μL(200 nmol/L),基因组DNA为1.0 μL(1 ng/μL),2×mix10 μL,50×ROX 0.4 μL,无菌水定容到20 μL;反应条件见表2。
1.5 耐乙酸乳杆菌定性检测
1.5.1 引物特异性验证
表2 耐乙酸乳杆菌种特异性PCR反应条件
Table 2 Speceis-specific PCR reaction conditions

反应阶段反应温度与时间设置恒温阶段step1 95 ℃10 min循环阶段step1 95 ℃15 sstep2 60 ℃60 s30cycle熔解曲线阶段step1 95 ℃step3 95 ℃15 s30 sstep2 60 ℃step4 60 ℃15 s5 s冷却阶段step1 60 ℃7 min
以提取的啤酒腐败菌耐乙酸乳杆菌基因组DNA为模板,使用设计的10对耐乙酸乳杆菌引物分别对每种菌的基因组进行扩增,筛选特异性最佳的耐乙酸乳杆菌引物用于后续混合菌中耐乙酸乳杆菌的定性和定量检测[19-20]。
1.5.2 混合菌中耐乙酸乳杆菌的定性检测
以本研究中使用的12株啤酒腐败菌的基因组DNA混合物作为模板,检测筛选的特异性引物在混合菌中耐乙酸乳杆菌的检出情况[21]。
1.5.3 同种属耐乙酸乳杆菌的检测
以同种属的耐乙酸乳杆菌2011-3(2011年3月在8°纯生啤酒中回收样品中检出)与2011-8(2011年8月在10°纯生微检留样中检出)的DNA为模板,检测引物特异性。
1.6 耐乙酸乳杆菌特异性探针的定量检测
使用细菌基因组提取试剂盒,提取耐乙酸乳杆菌的菌悬液(1.0×108 CFU/mL)基因组。经10倍系列梯度稀释,得到耐乙酸乳杆菌不同DNA浓度的标准品。再以该标准品为模板,使用筛选到的特异性耐乙酸乳杆菌引物进行RT-qPCR扩增,建立耐乙酸乳杆菌的标准曲线,得到标准曲线的相关性方程,用于后续的实际样品检测[22]。
1.7 耐乙酸乳杆菌特异性探针的最低检出限
将培养好的耐乙酸乳杆菌按照10倍系列梯度稀释后,分别接种到NBB-B液体培养基中,26 ℃厌氧培养2 d,提取基因组。利用优化的种特异性引物对目标菌上机检测,比对确认样品中耐乙酸乳杆菌的菌量与实际值之间的差异,完成样品中耐乙酸乳杆菌的种特异性定性和定量检测。
1.8 耐乙酸乳杆菌特异性引物检测实际样品
跟踪生产上的20批样品,利用膜过滤(0.45 μm)富集啤酒样品中的微生物。然后,将抽滤膜置于NBB-B培养基中,26 ℃厌氧培养2 d,取10 mL菌液提取基因组DNA,然后使用本研究方法进行检测。同时,对阳性样品进行菌株分离,然后送往华大基因公司进行测序,对比本研究建立的方法与实际样品的一致性。
2 结果与分析
2.1 特异性引物筛选和定性检测
2.1.1 耐乙酸特异性引物的筛选
按照1.3的方法,初步筛选10个基因作为备选靶标用于啤酒中耐乙酸乳杆菌的种特异性检测。以啤酒腐败菌耐乙酸乳杆菌CN247基因组DNA为模板,经RT-qPCR扩增后,剔除产生非特异性片段的引物,最终确定了3对特异性强的引物,引物序列如表3所示。
表3 本研究筛选的啤酒腐败菌耐乙酸乳杆菌的特异性引物序列
Table 3 Specific primer sequence for Lactobacillus acetotolerans

引物名称引物序列 5′—3′对应耐乙酸乳杆菌基因片段大小/bpYW5F:TAAAAGAAGTAACGCAAACCACGR:ACGGATACAAACGCTGCTCAACAP014808.1:603921-604277砷酸还原酶163YW6F:CGGAAATGTATTAGACGGTGTTR:ATGATGAAGCGTAAGAAGGATGAP014808.1:1509468-1510004保守预测蛋白150YW7F:TCTTATTGCTTCTGGTTTGGR:ACACGATGCGACTGATTTTAAP014808.1:529550-529885保守预测蛋白143
2.1.2 耐乙酸乳杆菌特异性引物定性检测单一DNA模板的结果
以本研究中12株菌的基因组为模板,分别用YW5、YW6和YW7三对耐乙酸乳杆菌特异性引物进行RT-qPCR扩增,以确认3对引物对耐乙酸乳杆菌检测的特异性。YW5、YW6和YW7特异性引物的RT-qPCR扩增曲线和熔解曲线如图1所示。

图1 YW5、YW6和YW7引物在RT-qPCR上检测单一DNA的扩增曲线和熔解曲线
Fig.1 Amplification curve and melting curve of detection of single DNA by YW5,YW6 and YW7 primers on RT-qPCR
如图1所示,使用1.4的反应体系和反应条件,YW5、YW6和YW7三对引物只有在扩增耐乙酸乳杆菌CN247时分别出现了对应的扩增曲线,其余菌株均无扩增曲线;同时,对应的熔解曲线出现单一尖锐的峰,没有杂峰,说明在本研究的反应体系和反应条件下,3对引物对于耐乙酸乳杆菌菌种CN247检测具有特异性,参照Ct值和熔解曲线可将CN247与其他11株腐败菌区分开来,因此这3对引物可以作为CN247的特异性引物用于12种啤酒腐败菌中耐乙酸乳杆菌菌种的定性检测。
2.1.3 耐乙酸乳杆菌特异性引物定性检测混合DNA模板
为了进一步验证所筛选引物的特异性,研究中将12个菌种的基因组混合后作为模板,利用筛选的3对引物对目标产物进行扩增,扩增结果如图2所示。

图2 YW5、YW6和YW7引物在RT-qPCR上检测混合DNA的扩增曲线和熔解曲线
Fig.2 Amplification curve and melting cuve of detection of mixed DNA by YW5、YW6 and YW7
primers on RT-qPCR
如图2所示,在分别使用3对特异性引物对混合DNA样品进行扩增时,在多个复杂模板及近缘乳酸菌菌种存在的情况下,检测体系只对耐乙酸乳杆菌的检测具有种特异性,说明引物特异性良好。
2.1.4 耐乙酸乳杆菌特异性引物定性检测同种属其他耐乙酸乳杆菌
为了进一步验证设计的特异性引物对另外2株鉴定的耐乙酸乳杆菌2011-3和2011-8检测的有效性,使用其中1对引物YW5对测试菌株DNA进行PCR扩增,结果如图3所示。
RT-qPCR测试结果表明,检测的2株菌2011-3与2011-8为耐乙酸乳杆菌,与分子鉴定的结果相同。说明设计的引物对耐乙酸乳杆菌菌种具有种特异性。
因此,以分离到的12株啤酒污染菌基因组为模板,无论是单一菌DNA还是混合菌的DNA,通过YW5、YW6和YW7三对引物在RT-qPCR上扩增后,均能对样品中的耐乙酸乳杆菌检测出唯一的扩增曲线和熔解曲线,同时也能对耐乙酸乳杆菌的菌种进行种特异性有效检出,表明了3对引物的特异性符合检测耐乙酸乳杆菌的检测要求。
2.2 定量检测结果
2.2.1 耐乙酸乳杆菌DNA标准曲线的建立
以108 CFU/mL耐乙酸乳杆菌提取的基因组为基础,通过10倍梯度稀释的DNA为模板,以特异性引物YW5对不同梯度的耐乙酸乳杆菌基因组DNA扩增,建立耐乙酸乳杆菌标准曲线,如图4所示。
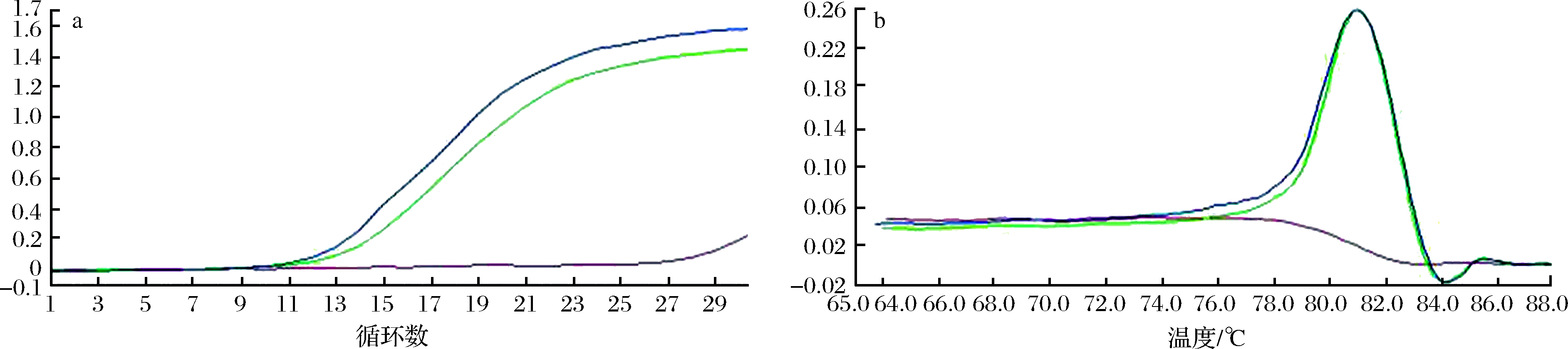
图3 YW5引物在RT-qPCR上检测耐乙酸乳杆菌2011-3和2011-8的扩增曲线和熔解曲线
Fig 3 Amplification curve and melting cuve of detection of strain 2011-3 and 2011-8 by YW5 primer on RT-qPCR

图4 耐乙酸乳杆菌DNA的标准曲线
Fig.4 Standard curve of Lactobacillus acetotolerans
DNA
如图4所示,7个浓度梯度的耐乙酸乳杆菌DNA样品扩增的标准曲线满足误差范围,其对应的回归方程y=-3.344X+33.56,R2>0.99,线性关系良好,满足后续对标的需要。
2.2.2 标曲的重复性和再现性验证
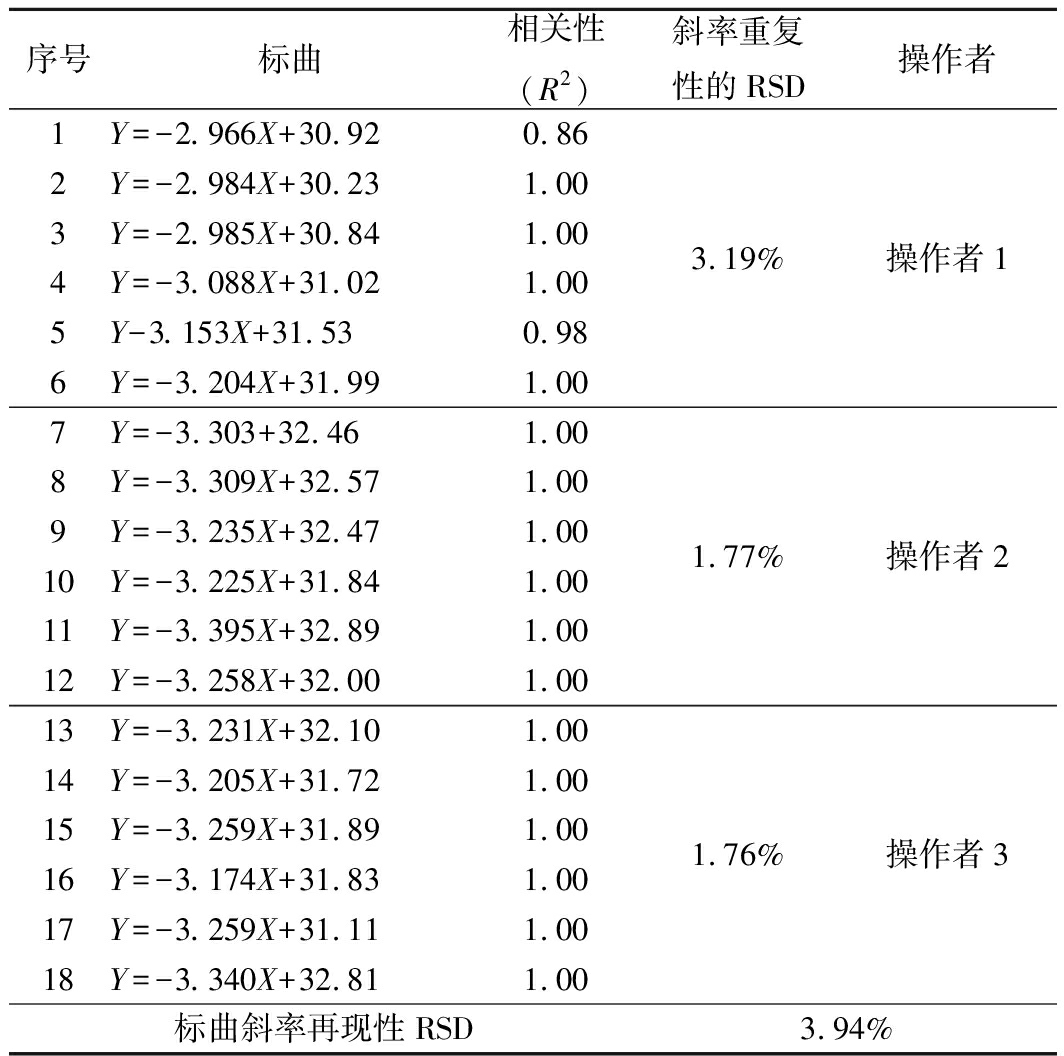
选取3人分别使用新开发的耐乙酸乳杆菌特异性探针YW5,验证定量标曲(从DNA稀释至上机为一次操作)的重复性和再现性,每人做6次标曲,实验结果如表4所示。
如表4所示,每个人测试6次标曲斜率的重复性RSD均在5%以内,3人测试标曲的再现性RSD为3.94%,在5%的合格范围内,说明标曲的重复性和再现性良好,可以用于实际的检测。
2.2.3 耐乙酸乳杆菌的最低检出限
为了进一步确定耐乙酸乳杆菌特异性引物的最低检出限,将浓度1×108 CFU/mL的耐乙酸乳杆菌CN247的原始菌液10倍梯度稀释,并以最后3个梯度分别进行平板计数,26℃厌氧培养2 d。再对各梯度的菌液平板计数,确定样品培养前后的菌体浓度。培养后的菌液分别取10 mL菌液提取DNA后在RT-qPCR进行检测,结果如表5所示。
不同梯度接种的样品经预培养2 d后,取10 mL预培养液提取的基因组DNA中均可以检测到目标菌。其中,经预培养的最低稀释度的4号样品平板检测结果为11 CFU/mL。因在提取DNA做RT-qPCR时使用的是10 mL菌液,故上机检测出来4号样品对应的菌数为102 CFU/mL。因此,新开发的特异性探针对耐乙酸乳杆菌的最低检出限为102 CFU,检测精度为101 CFU/mL。
表4 标曲的重复性和再现性结果
Table 4 Repeatability and reproducibility results offstand curve
序号标曲相关性(R2)斜率重复性的RSD操作者1Y=-2.966X+30.920.86 2Y=-2.984X+30.231.00 3Y=-2.985X+30.841.00 4Y=-3.088X+31.021.00 5Y-3.153X+31.530.98 6Y=-3.204X+31.991.00 3.19%操作者17Y=-3.303+32.461.00 8Y=-3.309X+32.571.00 9Y=-3.235X+32.471.00 10Y=-3.225X+31.841.00 11Y=-3.395X+32.891.00 12Y=-3.258X+32.001.00 1.77%操作者213Y=-3.231X+32.101.00 14Y=-3.205X+31.721.00 15Y=-3.259X+31.891.00 16Y=-3.174X+31.831.00 17Y=-3.259X+31.111.00 18Y=-3.340X+32.811.00 1.76%操作者3标曲斜率再现性RSD3.94%
表5 不同梯度CN247培养2 d后的菌液使用特异性探针RT-qPCR测试结果
Table 5 The results of RT-qPCR using the special probe to detecting the suspension of CN247 after cultivation

编号培养前平板计数结果(CFU·mL-1)培养后平板计数结果(CFU·mL-1)特异性探针RT-qPCR检测结果扩增曲线熔解曲线判定检出率1不可计数7.1×103++(78 ℃)+100.0%21155.4×102++(78 ℃)+100.0%31497++(78 ℃)+100.0%4111++(78 ℃)+100.0%
注:“+”表示结果正常,判定为阳性。
2.3 实际样品的检测
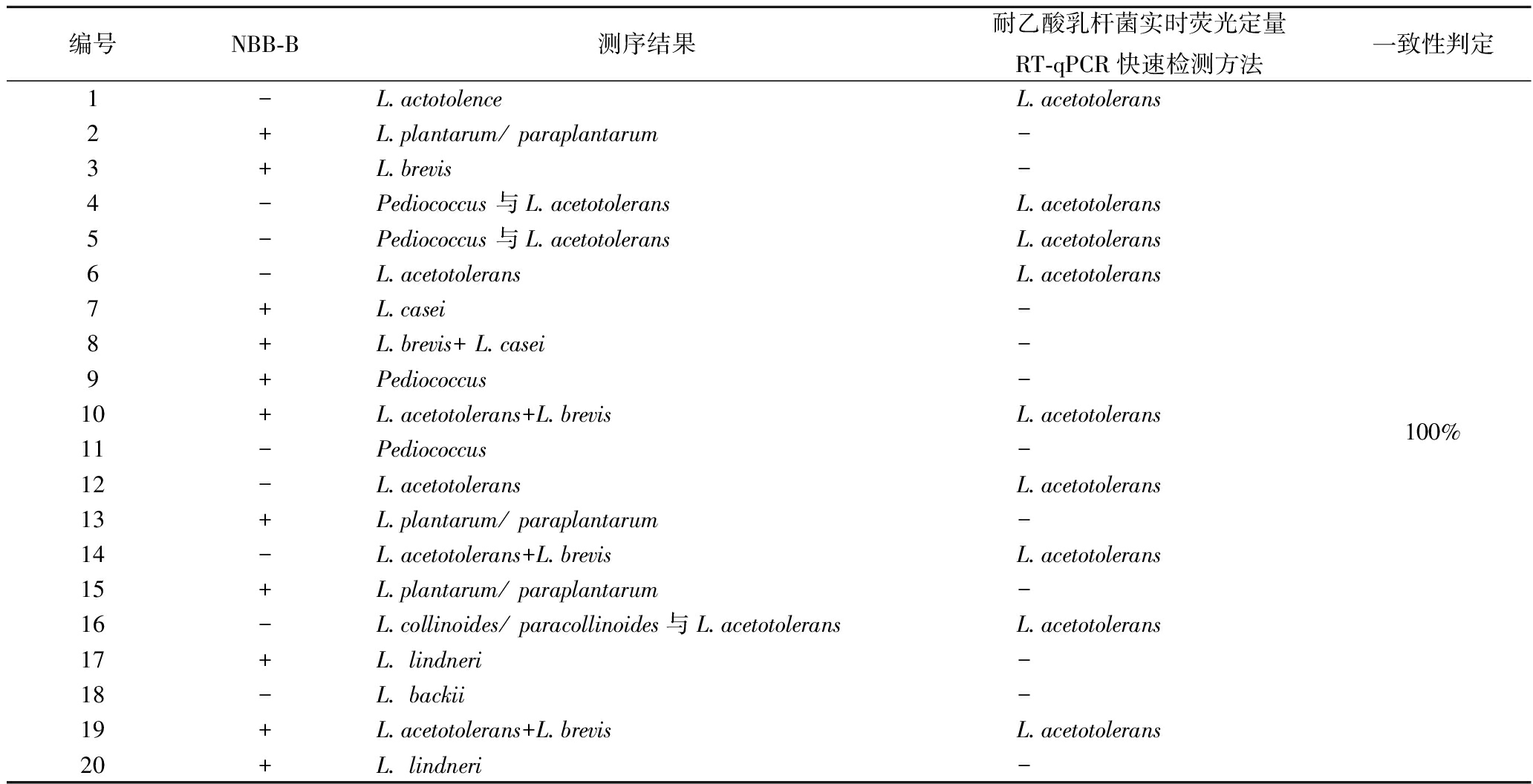
应用本研究开发的耐乙酸乳杆菌特异性引物检测生产上预培养2 d的20批样品,并同时进行菌株分析后的样品测序对比结果如表6所示。
表6 样品分离鉴定后的测序结果与新开发探针方法检测结果
Table 6 Sequencing results of isolated and identified sample and test results of newly developed method
编号NBB-B测序结果耐乙酸乳杆菌实时荧光定量RT-qPCR快速检测方法一致性判定1-L.actotolenceL.acetotolerans2+L.plantarum/ paraplantarum-3+L.brevis-4-Pediococcus 与L.acetotoleransL.acetotolerans5-Pediococcus 与L.acetotoleransL.acetotolerans6-L.acetotoleransL.acetotolerans7+L.casei-8+L.brevis+ L.casei-9+Pediococcus-10+L.acetotolerans+L.brevisL.acetotolerans11-Pediococcus-12-L.acetotoleransL.acetotolerans13+L.plantarum/ paraplantarum-14-L.acetotolerans+L.brevisL.acetotolerans15+L.plantarum/ paraplantarum-16-L.collinoides/ paracollinoides与L.acetotoleransL.acetotolerans17+L. lindneri-18-L. backii-19+L.acetotolerans+L.brevisL.acetotolerans20+L. lindneri-100%
注: “+”表示NBB-B液体培养基变色,判定为阳性;“-”表示不变色或未检测到,判定为阴性。
实验结果表明,检测的20批生产样品经预培养2 d后,只要样品中含有耐乙酸乳杆菌且达到检出限的样品,均可以使用本研究开发的方法进行有效检测。结果表明,本研究开发的耐乙酸乳杆菌特异性探针对实际样品中的耐乙酸乳杆菌检测具有特异性及准确性,检出效率为100%。
3 结论
本研究以耐乙酸乳杆菌3个种特异性基因为靶目标,成功设计并优化了3对种特异性引物用于啤酒腐败菌耐乙酸乳杆菌的检测。建立了基于SYBR GREEN实时荧光定量种特异性PCR方法及耐乙酸乳杆菌定量检测的标准曲线,实现了对耐乙酸乳杆菌的定性与定量检测。定量检测的最低检测限为102 CFU/mL,精度为10 CFU/mL。实际样品经富集培养2 d后,可以满足PCR检测需求,整个检测时间可控制3 d以内,特异性探针对生产上含有耐乙酸乳杆菌样品的检出率为100%。本研究开发的方法满足了啤酒生产企业对出厂产品进行质量控制的要求,为啤酒或其他食品中新发现的污染微生物检测提供了借鉴。
[1] 王伟, 刘雅文, 谷凤霞,等.啤酒腐败微生物与啤酒微生物稳定性研究进展[J].微生物学杂志, 2016, 36(1):80-88.
[2] 方贵权, 李惠萍, 涂京霞,等.中国啤酒易感微生物的研究[J]. 啤酒科技, 2011(2):25-28.
[3] DENG Y , LIU J , LI L , et al. Reduction and restoration of culturability of beer-stressed and low-temperature-stressed Lactobacillus acetotolerans strain 2011-8.[J]. International Journal of Food Microbiology, 2015, 206:96-101.
[4] 张全斌, 李艳琴. 啤酒腐败菌分子检测技术的研究进展[J]. 食品与药品, 2007, 9(6):55-57.
[5] COLLINS M D , RODRIGUES U , ASH C , et al. Phylogenetic analysis of the genus Lactobacillus and related lactic acid bacteria as determined by reverse transcriptase sequencing of 16S rRNA [J]. Fems Microbiology Letters, 2010, 77(1):5-12.
[6] GANGER M T , DIETZ G D , EWING S J . A common base method for analysis of qPCR data and the application of simple blocking in qPCR experiments[J]. BMC Bioinformatics, 2017, 18(1):534.
[7] 陈利娜. PCR技术用于啤酒中有害乳酸菌的检测和鉴定的研究[D]. 呼和浩特:内蒙古农业大学, 2008.
[8] SAKAMOTO K , MARGOLLES A , VAN VEEN H W , et al. Hop resistance in the beer spoilage bacterium Lactobacillus brevis is mediated by the ATP-binding cassette multidrug transporter HorA[J]. Journal of Bacteriology, 2001, 183(18):5 371-5 375.
[9] SAMI M , YAMASHITA H , HIRONO T , et al. Hop-resistant Lactobacillus brevis contains a novel plasmid harboring a multidrug resistance-like gene[J]. Journal of Fermentation and Bioengineering, 1997, 84(1):1-6.
[10] IIJIMA K, SUZUKI K, YAMASHITA H. horC confers beer-spoilage ability on hop-sensitive Lactobacillus brevis ABBC45cc[J]. Journal of Applied Microbiology, 2006,100(6): 1 282-1 288.
[11] DENG Y, LIU J, LI H, et al. An improved plate culture procedure for the rapid detection of beer-spoilage lactic acid bacteria[J]. Journal of the Institute of Brewing, 2014,120(2): 127-132.
[12] 任河山, 王雪涵, 姚金城, 等. 啤酒有害菌的PCR检测和SYBR Green实时PCR定量[J]. 啤酒科技, 2010(3):11-16.
[13] 栾春光, 罗娜, 郝建秦, 等. 啤酒腐败菌的分离鉴定和耐乙酸乳杆菌定量检测方法建立[J]. 中外酒业, 2018(23):14-18.
[14] 李凤云. NBB培养基在啤酒工厂中的应用[J]. 啤酒科技, 2003(12):20-25.
[15] HIDEHIRO T, HIDETOSHI M, HIROYUKI TSUJI, et al. Complete genome sequence of Lactobacillus acetotolerans RIB 9124 (NBRC 13120) isolated from putrefied (hiochi) Japanese sake[J]. Journal of Biotechnology, 2015, 214:214-215.
[16] DELCHER A L, BRATKE K A, POWERS E C, et al. Identifying bacterial genes and endosymbiont DNA with Glimmer[J]. Bioinformatics, 2007, 23(6):673-679.
[17] 姚纲, 陈永红, 胡红焱, 等. 棒状乳杆菌4个管家基因PCR引物的设计与验证[J]. 中国医学工程, 2015(8):86-87.
[18] 刘静. 实时荧光PCR方法快速检测鉴定啤酒有害菌的研究[J]. 啤酒科技, 2014(2):8-10.
[19] 杨怡姝, 孙晓娜, 王小利, 等. 实时荧光定量PCR技术的操作实践[J]. 实验室研究与探索, 2011(7):28-32.
[20] 谢鑫, 粱云, 郭立芸. 荧光定量PCR检测啤酒酿造过程中的戊糖片球菌[J]. 啤酒科技, 2016(4):19-24.
[21] 石晓路. 沙门氏菌荧光PCR快速检测方法的建立与应用[D]. 武汉:华中农业大学, 2003.
[22] 王燕梅, 符晓梅, 乔昕, 等. SYBR Green I实时荧光PCR检测食品中转基因成分方法的灵敏度和特异性研究[J]. 中国卫生检验杂志, 2012(1):69-71.