四川工业泡菜是一种高盐发酵蔬菜,多采用新鲜豇豆、萝卜、青菜、榨菜等为原料,通过利用蔬菜自身携带的微生物,在相对封闭的盐渍池中自然发酵而成的辅餐或调味食品[1],其中泡豇豆因酸爽脆嫩、风味独特而深受广大消费者的青睐[2-3],且豇豆中还含有丰富的氨基酸[4]等对人体有益的营养物质。近年来由于人们对食品的安全意识逐渐提高,国内外研究人员对发酵蔬菜潜在的风险因素也做了很多研究,如对生物胺的研究。
生物胺是一类含氮、具有生物活性的碱性低分子量有机化合物,主要通过机体细胞的代谢或微生物体内的脱羧反应形成[5]。生物胺是生物体内正常的活性成分,起着重要的生理作用,但当人体外源摄入过量生物胺就会造成一些不良症状[6],而且生物胺还是致癌性亚硝胺的前体物质[7-8]。已有研究表明,发酵蔬菜的生物胺含量可能存在安全隐患,而国内外对发酵蔬菜中生物胺的研究主要集中在含量测定,对其形成和降解研究较少,因此,本研究以发酵成熟的四川工业泡豇豆为研究对象,测定其生物胺和游离氨基酸的含量,采用Illumina HiSeq 4000 平台对其进行宏基因组测序,并在NR和KEGG数据库上进行注释,探明主要生物胺的形成和降解与微生物及酶的关系,为研究发酵蔬菜中生物胺的形成及降解提供参考。
1 材料与方法
1.1 材料与试剂
四川工业泡豇豆2份,四川省眉山市某知名泡菜企业,发酵成熟并贮藏1年以上;新鲜豇豆,四川省眉山市某超市;组胺、腐胺、酪胺、尸胺、色胺、苯乙胺、精胺、亚精胺生物胺标准品,美国Sigma-Aldrich公司;乙腈、甲醇等色谱纯溶剂,天津市致远化学试剂有限公司;Magnetic soil and stool DNA kit,北京天根生化科技(北京)有限公司;TruSeqTM DNA Sample Prep Kit、HiSeq 3000/4000 PE Cluster Kit、HiSeq 3000/4000 PE Cluster Kit、HiSeq 3000/4000 SBS Kits,美国Illumina公司;其他试剂均为国产分析纯。
1.2 仪器与设备
LC-2030高效液相色谱仪,日本岛津公司;MJ-54A高压灭菌锅,美国STIK公司;Nano Drop 2000分光光度计,美国Thermo公司;Covaris M220超声波DNA破碎仪,美国Illumina公司;Illumina Hiseq 4000测序平台,上海美吉生物医药科技有限公司。
1.3 实验方法
1.3.1 生物胺的测定
1.3.1.1 样品前处理
准确称取10 g样品于50 mL离心管中,加入1.25 mL 1.0 g/L的内标使用液(1,7-二氨基庚烷,10 g/L)与10 mL 0.1 mol/L的HCl溶液,振荡30 min,4 000 r/min离心20 min,取上清液;沉淀部分再加入15 mL 0.1 mol/L的HCl溶液,振荡30 min,4 000 r/min离心20 min,合并上述2次上清液,并用0.1 mol/L的HCl溶液定容至50 mL。
1.3.1.2 生物胺标准溶液制备与样品衍生
生物胺标准溶液的配制参照GB/T 5009.208—2016《食品中生物胺含量的测定》。根据FRIAS等[9]研究方法稍作改动,测定样品常见的8种生物胺。取1.0 mL标准溶液于50 mL离心管中,依次加入200 μL 2 mol/L NaOH溶液、300 μL饱和NaHCO3溶液缓冲,再加入2.0 mL丹酰氯衍生剂(10 g/L,溶剂为丙酮),振荡混匀后40 ℃避光水浴60 min,每15 min振荡1次。加入200 μL 100 g/L脯氨酸溶液,旋涡1 min后于室温避光放置15 min终止衍生反应,再加入0.4 g NaCl旋涡振荡1 min后加入1 mL乙醚,旋涡振荡30 s,静置分层后,吸出上层有机相,再萃取1次,合并有机相,45 ℃水浴挥干。用1.0 mL乙腈溶解残留物,0.22 μm有机膜过滤,于4 ℃避光保存,用于高效液相色谱测定。样品溶液衍生的条件及方法与标准溶液相同。
1.3.1.3 色谱条件
色谱柱为C18柱(250 mm×4.6 mm,5 μm),紫外检测波长254 nm,进样量20 μL,流速0.8 mL/min,柱温35 ℃,流动相A为乙腈,流动相B为去离子水,梯度洗脱条件见表1。
表1 梯度洗脱程序表
Table 1 Gradient elution program list

时间/min乙腈体积分数/%去离子水体积分数/%0604022851525100032100032.016040376040
1.3.2 游离氨基酸测定
1.3.2.1 样品前处理
取5 g样品,加水5 mL振荡混匀,50 ℃水浴30 min,离心,取上清液待用。
1.3.2.2 衍生试剂的制备和氨基酸衍生方法
根据HU等[10]研究方法稍作改动,测定样品中的游离氨基酸,取1 600 μL乙腈,200 μL三乙胺和20 μL异硫氰酸苯酯,混合即得衍生试剂。取100 μL氨基酸标准品或待测样品置于5 mL试管中;加入200 μL衍生试剂,旋涡混合20 s后放置60 min,加入2 mL水和1 mL正己烷,旋涡混合1 min除去上层溶液,再次加入1 mL正己烷,旋涡混合1 min后静置10 min,取下层溶液待用。
1.3.2.3 色谱条件
色谱柱AgilentXDB-C18柱(250 mm×4.6 mm,5 μm),检测波长为254 nm,进样量和流速分别为10 μL和1.0 mL /min;柱温40 ℃;流动相A:V[0.1 mol/L醋酸钠溶液(pH 6.50)]∶V(乙腈)=93∶7;流动相B:V(水)∶V(乙腈)=20∶80;梯度洗脱条件见表2。
表2 HPLC的梯度洗脱条件
Table 2 Gradient elution condition of HPLC
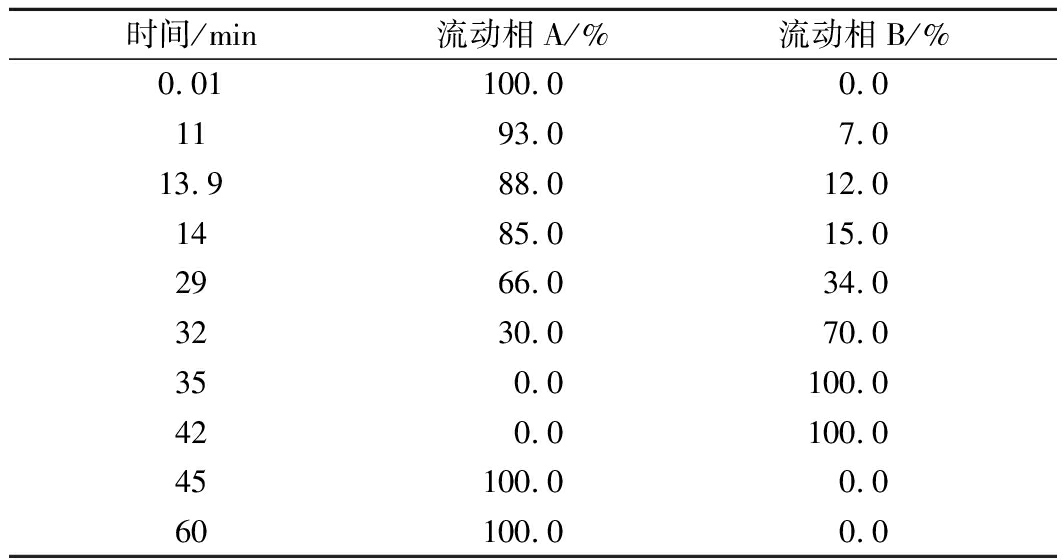
时间/min流动相A/%流动相B/%0.01100.00.01193.07.013.988.012.01485.015.02966.034.03230.070.0350.0100.0420.0100.045100.00.060100.00.0
1.3.3 DNA提取及宏基因组测序
样品滤液4 000 r/min离心5 min,收集2 g以上的菌泥。
根据Magnetic soil and stool DNA kit操作方法抽提样品基因组DNA,用Nano Drop2000分光光度计测定抽提的DNA浓度和纯度,利用1%琼脂糖凝胶电泳检测抽提基因组DNA;用Covaris M220超声波DNA破碎仪将DNA片段化至平均大小为300 bp左右,片段化后采用TruSeqTM DNA Sample Prep Kit试剂盒构建PE文库,将含有完整序列引物杂交位点的衔接子连接到片段末端;采用HiSeq 3000/4000 PE Cluster Kit进行桥式PCR,最后在上海美吉HiSeq测序平台,采用HiSeq 3000/4000 SBS Kits进行宏基因组测序。
1.2.4 序列质控和基因丰度计算
用Seqprep和Sickle软件对原始序列进行拆分、质量剪切等优化处理,在NCBI上检索样品微生物的基因组DNA,通过BWA软件比对已发表的基因组DNA序列,去除比对相似性高的污染序列;使用MEGAHIT软件对优化后的序列进行拼接[11],使用MetaGene软件对拼接结果进行开放性阅读框预测(ORFs)[12];使用CD-HIT软件对具有95%序列同源性(90%覆盖度)的预测基因进行聚类[13],选择每个簇的最长序列作为代表构建非冗余基因集,使用SOAPaligner软件分别将每个样品的高质量序列与非冗余基因集进行比对,统计基因在对应样品中的丰度信息[14]。
1.2.5 物种及功能注释
用BLASTP软件[15]将非冗余基因与NR数据库和京都基因百科全书(KEGG)基因数据库进行比对(BLAST比对参数设置期望值为10~5)。通过NR库对应的分类学信息数据库获得物种注释,构建相应分类学水平上的丰度谱;根据比对结果使用KOBAS 2.0进行功能注释,使用Pathway对应的基因丰度总和计算该功能类别的丰度。
2 结果与分析
2.1 氨基酸、生物胺检测结果
如图1所示,生物胺总含量大于1 000 mg/kg,对人体健康已经造成威胁,尸胺、酪胺、腐胺、组胺含量较高,分别占总生物胺含量的27.69%、19.05%、18.03%、17.53%,且样本中4种生物胺总含量在1 010 mg/kg左右,占8种生物胺总量的82.30%。可见样品中生物胺以尸胺、腐胺、组胺、酪胺为主,且与国内外研究发酵蔬菜中生物胺含量的报道一致[16-20]。
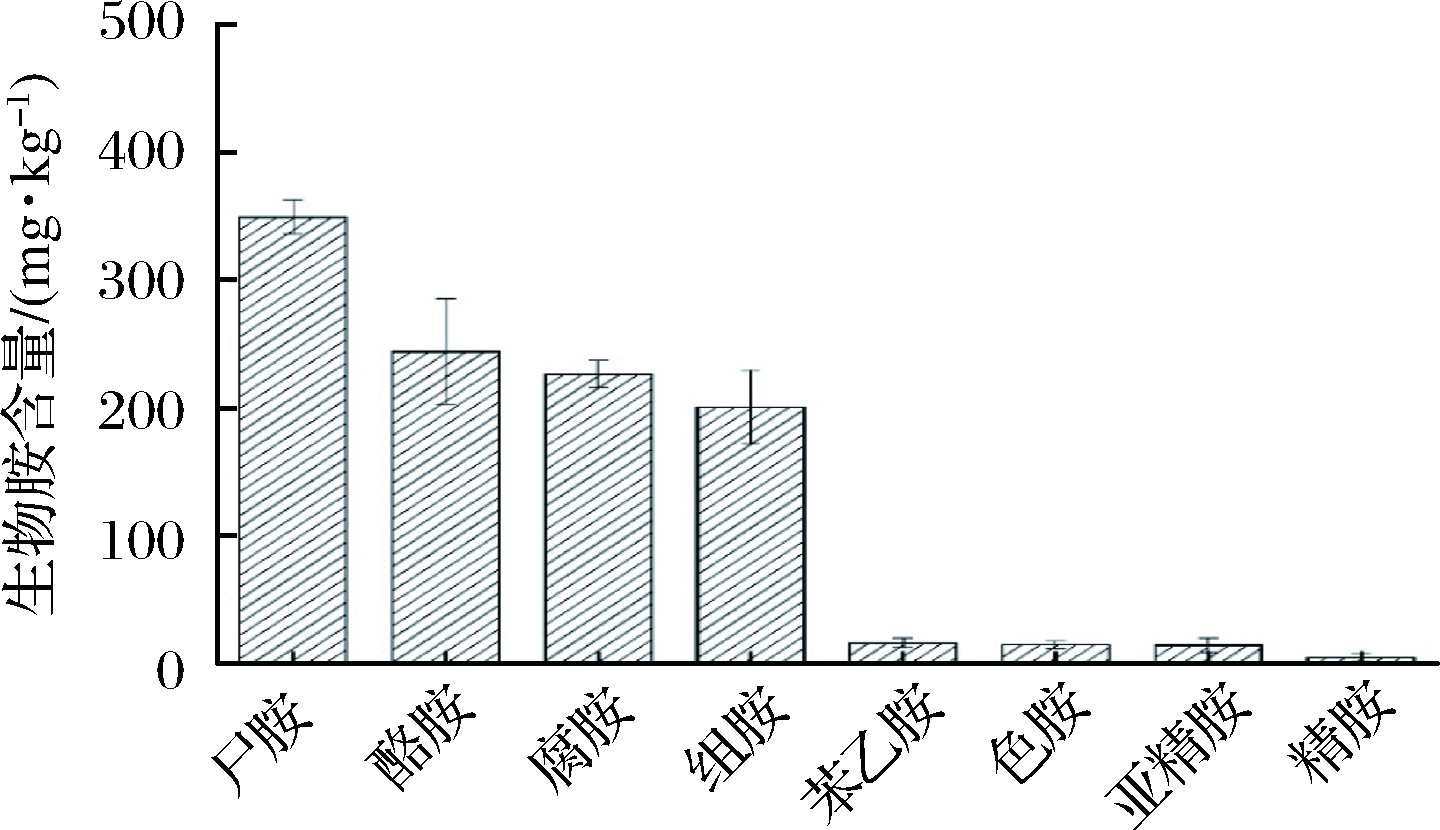
图1 样品生物胺含量
Fig.1 Contents of biogenic amines in samples
氨基酸是生物胺的前体物质,因此含有氨基酸脱羧酶基因的微生物可利用氨基酸产生相应的生物胺。如图2所示,豇豆发酵前后对比发现,组氨酸、精氨酸、酪氨酸、赖氨酸、鸟氨酸分别减少了153.58、114.02、57.48、85.85、24.46 mg/kg。样品中主要生物胺的主要前体氨基酸为赖氨酸、鸟氨酸、组氨酸、酪氨酸,但精氨酸酶作用于精氨酸后可产生鸟氨酸,于是可推测,泡豇豆体系中的微生物除利用豇豆的氨基酸供自身生长所需外,还可能利用氨基酸产生生物胺,这可能也是样品生物胺含量较高的原因之一。

图2 样品中5种氨基酸含量
Fig.2 Five kinds of amino acids in samples
2.2 主要生物胺形成及降解分析
2.2.1 生物胺关联微生物分析
通过KEGG数据库注释样品主要生物胺代谢相关酶,再筛选宏基因数据中这些酶关联到的微生物,并作生物胺关联物种相对丰度图,如图3所示。

图3 关联物种相对丰度
Fig.3 Relative abundance of associated species
样品中微生物主要以乳酸菌、酵母菌和芽孢菌为主,S.lignohabitans、酸鱼乳杆菌(Lactobacillus acidipiscis)、白地霉(G.candidum)的相对丰度较高,分别为10.60%、8.15%、6.19%。样品中生物胺关联微生物主要与腐胺形成与降解、尸胺降解、酪胺降解有关,且相对丰度>0.5%的物种主要与腐胺形成及降解、尸胺降解有关,与腐胺形成相关的微生物相对丰度较腐胺降解关联微生物高,同时具有生物胺形成及降解能力的物种相对丰度为51.05%。研究发现,L.acidipiscis、费斯莫尔德乳杆菌(Lactobacillus versmoldensis)、凝乳酶乳杆菌(Lactobacillus rennini)、短乳杆菌(Lactobacillus brevis)、戊糖乳杆菌(Lactobacillus pentosus)5种乳酸菌具有腐胺形成能力[21-22],Halomonas smyrnensis、Halomonas HG01、Halomonas salina 3种盐单胞菌具有腐胺和尸胺降解能力,Halomonas halodenitrificans具有酪胺降解能力[23]。推测样品中高含量的生物胺与样品中高相对丰度的生物胺关联微生物有关。
2.2.2 主要生物胺代谢通路
图4~图7分别表示样本中4种主要生物胺的代谢与微生物及酶的关系图(括号内数字表示该酶关联的物种数量)。
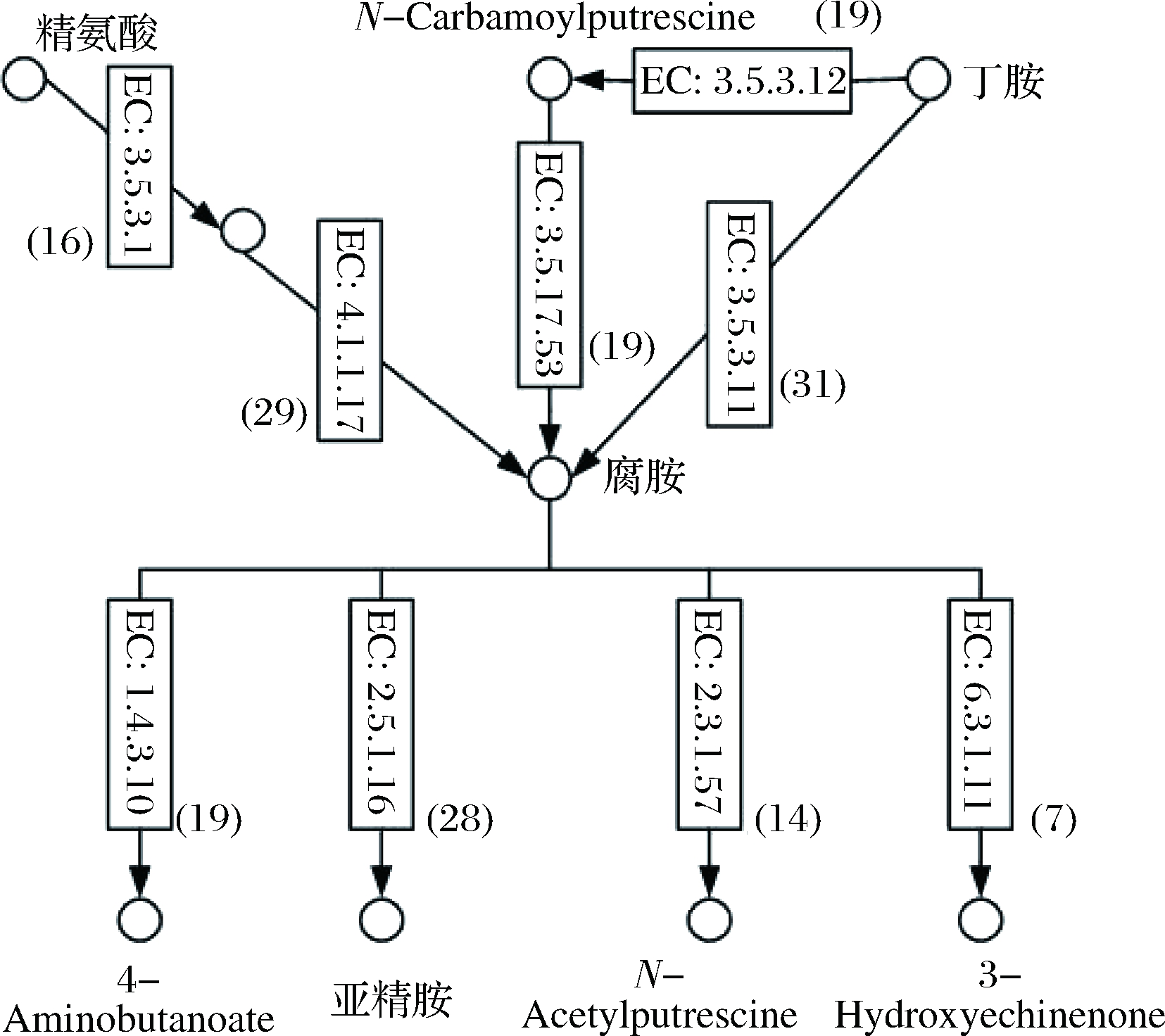
图4 腐胺代谢路径
Fig.4 Putrescine metabolic pathway
图4为结合KEGG PATHWAY中ko00480、ko00960、ko00330注释的腐胺代谢通路图。样品中腐胺的形成路径有3条,与微生物利用氨基酸或其他物质通过脱羧、转胺、合成有关,涉及到的酶有鸟氨酸脱羧酶(EC:4.1.1.17)、胍丁胺酶(EC:3.5.3.11)、EC:3.5.1.53等。丰度较高具有EC:4.1.1.17的微生物有L.acidipiscis、L.versmoldensis、假丝酵母(Debaryomyces hansenii)等,具有EC:3.5.3.11的微生物有S.lignohabitans、D.hansenii、Millerozyma farinosa等,具有EC:3.5.1.53的微生物有H.halodenitrificans、H.smyrnensis、Halomonas sp. HG01等。样品中共有89种微生物含有与腐胺形成相关的酶,其中包括泡菜体系常见的产生物胺乳酸菌[24-26],如L.brevis含有鸟氨酸脱羧酶和琼脂脱硫酶,L.lactis含有琼脂脱硫酶,L.pentosus含有精氨酸酶,且样本的菌群结构中乳酸菌的相对丰度较高。腐胺可通过氧化、转移、合成等途径得到降解,其中相对丰度较高具有亚精胺合成酶(EC:2.5.1.16)的微生物有S.lignohabitans、D.hansenii、H.smyrnensis等,具有EC:6.3.1.11的微生物有阴沟肠杆菌(Enterobacter cloacae)、unclassified f Enterobacteriaceae、桑肠杆菌(Enterobacter mori)等,具有EC:1.4.3.10的微生物有Corynebacterium variabile、金色棒状杆菌(Corynebacterium aurimucosum)、Arthrobacter sp. NIO—1057。样品中有49种微生物含有与腐胺降解相关的酶,但仅占腐胺形成关联微生物的一半,与腐胺形成和降解相关的微生物分别有70、45种,但只与腐胺形成、腐胺降解相关的微生物分别是48、13种,同时含有腐胺降解和形成相关酶的L. acidipiscis、D.hansenii、M.farinosa等8种微生物相对丰度较高,因此推测样品中高含量的腐胺不仅与腐胺形成微生物的数目及表达能力有关,还可能与微生物实际参与的代谢途径有关。
图5为结合KEGG PATHWAY中ko00340注释的组胺代谢通路图。组胺只由微生物通过组氨酸脱羧酶(EC:4.1.1.22)作用于组氨酸脱羧形成,样品中具有该能力的微生物有2种(Halomonas elongata、Lactobacillus hilgardii)。组胺可以通过酶氧化[27-29]、基团转移、化学合成3条途径得到降解,但未在样品中发现有与这3条降解路径相关的微生物。分析可能由于样品中的微生物含有产生组胺的酶,而没有降解组胺的酶,从而导致样品中检测出高含量的组胺。
图5 组胺代谢路径
Fig.5 Histamine metabolic pathway
![]() 图6为结合KEGG PATHWAY中ko00350、ko00680、ko00950注释的酪胺代谢通路图。酪胺可通过酪氨酸脱羧酶(EC:4.1.1.25)作用于酪氨酸脱羧酶形成,样本中有2种微生物(Natronococcus amylolyticus)、嗜盐球菌(Halococcus morrhuae)含有EC:4.1.1.25。酪胺降解的途径只有1条,涉及到的微生物有6种H.halodenitrificans、Propionibacterium acidipropionici、盐碱球菌(Natronococcus occultus)等,均为通过单胺氧化酶(EC:1.4.3.4)来降解酪胺。但有报道[30]称,生物胺本身也有抑制生物胺氧化酶活性的作用,如酪胺能抑制单胺氧化酶的活性,色胺抑制二胺氧化酶的活性[31],从而抑制某些生物胺的降解。LEUSCHNER等[32]认为酪胺氧化酶最适pH值为7~8,并且还受食盐和葡萄糖添加量的影响[33],因此在豇豆发酵过程中酪胺氧化酶、单胺氧化酶活性可能会受到抑制,单胺氧化酶作用于酪胺的强度受到限制,可能导致酪胺难以降解,并逐渐积累。
图6为结合KEGG PATHWAY中ko00350、ko00680、ko00950注释的酪胺代谢通路图。酪胺可通过酪氨酸脱羧酶(EC:4.1.1.25)作用于酪氨酸脱羧酶形成,样本中有2种微生物(Natronococcus amylolyticus)、嗜盐球菌(Halococcus morrhuae)含有EC:4.1.1.25。酪胺降解的途径只有1条,涉及到的微生物有6种H.halodenitrificans、Propionibacterium acidipropionici、盐碱球菌(Natronococcus occultus)等,均为通过单胺氧化酶(EC:1.4.3.4)来降解酪胺。但有报道[30]称,生物胺本身也有抑制生物胺氧化酶活性的作用,如酪胺能抑制单胺氧化酶的活性,色胺抑制二胺氧化酶的活性[31],从而抑制某些生物胺的降解。LEUSCHNER等[32]认为酪胺氧化酶最适pH值为7~8,并且还受食盐和葡萄糖添加量的影响[33],因此在豇豆发酵过程中酪胺氧化酶、单胺氧化酶活性可能会受到抑制,单胺氧化酶作用于酪胺的强度受到限制,可能导致酪胺难以降解,并逐渐积累。
图6 酪胺代谢路径
Fig.6 Tyramine metabolism pathway
![]() 图7为结合KEGG PATHWAY中ko00310、ko00960、ko00480注释的尸胺代谢通路图。样品中尸胺的合成路径只有1条,且注释到尸胺合成路径的微生物有10种,皆是通过自身的赖氨酸脱羧酶(EC:4.1.1.18)作用于赖氨酸产生尸胺,其中丰度较高的微生物有E.cloacae、癌杆菌(Oceanobacillus oncorhynchi)等。尸胺主要通过氧化、转移、合成3种途径降解,关联的微生物共有47种,丰度较高具有EC:2.5.1.16的微生物有S.lignohabitans、D.hansenii、H.smyrnensis等,具有伯胺氧化酶(EC:1.4.3.21)的微生物有S.lignohabitans、G.candidum、D.hansenii等,具有腐胺氨基转移酶(EC:2.6.1.82)的微生物有E.cloacae、乳酸乳球菌(Lactococcus lactis)、Klebsiella cf. Planticola B43等。数据显示高丰度的S.lignohabitans不仅含有酶EC:2.5.1.16,还含有酶EC:1.4.3.21。 E.cloacae不仅含有尸胺合成酶,还含有尸胺降解酶。
图7为结合KEGG PATHWAY中ko00310、ko00960、ko00480注释的尸胺代谢通路图。样品中尸胺的合成路径只有1条,且注释到尸胺合成路径的微生物有10种,皆是通过自身的赖氨酸脱羧酶(EC:4.1.1.18)作用于赖氨酸产生尸胺,其中丰度较高的微生物有E.cloacae、癌杆菌(Oceanobacillus oncorhynchi)等。尸胺主要通过氧化、转移、合成3种途径降解,关联的微生物共有47种,丰度较高具有EC:2.5.1.16的微生物有S.lignohabitans、D.hansenii、H.smyrnensis等,具有伯胺氧化酶(EC:1.4.3.21)的微生物有S.lignohabitans、G.candidum、D.hansenii等,具有腐胺氨基转移酶(EC:2.6.1.82)的微生物有E.cloacae、乳酸乳球菌(Lactococcus lactis)、Klebsiella cf. Planticola B43等。数据显示高丰度的S.lignohabitans不仅含有酶EC:2.5.1.16,还含有酶EC:1.4.3.21。 E.cloacae不仅含有尸胺合成酶,还含有尸胺降解酶。

图7 尸胺代谢路径
Fig.7 Cadaverine metabolic pathway
除综上所述有8种微生物同时与腐胺的形成及降解相关外,据宏基因数据显示,有6种微生物,如肠杆菌(Enterobacter asburiae)、unclassified g Enterobacter、E.cloacae[16]、催产克雷伯氏菌(Klebsiella oxytoca)、D.hansenii、油桃千酵母菌(Millerozyma arinosa),同时与腐胺、尸胺的形成与降解相关,其中包含肠杆菌,这与相关报道[21-23]肠杆菌产生物胺相一致。unclassified f Enterobacteriaceae与尸胺的形成和降解相关,E.hormaechei与腐胺和尸胺的形成相关,A.sp. NIO-1057与腐胺和尸胺的降解相关,N.occμLtus与尸胺和酪胺的降解相关。不难发现,一种微生物可能同时与不同生物胺的形成和降解相关,这与生物胺形成菌的相关报道[21-22]一致。有研究[34]发现菌株可以选择性释放胞内生物胺来保持环境pH值的平衡,且环境低pH值能激活细菌的脱羧代谢途径,在低pH值下增加生物胺产生量以提高对酸性环境的抵抗能力[35-36],此时,赖氨酸→尸胺,鸟氨酸→腐胺的产生途径相对其他生物胺产生途径更为有效。DAPKVICIUS等[33]研究表明,二胺氧化酶的最适pH为7.00,在5.00~10.00时,此酶都有活性,但当pH在5.00和10.00时酶活都很低。王振等[30]研究发现,pH 4.0~5.5的酸性环境有利于提高微生物体内的氨基酸脱羧酶活性,从而促进微生物产生生物胺,而微生物产胺氧化酶非常有限[37]。可见样品中生物胺的含量不仅与产生物胺微生物种类[24,38-39]、数量有关,还与参与途径时关联酶的表达能力有关。
3 结论
利用KEGG数据库分析样品宏基因数据,得到样品的主要生物胺代谢路径及关联的物种和酶,发现样品中主要生物胺腐胺、尸胺、酪胺、组胺代谢关联的物种相对丰度较高。具有主要生物胺形成及降解能力的物种相对丰度高达51.05%,且同一种微生物可能参与生物胺的多条代谢路径。样品中高含量的腐胺、尸胺、酪胺、组胺不仅来源于豇豆本身,而且可能更多地来源于高丰度的产生物胺微生物利用豇豆丰富的氨基酸资源产生生物胺,而微生物的种类、数量与其参与生物胺代谢时的表达能力也对生物胺含量有一定影响。本研究揭示了四川工业泡豇豆中生物胺形成及降解与关联微生物及酶的关系,确定了四川工业泡豇豆主要生物胺形成及降解的路径,不仅为企业实际应用提供数据支撑,还为发酵蔬菜生物胺的形成及降解奠定理论基础。
[1] 张林,罗陶,张华,等.四川泡菜分类归属及定义的分析与建议[J].食品安全质量检测学报,2019,10(5):1 250-1 253.
[2] 向文良, 车振明,陈功. 四川泡菜加工原理与技术[M]. 北京:中国轻工业出版社, 2015.
[3] YUSUF A,ZEYNAL T, METIN D. Biogenic amine and fermentation metabolite production assessments of Lactobacillus plantarum isolates for naturally fermented pickles[J]. LWT-Food Science and Technology, Elsevier Ltd, 2018,98: 322-328.
[4] 李安林, 熊双丽. 豇豆籽蛋白的氨基酸含量与营养价值评价[J]. 食品研究与开发, 2008, 29(6):147-150.
[5] SHRUTI SHUKLA,JONG SUK LEE,VIVEK K,et al. Toxicological evaluation of lotus, ginkgo, and garlic tailored fermented Korean soybean paste (Doenjang) for biogenic amines, aflatoxins, and microbial hazards[J]. Food and Chemical Toxicology,2019,133:110 729.
[6] 管世敏. 降解亚硝酸盐乳酸菌的分离筛选及其在泡菜发酵中的应用研究[D]. 上海:上海师范大学, 2009.
[7] 王光强, 俞剑燊,胡健, 等. 食品中生物胺的研究进展[J]. 食品科学, 2016, 37(1):269-278.
[8] JEON A R, LEE J H, MAH J H. Biogenic amine formation and bacterial contribution in, Cheonggukjang, a Korean traditional fermented soybean food[J]. LWT, 2018,92:282-289.
[9] J. FR AS, C. MART
AS, C. MART NEZ-VILLALUENGA, GΜLEWICZ P, et al. Biogenic amines and HL60 citotoxicity of alfalfa and fenugreek sprouts[J]. Food Chemistry, 2007, 105(3):959-967.
NEZ-VILLALUENGA, GΜLEWICZ P, et al. Biogenic amines and HL60 citotoxicity of alfalfa and fenugreek sprouts[J]. Food Chemistry, 2007, 105(3):959-967.
[10] 胡建鸿,邱利焱,王成润,等.柱前衍生反相高效液相色谱法同时测定氨基酸保健饮品中16种氨基酸的含量[J]. 食品科技,2008,29(10):211-214.
[11] LI D, LIU C M, LUO R, et al. MEGAHIT: anultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph[J]. Bioinformatics, 2015, 31(10): 1 674-1 676.
[12] NOGUCHI H, PARK J, TAKAGI T. MetaGene: prokaryotic gene finding from environmental genome shotgun sequences[J]. Nucleic Acids Research, 2006, 34(19): 5 623-5 630.
[13] FU L, NIU B, ZHU Z, et al. CD-HIT: accelerated for clustering the next-generation sequencing data[J]. Bioinformatics, 2012, 28(23): 3 150-3 152.
[14] LI R, LI Y, KRISTIANSEN K, et al. SOAP: short oligonucleotide alignment program[J]. Bioinformatics, 2008, 24(5):713-714.
[15] XIE C, MAO X, HUANG J, et al. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 2011, 39(suppl 2): W316-W322.
[16] HSIEN-FENG KUNG, YIING-HORNG LEE, DER-FENG TENG. Histamine formation by histamine-forming bacteria and yeast in mustard pickle products in Taiwan[J]. Food Chemistry, 2006,99(3): 579-585.
[17] 霍晓慧,宫瑞泽,王燕华,等.UPLC法测定桔梗泡菜中的生物胺类成分[J].食品科技,2018,43(9):325-329.
[18] LIU L, DU P, ZHANG G, et al. Residual nitrite and biogenic amines of traditional northeast sauerkraut in China[J]. International Journal of Food Properties, Taylor & Francis, 2017, 20(11): 2 448-2 455.
[19] KIM H J, RDA, SUWON, Republic of Korea. investigation on biogenic amines in plant- based minor Korean fermentated foods[J]. Journal of Applied Biological Chemistry, 2013, 56(2):113-117.
[20] 瞿凤梅,丁晓雯,王瑜. 市售泡菜中生物胺含量的比较分析[J]. 食品工业科技, 2014, 35(21): 316-319;323.
[21] 田丰伟, 孟甜,丁俊荣,等. 蔬菜发酵剂乳酸菌产生物胺的检测与评价[J]. 食品科学, 2010, 31(24):241-245.
[22] DEL R B, MARCOBAL A, CARRASCOSA A V, et al. PCR detection of foodborne bacteria producing the biogenic amines histamine, tyramine, putrescine, and cadaverine.[J]. Journal of Food Protection, 2006, 69(10):2 509-2 514.
[23] LUCAS P L A. Purification and partial gene sequence of the tyrosine decarboxylase of Lactobacillus brevis IOEB 9809[J]. FEMS Microbiology Letters, 2002, 211(1): 85-89.
[24] 谷静思. 传统发酵臭豆腐中微生物菌相结构解析及其与生物胺形成的关系[D].杭州:浙江大学,2018.
[25] 张吉明, 赵燕梅,许庆方. 产生物胺乳酸菌研究概述[J]. 山西农业科学, 2014, 42(5):521-525.
[26] 王长远, 王云光,于长青, 等. 产生物胺乳酸菌的检测及产生物胺量的测定[J]. 农产品加工(学刊), 2010(1):22-25.
[27] 李彬彬,徐晔,牛淑慧,等.食品中生物胺含量及生物胺氧化酶的研究进展[J]. 食品科学,2019,40(1):341-347.
[28] 燕慧. 发酵食品中的生物胺问题及其控制措施探讨[J]. 中国调味品, 2014, 39(7): 130-132.
[29] SANTOS-BUELGA C, PE A-EGIDO M J R J C. Changes in tyramine during chorizo-sausage ripening[J]. Journal of Food Science, 2015, 51(2): 518-519.
A-EGIDO M J R J C. Changes in tyramine during chorizo-sausage ripening[J]. Journal of Food Science, 2015, 51(2): 518-519.
[30] 王振. HPLC法检测葡萄酒中的生物胺[D]. 青岛:青岛科技大学, 2013.
[31] MARTUSCELLI M, CRUDELE M A, GARDINI F, et al. Biogenic amine formation and oxidation by Staphylococcus xylosus strains from artisanal fermented sausages[J]. Letters in Applied Microbiology, 2010, 31(3):228-232.
[32] LEUSCHNER R G, HEIDEL M, HAMMES W P. Histamine and tyramine degradation by food fermenting microorganisms[J]. International Journal of Food Microbiology, 1998, 39(1-2):1-10.
[33] DAPKEVICIUS M L N E, NOUT M J R, ROMBOUTSO F M, et al. Biogenic amine formation and degradation by potential fish silage starter microorganisms[J]. International Journal of Food Microbiology, 2000, 57(1-2):107-114.
[34] 钟凯. 基于NMR的乳酸菌生物胺通路的代谢组学分析及安全性评价中的应用[D]. 北京:中国疾病预防控制中心,2009.
[35] GALE E F. The bacterial amino acid decarboxylases[M]// Advances in Enzymology and Related Areas of Molecular Biology, Volume 6. John Wiley & Sons, Inc. 2006,6:1-32.
[36] MAR A F, LINARES D M, ANA R, et al. Factors affecting tyramine production in Enterococcus durans, IPLA 655[J]. Applied Microbiology and Biotechnology, 2007, 73(6):1 400-1 406.
A F, LINARES D M, ANA R, et al. Factors affecting tyramine production in Enterococcus durans, IPLA 655[J]. Applied Microbiology and Biotechnology, 2007, 73(6):1 400-1 406.
[37] 黄瑶,罗爱玲,彭铭烨,等.微生物胺氧化酶研究进展[J].中国酿造,2016,35(9):24-27.
[38] COSANSU S. Determination of biogenic amines in a fermented beverage, boza[J]. Journal of Food, Agriculture and Environment, 2009, 7(2): 54-58.
[39] XIA X L, LUO Y, ZHANG Q W, et al. Mixed starter culture regulate biogenic amines fournation via decarboxylation and transamination during Chinase rice wine fermentation[J]. Journal of Agricultural and Food Chemistry,2018,66(25): 6 348-6 356.