近年来,乳酸菌的开发成为食品和药品的研发热点,一些菌株经过研究发现具有良好的应用价值,而且越来越多不同种或不同菌株的乳酸菌被添加到婴儿配方食品、保健品和药品中。但是,最近的研究表明乳酸菌发挥特定的功能特性可能具有菌株特异性,因此FAO/WHO益生菌评价指南中明确指出,精确的菌株识别是益生菌使用的必要前提[1]。对于益生菌来说,精确的菌株识别有助于确定其特定的益生功能,也有利于进行群体进化研究,包括菌群结构调查和菌群遗传学分析。所以,乳酸菌的分类鉴定现在越来越受到各国科学家的重视和关注。在乳酸菌的分类鉴定方法中,传统的生理生化鉴定方法具有耗时长、花费高、准确率低、不能鉴定到株的缺点。近年来分子生物学鉴定方法由于其快速准确的特点已经被广泛应用于乳酸菌鉴定中,多位点序列分型(multiloci sequence typing,MLST)就是近年来发展较为迅速的鉴定手段之一。与其他鉴定方法相比,MLST具有操作简单、重复性好的优点,通过标准化处理实现数据共享,其分辨率可以达到亚种的水平,是一种良好的菌株生物标签制备方法。目前,已经有多种乳酸菌进行了MLST分型,如干酪乳杆菌、植物乳杆菌、德式乳杆菌等,均得到了丰富的分类和进化信息。
马乳酒样乳杆菌ZW3[2]是从高加索地区开菲尔粒中筛选分离得到的一株优良菌株,经过实验室的长期研究发现,此菌株具有耐酸、耐胆盐、高产胞外多糖[3]等良好的生物学特性和降解亚硝酸盐[4]、调节肠道微生态[5-6]、抗抑郁[7]等益生功能。
为了快速、准确检测出马乳酒样乳杆菌ZW3,了解其在种内和近源种间的遗传位置,本研究对马乳酒样乳杆菌ZW3进行了遗传生物标签特性研究,通过对多个不同来源的马乳酒样乳杆菌和开菲尔乳杆菌的MLST分型,确定了马乳酒样乳杆菌ZW3的生物遗传信息标签,能够对马乳酒样乳杆菌ZW3菌株进行精确识别。
1 材料与方法
1.1 材料
1.1.1 样品及来源
1株马乳酒样乳杆菌ZW3保藏在本实验室。9株乳杆菌分离自俄罗斯(3株)、西藏(4个样品分别来自不同家庭)和南奥塞梯(2株)的开菲尔粒和雪莲菌粒中,菌株具体来源信息见表1。
1.1.2 培养基
MRS培养基(g/L):葡萄糖20,牛肉浸粉5,蛋白胨10,酵母浸膏4,MgSO4·7H2O 0.2,MnSO4·4H2O 0.5,K2HPO42,柠檬酸三胺2,无水乙酸钠5,吐温-80 1,pH调至6.2,121 ℃灭菌20 min。
表1 菌株的来源信息表
Table 1 Resource information of Lactobacillus

菌株编号样品来源地区分离源an1西藏-1雪莲菌粒an2西藏-1雪莲菌粒an3西藏-1雪莲菌粒shan3俄罗斯开菲尔粒shan4俄罗斯开菲尔粒shan5俄罗斯开菲尔粒liao3南奥塞梯开菲尔粒liao7南奥塞梯开菲尔粒XL10西藏-2雪莲菌粒
1.1.3 主要试剂
细菌基因组提取试剂盒:北京康为世纪生物科技有限公司;核酸染料:美国Genview公司;DNA marker:北京全式金生物技术有限公司;2×Taq PCR Mix:北京奥赛博科技发展有限公司;琼脂糖(UP):法国Biowest公司;Tris(BR):北京索莱宝科技有限公司;过氧化氢(AR):天津市北方天医化学试剂厂;冰乙酸(AR):天津江天化工技术有限公司;EDTA(LR):天津北方天医化学试剂厂;引物合成及序列测定均由苏州金唯智生物科技有限公司完成。
1.1.4 仪器与设备
PCR仪,德国Eppendorf公司;DL-31DN水平电泳槽,北京东林昌盛生物技术有限公司; DYY-Ⅲ-6B 型稳压稳流电泳仪,北京六一仪器厂;Gel DocTM xR+凝胶成像装置,美国BIO-RAD公司;多功能厌氧培养箱,美国GENE-SCIENCE公司。
1.1.5 引物
所选取基因的名称、功能、引物序列(5′-3′)、扩增长度、退火温度详见表2,引物分别用无菌ddH2O稀释到100 μmol/L,置于-20℃保存。
表2 PCR扩增MLST目的基因片段引物
Table 2 Primers used for amplification of MLST gene sequences
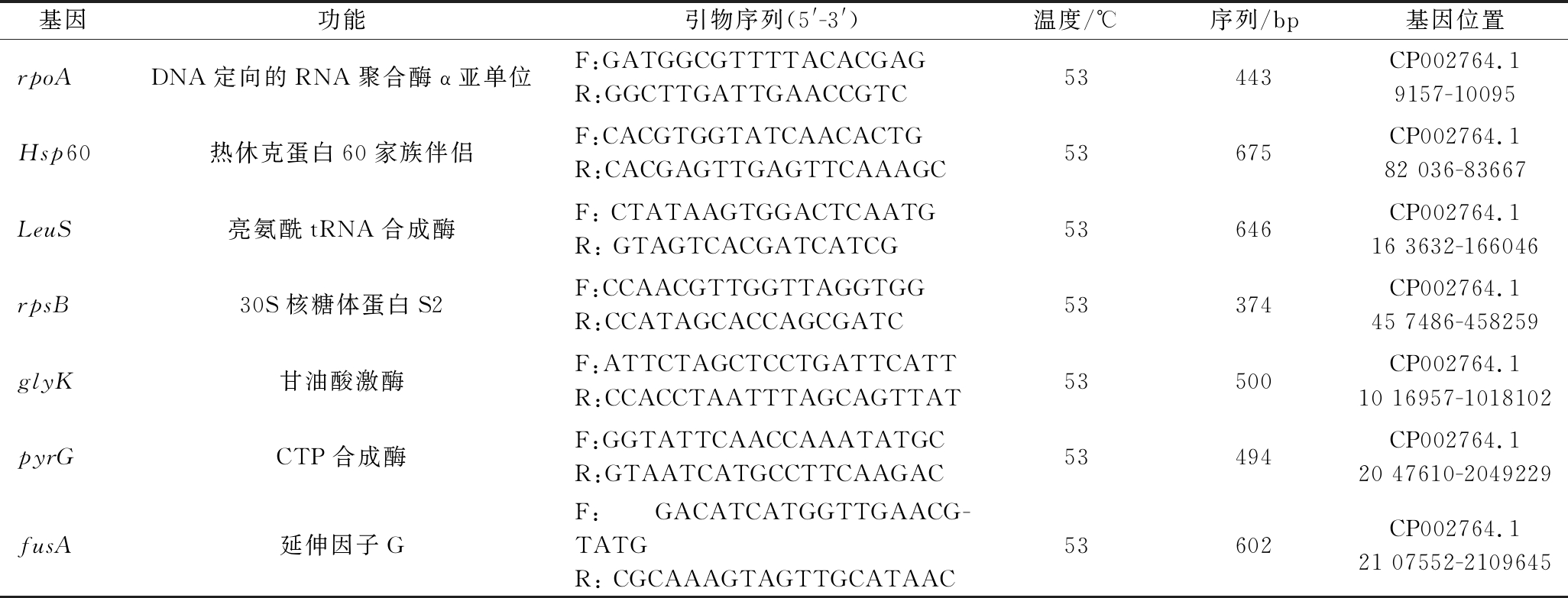
基因功能引物序列(5'-3')温度/℃序列/bp基因位置rpoADNA定向的RNA聚合酶α亚单位F:GATGGCGTTTTACACGAGR:GGCTTGATTGAACCGTC53443CP002764.19157-10095Hsp60热休克蛋白60家族伴侣F:CACGTGGTATCAACACTGR:CACGAGTTGAGTTCAAAGC53675CP002764.182 036-83667LeuS亮氨酰tRNA合成酶F: CTATAAGTGGACTCAATGR: GTAGTCACGATCATCG53646CP002764.116 3632-166046rpsB30S核糖体蛋白S2F:CCAACGTTGGTTAGGTGGR:CCATAGCACCAGCGATC53374CP002764.145 7486-458259glyK甘油酸激酶F:ATTCTAGCTCCTGATTCATTR:CCACCTAATTTAGCAGTTAT53500CP002764.110 16957-1018102pyrGCTP合成酶F:GGTATTCAACCAAATATGCR:GTAATCATGCCTTCAAGAC53494CP002764.120 47610-2049229fusA延伸因子GF:GACATCATGGTTGAACG-TATGR: CGCAAAGTAGTTGCATAAC53602CP002764.121 07552-2109645
注:表中以Lb.kefiranofaciens ZW3为参考基因组。
1.2 确定菌株序列型(ST型)
1.2.1 菌株活化及16S rRNA鉴定
取-80℃冻存的马乳酒样乳杆菌ZW3与9株同种或近源乳杆菌an1、an2、an3、shan3、shan4、shan5、liao3、liao7、XL10充分活化,按4%接种量接种于MRS液体培养基中厌氧培养,活化2代后,采用细菌全基因组提取试剂盒对各菌株细菌全基因组DNA进行提取。以提取的细菌全基因组DNA为模板,以细菌16S rRNA序列通用引物(27F:GAGTTTGATCCTGGCTCAG;1492R:TACCGCGGCTGCTGGCAC)进行PCR扩增。反应总体系为50.0 μL,DNA模板1.0 μL,2×Taq PCR Mix为25.0 μL,上下游引物(10 μmol/L) 各1.0 μL,ddH2O 为22.0 μL。PCR反应程序为:95 ℃预变性 8 min;95 ℃变性 45 s,55 ℃退火30 s,72 ℃延伸 2 min,共进行 35 个循环;最后 72 ℃延伸 10 min。反应结束后用0.8%(质量分数)的琼脂糖凝胶电泳分析。序列测序完成后,利用NCBI数据库进行序列相似性比对,并将所得序列结果上传至NCBI Genbank数据库中得到唯一的序列编号。
1.2.2 PCR扩增MLST目的基因片段
以提取的基因组DNA作为PCR模板,分别对7个基因(rpoA、Hsp60、LeuS、rpsB、glyK、pyrG、fusA)进行PCR扩增。反应总体系为50.0 μL:DNA模板1.0 μL,2×Taq PCR Mix为25.0 μL,上下游引物(10 μmol/l) 各1.0 μL,ddH2O 为22.0 μL。PCR反应程序为:95 ℃预变性 8 min;95 ℃变性 45 s,53 ℃退火30 s,72 ℃延伸 1 min,共进行 35 个循环;最后 72 ℃延伸 10 min。反应结束后用0.8%(质量分数)的琼脂糖凝胶电泳分析。序列测序完成后,利用NCBI数据库进行序列相似性比对,并将所得序列结果上传至NCBI Genbank数据库中得到唯一的序列编号。
1.2.3 确定菌株序列型(ST型)
人工对单个基因的SNP位点进行统计,根据SNP位点数目和位置的不同确定对应等位基因的编号,按照各基因在参考基因组(Lactobacillus kefiranofaciens ZW3)上的排列顺序得到等位基因图谱(基因排列顺序为rpoA-Hsp60-LeuS-rpsB-glyK-pyrG-fusA),每个菌株的等位基因图谱对应一个ST型。
1.3 等位基因和菌株遗传进化分析
1.3.1 等位基因多样性、连锁平衡、重组分析
利用START2.0[8](http://pubmlst.org/software/analysis/start2/)计算得出7个基因的GC含量、dN/dS比率(dN为非同义突变位点数目,dS为同义突变位点的数目)、单核苷酸多态性位点数目。利用LIAN 3.0[9](http://guanine.evolbio.mpg.de/cgi-bin/lian/lian.cgi.pl)计算7个基因的遗传多态性(mean genetic diversity,H)和标准关联指数(standardized index of association,ISA)值,对它们进行连锁平衡分析。利用SplitsTree 4.0[10]软件分析基因的重组情况,构建重组分解图。
1.3.2 不同菌株的聚类分析
采用eBURSTv3[11]程序(http://eburst.mlst.net/v3/enter_data/single/)进行克隆复合体计算,将各菌株的ST型划分为不同的克隆复合体,分析菌株间亲缘远近。将各菌株的ST型分别通过START2.0和MEGA6.0中的UPGMA进行系统发育树绘制,利用itol[12] (http://itol.embl.de/itol.cgi)在线绘图工具标记分离地区。
2 结果与分析
2.1 菌株鉴定结果
对9株不同来源的乳杆菌在MRS液体培养基中进行菌株活化,并进行菌株16S rRNA鉴定,结果如表3所示。9株菌中2株为开菲尔乳杆菌(L.kefir),另外7株为马乳酒样乳杆菌(L. kefiranofaciens)。两种乳杆菌均为开菲尔粒和雪莲菌粒中的优势菌株[13],根据文献报道[14]二者区别主要在于以下3点:(1)存在位置不同:L.kefir在菌粒表层,而L.kefiranofaciens在开菲尔粒内部;(2)是否产多糖,L.kefir不产糖,L.kefiranofaciens产多糖;(3)代谢类型:L.kefir为同型发酵,L.kefiranofaciens异型发酵;(4)GC含量:L.kefir为34.3%~35.4%,L. kefiranofaciens 为37%~42%。但是,目前尚未有研究报道二者遗传距离的关系,因此,后续的多位点序列分型实验包含2株L.kefir菌株进行。
表3 分离菌株16S rRNA鉴定结果
Table 3 Identification of isolates from16S rRNA sequences
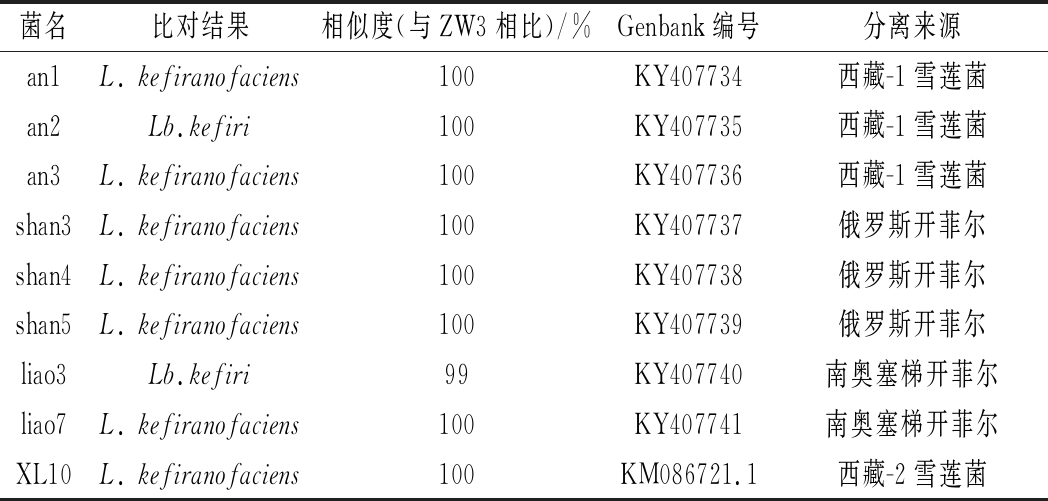
菌名比对结果相似度(与ZW3相比)/%Genbank编号分离来源an1L. kefiranofaciens100KY407734西藏-1雪莲菌an2Lb.kefiri100KY407735西藏-1雪莲菌an3L. kefiranofaciens100KY407736西藏-1雪莲菌shan3L. kefiranofaciens100KY407737俄罗斯开菲尔shan4L. kefiranofaciens100KY407738俄罗斯开菲尔shan5L. kefiranofaciens100KY407739俄罗斯开菲尔liao3Lb.kefiri99KY407740南奥塞梯开菲尔liao7L. kefiranofaciens100KY407741南奥塞梯开菲尔XL10L. kefiranofaciens100KM086721.1西藏-2雪莲菌
2.2 PCR扩增MLST片段
将马乳酒样乳杆菌ZW3与同种或近源的9株乳杆菌株一同进行总基因组提取,以提取的10株细菌的基因组为模板,利用设计好的引物扩增7个目的基因(rpoA、Hsp60、LeuS、rpsB、glyK、pyrG、fusA),得到与预期大小相一致的单一条带,证明7个目的基因均成功扩增(如图1)。
2.3 菌株ST分型结果
8株马乳酒样乳杆菌和2株开菲尔乳杆菌的rpoA、Hsp60、LeuS、rpsB、glyK、pyrG、fusA基因扩增产物经过双向测序后,由于测序结果中的前后20~50 bp的序列可信度较低,故利用Chromas软件检查峰图,依据测序峰图进行人工校对、剪切,并且对序列中的疑似SNP位点进行确认,经过人工校对,利用DNAMAN 6.0进行正向、反向序列拼接,得到准确的有效序列。最终用于MLST分析的序列长度分别为:rpoA:319 bp、Hsp 60:517 bp、LeuS:390 bp、rpsB:251 bp、glyK:361 bp、pyrG:378 bp、fusA:524 bp。在7个基因位点上,每个基因按照所在参考基因组的位置排列(rpoA-Hsp60-LeuS-rpsB-glyK-pyrG-fusA)等位基因数字,所组成的一个独特的等位基因模式即为一个ST型。最终将10株菌株共分为8个ST型,结果见表4。其中俄罗斯地区样品中分离的马乳酒样乳杆菌shan3、shan4、shan5同属一个ST型,其余菌株各不相同,说明不同分离地区的菌株间ST型大部分是不相同的,具有较高的遗传多样性。

1-an1; 2-an2; 3-an3; 4-shan3; 5-shan4; 6-shan5; 7-liao3; 8-liao7; 9-XL10; 10-ZW3; M:Trans 2K PlusII DNA marker
a-rpoA;b-Hsp60; c-LeuS; d-rpsB; e-glyK; f-pyrG; g-fusA
图1 七个目的基因PCR扩增凝胶电泳图
Fig.1 Agarose gel electrophoresis of amplification of 7 target genes
由表4可知,an2与an3的ST型只有LeuS和glyK上等位基因编号不同,说明二者相关性较近;an3与ZW3的ST型只有Hsp60和glyK上等位基因编号不同,说明此二者相关性较近;shan3、shan4、shan5同属的ST-4型与liao7所属的ST-6型只有LeuS上等位基因编号不同,说明它们相关性较近。
表4 菌株的ST型及对应的等位基因编号
Table 4 Alleles profiles and ST designation of strains
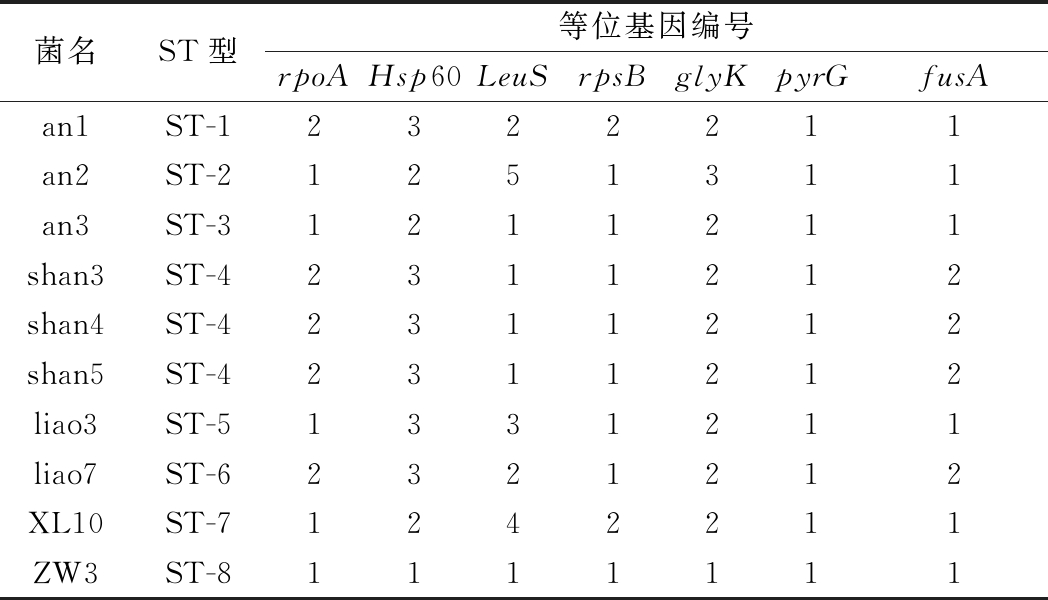
菌名ST型等位基因编号rpoAHsp60LeuSrpsBglyKpyrGfusAan1ST-12322211an2ST-21251311an3ST-31211211shan3ST-42311212shan4ST-42311212shan5ST-42311212liao3ST-51331211liao7ST-62321212XL10ST-71242211ZW3ST-81111111
从结果中还可以看出,不同管家基因中等位基因编号的个数不同,其中等位基因最多的为LeuS(5个)基因位点,其次为Hsp60和glyK(3个),说明三者对于菌株多态性的分辨力较强。等位基因数最少的为pyrG,说明其基因最为稳定,变化最小。同时,ZW3序列型为独有的ST8,上述结果也证明了利用MLST方法研究马乳酒样乳杆菌的菌株分类比传统的16S rRNA鉴定方法更准确、分辨率更高,可以作为快速识别ZW3菌株特异性的一种较为准确和简便的手段。
2.4 等位基因多样性分析
通过START2.0软件对7个管家基因的等位基因的多态性位点数、GC含量和dN/dS比率进行计算,结果如表5所示。从结果中可以看出LeuS的SNP数目最多,达到327个,其次为glyK(10个)。7个管家基因的GC含量在38.96%~45%,而参考基因组马乳酒样乳杆菌ZW3的染色体基因组GC含量为37.70%[2],低于7个管家基因的GC含量,较高的GC含量表明该基因可能处于不断进化的时期[15]。
氨基酸编码区的核苷酸同义替换的平均速率表示为dS;核苷酸的非同义替换的平均速率表示为![]() 当基因的非同义替换速率大于同义替换速率,可以反映该基因正在发生或者已经发生了大的变化,所以dN/dS值的统计是分子进化研究的重要内容[17]。由表5可知7个基因(pyrG无SNP位点)的潜在非同义位点上的非同义替换速率(dN)在0(rpoA)~0.972 3(LeuS);而潜在同义位点上的同义替换速率(dS)在0(rpsB、fusA)~1.421 1(LeuS);dN/dS值在0(rpoA)~0.684 2(LeuS),说明所有管家基因的非同义替换速率均小于同义替换速率。结果表明这些基因并没有受到外界明显的选择压力影响,位点的突变为中性替代速率,或受到净化选择压力[18]。
当基因的非同义替换速率大于同义替换速率,可以反映该基因正在发生或者已经发生了大的变化,所以dN/dS值的统计是分子进化研究的重要内容[17]。由表5可知7个基因(pyrG无SNP位点)的潜在非同义位点上的非同义替换速率(dN)在0(rpoA)~0.972 3(LeuS);而潜在同义位点上的同义替换速率(dS)在0(rpsB、fusA)~1.421 1(LeuS);dN/dS值在0(rpoA)~0.684 2(LeuS),说明所有管家基因的非同义替换速率均小于同义替换速率。结果表明这些基因并没有受到外界明显的选择压力影响,位点的突变为中性替代速率,或受到净化选择压力[18]。
表5 七个管家基因的等位基因多样性分析
Table 5 Alleles variation in 7 housekeeping genes
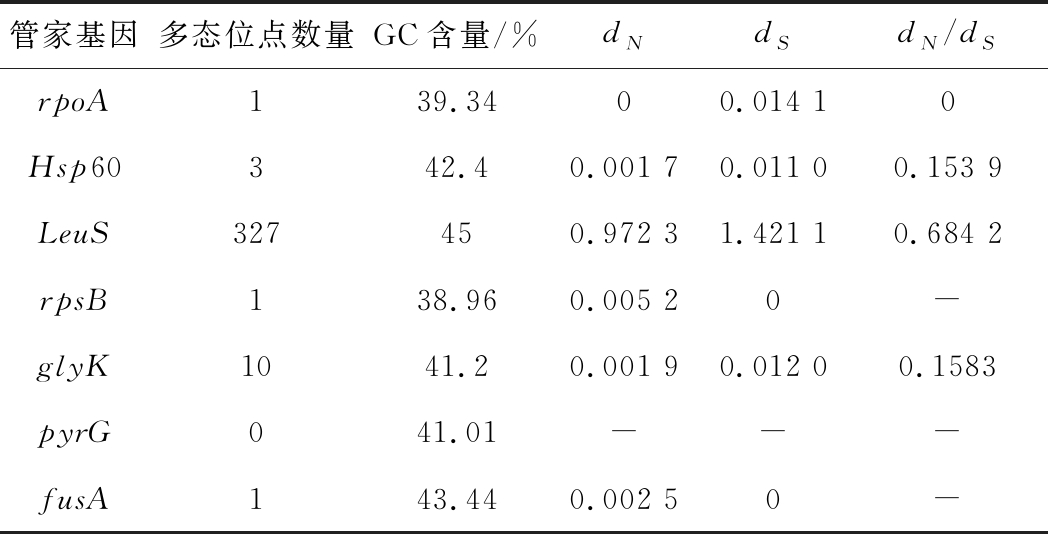
管家基因多态位点数量GC含量/%dNdSdN/dSrpoA139.3400.014 10Hsp60342.40.001 70.011 00.153 9LeuS327450.972 31.421 10.684 2rpsB138.960.005 20-glyK1041.20.001 90.012 00.1583 pyrG041.01---fusA143.440.002 50-
注:“-”表示无。
2.5 等位基因的连锁平衡分析
采用在线分析工具LIAN 3.7对等位基因的ST型进行分析,Nei′s遗传多样性指数(H)是衡量生物遗传多样性的参数[19],数值为0~1,数值越大说明其变异度越大[20]。结果显示7个管家基因位点的平均遗传多态性(H)为0.488 7±0.107 0。7个管家基因(rpoA、Hsp60、LeuS、rpsB、glyK、pyrG、fusA)的遗传多态性(H)分别为:0.555 6、0.6、0.755 6、0.355 6、0.621 1、0、0.533 3。本研究中LeuS和glyK位点变异度较大,pyrG位点变异度最小。
标准关联指数(ISA)通常被用来衡量一组序列的发生重组的数量和检测等位基因之间的相互关联,是分析连锁不平衡的重要参数。当重组事件发生时,ISA越趋近于0,相反,如果存在克隆结构,那ISA显著不等于0,存在连锁不平衡现象[21]。本研究中ISA值为0.105 2,说明存在连锁不平衡现象,每次重组事件不是在所有位点上同时发生的。
2.6 等位基因的重组分析
有关生物合成的管家基因发生的变异通常为中性选择,同源重组分析可以帮助我们了解其发生遗传变异的证据[22]。等位基因的重组通常使用网络结构来进行分析,当一个图谱为克隆结构,会形成树状结构,而明显的网络形状表明了同种菌中不同菌株发生基因重组交换[23]。本研究采用SplitsTree4.0工具对7个等位基因的ST型的数据进行分解分析,同时对于Hsp60和LeuS两个位点上等位基因的重组情况进行分析,如图2所示。从单个位点的分解分析结果中可以看出,Hsp60位点在进化过程中无明显重组现象,LeuS有轻微的重组现象。不同ST型分解分析图中显示, ST1、ST6、ST73个基因型有轻微重组现象,对应菌株分别为an1、liao7、XL10,此3株菌的分离来源分别为西藏-1、南奥塞梯、西藏-2,表明不同地区的马乳酒样乳杆菌存在一定的重组现象,这可能与环境适应有关。由图2-C可知,不同的ST型主要分为3个类群,ST2和ST5一组,ST3、ST4和ST8一组,这两组均为树状结构,说明存在克隆结构。
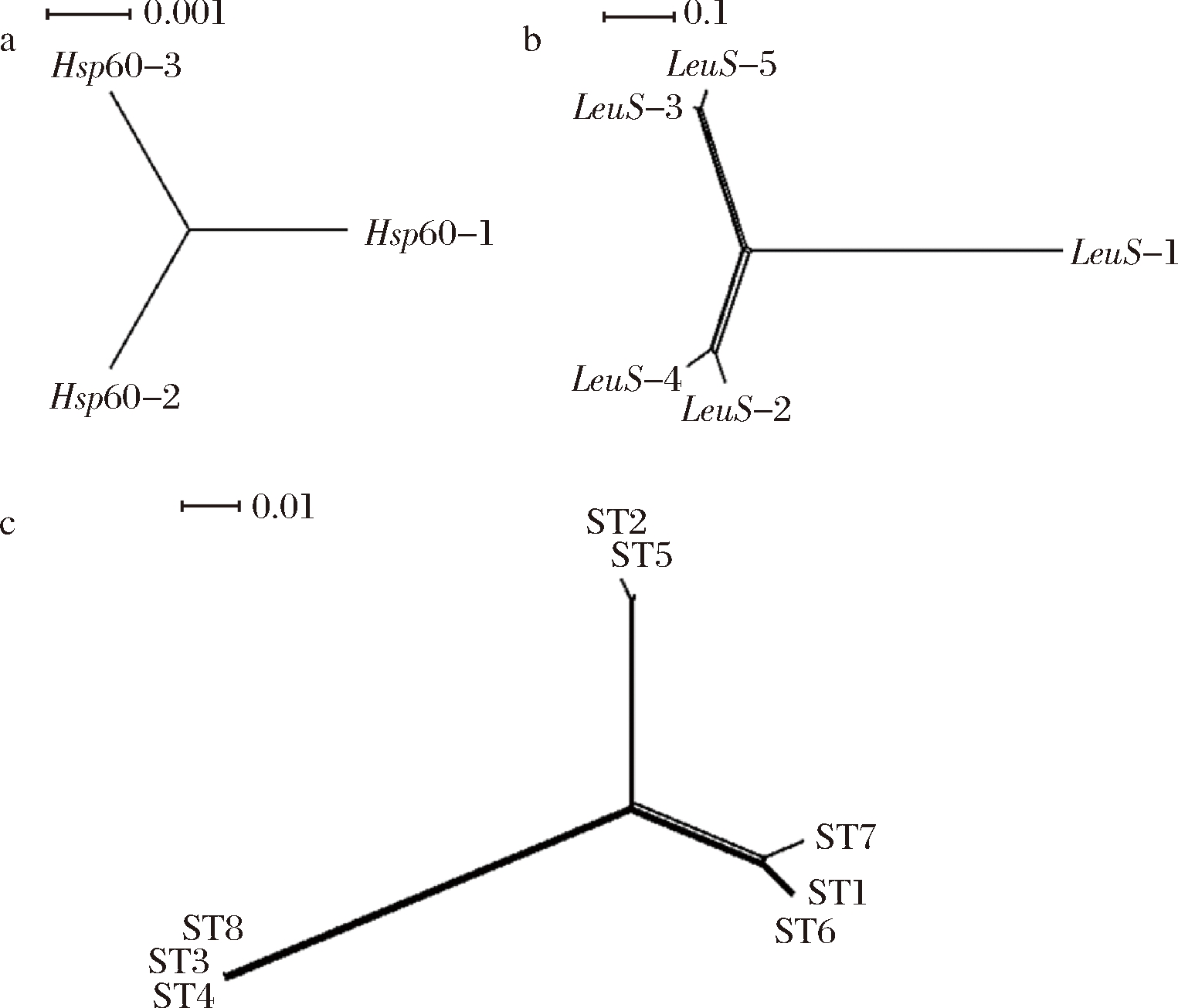
a-Hsp60; b-LeuS ; c-STs
图2 重组分解分析图
Fig.2 Split decomposition analysis
2.7 不同ST型的eBURST分析
eBURST是一种用来研究不同ST型进化关系的一种简单模型,它通常模拟归为一个型组的情形是指:同一祖先的基因型随着种群数目频繁的增加,形成相近的一系列基因型[24]。本研究对8个ST型序列进行eBURST分析,结果如图3所示,分为2个型组,其中型组1中包括ST2、ST3和ST8;型组2中包括ST4和ST6;其他ST1、ST7、ST5为独特型,即为独有的序列型。上述结果说明,ST2、ST3和ST8(对应菌株:an2、an3和ZW3)可能拥有共同的祖先;ST4和ST6(对应菌株:shan3、shan4、shan5和liao7)可能拥有共同的祖先。虽然菌株ZW3与an2和an3分离自不同地区的开菲尔粒和雪莲菌粒,但它们可能拥有共同的起源。
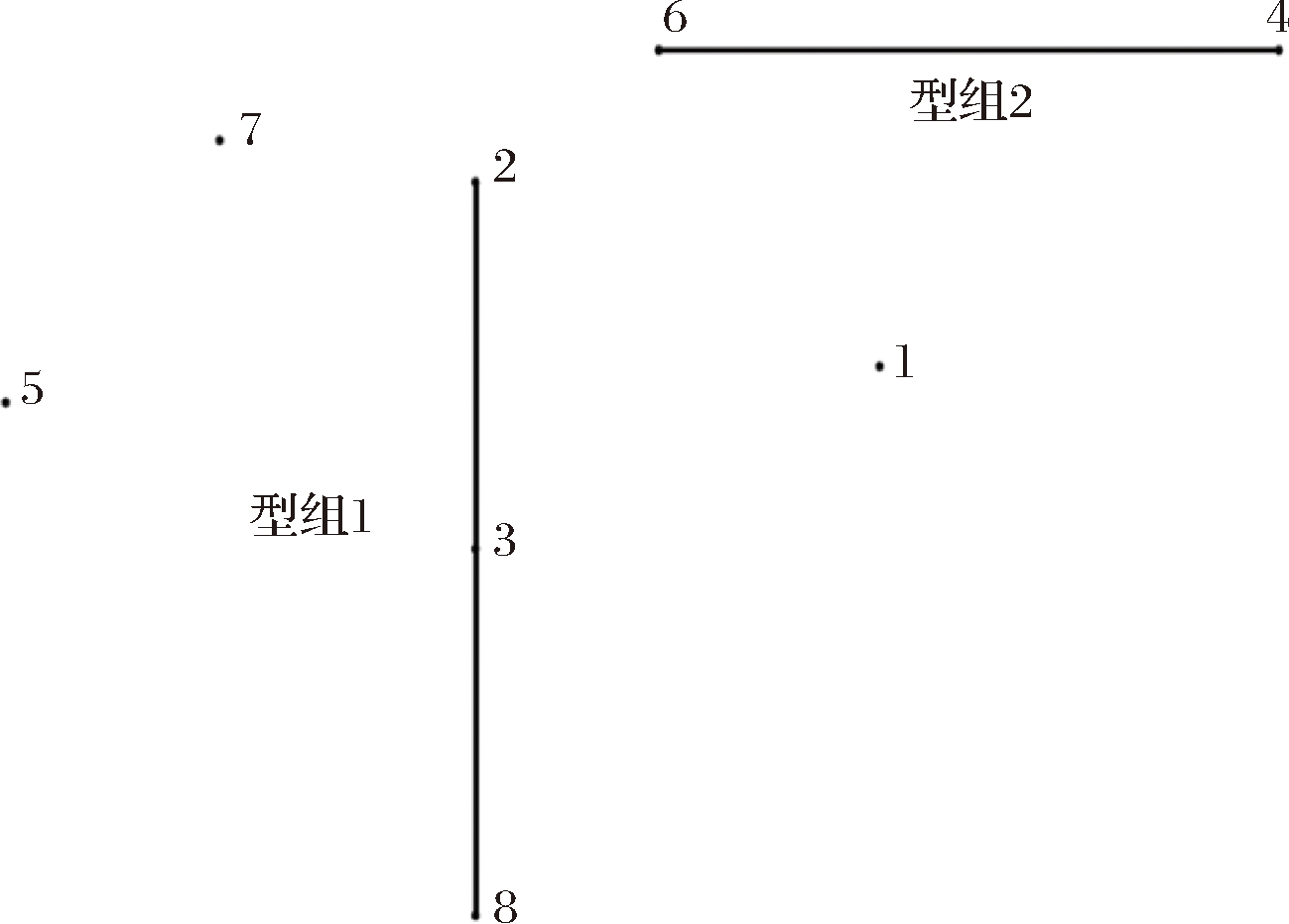
图3 eBURST分析图
Fig.3 eBURST analysis map
2.8 不同ST型和不同菌株的聚类分析
为了更好的研究ST型间以及菌株间的遗传关系,本研究分别采用START 2.0和MEGA 6.0中的非加权组平均聚类方法(unweighted pair-gruop method with arithmetic means,UPGMA)计算个体之间的遗传距离,并绘制遗传聚类图,结果如图4和图5。
遗传距离是研究物种基因多样性的基础,它反映了所研究群体的整体系统进化情况,用来描述群体的遗传结构和种间差异[25]。本研究中采用的UPGMA聚类分析法,为多序列位点分析中最为常用的一种方法[26],它计算简单,结果清晰,通常分支的长短代表了遗传距离的远近,而遗传距离较近的个体聚为一簇,可以直观解读遗传进化关系。由图4可以看出ST型主要分为两簇,一簇为ST1、ST2、ST5、ST6、ST7;另一簇为ST3、ST4、ST8,这与图5中分离菌株的UPGMA聚类分析结果相吻合。其中,ST1和ST6的遗传距离较短,ST2与ST5的遗传距离较短。菌株an2序列型为ST2,菌株liao3序列型为ST5,二者遗传距离较短,与表3中16S rRNA的序列鉴定结果均为开菲尔乳杆菌的结果相呼应。XL10菌株对应序列型为ST7,在聚类中与另外两个分支均有一定的距离,说明它的分化时间较长。如图5所示,虽然ZW3与an3、shan3、shan4和shan5的分离地点不同,但它们之间的遗传距离很近,说明它们的分化时间较短。由图5可知,西藏-1、西藏-2和南奥塞梯这3个地点分离的菌株遗传距离较近,俄罗斯和高加索地区分离菌株归为另一簇,这可能是由于居住在这些地区的人群主要是游牧民族,开菲尔粒和雪莲菌粒随着他们不断迁徙带到各地,随着环境的适应,基因不断进化,所以可能造成了这种遗传上的区域差异。

图4 ST型的UPGMA聚类图
Fig.4 The UPGMA dendrogram of the STs
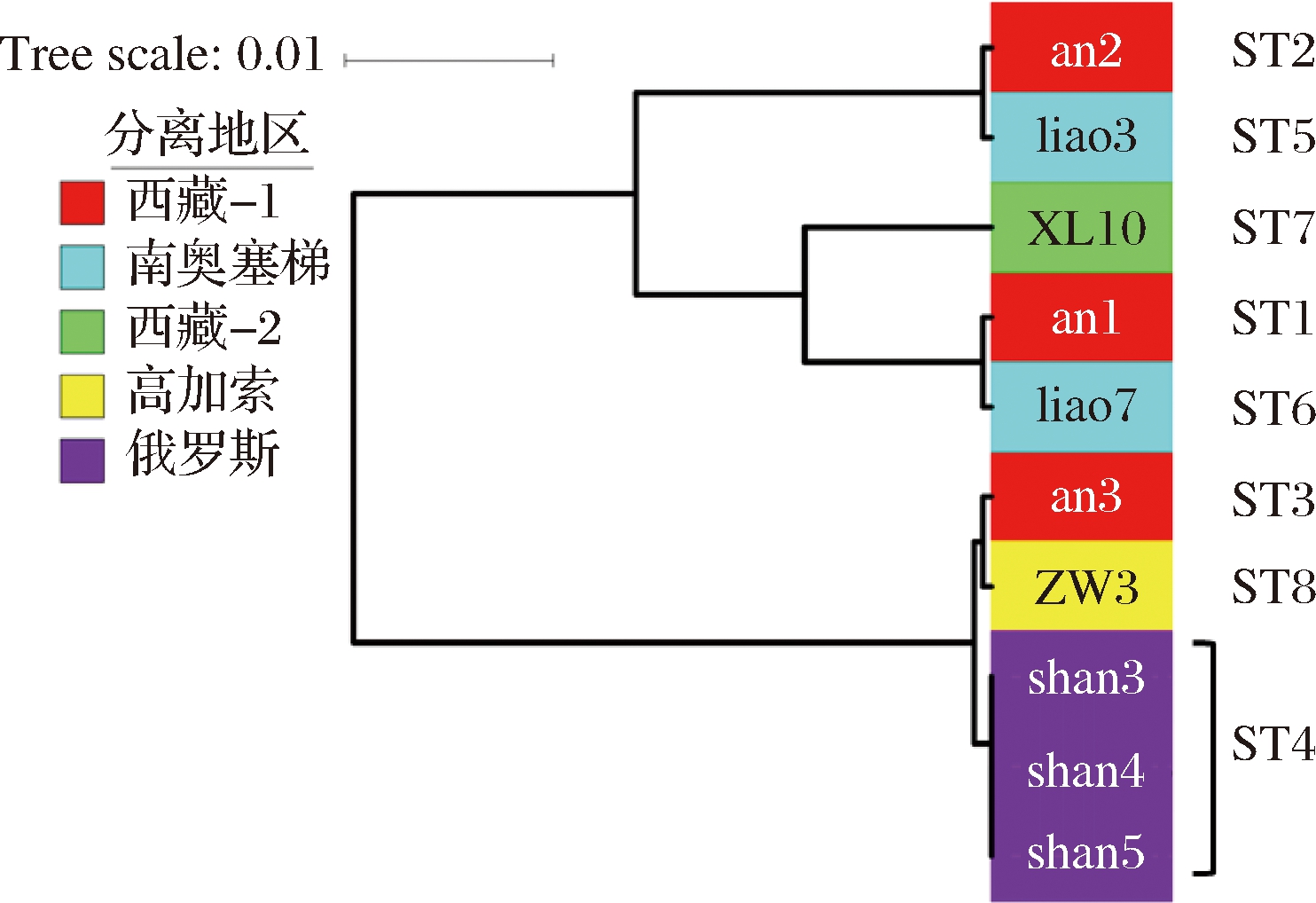
图5 分离菌株7个基因位点的UPGMA聚类图
Fig.5 The UPGMA dendrogram of all the isolates for 7 genes
3 讨论
管家基因通常主要编码代谢功能相关蛋白,普遍存在于每种微生物中,随着物种的不断进化、演变,它们以较慢的速度进化[27],多位点序列分型方法的关键是管家基因的选择,其次为基因数量,选择位点数量增加可以提高分辨率,但过多只能浪费人力物力[28],一般多位点序列分型的片段长度在400~700 bp[21],因此本研究中筛选出在乳杆菌中具有较好保守性、在不同种间又具有差异性的7个管家基因,涵盖了较全面的遗传信息,目的基因片段的扩增长度在374~675 bp,符合多位点序列分型实验设计要求。
多位点序列分型技术可以通过比较ST型发现菌株之间的相关性,例如密切相关的菌株具有相同的ST型或者仅有个别的等位基因编号不同;而不相关菌株的ST型至少有3个或3个以上的等位基因编号不同[29]。an2与an3相关性较近;an3与ZW3相关性较近;shan3、shan4、shan5同属的ST-4型与liao7所属的ST-6型相关性较近。另外,等位基因数与多态性位点的多少可以用来评判管家基因对菌株多态性的分辨能力[30]。LeuS、Hsp60和glyK,对于菌株多态性的分辨力较强;pyrG的基因,最为稳定,变化最小。同时,ZW3序列型为独有的ST8,上述结果也证明了利用MLST方法研究马乳酒样乳杆菌的菌株分类比传统的16S rRNA鉴定方法更准确、分辨率更高,可以作为快速识别ZW3菌株特异性的一种较为准确和简便手段。
通过一系列生物信息学软件对等位基因和遗传多态性进行分析,发现等位基因存在连锁不平衡现象,发生过轻微的基因重组现象,发生的变异通常为中性选择。eBURST分析显示an2、an3和ZW3归为同一型组,可能拥有共同的祖先。对不同的ST型和分离菌株分别进行UPGMA遗传聚类分析,发现二者结果一致,ZW3与an3、shan3、shan4和shan5之间的遗传距离很近,与另外5株菌遗传距离较远,分别属于两个群簇。
[1] 吴蜀豫,冉陆.FAO/WHO《食品益生菌评价指南》[J].中国食品卫生杂志,2003,15(4):377-379.
[2] WANG Y, WANG J, AHMED Z,et al.Complete genome sequence of Lactobacillus kefiranofaciens ZW3[J].J Bacteriol. 2011,193(16):4 280-4 281.
[3] 王艳萍,李超, ZAHEER AGMED.一株马乳酒样乳杆菌胞外多糖的理化性质[J].食品与发酵工业,2011,37(9):59-63.
[4] 郑永娜. 马奶酒样乳杆菌ZW3降解亚硝酸盐功能的研究与应用[D].天津:天津科技大学,2016.
[5] XING Z, TANG W, YANG Y, et al.Colonization and gut flora modulation of Lactobacillus kefiranofaciens ZW3 in the intestinal tract of mice[J].Probiotics Antimicrob Proteins,2018,10(2):374-382.
[6] XING Z, GENG W, LI C, et al.Comparative genomics of Lactobacillus kefiranofaciens ZW3 and related members of Lactobacillus. spp reveal adaptations to dairy and gut environments[J].Sci Rep,2017,7(1):12 827.
[7] SUN Y, GENG W, PAN Y, et al.Supplementation with Lactobacillus kefiranofaciens ZW3 from Tibetan Kefir improves depression-like behavior in stressed mice by modulating the gut microbiota[J].Food Funct,2019,10(2):925-937.
[8] JOLLEY K A, FEIL E J, CHAN M S, et al. Sequence type analysis and recombinational tests (START)[J]. Bioinformatics, 2001,17(12):1 230-1 231.
[9] HAUBOLD B, HUDSON R R. LIAN 3.0: Detecting linkage disequilibrium in multilocus data[J]. Bioinformatics, 2000,16(9):847-849.
[10] HUSON D H, BRYANT D. Application of phylogenetic networks in evolutionary studies[J]. Molecular Biology and Evolution, 2006,23(2):254-267.
[11] SPRATT B G, HANAGE W P, LI B, et al. Displaying the relatedness among isolates of bacterial species - the eBURST approach[J]. Fems Microbiology Letters, 2004,241(2):129-134.
[12] LETUNIC I, BORK P. Interactive tree of life (iTOL) v3: An online tool for the display and annotation of phylogenetic and other trees[J]. Nucleic Acids Research, 2016,44(W1):W242-W245.
[13] LEITE A M, MAYO B, RACHID C T, et al.Assessment of the microbial diversity of Brazilian kefir grains by PCR-DGGE and pyrosequencing analysis[J]. Food Microbiology, 2012,31(2):215-221.
[14] ARIHARA K, TOBA T, ADACHI S. Immunofluorescence microscopic studies on distribution of Lactobacillus kefiranofaciens and Lactobacillus kefir in kefir grains[J]. International Journal of Food Microbiology, 1990,11(2):127-134.
[15] O′SULLIVAN O, O′CALLAGHAN J, SANGRADOR-VEGAS A, et al. Comparative genomics of lactic acid bacteria reveals a niche-specific gene set[J]. Bmc Microbiology, 2009,9:50.
[16] 李易. 基因进化的同义与非同义替代计算及统计检验的比较分析[J]. 曲靖师范学院学报, 2006,25(6):1-8.
[17] NEI M, KUMAR S. Molecular evolution and phylogenetics[J]. Heredity, 2001,31(86):385.
[18] DOBZHANSKY T. Genetics of the evolutionary process[J]. Journal of Medical Genetics, 1971,8(4):545-546.
[19] GAO B J, GAO L J, HOU J H. Genetic diversity of Dendrolimus(Lepidoptera) population from different geographic area[J]. Acta Ecologica Sinica, 2008,28(2):842-848.
[20] 于洁. 中国、俄罗斯和蒙古国地区传统发酵乳制品中嗜热链球菌的多位点序列分型研究[D]. 呼和浩特:内蒙古农业大学, 2013.
[21] SMITH j M, SMITH N H, O′ROURKE M,et al. How clonal are bacteria[J].Processing of the National Acdemy of Science,1993,90(10):4 384-4 388.
[22] DE LAS RIVAS B, MARCOBAL A, MUNOZ R. Development of a multilocus sequence typing method for analysis of Lactobacillus plantarum strains[J]. Microbiology-Sgm, 2005,152(Pt1):85-93.
[23] FEIL E J, ENRIGHT M C, SPRATT B G. Estimating the relative contributions of mutation and recombination to clonal diversification: A comparison between Neisseria meningitidis and Streptococcus pneumoniae[J]. Research in Microbiology, 2000,151(6):465-469.
[24] FEIL E J, LI B C, AANENSEN D M, et al. eBURST: Inferring patterns of evolutionary descent among clusters of related bacterial genotypes from multilocus sequence typing data[J]. Journal of Bacteriology, 2004,186(5):1 518-1 530.
[25] 武艳平. 中国部分山羊品种多样性及系统进化分析[D].北京:中国农业科学院, 2008.
[26] KORNIENKO M, ILINA E, LUBASOVSKAYA L, et al.Analysis of nosocomial Staphylococcus haemolyticus by MLST and MALDI-TOF mass spectrometry[J].Infect Genet Evol, 2016, 39:99-105.
[27] PRAKASH A, SHARMA C, SINGH A, et al.Evidence of genotypic diversity among Candida auris isolates by multilocus sequence typing, matrix-assisted laser desorption ionization time-of-flight mass spectrometry and amplified fragment length polymorphism[J].Clin Microbiol Infect, 2016, 22(3):277.
[28] OZAWA M, HIKI M, KAWANISHI M, et al.Molecular typing of fluoroquinolone-resistant Campylobacter jejuni isolated from Broilers in Japan using multilocus sequence typing and pulsed-field gel electrophoresis[J].Foodborne Pathog Dis, 2016, 13(1):1-7.
[29] MUND N L A, MASANTA W O, GOLDSCHMIDT A M, et al.Association of Campylobacter jejuni ssp. jejuni chemotaxis receptor genes with multilocus sequence types and source of isolation[J].Eur J Microbiol Immunol, 2016, 6(3):162-177.
[30] 陈鲜宝,金向红,邵先兵,等. 138株临床分离肺炎克雷伯菌耐药性与多位点序列分型研究[J]. 全科医学临床与教育, 2015, 13(5):529-532.