玉米皮是玉米深加工的主要副产物之一,占玉米籽粒质量的15%左右[1],玉米皮中半纤维素含量可达到68%[2],与其他植物来源的半纤维素相比,其具有支链化程度高的特点,其主链的每一个木聚糖残基几乎都连接有支链残基[3]。玉米皮纤维的生物降解涉及到多种酶的协同作用,包括木聚糖的主链降解酶——木聚糖酶(xylanase)、木糖苷酶(xylosidase)以及多种支链降解酶[4]。目前,玉米皮多作为廉价的饲料出售,为提高其利用价值,已有应用玉米皮发酵培养黑曲霉[5]、酵母菌发酵玉米皮制备菌体蛋白[6]、玉米皮发酵培养琥珀酸放线杆菌产丁二酸[7]等方面研究。
裂褶菌(Schizophyllum commune)具有较强的半纤维素和纤维素的分解能力,能够在含有半纤维素的基质上良好地生长[8]。本课题组在前期研究中,已经测定了玉米皮纤维发酵培养裂褶菌的产酶情况,包括木聚糖酶、乙酰木聚糖酯酶、α-L-阿拉伯呋喃糖苷酶和α-葡萄糖醛酸酶[9];同时完成了1个木聚糖酶基因的克隆和异源表达[9]。但与玉米皮木聚糖连接的阿拉伯糖支链抑制了木聚糖水解酶的活性,而α-L-阿拉伯呋喃糖苷酶(α-L-arabinofuranosidase, EC 3.2.1.55)能从含有阿拉伯糖残基的多聚物的非还原末端水解1个阿拉伯糖分子,该酶同木聚糖酶复合后具有协同作用,能够促进玉米皮木聚糖降解。因此,本研究设计克隆裂褶菌α-L-阿拉伯呋喃糖苷酶基因,并完成其在毕赤酵母中的异源表达。
真菌转录组的测序分析具有速度快、信息全面的优点,能够全面分析特定培养条件下基因转录的情况[10]。例如,对灰树花的菌丝体进行转录组测序分析,获得了该菌株生长过程中参与多糖合成的基因的表达情况[11]。与真菌的全基因组测序分析相比,转录组的测序结果中不含内含子及其他非编码的核酸序列,可以更高效地分析与培养条件相关的基因转录情况。
本研究是以玉米皮纤维为唯一碳源发酵培养裂褶菌为样本,通过转录组测序技术分析与玉米皮纤维降解相关基因的转录情况;同时克隆了裂褶菌糖苷水解酶62家族蛋白基因Sabf32,并完成该基因在毕赤酵母中的表达,研究为木聚糖酶与α-L-阿拉伯呋喃糖苷酶协同降解玉米皮纤维提供了基础,同时为玉米皮的增值利用提供了理论基础。
1 材料和方法
1.1 材料
1.1.1 菌株
裂褶菌S. commune DB01,本实验室筛选并保存。
1.1.2 主要试剂
D-木糖、桦木木聚糖,Megazyme公司;PDA培养基,北京陆桥技术股份有限公司;3,5二硝基水杨酸、酒石酸钾钠、NaOH,国药集团化学试剂有限公司;蛋白质浓度测定试剂盒,北京索莱宝科技有限公司;对硝基苯阿拉伯呋喃糖苷(4-nitrophenyl α-L-arabinofuranoside,pNPAF),Sigma公司。
1.1.3 主要仪器
DK-8D电热恒温水槽,上海精宏实验设备有限公司;PL2002IC电子天平,Mettler公司;MQD-SIR单层小容量振荡培养箱,上海旻泉仪器有限公司;SynergvHT酶标仪,美国BioTek公司;ND OneC NanoDropTM微量紫外-可见光分光光度计,Thermo Fisher公司;电泳仪、凝胶成像仪,美国Bio-Rad公司。
1.2 实验方法
1.2.1 培养条件
玉米皮纤维诱导培养基成分[12](g/L):蛋白胨1、尿素0.2、CaCl2 0.3、MgSO4·H2O 0.3、KH2PO4 2、(NH4)2SO4 4.2、0.2%(v/v)微量元素(CoCl2 1.6、MnSO4·4H2O 1.6、ZnSO4·7H2O 1.4、FeSO4·7H2O 5),玉米皮纤维10,作为碳源。
菌株经PDA平板活化后,将裂褶菌接种于300 mL玉米皮纤维培养基中,30 ℃、220 r/min培养120 h。在培养过程中,取发酵液作为检测样品,取样过程中应尽量避免振荡,将发酵液于12 000×g离心5 min后取上清液待测。
1.2.2 发酵液中蛋白质、还原糖和酶活力测定
发酵液还原糖含量测定:将100 μL上清液加水稀释至200 μL,加入300 μL DNS试剂,混合后置于沸水浴5 min,迅速冷却后在540 nm波长下测定吸光值。
木聚糖酶活性测定[13],反应体系为:100 μL适量稀释的发酵液上清、100 μL底物(10 g/L,桦木木聚糖),在40 ℃保温10 min,然后加入300 μL试剂终止反应,沸水煮5 min并冷却,在540 nm波长下测定吸光值。
蛋白质浓度测定:根据蛋白质浓度测定试剂盒说明书测定。
1.2.3 裂褶菌总RNA提取及测序
菌株经PDA平板活化后,接种玉米皮纤维培养基,于30 ℃,220 r/min条件下培养48 h。抽滤液体培养基得到菌丝体,称取0.1~0.3 g菌丝体于研钵中,加入液氮,并在液氮保护下快速研磨,样品研磨至无颗粒呈粉状,将研磨好的粉末移入装有Trizol的离心管中。用Trizol法提取裂褶菌总RNA,并用琼脂糖凝胶电泳分析RNA的完整性。应用微量紫外-可见光分光光度计测定样品浓度及纯度(A260/A280 nm及A260/A230 nm)。
1.2.4 数据分析及注释分类
Illumina HiSeq测序后对原始序列进行过滤(测序工作由安诺优达基因科技有限公司完成),用组装软件Trinity(20140717版)[14]组装出全长转录本序列,将每个基因中最长的转录本作为Unigene。在NCBI数据库(https://www.ncbi.nlm.nih.gov/)对Unigene进行NR(non-redundant protein sequence database;non-redundant protein sequences from GenPept, Swissprot, PIR, PDF, PDB, and NCBI RefSeq)、NT(nucleotide sequence database;all traditional divisions of GenBank, EMBL, and DDBJ excluding bulk divisions)数据库的物种分布分析[16],同时进行BLASTP和BLASTX分析、统计、排序,确定数目前10的物种种类。其他能比对上的物种总称为Others。
Unigene的GO注释[17]是以基因NR注释信息为基础,获得每个Unigene的注释信息。对Unigene进行功能分类统计(WEGO),从生物过程(biological process)、细胞组成(cellular component)、分子功能(molecular function)3方面对裂褶菌Unigene的功能分布进行统计,并分析其分布特征。
1.2.5 α-L-阿拉伯呋喃糖苷酶基因的克隆
根据转录组测序结果,获得α-L-阿拉伯呋喃糖苷酶基因Sabf32的序列信息,应用SignalP 4.0(http://www.cbs.dtu.dk/services/SignalP-4.0/)预测Sabf32的信号肽序列,设计毕赤酵母表达引物:EcoRIsAfF:5′-CCG GAATTC CAGGCGTGCGACCTCCCGT-3′;NotIsAfR:5′-ATAGTTTA GCGGCCGC AGCCTGGGTAAGCAAGCCGGG-3′,引物由生工生物工程(上海)股份有限公司合成。
应用限制性内切酶:EcoR I和Not I分别将质粒pPIC9K和Sabf32进行双酶切;将酶切产物纯化后,应用T4 DNA连接酶构建重组表达质粒pPIC9K-Sabf32。应用PCR扩增验证阳性转化子,进一步通过测序验证核酸序列。
1.2.6 α-L-阿拉伯呋喃糖苷酶的毕赤酵母表达
应用电击转化将重组表达质粒pPIC9K-Sabf32和pPIC9K空载体转化毕赤酵母GS115感受态细胞,筛选获得阳性重组子。应用BMGY对重组子进行扩大培养,离心收集菌体后用BMMY培养基进行诱导培养;取诱导培养7 d的发酵液,7 000×g离心,取上清液进行SDS-PAGE电泳检测(转化、重组子筛选及发酵条件参照Invitrogen公司的毕赤酵母菌实验操作手册)。应用阴离子交换树脂纯化重组蛋白,并进行SDS-PAGE检测和分析重组蛋白的分子量。
1.2.7 重组酶Sabf32的酶活力测定
酶活力测定体系:25 μL 4 mmol/L pNPAF溶液,15 μL 0.1 mol/L pH 6.0 柠檬酸缓冲液混匀,加入10 μL适当稀释酶液。反应条件:将反应体系在40 ℃反应10 min,迅速加入150 μL 1 mol/L Na2CO3终止反应,于405 nm处测定OD值。将经过10 min沸水浴灭活的酶液做空白对照。用对硝基苯酚制作标准曲线,计算酶解反应体系中产生的对硝基苯酚含量。酶活的定义:每分钟产生1 μmol/L的对硝基苯酚所需的酶量为一个酶活力单位。
1.2.8 重组Sabf32的酶学性质研究
酶的最适温度及温度稳定性:在pH 6.0的条件下,用适当稀释的酶液分别在30、35、40、45、50、55、60、65、70、75、80、85、90 ℃下测定酶活,反应10 min,以最高酶活力为100%,计算各个温度下的相对酶活力。温度稳定性是将适当稀释的酶液分别置于40、50、60 ℃下,保温60 min,测定残余酶活。
酶的最适pH值及pH值稳定性:分别配制pH 2.0~8.0含pNPAF的缓冲液,在最适温度下测定不同pH值下的相对酶活,确定该酶的最适pH反应条件。pH值稳定性的测定是将浓缩后的酶液在上述不同pH条件下处理30 min后,调节至最适pH下,测定其残余酶活。
1.2.8 比活力和动力学参数的测定
应用Lowry法测定纯化重组酶Sabf32的蛋白浓度;在最适反应条件下(pH 5.0,40 ℃)测定重组酶Sabf32的活性,计算Sabf32的比酶活力。
在最适反应条件下,测定Sabf32催化不同浓度的pNPAF(1~4 mmol/L)释放的产物量,利用米氏方程(Lineweaver-Burk)绘制曲线,求出Km值和Vmax。
2 结果与分析
2.1 发酵液中蛋白质、还原糖和酶活力测定
将裂褶菌在以玉米皮纤维为唯一碳源的培养基中发酵培养,120 h发酵培养结果显示其生长良好,该菌株能够将玉米皮纤维转化为其菌体生长所必须的碳源。
通过测定发酵液中蛋白质浓度、还原糖含量和木聚糖酶酶活力,确定了裂褶菌转录组测序的取样条件。其中发酵液中还原糖含量在培养48 h后达到最高(图1),之后随着培养时间延长,浓度逐渐降低。推测可能是在菌种生长初期开始分泌胞外降解酶,水解玉米皮纤维,培养基中还原糖含量提高,但当菌体生长加快,菌株生长加速利用培养基中还原糖,因此含量开始逐渐降低。裂褶菌表达分泌胞外酶是其降解、利用玉米皮纤维的主要途径,当培养基上清液中还原糖含量快速上升并达到最高点时,推测其纤维降解酶也同时在进行高效地转录,因此选择培养48 h为RNA提取时间。当培养至72 h时,发酵液蛋白质含量、酶活力趋于稳定,还原糖含量逐步降低,推测裂褶已经开始利用培养基中已经降解的纤维维持生长,胞外酶的转录效率可能开始降低。发酵液中木聚糖酶酶活力随着培养时间延长逐渐提高(图2),但超过72 h后,酶活力增长趋于稳定。其在48~72 h间酶活力增长较快。发酵液中蛋白质浓度随着培养时间延长逐渐提高(图3),但超过60 h后,蛋白质浓度增长趋于稳定。
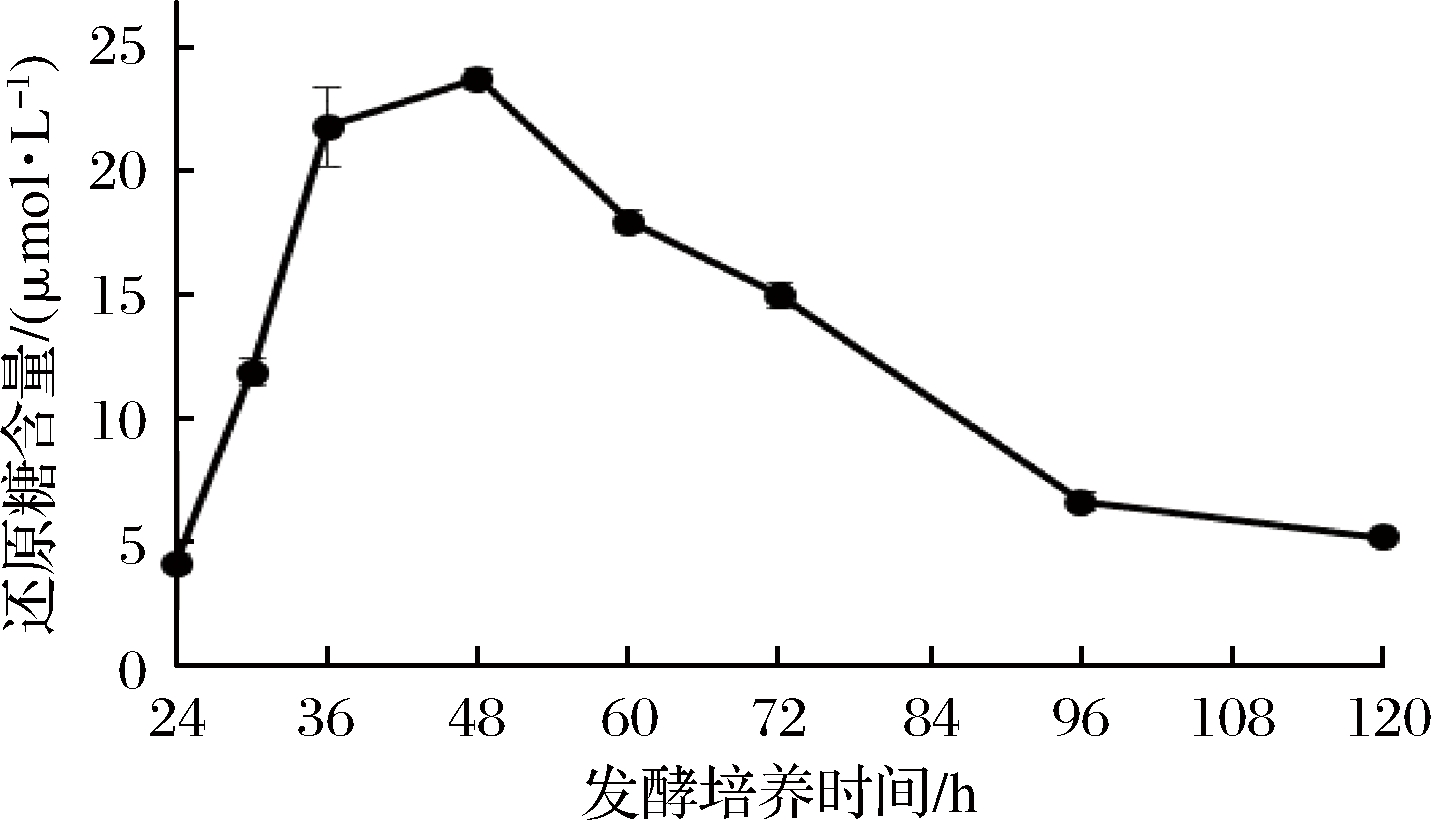
图1 发酵液中还原糖含量
Fig.1 Reducing sugar content in fermentation
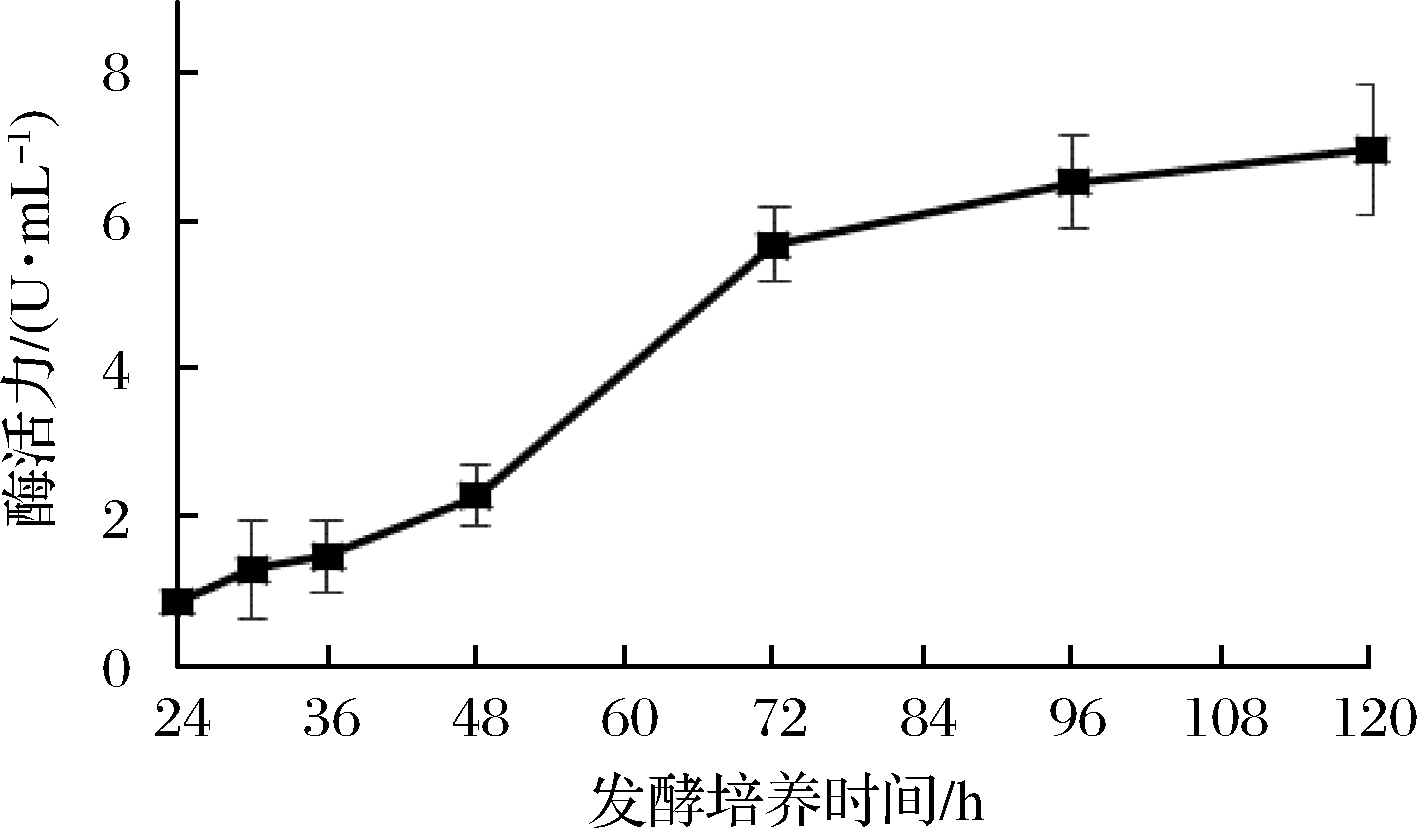
图2 发酵液中木聚糖酶酶活力
Fig.2 Activity of xylanase in fermentation
2.2 总RNA提取、测序及Unigene的组装
提取了发酵培养48 h时的裂褶菌总RNA,经过NanoDrop定量后,确定提取的总RNA浓度为395 ng/μL,260/280比值为2.07。提取的裂褶菌RNA完整,没有弥散(图4),28S∶18S的比值符合构建cDNA文库和转录组测序质量要求。
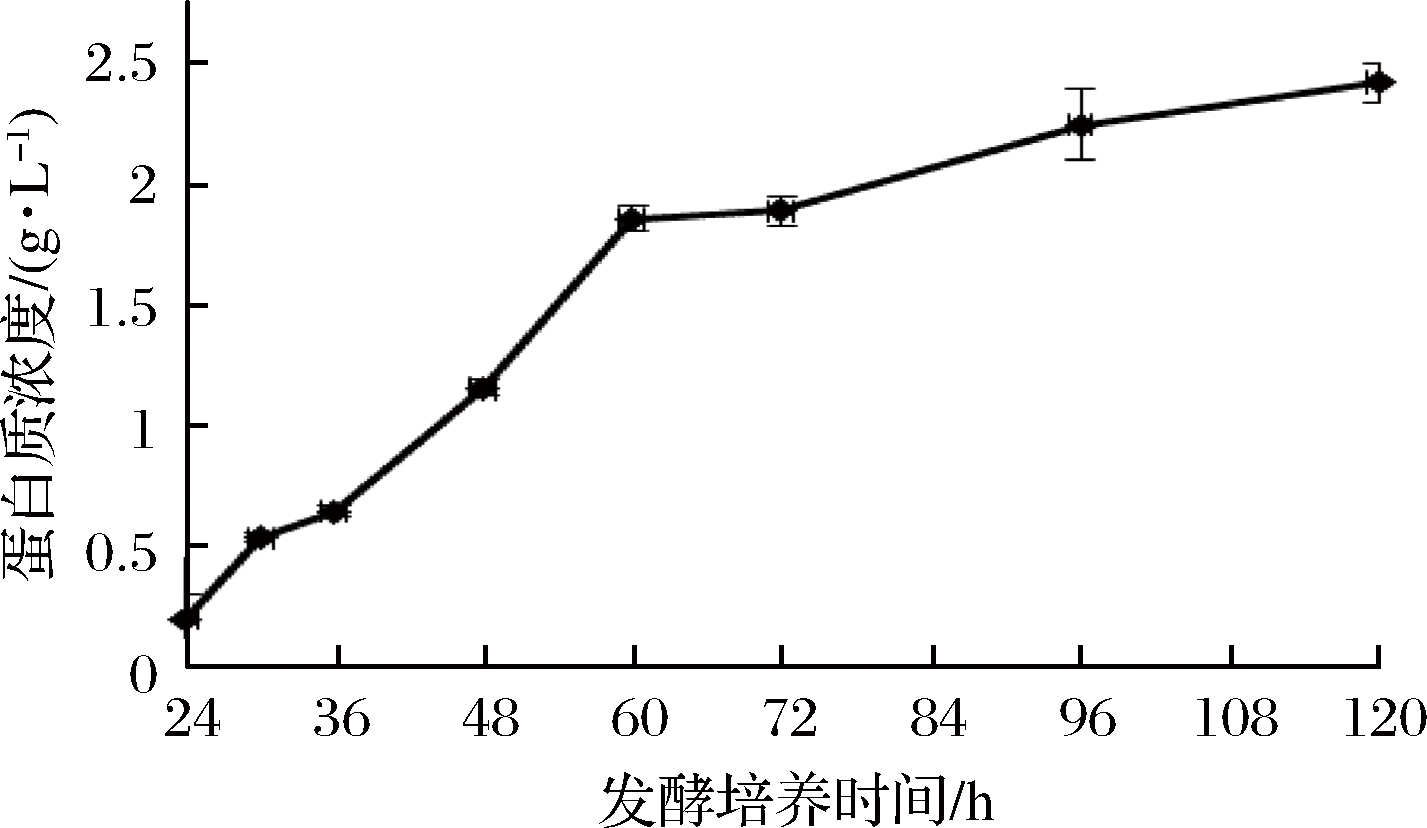
图3 发酵液中蛋白质浓度变化
Fig.3 Change of protein concentration in fermentation
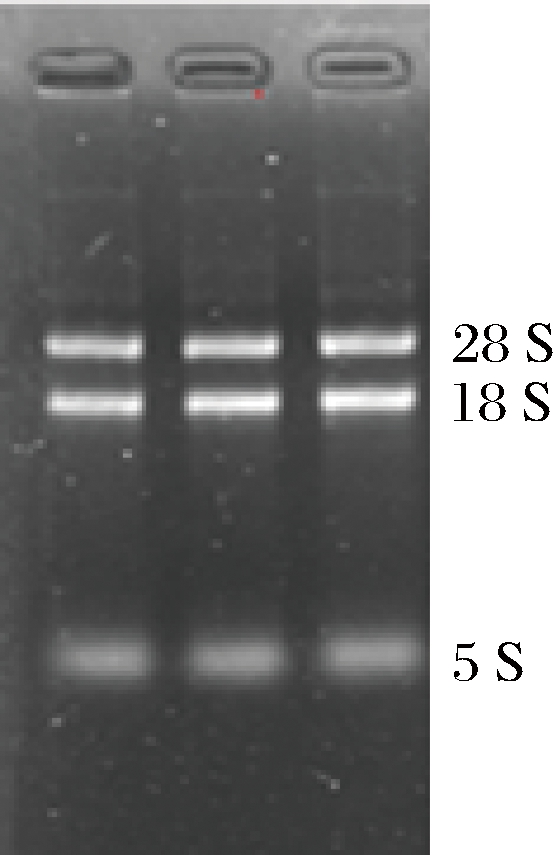
图4 裂褶菌总RNA电泳检测
Fig.4 The electrophoresis detection of maitake RNA
转录组测序数据经过滤统计、拼接,组装出全长转录本Unigene序列,共获得23 656个Unigene,最长为13 092 nt,最短为201 nt,Unigene序列的平均GC含量为0.589 7。
根据Unigene序列的长度进行分析,核酸序列长度在200~400 nt之间的Unigene占比最多,而长度在200~2 000 nt之间的占85.51%,为20 229个;长度在2 000~3 000 nt之间的为2 210个,占比为9.34%;长度大于3 000 nt的Unigene数为1 217个,占总数的5.14%。
2.3 Unigene的数据库比对分析
Unigene序列分别与NR、NT数据库进行BLAST比对分析,分别有7 281和1 559个能够比对成功,占总数目的30.78%和6.59%。Unigene序列分别进行BLASTP和BLASTX分析后,有7 353和7 084个能够比对成功,占总数的31.08%和29.95%。分析显示,共有5个作用于玉米皮半纤维素主链的木聚糖酶基因(表1),分别属于糖苷水解酶第10家族和第11家族,木聚糖酶基因在687~1 113 bp之间。玉米皮半纤维素的彻底降解需要多种酶的协同作用[18],而半纤维素主链的降解同样需要多种木聚糖酶的协同作用。有研究显示,通过裂褶菌基因组测序分析能够预测到6个木聚糖酶基因[19],其中5个为糖苷水解酶的第10家族,1个为第11家族。转录组测序分析预测木聚糖酶基因数量少于基因组测序分析数量,可能是由于在本研究的培养条件下,某些木聚糖基因没有转录或转录水平低造成的。按序列同源性,木聚糖酶分别属于糖苷水解酶第10家族、11家族和5家族,属于同一家族的木聚糖酶催化区域具有同源性。第10家族的木聚糖酶分子量高,结构复杂,主要作用于支链,通常生成较小的低聚糖;第11家族主要作用于主链,对木聚糖有很高的特异性;第5家族只作用于有阿拉伯糖集合的位点[20-21]。
表1 木聚糖酶基因序列分析
Table 1 Analysis of xylanase gene sequence
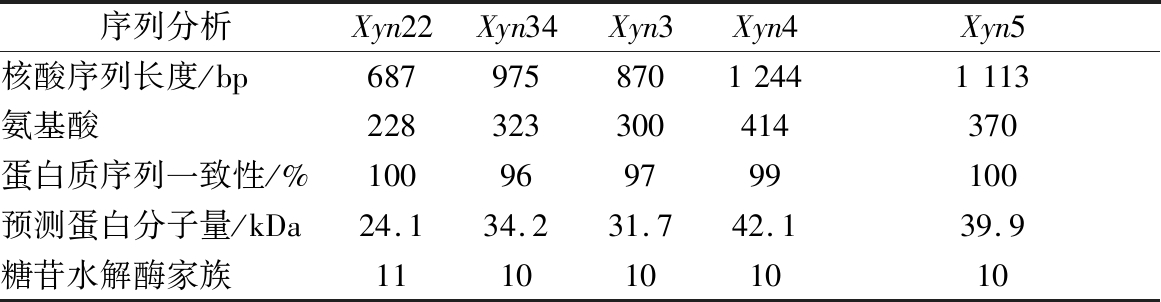
序列分析Xyn22Xyn34Xyn3Xyn4Xyn5核酸序列长度/bp68797587012441113氨基酸228323300414370蛋白质序列一致性/%100969799100预测蛋白分子量/kDa24.134.231.742.139.9糖苷水解酶家族1110101010
裂褶菌能够分泌表达多种半纤维素酶,并通过多种酶的协同作用将玉米皮纤维降解利用。转录组结果显示,裂褶菌还表达了多种半纤维素支链水解酶基因,包括木糖苷酶(β-xylosidase)、α-阿拉伯呋喃糖苷酶(α-arabinofuranosidase)、甘露糖苷酶(mannosidases)、阿魏酸酯酶(feruloyl esterase)、酯酶(esterase)等。其中与木聚糖主链连接的阿拉伯糖基侧链抑制了木聚糖水解酶的活性,而α-L-阿拉伯呋喃糖苷酶能水解木聚糖的阿拉伯糖残基,释放阿拉伯糖分子,促进半纤维素的降解[22]。根据α-L-阿拉伯呋喃糖苷酶氨基酸序列的相似性以及活性蛋白质催化中心的结构特征,将该酶归为糖苷水解酶的第3、43、51、54、62和127家族[23]。转录组测序显示,裂褶菌能够表达多种α-L-阿拉伯呋喃糖苷酶(表2),共有6个α-L-阿拉伯呋喃糖苷酶基因,分别属于糖苷水解酶第43、51和62家族。目前已有研究表明α-L-阿拉伯呋喃糖苷酶可同其他半纤维素酶协同作用,促进半纤维素水解[24],因此本研究设计克隆α-L-阿拉伯呋喃糖苷酶基因Sabf32。
2.4 Unigene的注释分类
通过GO注释和GO功能分类分析,Unigene被注释到生物过程(biological process)、细胞组成(cellular component)、分子功能(molecular function)3个部分,其中生物过程注释23个条目、细胞组成注释22个条目、分子功能注释22个条目。
表2 α-L-阿拉伯呋喃糖苷酶基因序列分析
Table 2 Analysis of α-L-arabinofuranosidase gene sequence
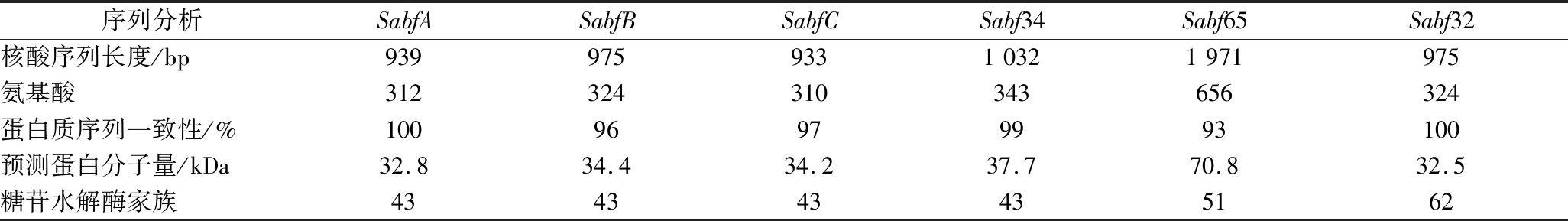
序列分析SabfASabfBSabfCSabf34Sabf65Sabf32核酸序列长度/bp93997593310321971975氨基酸312324310343656324蛋白质序列一致性/%10096979993100预测蛋白分子量/kDa32.834.434.237.770.832.5糖苷水解酶家族434343435162
将Unigene的蛋白质序列提交到NCBI,获得与Unigene编号相对应的COG编号,对COG每个类别Unigene的数目进行统计。数量最多的是一般功能预测蛋白,其他较多的分类包括与氨基酸转运和代谢、能量产生和转化、糖类转运和代谢、翻译/核糖体结构和发生、翻译后修饰/蛋白质转化和伴侣有关的基因,根据基因数量分布绘制裂褶菌的Unigene COG分类图(图5)。

图5 裂褶菌Unigene的COG分类
Fig.5 COG functional classifications of maitake Unigene
2.5 Sabf32基因的克隆及重组表达载体的构建
以裂褶菌cDNA为模版,PCR扩增Sabf32基因的编码序列,并进行测序分析。测序显示Sabf32编码序列全长975 bp,编码324个氨基酸和1个终止密码子;信号肽预测分析显示,Sabf32的N端具有21个氨基酸组成的信号肽序列。通过BLASTX比对,Sabf32与来源于Schizophyllum commune H4-8的糖苷水解酶62家族蛋白(XP_003038505.1)具有最高一致性(100%),目前还没有关于该基因的研究报道。
根据Sabf32序列设计合成表达引物,以cDNA为模板,PCR扩增获得Sabf32基因片段(图6)。
将基因片段Sabf32与表达载体pPIC9k分别进行双酶切;将纯化后的片段链接,构建重组表达载体pPIC9k-Sabf32。测序确定序列正确后,将重组表达载体pPIC9k-Sabf32与空载体pPIC9k分别电击转化毕赤酵母GS115感受态细胞,通过α-L-阿拉伯呋喃糖苷酶酶活力的测定筛选获得阳性重组子。
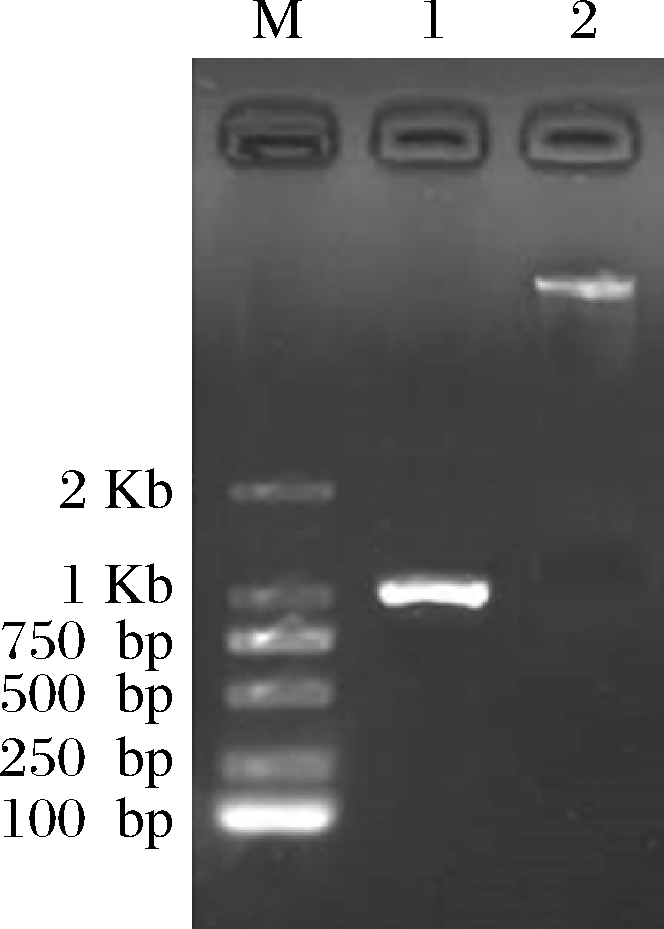
M-核酸分子量标准; 1- Sabf32;2-pPIC9k载体
图6 Sabf32和pPIC9k载体双酶切琼脂糖凝胶电泳检测
Fig.6 Agarose gel electrophoresis of Sabf32 and pPIC9k
2.6 重组毕赤酵母的诱导表达及酶活力测定
挑取转化pPIC9k-Sabf32的毕赤酵母,利用BMGY发酵培养,离心收集菌体后在BMMY培养基中进行甲醇诱导表达。取发酵7 d后的发酵液的上清,进行酶活力测定和SDS-PAGE电泳检测(图7)。重组酶Sabf32的相对分子量为32.5 kDa。
2.7 温度和pH对重组Sabf32活性的影响
重组Sabf32的最适反应温度为40 ℃(图8),在40 ℃条件下有较好的稳定性,在50 ℃条件下酶活力迅速降低,30 min后酶活完全丧失(图9)。重组Sabf32的最适pH值为5.0,在pH 3.5~5.5的缓冲液中处理30 min,能保存80%以上的酶活(图10)。在pH值低于2.5或高于7.0的缓冲液中处理30 min,酶活基本丧失(图11)。
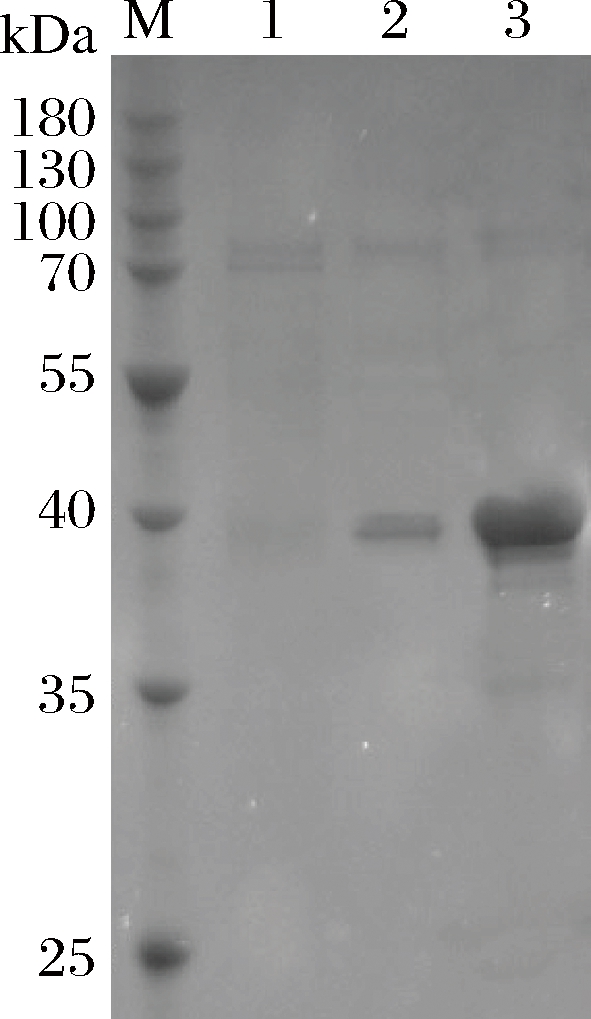
M-低分子量蛋白标准;1-对照组;2-Sabf32诱导表达发酵液;3-纯化的酶液
图7 重组酶Sabf32的SDS-PAGE电泳检测
Fig.7 SDS-PAGE analysis of Sabf32
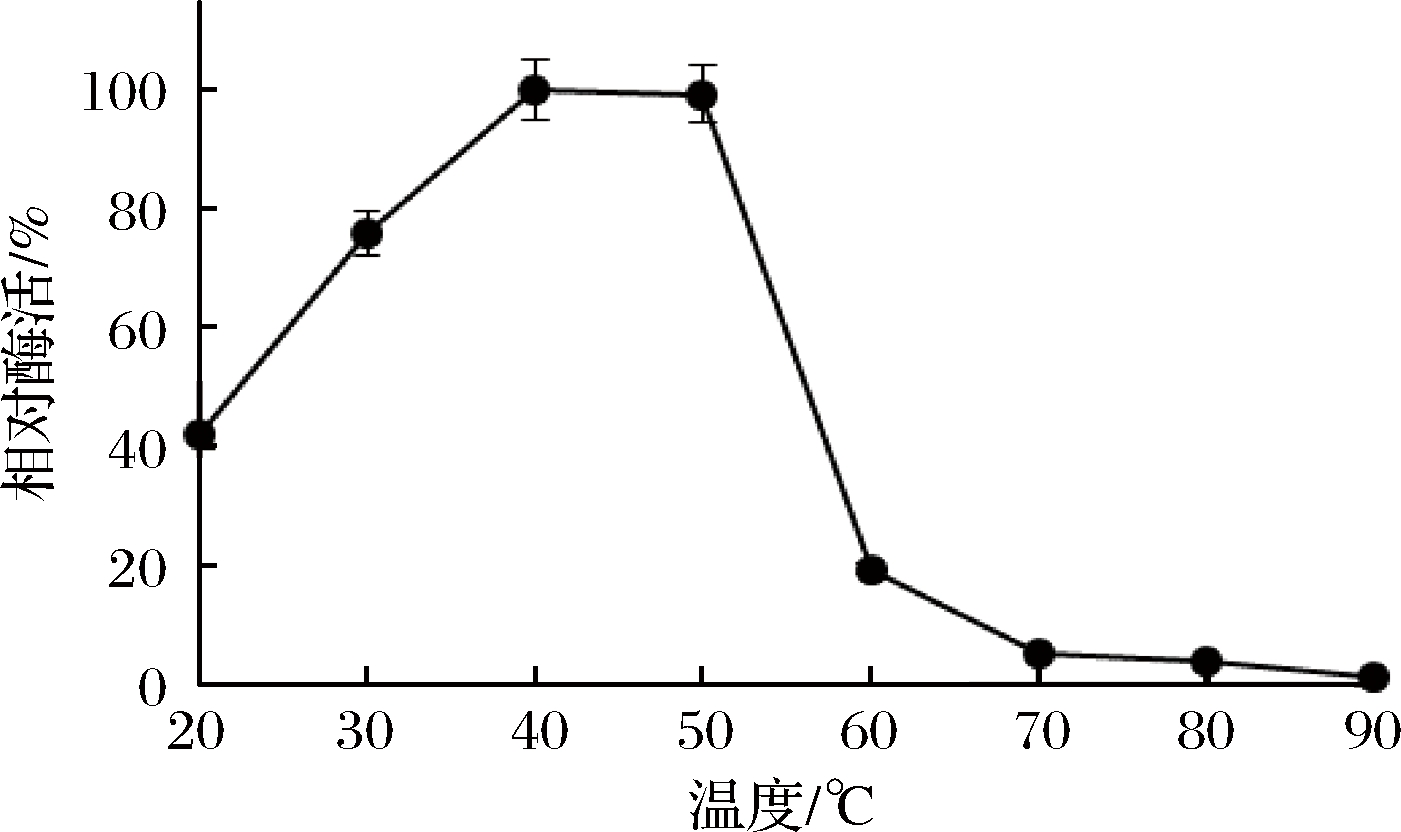
图8 重组Sabf32的最适温度
Fig.8 The optimum temperature of Sabf32
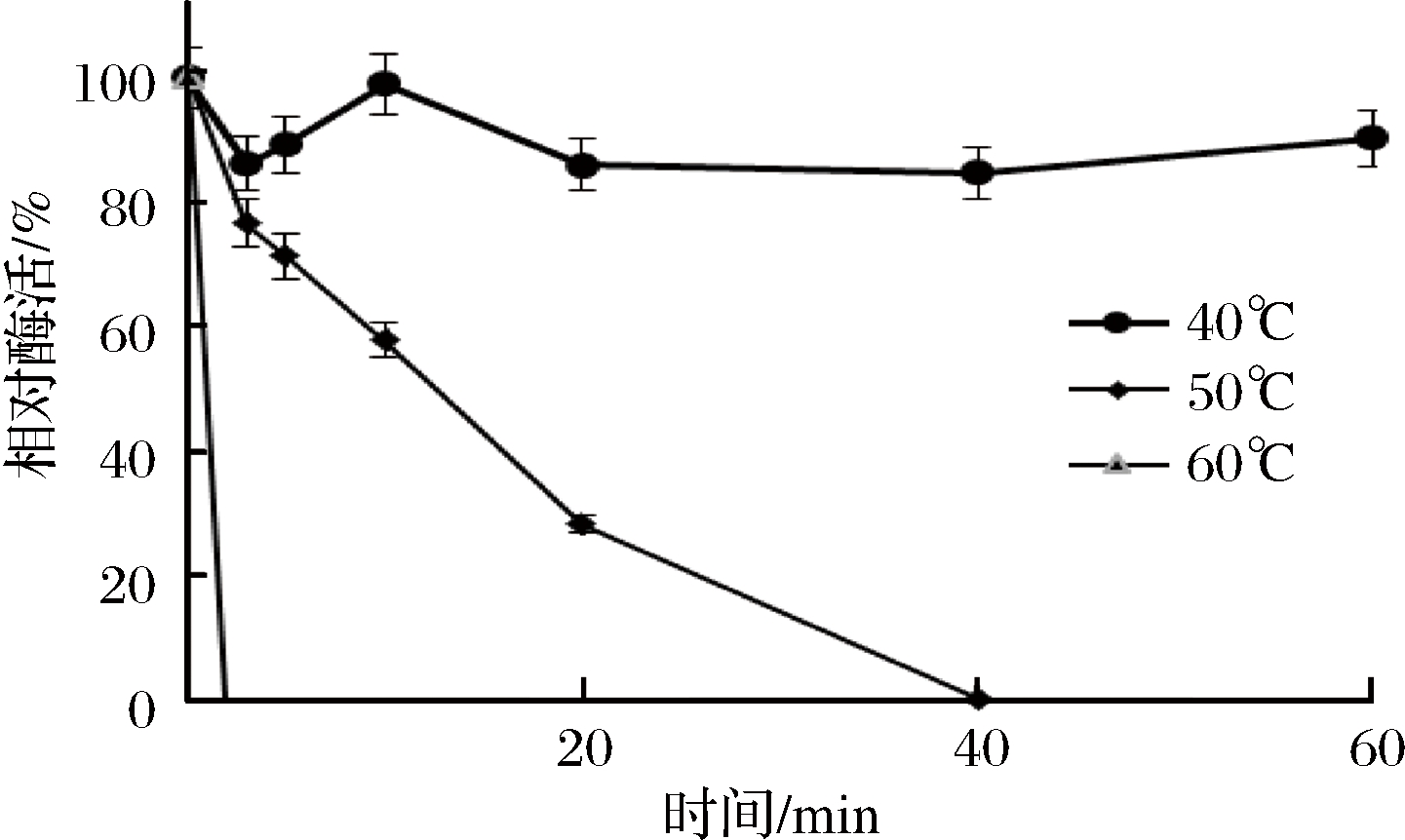
图9 重组Sabf32的温度稳定性
Fig.9 The temperature stability of Sabf32
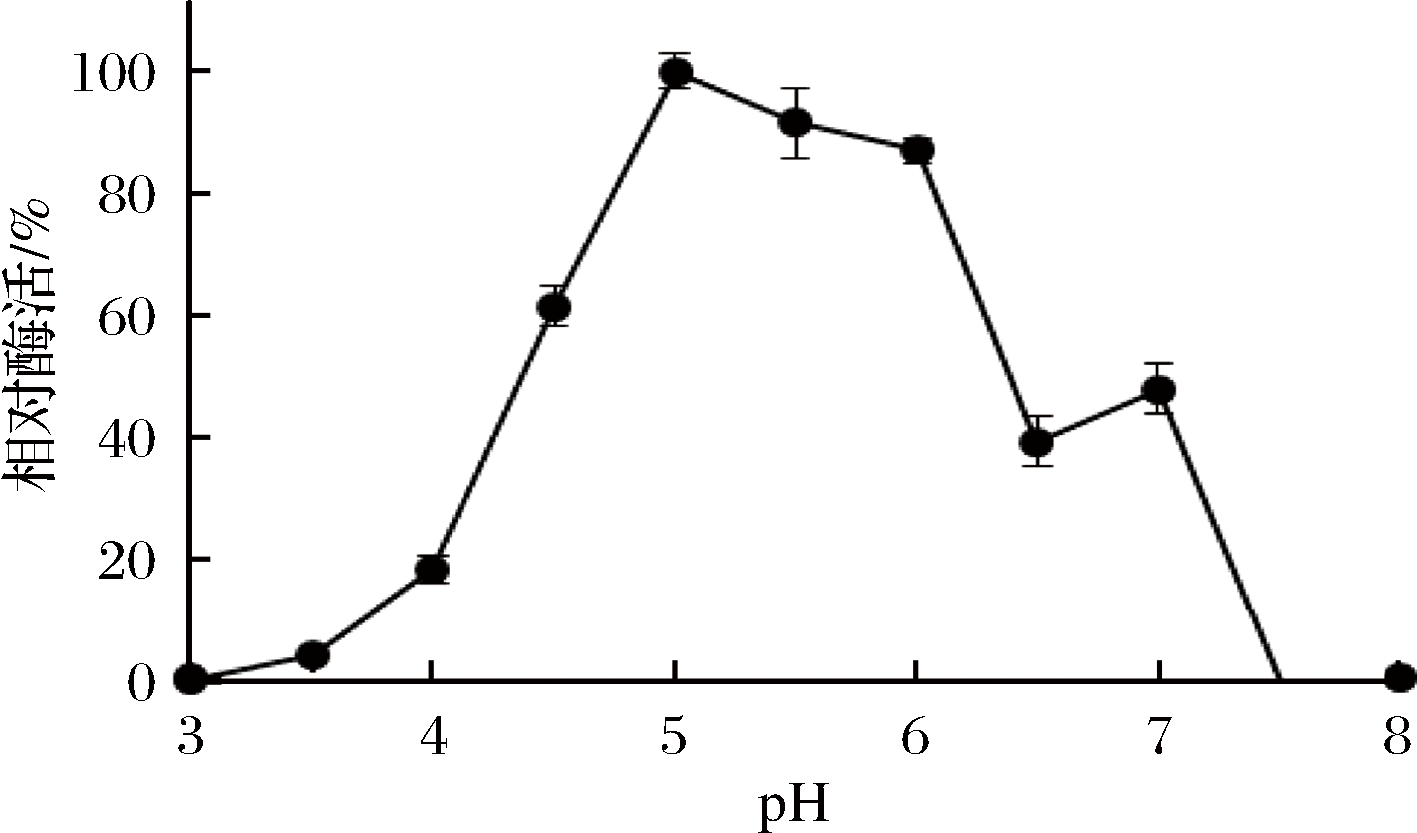
图10 重组Sabf32的最适pH
Fig.10 The optimum pH of Sabf32

图11 重组Sabf32的pH稳定性图
Fig.11 The pH stability of Sabf32
2.8 比活力和动力学参数的测定
经测定重组阿拉伯呋喃糖苷酶Sabf32比酶活力为16.18 U/mg,动力学常数Km和Vmax分别为(3.98±0.32) mmol/L和(2.59±0.09) μmol/(min·mg)。Sabf32比酶活力低于Talaromyces leycettanus JCM12802来源的阿拉伯呋喃糖苷酶TlAbf51,其比酶活力达到1 068 U/mg[25]。
3 结论
通过测定发酵液中蛋白质浓度、还原糖含量和木聚糖酶酶活力,确定了裂褶菌转录组测序的取样条件。其中发酵液中还原糖含量在48 h达到最高,之后随着培养时间延长逐渐降低;木聚糖酶活力随培养时间延长而提高,在48~72h间酶活力增长较快;蛋白质浓度随着培养时间延长逐渐提高,但超过60 h增长趋于稳定。
提取了发酵培养48 h后的裂褶菌总RNA,进行转录组测序,原始数据经分析后共获得23 656个Unigene,平均GC含量为0.589 7。序列分析显示,共有5个木聚糖酶基因,分别属于糖苷水解酶第10家族和11家族;有6个α-L-阿拉伯呋喃糖苷酶基因,分别属于糖苷水解酶第43、51和62家族。此外,裂褶菌还表达了多种半纤维素支链水解酶基因,包括木糖苷酶、甘露糖苷酶、阿魏酸酯酶、酯酶等。
克隆了α-L-阿拉伯呋喃糖苷酶基因Sabf32,编码序列全长975 bp。Sabf32的N端具有21个氨基酸的信号肽序列,与来源于Schizophyllum commune H4-8的糖苷水解酶62家族蛋白(XP_003038505.1),具有最高一致性100%。完成了该酶在毕赤酵母表达,其最适温度为40 ℃,最适pH值为4.0;在pH 3.5~5.5和40℃下较稳定。Sabf32比酶活力为16.18 U/mg,动力学常数Km和Vmax分别为(3.98±0.32) mmol/L和(2.59±0.09) μmol/(min·mg)。本研究为进一步开发玉米皮纤维降解酶以及多酶协同降解提供了基础。
[1] 鲁晓翔, 连喜军,唐津忠,等.玉米加工副产物高附加值利用研究进展[J].粮油加工, 2007, 4:84-87.
[2] LEE J E, VADLANI P V, FAUBION J. Corn bran bioprocessing: Development of an integrated process for microbial lipids production[J]. Bioresour Technol, 2017, 243:196-203.
[3] ROGOWSKI A, BASL A, FARINAS C S, et al. Evidence that GH115 α-glucuronidase activity, which is required to degrade plant biomass, is dependent on conformational flexibility[J]. J Biol Chem. 2014, 289(1):53-64.
[4] ROSE D J, INGLETT G E, LIU S X. Utilisation of corn (Zeamays) bran and corn fiber in the production of food components[J]. J Sci Food Agric, 2009, 90(6):915-924.
[5] 胡慧东, 许赣荣. 黑曲霉固态发酵及酶解玉米皮[J]. 生物加工过程, 2011, 2(9):1 672-1 678.
[6] 王雅波, 刘占英,兰辉,等. 酵母菌发酵玉米皮制备菌体蛋白饲料[J]. 中国饲料, 2017, 4:31-33.
[7] 王晶, 孙红哲,孙国英,等.玉米皮渣类纤维兼氧发酵产丁二酸[J]. 食品科技, 2012, 37(5):136-140.
[8] 潘欣, 邹立扣,岳爱玲,等. 裂褶菌培养、鉴定及氨基酸组成分析[J]. 生物技术, 2011, 21(1):54-56.
[9] 王靖宇, 刘玉春,韩伟,等. 玉米皮纤维发酵裂褶菌的产酶分析及木聚糖酶基因克隆、表达和酶学性质测定[J]. 食品与发酵工业, 2018, 44(5): 46-51.
[10] YANG F, XU B, LI J, et al. Transcriptome analysis of Termitomyces albuminosus reveals the biodegradation of lignocellulose[J]. Acta Microbiologica Sinica, 2012, 52(4):466-477.
[11] 聂文强, 吴天祥,钟敏,等. 真菌灰树花菌丝体转录组测序及分析[J].食品科学, 2017, 38(2): 6-11.
[12] SONG Y, LEE Y G, CHOI I S, et al. Heterologous expression of endo-1,4-β-xylanase A from Schizophyllum commune in Pichia pastoris and functional characterization of the recombinant enzyme[J]. Enzyme Microb Technol. 2013,52(3):170-176.
[13] BAILEY M J, BIELY P, POUTANEN K. Interlaboratory testing of methods for assay of xylanase activity[J]. J Biotechnol, 1992, 23(3):257-270.
[14] GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652.
[15] HAAS B J, PAPANICOLAOU A, YASSOUR M, et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nature Protocols, 2013, 8(8):1 494-1 512.
[16] QUEVILLON E, SILVENTOINEN V, PILLAI S, et al. InterProScan: protein domains identifier[J]. Nucleic Acids Research, 2005, 33(2):116-120.
[17] YE J, FANG L, ZHENG H K, et al. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research, 2006, 34(2):293-297.
[18] SAWHNEY N, CROOKS C, ST J F, et al. Transcriptomic analysis of xylan utilization systems in Paenibacillus sp. strain JDR-2[J]. Appl Environ Microbiol, 2015, 81(4):1 490-1 501.
[19] Alfaro M, Oguiza J A, Ramírez L, et al. Comparative analysis of secretomes in basidiomycete fungi[J]. J Proteomics, 2014, 102:28-43.
[20] COLLINS T, GERDAY C, FELLER G. Xylanases, xylanase families and extremophilic xylanases[J]. FEMS Microbiol Rev, 2005, 29(1):3-23.
[21] KOLENOVA K, VRSANSKA M, BIELY P. Mode of action of endo-β-1,4-xylanases of families 10 and 11 on acidic xylooligosaccharides[J]. J Biotechnol, 2006, 121(3):338-345.
[22] 解西柱,张明,林材,等. α-L-阿拉伯呋喃糖苷酶在毕赤酵母中的表达及其对大麦麦芽过滤性能影响[J]. 食品与发酵工业, 2018, 44(2):48-54.
[23] HENRISSAT B, DAVIES G J. Glycoside hydrolases and glycosyltransferases families, modules and implications for genomics[J]. Plant Physiology, 2000, 124:1 515-1 519.
[24] DE VRIES R P, KESTER H C M, POULSEN C H, et al. Synergy between enzymes from Aspergillus involved in the degradation of plant cell wall polysaccharides[J]. Carbohydrate Research, 2000. 327: 401-410.
[25] Tu T, Li X, Meng K, et al. A GH51 α-L-arabinofuranosidase from Talaromyces leycettanus strain JCM12802 that selectively drives synergistic lignocellulose hydrolysis[J]. Microb Cell Fact, 2019, 18(1):138.