人体蛋白质和微量元素的重要来源为肉及其制品。近年来,随着人们生活和消费水平的提高,肉类的权重在饮食中逐年增加[1]。但全球的肉制品安全现状令人担忧,欧洲的马肉风波[2],我国江苏地区羊肉掺假事件[3]等严重的食品安全问题不断暴露。国内部分地区开展肉及其制品的掺假情况调查[4],2013年的结果显示,25.6%的样品标识的源性成分与标签内容不符,肉制品掺假问题严重,熟制牛肉和羊肉卷成为掺假高危品。另外由于通过食用牛源性成分的食品可能引起疯牛病传播[5],以及宗教信仰问题,印度教徒不食用牛肉,而犹太和穆斯林人群饮食避免猪肉和马肉成分[6]。在这样的背景下,迫切需要建立针对食品基质的快速、经济、准确有效的肉源性鉴定的方法。
追溯到20世纪80年代,动物源性成分的鉴别已经引起国外人士的重视,并开展了相关研究。目前鉴别方法有通过感官的(配合仪器)物理法、化学法,基于蛋白质的免疫分析法、分子生物学法,如色谱法、酶联免疫吸附测定(enzyme-linked immunosorbent assay,ELISA)法[7-8]、结合电泳的普通聚合酶链式反应(polymerase chain reaction,PCR)法[9]、实时荧光PCR法、环介导等温扩增法(loop-mediated isothermal amplification,LAMP)[10-12]等。其中PCR方法最为准确,快速,灵敏,尤其实时荧光PCR法,操作简单,PCR产物不需要电泳,污染小,在食品检测中应用广泛[13-20],但是考虑荧光PCR仪器昂贵,对实验设备要求高,本研究采用普通PCR方法进行,选择新标识性基因位点,在保证结果准确、稳定性高的前提下,可提高检测灵敏度,极大节约了实验设备、荧光试剂费用,检测成本低廉,为牛肉及其制品的掺假检测提供方法保障。
1 材料与方法
1.1 食品样品
牛(Bovidae)样本生鲜肉分别来自江苏省南京市当地大型超市、大型菜市场,部分生鲜牛肉及其制品购自大型连锁餐饮店或通过网络渠道获得。收集到的24个样本信息详见表1。其中生鲜牛肉是直接购买的未切分的产品,加工制品的配料来源于产品标签标识。
同时市售购买牛近源哺乳动物肉类样品,山羊(Capra hircus)、绵羊(Ovis aries)、马(Equus caballus)、驴(Equus asinus)、兔(Leporidae)、猪(Sus domesticus)及禽类鸡(Gallus domesticus)、鸭(Anatinae)、鹅(Anser cygnoides orientalis),其他常见动物草鱼(Ctenopharyngodon idellus)、鲫鱼(Carassius aur tus)、小黄鱼(Larimichthys polyactis),常见植物材料大豆(Glycine max)、玉米(Zea mays)种子颗粒,用于建立的牛成分方法的特异性验证。
表1 收集到的24个样本信息
Table 1 Species information of 24 Bovidae samples collected for the study
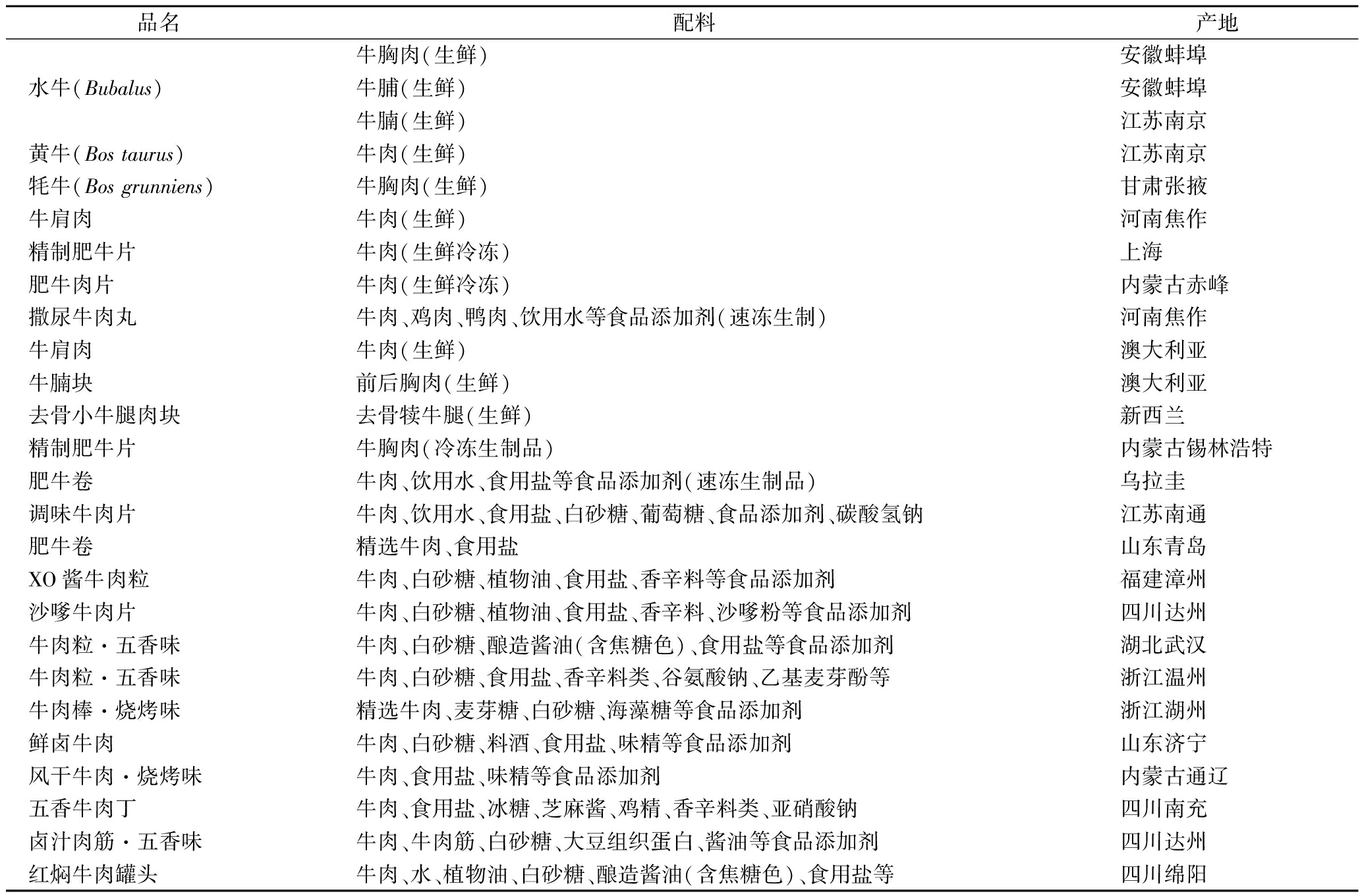
实验样品除驴肉、兔肉、鹅肉购自河南省外,其他样本均购自南京市各大超市或大型农贸市场,包括生鲜山羊、绵羊、马、猪、鸡、鸭、鱼肉、大豆、玉米,生鲜牛肉片、牛肉卷、冷冻牛肉丸及牛肉卷,肉脯类(牛肉片、牛肉粒)加工制品购自网络畅销商家,所有牛及其制品的样本均标注含有牛成分或为牛肉制品,其他样本均需通过国家现行有效的标准方法[21-28]鉴定其源性真实。.
1.2 试剂与仪器
TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0,宝日医生物技术(北京)有限公司;10×Reaction Buffer、BioReady rTaq、dNTP Mixture,杭州博日技术有限公司;GelRedTMNucleic Acid Gel Stain,10 000×上海开放生物科技有限公司;DL500 DNA Marker、6×Loading Buffer,宝生物工程(大连)有限公司;PCR上下游引物合成,上海生工生物有限公司。
Life Express Thermal Cycler PCR仪,杭州博日技术有限公司;Biophotometer D30核酸蛋白测定仪,德国Eppendorf公司;JY300C电泳仪,北京君意东方电泳设备有限公司;WD-9413B凝胶成像分析仪,北京六一生物科技有限公司。
1.3 实验方法
1.3.1 样本的预处理
对于生鲜的肉样品,用无菌、擦拭干净的手术剪刀除去表层组织及肉皮、筋、脂肪层等,从内部剪取肉块(避免表层污染),于高速粉碎机碾拌成均匀肉浆;对于植物样品,于高速粉碎机研磨成均匀的粉末,-20 ℃保存备用。
1.3.2 灵敏度测试样品制备
肉含量灵敏度试验拟定4种食品基质(猪肉、羊肉、驴肉、大豆)与黄牛肉制备混合样品[29]。考虑大豆为干粉,制备大豆以及牛肉混合样品时,首先将牛肉、大豆粉真空40 ℃干燥,后用高速粉碎机将牛肉研磨成均匀的干粉。之后按照质量梯度配制混合样品,总质量为100.00 g。将黄牛肉分别与大豆、猪肉、羊肉、驴肉混合,制备含质量分数为5%、1%、0.5%、0.1%、0.05%、0.01%牛肉的混合样品,依次分别记为b1~b6、p1~p6、s1~s6、d1~d6,并将混合样品均分为2份,1份不做处理,另1份模拟深加工产品进行121 ℃、0.1 MPa,20 min高温处理[15], -20 ℃贮存待用。
1.3.3 DNA提取及确证方法
准确称取植物组织(种子部分)100 mg,动物组织或实际样品25 mg,按照DNA提取试剂盒说明书步骤提取动植物组织DNA,每个样品设置3个平行用于提取DNA,同时设置提取空白,并于核酸蛋白仪测定所提取DNA的浓度及纯度,后-20 ℃保存待用。本研究使用的DNA浓度范围60~800 ng/μL,纯度(A260nm/A280nm)1.70~2.15、(A260nm/A230nm)1.87~2.40。提取的DNA在PCR扩增之前,均采用1.2%的琼脂糖,100 V电压参数下50 min琼脂糖凝胶电泳确证长度有效,满足PCR扩增要求。
1.3.4 引物设计
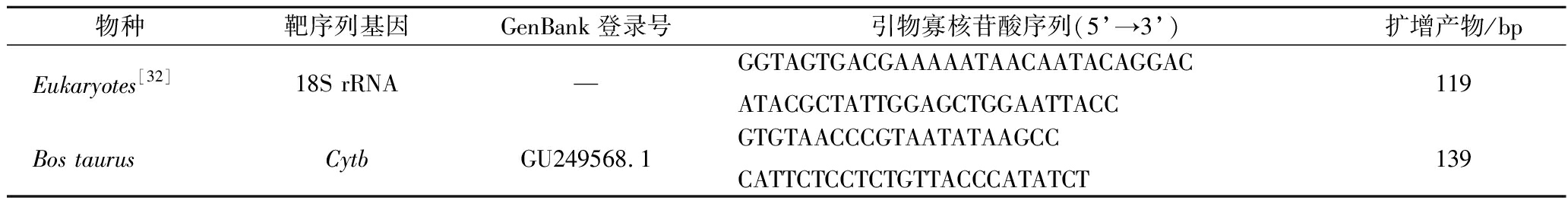
选取牛种属线粒体细胞色素b(cytochrome b,Cytb)基因作为靶标基因,以牛种属中水牛(Bubalus)、黄牛(Bos taurus)、牦牛(Bos grunniens)以及非牛动物(包括山羊、绵羊、马、猪、鸡、鸭、鱼、鹅、驴、兔)的拉丁学名为关键词,在NCBI上搜集Cytb基因序列,通过同源性比对分析[30],确定参考基因序列的GenBank登录号,并根据引物设计原则完成引物设计。所设计引物的特异性通过BLAST验证,并委托生工生物工程有限公司合成。
1.3.5 PCR反应体系及条件
反应体系(25 μL):2.5 μL 10×Reaction Buffer、1.0 μL dNTP Mixture(各2.5 mmol/L)、0.3 μL(2U) BioReady rTaq,0.5 μL上游引物(10 μmol/L)、0.5 μL下游引物(10 μmol/L)、5 μL Template DNA、超纯水补足至25 μL。
18S rRNA PCR反应条件:模板预变性94 ℃,2 min;模板变性94 ℃,45 s,退火57 ℃,45 s,延伸72 ℃,45 s,30个循环;72 ℃,5 min。
Cytb PCR反应条件:模板预变性94 ℃,2 min;模板变性94 ℃,45 s,退火57 ℃,45 s,延伸72 ℃,45 s,30个循环;72 ℃,5 min。
1.3.6 PCR产物检测方法
取7 μL PCR扩增产物与1.5 μL 6×Loading buffer混合,于1.5%琼脂糖凝胶和0.5×TBE缓冲液中电泳(电压160 V,30 min),并采用凝胶成像分析仪查看拍照。
1.3.7 PCR产物测序
采用上述设计的引物进行PCR扩增,产物取7 μL用于凝胶电泳,得到阳性扩增条带后,取剩余的PCR产物40 μL送上海生工生物有限公司测序部进行测序。
1.3.8 引物特异性和灵敏度分析
特异性检测选用3种牛品种;4种不同产地的近源哺乳动物: 1种反刍动物(羊)、3种非反刍哺乳动物(猪、马、驴);3种常见的禽类(鸡、鸭、鹅)以及兔、鱼;2种植物(大豆、玉米)样品的DNA为模板,采用所建立的鉴定方法进行PCR扩增,同时设置空白组。
灵敏度检测包括肉含量和DNA用量。肉含量灵敏度检测的样品处理和制备方法参照1.3.1。DNA用量的灵敏度检测选择黄牛和羊、马、猪、鸡、鸭、鱼、鹅、驴、兔、大豆、玉米,分别将提取到的DNA稀释到10 ng/μL,然后按照体积梯度,将黄牛DNA分别与上述非牛动植物DNA混合至黄牛DNA为体积分数5%、1%、0.5%、0.1%、0.05%、0.01%,进行普通PCR扩增,以测试该方法的最低检测限(limit of detection,LOD)。采用GB/T 25165—2010标准[31],稀释阳性DNA质量浓度分别为:100、10、1.0、0.1、0.01、0.001 ng/μL,建立PCR方法标准曲线,确定所建立方法检测下限的DNA量。
1.3.9 方法适用性评价
采用所建立的方法,对市售的配料中含“牛肉”的26份样品进行检测。每种样品取2份,提取DNA后用18S rRNA引物验证所提取DNA的有效性,避免假阴性结果。18S rRNA引物扩增阳性后,采用所建立的方法PCR扩增。每个样品做3个平行,减少随机误差。
同时采用GB/T 25165—2010[31]和GB/T 20190—2006[21]标准方法检测上述26份样品的牛源性成分,比较所建立的牛源性PCR鉴定方法与标准方法的一致性。
2 结果与分析
2.1 引物设计结果
通过比对分析,确定参考基因序列的GenBank登录号为:牛(KX575711.1),绵羊(AF010406.1)[14],马(JF010406.1)、驴(JF718884.1)、兔(AJ001588)、猪(X56295.1)及禽类鸡(X52391.1)、鸭(EU755252.1)、鹅(AY427816.1)等。最终选择以牛的Cytb基因为靶基因,确定GenBank登录号为GU249568.1的序列为参考序列设计引物。采用Primer 5.0共设计了9对引物,结果如表2所示。将各组引物的预期序列与上述参考的非牛基因序列(绵羊、马、驴、兔、猪、鸡、鸭、鹅)进行比对,选取引物3’端与牛源序列高度匹配而同非牛源序列不互补的1对引物,如图1所示。结合后续实验结果,最终确定的引物及参考的真核生物18S rRNA引物(如表3)。
表2 本研究设计PCR引物
Table 2 PCR primer used in this study
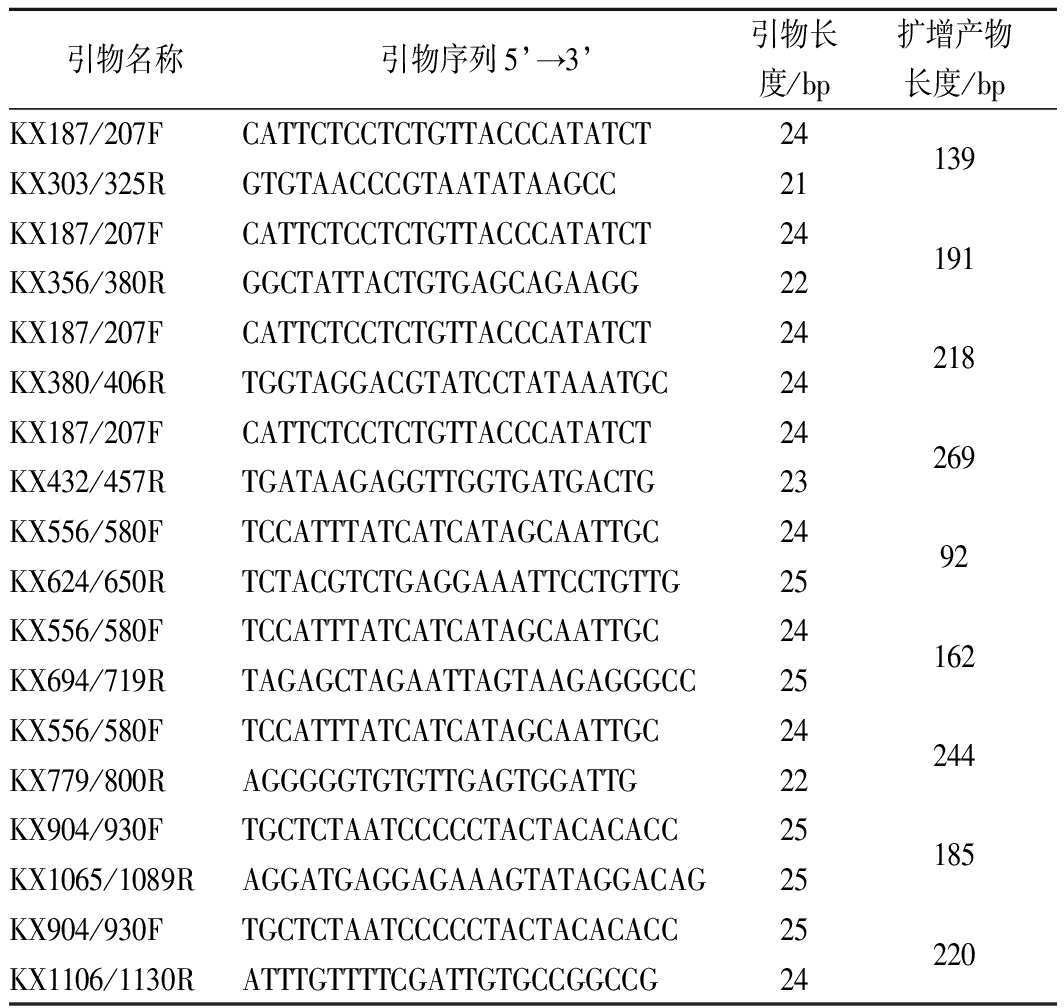

图1 牛源性成分检测引物KX187/207F和KX303/325
扩增片段与其他物种线粒体DNA相应片段一致性比对
Fig.1 Identity comparison sequences amplified by cow
specific primers(KX187/207F and KX303/325R) and
corresponding region of mtDNA of other animals
2.2 引物特异性分析
本实验中用到的动物源DNA均已采用现行有效的标准方法验证其源性真实,排除异质污染,为特异性试验作保障。
为了验证引物对牛源性成分的特异性,将3种牛品种,9种非牛动物(羊、猪、马、驴、兔、鸡、鸭、鹅、鱼),2种植物(大豆、玉米)样品的DNA为模板,进行PCR扩增。结果如图2所示,牛样品有明显的扩增条带,长度为139 bp,与引物设计预期一致,其他11种非牛动植物均无扩增条带(如图2-A)。研究中同时对选用的所有样品采用18S rRNA引物扩增,均出现目标条带并对应其长度为119 bp(如图2-B)。确证所提取DNA满足PCR要求,避免假阴性结果。综上,该对引物针对牛源性成分特异性良好。
表3 牛种属及真核生物的寡核苷酸引物
Table 3 Oligonucleotide primers for eukaryotes and cow species

A-采用特异性引物扩增得到的PCR产物的电泳结果;B-采用18S rRNA引物扩增得到的PCR产物的电泳结果;
M-500 bp Maker;1~15依次为牦牛、黄牛、水牛、羊、鸡、鸭、猪、驴、马、兔、鹅、鱼、玉米、大豆、空白对照
图2 普通PCR方法的特异性分析
Fig.2 Specificity of the conventional PCR method
2.3 设计引物PCR产物测序结果
对所设计的引物扩增获得的阳性结果进行核苷酸序列分析,结果如表4所示。表4中测序片段序列是通过对正、反向测序结果进行接拼而成的完整序列。结果表明,目标基因正向测序结果与预期目的基因序列一致性为100%,反向测序结果与预期目的基因序列一致性为100%。在NCBI(national center forbiotechnology information(https://www.ncbi.nlm.nih.gov/)上BLAST结果显示,测序得到的核苷酸序列与引物设计所依据的牛(KX575711.1)片段100%同源。综上,所设计的引物能达到扩增预期目标基因的目的。
表4 牛PCR产物预期序列及测序结果
Table 4 PCR products expected sequence and sequencing results

2.4 所设计引物灵敏度分析
2.4.1 肉含量
肉含量为相对灵敏度检测,将黄牛肉样品按照肉的含量梯度混入反刍动物基质(羊肉)、非反刍哺乳动物(猪、驴)、植物基质(大豆粉)中,分析可以稳定扩增的最低肉含量。根据GMO实验室欧洲网络指南[33],LOD值被定义为当至少总重复的95%PCR结果中获得阳性信号时,目标肉含量的最低浓度。本试验中LOD使用9次实验重复分析,每个样品同次设置3个PCR平行反应。肉含量(生鲜)检测限测试结果如图3所示。

M-500 bp Maker;1-100%黄牛;2~7依次为黄牛肉含量梯度5%,1%,0.5%,0.1%,0.05%,0.01%;8-阴性对照;9-空白对照;A~D依次为黄牛肉混合羊肉、驴肉、猪肉、大豆粉的电泳结果
图3 肉含量检测限测试结果
Fig.3 The limit of detection test results of meat content
从图3可以看出,黄牛肉混合羊肉、驴肉、猪肉、大豆粉的样品(生鲜)的检测限均为牛肉含量0.01%,27次PCR结果显示,大于总重复95%的PCR结果显示可检测到的最低牛含量为0.01%;121 ℃、0.1 MPa,20 min处理的混合样品,仍有高于总重复95%的PCR结果显示,可检测到牛肉含量达0.01%(结果未展示)。综上,所建立的牛源性成分特异性检测PCR方法的肉含量检测限可达0.01%。
2.4.2 DNA用量
DNA用量的灵敏度为绝对灵敏度测试,选择黄牛和羊、马、猪、鸡、鸭、鱼、鹅、驴、兔、大豆、玉米,将黄牛DNA按照体积梯度与其他非牛动植物DNA混合,分析可以稳定扩增的DNA用量稀释倍数。根据GMO实验室欧洲网络指南[33]中对LOD的定义,DNA用量的LOD值使用9次重复分析确定,每个样品同次设置3个PCR平行反应。检测结果见图4。
如图4所示,黄牛DNA掺入羊、鸡、猪、兔、鹅、玉米、大豆DNA的检测限均为DNA用量0.01%(总DNA量50 ng),黄牛DNA掺入鸭、驴、马、鱼DNA的检测限均为DNA用量0.05%,27次PCR结果显示,大于总重复95%的PCR结果显示可检测到的最低牛含量为总DNA的0.01%(结果未展示)。综上,所建立的特异性牛源检测PCR方法的DNA用量检测限可达0.01%。采用GB/T 25165—2010标准[31],按照1.3.7进行标准曲线的制作(标准曲线如图5),通过30次PCR重复反应,得到所建立PCR方法可稳定检测的牛DNA用量为0.2 pg。

M-500bp Maker;1-100%牛;2~7依次为黄牛DNA体积梯度5%,1%,0.5%,0.1%,0.05%,0.01%;8-阴性对照;9-空白对照;A~K依次为黄牛DNA混合羊、鸡、鸭、猪、驴、马、兔、鹅、鱼、玉米、大豆
DNA扩增后的电泳结果
图4 DNA用量检测限测试结果
Fig.4 The limit of detection test results of DNA content
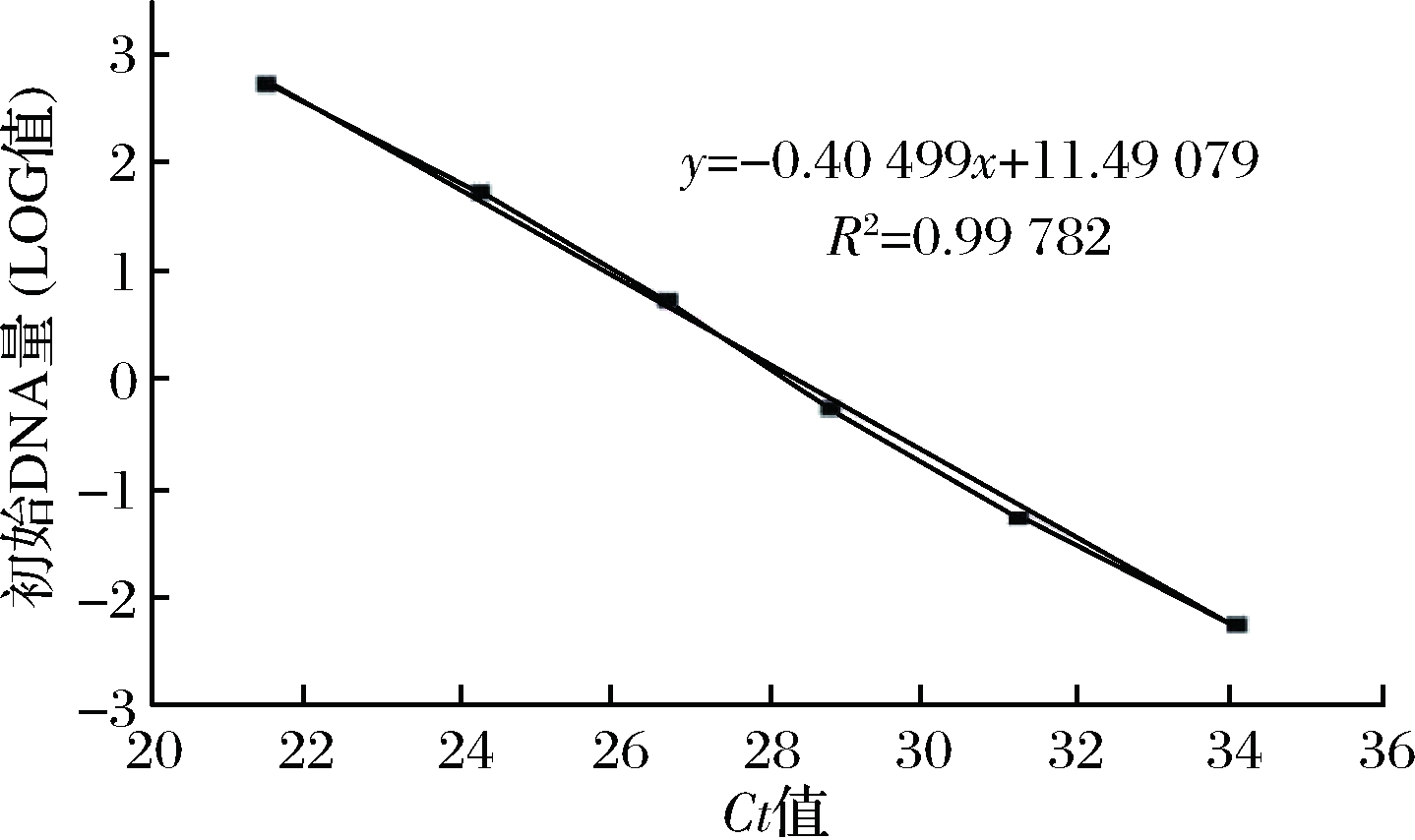
图5 黄牛DNA标准曲线
Fig.5 The standard curve line of cattle DNA
2.5 适用性评价
将26份市场上购买的标签中含“牛肉”成分的样品分别编号1~26,并进行检测,结果如表5。
分析表5结果,表明采用所设计的特异引物鉴定样品牛源性成分,均可扩增出目的条带;同时采用标准方法[31]比对,同所设计方法的结果一致率100%。表明所建立的特异性牛源成分鉴定方法适用性良好。其中22号样品,采用所建立方法与标准方法[31]均未检测出牛成分(同时以黄牛DNA为阳性对照),表明市场上存在食品成分与标签不符或掺假的问题。所检测26份样品的18S rRNA引物扩增结果均为阳性(结果未展示),验证所提取的DNA满足PCR扩增要求。
表5 市售样品PCR检测结果
Table 5 The results of PCR detection of commercial
samples
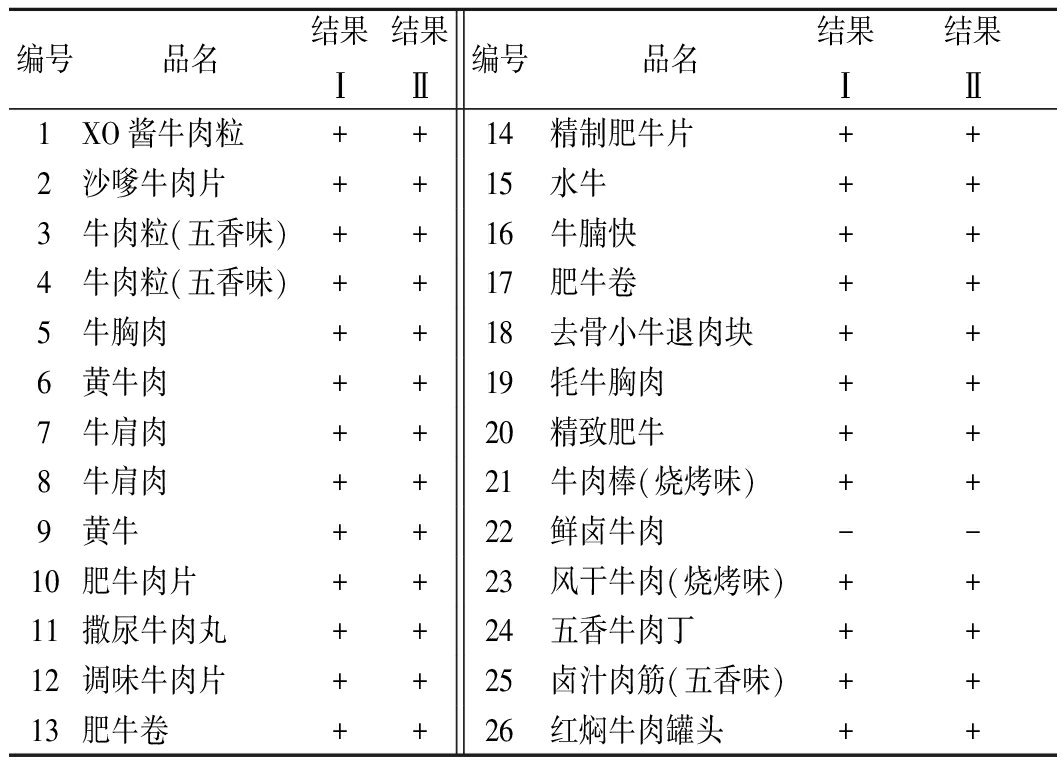
注:表中结果Ⅰ,采用本研究所建立的方法;结果Ⅱ,采用GB/T 25165—2010标准方法[31];+,检出牛成分;—,未检出牛成分。
3 讨论
比较本研究建立的特异性牛源成分检测方法,目前标准GB/T 20190—2006[21]选择ATPase subunit 6&8基因设计引物的普通PCR牛成分检测方法,得到牛成分的最低检测限达质量分数为0.25%;按照GB/T 25165—2010[31],选取生长激素基因进行引物设计的荧光定量PCR方法,检测限达0.1%(质量分数);NANAS PASCOAL[34]、JEOMG CHUL HA[35]针对Cytb和12S rRNA建立牛源测试方法,检测限分别可达0.025%、0.01%(质量分数);ZHANG等[36]选用非内部重复的、含有至少3个不同牛家族成员的1.709卫星DNA作为靶基因,设计牛源成分引物,检测限达33.6 fg DNA(生肉)、0.32 pg DNA(加工)。本研究建立的方法检测限达0.2 pg DNA,0.01%牛肉含量,检测限低于所述普通PCR方法,与现有的实时荧光PCR方法的检测限相当,达到了荧光PCR的高灵敏度,同时极大地降低了检测成本。因此所建立的牛源成分鉴定方法达到了高特异性、高灵敏性的要求,具有成本低的优势。
本研究从GenBank中检索获得不同牛种属的线粒体色素b(Cytb)基因,与其他畜类、植物等进行同源性分析比对后,选择了新标识性基因位点,设计了引物,建立了高效新标识基因位点的扩增体系。所选取的Cytb基因种内高度保守,种间高度特异,进而提高了检测方法的灵敏性、稳定性和适用性。
参考文献
[1] 陈琼,王济民.我国肉类消费现状与未来发展趋势[J].中国食品与营养,2013(6):45-49.
[2] 徐剑梅.欧洲马肉冒充牛肉事件席卷英法德等16国[EB/OL].(2013-02-13)[2018-04-22].http://finance.ifeng.com/news/hqcj/20130213/7665518.shtml.
[3] 白羽.江苏假羊肉案嫌犯自称没用过老鼠肉[EB/OL]. (2013-05-07)[2018-04-22].http://sh.eastday. com/m/20130507/u1a7372866.html.
[4] 金萍,丁洪流,李培,等.2013年苏州地区肉及其制品掺假情况调查[J].中国食品卫生杂志,2014,26(2):168-172.
[5] HUESTON W D.BSE and variant CJD:Emerging science,public pressure and the vagaries of policy-making[J]. Preventive Veterinary Medicine,2013,109(3/4):179-184.
[6] YASEMIN D,PELIN U,HAMIDE Z.Detection of porcine DNA in gelatine and gelatine-containing processed food products-Halal/Kosher suthentication[J].Meat Science,2012,90(3):686-689.
[7] 侯瑾,李迎秋.酶联免疫吸附技术在食品安全检测中的应用[J].中国调味品,2017,42(6):165-169.
[8] 马永征,马冬,白娣斯,等.免疫学检测肉类制品掺假研究进展[J].肉类研究,2012(9):26-29.
[9] KUMAR D, SINGH S, SINGH R, et al. A highly specific PCR assay for identification of goat (Capra hircus), meat[J]. Small Ruminant Research, 2011, 97(1):76-78.
[10] ZAHRA DNIK C, MARTZY R, MACH R L, et al. Loop-mediated isothermal amplification (LAMP) for the detection of horse meat in meat and processed meat products[J]. Food Analytical Methods, 2015, 8(6):1 576-1 581.
[11] AARTSE A, SCHOLTENS I M J, HANS J G V D A, et al. Evaluation of a loop-mediated isothermal amplification (LAMP) method for rapid on-site detection of horse meat[J]. Food Control,2017,81:9-15.
[12] ABDULMAWJOOD A, GRABOWSKI N, FOHLER S, et al. Development of loop-mediated isothermal amplification (LAMP) assay for rapid and sensitive identification of ostrich meat[J]. Plos One, 2014, 9(6):e100 717.
[13] CAI Yi-Cun, WANG Qiang, HE Yu-ping, et al. Interlaboratory validation of a real-time PCR detection method for bovineand ovine-derived material[J].Meat Science,2017,134:119-123.
[14] MONTSERRAT ESPIFIEIIRA, JUAN M VIEITES. Detection of dog and cat traces in food, pet food and farm animal feed by real-time PCR[J]. European Food Research & Technology, 2015, 241(2):233-238.
[15] NICOLETTE PEGELS,TERSA GARCIA, ROSARIO MARTIN, et al. Market analysis of food and feed products for detection of horse DNA by a TaqMan real-time PCR[J]. Food Analytical Methods, 2015, 8(2):489-498.
[16] 史艳宇,刘金华,逄晓阳,等.实时荧光PCR方法检测食品中痕量荞麦成分[J].食品科学,2009,30(20): 312-315.
[17] HERRERO B, VIEITES J M, ESPI EIRA M. Authentication of Atlantic salmon (Salmo salar) using real-time PCR[J]. Food Chemistry,2011,127(3): 1 268-1 272.
[18] SONIA SOARES, JOANA S AMARAL, M BATRIZ P P OLIVEIRA,et al. Quantitative detection of soybean in meat products by a TaqMan real-time PCR assay[J]. Meat Science, 2014,98(1): 41-46.
[19] ERGUN  , FATIH A M. The development of duplex real-time PCR based on SYBR green florescence for rapid identification of ruminant and poultry origins in foodstuff[J].Food Chemistry, 2012, 130(4):1 050-1 054.
, FATIH A M. The development of duplex real-time PCR based on SYBR green florescence for rapid identification of ruminant and poultry origins in foodstuff[J].Food Chemistry, 2012, 130(4):1 050-1 054.
[20] MUHAMMAD SAFDAR, YASEMEEN JUNJO. Development and validation of fast duplex real-time PCR assays based on SYBER Green florescence for detection of bovine and poultry origins in feedstuffs[J]. Food Chemistry, 2015, 173:660-664.
[21] 国家质量监督检验检疫总局,中国国家标准化管理委员会. GB/T 20190—2006, 饲料中牛羊源性成分的定性检测定性检测聚合酶链式反应(PCR)法[S].北京:中国标准出版社,2006.
[22] 国家质量监督检验检疫总局,中国国家标准化管理委员会. GB/T 21101—2007, 动物源性饲料中动物源性成分定性检测方法PCR方法[S].北京:中国标准出版社,2007.
[23] 国家质量监督检验检疫总局. SN/T 2978—2011, 动物源性产品中鸡源性成分PCR检测方法[S].北京:中国标准出版社,2011.
[24] 吉林省卫生厅.DBS 22/018—2013, 食品安全地方标准鲜(冻)畜肉中鸭源成分的定性检测PCR方法[S].北京:中国标准出版社,2013.
[25] 国家质量监督检验检疫总局. SN/T 3731.2—2013, 食品及饲料中常见禽类品种的鉴定方法第2部分:鹅成分检测PCR法 [S].北京:中国标准出版社,2013.
[26] 国家质量监督检验检疫总局,中国国家标准化管理委员会. GB/T 21107—2007, 动物源性饲料中马、驴源性成分定性检测方法PCR方法[S]. 北京:中国标准出版社,2007.
[27] 国家质量监督检验检疫总局,中国国家标准化管理委员会. GB/T 21102—2007, 动物源性饲料中兔源性成分定性检测方法实时荧光PCR方法[S]. 北京:中国标准出版社,2007.
[28] 国家质量监督检验检疫总局. SN/T 2867—2011, 饲料中鱼源性成分定性检测方法PCR方法[S]. 北京:中国标准出版社,2011.
[29] 李进波,鸿超,谌鸿超,等. 实时荧光PCR法鉴定食品中鲑亚科鱼成分[J].食品科学, 2013,34(20):195-196.
[30] 冯涛.动物源性成分交叉引物等温扩增快速检测方法的研究和建立[D].杭州:中国计量大学,2016.
[31] 国家质量监督检验检疫总局,中国国家标准化管理委员会. GB/T 25165—2010, 明胶中牛、羊、猪源性成分的定性测定检测方法实时荧光PCR法[S]. 北京:中国标准出版社,2010.
[32] FAJARDO V,GONZALEZ, I,MARTIN,I,et al.Real-time PCR for detection and quantification of red deer (Cervus elaphus), fallow deer(Dama dama),and roe deer(Capreolus capreolus) in meat mixtures[J].Meat Science,2008,79(2),289-298.
[33] European network of GMO Laboratories (ENGL). Definition of minimum performance requirements for analytical methods of GMO testing[EB/OL].(2008-10-13)[2018-04-22].http://gmo-crl.jrc.ec.europa.eu/doc/Min_Perf_Requir_Analyt_methods.pdf.
[34] ANANAS PASCOAL,MARTA PRADO,PILAR CALO,et al. Detection of bovine DNA in raw and heat-processed foodstuffs,commercial foods and specific risk materials by a novel specific polymerase chain reaction method[J].European Food Research & Technology,2005, 220(3/4):444-450.
[35] JEONG CHUL HA, WAN TAE JUNG, YONG SUK NAM,et al. PCR identification of ruminant tissue in raw and heat-treated meat meals[J].Journal of Food Protection,2006, 69(9):2 241-2 247.
[36] ZHANG Guoli, ZHENG Mingguang, ZHOU Zhijiang, et al. Establishment and application of a polymerase chain reaction for the identification of beef[J]. Meat Science,1999,51(3):233-236.