异常汉逊酵母菌具有较强的发酵力和酯化力,是酿造食品香气成分的主要贡献者之一[1-3],在其发酵过程中还能积累色氨酸[4]。色氨酸是动物的必需氨基酸之一,其生理作用不可替代[5]。此外,在离子注入介导外源DNA大分子的遗传转化研究中,异常汉逊酵母菌常作为外源基因的受体菌[6-7]。
转录组对于解释基因组的功能元件、揭示细胞和组织的分子成分以及对发育和进化的理解具有重要作用。近年来,转录组测序(RNA-seq)技术成熟,测序数据具有高度重现性,在技术重复方面几乎没有系统差异,同时具有信噪比高、分辨率高、应用范围广等优势,正成为研究基因表达水平、新基因的检测及注释的重要手段[8]。
本研究基于RNA-seq,利用生物信息学方法分析异常汉逊酵母菌不同发酵时期的差异表达基因的信息及其功能,以期为人们深入认识异常汉逊酵母菌的基因表达与调控及其色氨酸代谢机制提供线索。
1 材料与方法
1.1 菌株
异常汉逊酵母(Hansenula anomala),来源于中国普通微生物菌种保藏管理中心(编号2.340,产色氨酸),由陕西科技大学食品与生物工程学院功能微生物研究室保存。
1.2 培养基
YPD斜面培养基[9](g):酵母膏10,蛋白胨20,葡萄糖20,琼脂粉10,蒸馏水1 000 mL。
液体培养基(g):一水葡萄糖100,NaNO3 10,酵母膏粉 5,K2HPO4·3H2O 1,MgSO4·7H2O 0.5,蒸馏水1 000 mL,用浓度100 g/L的NaOH调pH至7.0。
1.3 试验方法
将YPD斜面培养基上培养48 h的新鲜菌种接种至液体培养基中,置摇床230 r/min、28~30 ℃发酵96 h。每间隔24 h取样(含0 h),于4 ℃、8 000×g离心5 min,充分弃上清,迅速将菌体置于液氮速冻保存。本研究设置3个生物学重复,每个生物学重复的0、24、48、72、96 h的样品均设置6个技术重复。
分别制备各发酵时间点菌体样本的mRNA,并应用Illumina进行RNA-seq测序,每个样本的测序数据量5G。将获得的每个样本的转录组文库经过CASAVA碱基识别分析转化为原始测序序列(raw data),再经过去除接头和过滤低质量reads处理,所获得的RNA序列(clean data),作为本研究的基本数据。同时,制备异常汉逊酵母菌基因组DNA,并应用PacBio单分子测序技术进行全基因组De novo测序,所获得的全基因组DNA序列,作为RNA-seq的参考基因组。
根据RNA-seq规程[10],选取HISAT2方法[11]和HTSeq方法[12]分别作为异常汉逊酵母不同发酵时间点差异表达基因的表达量的比对分析方法和FPKM(fragments per kilobase of transcript per million fragments mapped)定量方法。
本研究应用HTSeq方法对差异表达基因的表达量进行FPKM定量和count定量,对表达基因通过BLAST2GO[13]和KAAS(KEGG automatic annotation server)[14]分别比对到GO(gene orthology)数据库和KEGG(kyoto encyclopedia of genes and genomes)数据库,利用DESeq2方法[15]对定量结果进行差异表达分析并获得差异表达基因列表,再通过GOSeq方法[16]采取超几何分布样本抽取方法进行差异表达的基因功能的GO富集和KEGG代谢通路(KEGG pathway)富集。
2 结果与分析
2.1 差异表达基因(DEGs)筛选
利用HISAT2方法将RNA序列比对到异常汉逊酵母的基因组后得到sam数据,比对结果均高于90%,表明相关实验不存在污染。
以FC(fold change,差异表达倍数)和p-adjusted(校正后的概率P值)作为衡量各发酵时间点基因表达的差异程度及显著性的尺度,以FC>2(|log2FC|>1)、Padj<0.05判定基因是否在样本间存在差异表达。
通过各发酵时间点的表达基因的相互对比,获得的DEGs如图1所示。各发酵时间点与其相邻的上一个时间点相比,发酵24、48、72、96 h的上调表达的基因数量分别为585、487、154、615,下调表达的基因数量分别为1 112、725、5、245;与0 h相比,发酵48、72、96 h的上调表达基因数量分别为701、639、639,下调表达的基因数量分别为1 315、1 211、1 023;与24 h相比,发酵72 h和96 h的上调表达基因数量分别为581和689,下调表达基因数量分别为587和471(图1)。
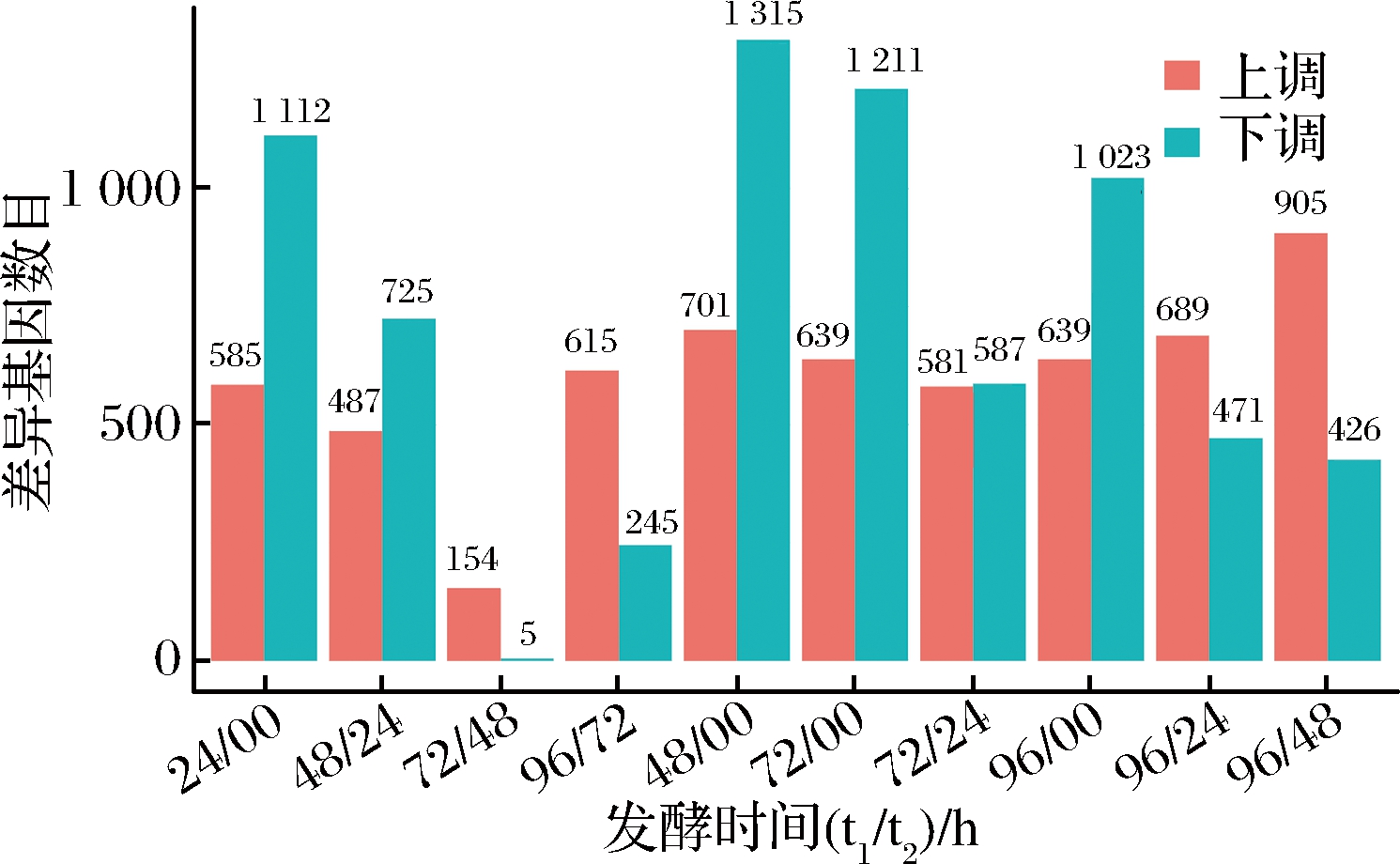
图1 异常汉逊酵母液体发酵不同时间点间的差异表达基因
Fig.1 Differential expressed genes of different culture time at Hansenula anomala liquid culture
注:t1/t2表示以t1时间样本为处理组、t2为对照组。
从各发酵时间点与其相邻的上一个时间点的比较来看,无论是上调还是下调的差异表达基因,其数量从24~72 h呈下降趋势,然后增加。发酵96 h时,其上调的差异表达基因数量高于24 h,而下调的差异表达基因数量只有24 h的22.03%。
从各发酵时间点与0 h的比较来看,下调表达的基因数量均超过1 000,其数量在24~48 h上升,然后下降;而上调表达的基因数量在24~48 h呈上升趋势,48~72 h下降,然后趋于平稳。这些分析结果表明,异常汉逊酵母从新鲜斜面接入液体发酵时(0 h),其基因表达水平已进入一次峰值,在液体发酵96 h时再进入新一轮基因表达峰值。
2.2 差异表达基因功能的GO富集分析
通过FPKM表达矩阵得到异常汉逊酵母菌表达的基因数目为6 086个,与GO数据库的比对结果显示,共有3 455个基因对应到3 686种GO功能。
将异常汉逊酵母菌差异表达基因通过GO功能富集(P<0.05)得到的功能与GO数据库(http://www.geneonthology.org)进行比对,寻找其二级分类,发现其共涉及43个生物学过程(BP,biological process)、13个细胞组分(CC,cellular component)和13个分子功能(MF,molecular function)。在生物学过程的功能分类中,差异表达基因的功能主要涉及细胞过程(cellular process)、代谢过程(metabolic process)、细胞成分(cellular component of organization or biogenesis);在细胞组分的功能分类中,差异表达基因的功能主要涉及细胞(cell)、细胞成分(cell part)、蛋白质复合物(protein-containing complex);在分子功能的分类中,差异表达基因功能主要涉及催化活性(catalytic activity)、结合(binding)、转录调节活性(transcription regulator activity)。
对异常汉逊酵母菌各个发酵时间点均含有GO富集的差异基因的功能进行分析,结果表明,rDNA沉默(GO: 0000183)、过氧化物酶(GO: 0002215)、ADP磷酸化(GO: 0006119)、蛋白质接合作用(GO: 0045116)、质子运输(GO: 0015992)、对热刺激反应(GO: 0009408)、胞质小核糖体亚基(GO: 0022627)、层黏连蛋白-3复合物(GO: 005608)、蛋白质结合(GO: 0030674)均在24 h和48 h下调富集、72 h和96 h上调富集,其变化趋势均为从0~48 h下降,48~96 h上升(图2)。

图2 异常汉逊酵母液体发酵的各时间点差异表达基因的GO功能类别
Fig.2 Differential gene GO enrichment in different culture time at Hansenula anomala liquid culture
注:t1/t2表示以t1时间样本为处理组、t2为对照组。
除去发酵时间连续的GO富集结果之后,对剩余GO功能类别进行分析,上调富集的GO功能大部分与细胞分裂有关,其中凋亡DNA片段化(GO: 0006309)、中心体循环(GO: 0007098)、DNA双链断裂处理(GO: 0000729)、DNA双链断裂修复(GO: 0006302)、微管解聚负调控(GO: 0007027)、正向调节弹性蛋白(GO: 0051544)、蛋白质泛素化的调节(GO: 0031396)、内膜的组成成分(GO: 0031356)在96 h对比0 h或24 h时均上调富集。将其余富集的GO Term与GO数据库比对寻找其二级分类发现功能主要分布在催化活性和代谢过程,涉及催化活性相关的有α-1,2-甘露糖基转移酶(GO:000026)、ADP-L-甘油基-d-甘露-庚糖-6-差向异构酶(GO:0008712)、谷氨酸合酶(GO:0016040)、ATP酶(GO:0016887)、3-羟基-N-甲基-(S)-丙氨酸4-O-甲基转移酶(GO:0030784)、[核酮糖二磷酸羧化酶]-赖氨酸N-甲基转移酶(GO:0030785)、质子转运ATP酶(GO:0046961)、邻二羟基香豆素7-O-葡糖基转移酶(GO:0047208);而和代谢相关的有三环化合物rRNA转录物的内切核酸切割(GO:0000449)、ITS1内核裂解性裂解(GO:0000464)、外切核酸修剪(GO:0000465)、产生成熟的LSU-rRNA(GO:0000468)、来自四顺反子rRNA转录物的SSU-rRNA成熟(GO:0000474)、5S rRNA的成熟(GO:0000481)、来自四顺反子rRNA转录物的5S rRNA成熟(GO:0000482)、腺嘌呤分解代谢(GO:0006146)、鸟嘌呤分解代谢(GO: 0006147)、dUMP生物合成(GO: 0006226)、蛋白水解(GO:0006508)、谷氨酸生物合成(GO:0006537)、异亮氨酸分解代谢(GO:0006550)、亮氨酸分解代谢(GO:0006552)、赖氨酸分解代谢(GO:0006554)、脯氨酸代谢(GO:0006560)、色氨酸代谢(GO:0006568)、缬氨酸分解代谢(GO:0006574)、脂肪酸生物合成(GO:0006633)、谷胱甘肽代谢(GO:0006749)、脂多糖生物合成(GO:0009103)、生物碱生物合成(GO:0009821)、苯甲酸盐代谢(GO:0018874)、去甲肾上腺素生物合成(GO:0042421)、去甲肾上腺素分解代谢(GO:0045422)、转录调节(GO:0045449)。
2.3 KEGG富集的差异表达基因的功能
将异常汉逊酵母菌表达的6 086个基因与KEGG数据库进行比对,结果显示,共有3 275个基因注释到2 865种功能。
KEGG富集(FDR<0.05)分析结果显示,DEGs共涉及50条通路,其中与氨基酸代谢相关的通路有11条,包括苯丙氨酸代谢(Phenylalanine metabolism,ko00340)、色氨酸代谢(Tryptophan metabolism,ko00360)、芳香族氨基酸(苯丙氨酸、酪氨酸和色氨酸)的生物合成(Phenylalanine,tyrosine and tryptophan biosynthesis,ko00380)。
异常汉逊酵母菌具有积累色氨酸的代谢特性,对其色氨酸的代谢通路的分析表明,参与色氨酸合成的为色氨酸合成酶(EC: 4.2.1.20)、参与其分解的为色氨酸氨基转移酶(EC: 2.6.1.27)和吲哚胺2,3-二氧化酶(EC: 1.13.11.52)。由酶的表达图(图3)可知,色氨酸的合成速率在0~24 h上升,随后下降;而分解速率从0~48 h上升,随后下降。
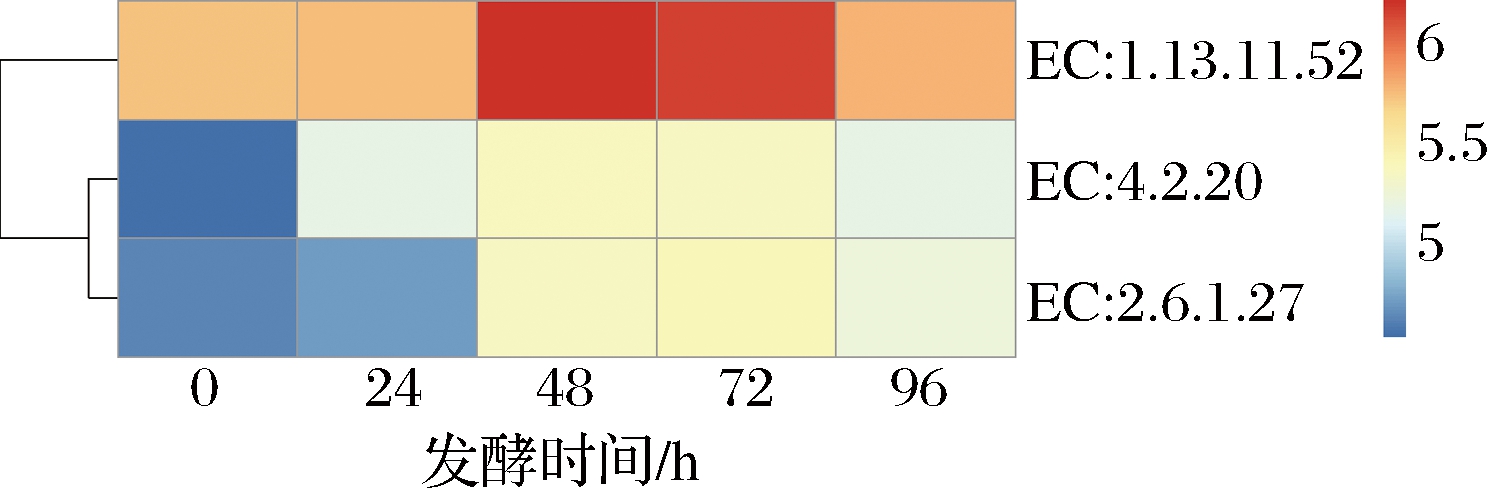
图3 异常汉逊酵母液体的各时间点色氨酸代谢相关相关酶
Fig.3 The synthesis for metallic of tryptophan at Hansenula anomala liquid culture
对异常汉逊酵母菌各个发酵时间点均含有KEGG富集的差异基因的功能进行分析,结果表明,核糖体(ribosome,ko03010)的代谢趋势为0 h至 48 h 下降,48 h至96 h上升,这与各个发酵时间点均含有的GO富集的差异基因的功能分析结果一致,说明了异常汉逊酵母菌的合成代谢强度在0 h至48 h下降,48 h至96 h上升。
3 讨论
本研究通过Illumina测序平台首次对异常汉逊酵母5个发酵时间点共15个生物学样本进行转录组测序,共获得6 086个表达基因。对5个时间点的异常汉逊酵母表达谱的分析显示,其在生长过程中的基因表达存在显著差异。与0 h相比,其他发酵时间点均存在大量下调基因,这是由于接入液体培养基的新鲜斜面菌种的代谢旺盛,导致0 h之后的表达基因出现大量下调。另外,本研究在选取差异表达的基因时,差异表达倍数设置为2,使得时间上连续的基因功能富集的种类较少。
差异表达基因的变化趋势与GO功能富集结果的关联分析结果表明,下调表达基因数量24~48 h上升,然后下降;而上调表达的基因数量在24~48 h呈上升趋势,48~72 h下降,然后趋于平稳。0~48 h下调的基因富集功能涉及多种代谢过程和催化活性,而上调基因功能富集的只有蛋白水解和信号识别粒子;48~96 h上调的基因功能也涉及多种代谢过程和催化活性,而其下调基因的功能富集只有谷胱甘肽代谢和脂肪调节酶。可能由于基因表达功能较多,导致即使存在大量差异表达基因,功能富集项也较少,而未富集的基因功能可能与细胞繁殖与衰老相关。异常汉逊酵母菌在细胞整体转录组变化和细胞数量变化的共同作用下,导致差异基因表达结果与酵母菌细胞的生长曲线存在差别,其内在机制有待进一步研究。
在异常汉逊酵母菌的差异表达基因功能的GO富集结果中,ADP磷酸化和质子运输的变化趋势代表其代谢强度的变化[17],即菌体代谢强度0~48 h下降,48~96 h上升。rDNA沉默的变化趋势和代谢强度变化趋势一致,说明菌体在48 h时,细胞已储存足够多的核糖体蛋白用于后续蛋白质合成,因此在48 h之后rDNA沉默也呈上升趋势。除此之外,其他与合成代谢产物前体相关的富集的GO功能类别,其变化趋势与核糖体蛋白作用速度的变化一致,这表明48 h后菌体代谢产物的合成速度上升。
在96 h上调的微管解聚基因的负调控与酵母菌的有丝分裂相关[18],弹性蛋白基因的正向调节与细胞有丝分裂末期形成细胞膜的组成成分相关[19];而凋亡DNA片段化的基因表达上调表明96 h时细胞死亡进入一个峰值,这与细胞生长曲线上的细胞进入衰老期相符;DNA双链断裂处理、修复上调说明大部分细胞进入分裂间期,表明其将进入第二轮基因表达。KEGG富集结果中的核糖体变化趋势与此一致,可为异常汉逊酵母菌代谢相关研究提供依据。
在异常汉逊酵母菌的差异表达基因功能的KEGG富集的通路中,合成色氨酸的酶为色氨酸合成酶(EC:4.2.1.20);而分解色氨酸的酶分别为色氨酸氨基转移酶(EC: 2.6.1.27)和吲哚胺2,3-二氧化酶(EC: 1.13.11.52)。基因表达矩阵的分析结果说明了色氨酸的合成速率在0~72 h下降,随后上升;而分解速率从0~48 h上升,随后下降。本研究从基因表达角度印证了此前关于酵母菌发酵色氨酸的相关实验[20]。
差异表达基因功能的GO和KEGG的富集结果显示,在本研究条件下,异常汉逊酵母的代谢强度在0~48 h下降,48~96 h上升,并且在96 h时其转录、复制和翻译等基因表达丰度较高,具有较强的代谢活动和遗传信息处理能力。本研究结果将为异常汉逊酵母菌的分子育种及其代谢调控研究提供理论依据。
参考文献
[1] COMITINI F,GOBBI M,DOMIZIO P,et al. Selected non-Saccharomyces wine yeasts in controlled multistarter fermentations with Saccharomyces cerevisiae [J]. Food Microbiology,2011,28(5): 873-882.
[2] LEE P R,CHONG I S M,YU B,et al. Effects of sequentially inoculated Williopsis saturnus and Saccharomyces cerevisiae on volatile profiles of papaya wine [J]. Food Research International,2012,45(1): 177-183.
[3] 刘景,王欣,辛红鸿,等. 非酿酒酵母的筛选及其发酵低醇苹果汁的研究[J]. 中国食品学报,2017,17(1): 134-140.
[4] EBIHARA Y,NIITSU N,TERUI G. Fermentative production of tryptophan from indole by Hansenula anomala[J]. Ferment Technol,1969,47(12): 733-738.
[5] WAKASA K,ISHIHARA A. Metabolic engineering of the tryptophan and phenylalanine biosynthetic pathways in rice [J]. Plant Biotechnology,2009,26(5): 523-533.
[6] 毛培宏,吕杰. 离子束重组酵母菌Ar_Han0458的RAPD与SSH的初步研究[J]. 基因组学与应用生物学,2015,34(3): 449-453.
[7] LU Jie,JIN Xiang,MAO Pei-hong,et al. Transfer of Ephedra genomic DNA to yeasts by ion implantation [J]. Appl Biochem Biotechnol,2009,158(3): 571~581
[8] WANG Z,GERSTEIN M,SNYDER M. RNA-Seq: A revolutionary tool for transcriptomics [J]. Nature reviews genetics,2009,10(1): 57.
[9] BURKE D,DAWSON D,STEARNS T. Methods in yeast genetics: A cold spring harbor laboratory course manual [M]. New York: Cold Spring Harbor Lab Press,2000: 171-172.
[10] SAHRAEIAN S M E,MOHIYUDDIN M,SEBRA R,et al. Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis [J]. Nature communications,2017,8(1): 59.
[11] KIM D,LANGMEAD B,SALZBERG S L. HISAT: A fast spliced aligner with low memory requirements [J]. Nature methods,2015,12(4): 357.
[12] ANDERS S,PYL P T,HUBER W. HTSeq-a Python framework to work with high-throughput sequencing data [J]. Bioinformatics,2015,31(2): 166-169.
[13] GÖTZ S,ARNOLD R,SEBASTI N-LE
N-LE N P,et al. B2G-FAR,a species-centered GO annotation repository [J]. Bioinformatics,2011,27(7):919-924.
N P,et al. B2G-FAR,a species-centered GO annotation repository [J]. Bioinformatics,2011,27(7):919-924.
[14] MORIYA Y,ITOH M,OKUDA S,et al. KAAS: An automatic genome annotation and pathway reconstruction server [J]. Nucleic acids research,2007,35:W182-W185.
[15] LOVE M I,HUBER W,ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2 [J]. Genome biology,2014,15(12): 550.
[16] YOUNG M D,WAKEFIELD M J,SMYTH G K,et al. Gene ontology analysis for RNA-seq: Accounting for selection bias [J]. Genome biology,2010,11(2): R14.
[17] MALDONADO E N,LEMASTERS J J. ATP/ADP ratio,the missed connection between mitochondria and the Warburg effect[J]. Mitochondrion,2014,19: 78-84.
[18] WEI San-hua,LIN Fang,WANG Xi,et al. Prognostic significance of stathmin expression in correlation with metastasis and clinicopathological characteristics in human ovarian carcinoma[J]. Acta Histochemica,2008,110(1): 59-65.
[19] BRAND O I D S L,OLIVEIRA-MORAES H M D S,SOUZA M C M D,et al. Elastin increases biofilm and extracellular matrix production of Aspergillus fumigatus[J]. Brazilian Journal of Microbiology,2018,49(3): 675-682.
O I D S L,OLIVEIRA-MORAES H M D S,SOUZA M C M D,et al. Elastin increases biofilm and extracellular matrix production of Aspergillus fumigatus[J]. Brazilian Journal of Microbiology,2018,49(3): 675-682.
[20] 李玲阁,唐任天,傅妙福,等.色氨酸发酵的初步研究[J].微生物学报,1975,15(3): 212-216.