耐酸乳酸杆菌物种特异性引物设计及其在古井贡酒窖泥酒醅质量评价中的初步应用
刘倩倩1,李俊薇2,曹润洁2,张会敏2,何宏魁2*,李安军2,张治洲1*
1(哈尔滨工业大学(威海) 海洋科学与技术学院,山东 威海,264209)2(安徽古井贡酒股份有限公司,安徽 毫州,236800)
摘 要 古井贡酒的酿造过程中,窖泥和酒醅中发现大量耐酵乳杆菌(Lactobacillus acetotolerans),对该菌快速特异性检测非常重要。该文利用L.acetotolerans菌及大量Lactobacillus类似菌种的全基因组序列与PRIMER-BLAST方法,设计了几对L.acetotolerans特异性引物,其中2对经过系列验证具有较好的特异性,可结合QPCR技术对不同质量和不同发酵阶段的窖泥进行L.acetotolerans含量的快速检测。结果表明,古井贡酒窖泥中L.acetotolerans 含量总体明显低于酒醅,老窖泥中L.acetotolerans含量总体明显低于新窖池窖泥;而酒醅中老厂区和新厂区L.acetotolerans含量平均来看都很高,但样本之间存在明显波动。所用L.acetotolerans特异性引物可对浓香型白酒发酵样本中L.acetotolerans的相对含量测定并辅助评估窖泥酒醅质量。
关键词 Lactobacillus acetotolerans;古井贡酒;特异性引物;窖泥;酒醅
DOI:10.13995/j.cnki.11-1802/ts.019007
第一作者:硕士研究生(何宏魁高级工程师和张治洲教授为共同通讯作者,E-mail:hehongkui@gujing.com.cn,zhangzzbiox@hitwh.edu.cn)。
基金项目:国家自然科学基金(31071170);山东省自然科学基金(ZR2018MC002);GujingTribute Fund (201601)
收稿日期: 2018-10-09,改回日期:2018-11-07
白酒是我国传统的发酵食品,有着悠久的历史,从古至今一直备受青睐[1]。中国传统白酒是以富含淀粉质的粮谷类为原料,以酒曲为糖化剂,采用固态、半固态或液态发酵,经蒸馏、贮存和勾调而成的乙醇饮料[2]。由于白酒的酿造厂区建设时间以及地域的差异,不同窖池中酸类物质组成成分和含量有较大差异[3],其中乳酸杆菌(Lactobacillus)的含量在新窖池和老窖池之间存在较大差异[3-5];该菌属的含量超过一定阈值后将严重影响酒质[6]。在浓香型白酒领域,乳酸杆菌及其具体系列物种的生物学功能、尤其是其在固态发酵各个阶段中的具体功能,尚未得到充分的探索。
在白酒生产中,乳酸菌在白酒口味及香味上发挥很重要的作用。乳酸菌能发酵糖类生成乳酸,在酒醅内通过酯化作用产生乳酸乙酯,乳酸乙酯被蒸入酒中,使白酒具有独特的香味[7]。乳酸杆菌也是窖泥中主要的微生物之一,往往与丁酸菌、己酸菌、甲烷菌、硫酸盐还原菌、硝酸盐还原菌等菌类共存“共生”,当乳酸菌混入入窖的料醅后,会争夺葡萄糖等糖类物质为发酵原料而生成乳酸。有的乳酸菌还同时生成少量的乙酸、乙醇、丙酸、丁酸、丙酮酸等物质[8],共同调节酒体风味。在酒类发酵过程中由于酒精含量等环境压力使微生物的生长受限,少数种类的细菌在这种条件下保持活力,可能被认定为酒类腐败微生物。L.acetotocerans被证实是一种培养较困难,且经常出现在中国南方的酿酒厂中的一种微生物;它具有产生乳酸的能力[9]。一方面,白酒的生产需要适量的乳酸;另一方面,这种菌含量过多时带来的综合效果严重影响酒类口感[6]。啤酒类发酵对于该菌的研究也不在少数,乳酸杆菌是啤酒发酵过程中常见的菌种[10-11]。我国四川理工学院四川省酿酒生物技术及应用重点实验室通过对四川某浓香型白酒股份有限公司20年窖龄的窖池上、中、下取样后分析得到,其中优势菌群Clostridium diolis及Lactobacillus acetotolerans在上、中、下层窖泥中所占比例分别为34.9%和39.8%、25.2%和20.6%以及23.8%和36.6%[12]。2015年Elsevier B.V.公司发表了一篇文章[13],从日本清酒中分离出Lactobacillus acetotolerans RIB 9124 (NBRC 13120)株菌,在这篇报道中首次公开了L. acetotolerans strain的全基因组序列,并对其进行命名[9]。该文章公开的L. acetotocerans全基因组序列为本实验中物种特异性引物设计奠定了基础。
根据古井贡酒窖泥和酒醅样本中已获悉的菌群信息[14-18],我们发现其中L. acetotocerans在酒醅中占主导地位,含量非常高,且明显高于窖泥中L. acetotocerans的含量。国内外对该乳酸杆菌的研究已经很多,主要针对微生物的功能性研究,而通过特异性引物对该菌含量的快速检测方法报道非常少。另外,数据库中显示Lactobacillus这个属不同的物种已经有70多个基因组全序列,这为本研究中设计L. acetotocerans的物种特异性引物提供了更多的背景信息,而PRIMER-BLAST这个引物设计平台可以把这70多个全基因组全部考虑进去,这样设计的引物就会有更好的特异性,可以用于窖泥酒醅等发酵样本中L. acetotocerans含量的快速测定,其数据可为窖泥等酿酒样本质量评估提供参考。
1 材料与方法
1.1 材料与仪器
该试验涉及的材料取自安徽省亳州市古井贡酒公司不同等级窖池的12个窖泥样本和12个酒醅样本。
利用基因组提取试剂盒提取上述样本的宏基因组以及纯菌种的基因组。基因组DNA经紫外分光光度计测定浓度后统一稀释为10 ng/μL用作PCR的模板以及QPCR的定量模板。
所用的纯菌菌种均购自中国微生物菌种保藏管理委员会普通微生物中心(China General Microbiological Culture Collection Center,CGMCC)。
Step-One Real-Time PCR系统(QPCR)、HC-3018R高速冷冻离心机,安徽中科中佳科学仪器有限公司;JY-300C电泳仪,北京君意东方电泳设备有限公司;WD-9413凝胶成像仪,北京六一仪器厂;TP600(TaKaRa)Q-PCR仪、Ezup柱式基因组提取试剂盒(B518251),上海生工;NPK02、NPK62 PCR试剂盒,威海晓东生物;pMD19-T kit TA克隆试剂盒,Takara。
1.2 利用Primer-Blast设计特异性引物
检索EBI中Genome数据库(http://www.ebi.ac.uk/genomes/),检索到70个除L. acetotocerans外的乳杆菌菌种,表1用作引物设计的背景序列,利用Primer-Blast[19]对L. acetotocerans的全基因组序列设计物种特异性引物,并用Blast对已设计的引物进行验证,筛选出2对优质引物。设计L. acetotocerans物种的特异性引物的目的一般基于两个,一是鉴定该物种,以及检测某一生物群落中是否包含该物种,二是确定该物种在整个微生物群落中的丰度[20-22]。
表1 EBI数据库中71个有全基因组序列的乳酸杆菌菌株信息
Table 1 71 Lactobacillus strains with whole genome sequences found in the EBI database

菌株名称检索号菌株名称检索号1Lactobacillus acetotolerans DNA strain: NBRC 13120AP01480837Lactobacillus hokkaidonensisAP0146802Lactobacillus acidophilus 30SCCP00255938Lactobacillus johnsonii DPC 6026CP0024643Lactobacillus acidophilus La-14CP00592639Lactobacillus johnsonii FI9785FN2984974Lactobacillus acidophilus NCFMCP00003340Lactobacillus johnsonii N6.2CP0068115Lactobacillus amylovorus GRL1118CP00260941Lactobacillus johnsonii NCC 533AE0171986Lactobacillus amylovorus GRL 1112CP00233842Lactobacillus kefiranofaciens ZW3CP0027647Lactobacillus brevis plasmid pLB925A01AB37033443Lactobacillus mucosae LM1CP0110138Lactobacillus brevis plasmid pLB925A02AB37033544Lactobacillus paracasei N1115CP0071229Lactobacillus brevis plasmid pLB925A03AB37033645Lactobacillus paracasei subsp. paracasei 8700:2CP00239110Lactobacillus brevis plasmid pLB925A04AB37033746Lactobacillus paracasei subsp. paracasei JCM 8130AP01254111Lactobacillus brevis ATCC 367CP00041647Lactobacillus plantarum 16CP00603312Lactobacillus brevis KB290AP01216748Lactobacillus plantarum DOMLaCP00440613Lactobacillus buchneri CD034CP00304349Lactobacillus plantarum JDM1CP00161714Lactobacillus buchneri NRRL B-30929CP00265250Lactobacillus plantarum subsp. plantarum P-8CP00594215Lactobacillus casei 12ACP00669051Lactobacillus plantarum subsp. plantarum ST-ⅢCP00222216Lactobacillus casei ATCC 334CP00042352Lactobacillus plantarum WCFS1AL93526317Lactobacillus casei BD-IICP00261853Lactobacillus plantarum ZJ316CP00408218Lactobacillus casei BL23FM17714054Lactobacillus reuteri DSM 20016CP00070519Lactobacillus casei LC2WCP00261655Lactobacillus reuteri I5007CP00601120Lactobacillus casei LOCK919CP00548656Lactobacillus reuteri JCM 1112AP00728121Lactobacillus casei str. ZhangCP00108457Lactobacillus reuteri SD2112CP00284422Lactobacillus casei subsp. casei ATCC 393AP01254458Lactobacillus reuteri TD1CP00660323Lactobacillus casei W56HE97076459Lactobacillus rhamnosus ATCC 8530CP00309424Lactobacillus crispatus ST1FN69203760Lactobacillus rhamnosus GG ATCC 53103AP01154825Lactobacillus delbrueckii subsp. bulgaricus 2038CP00015661Lactobacillus rhamnosus GGFM17932226Lactobacillus delbrueckii subsp. bulgaricus ATCC 11842=JCM 1002CR95425362Lactobacillus rhamnosus Lc 705FM17932327Lactobacillus delbrueckii subsp. bulgaricus ATCC BAA-365CP00041263Lactobacillus rhamnosus LOCK900CP00548428Lactobacillus delbrueckii subsp. bulgaricus ND02CP00234164Lactobacillus rhamnosus LOCK908CP00548529Lactobacillus gasseri 130918CP00680965Lactobacillus ruminis ATCC 27782CP00303230Lactobacillus gasseri ATCC 33323 = JCM 1131CP00041366Lactobacillus sakei subsp. sakei 23KCR93650331Lactobacillus helveticus strain KLDS1.8701CP00990767Lactobacillus salivarius strain JCM1046CP00764632Lactobacillus helveticus CNRZ32CP00208168Lactobacillus salivarius CECT 5713CP00203433Lactobacillus helveticus DPC 4571CP00051769Lactobacillus salivarius UCC118CP00023334Lactobacillus helveticus H9CP00242770Lactobacillus sanfranciscensis TMW 1.1304CP00246135Lactobacillus helveticus H10CP00242971Lactobacillus sp. wkB8CP00953136Lactobacillus helveticus R0052CP003799
1.3 利用普通PCR检测引物特异性
利用聚合酶链式反应(polymerase chain reaction,PCR)对两对引物的特异性进行验证。分别采用12个窖泥基因组、12个酒醅基因组DNA作为实验模板, PCR反应体系为:NPK02 buffer(2×) 6 μL,物种特异性引物LA2 (LA3)1.5 μL,模板0.5 μL,Taq酶0.3 μL,ddH2O 3.7 μL。PCR循环参数:94 ℃、4 min; 94 ℃、30 s,60 ℃(引物LA2) 或63 ℃(引物LA3)40 s, 72 ℃、40 s,37个循环;72 ℃, 2 min。取8 μL 反应液进行10 g/L琼脂糖凝胶(120V,20 min)电泳。
1.4 利用PCR产物测序进行引物特异性评估
上述PCR产物可以直接用于TA克隆。pMD19-T载体 0.3 μL,LA2(或LA3)扩增的PCR产物0.5 μL,灭菌ddH2O 4.2 μL,Solution I 5 μL,混匀,室温连接1小时后放入冰浴,加入冰冷的30 μL大肠杆菌感受态细胞DH5α并混匀,冰浴放置40 min。再加入1 mL普通LB培养基溶液,于37 ℃恒温振荡器中培养活化1 h。将X-gal 40 μL(10 mg/mL),IPTG 40 μL(10 mg/mL), 160 μL dH2O混匀,涂布于LBA固体培养基,待其干燥,将活化的菌液200 μL继续涂布于X-gal和IPTG干燥后的LBA固体培养基上。37 ℃ 恒温过夜培养。筛选单克隆(图1)。将筛选的单克隆37 ℃恒温过夜培养后,送至生工生物工程(上海)股份有限公司进行单菌落测序。利用NCBI网站的BLAST工具,比对测序结果,根据测序结果鉴定筛选的单克隆是否含有L. acetotolerans的特异性DNA序列。
A-LA2;B-LA3
图1 利用两对引物LA2和LA3扩增产物得到的TA克隆
Fig.1 TA clones of PCR amplicons by primer pair LA2 and LA3
1.5 利用纯Lactobacillus菌株验证L. acetotolerans引物特异性
涉及到的纯菌株为:(P1)耐酸乳杆菌Lactobacillus acetotolerans(实验室自备);(P2)植物乳杆菌Lactobacillus plantarum (CICC20265);(P3)发酵乳杆菌Lactobacillus fermentum (CICC22808);(P4)干酪乳杆菌Lactobacillus casei(1.320 6);(P5)嗜酸乳杆菌Lactobacillus acidophilus(1.334 2);(P6)德式乳杆菌乳亚种Lactobacillus delbrueckii subsp. Lactis(1.262 5)。
1.6 QPCR测定
使用筛选出来的两对特异性引物对12个窖泥样本和12个酒醅样本基因组DNA做QPCR定量实验。根据标准曲线、熔解曲线、指数增长曲线,以及copy数计算来对L. acetotolerans的含量做初步检测及综合质量评估。以用于测序的PCR纯化产物连续稀释10倍作为标准。阴性对照、每个用于制作标准曲线的标准样本以及待测样本均重复3次。使用的PCR扩增体系为NPK62(含荧光染料)6 μL,特异性引物1.5 μL,Taq酶0.25 μL,基因组模板4.25 μL。QPCR条件为94 ℃,4 min (94 ℃,30 s;57 ℃,40 s;72 ℃,40 s),42个循环,72 ℃,2 min。
2 结果与分析
2.1 引物设计与分析
通过对L. acetotolerans全基因组进行物种特异性引物设计,在1435181处设计上游引物LA2F:5′-GTG CAG CTT AAG GGC TTC CT-3′(20 bp),在1435320处设计下游引物LA2R:5′- ATC TGT GCA GAT TGT TCC CTT-3′(21bp),扩增片段长度194 bp;在1702025处设计上游引物LA3F:5′- AGA GTG TCC CGA GTA GTC CC-3′(20bp),在1702133处设计下游引物LA3R:5′- AAC TGC TCA AGA AGG GAC CG-3′(20bp),扩增片段长度为150 bp。
2.2 PCR扩增兼TA克隆/测序鉴定引物特异性
使用TA克隆技术手段对古井实际样本做了特异性引物验证并挑选阳性克隆,并对阳性克隆进行单菌落测序,测序结果如表2、表3所示。结果显示,对LA2 15个克隆进行测序,克隆序列均为目标序列,有效序列的成功率为100%;对LA3 14个克隆进行测序,克隆序列均为目标序列,有效序列的成功率也是100%。
2.3 利用乳杆菌纯菌株进一步验证引物特异性
以LA2、LA3这两对物种特异性引物分别对上述6株乳酸杆菌纯菌基因组(样本顺序依次为A,B,C,D,E,F)进行PCR扩增实验,结果如图2,两对引物均只从P1样本(L. acetotolerans)中扩增出来目的条带,说明引物的特异性较好。
表2 引物LA2 TA克隆Blast结果
Table 2 Primer LA2 TA cloning blast results

菌落号描述相似度/%序列号222-1Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-2Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-3Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312099AP014808.1222-4Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-5Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-6Lactobacillus acetotoleransDNA, complete genome, strain: NBRC 13120100AP014808.1222-7Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312099AP014808.1222-8Lactobacillusacetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-9Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-10Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-11Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312093AP014808.1222-12Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-13Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-14Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1222-15Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1
表3 引物LA3 TA克隆Blast结果
Table 3 Primer LA3 TA clone Blast results

菌落号描述相似度/%序列号333-1Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1333-2Lactobacillus acetotoleransDNA, complete genome, strain: NBRC 13120100AP014808.1333-3Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-4Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312097AP014808.1333-5Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-6Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-7Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 13120100AP014808.1333-8Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-9Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-10Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-11Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-12Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312097AP014808.1333-13Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312098AP014808.1333-14Lactobacillus acetotolerans DNA, complete genome, strain: NBRC 1312097AP014808.1
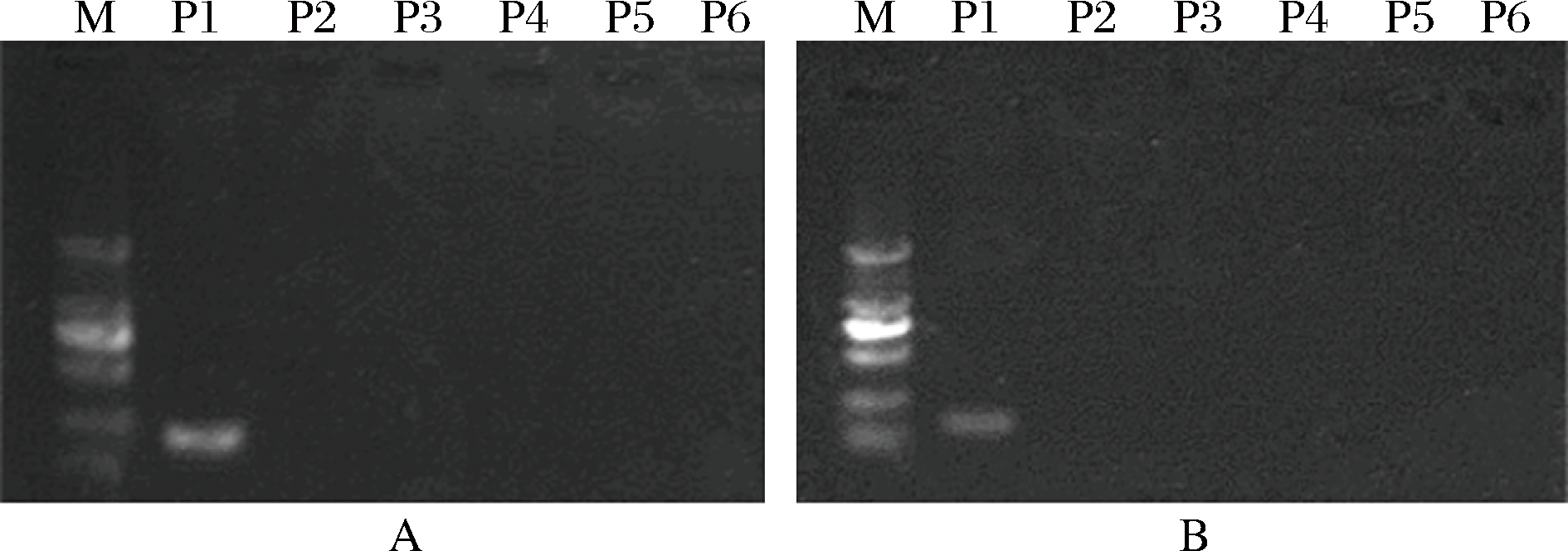
图2 L.a菌特异性引物LA2(A)和LA3(B)对6株乳酸杆菌纯菌基因组的PCR扩增实验
Fig.2 PCR amplification using L.a-specific primer LA2 (A) orLA3 (B) and genomic DNA of 6 pure Lactobacillus strains
注:M-DL2000 DNA marker (2 000, 1 000, 750,500,250和100 bp),图3同。
2.4 使用2对特异性引物对窖泥和酒醅中L.acetotolerans进行检测
PCR扩增结果(图3)显示,在相同的扩增条件下(其中PCR热循环数为37),普通窖泥的条带较优质窖泥更清晰明亮,普通酒醅的条带较优质酒醅更清晰明亮,酒醅条带较窖泥更清晰明亮,与理论预期一致。
A-窖泥样品LA3;B-窖泥样品LA2;C-酒醅样品LA3;D-酒醅样品LA2
图3 两对引物的普通PCR扩增结果
Fig.3 PCR results of two pairs of primers
注:A、B代表老窖池,C代表新窖池;N代表窖泥样,P代表酒醅样本。
结果为酒醅中该菌含量明显多于窖泥,普通酒醅中该菌含量明显多于优质酒醅,普通窖泥中该菌含量明显多于优质窖泥。在循环数较小的前提下,使用普通PCR可以对样本中的L.acetotolerans含量进行半定量。
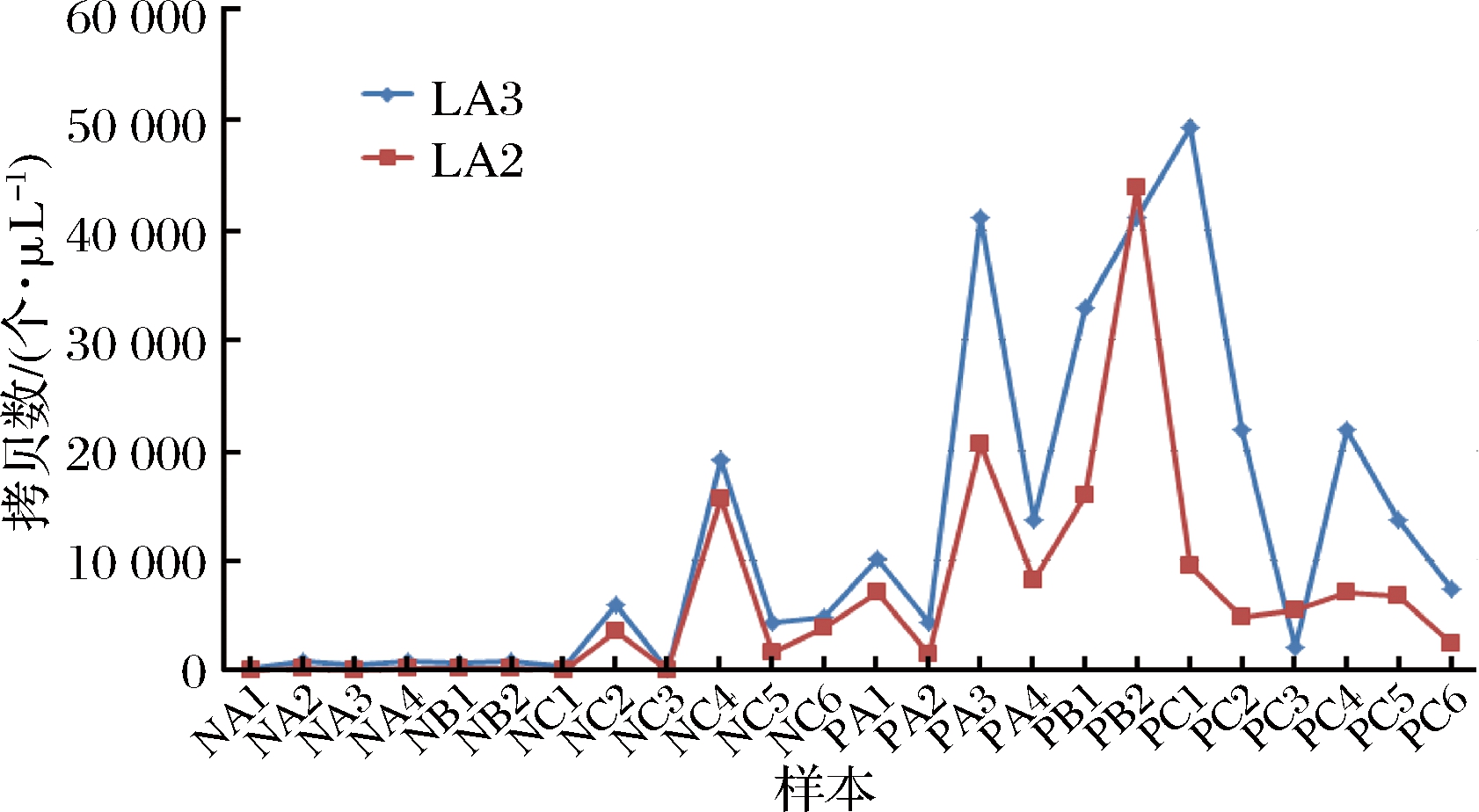
2.5 利用QPCR定量比较样本中L.acetotolerans含量的相对多寡
作为两对引物在古井发酵样本质量评估中的初步应用,将古井贡酒12个窖泥样本和12个酒醅样本进行QPCR实验,以此来对窖泥和酒醅样本中L.acetotolerans的含量进行检测,结果如图4所示。熔解曲线(未提供)均为比较集中接近的单峰,说明这两对引物特异性符合QPCR的定量要求。两对引物的定量结果总体趋势一致,即酒醅中L.acetotolerans的相对含量明显高于窖泥,尤其是远高于老窖泥。
图4 利用两对引物和QPCR测定12个窖泥样本和12个酒醅样本中菌的(相对)含量
Fig.4 QPCR quantification results of L. acetotoleransin 12 pitmud and 12 Zaopei samples
3 讨论
目前,窖泥的质量评估主要根据其色泽、气味、手感以及质地等感官特征为判别的重要指标[23],如优质老窖窖泥质感细腻,气味浓郁芳香,老化窖泥则较为干燥,容易板结成块,无芳香气味[24]。理化指标和菌群指标都可以用来衡量窖泥、醩醅等发酵样本的质量特征,以避免使用感官特征对窖泥进行实时评估与检测所对应的局限性。目前可以检测菌群特征的技术非常多,比如PCR-SSCP法可对窖泥微生物群落结构进行分析[25]。通过检测单一菌种来辅助判断发酵样本的质量好坏也是一种重要的方法,比如徐岩等设计了特异性引物可以对发酵食品中耐酸乳杆菌进行定性定量[26],但是其设计的引物与本研究设计的引物在序列内容和在L.acetotolerans基因组中的位置均不同。
本文中的QPCR测定结果表明老窖泥中L.acetotolerans的含量明显低于新窖泥,但是不是绝对的。个别新窖池的窖泥也具有很低的含量,原因尚不清楚,需要进一步观察研究。但是如果L.acetotolerans在窖泥中的含量很高则肯定不是老窖泥。图3中的NC3样本(新窖池窖泥样本)使用两个引物对扩增的结果都很低,但是应该不是取样误差带来的问题,因为每一个窖泥样本都是一个窖池内进行9点等量取样后再等量均匀混合得到的混合样本[5]。另外,个别酒醅样本利用LA2引物对测定的L.acetotolerans含量也较低,但是使用LA3测定的结果很高(如PC1样本)。不过这些不一致暂时还说明不了LA2和LA3引物对到底哪个定量更准确,可以在使用的过程中逐步确定最佳的定量引物。
高通量测序结果显示[3],古井贡酒酒醅中至少含有49种不同的乳酸杆菌(Lactobacillus),它们中间85%以上都是L.acetotolerans。酒醅中如此分布的乳酸杆菌(Lactobacullus)之间如何相互作用是一个很有趣的问题,值得研究人员思索和探究。其他种类的乳酸杆菌也可以使用相同的方法设计物种特异性引物并测定各自在发酵过程中的含量变化。本研究设计的引物以71种不同的乳酸杆菌基因组为背景,所以本文中的两对引物测定的绝大部分只是L.acetotolerans的含量,基本不包含其他乳酸杆菌的含量信息。
本研究中使用的12个窖池的出酒质量都经过仔细的品评和香味成分分析[3]。白酒的质量总体上是B厂区(百年窖池)好于A厂区(40年窖池),C厂区属于新厂区,出酒质量排在最后。总体来讲,窖泥中耐酸乳酸杆菌的含量与白酒品质存在明确的负相关关系。而酒醅中L.acetotolerans的含量与酒体质量之间的关系尚未见明显的规律。C厂区不同池子的窖泥中L.acetotolerans的含量差异与酒体质量并不完全对应,提示新厂区的窖池整体菌群尚处于调整阶段,菌群结构上不像老厂区那样稳定。这一点可能反映在L.acetotolerans含量的波动上。
徐岩等申报的专利[26]提供了一对L.acetotolerans的特异性引物,其设计方法的特异性验证手段与本文类似。从其专利公开的数据来看,引物的特异性和QPCR定量效果都非常好。但是本文并没有比较他们的这对引物与本文中2对引物在测定24个样本中的特异性差异和QPCR定量结果差异。主要原因在于,不同白酒的不同发酵样本中菌群的组成差异较大,L.acetotolerans的几对引物面向不同的样本中菌群基因组背景差异很大时,各自表现出来的物种特异性程度肯定是不同的。或者说,到底哪一对引物的物种特异性最好取决于使用的具体的样本,研究人员需要在实际的测试过程中找到适合所用样本的最佳引物。
参考文献
[1] 敞颜, 赵辉, 凌宏志. 传统白酒发酵微生物的研究进展[J]. 食品工业科技,2010, 31(9):425-427; 431.
[2] 余乾伟. 传统白酒酿造技术[M]. 北京:中国轻工业出版社, 2010.
[3] 张会敏,古井贡酒微生物群落结构及其与主要风味物质的关联研究[D]. 哈尔滨:哈尔滨工业大学,2016.
[4] HU X, DU H, REN C, et al. Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J]. Applied and Environmental Microbiology, 2016, 82(8):2 506-2 515.
[5] LI Junwei, HE Hongkui, CAO Runjie, et al. Significant difference of the culturable anaerobic microbial species in GujingTribute pitmud with different ages[C]//IOP Conference Series: Materials Science and Engineering, 2018 5th International Conference on Advanced Composite Materials and Manufacturing Engineering,2018-08-08.
[6] HONG X, CHEN J, LIU L, et al. Metagenomic sequencing reveals the relationship between microbiota composition and quality of Chinese rice wine[J]. Scientific Reports, 2016, 6: 26 621.
[7] 胡佳. 利用免培养法对浓香型白酒曲药细菌菌群的解析研究[D]. 成都:四川大学, 2007.
[8] 李维青. 浓香型白酒与乳酸菌、乳酸、乳酸乙酯[J]. 酿酒, 2010, 37(3): 90-93.
[9] WANG H Y, ZHANG X J, ZHAO L P, et al. Analysis and comparison of the bacterial community in fermented grains during the fermentation for two different styles of Chinese liquor[J]. Journal of Industrial Microbiology & Biotechnology, 2008 , 35(6):603-609.
[10] HARUTA S, UENO S, EGAWA I, et al.Succession of bacterial and fungal communities during a traditional pot fermentation of rice vinegar assessed by PCR-mediated denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2006,109(1):79-87.
[11] LIU Junyan, DENG Yang, PETERS B M, et al. Transcriptomic analysis on the formation of the viable putative non-culturable state of beer-spoilage Lactobacillus acetotolerans[J]. Scientific Reports, 2016, 6: 36 753.
[12] 刘森, 李林光, 李可,等. 中国浓香型白酒窖池窖泥中原核微生物群落的空间异质性[J]. 食品科学, 2013, 34(21): 221-226.
[13] TOH H, MORITA H, TSUJI H, et al. Complete genome sequence of Lactobacillus acetotolerans RIB 9124 (NBRC 13120) isolated from putrefied (hiochi) Japanese Sake.[J]. Journal of Biotechnology, 2015, 214:214-215.
[14] 张会敏, 束莹, 周庆伍,等. 利用非培养技术初步研究古井贡酒大曲中的细菌群落结构[J]. 现代食品科技, 2014,30(4):44-49.
[15] 张会敏, 李天婵, 孙美青, 等. 利用非培养技术初步研究古井贡酒窖泥细菌群落结构[J].食品工业科技, 2014, 35(13):200-206;228.
[16] 张会敏, 王杰, 周庆伍,等. 利用非培养技术初步解析古井贡酒大曲真核微生物群落结构[J]. 食品工业, 2014, 35(6):158-162.
[17] ZHANG Huimin, HE Hongkui, YU Xiujuan, et al. Employment of near full-length ribosome gene TA-cloning and Primer-Blast to detect multiple species in a natural complex microbial community using species-specific primers designed with their genome sequences[J]. Molecular Biotechnology, 2016, 58:729-737.
[18] LIU Zuolin, HAN Ying, LI Junwei, et al. Analysis of microbial community structure of pit mud for Chinese strong-flavor liquor fermentation using next generation DNA sequencing of full-length 16S rRNA[J]. bioRxiv,2018.31.DOI: http://dx.doi.02g/10.1101/380949.
[19] YE J, COULOURIS G, ZARETSKAYA I, et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction[J]. BMC Bioinformatics, 2012, 18(13):134.
[20] LI K Q, ZHANG H M, LI A J, et al. Differential microbial community complexity levels between different locations in the GuJingGong pits[J].International Conference on Medicine Sciences and Bioengineering, ICMSB 2014, 2015: 641-646.
[21] LUO Ximei, GAO Zhilei, ZHANG Huimin, et al. Phylum-specific primer design and implication in quantification of the microbial community structure in GuJingGong pit mud[J]. Advanced Materials Research, 2014, 3 607(1 051): 311-316.
[22] ZHANG Huimin, GUO Changlu, HE Hongkui, et al. QPCR quantification strategy of minor species in a complex microbial community using species-specific primers[J]. Journal of Pure & Applied Microbiology, 2013, 7: 601-610.
[23] 赵长青,杨秦欢,邓静,等.窖泥的评定指标检测[J].生物技术进展,2012,2(3): 212-216.
[24] 张强,沈才洪,刘清斌,等.窖泥质量评价研究进展[J].酿酒科技,2013(7): 84-86.
[25] 卫春会,黄治国,甄攀.窖泥微生物群落SSCP分析条件优化的研究[J].四川理工学院学报(自然科学版),2010,23(6): 695-698.
[26] 徐岩,吴群,王石垒. 一种快速筛选、鉴定及定量发酵食品中耐酸乳杆菌的方法[P]. 江苏:CN106906302A, 2017-06-30.
Employment of PRIMER-BLAST to design species-specific primers for Lactobacillus acetotolerans and their implications in quality assessment of pitmud or Zaopei samples in Gujing Tribute liquor
LIU Qianqian1, LI Junwei2, CAO Runjie2, ZHANG Huimin2, HE Hongkui2*, LI Anjun2, ZHANG Zhizhou1*
1(Harbin Institute of Technology (Weihai), College of Marine Science and Technology, Weihai 264209, China) 2(Anhui Gujing Tribute Co., Ltd., Bozhou 236800, China)
ABSTRACT When brewing Gujing Tribute liquor, a large number of Lactobacillus acetotolerans were found in both pitmud and Zaopei. Therefore, it is important to rapidly detect L. acetotolerans specifically. In this paper, whole genome sequences of Lactobacillus bacteria and PRIMER-BLAST method were used to design L. acetotolerans-specific primers. Two pairs of primers were confirmed with decent specificity and combined with quantitative polymerase chain reaction (qPCR) to rapidly measure the amount of L. acetotolerans with different qualities and at different fermentation stages in pitmud and Zaopei. The results showed that the population of L. acetotolerans in Gujing Tribute pitmud was apparently lower than that of Zaopei and was apparently lower in old pitmud than that in pitmud of new plant. Moreover, L. acetotolerans in Zaopei from both new and old plants were generally high with apparent fluctuations among samples. In general, QPCR mediated by L. acetotolerans-specific primers can be used for L. acetotolerans quantification and facilitate quality assessment of pitmud or Zaopei in strong-aroma liquor production.
Key words Lactobacillus acetotolerans; Gujing Tribute liquor; specific primers; pitmud; Zaopei