表1 选择性培养基及培养条件
Table 1 Incubatory conditions of different selective media

阮征1,2,魏力1,张延杰3,夏雨3,李汴生1,2*
1(华南理工大学 食品科学与工程学院,广东 广州,510640) 2(广东省天然产物绿色加工与产品安全重点实验室,广东 广州,510640) 3(咀香园健康食品(中山)有限公司,广东 中山,528400)
摘 要 为揭示油沙冰皮月饼菌群结构,采用高通量测序和传统培养方法进行分析。结果表明,2种方法分析菌群结构具有较好的一致性;高通量测序可以全面反映菌群的组成及丰度信息,以蓝细菌属(Cyanobacteria),芽孢杆菌属(Bacillus)和葡萄球菌属(Staphylococcus)为主,随着贮藏时间的延长,菌落总数,芽孢杆菌和霉菌呈现显著性增加(P<0.05)的趋势,葡萄球菌呈显著性降低趋势。传统培养方法分离得到55株细菌和6株霉菌,芽孢杆菌属(58.18%)和葡萄球菌属(12.72%)比例较高,毛霉33.33%,青霉16.67%,曲霉50.00%,并发现腐败菌主要来自于原料粉料和馅料。研究结果可以为冰皮月饼的安全性控制提供参考。
关键词 冰皮月饼;高通量测序;传统培养;菌群结构
DOI:10.13995/j.cnki.11-1802/ts.019386
第一作者:博士,副教授(李汴生教授为通讯作者,E-mail:febshli@scut.edu.cn)。
基金项目:2017年度国家重点研发计划项目“食品绿色节能制造关键技术及装备(2017YFD0400400)
收稿日期:2018-11-20,改回日期:2018-12-20
根据GB19855—2015中的定义,冰皮月饼以糯米粉,大米淀粉,玉米淀粉等为饼皮的主要原料,经熟制后制成饼皮,包裹馅料,并经成型,冷藏(或不冷藏)等冷加工工艺制成的口感绵软的月饼[1-2]。冰皮月饼相比于传统月饼,为了保持其本真原料的外观色泽(例如熟糯米粉制备的月饼外观洁白如雪,故以“冰皮”命名),一般不经过烤制处理,采用冷加工工艺,要求在洁净度较高的环境下加工。然而,即便如此,由于原辅料营养丰富,加工过程缺乏杀灭或抑制微生物生长的控制措施,导致冰皮月饼,尤其是常温贮存的产品存在较大的安全隐患,往往需要通过添加抑菌物质等手段来提高其保藏性。其中月饼中芽孢杆菌和霉菌对环境的耐受力很强,较难控制,对其原辅料和加工过程的菌群多样性进行分析,才能更有效地进行追溯和防控。
随着科学技术的发展,研究食品腐败微生物的方法越来越多。高通量测序作为新一代测序技术的代表,所包含信息量大,但是信息的归类整合模糊,无法区分死菌与活菌,因此常常需要辅助另外一些手段进行研究[3-6]。传统培养技术可培养的条件有限,对于绝大多数微生物并不能很好地富集和分离,而且培养周期长,但成本低、分离出的菌体纯度高,在微生物研究中仍有重要地位,可以作为高通量测序技术的补充,同时传统培养技术能更好更准确地区分活菌和死菌,因此高通量测序技术结合传统培养技术能更加真实准确地反映系统中的活菌情况[7-10],这样有助于更准确地研究系统中的腐败微生物。曹荣等[9]利用高通量测序技术和传统纯培养技术研究牡蛎微生物群落,发现2种方法得到的结果一致。
关于月饼腐败菌的研究主要集中在传统烘焙型月饼的霉菌群系上,黄恺婷[11]对广式月饼腐败霉菌进行了分离,发现导致广式月饼败坏的霉菌以曲霉菌和青霉菌为主;陈盼[12]对广式月饼中的微生物区系进行研究,发现微生物群系以细菌为主,其次是霉菌和酵母菌;顾媛等[13]研究肉制品月饼微生物群系,发现霉菌主要有灰绿曲霉、杂色曲霉和黄曲霉。目前对于冰皮月饼腐败微生物的群系研究鲜有报道。本研究选择未添加抑菌物质的油沙冰皮月饼(馅料为油性豆沙)为研究对象,以高通量测序和传统培养为手段,研究常温贮藏样品的微生物菌群结构和主要来源,在不影响冰皮月饼特征感官品质的前提下,探究包装前短时间热处理和紫外线处理对菌群状况的影响,以期为常温冰皮月饼的安全性控制提供参考。
油性豆沙馅料、冰皮月饼预拌粉、麦芽糖浆、白奶油,市售。
冰皮月饼制作流程:清水与麦芽糖浆混合煮沸;取冰皮预拌粉与混合液混合慢速搅拌,分2次加入白奶油,搅拌成型;取出面团,整形分割;包裹馅料,模具成型;包装(添加脱氧剂)。
鉴于热处理和紫外线处理是常用的微生物控制的物理方法,在不影响冰皮月饼固有外观品质的前提下,对包装前的样品进行短时热处理(170 ℃, 8 min)和紫外处理(250~270 nm, 30 min),探讨2种手段对常温冰皮月饼微生物控制的可行性(操作环境经过30 min紫外处理,所有接触表面经过酒精消毒,经环境检验符合国家标准)。
样品标注为对照样,紫外线处理,热烘+紫外线处理。样品未添加抑菌物质,置于25 ℃下贮存备用。
1.3.1 腐败菌的分离纯化及鉴定[14-17]
常温冰皮月饼腐败菌分离纯化:过程样及成品于无菌操作台上用体积分数75%酒精擦拭无菌包装表面后,共取25 g样品置入225 mL的无菌生理盐水中,均质5 min,无菌生理盐水做梯度稀释10-1、10-2、10-3、10-4。从以上稀释度菌液中各取0.1 mL在选择性培养基平板上涂布均匀,培养条件及培养基种类如表1所示。然后观察各平板菌落形态及分布,挑取典型菌落平板划线直至出现单一菌落,挑取纯菌落于肉汤培养过夜,4 ℃保存备用。纯培养腐败菌的生化鉴定:分离纯化菌株依据伯杰氏细菌手册和真菌鉴定手册进行鉴定,结果如表1所示。
表1 选择性培养基及培养条件
Table 1 Incubatory conditions of different selective media

1.3.2 高通量测序技术检测微生物多样性[18-20]
1.3.2.1 DNA抽提和纯度检测
在超净工作台称取25 g成品样品放入装有225 mL 生理盐水的无菌均质袋中,均质5 min,无菌操作取2 mL均质液至无菌离心管,放入离心机3 000 r/min离心1 min,吸取离心管中上清液至2 mL无菌离心管中,放入高速冷冻离心机,4 ℃,12 000 r/min离心2 min, 弃去上清液,离心沉淀按照细菌基因组试剂盒方法提取细菌基因组DNA,并利用10 g/L琼脂糖凝胶电泳检测抽提的基因组DNA纯度。
1.3.2.2 PCR扩增及测序
基因组DNA采用16SrDNA的引物338F_806R(338F,5′-ACTCCTACGGGAGGCAGCAG- 3′,806R,5′-GGACTACHVGGGTWTCTAAT- 3′)对V3可变区进行扩增,PCR扩增条件预变性1个循环(95 ℃,3 min), 扩增30个循环(变性95 ℃,30 s;退火60 ℃, 30 s;延伸72 ℃,45 s),最后72 ℃延伸10 min;PCR反应体系为5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL,Forward Primer(338F,5 μmol/L) 0.8 μL,FastPfu Polymerase 0.4 μL,BSA 0.2 μL,ddH2O 11.8 μL。PCR扩增产物用质量分数为2%的琼脂糖凝胶电泳进行检测。参照电泳初步定量结果,将PCR产物进行定量检测,并进行混样,用质量分数为2%的琼脂糖凝胶电泳进行纯度检测,使用试剂盒回收PCR产物。由上海美吉生物构建PE扩增子文库,并使用Illumina Miseq平台进行测序。
1.3.3 单种纯化菌落鉴定方法[21-24]
1.3.3.1 纯化菌种DNA提取
取37 ℃肉汤培养的菌液3 mL,12 000 r/min离心1 min,弃去上清液,加入0.5 mL TE悬浮沉淀,并加入75 μL,50 mg/mL溶菌酶溶液,30 ℃保温10 min, 加入体积分数10% SDS,5 μL蛋白酶K,混匀,37 ℃保温30 min,加入0.75 mL,5 mol/L NaCl,混匀,用V(苯酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1抽提,12 000 r/min离心1 min,将上清液移至干净离心管,用V(氯仿)∶V(异戊醇)=24∶1抽提,静置10 min, 12 000 r/min离心1 min,转移上清液干净管,加2倍体积乙醇沉淀DNA,混合,室温静置10 min,离心沉淀DNA,70%乙醇漂洗DNA后,用3050 TE溶解DNA备用。
1.3.3.2 PCR扩增及测序
基因组DNA采用引物27F_1492R 引物(27F:5′-AGAGTTTGATCCGGCTCAG- 3′,1492R:5′-GGTTACCTTGTTACGACTT- 3′)进行扩增,PCR反应体系为2×Taq Plus Master Mix 10 μL,5p 27F 0.8 μL,5p 1492R 0.8 μL,Template 1.0 μL,ddH2O 7.4 μL;PCR扩增条件预变性1个循环(95 ℃,5 min),扩增35个循环(变性95 ℃,30 s;退火55 ℃,30 s;延伸72 ℃,90 s), 最后72 ℃延伸10 min;PCR扩增产物用质量分数为1%的琼脂糖凝胶电泳进行检测,使用试剂盒回收PCR产物并测序,结果在NCBI文库中进行比对。
分析图采用Excel 2016,Origin 7进行处理;采用SPSS 21.0进行显著性分析(P<0.05)。
图1反应的是对照样高通量测序的结果,该结果反映从样品中共分离鉴定出16个属,分别为蓝细菌属(Cyanobacteria),芽孢杆菌属(Bacillus),葡萄球菌属(Staphylococcus),不动杆菌属(Acinetobacter),链球菌属(Streptococcus),黄单胞菌属(Xanthomonas),乳球菌属(Lactococus),肠杆菌属(Enterobacter),红球菌属(Rhodococcus),气单胞菌属(Aeromonas)以及金黄杆菌属(Chryseobacterium)等。其中微生物属丰度前3的属为蓝细菌属,葡萄球菌属和不动杆菌属。对照样进行传统分离培养,共分离得到6株菌株,经鉴定得到葡萄球菌属占16.67%,芽孢杆菌属占50.00%,未知属占33.33%;结合高通量测序,可知样品中存在的葡萄球菌属和芽孢杆菌属占有较大的比例。传统分离培养技术从对照样中分离得到3株霉菌,分别为毛霉,青霉和曲霉;而食品中的腐败菌主要包括细菌,酵母和霉菌,其中细菌主要包括芽孢杆菌属,假单胞菌属,葡萄球菌属和黄杆菌属等,这些微生物属对于蛋白质类食品,碳水化合物类食品和脂肪类食品表现为不同的分解能力,但是作为腐败菌总体皆表现为对营养物质和感官的破坏。结果表明,本研究中冰皮月饼的主要腐败细菌为芽孢杆菌属和葡萄球菌属。

图1 样品微生物属的相对丰度
Fig.1 The relative abundance of different samples
图2反映的是经过短时热处理和紫外处理的成品样品高通量测序的结果,该结果反映从样品中共分离鉴定出14个属,结果同对照样的菌群结构相似,表明短时热处理和紫外处理对菌群结构影响不大;其中经过短时热处理和紫外处理的成品样品中微生物属丰度前三的属为蓝细菌属,相对丰度分别为29.89%,39.79%;芽孢杆菌属,相对丰度分别为20.62%,19.38%; 葡萄球菌属,相对丰度分别为11.70%,9.58%;结果与对照样有差异,说明短时热处理和紫外线处理虽然对菌群结构组成影响不大,但是能够影响不同菌群之间的相对丰度。同样传统分离培养技术从经过短时热处理和紫外处理的成品样品中分离得到3株霉菌,分别为毛霉,青霉和曲霉,说明短时热处理和紫外处理对于霉菌的种类影响不大。
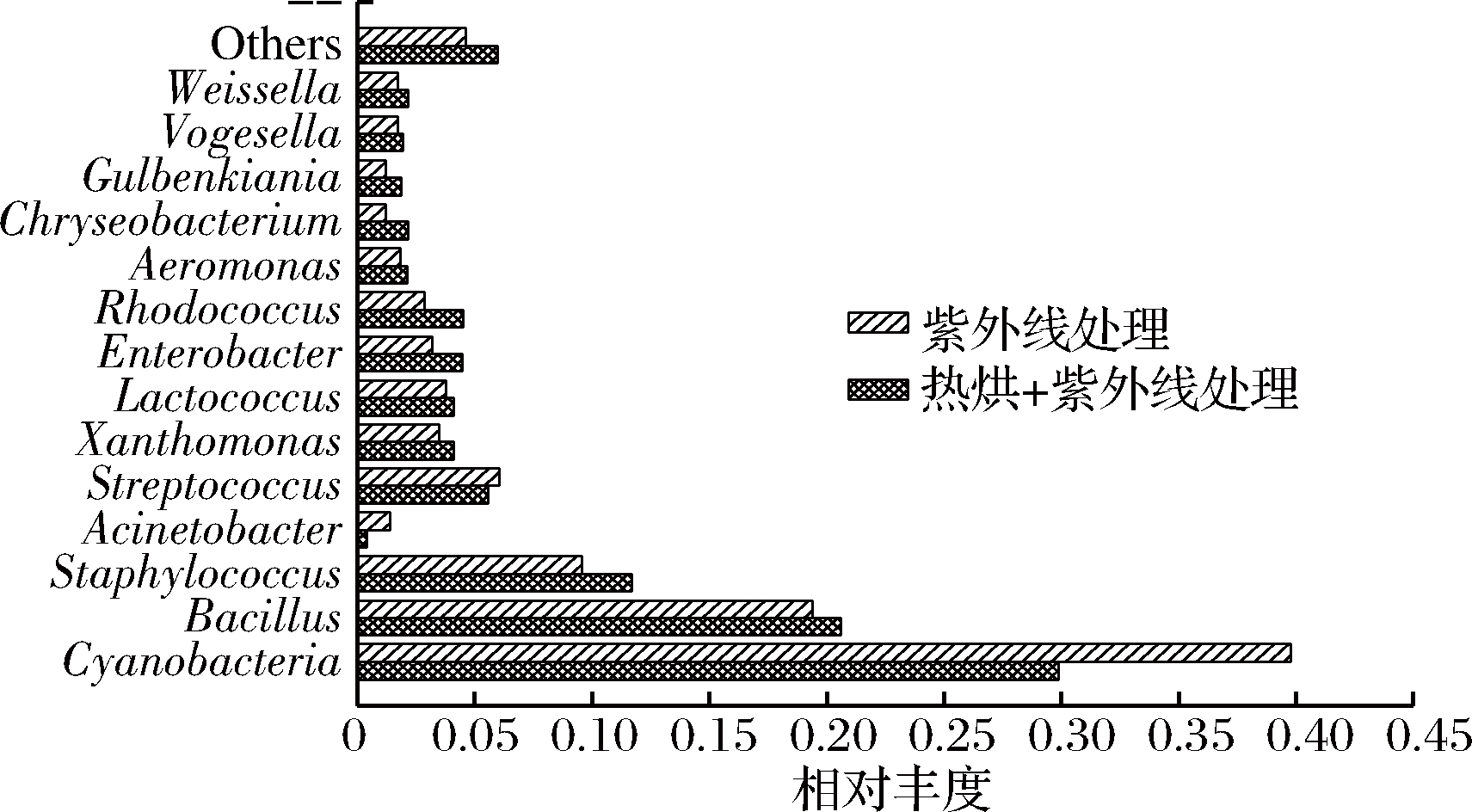
图2 样品微生物属的相对丰度
Fig.2 The relative abundance of different samples
图3反映的是样品贮藏过程中微生物数量的变化情况。根据高通量测序结果和传统培养技术得出,实验用冰皮月饼主要存在的腐败菌为葡萄球菌属和芽孢杆菌属,因此探究腐败菌属葡萄球菌和芽孢杆菌在贮藏过程中的变化对于了解和控制腐败菌的生长有利。(1)反映随贮藏时间的延长样品中菌落总数、芽孢杆菌和霉菌呈现显著性增长(P<0.05),而葡萄球菌属的数量呈现显著性降低的趋势(P<0.05);(2)反映了微生物的变化随贮藏时间变化的关系,同样得出与(1)一样的变化趋势;经过短时间的热处理和紫外处理,样品中的菌落总数增加,而葡萄球菌数量呈现下降的趋势,但是短时间的热处理对于霉菌和芽孢杆菌的影响较小。未经过热处理的样品在贮藏30 d时,菌落总数达到1 280 CFU/g,超过标准(≤1 000 CFU/g)的1.2倍,而经过短时间热处理的样品在40 d后微生物才超标(1 039 CFU/g),表明短时热处理对微生物的生长有抑制作用。通过前面的分析,样品中主要存在的腐败微生物芽孢杆菌属有较强的抗逆性,在贮藏过程中呈现显著性增长趋势,因此采取有效的措施从原料入手,才能减少样品中的微生物污染。
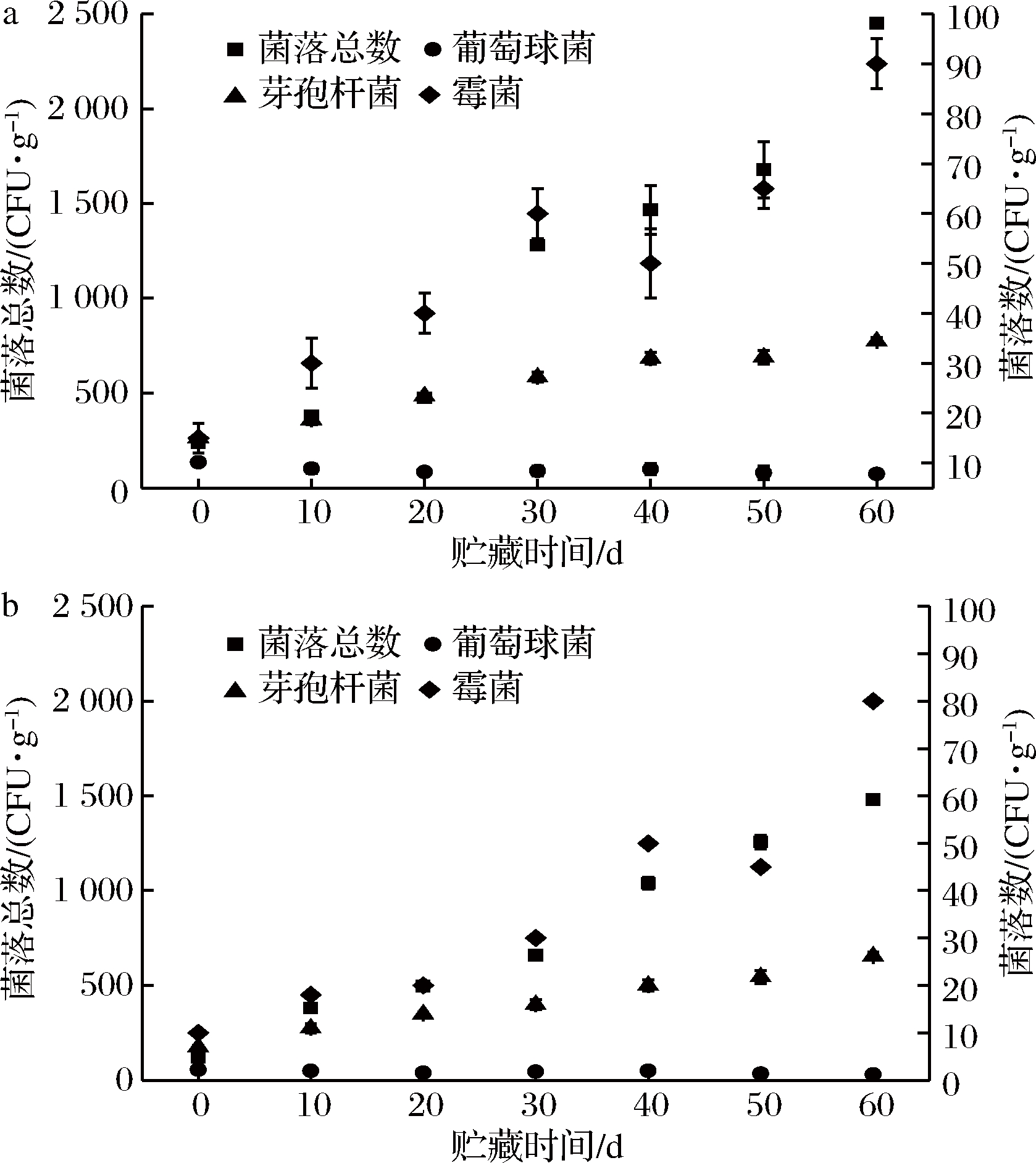
a-紫外线处理;b-热烘+紫外线处理
图3 样品贮藏过程中微生物变化
Fig.3 Microbial changes of samples during storage
贮藏过程中,冰皮月饼皮料和馅料水分活度的变化如图4所示。

a-皮料水分活度;b-馅料水分活度
图4 样品贮藏期间水分活度变化
Fig.4 Changes in water activity during storage of samples
其中在贮藏过程中,皮料水分活度呈现显著降低的趋势,而馅料水分活度变化不显著。食品贮藏稳定性与水分活度存在一定关系,其中对于微生物来说,大部分细菌在水分活度低于0.90时无法繁殖,大部分霉菌可在0.80~0.87生长;且样品的皮料的水分活度分别从0.836下降到0.756(P<0.05),从0.833到0.762 (P<0.05),说明实验样品在贮藏过程中水分活度呈现显著性降低的趋势,大部分微生物的生长受到抑制。
表2反映的是微生物生理生化鉴定结果与NCBI文库序列比对结果。传统微生物培养技术从原料,过程样和成品共分离得到55株细菌和6株霉菌(其中毛霉占33.33%,青霉占16.67%,曲霉占50.00%);经形态学检验和生理生化鉴定,发现芽孢杆菌属(Bacillus)和葡萄球菌属(Staphylococcus)比例较高,分别为58.18%,12.72%;依据初步鉴定结果划分为 11株不同细菌,其中从原料到成品都存在的微生物包含4株,分别为Ⅰ,Ⅱ,Ⅲ,Ⅳ。
表2 NCBI序列对比结果
Table 2 NCBI sequence comparison results
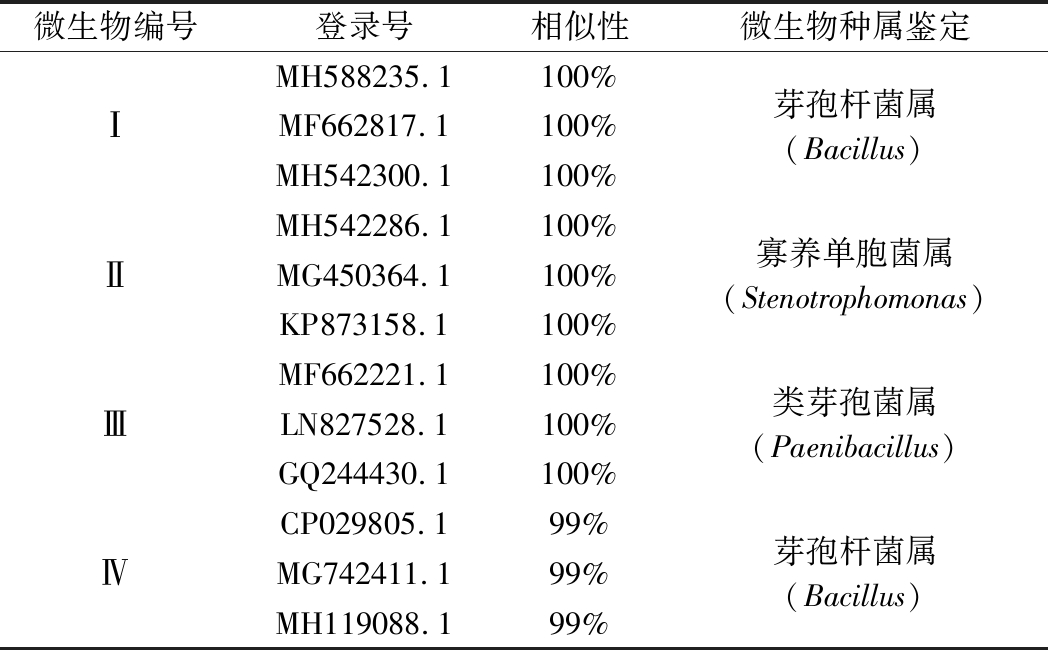
其中Ⅰ,Ⅲ及毛霉分离来源于冰皮月饼预拌粉;Ⅱ,Ⅳ,青霉和曲霉分离来源于馅料。根据伯氏细菌手册鉴定Ⅰ,Ⅱ,Ⅲ,Ⅳ,分属于芽孢杆菌属(Bacillus),黄单胞菌属(Xanthomonas),芽孢杆菌属(Bacillus)和芽孢杆菌属(Bacillus),且分离得到的这些微生物是样品中的有害微生物。考虑到传统生理生化鉴定存在一定的局限性,对纯化的Ⅰ,Ⅱ,Ⅲ,Ⅳ菌株进行进一步的鉴定,通过基因提取,PCR扩增,其结果登录NCBI进行序列比对,如表2所示,发现在NCBI文库中,Ⅰ鉴定属于芽孢杆菌属(Bacillus),Ⅱ鉴定属于寡养单胞菌属(Stenotrophomonas),Ⅲ鉴定属于类芽孢杆菌属(Paenibacillus),Ⅳ鉴定属于芽孢杆菌属(Bacillus),且与NCBI其他已知微生物基因序列相似性达到99%,此结果与传统纯培养和高通量测序结果相似,发现从样品中存在的腐败微生物主要为芽孢杆菌属和葡萄球菌属,且一直存在于整个加工过程中的腐败细菌为芽孢杆菌属,寡样单胞菌属和类芽孢杆菌属,并主要来自于原料粉料和馅料,所以原料中原有的微生物是导致样品微生物污染的主要来源。
本文采用高通量测序和传统分离培养的方法分析了常温油沙冰皮月饼的微生物菌群结构,发现两种方法的检测结果具有较好的一致性。
从研究结果上看,在属的水平上,经过短时热处理和紫外处理的油沙冰皮月饼菌群中丰度前三位的菌属为蓝细菌属(Cyanobacteria),芽孢杆菌属(Bacillus)和葡萄球菌属(Staphylococcus),且芽孢杆菌属和葡萄球菌属是其主要腐败有害微生物,而蓝细菌属对样品腐败几乎没有影响;且经过不同处理,尽管菌群结构相似,但不同种属之间相对丰度有差异,两种处理方式能降低样品中的微生物数量。结合传统培养技术,分析成品中微生物的来源发现,分离得到的微生物主要来源于各种原料,其中含量较多的芽孢杆菌属主要来源于粉料,霉菌主要来源于粉料和馅料;随着贮藏时间的延长,其中样品的菌落总数,芽孢杆菌和霉菌呈现显著性增加的趋势,而葡萄球菌呈现显著性降低的趋势,因为随着贮藏时间的延长,样品的水分活度呈现降低的趋势,对于抗逆性较强的霉菌和芽孢杆菌,更加能适应较恶劣的环境。
包装前短时间热处理和紫外线处理对于冰皮月饼中的微生物有一定的影响,但是处理强度过高会影响冰皮月饼的特有感官品质,因此仅仅依靠强度有限的热烘和紫外处理难以达到常温贮存所需的杀菌或抑菌程度。一方面,鉴于芽孢杆菌、霉菌等主要污染微生物大多属于好氧微生物,在月饼包装袋中添加脱氧剂可以提供一种有效的保藏途径;另一方面,有针对性地控制冰皮月饼的初始微生物污染程度将有助于贮存期的延长。结果显示原辅料是常温冰皮月饼的主要微生物污染源,因此皮料馅料等的卫生安全水平将直接影响终产品的安全性和贮存期。
参考文献
[1] 张宏康,黄育杭,李笑颜,等.冰皮月饼加工研究进展[J].食品工业,2018,39(7):275-278.
[2] 曾习.速冻冰皮月饼皮的工艺研究[J].食品科技,2017,42(4):157-161.
[3] HUISINTVELD J H.Microbial and biochemical spoilage of foods:an overview[J].International Journal of Food Microbiology,1996,33(1):1-18.
[4] ENGLER K,PALSSON B O.A road map for the development of community systems(Co Sy)biology[J].Nature Review Microbiology,2012,10(5):366-372.
[5] LEDERBERG J.Infectious history[J].Science,2000,288(5 464):287-293.
[6] NERSS, GOODIN D B, PIELAK G J, et al.A rapid droplet method for Sanger dideoxy sequencing[J].Biotechniques,1988,6(5):408-412.
[7] BROEKAERT K, HEYNDRICKX M, HERMAN L, et al. Seafood quality analysis: Molecular identification of dominant microbiota after ice storage on several general growth media[J].Food Microbiology, 2011,28(6): 1 162-1 169.
[8] JIANG Yun,GAO Feng,XU Xinglian, et al. Changes in the composition of the bacterial flora on tray-packaged pork during chilled storage analyzed by PCR-DGGE and real-time PCR[J]. Journal of Food Science, 2011, 76(1):27-33.
[9] 曹荣,张井,孟辉辉,等.高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J].食品科学,2016,37(24):137-141.
[10] 曹荣,刘淇,赵玲,等.基于高通量测序的牡蛎冷藏过程中微生物群落分析[J].农业工程学报,2016,32(20):275-280.
[11] 黄恺婷.广式月饼腐败霉菌分离与控制研究[D].广州:华南理工大学,2012.
[12] 陈盼. 广式月饼中微生物区系的研究[D].重庆:西南大学,2009.
[13] 顾媛,贺稚非,李洪军,等.肉制品月饼的细菌及霉菌的检测研究[J].食品科学,2008,29(11):555-558.
[14] WAITE J G, JONES J M, YOUSEF A E.Isolation and identification of spoilage microorganisms using food-based media combined with rDNA sequencing: ranch dressing as a model food[J]. Food Microbiology, 2009, 26 (3):235-239.
[15] MATAMOROS S, PILET M F, GIGOUT F,et al.Selection and evaluation of seafood-borne psychrotrophic lactic acid bacteria as inhibitors of pathogenic and spoilage bacteria[J]. Food Microbiology, 2009, 26 (6):638-644.
[16] 黄林,陈全胜,张燕华,等.冷却猪肉优势腐败菌分离鉴定及致腐能力测定[J].食品科学,2013,34(1):205-209.
[17] 唐文静,王楚文,柳云龙,等.冷藏海鲈鱼优势腐败菌的筛选和鉴定[J].食品科学,2016,37(3):170-174.
[18] JUSTYNA P, REBECCHI A, PISACANE V, et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J]. Food Microbiology,2015,46:342-356.
[19] BERRY D,MAHFOUDH B K,WAGNER M,et al.Barcoded primers used in multiplex amplicon pyrosequencing bias amplification[J]. Applied and Environmental Microbiology,2012,78(2):612.
[20] BARTRAM A K, LYNCH M D, STEARNS J C, et al.Generation of multimillion-sequence 16S rRNA gene libraries from complex microbial communities by assembling paired-end illumina reads[J].Applied & Environmental Microbiology, 2011, 77 (11):3 846-3 852.
[21] CRISAFI F, DENARO R, GENOVESE M, et al. Comparison of 16SrDNA and toxR genes as targets for detection of Vibrio anguillarum in Dicentrarchus labrax kidney and liver[J]. Research in Microbiology,2010,162(3): 223-230.
[22] TOURNIER E, AMENC L, PABLO A L, et al. Modification of a commercial DNA extraction kit for safe and rapid recovery of DNA and RNA simultaneously from soil, without the use of harmful solvents[J]. Methodsx,2015,2(2):182-191.
[23] FU Zeyu,SONG Jiancheng,JAMESON P E.A rapid and cost effective protocol for plant genomic DNA isolation using regenerated silica columns in combination with CTAB extraction[J]. Journal of Integrative Agriculture,2017,16(8):1 682-1 688.
[24] 吴银宝,史金才,莫测辉,等.猪粪和土壤样品中微生物DNA提取方法的比较[J].农业工程学报,2006(S2):10-13.
[25] 李晓然,龚福明,李洁,等.基于混合培养和高通量测序分析云南传统发酵豆豉中活性细菌群落[J].食品科学,2014,35(7):90-94.
RUAN Zheng1,2, WEI Li1, ZHANG Yanjie3, XIA Yu3, LI Biansheng1,2*
1(School of Food Science and Engineering, South China University of Technology, Guangzhou 510640, China) 2(Guangdong Key Laboratory of Green Processing and Product Safety of Natural Products, Guangzhou 510640, China) 3(Juxiangyuan Health Food (Zhongshan) Co.,Ltd., Guangzhou 528400, China)
ABSTRACT In order to reveal the microbial flora structure of oil-sand bean paste stuffing snowy moon cake, the flora were analyzed by high-throughput sequencing and traditional culture isolation. High-throughput sequencing fully reflected the members and abundance of the flora, which was dominated by the species from genera of Cyanobacteria, Bacillus and Staphylococcus. With the extension of storage time, the total number of colonies, Bacillus, and molds increased significantly (P<0.05), while Staphylococcus showed a significant decrease trend. There were fifty-five strains of bacteria and six strains of molds isolated by traditional culture methods, with Bacillus (58.18%) and Staphylococcus (12.72%) had higher ratio, as well as 33.33% Mucor, 16.67% Penicillium, and 50% Aspergillus. It was found that spoilage bacteria were mainly from raw material powders and fillings. This research can provide reference for the safety control of snowy moon cake.
Key words snowy moon cake; high-throughput sequencing; traditional culture method; flora structure