β-半乳糖苷酶的微生物细胞表面展示及其应用
蒋晓敏1,王贺2*,王允祥1,2*,钱永常3,尹良鸿3,范丽2
1(浙江农林大学 农业与食品科学学院,浙江 杭州,311300) 2(浙江农林大学 暨阳学院,浙江 绍兴,311800) 3(浙江农林大学 林业与生物技术学院,浙江 杭州,311300)
摘 要 β-半乳糖苷酶是一种在乳制品生产、医药、生化分析等领域得到广泛应用的酶制剂。商品化β-半乳糖苷酶多以游离形式存在,但游离的β-半乳糖苷酶制造过程复杂、稳定性低、易失活、有机溶剂耐受性差、重复利用率低,这些制约因素限制了β-半乳糖苷酶的进一步应用。将β-半乳糖苷酶通过基因工程技术固定在微生物细胞表面,该系统的优点有:1)在工业生产过程中,无需经复杂的分离纯化和固定化操作,成本低;2)稳定性高、有机溶剂耐受性强,既可用于反应条件温和又能满足复杂体系、反应条件剧烈的催化需求;3)拓展β-半乳糖苷酶在蛋白质工程研究、生物传感器制造等领域的应用。β-半乳糖苷酶表面展示所用宿主包括乳酸菌、酵母和芽孢,该文分别综述这3种宿主中β-半乳糖苷酶表面展示系统的研究进展及其在酶的生产、作为报告蛋白和全细胞生物催化方面的应用。
关键词 β-半乳糖苷酶;表面展示;全细胞催化剂
第一作者:硕士研究生(王贺博士和王允祥教授共为通讯作者,E-mail:wh2989@zafu.edu.cn,wyx@zafu.edu.cn)。
基金项目:国家自然科学基金青年科学基金项目(31701535)
收稿日期:2017-04-13,改回日期:2018-07-24
DOI:10.13995/j.cnki.11-1802/ts.017534
β-半乳糖苷酶(EC 3.2.1.23 β-galactosidase)不仅能水解乳糖生成半乳糖和葡萄糖,也会发生转糖苷反应产生非消化性的低聚半乳糖(galactooligosaccharides,GOS),具水解和转糖苷双重活力。因此,该酶广泛应用于食品工业、医学和生化分析等领域。目前,商用β-半乳糖苷酶的主要生产方式是微生物发酵[1],且多呈游离态。但游离的β-半乳糖苷酶存在提纯过程繁琐、传质阻力大等缺陷。虽然固定化酶技术在一定程度上有所改进,但固定化仍要提供纯化的酶源,还有固定化过程会造成大量的酶损失和活性降低等问题,因而限制了β-半乳糖苷酶在工业上的进一步应用。
近年来,微生物细胞表面展示技术的迅速发展为β-半乳糖苷酶的研究提供了新的视角。微生物细胞表面展示技术的基本原理是通过DNA重组技术把外源蛋白或多肽基因与宿主特定的锚定蛋白基因融合导入宿主中,利用该锚定蛋白的定位作用从而将外源蛋白或多肽固定化表达在细胞表面[2]。利用微生物细胞表面展示技术将β-半乳糖苷酶定位展示在细胞表面,不但省去了复杂的分离纯化和固定化操作,而且展示在细胞表面的β-半乳糖苷酶显示出优良的稳定性和有机溶剂耐受性[3]。
由于β-半乳糖苷酶是由4个亚基组成的四聚体蛋白,相对分子质量较大。鉴于此,该展示系统宿主的研究主要集中于乳酸菌、酵母、芽孢这3类。本文将综述不同宿主中β-半乳糖苷酶表面展示系统及其应用的进展。
1 不同的β-半乳糖苷酶微生物细胞表面展示系统
根据宿主的不同,β-半乳糖苷酶微生物细胞表面展示系统可分为乳酸菌细胞表面展示、酵母细胞表面展示和芽孢表面展示(表1)。
1.1 β-半乳糖苷酶的乳酸菌细胞表面展示系统
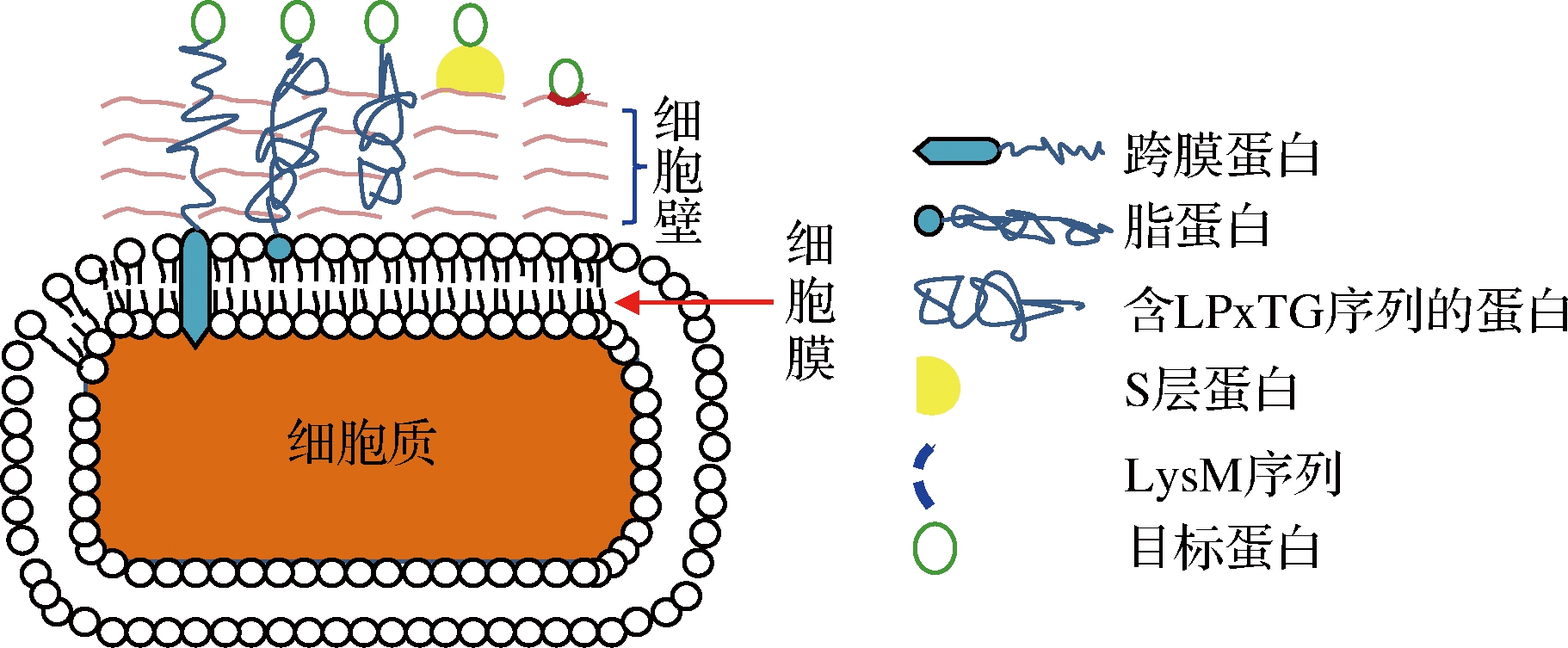
乳酸菌(lactic acid bacteria,LAB)是将活性分子传递到黏膜组织的理想载体,有些菌株具有益生菌的特性,被世界卫生组织公认为是安全的。构建乳酸菌表面展示系统需要有信号肽和锚定结构域基本元件。目标蛋白可以与跨膜蛋白或脂蛋白重组共价锚定在细胞膜上,或者利用被分选酶识别的保守基序(Leu-Pro-X-Thr-Gly,LPxTG)以共价或通过表层蛋白(S层蛋白)、自溶素基序(Lysine motif,LysM)结构域等非共价结合到细胞壁上[4],见图1所示。
表1 β-半乳糖苷酶微生物细胞表面展示系统
Table 1 Summary of β-galactosidase-oriented microbial cell surface display system
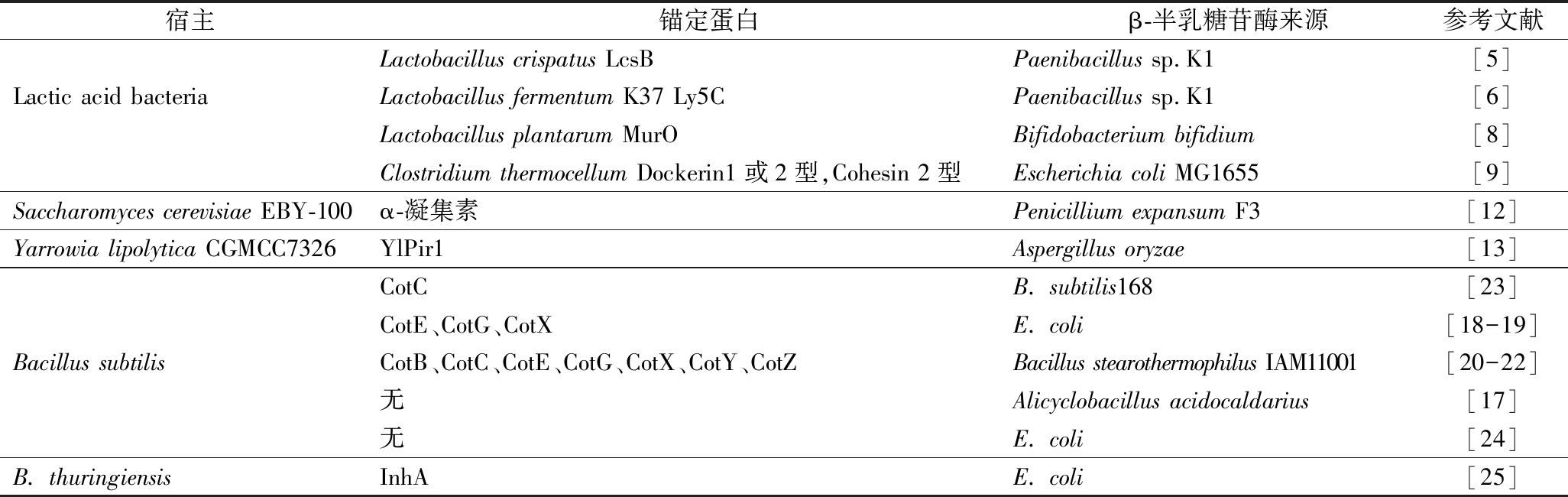
宿主锚定蛋白β-半乳糖苷酶来源参考文献Lactic acid bacteriaLactobacillus crispatus LcsBPaenibacillus sp.K1[5]Lactobacillus fermentum K37 Ly5CPaenibacillus sp.K1[6]Lactobacillus plantarum MurOBifidobacterium bifidium[8]Clostridium thermocellum Dockerin1或2型,Cohesin 2型Escherichia coli MG1655[9]Saccharomyces cerevisiae EBY-100 α-凝集素Penicillium expansum F3[12]Yarrowia lipolytica CGMCC7326YlPir1Aspergillus oryzae[13]Bacillus subtilisCotCB. subtilis168[23]CotE、CotG、CotXE. coli[18-19]CotB、CotC、CotE、CotG、CotX、CotY、CotZBacillus stearothermophilus IAM11001[20-22]无Alicyclobacillus acidocaldarius[17]无E. coli[24]B. thuringiensisInhAE. coli[25]
图1 乳酸菌细胞表面展示系统
Fig.1 Methods for target protein display in LAB using different anchor proteins
由于β-半乳糖苷酶是大分子蛋白,图1中跨膜蛋白、脂蛋白、LPxTG基序3种表面展示模型都需要目标蛋白穿越细胞膜与细胞壁,这一过程极有可能导致蛋白无法正确折叠或者无法正常分泌。所以,目前在乳酸菌表面展示β-半乳糖苷酶的策略主要集中在S层蛋白和内溶素的LysM重复序列这2种体外展示机制。S层蛋白是乳酸菌细胞壁上的表层蛋白,通常包含2个功能区域:细胞壁结合区和自组装区。细胞壁结合区主要利用该部分的碱性氨基酸与带负电荷的细胞壁次级聚合物之间的相互作用来介导细胞壁结合。自组装区则能在不受融合模块影响的同时将功能序列展示在S层蛋白最外层表面上[5]。这2个特殊功能区域使得β-半乳糖苷酶这类大分子蛋白展示在乳酸菌细胞表面成为可能。
目前,以LAB作为β-半乳糖苷酶的展示宿主主要有2种策略,一是目标蛋白与LAB锚定蛋白融合后经E. coli表达后,通过体外吸附方式固定在由三氯乙酸处理得到的LAB静息细胞表面;另一种方式是利用热纤梭菌(Clostridium thermocellum)纤维小体中的黏附模块(Cohesin)与锚定蛋白融合而固定在LAB细胞表面,目标蛋白与对接模块(Dockerin)进行融合表达使目的酶基因携带对接模块的分子标签,然后通过黏附模块和对接模块之间的特异性相互作用,引导带有对接模块分子标签的β-半乳糖苷酶定量地展示在LAB细胞表面(图2)。HU等[6]利用卷曲乳杆菌(Lactobacillus crispatus K2-4-3)的S层蛋白(SlpB)的C端区域(LcsB)作为锚定基序,以N-端融合方式与类芽孢杆菌(Paenibacillus sp. K1)β-半乳糖苷酶基因重组,在E. coli Origami B (DE3)胞内表达获得含融合蛋白的细胞裂解物,在37 ℃、1 h条件下与经过化学物质处理的Lactococcus lactis、Lb. crispatus、Lactobacillus brevis、Lactobacillus delbrueckii、Lactobacillus helveticus 等乳酸菌静息细胞进行体外吸附结合试验,发现该酶可成功锚定在上述乳酸菌细胞表面,并检测到一定量的酶活。有意思的是,但却无法通过基因重组技术以体内方式将β-半乳糖苷酶固定在乳酸菌细胞表面,推测可能与该酶在展示表达过程中无法正确折叠或分泌效率极低有关。该课题组[7]也根据LysM序列能与肽聚糖和甲壳素非共价结合这一特性[8],将类芽孢杆菌β-半乳糖苷酶基因与发酵乳杆菌(Lactobacillus fermentum K37)温和噬菌体фPYB5内溶素(Ly5C)基因构建融合蛋白,以体外方式成功展示在L. lactis、Lb. brevis、Lactobacillus casei和Lactobacillus plantarum等乳酸菌细胞表面,但表现出的活性显著低于LcsB构建的。此外,XU等[9]以植物乳杆菌细胞壁蛋白MurO为锚定蛋白成功将两歧双歧杆菌(Bifidobacterium bifidium)β-半乳糖苷酶定位在活的Lb. plantarum细胞表面。从这些研究结果可知,对β-半乳糖苷酶进行乳酸菌细胞表面展示更多地是采用体外吸附展示策略,但这一策略不仅要提供融合有锚定蛋白的β-半乳糖苷酶酶液,还需要在吸附结合前对乳酸菌细胞进行诱导处理,且目标酶蛋白易于脱落。
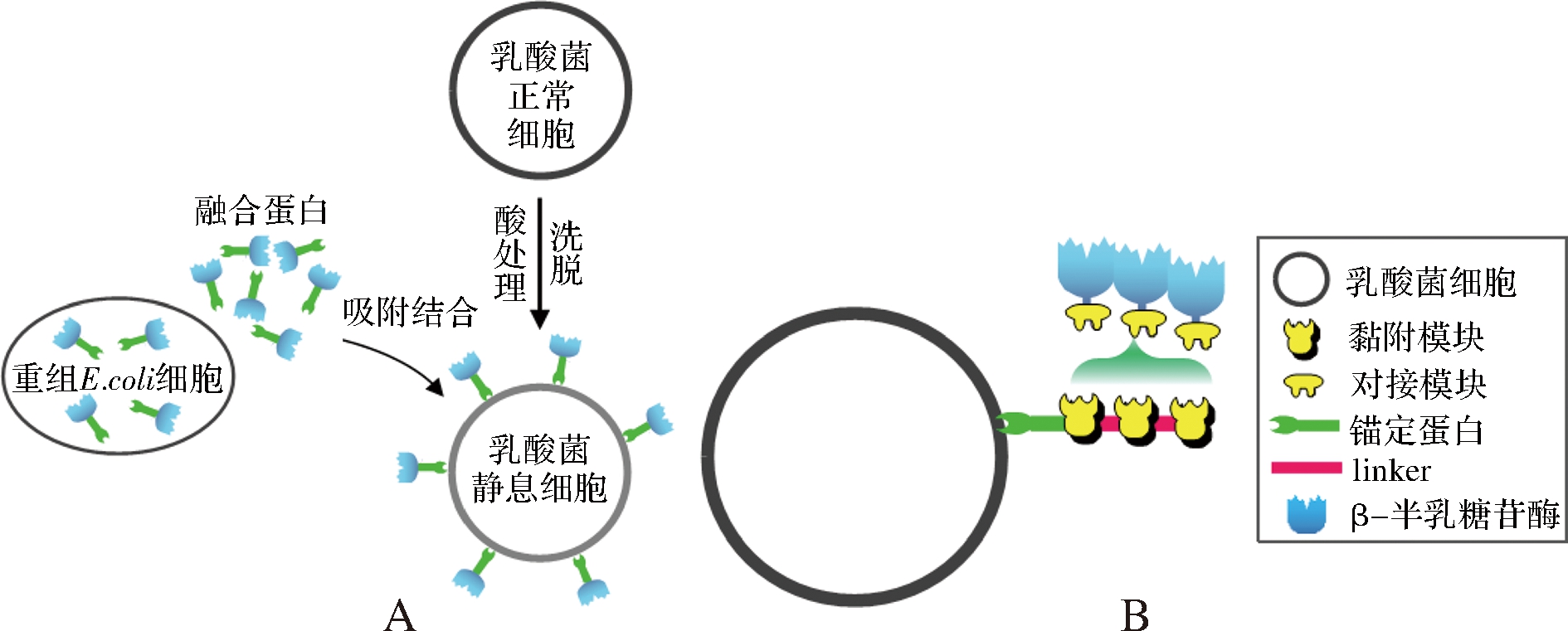
A-通过体外吸附方式展示在LAB细胞表面;B-利用“Cohesin-Dockerin”特异性相互作用原理展示在LAB细胞表面
图2 β-半乳糖苷酶乳酸菌细胞表面展示策略
Fig.2 Schematic representation of strategies for β-gala- ctosidase surface display in LAB
围绕着如何提高β-半乳糖苷酶的展示效率,研究者们亦开发了一些新型乳酸菌表面展示系统。WIECZOREK等[10]利用热纤梭菌纤维小体中的对接模块和黏附模块相互作用原理,把E. coli MG1655的β-半乳糖苷酶融合到1型或2型的对接模块,再与展示在L. lactis htra NZ9000细胞表面的1型或2型黏附模块蛋白结合,从而将β-半乳糖苷酶复合物成功展示在乳酸菌细胞表面,且1型的酶活达到1.4×10-8μmol/L PNP/min/cell,约为2型的2倍。研究表明,β-半乳糖苷酶的分子尺寸、结合顺序、在支架内的位置不同所对应的酶活确实存在差异,这为探索β-半乳糖苷酶表面展示领域提供了有价值的参考。
1.2 β-半乳糖苷酶的酵母细胞表面展示系统
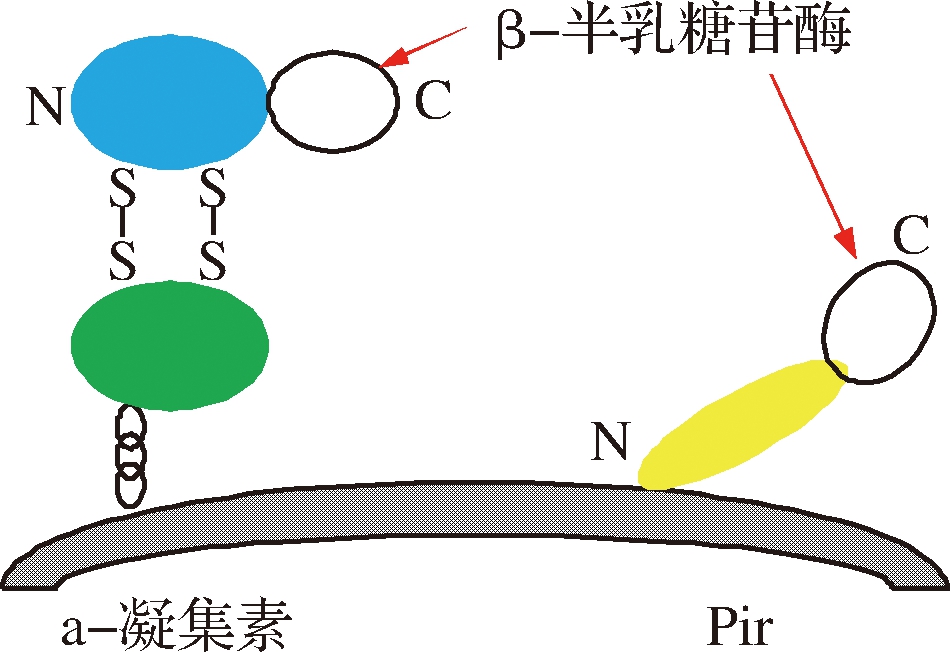
与乳酸菌非共价结合β-半乳糖苷酶的展示方式不同,酵母细胞表面展示系统拥有真核宿主分泌表达的优良特性,能促进β-半乳糖苷酶正确折叠和有效分泌,不需要额外的纯化和固定化步骤[11]。β-半乳糖苷酶的酵母细胞表面展示系统由β-半乳糖苷酶、锚定蛋白、连接肽、酵母细胞4个基本元件组成,其中已报道的锚定蛋白有α-凝集素和Pir蛋白(图3)。
图3 β-半乳糖苷酶酵母细胞表面展示系统
Fig.3 Cell surface display of β-galactosidase in yeast
β-半乳糖苷酶与锚定蛋白彼此间通过一小段连接肽组成融合蛋白,利用锚定蛋白与细胞壁的结合结构域成功展示在酵母细胞表面。目前,α-凝集素是酿酒酵母细胞表面展示体系中最常用的锚定蛋白[12]。LI等[13]将扩展青霉(Penicillium expansum F3)β-半乳糖苷酶编码基因(bgaF3)与α-凝集素Aga2亚基的C端融合,然后通过以二硫键共价结合的Aga1p而固定表达在酿酒酵母(Saccharomyces cerevisiae EBY-100)细胞表面,免疫荧光显微镜和酶活测定结果确证了β-半乳糖苷酶的成功展示。
此外,有学者利用解脂耶罗维亚酵母(Yarrowia lipolytica)细胞壁蛋白Pir作为锚定蛋白用来展示米曲霉(Aspergillus oryzae)β-半乳糖苷酶[14]。该研究小组还发现,当以乳糖作为碳源时可筛得具有较高赤藓糖醇合成能力的重组子,而且引入的2个组成型启动子FABIN和hp4d在增强β-半乳糖苷酶诱导表达水平方面无显著差异。
1.3 β-半乳糖苷酶的芽孢表面展示系统
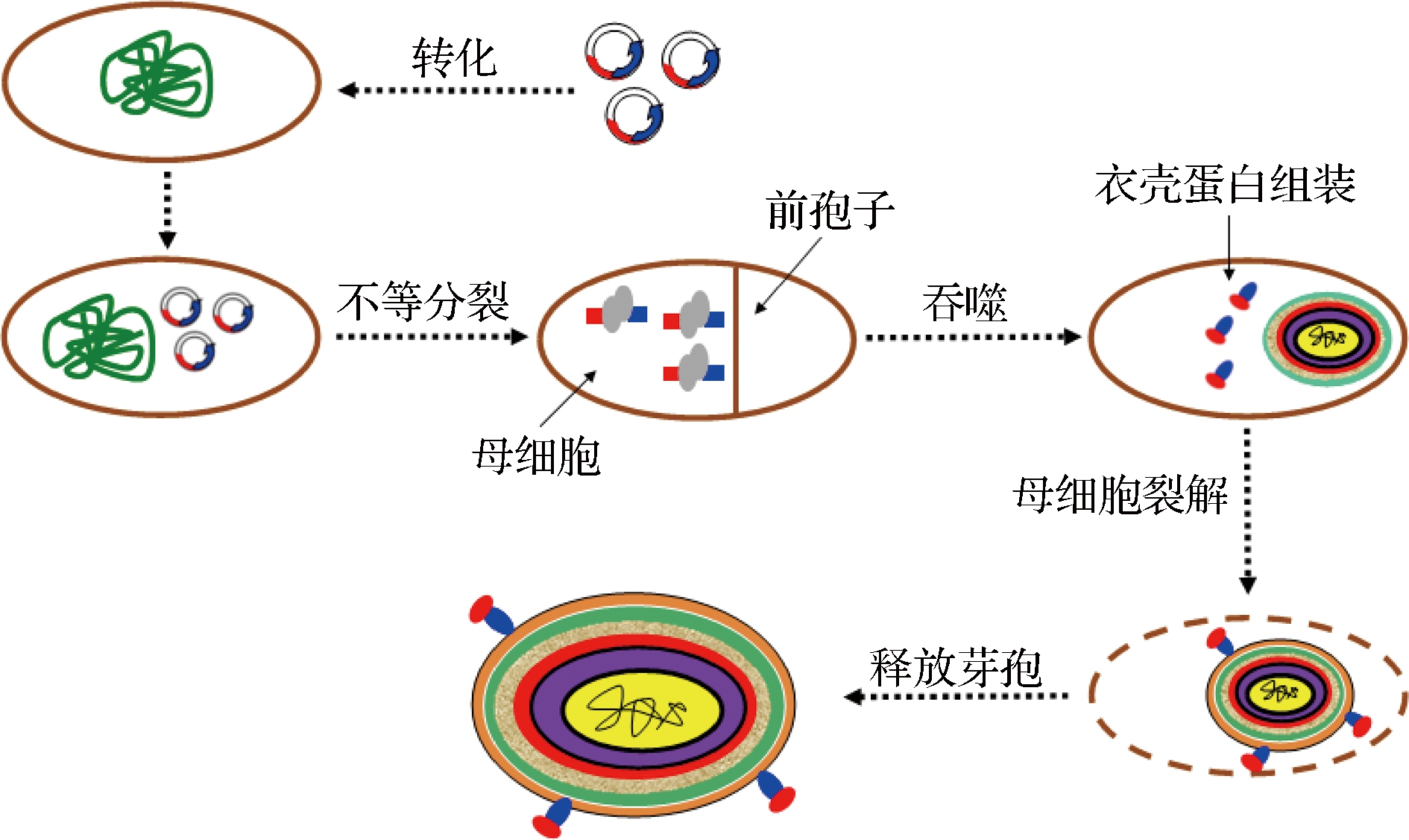
作为一种新型的微生物表面展示系统,芽孢表面展示技术以其独特的形成机制和理化特性赋予了外源蛋白特殊的展示过程和优良的抗逆性,并在相对分子质量高的多聚体蛋白生产、疫苗制备、工业用酶生产等方面展现出众多优势和巨大应用潜力,日益成为国内外科研人员研究的热点[15-17]。在芽孢杆菌属中,枯草芽孢杆菌(Bacillus subtilis)因其清晰的遗传背景、成熟的表达系统以及食用安全性等优势,使之成为进行芽孢表面展示的理想平台。推测的芽孢表面展示过程见图4所示[17]。
蓝色-锚定蛋白;红色-目标蛋白
图4 推测的芽孢表面展示过程示意图
Fig.4 Schematic of a possible process for the development of spore-surface display
由图4可知,外源目的蛋白编码基因与含有自身启动子的锚定蛋白基因融合,得到融合基因表达载体,之后转入芽孢杆菌胞内,经诱导培养后,融合基因在孢子母细胞内表达,并通过锚定蛋白的定位而完成在芽孢表面的附着,最后母细胞裂解释放出成熟的芽孢。近年来,围绕如何提高枯草芽孢杆菌芽孢表面展示效率进行了大量研究,提出了选择合适的锚定蛋白,引入诱导型启动子和适宜的linker调控表达,增加外源基因拷贝数,敲除部分高丰度、非必须型衣壳蛋白编码基因等策略。
由于芽孢外层的衣壳蛋白的特性,使得芽孢对恶劣环境具有高度抵抗能力,并且具有长时间保持休眠的能力。这一特性使得融合蛋白的热稳定性、有机溶剂耐受性等得以提高。有研究表明[18],β-半乳糖苷酶的芽孢表面展示系统中芽孢的存在不但不影响酶的最适pH和温度,而且还保护被吸附的β-半乳糖苷酶不受酸性pH条件的影响。相比于乳酸菌、酵母等展示系统要求目标蛋白穿过细胞膜,芽孢结构比较特殊无细胞膜结构,克服了目标蛋白折叠不正确和定位不准确等问题[15]。这一特殊机制使得β-半乳糖苷酶这类大分子蛋白在芽孢表面高效表达成为可能。
一般地,芽孢表面展示体系分为重组型芽孢表面展示体系、非重组型芽孢表面展示体系和无锚定蛋白型芽孢表面展示体系。近年来,开展β-半乳糖苷酶重组型芽孢表面展示的研究很多,已报道的锚定蛋白有CotB、CotC、CotE、CotG、CotX、CotY、CotZ和InhA,其分布位置主要集中在芽孢的外层与crust层。早在2009年,KIM等[19]利用枯草芽孢杆菌的衣壳蛋白CotE作为分子载体展示E. coli β-半乳糖苷酶,结果显示其酶活远低于CotG作为分子载体得到的。HWANG等[20]以CotE和CotG为锚定蛋白展示E. coli β-半乳糖苷酶时,发现当使用CotE时芽孢壁的完整性会受到影响,然而当锚定蛋白是CotG时芽孢壁则完全不受影响,表明CotG是展示E. coli β-半乳糖苷酶的较佳锚定蛋白。有国内学者[21-23]将嗜热脂肪芽孢杆菌(Bacillus stearothermophilus IAM11001)β-半乳糖苷酶分别与CotB、CotC、CotG、CotX、CotY、CotZ融合,在B. subtilis 168芽孢上成功展示β-半乳糖苷酶,测得酶活依次为0.30、0.14、0.46、0.42、0.12和0.25 U/mg芽孢(干重)。通过上述分析可知,重组芽孢表面展示的β-半乳糖苷酶活力与锚定蛋白的分布位置、分子尺寸大小有直接关系,表明锚定蛋白成为影响β-半乳糖苷酶展示效率的关键因素。
根据推测的芽孢表面展示过程及芽孢结构,目标蛋白与锚定蛋白形成的融合蛋白由于在芽孢表层的位置分布特点,其表达效率会在一定程度上受到某些丰富的、不完全协同的芽孢衣壳蛋白的影响。例如,TAVASSOLI等[24]利用CotC将B. subtilis 168 β-半乳糖苷酶分别展示在野生型和CotC缺失型芽孢表面,结果发现前者重复使用3次后残留酶活仅保留了50%,而后者仍达初始酶活的80%,说明CotC的缺失有助于进一步增强β-半乳糖苷酶的热稳定性。表达载体的类型也是影响展示效率的因素之一。WANG等[23]以CotX为锚定蛋白研究了整合型和游离型表达方式对B. stearothermophilus IAM11001 β-半乳糖苷酶在芽孢表面展示效果的影响,两种表达方式得到的活力分别为0.42和1.34 U/mg芽孢(干重),表明游离型表达方式更有利于β-半乳糖苷酶在芽孢表面进行展示。
相较于重组型芽孢表面展示体系,非重组型芽孢表面展示体系和无锚定蛋白型芽孢表面展示体系的提出与构建为目标蛋白的表达提供了新思路。非重组型芽孢表面展示体系主要利用在酸性介质中外源蛋白与芽孢表面之间的吸附作用,这种展示策略类似于以芽孢为基质的固定化。SIREC等[18]利用芽孢吸附作用原理把酸热脂环酸杆菌(Alicyclobacillus acidocaldarius)β-半乳糖苷酶展示在芽孢表面,通过对crust层蛋白缺失的突变型与野生型芽孢进行深入分析,发现缺失任一crust层蛋白、外层结构完全缺失的芽孢比野生型芽孢具有更高的吸附效率。与非重组型芽孢表面展示体系相比,无锚定蛋白型芽孢表面展示体系是基于σE和σK控制的时期特异性型启动子Pcry1Aa调控外源蛋白在芽孢形成后期的芽孢母细胞中表达进而分泌在芽孢表层策略,芽孢结构的疏水性、高电荷或静电的芽孢表层对外源蛋白的吸附也起到促进作用[25]。通过这种方式构建的展示体系既省去了酶制剂的提纯步骤,也克服了目标蛋白易于脱离芽孢的问题。2014年,PAN等[25]首次报道了在不使用锚定蛋白情况下利用Pcry1Aa启动子将E. coli β-半乳糖苷酶展示在芽孢表面,流式细胞仪、免疫电镜和蛋白酶耐受性分析实验确证了该酶的成功表达。然而目前仅有上述一篇报道文献,说明其潜力仍有待进一步探究。
枯草芽孢杆菌被认为是芽孢表面展示体系的优选宿主,但也有学者[26]发现苏云金芽孢杆菌(B. thuringiensis)外层蛋白InhA亦可将E. coli β-半乳糖苷酶成功表达在芽孢表面,测得的酶活为0.4 U/mg芽孢(干重)。该研究丰富了β-半乳糖苷酶芽孢表面展示系统,也为其他外源蛋白提供了一条新的展示途径。
2 β-半乳糖苷酶微生物细胞表面展示系统的应用
β-半乳糖苷酶微生物细胞表面展示系统的应用,主要集中在酶的制造、作为报告蛋白和全细胞催化剂。
2.1 生产β-半乳糖苷酶
将β-半乳糖苷酶锚定在微生物细胞或芽孢表面类似于对β-半乳糖苷酶进行固定化,但这种表面展示策略省去了冗繁的酶提纯步骤及传统的固定化操作,并且表现出更好的稳定性,使得工业化生产β-半乳糖苷酶在不久将来变成可能。近年来,国内外学者对β-半乳糖苷酶的微生物细胞表面展示体系进行了很多有益探究。例如,SIREC等[18]基于芽孢表层结构特点和目的酶蛋白特性将A. acidocaldarius β-半乳糖苷酶吸附固定在野生型和突变型芽孢上,得到的展示酶在酸性介质中的热稳定性显著增强。TAVASSOLi等[24]发现由CotC直接融合得到的、表面展示有枯草芽孢杆菌β-半乳糖苷酶的重组芽孢经-20 ℃冷冻或37 ℃干燥复苏后,仅有小部分酶活损失,重复使用3次后仍可保持80%的活性。
2.2 作为报告蛋白
β-半乳糖苷酶作为一种相对分子质量大的四聚体蛋白,常作为模式蛋白质广泛用于生化检测、科研分析等领域。基于β-半乳糖苷酶而开发的各类微生物细胞表面展示系统已在评估锚定蛋白锚定能力[6-7,9,22],分析细胞壁、芽孢壁结构组成[19-20]等方面取得了良好的应用效果。
2.3 全细胞催化剂
据报道,来自韩国的PAN课题组是最早开展β-半乳糖苷酶微生物细胞表面展示的研究团队,他们做了很多原创性的工作,其中影响力较大则是有关表面展示β-半乳糖苷酶的重组芽孢在两相体系中转糖基化反应方面的研究[3]。该文报道了E. coli β-半乳糖苷酶经CotG锚定展示后,得到的重组芽孢酶活力达5×103 U/g(干重),热稳定性和抗有机溶剂(如正己烷、乙醚、甲苯)的能力也显著增加,而且重组芽孢在磷酸缓冲液-乙醚双相反应体系中可催化100 mmol/L乳糖和100 mmol/L正辛醇产生27.7 mmol/L辛烷基-β-D-吡喃半乳糖苷。该研究同时指出目标产物产量增加的原因与重组芽孢主要分散在水相-有机相的界面有直接关系。在此基础上,该课题组通过向上述两相体系中加入1, 2-二甲氧基乙烷作为助溶剂的策略,在相同浓度底物条件下,使得辛烷基-β-D-吡喃半乳糖苷的产量提高到33.7 mmol/L[27]。新近研究[28]显示,基于“Cohesin-Dockerin”特异性相互作用原理构建的β-半乳糖苷酶枯草芽孢杆菌芽孢表面展示体系,通过优化纤维小体中黏附模块-对接模块的组成和空间结构,使得表面展示的酶蛋白高达1.4×103/芽孢,酶的热稳定性、重复利用性和贮藏稳定性也进一步提高,其在两相体系中合成烷基糖苷的转化率达35%(质量分数)。β-半乳糖苷酶在芽孢表面展示后不仅使其可重复利用性提升,其对有机溶剂的耐受性也大大增强。这些优良的特质使得β-半乳糖苷酶的芽孢表面展示系统在两相体系催化领域有着广阔的发展空间。
生物酶法合成功能性低聚糖一直是研究的热点。WANG等[23]以CotX介导的β-半乳糖苷酶芽孢表面展示体系作为全细胞生物催化剂,能催化200 g/L乳糖和100 g/L果糖合成8.8 g/L乳果糖,可重复利用测试结果显示该重组芽孢在连续转化8次后仍保持初始活性的30.3%。LI等[13]利用酿酒酵母展示的P. expansum β-半乳糖苷酶催化乳糖(100 g/L)合成GOS,其转化率达43.64%(质量分数),重复使用9次后,GOS产量和残留的β-半乳糖苷酶活力均保持在较高水平。同时,该催化体系产生的反应产物葡萄糖和半乳糖还可分别用作酵母的碳源和表达β-半乳糖苷酶基因的诱导剂,因此实现了真正意义上的生物循环。在赤藓糖醇发酵工业中,每年都有数千吨的废酵母膏因无法处理而带来环境问题。AN等[14]以表面展示有米曲霉β-半乳糖苷酶的Y. lipolytica CGMCC 11369为全细胞催化剂,在500 g/L乳糖、5 mg/mL细胞(干重)、pH 5.5、60 ℃条件下,GOS产量达160 g/L,转化率为51%(质量分数),且循环利用10次后重组酵母细胞仍能保留初始酶活的80%。这些研究结果表明β-半乳糖苷酶的微生物表面展示体系作为全细胞催化剂在资源循环利用方面有着巨大的潜力。
3 结语
相比于游离酶与固定化酶,β-半乳糖苷酶的微生物表面展示技术克服了诸如稳定性差、有机溶剂不耐受、难以回收、纯化过程复杂等问题。综合上述3大微生物表面展示系统各自的优缺点,不难发现,虽然乳酸菌表面展示系统能利用表层蛋白和一些细胞壁结合结构域展示β-半乳糖苷酶,但是原核表达的缺陷以及较厚的细胞壁始终限制着β-半乳糖苷酶的体内表达。酵母细胞由于是真核宿主,拥有促进蛋白折叠和正常分泌的生物合成装置,较之乳酸菌,酵母细胞表面展示β-半乳糖苷酶有着很大的优势。但是较差的遗传稳定性和较少的可供选择的锚定蛋白等因素严重制约着该展示系统的进一步开发与应用。与前两者展示系统不同的是,凭借着芽孢自身独特的抗逆性与特殊的无细胞膜结构,β-半乳糖苷酶的芽孢表面展示系统克服了诸如β-半乳糖苷酶表达折叠不正确、与宿主结合不够牢固等缺点,使得其在工业用酶的生产、作为报告蛋白和全细胞催化剂领域有着十分乐观的应用前景。不过目前成功实现芽孢表面展示的β-半乳糖苷酶酶活较低,影响展示效率的机制也不明确,深入研究芽孢的结构与衣壳蛋白的组装机理,开发新的锚定蛋白和展示策略将有利于提高β-半乳糖苷酶展示效率。构建人工芽孢颗粒也为表面展示技术在β-半乳糖苷酶领域的广泛应用提供了新思路。可以预见,β-半乳糖苷酶的微生物表面展示系统尤其是芽孢表面展示系统将会成为未来β-半乳糖苷酶产业应用和发展的主流。
参考文献
[1] 贺璐,龙承星,刘又嘉,等.微生物乳糖酶研究进展[J].食品与发酵工业,2017,43(6):268-273.
[2] LEE S Y,CHOI J H,XU Zhaohu.Microbial cell-surface display[J].Trends in Biotechnology,2003,21(1):45-52.
[3] KWON S J,JUNG H C,PAN J G.Transgalactosylation in a water-solvent biphasic reaction system with β-galactosidase displayed on the surfaces of Bacillus subtilis spores[J].Applied and Environmental Microbiology,2007,73(7):2 251-2 256.
[4] MICHON C,LANGELLA P,EIJSINK V G H,et al.Display of recombinant proteins at the surface of lactic acid bacteria: strategies and applications[J].Microbial Cell Factories,2016,15(1):70.
[5] HYNÖNEN U,PALVA A.Lactobacillus surface layer proteins: structure, function and applications[J].Applied Microbiology and Biotechnology,2013,97(12):5 225-5 243.
[6] HU Shumin,KONG Jian,SUN Zhilan,et al.Heterologous protein display on the cell surface of lactic acid bacteria mediated by the s-layer protein[J].Microbial Cell Factories,2011,10(1):86.
[7] HU Shumin,KONG Jian,KONG Wentao,et al.Characterization of a novel LysM domain from Lactobacillus fermentum bacteriophage endolysin and its use as an anchor to display heterologous proteins on the surfaces of lactic acid bacteria[J].Applied and Environmental Microbiology,2010,76(8):2 410-2 418.
[8] VISWESWARAN G R,LEENHOUTS K,VAN R M,et al.Exploiting the peptidoglycan-binding motif, LysM, for medical and industrial applications[J].Applied Microbiology and Biotechnology,2014,98(10):4 331-4 345.
[9] XU W,HUANG M,ZHANG Y,et al.Novel surface display system for heterogonous proteins on Lactobacillus plantarum[J].Letters in Applied Microbiology,2011,53:641-648.
[10] WIECZOREK A S,MARTIN V J.Effects of synthetic cohesin-containing scaffold protein architecture on binding dockerin-enzyme fusions on the surface of Lactococcus lactis[J].Microbial Cell Factories,2012,11(1):160.
[11] GAI S A,WITTRUP K D.Yeast surface display for protein engineering and characterization[J].Current Opinion in Structural Biology,2007,17(4):467-473.
[12] KÖNNING D,KOLMAR H.Beyond antibody engineering: directed evolution of alternative binding scaffolds and enzymes using yeast surface display[J].Microbial Cell Factories,2018,17(1):32.
[13] LI Yumei,LU Lili,WANG Hongmei,et al.Cell surface engineering of a β-galactosidase for galactooligosaccharide synthesis[J].Applied and Environmental Microbiology,2009,75(18):5 938-5 942.
[14] AN Jin,ZHANG Lebin,LI Lijuan,et al.An alternative approach to synthesizing galactooligosaccharides by cell-surface display of β-galactosidase on Yarrowia lipolytica[J].Journal of Agricultural and Food Chemistry,2016,64(19):3 819-3 827.
[15] KIM J,SCHUMANN W.Display of proteins on Bacillus subtilis endospores[J].Cellular and Molecular Life Sciences,2009,66(19):3 127-3 136.
[16] ISTICATO R,RICCA E.Spore surface display[J].Microbiology Spectrum,2014,2(5):TBS-0011-2012.doi:10.1128/microbiolspec.TBS-0 011-2 012.
[17] WANG He,WANG Yunxiang,YANG Ruijin.Recent progress in Bacillus subtilis spore-surface display: concept, progress, and future[J].Applied Microbiology and Biotechnology,2017,101(3):933-949.
[18] SIREC T,STRAZZULLI A,ISTICATO R,et al.Adsorption of β-galactosidase of Alicyclobacillus acidocaldarius on wild type and mutants spores of Bacillus subtilis[J].Microbial Cell Factories,2012,11(1):100.
[19] KIM J H,KIM B G,CHOI S K,et al.Method for expression of proteins on spore surface:US,US7582426[P].2009-09-01.
[20] HWANG B Y,PAN J G,KIM B G,et al.Functional display of active tetrameric β-galactosidase using Bacillus subtilis spore display system[J].Journal of Nanoscience and Nanotechnology,2013,13(3):2 313-2 319.
[21] 王贺,杨瑞金,华霄,等.利用枯草芽孢衣壳蛋白表面展示β-半乳糖苷酶[J].食品与发酵工业,2012,38(7):1-5.
[22] WANG He,YANG Ruijin,HUA Xiao,et al.Functional display of active β-galactosidase on Bacillus subtilis spores using crust proteins as carriers[J].Food Science and Biotechnology,2015,24(5):1 755-1 759.
[23] WANG He,YANG Rui-jin,HUA Xiao,et al.An approach for lactulose production using the CotX-mediated spore-displayed β-galactosidase as a biocatalyst[J].Journal of Microbiology and Biotechnology,2016,26(7):1 267-1 277.
[24] TAVASSOLI S,HINC K,IWANICKI A,et al.Investigation of spore coat display of Bacillus subtilis β-galactosidase for developing of whole cell biocatalyst[J].Archives of Microbiology,2013,195(3):197-202.
[25] PAN J G,CHOI S K,JUNG H C,et al.Display of native proteins on Bacillus subtilis spores[J].FEMS Microbiology Letters,2014,358(2):209-217.
[26] PARK T J,CHOI S K,JUNG H C,et al.Spore display using Bacillus thuringiensis exosporium protein InhA[J].Journal of Microbiology and Biotechnology,2009,19(5):495-501.
[27] BAE J,CHOI E H,PAN J G.Efficient synthesis of octyl-β-D-galactopyranoside by Bacillus spore-displayed β-galactosidase using an amphiphilic 1,2-dimethoxyethane co-solvent[J].Enzyme and Microbial Technology,2011,48(3):232-238.
[28] CHEN Long,HOLMES M,SCHAEFER E,et al.Highly active spore biocatalyst by self-assembly of co-expressed anchoring scaffoldin and multimeric enzyme[J].Biotechnology and Bioengineering,2018,115(3):557-564.
Overview of microbial cell surface display of β-galactosidase and its application
JIANG Xiaomin1, WANG He2*, WANG Yunxiang1,2*,QIAN Yongchang3, YIN Lianghong3, FAN Li2
1(School of Agricultural and Food Sciences, Zhejiang A&F University, Hangzhou 311300, China) 2(Jiyang College, Zhejiang A&F University, Shaoxing 311800, China) 3(School of Forestry and Biotechnology, Zhejiang A&F University, Hangzhou 311300, China)
Abstract β-galactosidase is an important industrial enzyme with broad applications in dairy production, medicine and biochemical analysis. The most commercially available β-galactosidase is generally made in free form. However, the free β-galactosidase requires complex production steps and often shows low stability, easy inactivation, poor tolerance to organic solvents, and poor reusability, which restrict its use in industrial application. The microbial cell surface display technology that enables the expression of β-galactosidase on the cell surface through genetic engineering confers several advantages: 1) there is no need for complicated purification and immobilization steps during industrial production process, thus lowering the cost; 2) owing to its good stability and excellent resistance to organic solvents, cell-surface displayed β-galactosidase can be used as an catalyst under both mild and harsh conditions; 3) widens its application feilds in protein engineering and biosensor. The hosts used for surface display of β-galactosidase include lactic acid bacteria, yeast and spore. In this paper, recent progress on microbial cell surface display for β-galactosidase and its application in enzyme production, acting as an report protein as well as whole cell biocatalysis were summarized.
Key words β-galactosidase;surface display;whole cell catalyst