多组学技术联用在传统发酵乳品风味代谢调控中的应用研究进展
周钺1,李键2,张玉1,王洪伟1,赵欣3,刘士健1,索化夷1*
1(西南大学 食品科学学院,重庆,400715) 2(西南民族大学 生命科学与技术学院,四川 成都,610041) 3(重庆第二师范学院,重庆市功能性食品协同创新中心,重庆,400067)
摘 要传统发酵乳制品具有独特的风味品质、营养价值和丰富的菌种资源。由于发酵微生物的区系复杂多样,传统发酵乳制品的风味品质形成机理不清,质量安全无法保证,阻碍了传统发酵乳制品的规模化生产。基于高通量组学技术的发展,多组学技术联用为分析传统发酵乳品的风味形成条件与菌种代谢通路提供了有利手段。该文综述了国内外多组学技术联用在传统发酵乳品的风味代谢调控中的研究应用并对此展望,为传统发酵乳品的品质调控和生产研发提供新思路。
关键词传统发酵乳品;多组学技术联用;特征风味成分;代谢通路;功能基因
发酵乳品源于我国游牧民族,多以牛羊驼马等动物乳为原料,涉及多种微生物共栖生长,发酵形成独特的风味品质和功能成分,同时延长乳品的保质期[1]。区别于现代发酵方式,传统发酵没有固定的发酵“起子”,发酵过程更加复杂多样。表1归纳了不同类型的传统发酵乳品及发酵优势菌种[2-3],发酵乳品中微生物群落存在个体代谢和复杂的相互作用,微生态的多样性和行为差异无法保证最终发酵产品的品质稳定和安全[4]。为了阐明传统发酵乳品风味品质的形成机理,对其实现定向调控,势必要解析多菌种相互作用与风味代谢通路间的相关性。
当前,多组学技术联用可以涵盖环境所有微生物的基因表达、蛋白或代谢物差异的多层面分析,快速获取海量数据。利用多组学联合揭示传统发酵乳品特征风味物质的形成机理,并以此为导向筛选功能微生物,从而对发酵生产进行指导[5]。我国传统发酵乳品的种类丰富,大量的菌种资源有待挖掘。本文总结了近年来多组学技术在解析传统发酵乳品特征风味成分的形成机理及其代谢调控的研究进展,并对此领域的应用前景进行展望(表1)。
表1 传统发酵乳制品类型及主要发酵剂
Table 1 The type of traditional fermented dairy products and main starter

发酵类型 原料乳类型 发酵乳品 主要菌种细菌乳酸发酵脱脂乳酪乳乳酸乳球菌、肠膜明串珠菌乳脂亚种酸奶德氏保加利亚乳杆菌亚种、嗜热链球菌、瑞氏乳杆菌真菌乳酸发酵全脂乳山羊乳、牛乳脱脂乳马乳维利乳酸乳球菌、肠膜明串珠菌乳脂亚种、白地霉卡门培尔干酪乳酸乳球菌乳酸亚种、干酪乳杆菌、乳酸克鲁维酵母、白地霉、青霉开菲尔酸奶马乳酒样乳杆菌、开菲尔乳杆菌、肠膜明串珠菌、酿酒酵母、马克思克鲁维酵母曲拉干酪乳杆菌、植物乳杆菌、乳酸乳球菌、酿酒酵母、发酵毕赤酵母蒙古Koumiss嗜酸乳杆菌、德氏保加利亚乳杆菌亚种、乳酸酵母、圆酵母
1 多组学技术在传统发酵食品的应用现状
近年来,微生态多样性的鉴定由单菌落传统分离培养法及其核酸片段的PCR-DGGE电泳图谱向高通量测序技术转变[6]。基于高通量测序的宏基因组学以环境中所有微生物的基因组为研究对象,分析菌群多样性并对不同代谢通路的功能基因聚类。LIU等首次利用功能基因组的多位点序列分型技术,成功将保加利亚乳杆菌发酵的酸化速率、产乙醛等表型特征与其功能基因序列结合,建立了一种可快速鉴定发酵菌是否具有良好发酵性能的方法[7]。利用色谱、质谱串联分析和光谱非靶向高精度定量的特点,使蛋白质组学和代谢组学作为表征菌落代谢产物的实时原位检测和定量分析的有力方法,已用于检测指导发酵乳制品中微生物和代谢物的实时变化[8-9]。
单个组学通常仅代表核酸、蛋白质或代谢物一个层面的研究结果,对于物种之间的相互作用和表型预测缺少可靠的数据支撑。多组学联合将各个水平的物质分析“打通”,在探索生物于环境变化中细胞间的发育分化、信号传导和互作网络,为宿主响应环境改变做出的应激扰动、宿主内部代谢通路的调控提供有效调控靶点,是系统生物学研究发展的一大趋势[10]。当前,多组学技术联用作为解析传统发酵食品“菌群-基因-代谢产物”关联性的主要手段,从多水平剖析验证食品微生物的组成对发酵风味品质的形成机理[11]。
2 传统发酵乳制品主要风味成分
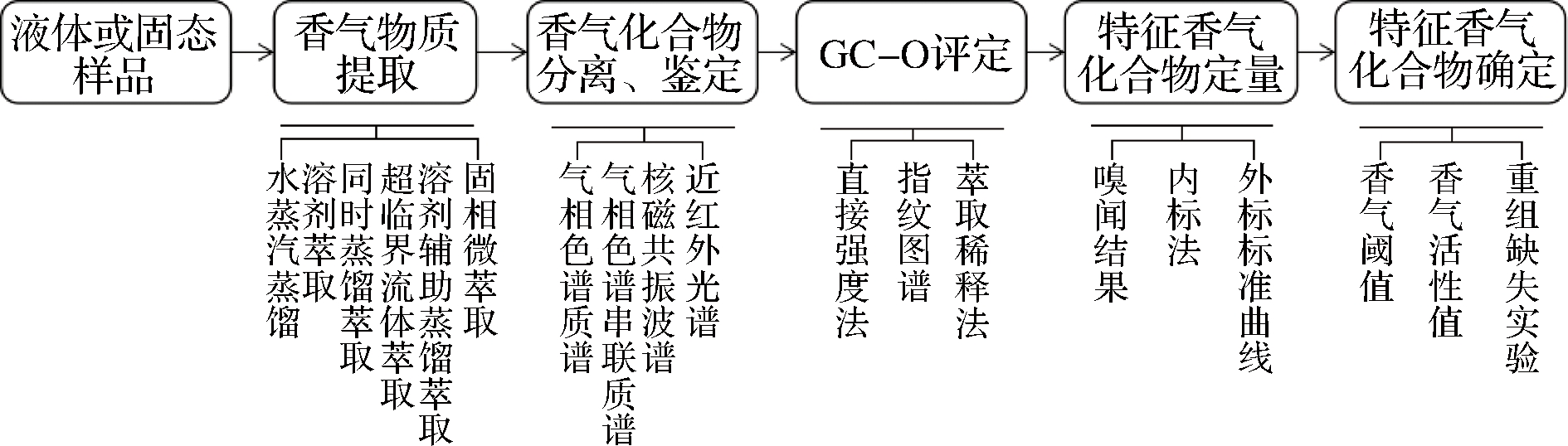
特征风味作为发酵食品的“骨架”,是传统发酵食品高附加值的体现。由于每种风味成分具有不同的物性,仅凭单一的手段无法将所有风味成分完整地提取出来[12]。随着近几年对传统发酵食品的风味导向调控成为研究热点,利用代谢组学对痕量风味物质定性、定量,并结合电子鼻或嗅辨仪挖掘特征风味成分(或关键活性风味物质),为进一步阐明传统发酵食品的风味形成机理和核心微生物的代谢机制奠定了基础[13]。本文参考徐岩等[14]对中国白酒、精油特征风味成分的挖掘,绘制了特征风味成分判定的技术路线(如图1所示)[14-16]。
乳制品发酵风味取决于乳源中脂肪、蛋白和碳水化合物的相对平衡,其中乳酸、乙醛、双乙酰、丁二酮和乙偶姻被证实对风味有很大影响。此外,在开菲尔、酸马奶、意大利干酪等有酵母参与的发酵乳制品中,醇类物质是清香和酒香的重要贡献者[17-18]。表2参考CHEN等总结列出了发酵乳制品中主要风味成分及特征描述[19]。
表2 发酵乳品中主要风味化合物、香气描述及阈值
Table 2 Main flavor compounds, aroma description and threshold in fermented dairy products

类别风味化合物香气描述阈值/(mg·kg-1)酮类双乙酰、乙偶姻、2,3-戊二酮、甲基酮黄油、奶油味0.001~0.020丙酮、2-庚酮、2-壬酮、1-壬烯-3-酮、1-辛烯-3-酮果香、甜味8×10-9~0.832醛类己醛、正辛醛、反式-2-壬烯醛、3-甲基丁醛草香、脂肪0.000 4~0.009 0乙醛、苯乙醛果香、花香0.004~0.039 0酸类异丁酸、戊酸、己酸香甜、花香0.05~1.84辛酸、3-甲基丁酸奶酪味0.1~2.0酯类乙酸乙酯、乙酸丁酯、γ-癸内酯、丁酸乙酯果香、花香0.001~3.300
挥发性醛、酮等羰基类化合物的含量对发酵乳制品的香气有显著影响,乙醛和2,3-戊二酮的形成与苏氨酸代谢通路有关[20-21]。挥发性醛类化合物是发酵乳中的微量香气组分,微量的乙醛(23~40mg/kg)赋予酸乳青苹果或坚果味,过多的醛类会带来青草臭[22]。酮类物质多具有果香、花香和奶香味,其中双乙酰是酸乳的特征风味物质,易被微生物的双乙酰还原酶还原成乙偶姻。乙偶姻和双乙酰具有相似的甜奶油香气,但乙偶姻的风味明显弱于双乙酰,且在适当范围(1.2~28.2mg/kg)可以减弱丁二酮的刺激性[23-24]。酯酶和醇酰基转移酶的活性以及含醇量是发酵乳制品中酯类成分含量的限制因素,酯类的香气阈值较低而对发酵乳的果味形成有很大贡献[25]。不同浓度的酯类含量对发酵乳风味的贡献不同,低浓度的酯类物质对整体风味的平衡有积极作用,还可以掩盖或弱化过多的短链脂肪酸造成的异味或“不洁”味道[26]。
3 多组学联用在微生物代谢与风味形成机理研究的应用
传统发酵乳品是以乳酸菌、酵母和霉菌为主的多菌种共栖发酵,发酵风味的形成与菌群相互作用、发酵环境改变引起的代谢通路相关酶的基因表达变化有关。通过多组学联用对微生态多样性、基因和代谢水平的差异分析,明确与特征风味成分形成相关的微生物及其基因的转录表达,表征了发酵风味物质代谢调控关键的“生物标志”。基于传统发酵乳制品品种多样,制作工艺和发酵菌种地域性差异较大,其中以干酪微生物研究最多。后文以传统发酵干酪中的微生物代谢研究为例,介绍近年组学联合在发酵微生物和风味代谢有关基因的研究应用。
3.1 发酵优势菌种及代谢功能基因
同所有传统发酵食品一样,干酪中的微生物种类随发酵成熟而变化。利用宏基因组联合转录组测序对菌群演替和相关基因表达的实时监测,取代了传统菌落计数法无法测量死细胞和不可培养菌的弊端,明确了样本中活细胞和死细胞的DNA分子比例,便于了解不同发酵阶段的优势菌种[27]。
研究表明,意大利干酪由Lb.casei和Lb.buchneri等非“起子”乳杆菌驱动成熟,其丰度随成熟环境温度的升高而增加[28]。Geotrichum candidum和Penicillium camemberti等真菌是卡门培尔干酪成熟的主要贡献菌,前者具有代谢氨苦味肽、增强干酪硫磺味的能力。功能基因注释表征了干酪微生物含多种酪蛋白和氨基酸的分解酶系,其中P.freudenreichii subsp.shermanii JS含有编码丝氨酸蛋白酶HtrA和丝氨酸脱氨酶基因,以及寡肽转运蛋白基因和参与支链脂肪酸形成的支链氨基酸氨基转移酶基因ilve[29]。同时,P.freudenreichii含有一些胞内肽酶基因,如pepO,pepC,pepE,然而尚未阐明这些肽酶基因的存在对乳品苦味肽的降解有所贡献[30]。
干酪内部的碳水化合物代谢以乳糖分解和柠檬酸盐代谢为主,最终形成乙醇、乳酸和一些C4挥发性风味成分。然而很少有乳酸菌可直接利用柠檬酸盐,利用宏基因组学可以判断柠檬酸代谢基因的有无,或通过编码转录调节因子草酰乙酸脱羧酶和柠檬酸转运蛋白基因对其分类[31]。尽管在Lb.rhamnosus和Lb.helveticus均有编码柠檬酸裂解酶的基因citD,citF和citE,但仅有Lb.rhamnosus拥有草酰乙酸脱羧酶基因,能够将柠檬酸盐降解为丙酮酸。
3.2 风味物质形成途径与菌种代谢功能基因表达
干酪中的微生物区系及其在发酵过程的活性决定了最终的发酵风味,风味物质需要通过代谢有关基因的转录表达与发酵微生物建立关系。基于宏基因组、转录组和代谢组联合对菌群代谢基因表达和风味物质的产生时期进行关联分析,以揭示发酵风味的形成机理。
干酪中乳糖的分解代谢是形成C4挥发性成分的主要途径。联合转录组测序研究,发现乳糖转运和利用的相关基因片段在成熟早期由霉菌显著表达,未发现酵母含有乳糖代谢基因,证实G.candidum不具有乳糖分解能力。编码乳糖特异性磷酸烯醇式丙酮酸转运系统的基因lacEF和ptsl以及参与糖酵解和异生途径的转录基因主要在Lc.lactis中高丰度表达[27],此外,P.camemberti等真菌中的磷酸烯醇式丙酮酸激酶和果糖-1,6-二磷酸酶基因在干酪成熟早期高度表达,基于真菌代谢对干酪成熟的重要性,将磷酸烯醇式丙酮酸激酶基因是否表达作为评判卡门培尔奶酪成熟过程正常与否的生物标志物[32]。干酪内部的碳水化合物代谢以乳糖分解和柠檬酸盐代谢为主,最终形成乙醇、乳酸和一些C4挥发性风味成分。柠檬酸盐作为乳制品的特征风味化合物双乙酰和乙偶姻的前体物质,也可以通过微生物降解。研究发现,瑞士干酪冷藏后熟期间,Lb.rhamnosus中乳糖和柠檬酸盐分解相关的基因下调,使得后熟风味物质双乙酰、乙醛降低。然而,Lc.lactis的代谢基因与之相反,其L-乳酸脱氢酶基因(ldh)在后熟期间显著上调,促使同型乳酸发酵的进行[30]。
酪蛋白和氨基酸是多种挥发性化合物的前体,前者通过几种膜蛋白酶PrtP,PrtH和PrtR降解为肽,经寡肽,二肽和三肽蛋白转运家族转运进入细胞中被肽酶水解成氨基酸;后者通过转氨基、脱氢、脱羧和C-S键断裂等反应酶系分解[30]。研究发现Lc.lactis具有两条氨基酸分解途径,其一由氨基转移酶bcaT和araT催化支链氨基酸和芳香族氨基酸形成谷氨酸。当支链氨基酸含量较低时,乳酸乳球菌IFPL730产生3-甲基丁醛及其还原物质3-甲基丁醇等代谢通路上的有关基因araT,bcaT,kivD,ytjE和panE的表达增加,2种物质均为切达奶酪的特征风味成分。Lc.lactis中存在另一条由β-或γ-胱硫醚裂解酶催化甲硫氨酸脱氨、脱甲硫醇反应[33]。含硫化合物的阈值低,微量即可具有强烈风味。通过宏基因组功能聚类关联风味化合物丰度分析,表征了切达干酪成熟后期Brevibacterium linens、G.candidum和Staphylococcus xylosus的出现与甲硫醇及衍生物二甲硫醚的形成显著相关[34]。
干酪熟化期间硬壳表面以碳水化合物代谢和细胞分裂代谢为主,而内部以氨基酸和脂质代谢为主。通常,脂肪分解与氨基酸代谢遵循相同的动力学,即在干酪成熟第一周显著表达,随后趋于缓慢稳定,产生的游离脂肪酸有利于干酪风味品质的形成。宏转录组数据显示G.candidum中脂肪代谢相关酶的基因高度表达,且表达水平随干酪成熟而提高,是干酪脂肪分解的主要贡献菌[27]。较高的熟化温度促进了干酪表面的乙偶姻和双乙酰合成酶显著表达,使得由丙酮酸合成脂肪酸代谢通路的基因表达上调,同时导致干酪内部参与脂肪酸β-氧化的基因过表达,增加干酪内部和表面的丁酸、戊酸等短链脂肪酸形成[28]。然而,高水平的脂肪酸带来脂肪酸败和乳臭味等不良感官影响,研究表明L.lactis中游离脂肪酸合成的代谢通路受长链酰基载体蛋白抑制物调控,避免游离脂肪酸的积累导致整体风味的失衡[35]。
4 展望
多组学技术联用被视为微生物研究的未来,在分析菌群演替、确定功能基因、发掘活性成分的代谢网络等具有“1+1>2”的显著优势。但是,国内外利用多组学技术在解析传统发酵乳制品微生物演替或代谢机理的研究尚处于起步阶段。一方面,传统发酵乳品的制作工艺存在差异性,发酵环境具有空间异质性和动态可变性,发酵菌群不均匀分布,导致不同生长点的微生物通过不同机制的相互作用各自演化,出现多种表型,使得多组学联用的结论缺少代表性。其次,测序平台和质谱仪器的研发速度超过了分析软件或数据库的开发,导致迄今仍有海量的数据无法得到充分解析。
随着组学检测成本的降低,通过多组学实时监测发酵进程中微生物的动态变化、差异基因表达和代谢物的组成来构建发酵菌群预测模型,势必成为传统发酵食品主要的研究方向。将环境因子纳入多组学的大数据分析,来预测特定环境中微生物群落的生理特性和结构演替,使多组学数据能更客观地揭示发酵菌群的互作机制。对于获取的多组学海量数据,配套的数据库和多元统计分析模型的建立也需及时跟进。最后,传统分离培养是验证多组学数据预测微生态结构最直观的方法,对于新基因的注释、功能表征和物种的生理鉴定都是必要的,目前多组学数据结合传统分离培养的结果仍是系统生物学中缺失的一环[36]。综上,多组学联用正在各种传统发酵食品的研究中逐步普及,然而面对大数据如何分析挖掘、如何使结果能客观还原传统发酵体系,以表征传统发酵食品的品质成熟机理是首要问题。
参考文献
[1] 董杰, 张和平. 中国传统发酵乳制品发展脉络分析[J]. 中国乳品工业, 2014, 42(11): 26-30.
[2] 王豪, 刘振民, 章慧. 酸马奶和维利的研究进展[J]. 食品科学, 2016, 37(3): 247-252.
[3] 陆东林, 刘朋龙, 李新玲,等. 传统发酵乳及其发展前景[J]. 中国乳业, 2018,202(10): 62-68.
[4] MONTEL M C, BUCHIN S, MALLET A, et al. Traditional cheeses: Rich and diverse microbiota with associated benefits[J]. Int J Food Microbiol, 2014,177(54): 136-154.
[5] 马海霞, 张丽丽, 孙晓萌, 等. 基于宏组学方法认识微生物群落及其功能[J]. 微生物学通报, 2015, 42(5): 902-912.
[6] COCOLIN L, ERCOLINI D. Zooming into food-associated microbial consortia: a ‘cultural’ evolution[J]. Current Opinion in Food Science, 2015, 2:43-50.
[7] LIU W, YU J, SUN Z, et al. Relationships between functional genes inLactobacillus delbrueckii ssp.bulgaricus isolates and phenotypic characteristics associated with fermentation time and flavor production in yogurt elucidated using multilocus sequence typing[J]. Journal of Dairy Science, 2016, 99(1):89-103.
[8] KARADUMAN A, OZASLAN M O, KILIC I H, et al. Identification by using MALDI-TOF mass spectrometry of lactic acid bacteria isolated from non-commercial yogurts in southern Anatolia, Turkey[J]. International Microbiology, 2017, 20(1): 25-30.
[9] LU Y , ISHIKAWA H , KWON Y, et al. Real-Time monitoring of chemical changes in three kinds of fermented milk products during fermentation using quantitative difference nuclear magnetic resonance spectroscopy[J]. Journal of Agricultural and Food Chemistry, 2018, 66(6): 1 479-1 487.
[10] KOPCZYNSKI D, COMAN C, ZAHEDI R P, et al. Multi-OMICS: A critical technical perspective on integrative lipidomics approaches[J]. BBA - Molecular and Cell Biology of Lipids, 2017, 1 862(8):808-811.
[11] ELEONORA S, ANDREANI N A, LISA C, et al. A multi-omics approach to evaluate the quality of milk whey used in ricotta cheese production[J]. Frontiers in Microbiology, 2016, 7: 1-13.
[12] 孙宝国,吴继红,黄明泉, 等. 白酒风味化学研究进展[J]. 中国食品学报, 2015, 15(9): 1-8.
[13] NIU Y, YAO Z, XIAO Q, et al. Characterization of the key aroma compounds in different light aroma type Chinese liquors by GC-olfactometry, GC-FPD, quantitative measurements, and aroma recombination[J]. Food Chemistry, 2017, 233:204-215.
[14] 徐岩. 基于风味导向技术的中国白酒微生物及其代谢调控研究[J]. 酿酒科技, 2015(2): 9-19;24.
[15] SUN Jinyuan, LI Qinya, LUO Siqi, et al. Characterization of key aroma compounds in Meilanchun sesame flavor style baijiu by application of aroma extract dilution analysis, quantitative measurements, aroma recombination, and omission/addition experiments[J]. RSC Advances, 2018, 8(42): 23 757-23 767.
[16] 肖作兵,李静,牛云蔚, 等. GC-O/AEDA法结合OAV及香气重组试验鉴定玫瑰精油中特征香气化合物[J].中国食品学报, 2018, 18(4): 319-324.
[17] 丹彤, 张和平. 发酵乳中风味物质的研究进展[J]. 中国食品学报, 2018(11): 287-292.
[18] MARIA B P, PAOLA S, ANTONIO M, et al. Metabolomics and microbiological profile of Italian mozzarella cheese produced with buffalo and cow milk[J]. Food Chemistry, 2016, 192: 618-624.
[19] CHEN C, ZHAO S, HAO G, et al. Role of lactic acid bacteria on the yogurt flavour: A review[J]. International Journal of Food Properties, 2017, 20(sup1):S316-S330.
[20] ROUTRAY W, MISHRA H N. Scientific and technical aspects of yogurt aroma and taste: A review[J]. Comprehensive Reviews in Food Science & Food Safety, 2011, 10(4):208-220.
[21] SMID E J, KLEEREBEZEM M. Production of aroma compounds in lactic fermentations.[J]. Annu Rev Food Sci Technol, 2014, 5(1):313-326.
[22] CHENG H. Volatile flavor compounds in yogurt: A review[J]. Critical Reviews in Food Science & Nutrition, 2010, 50(10):938-950.
[23] GIBSON B, VIDGREN V, PEDDINTI G, et al. Diacetyl control during brewery fermentation via adaptive laboratoyr engineering of the lager yeastSaccharomyces pastorianus[J]. Journal of Industrial Microbiology & Biotechnology, 2018,10(45):1 103-1 112.
[24] 杜晓敏, 刘文俊, 张和平.传统酸马奶发酵过程中挥发性风味物质动态变化研究[J]. 中国乳品工业, 2017, 45(5): 4-9.
[25] HONG Q, LIU X M, HANG F, et al. Screening of adjunct cultures and their application in ester formation in Camembert-type cheese.[J]. Food Microbiology, 2018, 70: 33-41.
[26] MEI J, GUO Q, WU Y, et al. Study of proteolycis, lipolysis, and volatile compoumds of a Camenmbert-type cheese mangfactured using a freezidried Tibetan kofir co-cultureduring ripening[J]. Food Science and Biotechnology, 2015, 24(2): 393-402.
[27] DUGATBONY E, STRAUB C, TEISSANDIER, AURÉLIE, et al. Overview of a surface-ripened cheese community functioning by meta-omics analyses[J]. Plos One, 2015, 10(4): e0124360.
[28] DEFILIPPIS, GENOVESE A, FERRANTI P, et al. Metatranscriptomics reveals temperature-driven functional changes in microbiome impacting cheese maturation rate[J]. Scientific Reports, 2016, 6:21 871.
[29] OJALA T, LAINE P K S, AHLROOS T, et al. Functional genomics provides insights into the role ofPropionibacterium freudenreichii ssp.shermanii JS in cheese ripening[J]. International Journal of Food Microbiology, 2017, 241: 39-48.
[30] DURU I C, LAINE P, ANDREEVSKAYA M, et al. Metagenomic and metatranscriptomic analysis of the microbial community in Swiss-type Maasdam cheese during ripening[J]. International Journal of Food Microbiology, 2018, 281: 10-22.
[31] MARTINO G P, QUINTANA I M, ESPARIZ M, et al. Aroma compounds generation in citrate metabolism ofEnterococcus faecium: Genetic characterization of type I citrate gene cluster[J]. International Journal of Food Microbiology, 2016, 218:27-37.
[32] MARIEHÉL NE L, VIEL C, BOYLE B, et al. Metatranscriptome analysis of fungal strainsPenicillium camemberti andGeotrichum candidum reveal cheese matrix breakdown and potential development of sensory properties of ripened Camembert-type cheese[J]. BMC Genomics, 2014,15:235.
NE L, VIEL C, BOYLE B, et al. Metatranscriptome analysis of fungal strainsPenicillium camemberti andGeotrichum candidum reveal cheese matrix breakdown and potential development of sensory properties of ripened Camembert-type cheese[J]. BMC Genomics, 2014,15:235.
[33] GARC ACAYUELA T, LP G D C, PEL
ACAYUELA T, LP G D C, PEL EZ C, et al. Expression inLactococcus lactis of functional genes related to amino acid catabolism and cheese aroma formation is influenced by branched chain amino acids[J]. International Journal of Food Microbiology, 2012, 159(3): 207-213.
EZ C, et al. Expression inLactococcus lactis of functional genes related to amino acid catabolism and cheese aroma formation is influenced by branched chain amino acids[J]. International Journal of Food Microbiology, 2012, 159(3): 207-213.
[34] BERTUZZI A S, WALSH A M, SHEEHAN J J, et al. Omics-Based insights into flavor development and microbial succession within surface-eipened cheese[J]. mSystems, 2018, 3(1): e00211-e00217.
[35] VAN MASTRIGT O, GALLEGOS TEJEDA D, KRISTENSEN M N, et al. Aroma formation during cheese ripening is best resembled byLactococcus lactis retentostat cultures[J]. Microbial Cell Factories, 2018, 17(1): 1-8.
[36] JANSSON, JANET K, BAKER E S,et al. A multi-omic future for microbiome studies[J]. Nature Microbiology, 2016, 1(5):16 049.
Research progress on multi-omics techniques applied in flavor metabolism regulation of traditional fermented dairy products
ZHOU Yue1, LI Jian2, ZHANG Yu1, WANG Hongwei1, ZHAO Xin3, LIU Shijian1, SUO Huayi1*
(College of Food Science, Southwest University, Chongqing 400715, China) 2 (College of Life Science and Technology, Southwest Minzu University, Chengdu 610041, China) 3(Chongqing Collaborative Innovation Center for Functional Food, Chongqing University of Education, Chongqing 400067, China)
Abstract Traditional fermented dairy products have unique flavor, nutritional value, and abundant microorganism resources. Due to the complexity of microbial flora during fermentation, the mechanisms of flavor formation in traditional fermented dairy products are unclear and therefore cannot guarantee their safety, which has hindered the large-scale standardized production of traditional fermented dairy products. Based on the development of high-throughput omics technology, multi-omics technology is a favorable method to analyze the flavor formation conditions and microbial metabolic pathways of traditional fermented dairy products. This paper reviewed recent research progress on multi-omics techniques for regulating flavor metabolisms of traditional fermented dairy products. It could provide new ideas for quality control and production research and development of traditional fermented dairy products.
Key words traditional fermented dairy products; multi-omics technology; characteristic flavor compounds; metabolism pathways; functional genes
第一作者:硕士研究生(索化夷副教授为通讯作者,E-mail:birget@swu.edu.cn)。
基金项目:国家重点研发计划项目(2018YFD0502404)
收稿日期:2018-12-18,改回日期:2019-01-16
DOI:10.13995/j.cnki.11-1802/ts.019661


 NE L, VIEL C, BOYLE B, et al. Metatranscriptome analysis of fungal strains
NE L, VIEL C, BOYLE B, et al. Metatranscriptome analysis of fungal strains ACAYUELA T, LP G D C, PEL
ACAYUELA T, LP G D C, PEL EZ C, et al. Expression in
EZ C, et al. Expression in