表1 拼装统计结果
Table 1 The results of assemble

统计类别数目reads数(paired)8 896 966覆盖度(coverage)466xContig数78Contig总长度/bp5 729 055N50 长度/bp326 672Contig平均长度/bp73 449最长的Contig长度/bp683 470
我国是畜产品生产大国,牛骨资源丰富,然而只有少数的蛋白酶可以降解牛骨胶原蛋白,导致长期以来,我国绝大多数牛骨资源尚未得到有效地开发利用[1-2]。研究生物酶工程技术开发出功能性骨蛋白肽,可解决牛骨进行深加工和牛骨资源浪费问题,并具有一定的经济效益,也为我国畜禽骨骼资源的深度开发提供新思路。牛骨骼中的胶原蛋白经酶解后,可以得到骨胶原多肽,它不仅能够抗氧化、抗衰老、促进矿物质吸收等;且在营养吸收方面,比氨基酸或胶原蛋白效果更佳[3-5]。但由于牛骨胶原蛋白溶解性差,以及其特殊的三股螺旋结构,使得牛骨胶原不易被普通的蛋白酶降解。此外,目前能降解骨胶原蛋白的蛋白酶具有底物专一性强,水解程度低的特点,与进入工业化大生产还有一定距离。
目前微生物源牛骨胶原蛋白酶来源有溶组织梭菌[6],Bacillus sp. DPUA 1728[7],Clostridium perfringens[8],Vibrio vulnificus[9],和Vibrio vulnificus CYK279H[10]等,但是这些菌株大部分为致病菌,会与胶原蛋白酶同时产生相应的毒素,不适合大规模生产。因此,目前市售的胶原蛋白酶菌株很少[11-12]。所以,找出安全有效的胶原蛋白酶资源,并提高酶的降解效率成为牛骨胶原蛋白资源深度利用的关键问题。研究人员尝试通过构建工程菌株、基因定向改造、染色体重组等方式来提高原始菌株的产酶效率[13-19]。本研究以课题组筛选的高效降解骨胶原蛋白的菌株B.cereus MBL13-U为出发菌,测定其全基因组序列,然后采用软件对产酶功能基因的二级结构和三维结构进行预测,并将目的基因转入大肠杆菌宿主菌株BL21,构建工程菌。通过此项研究以期为我国畜禽骨骼资源开发利用提供了新的研究思路和途径。
1.1.1 菌种和质粒
蜡样芽孢杆菌(Bacillus cereus,B.cereus) MBL13-U、宿主菌为大肠杆菌(Escherichia coli BL21(DE3))、表达载体pET-30a均为课题组保藏菌株。
1.1.2 PCR引物
根据目的基因和表达载体设计出聚合酶链式反应(polymerase chain reaction, PCR)扩增目的基因的引物。
上游引物序列:CGCGGATCC GCTCTCCCCT ACCTTACATT T(划线处为BamH I的酶切位点)。
下游引物序列:CCGCTCGAG TAAAAAGGTCGCCGTGAGTGG(划线处为Xho I的酶切位点)。
1.1.3 工具酶与主要试剂
ExTaq DNA聚合酶、内切酶(BamHⅠ和XhoⅠ)、T4 DNA连接酶、DNA Marker(DL5 000、DL12 000)、低分子质量蛋白质,Marker TaKaRa公司;卡那霉素(Kana)、异丙基硫代半乳糖苷(IPTG),北京索莱宝公司;DNA快速纯化回收试剂盒、质粒提取试剂盒,北京GENVIEW公司;基因组提取试剂盒,德国Analytik Jena公司;牛血清白蛋白,众一生物公司;Agarose(AR),德国Analytik Jena公司;50 X TAE(AR),吉格生物科技(广州)有限公司;牛跟腱Ⅰ型胶原蛋白,源叶生物有限公司。
1.1.4 主要培养基
B.cereus MBL13-U种子培养基:100 mL去离子水中分别加入牛肉膏1.0 g,蛋白胨1.0 g,NaCl 0.5 g,自然pH。在121 ℃下灭菌20 min,冷却待用。
LB培养基:在100 mL的去离子水中分别加入NaCl 1.0 g,酵母粉0.5 g,胰蛋白胨1.0 g,在121 ℃下灭菌20 min,冷却待用。
LB培养基(含卡那霉素):向100 mL的去离子水中加入NaCl 1.0 g,酵母粉0.5 g,蛋白胨1.0 g,于121 ℃灭菌锅中灭菌20 min,冷却后向其中加入50 mg/mL 卡那霉素100 μL。
固体培养基:加1.5%(质量分数)琼脂于上述培养基中,即成相应固体培养基。
1.1.5 仪器与设备
GeneAmp9700 PCR仪,美国Applied Biosystems公司;DY04-13立式压力灭菌锅,上海东亚压力容器制造有限公司;JY92-IIN超声波细胞破碎仪,宁波新芝科技有限公司;LGJ-25C冷冻离心机,湘仪仪器(湖南)开发有限公司;JY600E SDS电泳仪、WD-9413A凝胶成像仪,美国BIO-RAD公司;H1650离心机,湘仪仪器(湖南)开发有限公司;UV-1800紫外光谱扫描仪,日本岛津公司;TM3030Plus扫描电镜,日本Jeol有限公司。
1.2.1 全基因组测序
提取收集菌株的基因组送至上海源序生物科技有限公司测序。所得Solexa数据采用velvet V1.2.03软件进行拼接。
1.2.2 基因组数据分析
搜寻NCBI的NR库、KEGG蛋白数据库以及SEED蛋白数据库进行基因功能注释,将拼接生成的基因组序列进行开放阅读框(ORF)预测、基因功能注释分析、KEGG代谢途径分析。
1.2.3 胶原蛋白酶的结构预测
在上述研究获得的胶原蛋白酶功能基因序列的前提下,先采用DNA翻译工具ExPASy将基因序列翻译为相对应的氨基酸序列,用SOPMA在线预测该蛋白酶的二级结构,利用NCBI的BLAST工具以及Phyre2在线分析平台等工具对胶原蛋白酶的三维结构进行预测[9]。
1.2.4 ColM13基因克隆
以提取的B.cereus MBL13-U基因组DNA为模板,使用设计出的一对特异性引物进行目的片段的扩增。PCR反应的体系为:10×Buffer 2.5 μL,dNTP 2.0 μL, 上游引物 1.0 μL,下游引物 1.0 μL,模板DNA 1.0 μL,ExTaqDNA DNA聚合酶 0.15 μL,ddH2O 17.5 μL,总体积25 μL。PCR扩增过程为在94 ℃条件下反应4 min进行预变性;在95 ℃条件下反应1 min进行变性;在51 ℃条件下反应1 min进行退火;在72 ℃条件下反应1.5 min进行延伸;在72 ℃条件下反应10 min终止延伸,35个循环。反应结束在4 ℃条件下保存一段时间并抽取PCR产物,用1%(质量分数)琼脂糖凝胶电泳检测。
1.2.5 大肠杆菌BL21感受态的制备
感受态细胞的制备在代军[20]、张迹[21]的基础上略加改善。本试验中采用预冷的已灭菌80 mmol/L MgCl2-20 mmol/L CaCl2溶液对菌体细胞进行重悬,冰上冷却25 min后离心;再采用已灭菌且预冷的0.1 mol/L CaCl2溶液(含30%(体积分数)甘油),轻轻振荡使细胞重悬,然后将菌液以200 μL/管进行分装(冰上操作),-70 ℃保存备用。
1.2.6 重组表达载体pET30a-ColM13及工程菌的构建
在37 ℃条件下用BamHI、XhoI对纯化的PCR产物和表达载体pET-30a进行双酶切,用T4 DNA连接酶对双酶切的产物(酶切产物已进行胶回收纯化)进行连接,完成重组表达载体pET30a-ColM13的构建。在16 ℃下酶连3 h左右,将重组表达载体pET30a-ColM13转化至感受态细胞BL21混匀,完成工程菌pET30a-ColM13/BL21的构建。其中,转化条件[22]为:冰上预冷30 min后在42 ℃条件下水浴热激90 s;再次冰上预冷2~5 min后转入1.5 mL EP管中并加入已灭过菌的LB培养基,以37 ℃、180 r/min恒温培育60 min。无菌条件下,抽取200 μL菌液涂于LB固体平板(含Kana)上,37 ℃恒温过夜培养。用接种环挑取单克隆菌落,接入5 mL LB培养液(含Kana),37 ℃、180 r/min振荡培养过夜。然后将培养物离心,收集菌体并按质粒提取试剂盒的说明书(GENVIEW公司)从工程菌中提取重组质粒,对重组质粒进行酶切鉴定及序列测定。
1.2.7 重组蛋白酶ColM13的诱导表达
为检验构建出的工程菌是否能够表达所需要的重组蛋白酶ColM13。诱导表达时采用IPTG诱导法,主要是在处于对数生长期的工程菌培养液中加入0.1%(体积分数)为100 mmol/mL的IPTG,在37 ℃的恒温振荡器以180 r/min的条件培养4 h,然后离心收集菌体进行超声破碎,再次离心,上清即为重组蛋白酶。最后进行SDS-PAGE电泳检测[23-24]。
1.2.8 重组蛋白酶ColM13 酶解骨胶原蛋白的结构分析
1.2.8.1 紫外(UV)扫描分析
将骨胶原蛋白分别用BSC和ColM13进行处理,在近紫外区对处理过的样品进行200~400 nm的全波长扫描。
1.2.8.2 扫描电镜(SEM)分析
分别将胶原蛋白原样与用ColM13处理的胶原蛋白经真空冷冻干燥后进行喷金处理后用扫描电镜观察表面结构的变化。
2.1.1 Solexa数据拼装
采用软件velvet V1.2.03对Solexa数据进行拼装,拼装后共得到78个重叠群(Contig),Contig的平均长度为73 449 bp,总长为5.7 Mb,其中最长的Contig为683 kb。拼装结果详见表1。
表1 拼装统计结果
Table 1 The results of assemble

统计类别数目reads数(paired)8 896 966覆盖度(coverage)466xContig数78Contig总长度/bp5 729 055N50 长度/bp326 672Contig平均长度/bp73 449最长的Contig长度/bp683 470
2.1.2 基因预测
通过glimmer 3.02 对出发菌株的基因组进行基因预测,结果如表2。
表2 基因预测的结果
Table 2 The results of gene prediction

统计类别数目基因数5 989编码区大小/bp4 774 974编码区GC含量36.0%基因平均长度/bp797最长基因/bp15 054最短基因/bp114
可知其含5 989个基因;通过tRNA-scan发现84个tRNA序列;使用RNA mmer分析软件从菌株的基因组中预测出6个rRNA序列。图1显示基因长度的分布情况。
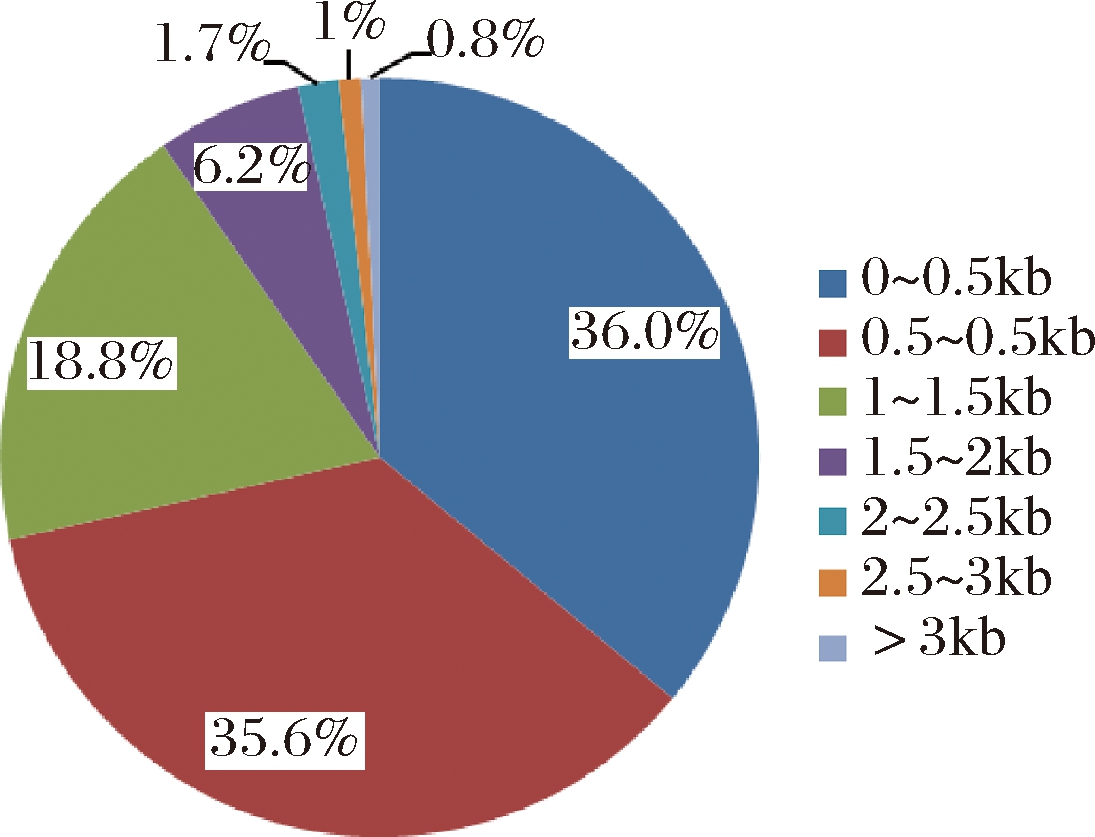
图1 基因长度分布
Fig.1 The fraction of gene length
由图1可知,基因长度在0.5 kb以下的为36.0%,基因长度在0.5~1 kb以下的为35.6%,基因长度在1~1.5 kb以下的为18.8%。综上可知,90%以上的基因长度都在1.5 kb以下。
搜寻NCBI的NR库、KEGG蛋白数据库以及SEED蛋白数据库对所测胶原蛋白酶进行基因功能注释,利用CDD数据库进行COG分类。通过KEGG数据库构建代谢通路。分析可知共有5 831个蛋白具有生物学功能。3 715个蛋白具有COG分类,结果见图2;1 610个蛋白具有KEGG的同源基因(ortholog),结果见图3;所有蛋白匹配的同源蛋白来源于341个物种,分类情况如图4所示。
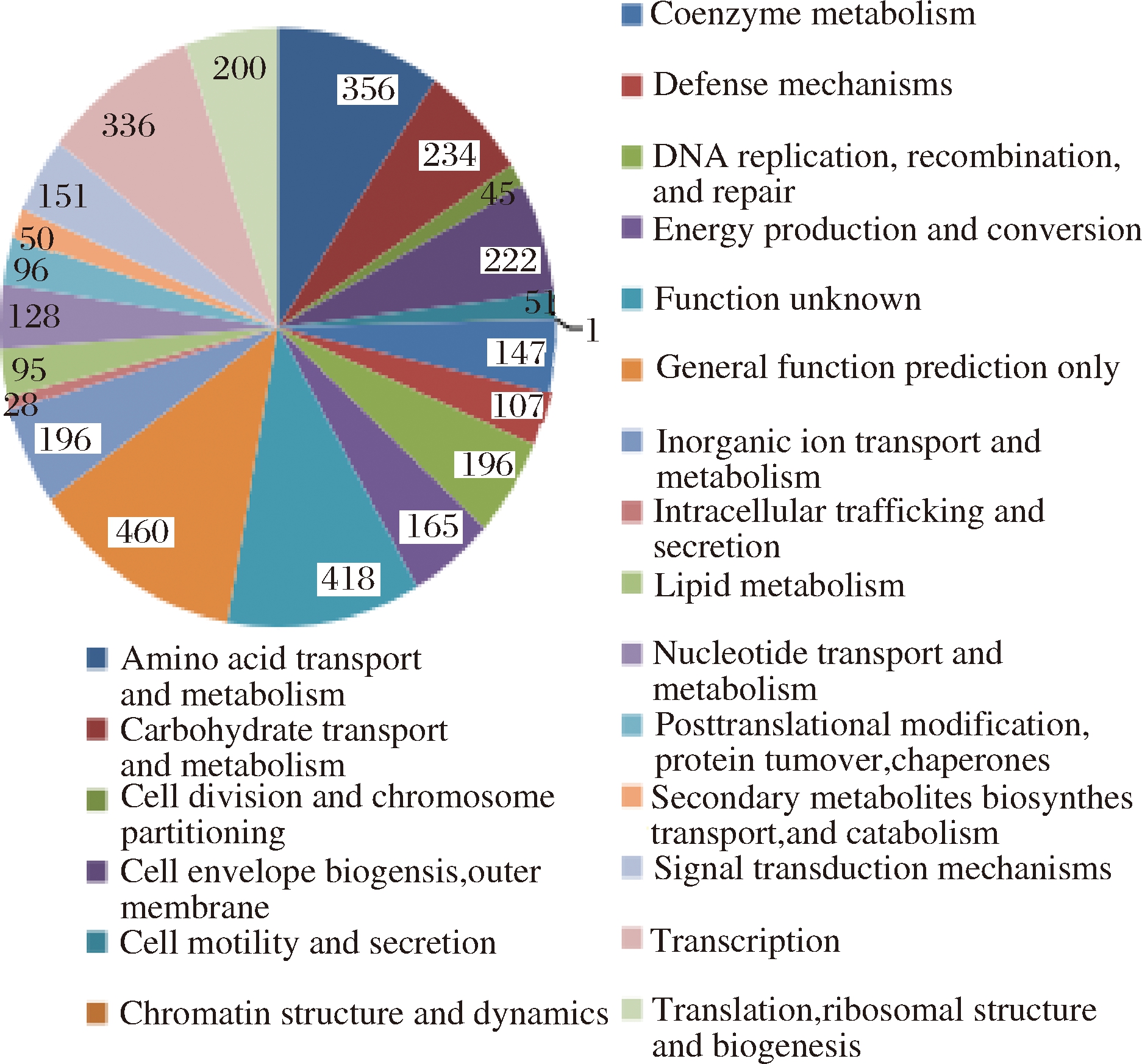
图2 COG功能统计
Fig. 2 The function statistics of COG

图3 KEGG代谢通路的类型
Fig. 3 The type of KEGG metabolic pathway
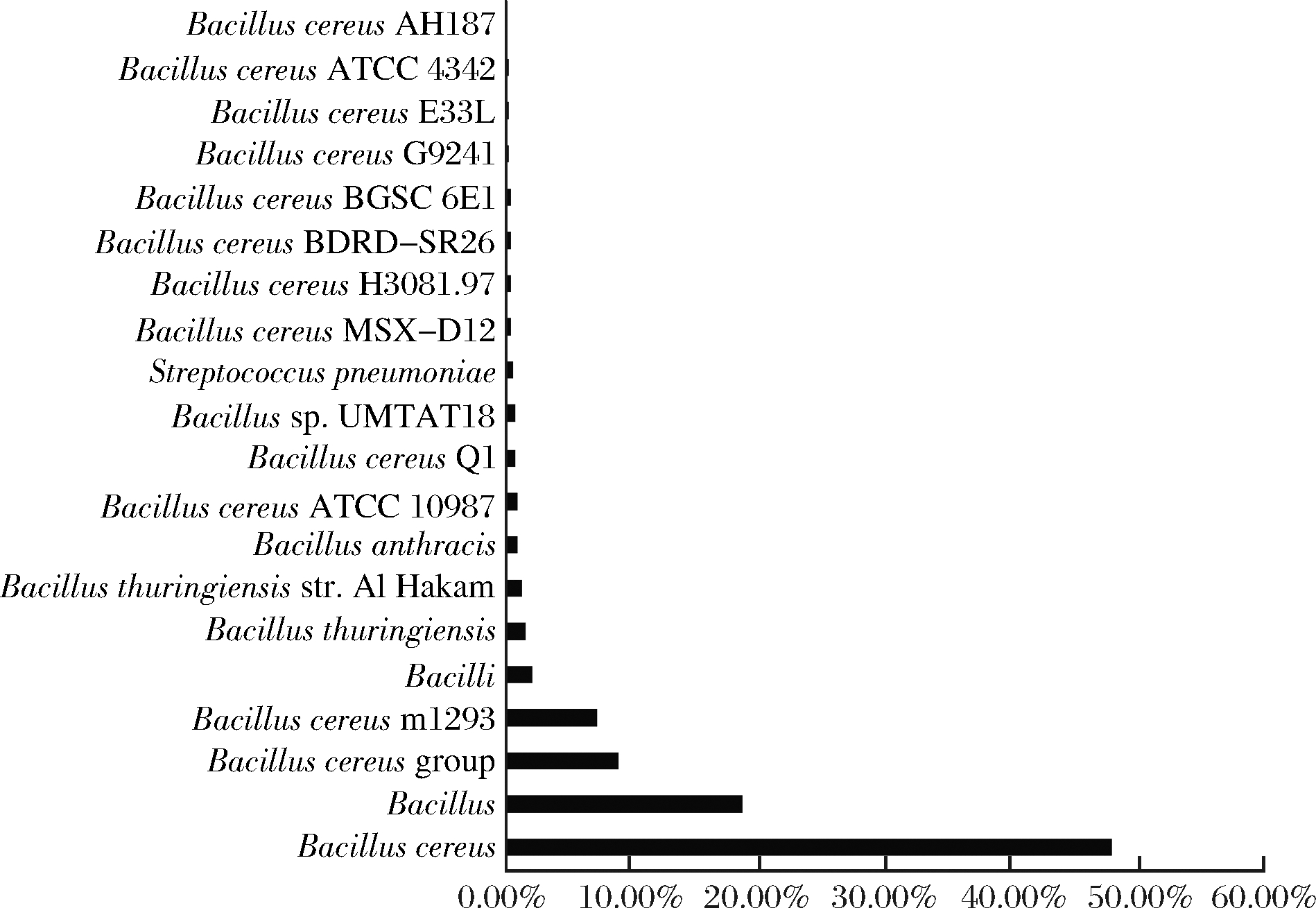
图4 同源蛋白的类型
Fig. 4 The type of homologous protein
由图2可知,COG功能主要包括:氨基酸、碳水化合物的转运与代谢;细胞与染色体的分裂等。由图3可知,KEGG代谢通路的类型主要分为四大类:(1)代谢;(2)遗传信息处理;(3)环境信息处理;(4)细胞过程。
其中,代谢主要为各种物质及能量的代谢;遗传信息处理主要指的是:转录,翻译,折叠、排序和退化,复制和修复;环境信息处理包括:膜运输、信号转导、信号分子和交互;细胞过程指的是细胞运输和分解代谢、细胞活性、细胞生长和死亡、细胞群体。由图4可知,所有蛋白匹配的同源蛋白中Bacillus cereus比例最高,达到47.9%。
经过上述基因组数据分析,从全基因组序列中得到胶原蛋白酶的功能基因序列长度为2 916 bp。序列号为CP023179.1。
根据获得的目的基因序列,通过SOPMA在线预测该蛋白的二级结构,结果表明该功能基因中α螺旋占总氨基酸的37.1%,β转角占总氨基酸的12.8%,无规则卷曲占总氨基酸的26.0%。利用NCBI的BLAST工具以及Phyre2在线分析平台等工具对胶原蛋白酶的三维结构进行预测,结果如图5所示。

A-模块结构;B-三级结构
图5 胶原蛋白酶的模块结构和胶原蛋白酶的三级结构预测
Fig.5 Module structure of collagenase and prediction of the tertiary structure of collagenase
由图5-A可知,NCBI的BLASTp分析结果表明胶原酶含有的4个保守的结构域:氨基酸残基序列中97~279位是M9肽酶家族N端结构域(peptidase family M9N-terminal),该结构域最初从微生物来源的胶原酶-金属蛋白酶中发现;氨基酸残基序列中从359~621位是谷氨酸-锌肽酶家族结构域(collagenase),这个酶家族主要用于分解胶原蛋白;氨基酸残基序列中的774~843位是多囊性肾病蛋白结构域(PKD),PKD结构域可能是蛋白质-蛋白质或蛋白质-碳水化合物相互作用中的配体,在结合位点处起作用;氨基酸残基序列891~956位是细菌前肽酶C末端结构域(bacterial pre-peptidaseC-terminal domain),该结构域通常在分泌的细菌肽酶的C端发现,它们可能与PKD(pfam00801)结构域所发挥的作用相类似。
将所得的胶原蛋白酶基因序列在Phyre2在线分析平台模拟其三维结构,由图5-B可知,该蛋白酶的三维结构呈现出镊子状结构。
由图6可知,在2号泳道均出现目的条带,大小约2 916 bp与预期的ColM13基因片段大小相符。采用DNA回收试剂盒快速纯化回收PCR扩增的产物,抽取样品送至北京六合华大公司进行测序,结果表明PCR结果正确。

M-DL 5 000 Marker;1-扩增的目的片段
图6 PCR扩增产物检测
Fig.6 PCR amplification product detection
由图7-A可知,泳道2和3种酶切重组子后并未出现两条条带;泳道4出现2条条带,说明挑取的3个工程菌的菌落中泳道4的菌落可能重组成功。因此就泳道4提取的工程菌的质粒增加对照类型,电泳验证结果如图7-B所示。结合图7-B可知,泳道7双酶切后的工程菌的质粒出现两条条带,其中上方条带为酶切后的质粒条带,下方条带为酶切后目的基因条带,并且这两条条带与pET-30a(+)空载体和PCR扩增出ColM13的基因片段大小基本一致。由此得出,工程菌pET-30a-ColM13/BL21(DE3)已成功构建。
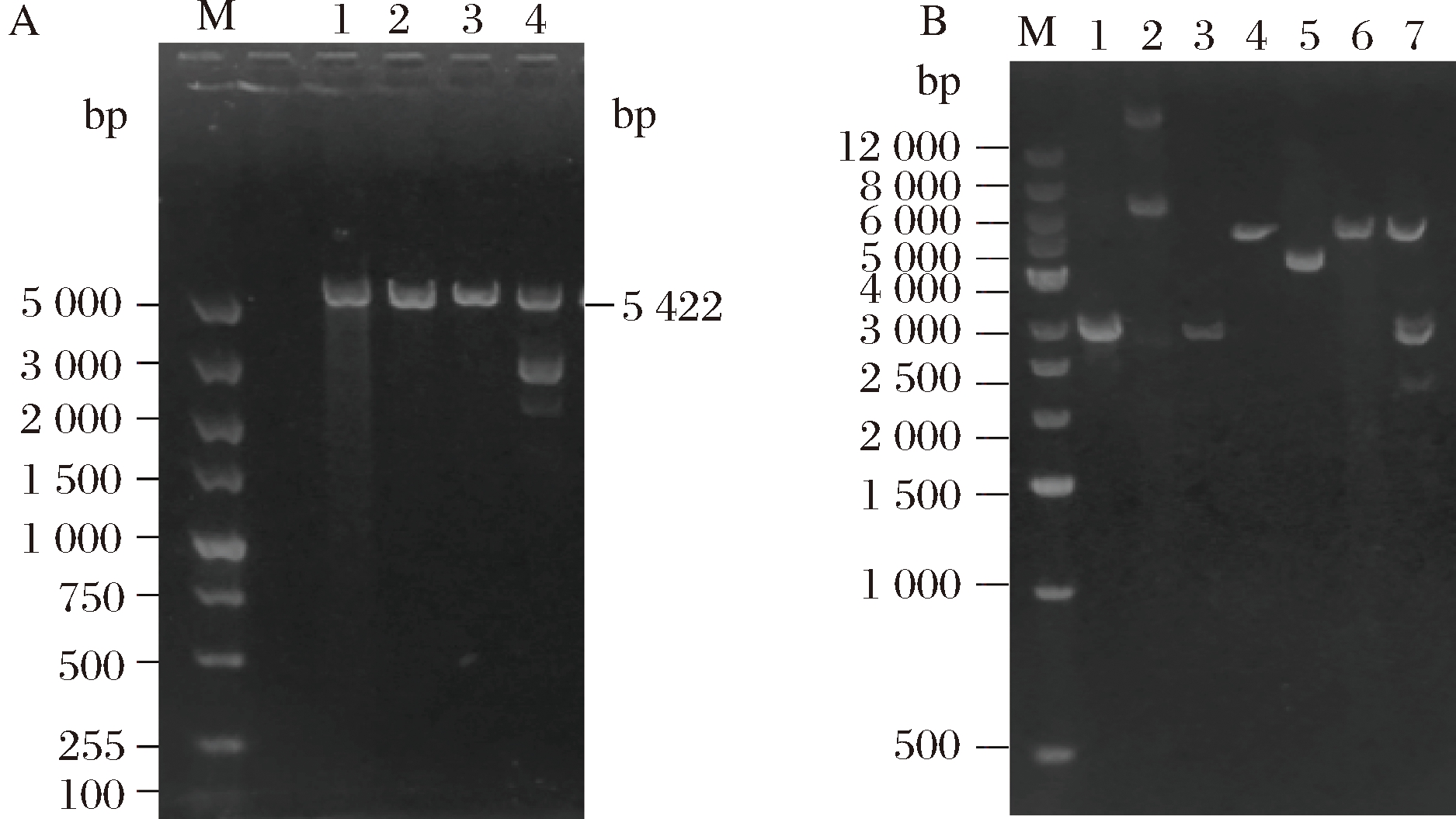
A-M:DL 5 000 Marker;1:单酶切后pET-30a(+)质粒载体;
2-4:双酶切后重组质粒;B-M:DL 12000 Marker;泳道1:原目
的片段;泳道2:pET-30a(+)空质粒;泳道3:双酶切后目的片段;
泳道4:双酶pET-30a(+)质粒;泳道5:重组质粒;泳道6:单
酶切后pET-30a(+)质粒;泳道7:双酶切后重组质粒
图7 重组子酶切验证
Fig. 7 The enzyme digestion verification of the recombinant
经过对ColM13诱导表达的优化,将工程菌pET30a-ColM13/BL21和空质粒菌pET30a/BL21在37 ℃的条件下加入为6‰的IPTG进行诱导,诱导时间为6 h。ColM13表达情况如图8所示。
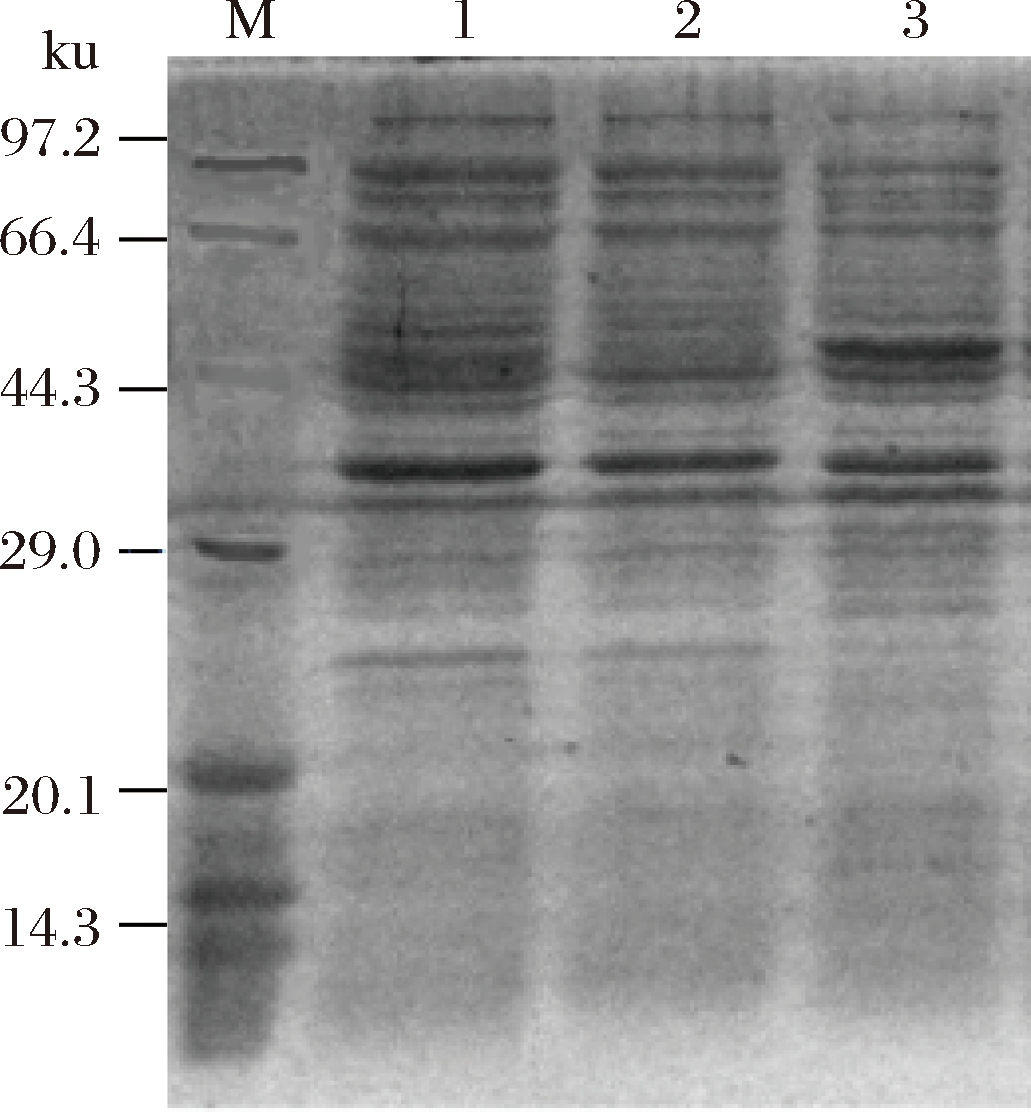
M-蛋白质分子质量Marker;1-pET-30a(+)质粒转入后
经IPTG诱导;2-重组子转入后未经IPTG诱导;
3-重组子转入后经IPTG诱导
图8 SDS-PAGE检测
Fig. 8 SDS-PAGE detect
由图8可知,与未经IPTG诱导的工程菌相比,加入IPTG诱导后构建的工程菌中的目的蛋白质得到表达。诱导表达的蛋白与Marker进行对比,其分子质量约为46 kDa。
胶原蛋白肽链中的-C=O、-COOH、-CONH2为生色基团,蛋白质溶液形成紫外吸收光谱即是蛋白分子中各种紫外生色基团加和的结果[25]。本实验的紫外图谱检测结果如图9所示。
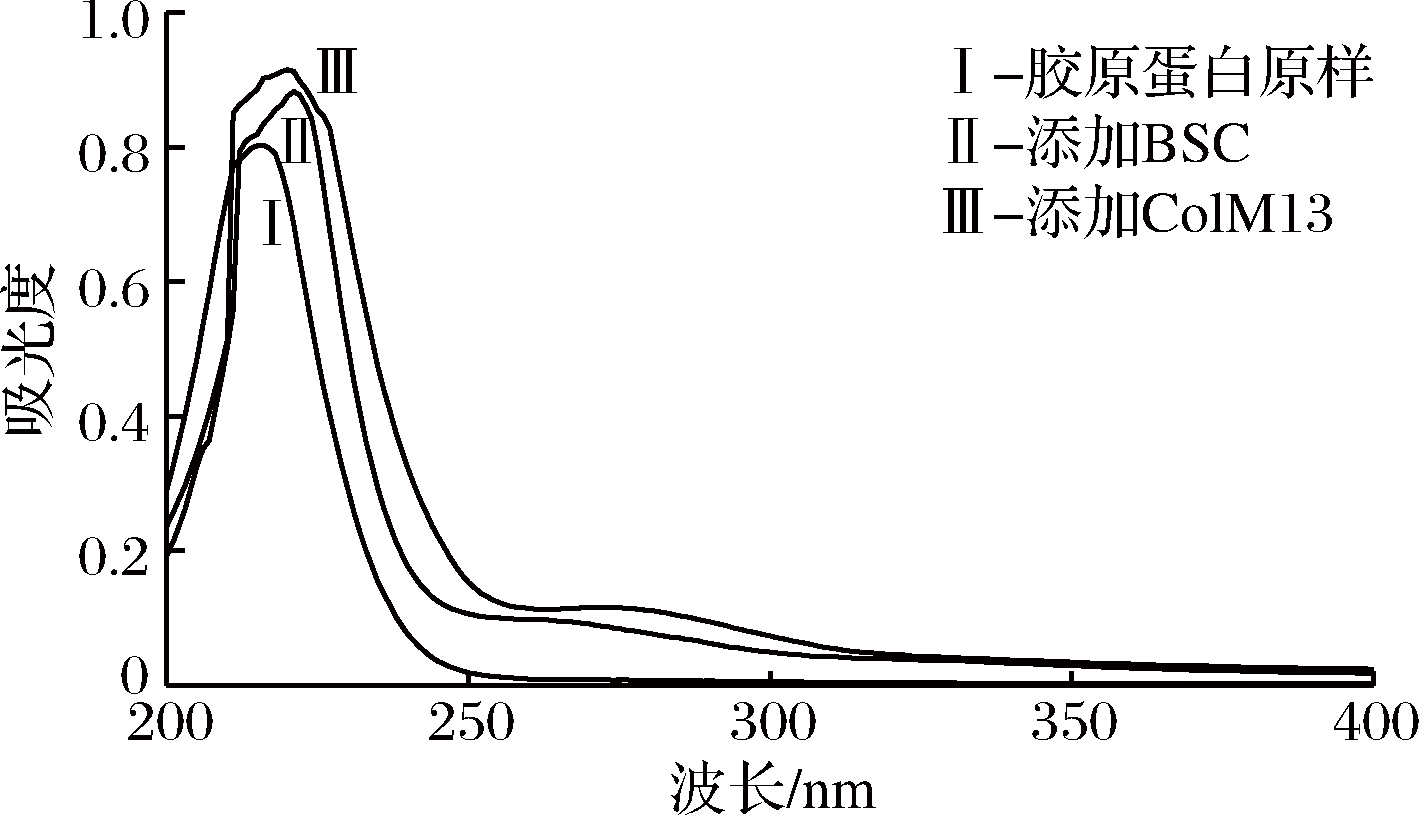
图9 胶原蛋白酶解前后的紫外光谱分析
Fig.9 Analysis of UV spectrum before and after enzymatic hydrolysis of collagen
胶原蛋白的近紫外吸收光谱可用于测量它的酪氨酸含量和非螺旋端肽的完整性。LIN等[26]提出哺乳动物或鱼类的I型胶原蛋白的特征吸收光谱在218 nm处,主要是因为肽键C![]() O的n→π*跃迁所致。由图9可知,Ⅰ、Ⅱ、Ⅲ三者均在220~230 nm出现特征吸收,经ColM13处理的胶原蛋白峰值比经BSC处理的偏高,说明ColM13较BSC有更好的降解效果。酶处理过的溶液紫外吸收峰右移,表明反应过程中胶原蛋白肽中生色基团分布发生了变化,导致峰值逐渐升高。此外,ColM13处理过的溶液在280 nm处有微弱吸收,这可能与胶原蛋白中酪氨酸(278 nm),苯丙氨酸(259 nm)的存在相关,这些吸收峰的存在表明胶原蛋白非螺旋端肽具有完整性,这与张晓洁等[27]的研究结果相一致。
O的n→π*跃迁所致。由图9可知,Ⅰ、Ⅱ、Ⅲ三者均在220~230 nm出现特征吸收,经ColM13处理的胶原蛋白峰值比经BSC处理的偏高,说明ColM13较BSC有更好的降解效果。酶处理过的溶液紫外吸收峰右移,表明反应过程中胶原蛋白肽中生色基团分布发生了变化,导致峰值逐渐升高。此外,ColM13处理过的溶液在280 nm处有微弱吸收,这可能与胶原蛋白中酪氨酸(278 nm),苯丙氨酸(259 nm)的存在相关,这些吸收峰的存在表明胶原蛋白非螺旋端肽具有完整性,这与张晓洁等[27]的研究结果相一致。
将表达的ColM13作用于胶原蛋白,对酶解前后的胶原蛋白进行扫描电镜分析,结果如图10所示。

A-胶原蛋白原样;B-ColM13处理的胶原蛋白
图10 胶原蛋白酶解前后的扫描电镜图
Fig.10 The SEM of collagen before and after enzymatic hydrolysis
由图10可知,将诱导表达出的ColM13酶作用于胶原蛋白后,胶原蛋白的结构发生明显变化。其中,胶原蛋白呈现为颗粒状结构,表面凹凸不平,但颗粒的整体性较好;但经ColM13处理后胶原蛋白的结构由颗粒状转变成层状或者片段分列,表面均匀、细腻光滑,这是由于ColM13将胶原蛋白降解为小分子多肽,破坏了其分子表面结构[28],从而表明ColM13在工程菌中已成功表达。
(1)本研究将课题组筛选的B.cereus MBL13-U作为目标菌株,根据其全基因组测序分析获得产酶的功能基因序列,采用velvet V1.2.03对数据进行拼装,搜寻NCBI的NR库、KEGG蛋白数据库以及SEED蛋白数据库进行基因功能注释,确定胶原蛋白酶的基因序列长度为2 916 bp。
(2)采用SOPMA在线预测可知该蛋白的二级结构,结果表明该功能基因中α螺旋占总氨基酸的37.1%, β转角占总氨基酸的12.8%,无规则卷曲占总氨基酸的26.0%。利用NCBI的BLAST工具以及Phyre2在线分析平台等工具对胶原蛋白酶的三维结构进行预测,表明该胶原蛋白酶呈现为镊子状三维结构。
(3)以B.cereus MBL13-U基因组DNA为模板进行PCR扩增,将目的片段克隆至质粒pET-30a,成功构建出表达型工程菌大肠杆菌(E.coli BL21(DE3)),通过全波长紫外扫描光谱和扫描电镜对工程菌所产的工程蛋白酶ColM13酶解Ⅰ型骨胶原蛋白后的结构进行分析。表明工程菌构建成功,这对骨胶原蛋白酶结构特性的相关研究及其在生产领域的应用具有重要意义。
[1] 安广杰,胡加松,王章存,等. 猪骨蛋白酶解制取多肽钙的研究综述[J]. 郑州轻工业学院学报(自然科学版), 2013,28(6):27-31.
[2] 刘丽莉, 马美湖,余秀芳,等. 胶原蛋白酶产生菌的筛选及酶的分离纯化[J]. 生物工程学报, 2010, 26(2):194-200.
[3] GELSE K, PÖSCHL E, AIGNER T. Collagens-structure, function, and biosynthesis[J]. Advanced Drug Delivery Reviews, 2003, 55(12):1 531-1 546.
[4] HORN M M, MARTINS V C A, PLEPIS A M D G. Interaction of anionic collagen with chitosan: Effect on thermal and morphologi cal characteristics[J]. Carbohydrate Polymers, 2009, 77(2):239-243.
[5] 柳林, 韦术敏,程仕伟,等. 产胶原酶的琥珀葡萄球菌分离鉴定及其培养优化研究[J]. 中国酿造, 2016, 35(1):62-67.
[6] BICSAK T A, HARPER E. Purification of nonspecific protease-free collagenase fromClostridium histolyticum[J]. Analytical Biochemistry, 1985, 145(2):286-291.
[7] LIMA L A, CRUZ R F, SANTOS J G D, et al. Protease with collagenolytic activity produced byBacillus sp. DPUA 1728 from Amazonian soil[J]. Brazilian Journal of Microbiology, 2015, 46(4):1 217-1 223.
[8] ABFALTER C M, SCHÖNAUER E, PONNURAJ K, et al. Cloning, purification and characterization of the collagenaseColA expressed byBacillus cereus ATCC 14579[J]. Plos One, 2016, 11(9):e0162 433.
[9] WEI Shuang, ZHAO Hui, XIAN Yuyin, et al. Multiplex PCR assays for the detection ofVibrio alginolyticus,Vibrio parahaemolyticus,Vibrio vulnificus, andVibrio cholerae with an internal amplification control[J]. Diagnostic Microbiology & Infectious Disease, 2014, 79(2):115-118.
[10] KANG S I, JANG Y B, KONG C J Y. Purification and properties of a collagenolytic protease produced byMarine Bacterium Vibrio vulnificus CYK279H[J]. Biotechnology and Bioprocess Engineering, 2005, 10(6):593-598.
[11] ZHANG X X, LI Y, WANG S Y, et al. Identification of a collagenase produced byBacillus cereus R75E isolated from human colostrum[J]. Applied Biochemistry and Microbiology, 2015, 51(5):511-521.
[12] 杨光垚. 胶原蛋白酶的纯化及其基因的克隆与表达研究[D]. 成都:四川大学, 2004.
[13] ROCHIMA E, SEKAR N, BUWONO I D, et al. Isolation and characterization of collagenase fromBacillus Subtilis (Ehrenberg, 1835); ATCC 6633 for degrading fish skin collagen waste from cirata reservoir, indonesia[J]. Aquatic Procedia, 2016, 7:76-84.
[14] SUN Qian, CHEN Fusheng, GENG Fang, et al. A novel aspartic protease fromRhizomucor miehei expressed inPichia pastoris and its application on meat tenderization and preparation of turtle peptides[J]. Food Chemistry, 2018, 245:570-577.
[15] TURSUNOV K, BEGALIYEVA A, INGIRBAY B, et al. Cloning and expression of fragment of the rabiesvirus nucleoprotein gene inEscherichia coli and evaluation of antigenicity of the expression product[J]. Iranian Journal of Veterinary Research, 2017, 18(1):36-42.
[16] TERAMURA N, TANAKA K, IIJIMA K, et al. Cloning of a novel collagenase gene from the gram-negative bacteriumGrimontia (Vibrio)hollisae 1706B and its efficient expression inBrevibacillus choshinensis[J]. Journal of Bacteriology, 2011, 193(12):3 049-3 056.
[17] KATO T, TAKAHASHI N, KURAMITSU H K. Sequence analysis and characterization of thePorphyromonas gingivalis prtC gene, which expresses a novel collagenase activity[J]. Journal of Bacteriology, 1992, 174(12):3 889-3 895.
[18] LEE J M, LEE J, NAM G H, et al. Heterologous expression and enzymatic characterization of γ-glutamyltranspeptidase fromBacillus amyloliquefaciens[J]. Journal of Microbiology, 2017, 55(2):147-152.
[19] TSURUOKA N, NAKAYAMA T, ASHIDA M, et al. Collagenolytic serine-carboxyl proteinase fromAlicyclobacillus sendaiensis Strain NTAP-1: purification, characterization, gene cloning, and heterologous expression[J].Applied and Environmental Microbiology 2003, 69(1):162-169.
[20] 代军. 大肠杆菌感受态细胞制备及转化条件优化[J]. 江苏农业科学, 2015, 43(4):53-54.
[21] 张迹, 李智,张宇,等. 一步法制备大肠杆菌BL21(DE3)菌株感受态细胞及转化条件优化[J]. 江苏农业科学, 2016, 44(12):529-532.
[22] KIM H D, KIM S M, CHOI J I. Purification, characterization, and cloning of a cold-adapted protease fromAntarctic Janthinobacterium lividum[J]. Journal of Microbiology & Biotechnology, 2018, 28(3):448-453.
[23] TURSUNOV K, BEGALIYEVA A, INGIRBAY B, et al. Cloning and expression of fragment of the rabies virus nucleoprotein gene inEscherichia coli and evaluation of antigenicity of the expression product[J]. Iranian Journal of Veterinary Research, 2017, 18(1):36-42.
[24] AOKI E, FUJIWARA K, SHIMIZU A, et al. Optimization ofHaemophilus influenzae adhesin transmembrane domain expression inEscherichia coli.[J]. Protein Expression & Purification, 2018, 145:19-24.
[25] 李飞, 周帅,董慧,等. 金黄色葡萄球菌肠毒素B基因在大肠杆菌中的克隆及表达[J]. 检验医学与临床, 2016, 13(18):2 553-2 555.
[26] LIN Y K, DENG C L. Comparison of physical-chemical properties of type I collagen from different species[J]. Food Chemistry, 2006, 99(2):244-251.
[27] 张晓洁, 张宇昊,马良,等. 超声辅助提取兔皮胶原蛋白及其理化特性[J]. 食品与机械, 2017, 33(1):167-171.
[28] BARATHIRAJA S, PAV G, UMAPATHI V, et al. Expression and purification of biologically active bovine Interferon λ3 (IL28B) inPichia pastoris[J]. Protein Expression & Purification, 2018, 145:14-18.